2. 甘肃省黄河水环境重点实验室, 兰州 730070;
3. 甘肃农业大学资源与环境学院, 兰州 730070;
4. 烟台大学环境与材料工程学院, 烟台 264005
2. Key Laboratory of Yellow River Environment in Gansu Province, Lanzhou 730070, China;
3. College of Natural Resources and Environment, Gansu Agricultural University, Lanzhou 730070, China;
4. School of Environmental and Material Engineering, Yantai University, Yantai 264005, China
人类利用雨水的历史已有近千年, 发达国家在雨水资源的回收利用方面积累了相当的经验和案例, 如以色列的“花园计划”, 德国1989年制定的《雨水利用工程设施标准》等.中国的西北地区由于水资源匮乏, 水窖经常被用来收集和贮存雨水, 用于日常人畜饮用水.相较于河流和湖泊等开放式水体, 窖水的贮存环境较为封闭, 集雨面不断经历集流雨水的冲刷, 致使大量的污染物随雨水径流进入水窖并富集, 进而通过物理和生物作用在水-沉积物两相之间交换迁移[1].沉积物作为窖水生态系统中的重要组成部分, 不仅承载上覆水, 吸纳部分污染物, 更为微生物提供了一个繁衍生息的稳定环境[2, 3].
真菌是一种种类繁多、分布广泛的真核微生物, 可作为特定生态系统的有效标志物, 在极端环境下具有较强的生长繁殖能力, 其胞外酶具有极强的分解能力, 能降解大部分有机物[4~6], 在窖水生态系统中与其它微生物一同推动能量流动与物质转换[7].此外真菌作为生态系统中的分解者还具有生物防治、重金属吸附和富集、农药降解等功能.常敏等[8]发现Aspergillus awamori的代谢产物大黄素对金黄色葡萄球菌和枯草芽孢杆菌具有明显抑制作用, Sarkar等[9]发现哈茨木霉菌丝体对Cr6+具有显著移除作用, 田连生等[10]发现木霉菌株对多菌灵、速克灵和三唑酮这3种化学农药具有极高的降解率.在研究方法上, 高通量测序具有通量高, 深度大, 准确度高的优势, 可为生态、资源和环境等领域的深度研究提供极大的便利和技术支持[11~14].譬如Panzer等[15]总结了基于18S rRNA的环境真菌群落研究, 发现Ascomycota、Basidiomycota和Chytridiomycota是淡水真菌群落的主要组成; 商潘路等[16]发现金盆水库水体真菌主要为Chytridiomycota和未知真菌.赵彦玲等[17]发现湖泊沉积物中不同深度真菌群落结构虽不同, 但主要菌门依然为Ascomycota和Basidiomycota.
然而诸如以上基于高通量测序卓有成效和值得借鉴的研究, 更多聚焦于大区域的开放式生态系统, 对窖水这种封闭式水体的研究鲜有报道.解析窖水沉积物中真菌多样性与群落结构, 将有助于明晰窖水中物质循环及能量转化的基本过程, 对揭示窖水水质变化机制、水体微生物病原体污染状况和改善集雨环境等具有重要意义.
1 材料与方法 1.1 样本采集与处理本研究根据我国干旱半干旱典型集雨地区集雨现状, 针对不同集流面(黄土地面、混凝土地面)和水窖类型(水泥窖)的组合, 使用彼得森采泥器, 采集了位于甘肃省会宁县6户农户庭院中的6口水窖中的沉积物.采样深度5 cm, 以每个采样点为中心采集3个平行样, 现场混为一个待测样品并编号, CSC1、CSC2和CSC3号样品取自黄土地集流面+混凝土水窖, CSR1、CSR2和CSR3号样品取自混凝土集流面+混凝土水窖.样品置于冰盒带回实验室, 在冰箱快速冷冻(-80℃)保存用于后续微生物DNA提取.
1.2 真菌基因组DNA的提取与测序水窖沉积物样品的真菌基因组DNA采用试剂盒(OMEGA Soil DNA Kit)提取, 待微生物DNA溶解完全后, 采用美国Quawell公司的Q5000型紫外可见分光光度计, 依据D260/D280值评估其纯度, 使用电泳仪进行琼脂糖电泳(0.8%)检测, 并通过凝胶成像系统拍照、观察和分析样品微生物DNA的完整性.采用通用引物序列(上游引物: GGAAGTAAAA GTCGTAACAAGG, 下游引物: GCTGCGTTCTTCATCG ATGC)[18], 以提取到的6个DNA原液为PCR模板, 进行ITS1区扩增.待反应完全结束后, 使用电泳仪(北京六一生物科技有限公司DYY-6C型)进行琼脂糖电泳(2.0%)检测PCR产物.经检测, PCR扩增图谱条带清晰明显, 且条带位置一致, 背景干净, 可用于后续分析.高通量测序(Illumina MiSeq)由上海派森诺生物科技有限公司完成.
1.3 生物信息学分析使用Qiime对原始数据进行分析, 包括双末端序列装配、去除低质量序列和去重复, 运用Mothur检查并剔除嵌合体序列, 并统计每个样本的优质序列; 将有效序列按照97%的序列相似度作为OTU划分阈值[19, 20], 对获得的每个OTU代表序列进行分类学信息注释, 剔除丰度值低于全体样本测序总量0.001%的稀有OTU[21], 得到精简后的OTU列表.根据各样品OTU丰度情况, 应用软件Mothur中的summary和single命令, 计算种群丰富度指数和多样性指数, 使用Origin2018绘制群落分类学组成和丰度图, 使用Galaxy在线平台进行LEfSe分析不同分类水平的组间差异, 使用Gephi软件构建微生物群落-环境因子网络图, 采用Canon5.0软件绘制RDA分析.
2 结果与讨论 2.1 水窖沉积物理化性质水窖沉积物的物理化学性质分析结果如表 1所示.6个样品的物理化学性质有明显差异, 其中样品CSR2中有机质与NH4+-N含量最高, 样品CSC1和样品CSC3中NO3--N含量最高, NO2--N和TP分别在样品CSC2和样品CSC1中含量最高, 样品CSR1中TN含量最高.总体而言, 水窖沉积物中有机质、氮和磷等营养盐含量丰富.
|
|
表 1 水窖沉积物物理化学指标 Table 1 Physicochemical parameters of sediment in cellar water |
2.2 沉积物微生物群落多样性分析
6个沉积物样品共得到可用于后续分析的优质序列313 142条, 占有效序列的98.37%.优质序列长度多分布在200~300 bp, 以256 bp的序列居多.其中样品CSR2(58 631条)最多, CSR3(45 242条)最少, 其余样品高质量序列数量差异较小, 结果见表 2.
|
|
表 2 窖水沉积物真菌16S rRNA序列、丰富度和多样性 Table 2 High-throughput sequencing data, biodiversity, and diversity of fungi in cellar sediment |
在群落生态学中, α多样性可以解释微生物群落中的物种丰富度和多样性.使用Chao1指数和ACE指数用来表征物种的丰富度[22, 23], Shannon指数和Simpson指数用来表征物种的多样性及均匀性[24, 25].Shannon指数对群落丰富度以及稀有OTU更敏感, 而Simpson指数对均匀度和群落中的优势OTU更敏感; 一般而言, Chao1或ACE指数越大, 表明群落的丰富度越高.Shannon指数或Simpson指数越大, 表明群落多样性和均匀性越好.
表 2中为取自6个水窖沉积物样本的真菌群落丰富度和多样性指数, Chao1指数均值分别为318.66(CSC1~CSC3)和328.5(CSR1~CSR3), ACE指数均值分别为324.06(CSC1~CSC3)和308.37(CSR1~CSR3), 其中ACE指数最大值(CSC2)与最小值(CSC3)均来自于黄土地集流面, 在Chao1指数与ACE指数中, 样品CSC1与CSR3相差最小, 说明样品CSC1与样品CSR3的微生物总数差异较小, 样品CSC2的Chao1指数和ACE指数最大, 说明样品CSC2中所含物种总数最多、物种丰富度最大, 反之样品CSC3的Chao1指数和ACE指数最小, 表明CSC3所含物种总数最少, 且丰富度最小; 除CSC3采样点卫生条件较差外, 其余5处采样点卫生条件均较好, 因此在同一集流面下, 不同的环境和卫生条件也会最终影响集雨窖水中沉积物的真菌群落丰富度.Shannon指数平均值分别为5.336(CSC1~CSC3)和5.448(CSR1~CSR3), Simpson指数平均值分别为0.887(CSC1~CSC3)和0.924(CSR1~CSR3), 混凝土集流面的Shannon指数均值和Simpson指数均值都大于黄土地集流面; 样品CSR2和CSR3的Shannon指数相差较小且高于大部分样品, 说明CSR2和CSR3的真菌多样性较高.同样, 样品CSC3的Shannon指数和Simpson指数在6个样品中依然为最小, 说明样品CSC3的真菌群落多样性最差, 考虑CSC3采样点的卫生条件, 表明良好的集流面环境和卫生条件使得真菌群落丰富度和多样性更高, 水体也更适合真菌生长繁殖[26].
2.3 沉积物真菌群落结构特征 2.3.1 沉积物真菌群落门水平结构特征不同采样点的沉积物真菌群落在门水平上组成差异见图 1所示, Ascomycota为绝对优势菌门, 除CSC2丰度略低, 其余样品Ascomycota丰度在78% ~88%之间, 在Ascomycota中(图 2), 黄土地集流面的优势纲(平均丰度大于1%)为Sordariomycetes(23.74%), Saccharomycetes(13.26%), Eurotiomycetes(9.41%), Dothideomycetes(5.29%), Archaeorhizomycetes(1.03%)和无法分类的Ascomycota(19.08%), 混凝土集流面的优势菌纲为Sordariomycetes(22.04%), Saccharomycetes(16.86%), Eurotiomycetes(33.57%), Dothideomycetes(9.57%), Archaeorhizomycetes(7.41%)和无法分类的Ascomycota(5.37%), 两种集流面条件下的Ascomycota真菌群落组成结构基本保持一致.丰度第二的为Basidiomycota, 6个样本的平均丰度为10.7%; 样品CSC2中Basidiomycota的丰度达到了27.3%, 是其余样品的2~5倍; 丰度第三的为Zygomycota, 在CSC和CSR中的平均丰度分别为4%和2%, 在黄土地集流面条件下Zygomycota的丰度普遍大于混凝土集流面.除此之外, 样品中还存在少量原生动物Cercozoa和Ciliophora, 它们直接参与真菌和其他微生物的群落调节过程并在能量转化和生态系统稳定性方面发挥重要作用[27, 28].淡水真菌被认为是陆地真菌以孢子的形式进入水环境, 并在水中繁殖扩散.在窖水生境中, 从地表集流面至水窖内部之间物质交换频率可能伴随降水事件而发生周期波动, 因此很难精确地确定某一真菌的真正环境来源, 文献[29, 30]发现水生真菌以Ascomycota和Chytridiomycota为主, 陆生真菌以Ascomycota和Basidiomycota为主, 本次研究窖水沉积物中Basidiomycota丰度远高于Chytridiomycota, Ascomycota、Basidiomycota和Zygomycota共同构成了真菌群落门水平丰度的90%以上.这与其它关于土壤真菌群落的研究结果基本一致[31~34].Ascomycota分布广泛, 包括腐生和寄生的种类, 对降解土壤有机质具有重要作用, Basidiomycota是陆生真菌的重要组成部分, 此次样本中Basidiomycota以Agaricales为主, 而Agaricales被认为是外生根菌真菌的典型代表[35].Zygomycota大多为陆生腐生物, 生长在富营养环境中, 因此相较于混凝土集流面, 在自然成型的黄土地集流面条件下更易生长, 不同类型的集流面在一定程度上影响了真菌群落的多样性[36].有研究认为Ascomycota和Basidiomycota是土壤中主要的真菌分解者, 在群落演替中处于前期阶段[37], 表明窖水生态系统中真菌群落结构与陆生真菌更为相似; 这可能是由于窖水并不是一个完整独立的水生生态系统, 在农村地区的集流面周围通常存在枯死树叶和家禽粪便等, 以上腐殖质同时也为陆生真菌提供了天然的生长基质和繁殖环境, 而集流雨水的外源输送作用使窖水与陆生真菌有着密不可分的联系, 沉积物中较高的碳氮含量也证明了较多的外源输入.总的来说, 两种集流面下的水窖沉积物真菌群落结构特征相似之处表现为Ascomycota、Basidiomycota和Zygomycota均为优势菌门, 不同之处为黄土地集流面中各样本优势门之间丰度差异较大; 同时说明混凝土集流面条件下沉积物的真菌群落较黄土地集流面条件下更为稳定, 抗冲击能力也更强.
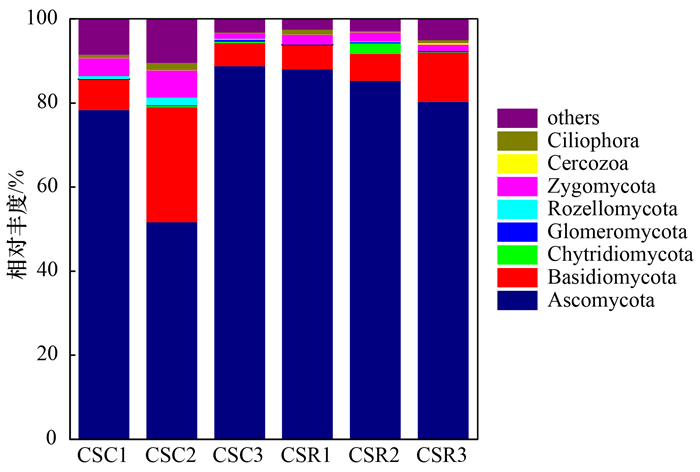
|
图 1 门水平的真菌群落结构及分布 Fig. 1 Fungal communities and distribution of the samples at the phylum level |
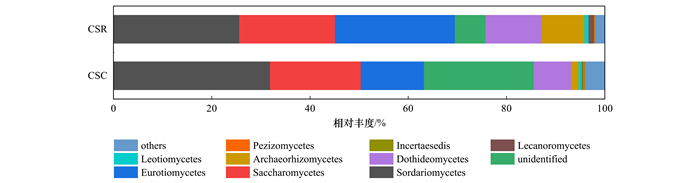
|
图 2 真菌群落Ascomycota的结构及分布 Fig. 2 Fungal communities and distribution of the samples at the Ascomycota |
使用Bray-Curtis相似度[38], 通过聚类热图(图 3)对丰度前50的属水平真菌群落进行比较, 可以看出同一集流面条件下的样本更易被聚类在一起, 并且在6个样本中可以观察到6个明显的具有较高丰度的真菌属簇, 分别是Myrothecium-Mycoleptodiscus, Aureobasidium-Verticillium, Leptosphaeria-Sporobolomyces, Gibberella-Pichia, Penicillium-Mycosphaerella和Basidiobolus-Schizophyllum, 这些组也代表对应样本的中心功能属.红曲霉属(Monascus)相对丰度最大(33.6%), Monascus是一种丝状真菌, 红曲菌科中唯一的隶属分类单元[39, 40]; Monascus广泛存在于各种发酵过程, 且Monascus具有抑菌防腐的作用并早在明代著作《天工开物》中就被记载[41], 这或许能部分解释窖水中在贮存过程中水质并未出现恶化的原因, 除此之外, 6个样本中有19.41% ~62.11%的真菌序列无法分类, Zhang等[42]通过对3座水库沉积物的研究发现在门分类水平上多达65%的真菌序列无法分类, 在目前已知的100 000种真菌中仅有3 000种来自水生环境[43], 这表明在水窖沉积物中有大量的未分类的功能属真菌维持着水窖中的生境平衡.
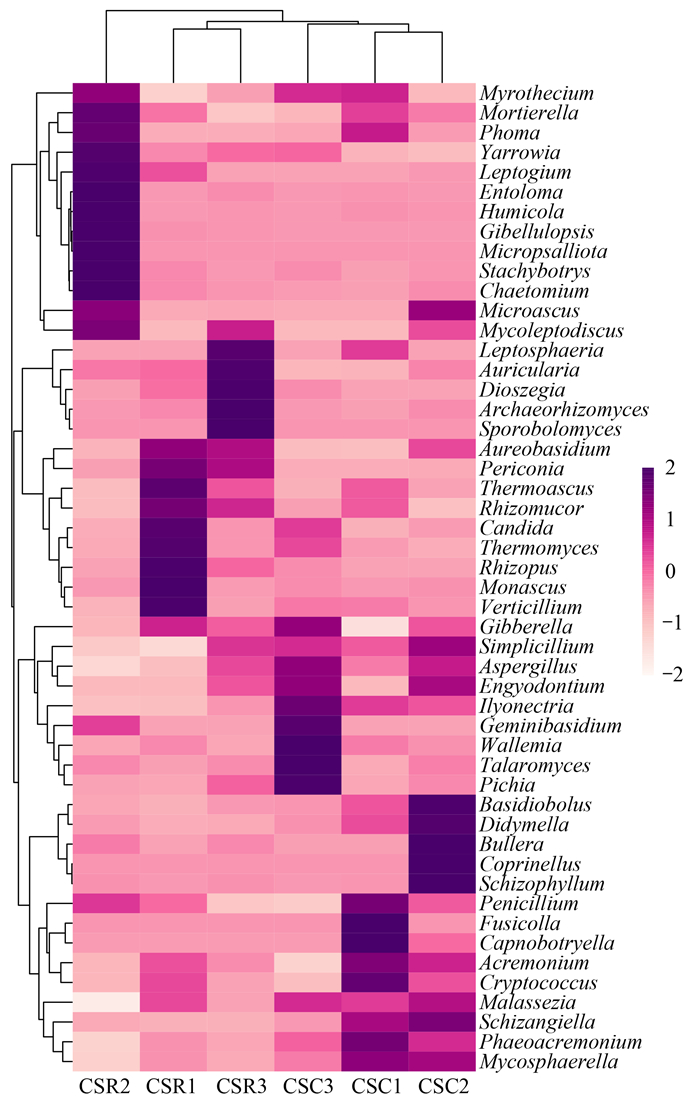
|
图 3 分类前50属的热图 Fig. 3 Heat-map of the top 50 classified genera |
图 4显示为6个样品OTU种类差异性的UpSet图, 整体而言, 黄土地集流面平均OTU序列312个, 混凝土集流面平均OTU序列374个, 表明混凝土集流面比黄土地集流面的真菌群落多样性更高, 这一点从α多样性指数中也可以得到印证.6个样品中OTU序列数目大小为: CSR2>CSC2>CSR1>CSC1>CSC3>CSR3, 样品CSR2中OTU序列数最多为578个, 无论是组内还是组间OTU聚类分析, 都预示着样品CSR2有着较多的真菌种类, 样品CSR3的OTU序列数最少为168, 反之则预示着样品CSR3真菌种类较少, 表明同一集流面环境也存在着较大的个体差异.6个样品独有的OTU序列数由大到小分别为: CSR2(331)>CSC2(301)>CSR1(197)>CSC1(115)>CSR3(57)>CSC3(56), 基本与6个样品的OTU序列数量顺序一致, 混凝土集流面的环境条件和窖水理化性质较黄土地集流面更好, 水体污染相对较轻, 也就更适应真菌的生长繁殖, 真菌多样性也就更高, 独有的OTU序列也就更多[44].分析结果表明, 6个样品共有OTU序列31个, 组间样品CSC1、CSC2和CSC3共有OTU序列2个, CSR1、CSR2和CSR3共有OTU序列7个, 混凝土集流面下的真菌群落结构更为相似.
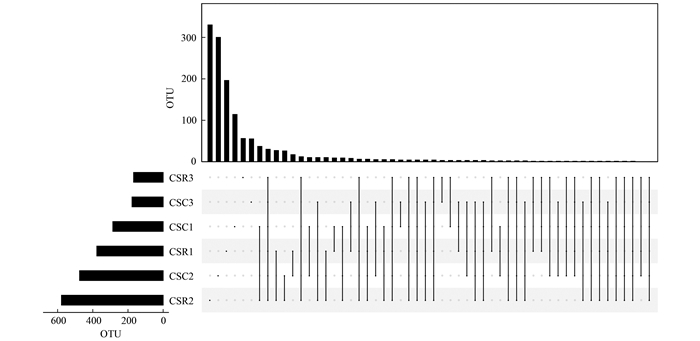
|
左侧的柱状图表示每个样品所包含OTU序列数, 右侧黑色圆点表示样品独有OTU序列数, 连线表示对应样本共有OTU序列数, 与上方柱状图一一对应 图 4 OTU分布UpSet图 Fig. 4 UpSet diagram showing the distribution of OTUs |
为了证明在不同集流面下的样本菌群具有显著丰度差异, 运用LEfSe分析方法进行生物标志物分析[45], 研究与物种层级关系相一致的差异物种.LEfSe的分析结果为一个包含5级分支(由内至外依次为门、纲、目、科和属)的圆圈图, 显示了各个分类水平上一些具有统计学意义的显著差异物种, 并在其中用不同颜色的圆圈标识以区别于大量其它的非显著物种, 其中LDA值越高则说明该菌群在该分类单元下的显著差异中贡献值也越大[46].
对6个沉积物样本使用LEfSe分析(图 5), 共得到24个不同分类的差异物种且得分超过3.0, 其中11个菌群显著富集在CSC样本, 包括1个门(Rozellomycota)、2个目(Diaporthales、Basidiobolales)、2个科(Togniniaceae、Basidiobolaceae)和6个属(Mycosphaerella、Ilyonectria、Apiosordaria、Didymella、Schizangiella、Phaeoacremonium); 13个菌群显著富集在CSR样本中, 包括2个纲(Saccharomycetes、Lecanoromycetes)、4个目(Saccharomycetales、Peltigerales、Thelephorales、Auriculariales)、3个科(Thelephoraceae、Herpotrichiellaceae、Incertaesedis)和4个属(Gibellulopsis、Chaetomium、Auricularia、Periconia), 其中CSC中对差异性贡献最大的菌群是Basidiobolales, 贡献最小的是Mycosphaerella; CSR中对差异性贡献最大的菌群是Saccharomycetales, 贡献最小的是Periconia.以Basidiobolales和Saccharomycetales为例, 在两种环境下都检测到了Basidiobolales和Saccharomycetales, 在目分类水平上, Basidiobolales在CSC中的平均丰度是CSR的9倍, 相比之下, 在CSR中观测到的Saccharomycetales平均丰度却高于CSC, 以上结果表明, 在不同的集流面环境下有利于不同的生物标志物生长.
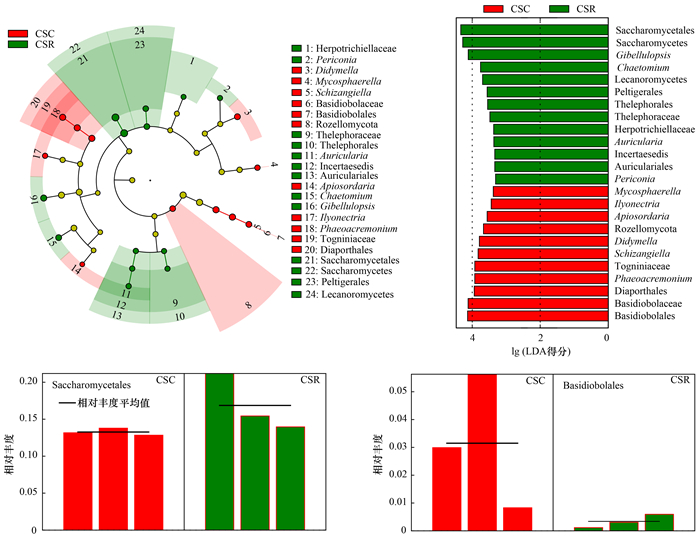
|
图 5 窖水沉积物真菌群落的LEfSe分析 Fig. 5 LEfSe analysis of fungal communities in cellar water |
微生物会对生态系统中环境的细微变化做出敏感反应[47], 因此微生物群落特征与环境的改变有着密不可分的联系.研究窖水沉积物中真菌群落与环境因子之间的相关联系对探索窖水水质变化有重大意义.运用Canon5.0软件对窖水沉积物真菌群落与环境因子之间的关联性进行RDA分析, 如图 6(a)所示.门水平真菌群落与环境因子RDA分析图, TP与Ciliophora、Cercozoa和Zygomycota为正相关关系, 与Ascomycota、Chytridiomycota和Glomeromycota为负相关关系, 与Basidiomycota和Rozellomycota几乎无联系; NO3--N、TN和含水率与Ciliophora、Zygomycota、Rozellomycota和Basidiomycota为正相关关系, 与Cercozoa、Ascomycota和Glomeromycota为负相关关系, 与Chytridiomycota几乎无联系, 对比属水平上真菌群落与环境因子RDA分析可以看出[图 6(b)], 在总体相关性基本一致的情况下仍有少部分物种表现出与所在门水平相反的相关性, 如monascus和candida与TN成正相关, 但是所在门分类Ascomycota与TN为负相关, 表明在窖水生态系统中不同分类水平下的真菌群落与环境因子间的相关性并不完全相同.
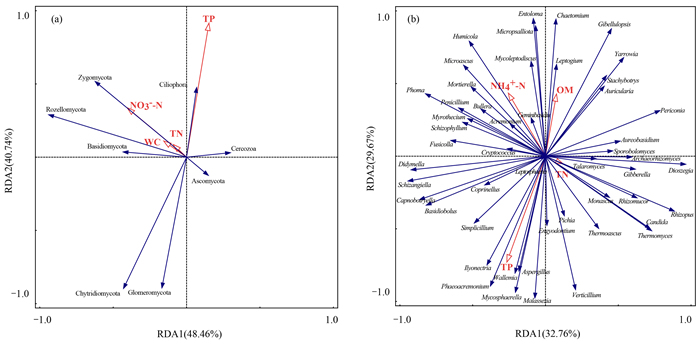
|
空心箭头表示环境因子, 箭头方向表示环境变量值增长最陡的方向, 实心箭头表示物种, 每个箭头指向对应物种丰度增加最大的方向; 两箭头之间的夹角为锐角时表示正相关, 钝角时表示负相关[48], 箭头长度表示相关性强弱; WC: 含水率, OM: 有机质 图 6 真菌群落与环境因子的RDA分析 Fig. 6 RDA analysis of fungal community and environmental factors |
群落-环境因子共现性网络是由节点和边组成的在时间或空间状态上关于生态系统内各研究对象之间相互关系的快照体现, 点线的形式包含非常丰富的信息.通常节点代表微生物或环境因子.边表示节点之间具有统计学意义的关联关系, 网络分析可以为复杂环境样本中的微生物相关分析提供新思路.通过选取斯皮尔曼相关系数|r|>0.8, 显著性P<0.01的物种构建共现性网络, 图中节点大小与节点自身的度成比例, 节点与节点之间边的颜色表明了物种间和物种与环境因子间的相互作用关系: 红色表明两节点间为正向关系, 绿色表明为负向关系, 网络参数统计如表 3所示, 在不同分类水平的群落-环境因子共现性网络中均观察到真菌之间强大的共生模式和由真菌与环境因子共同组成的相互关系(图 7), 结果显示正向边占比大于负向边, 表明真菌群落与环境因子之间有明显的正相关性, 真菌群落间合作关系大于竞争关系[49], 这种微生物之间和微生物与环境因子之间的协作机制对于维持生境平衡具有至关重要的作用.随着分类水平的不断递进, 真菌群落的模块化水平也越来越高, 模块化值高于0.4则认为该网络具有模块结构, 即代表拥有相似的生态位[50], 不同分类水平的网络参数如表 3所示; 此外, 在这些节点中, 各分类水平的最大节点度分别为门水平的Basidiomycota(4个节点), 纲水平的Microbotryomycetes(6个节点), 目水平的Dothideales和Helotiales(8个节点), 科水平的Incertaesedis(8个节点), 属水平的Phoma、Thermomyces、Candida、Humicola、Fusicolla、Trichosporon、Microascus和Rhizopus(9个节点).Ascomycota一般都有较厚的细胞壁用以维持细胞温度和正常的生理活动, 所以对较低的窖水温度并无显著变化[51], 且外源输入为Ascomycota提供了充足的氮源和有机质使得Ascomycota成为优势菌门; 而在网络图中可以看到Ascomycota和Basidiomycota之间具有更强的竞争关系, 且过多氮源输入抑制了Basidiomycota的生长, 但Basidiomycota对复杂环境的适应能力和对碳的利用能力也使得其丰度也较高[52]; Zygomycota虽与其它菌群无较强竞争关系, 但Zygomycota主要是在富营养型环境中生长, 气候较冷会降低其相对丰度.还有一种可能, Ascomycota和Basidiomycota较强的生长繁殖能力是竞争作用, 使得局部形成竞争环境不利于Zygomycota的生长.
|
|
表 3 群落-环境因子共现性网络参数统计 Table 3 Parameter statistics of co-occurrence network of community and environment factors |
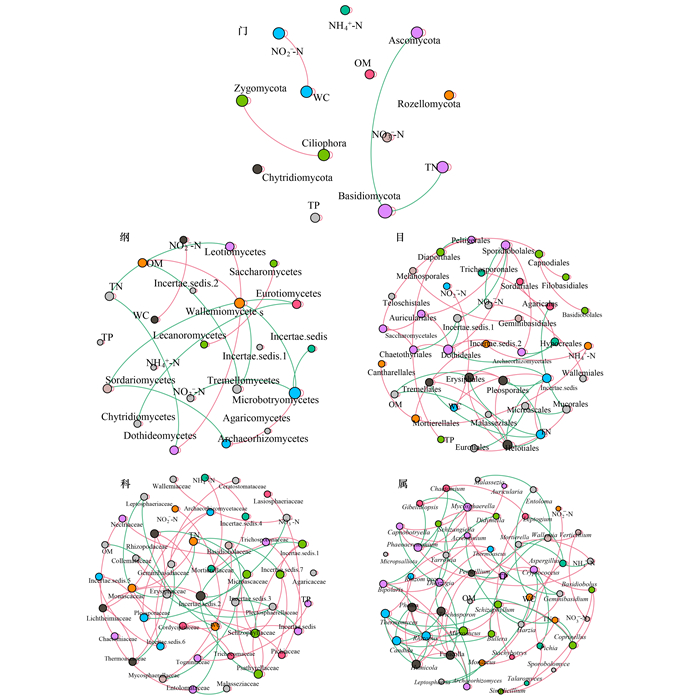
|
图 7 群落-环境因子共现性网络 Fig. 7 Co-occurrence network of community and environment factors |
(1) 混凝土集流面的真菌群落多样性与稳定性高于黄土地集流面.不同材质集流面的真菌群落结构组成相似性较高, 但丰度差异较大.Ascomycota和Basidiomycota为优势门, 真菌群落结构与陆生土壤真菌结构较为相似, 且存在大量未知真菌.
(2) 不同集流面条件下真菌群落存在差异, 黄土地集流面中对差异贡献最大的菌群是Basidiobolales, 最小的是Mycosphaerella; 混凝土集流面中对差异性贡献最大的菌群是Saccharomycetales, 贡献最小的是Periconia.不同集流面的环境与卫生条件对真菌存在显著影响, 改善窖水集流环境与卫生条件, 对于提高窖水水质具有积极作用.
(3) 集雨水窖沉积物中真菌群落间合作关系大于竞争关系, 真菌丰富度与多种环境因子相关, 在门分类水平上, TP和NO3--N为主要影响因素, 而在属分类水平NH4+-N、TP和有机质为主要影响因素, 并且沉积物真菌不仅与外部环境因子相关, 与微生物间复杂的相互作用也具有密不可分的联系.
| [1] |
彭柯, 董志, 邸琰茗, 等. 基于16S rRNA高通量测序的北运河水体及沉积物微生物群落组成对比分析[J]. 环境科学, 2021, 42(11): 5424-5432. Peng K, Dong Z, Di Y M, et al. Contrasting analysis of microbial community composition in the water and sediments of the North Canal based on 16S rRNA high-throughput sequencing[J]. Environmental Science, 2021, 42(11): 5424-5432. |
| [2] | Perkins T L, Clements K, Baas J H, et al. Sediment composition influences spatial variation in the abundance of human pathogen indicator bacteria within an estuarine environment[J]. PLoS One, 2014, 9(11). DOI:10.1371/journal.pone.0112951 |
| [3] | Jamieson R, Gordon R, Joy D, et al. Assessing microbial pollution of rural surface waters: A review of current watershed scale modeling approaches[J]. Agricultural Water Management, 2004, 70(1): 1-17. DOI:10.1016/j.agwat.2004.05.006 |
| [4] | Floudas D, Binder M, Riley R, et al. The Paleozoic origin of enzymatic lignin decomposition reconstructed from 31 fungal genomes[J]. Science, 2012, 336(6089): 1715-1719. DOI:10.1126/science.1221748 |
| [5] | Taylor D L, Sinsabaugh R L. The soil fungi: occurrence, phylogeny, and ecology[A]. In: Paul E A (Ed.). Soil Microbiology, Ecology and Biochemistry[M]. New York: Academic Press, 2015. 77-109. |
| [6] | Schadt C W, Martin A P, Lipson D A, et al. Seasonal dynamics of previously unknown fungal lineages in tundra soils[J]. Science, 2003, 301(5638): 1359-1361. DOI:10.1126/science.1086940 |
| [7] | Fries J S, Characklis G W, Noble R T. Sediment-water exchange of Vibrio sp. and fecal indicator bacteria: implications for persistence and transport in the Neuse River Estuary, North Carolina, USA[J]. Water Research, 2008, 42(4-5): 941-950. DOI:10.1016/j.watres.2007.09.006 |
| [8] |
常敏, 王娟, 田峰, 等. 红海榄根际土壤来源的泡盛曲霉(Aspergillus awamori)F12及其代谢产物的抗菌活性分析[J]. 微生物学报, 2010, 50(10): 1385-1391. Chang M, Wang J, Tian F, et al. Antibacterial activity of secondary metabolites from Aspergillus awamori F12 isolated from rhizospheric soil of Rhizophora stylosa Griff[J]. Acta Microbiologica Sinica, 2010, 50(10): 1385-1391. |
| [9] | Sarkar S, Satheshkumar A, Premkumar R. Hexavalent chromium (Cr(Ⅵ)) removal by live mycelium of a Trichoderma harzianum strain[J]. Molecular Soil Biology, 2013, 4(1). DOI:10.5376/msb.2013.04.0001 |
| [10] |
田连生, 陈菲. 木霉对多菌灵的生物降解特性研究[J]. 土壤学报, 2009, 46(6): 1127-1131. Tian L S, Chen F. Carbendazim biodegradation characteristics of Trichoderma[J]. Acta Pedologica Sinica, 2009, 46(6): 1127-1131. DOI:10.3321/j.issn:0564-3929.2009.06.022 |
| [11] |
杨浩, 张国珍, 杨晓妮, 等. 典型集雨人饮地区窖水微生物群落多样性及差异解析[J]. 环境科学, 2017, 38(11): 4733-4746. Yang H, Zhang G Z, Yang X N, et al. Microbial community diversity and differences in cellar water of typical rainwater harvesting area[J]. Environmental Science, 2017, 38(11): 4733-4746. DOI:10.13227/j.hjkx.201702128 |
| [12] |
刘驰, 李家宝, 芮俊鹏, 等. 16S rRNA基因在微生物生态学中的应用[J]. 生态学报, 2015, 35(9): 2769-2788. Liu C, Li J B, Rui J P, et al. The applications of the 16S rRNA gene in microbial ecology: current situation and problems[J]. Acta Ecologica Sinica, 2015, 35(9): 2769-2788. |
| [13] | Margulies M, Egholm M, Altman W E, et al. Genome sequencing in microfabricated high-density picolitre reactors[J]. Nature, 2005, 437(7057): 376-380. DOI:10.1038/nature03959 |
| [14] | Chen Z J, Tian W, Li Y J, et al. Responses of rhizosphere bacterial communities, their functions and their network interactions to Cd stress under phytostabilization by Miscanthus spp.[J]. Environmental Pollution, 2021, 287. DOI:10.1016/j.envpol.2021.117663 |
| [15] | Panzer K, Yilmaz P, Weiß M, et al. Identification of habitat-specific biomes of aquatic fungal communities using a comprehensive nearly full-length 18S rRNA dataset enriched with contextual data[J]. PLoS One, 2015, 10(7). DOI:10.1371/journal.pone.0134377 |
| [16] |
商潘路, 陈胜男, 黄廷林, 等. 深水型水库热分层诱导水质及真菌种群结构垂向演替[J]. 环境科学, 2018, 39(3): 1141-1150. Shang P L, Chen S N, Huang T L, et al. Vertical distribution of fungal community composition and water quality during the deep reservoir thermal stratification[J]. Environmental Science, 2018, 39(3): 1141-1150. DOI:10.13227/j.hjkx.201706291 |
| [17] |
赵彦玲, 郑世学, 方治伟, 等. 洪湖沉积物中真菌群落结构的垂直分布[J]. 菌物学报, 2011, 30(5): 721-728. Zhao Y L, Zheng S X, Fang Z W, et al. Vertical distribution of fungal communities in Honghu Lake sediment[J]. Mycosystema, 2011, 30(5): 721-728. DOI:10.13346/j.mycosystema.2011.05.016 |
| [18] | Liu Z B, Wang Z Y, Sun J Y, et al. The dynamics of volatile compounds and their correlation with the microbial succession during the traditional solid-state fermentation of Gutian Hong Qu glutinous rice wine[J]. Food Microbiology, 2020, 86. DOI:10.1016/j.fm.2019.103347 |
| [19] | Blaxter M, Mann J, Chapman T, et al. Defining operational taxonomic units using DNA barcode data[J]. Philosophical Transactions of the Royal Society B: Biological Sciences, 2005, 360(1462): 1935-1943. DOI:10.1098/rstb.2005.1725 |
| [20] |
杨浩, 杨晓妮, 张国珍, 等. 窖水中微生物降解污染物的关键细菌[J]. 环境科学, 2018, 39(10): 4766-4777. Yang H, Yang X N, Zhang G Z, et al. Key bacteria for the microbial degradation of pollutants in cellar water[J]. Environmental Science, 2018, 39(10): 4766-4777. DOI:10.13227/j.hjkx.201711123 |
| [21] | Bokulich N A, Subramanian S, Faith J J, et al. Quality-filtering vastly improves diversity estimates from Illumina amplicon sequencing[J]. Nature Methods, 2013, 10(1): 57-59. DOI:10.1038/nmeth.2276 |
| [22] |
赵炜, 李杰, 谢慧娜, 等. 兰州市春季微生物气溶胶浓度、粒径及细菌群落结构分布特性[J]. 环境科学, 2021, 42(4): 1668-1678. Zhao W, Li J, Xie H N, et al. Concentration and particle size distribution characteristics of microbial aerosol and bacterial community structure during spring in Lanzhou City, China[J]. Environmental Science, 2021, 42(4): 1668-1678. DOI:10.13227/j.hjkx.202006023 |
| [23] |
张紫薇, 陈召莹, 张甜娜, 等. 岗南水库沉积物好氧反硝化菌群落时空分布特征[J]. 环境科学, 2022, 43(1): 314-328. Zhang Z W, Chen Z Y, Zhang T N, et al. Spatial and temporal distribution of aerobic denitrification bacterial community in sediments of Gangnan reservoir[J]. Environmental Science, 2022, 43(1): 314-328. DOI:10.13227/j.hjkx.202104196 |
| [24] |
赵鑫宇, 剧泽佳, 陈慧, 等. 石家庄市土壤中喹诺酮类抗生素空间分布特征及其与微生物群落相关性[J]. 环境科学, 2022, 43(9): 4684-4696. Zhao X Y, Ju Z J, Chen H, et al. Spatial distribution of quinolone antibiotics and its correlation relationship with microbial community in soil of Shijiazhuang City[J]. Environmental Science, 2022, 43(9): 4684-4696. |
| [25] |
许晴, 张放, 许中旗, 等. Simpson指数和Shannon-Wiener指数若干特征的分析及"稀释效应"[J]. 草业科学, 2011, 28(4): 527-531. Xu Q, Zhang F, Xu Z Q, et al. Some characteristics of Simpson index and the Shannon-Wiener index and their dilution effect[J]. Pratacultural Science, 2011, 28(4): 527-531. DOI:10.3969/j.issn.1001-0629.2011.04.001 |
| [26] |
王晓丽, 其勒格尔. 黄河内蒙古段表层沉积物细菌多样性及群落结构类型[J]. 生态学报, 2020, 40(2): 578-589. Wang X L, Qi L G E. Bacterial diversity and community structure in surface sediments of Yellow River from Inner Mongolia section[J]. Acta Ecologica Sinica, 2020, 40(2): 578-589. |
| [27] | Geisen S, Mitchell E A D, Adl S, et al. Soil protists: a fertile frontier in soil biology research[J]. FEMS Microbiology Reviews, 2018, 42(3): 293-323. DOI:10.1093/femsre/fuy006 |
| [28] |
罗正明, 刘晋仙, 周妍英, 等. 亚高山草地土壤原生生物群落结构和多样性海拔分布格局[J]. 生态学报, 2021, 41(7): 2783-2793. Luo Z M, Liu J X, Zhou Y Y, et al. Community structures and diversity patterns of the soil protist communities along an altitudinal gradient in a subalpine grassland[J]. Acta Ecologica Sinica, 2021, 41(7): 2783-2793. |
| [29] | Bärlocher F, Boddy L. Aquatic fungal ecology-How does it differ from terrestrial?[J]. Fungal Ecology, 2016, 19: 5-13. |
| [30] | Buée M, Reich M, Murat C, et al. 454 Pyrosequencing analyses of forest soils reveal an unexpectedly high fungal diversity[J]. New Phytologist, 2009, 184(2): 449-456. |
| [31] |
朱书红, 辉朝茂, 赵秀婷, 等. 甜龙竹不同种植年限对土壤真菌群落的影响[J]. 环境科学, 2023, 44(6): 3408-3417. Zhu S H, Hui C M, Zhao X T, et al. Effects of different planting years of Dendrocalamus brandisii on soil fungal community[J]. Environmental Science, 2023, 44(6): 3408-3417. |
| [32] |
李明美, 全飞, 孙树晴, 等. 近自然管理方式对橡胶林根际土壤真菌群落结构和多样性的影响[J]. 南方农业学报, 2022, 53(4): 1121-1130. Li M M, Quan F, Sun S Q, et al. Effects of close-to-natural management on the community structure and diversity of fungi in the rhizosphere soil of rubber plantation[J]. Journal of Southern Agriculture, 2022, 53(4): 1121-1130. |
| [33] |
张健, 徐明, 陈进, 等. 黔中不同地区马尾松林土壤真菌群落分布特征[J]. 东北林业大学学报, 2022, 50(10): 84-89. Zhang J, Xu M, Chen J, et al. Distribution characteristics of soil fungal community in Pinus massoniana forest among different areas in the Central Guizhou[J]. Journal of Northeast Forestry University, 2022, 50(10): 84-89. |
| [34] | Yao Q, Liu J J, Yu Z H, et al. Three years of biochar amendment alters soil physiochemical properties and fungal community composition in a black soil of northeast China[J]. Soil Biology and Biochemistry, 2017, 110: 56-67. |
| [35] | Khomich M, Davey M L, Kauserud H, et al. Fungal communities in Scandinavian lakes along a longitudinal gradient[J]. Fungal Ecology, 2017, 27: 36-46. |
| [36] |
徐飞, 张拓, 怀宝东, 等. 土地利用变化对松花江下游湿地土壤真菌群落结构及功能的影响[J]. 环境科学, 2021, 42(5): 2531-2540. Xu F, Zhang T, Huai B D, et al. Effects of land use changes on soil fungal community structure and function in the riparian wetland along the downstream of the Songhua River[J]. Environmental Science, 2021, 42(5): 2531-2540. |
| [37] |
赵帆, 赵密珍, 王钰, 等. 基于高通量测序研究草莓根际微生物群落结构和多样性[J]. 土壤, 2019, 51(1): 51-60. Zhao F, Zhao M Z, Wang Y, et al. Microbial community structures and diversities in strawberry rhizosphere soils based on high-throughput sequencing[J]. Soils, 2019, 51(1): 51-60. |
| [38] | Bray J R, Curtis J T. An ordination of the upland forest communities of Southern Wisconsin[J]. Ecological Monographs, 1957, 27(4): 325-349. |
| [39] |
周康熙, 陈颖, 倪莉. 红曲霉分离纯化、分类和鉴定研究进展[J]. 中国酿造, 2021, 40(1): 7-13. Zhou K X, Chen Y, Ni L. Advances in isolation, purification, classification and identification of Monascus[J]. China Brewing, 2021, 40(1): 7-13. |
| [40] |
索昊, 张佳兰, 李利, 等. 光照调控红曲霉生长发育及聚酮类代谢产物合成的研究进展[J]. 生命科学, 2023, 35(4): 509-518. Suo H, Zhang J L, Li L, et al. Research progress on the growth and development of Monascus and the synthesis of polyketide metabolites regulated by light[J]. Chinese Bulletin of Life Sciences, 2023, 35(4): 509-518. |
| [41] |
李沅达, 邓秀娟, 吴婷, 等. 红曲霉发酵食品研究现状与分析[J]. 食品安全质量检测学报, 2022, 13(3): 688-696. Li Y D, Deng X J, Wu T, et al. Research status and analysis of Monascus fermented food[J]. Journal of Food Safety and Quality, 2022, 13(3): 688-696. |
| [42] | Zhang H H, Huang T L, Chen S N. Ignored sediment fungal populations in water supply reservoirs are revealed by quantitative PCR and 454 pyrosequencing[J]. BMC Microbiology, 2015, 15(1). DOI:10.1186/s12866-015-0379-7 |
| [43] | Tsui C K M, Baschien C, Goh T. Biology and ecology of freshwater fungi[A]. In: Li D W (Ed.). Biology of Microfungi[M]. Cham: Springer, 2016. |
| [44] |
郭芝芝, 苏振华, 邸琰茗, 等. 北运河京津冀段河道浮游微生物群落多样性变化分析[J]. 环境科学, 2022, 43(2): 803-812. Guo Z Z, Su Z H, Di Y M, et al. Analysis on diversity of plankton microbial community in the Beijing-Tianjin-Hebei section of the North Canal River[J]. Environmental Science, 2022, 43(2): 803-812. |
| [45] | Segata N, Izard J, Waldron L, et al. Metagenomic biomarker discovery and explanation[J]. Genome Biology, 2011, 12(6): 1-18. |
| [46] |
刘海燕, 王敬敬, 赵维, 等. 塔里木河中下游流域棉田及胡杨林土壤细菌群落结构及多样性研究[J]. 微生物学通报, 2019, 46(9): 2214-2230. Liu H Y, Wang J J, Zhao W, et al. Structure of soil bacteria community and diversity in cotton field and Euphrates poplar forest in the middle and lower reaches of Tarim river basin[J]. Microbiology, 2019, 46(9): 2214-2230. |
| [47] |
张彦军, 郭胜利. 环境因子对土壤微生物呼吸及其温度敏感性变化特征的影响[J]. 环境科学, 2019, 40(3): 1446-1456. Zhang Y J, Guo S L. Effect of environmental factors on variation characteristics of soil microbial respiration and its temperature sensitivity[J]. Environmental Science, 2019, 40(3): 1446-1456. |
| [48] |
王能, 张瑞雪, 吴攀, 等. 贵州某废弃煤矿酸性废水处理系统中细菌群落结构及功能分析[J]. 环境科学研究, 2021, 34(9): 2154-2163. Wang N, Zhang R X, Wu P, et al. Analysis of bacterial community structure and function in acid wastewater treatment system of an abandoned coal mine in Guizhou Province[J]. Research of Environmental Sciences, 2021, 34(9): 2154-2163. |
| [49] |
周石磊, 孙悦, 岳哿丞, 等. 雄安新区-白洋淀冬季冰封期水体好氧反硝化菌群落空间分布特征及驱动因素[J]. 环境科学, 2020, 41(5): 2177-2187. Zhou S L, Sun Y, Yue G C, et al. Spatial distribution characteristics and driving factors of aerobic denitrification bacterial community structure from Baiyangdian Lake in Xiong'an New Area during the winter freezing period[J]. Environmental Science, 2020, 41(5): 2177-2187. |
| [50] |
沈烽, 黄睿, 曾巾, 等. 分子生态网络分析研究进展[J]. 环境科学与技术, 2016, 39(S1): 94-98, 173. Shen F, Huang R, Zeng J, et al. Progress of the molecular ecology network analysis[J]. Environmental Science & Technology, 2016, 39(S1): 94-98, 173. |
| [51] | Treseder K K, Maltz M R, Hawkins B A, et al. Evolutionary histories of soil fungi are reflected in their large-scale biogeography[J]. Ecology Letters, 2014, 17(9): 1086-1093. |
| [52] | Ma A Z, Zhuang X L, Wu J M, et al. Ascomycota members dominate fungal communities during straw residue decomposition in arable soil[J]. PLoS One, 2013, 8(6). DOI:10.1371/journal.pone.0066146 |
 2023, Vol. 44
2023, Vol. 44

