2. 河海大学环境学院, 南京 210098
2. College of Environment, Hohai University, Nanjing 210098, China
我国水资源季节分配不均且年际变化大, 为了实现水资源在时间上的再分配, 近几十年来我国兴建了大批水库以缓解水资源短缺等问题.目前, 全国已建成各类水库98 112座, 总库容达8 983亿m3, 其中, 大型水库744座, 中型水库3 978座[1].水源水库通常具有水深较大、流动性差和水温分层等特点.水库水质相应特征的相关研究已经成为水库水环境变化的热点问题[2].王禹冰等[3]对北京密云水库的研究发现, 水库水深变化会影响pH、溶解氧、电导率和氨氮等水体理化性质, 从而改变不同水深处的水环境质量.因此, 探究水库不同水深对水环境的影响, 对于保障周边地区饮水安全、生态健康和经济发展具有举足轻重的作用.
作为水生态系统里的生产者、消费者和分解者, 微生物种类多样、数量庞大, 且与各类物质代谢循环密切相关.Zhao等[4]的研究发现泸沽湖细菌群落的丰富度随水深增加呈非线性下降趋势, 水深是细菌群落丰富度和更替的主要驱动因素.Tran等[5]对坦噶尼喀湖微生物在垂直深度上的分布和功能进行了系统性的研究, 发现有氧混合表层和缺氧深层水体的微生物组成和代谢潜力有明显的差异, 这与不同深度微生物在物质循环过程中的作用密切相关.王雪等[6]的研究发现宁武亚高山湖泊公海不同深度之间的细菌群落具有明显不同的空间分布格局.值得注意的是, 微生物不是单独存在于环境中, 总是有较多种群聚集在一起.微生物通过直接或间接的相互作用能实现个体无法实现的系统级功能, 例如营养循环和维持生态稳定性等, 因此解释和预测物种间相互作用是生态学的重要内容[7].探索种群间的关联作用有助于弄清群落成员共享的生态位, 全面了解微生物群落结构, 并揭示群落组成的生态规律[8].
然而, 目前对于水库微生物的研究主要集中在群落多样性和环境影响因子.例如, 秦宇等[9]的研究关注三峡库区中段的微生物群落结构特征与关键驱动因子; 王欢等[10]的研究探究不同季节里碧流河水库微生物多样性和影响因素的差异; Wang等[11]的研究阐明了受向家坝建设影响, 细菌群落结构的分布特征及与环境因子的相关性.目前利用数据集直接研究微生物种间相互作用和关键种的研究较少, 这限制了对群落互作关系和功能组成方面的认识, 因而提供的生物关联信息不全面.
生态网络模型是阐明微生物群落相互作用的重要工具.微生物之间往往存在着复杂的互作关系, 利用可视化网络对其进行分析, 可以为复杂微生物群落的环境响应问题提供一些新见解, 补充传统研究方法难以给予的额外信息[12].在一个生态网络中, 节点表示微生物物种, 连线表示物种间的相互作用关系, 一系列拓扑参数如模块化、聚类系数和连接度等能够反映出该群落的系统特征.Mo等[13]的研究利用共生网络阐明盐度对杏林湾水库微真核浮游生物群落的影响机制.其中, 根据节点在网络中的位置和连通度可识别关键种, 并分析其在网络中的潜在功能.例如, 曾有学者在构建小麦根际和土壤的细菌共现网络时, 发现土壤的细菌模块枢纽是Gemmatimonas phototrophica, 其含有紫色细菌的光合反应中心[14], 推断其可能控制特定模块内的碳合成[15].关键种一旦从群落中消失, 就会导致生态网络系统发生重大改变[16].
位于江苏省南京市的横山水库是一座以防洪和灌溉为主的水库, 其水质状况对于周边区域生态系统稳定具有重要影响.本研究采集横山水库水深0.5 m(表层)和5 m(深层)处水样, 利用16S rRNA扩增子高通量测序揭示不同深度微生物群落结构, 借助生物信息学分析方法构建微生物群落生态网络, 并识别不同深度微生物群落关键物种, 对深入解析和预测水库生态系统中微生物群落生存模式具有重要意义.
1 材料与方法 1.1 研究区域概况横山水库位于江苏省南京市, 水库集水面积3.7 km2, 大坝坝长440 m, 最大坝高16 m, 坝顶宽度5.6~6.5 m.水库库容273万m3, 属于小(I)型水库.根据现场调研, 该水库最小水深6 m左右.该水库西北部人类活动较少, 东南部紧邻住宅区, 人类活动较多.
1.2 样品采集和实验室测定本研究于2021年1月在横山水库进行野外采样.由于现场采样条件的限制, 4号点未采集, 因此只有7个样点.7个采样点的具体坐标位置如图 1和表 1所示.本研究采用无人机水体取样设备, 采集每个采样点水面下0.5 m和5 m两层水样, 即表层(surface, S)和深层(bottom, B)水样.共采集14个点位, 每个点位采集水样5 L.采用HQ40d便携多参数分析仪(Hach, CO, USA)现场测定水体的pH、水温(T)、电导率(EC)、溶解氧(DO)和氧化还原电位(ORP).浊度通过Hach 2100P浊度计(CO, USA)现场进行测定.
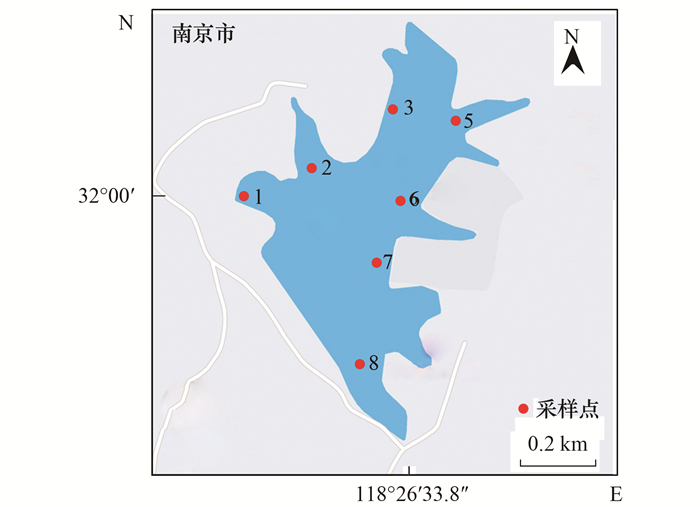
|
图 1 横山水库采样点位置 Fig. 1 Location map of sampling points of Hengshan Reservoir |
|
|
表 1 采样位点经纬度 Table 1 Longitude and latitude of sampling sites |
在实验室中, 水样先经过孔径0.45 μm的Whatman滤膜(NJ, USA)过滤, 再采用Liquid-TOC分析仪(Elementar, Frankfurt, Germany)测定总有机碳(TOC)浓度, 采用钼酸盐蓝分光光度法测量溶解性硅酸盐(SiO44-)浓度, 利用过硫酸盐消化酸水解后比色法测定总氮(TN)和总磷(TP)[17].硝酸盐(NO3-)、亚硝酸盐(NO2-)、铵盐(NH4+)和磷酸盐(PO43-)均由AA3 Seal自动分析仪(Norderstedt, Germany)测定.各指标测定均设置3个平行样品, 共测定42个水样.
用于进行16S rRNA扩增子高通量测序的水样于48 h内经滤膜(0.22 μm, Milipore)抽滤, 每张滤膜抽滤500 mL, 每个水样获得6张滤膜.抽滤后获得的滤膜于-80℃冷冻保存, 用于下一步DNA的提取.样点编号中S表示表层水样, B表示深层水样.样品编号为S-1(1号点表层水样)、B-1(1号点深层水样)、S-2和B-2等.本研究每个采样点每层水样均采集3个平行样品, 共获得42个微生物样品.
1.3 水体浮游微生物16S rRNA扩增子高通量测序本研究采用MoBio PowerWater DNA提取试剂盒(CA, USA)对浮游微生物群落DNA完成抽提, 并利用1%琼脂糖凝胶电泳检测DNA的完整性和纯度, 同时利用NanoDrop One(Thermo Scientific, DE, USA)检测DNA的浓度和纯度.
以基因组DNA为模板, 选择引物515F(5′-GT GCCAGCMGCCGCGG-3′)和806R(5′-GGACTACH VGGGTWTCTAAT-3′), 对V4可变区进行PCR扩增.反应条件为: 94℃初始变性5 min, 然后在94℃下变性30 s, 52℃下退火30 s, 72℃下延伸30 s, 共30个循环, 最后在72℃下延伸10 min.扩增产物使用1%琼脂糖凝胶电泳检测, 主带长度在290~310 bp的样品进行下一步实验.
利用GeneTools Analysis Software(Version4.03.05.0, SynGene)对PCR产物的浓度进行对比, 根据等质量原则计算各样品所需体积, 将各PCR产物充分均匀地混合.使用E.Z.N. A.Ⓡ Gel Extraction Kit凝胶回收试剂盒(Omega, USA)回收PCR混合产物.按照NEBNextⓇ UltraTM DNA Library Prep Kit for IlluminaⓇ标准流程进行建库操作.使用Illumina HiSeq2500平台对构建的扩增子文库进行PE250测序.
浮游微生物16S rRNA基因高通量测序序列已上传至美国国立生物技术信息中心(National Center for Biotechnology Information, NCBI)的序列片段数据库(Sequence Read Archive, SRA), 其序列登录号为PRJNA756625.
1.4 数据预处理及分析测序数据预处理包括: 对原始测序的reads进行裁剪(Trimmomatic, V0.33), 丢弃含N、质量值低于20及小于100 bp的reads.随后使用FLASH(V1.2.11)完成拼接, 用Mothur(V1.35.1)完成分配, 获得有效的拼接片段.并利用usearch(V8.0.1517)对有效的拼接片段进行聚类, 相似度为97%的reads聚类为操作分类单元(operational taxonomic units, OTUs).
为了解样品内物种多样性, 本研究采用Chao1指数、Equitability指数和Simpson指数对群落α多样性进行表征, 采用PCoA分析对群落β多样性进行表征, 并利用ANOSIM函数进一步表明表层和深层浮游微生物群落是否存在差异.为分析表层和深层水体间理化及生源物质指标的差异性, 首先进行PCA分析以直观表示.再计算环境因子矩阵在不同水深处间的差异(即欧氏距离, Euclidean距离), 以1号样点表层水样为基准, 获得各样点的每层水样与其的欧氏距离, 对同一个采样点不同水深进行差异显著性检验, 以说明水深对环境异质性的影响.为探究环境异质性对浮游微生物的影响, 分别计算每个点位浮游微生物群落结构在两个水深处的差异性(即bray-curtis距离), 以全部欧氏距离为横坐标, 群落差异性为纵坐标, 进行相关性分析, 并建立两者间的线性关系.
同时, 使用Mantel分析、膨胀系数分析和冗余分析(redundancy analysis, RDA)识别影响微生物群落结构变化的重要因子.首先通过Mantel和膨胀系数分析初筛影响群落结构的环境因子, 留下P < 0.05且vif < 20的环境因子, 再进行RDA分析筛选影响最为显著的因子.以上分析均通过R语言(V3.2.1)中vegan包实现.
网络构建方法为采用Cytoscape软件(V3.8.0)中插件Conet进行计算确定节点和边的信息.随后利用Gephi(V0.9.2)软件进行可视化处理, 构建生态网络图, 并计算获得网络节点数、平均度、网络直径和平均路径长度等拓扑结构参数.同时, 利用R语言WGCNA包和rnetcarto包分别计算相关矩阵的P值和分析各点在网络中的位置, 获得各物种的模块内连通性(within-module connectivity, Zi)和模块间连通性(connectivity among modules, Pi), 由此识别微生物群落关键物种.
2 结果与分析 2.1 水库不同深度水体理化指标变化横山水库7个采样点的水体理化指标见图 2.水质的酸碱度为中性, 表层水体的电导率和水温比深层高.水库的溶解氧浓度整体较高, 采样点1~3的溶解氧浓度明显高于采样点5~8. 除采样点2和6, 其余点位表层水体的氧化还原电位均高于深层.除采样点2外, 其余各点表层水体的浊度均比深层水体高, 采样点2呈现相反趋势则是因为该处水深较浅, 导致取深为5 m的水样时扰乱底部沉积物, 造成浊度较表层高.
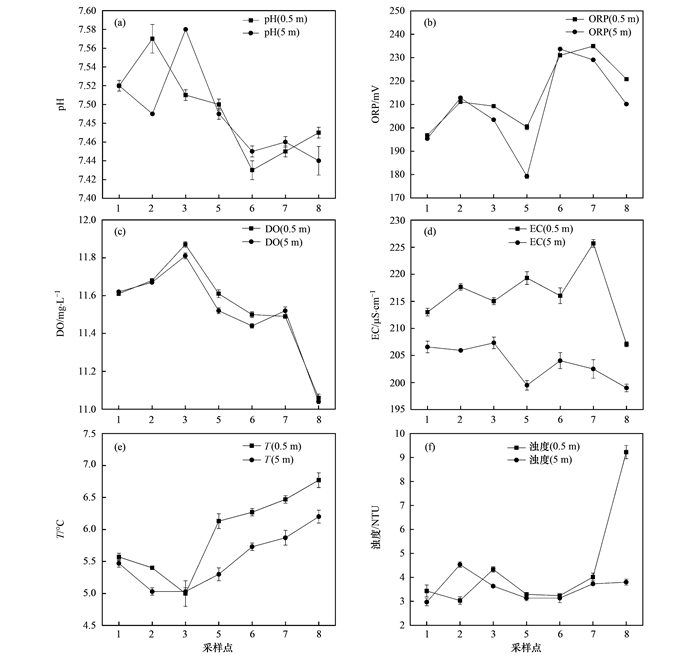
|
图 2 横山水库各采样点水体理化指标 Fig. 2 Physical and chemical indexes of water body at each sampling point in Hengshan Reservoir |
生源物质指标结果如图 3所示, 水库的ρ(TN)普遍较高, 范围为0.859~1.723 mg·L-1, ρ(TP)在0.006~0.085 mg·L-1.其中, 采样点5~8的TN和TP浓度明显高于采样点1~3.深层水体的TOC浓度和NO2-浓度均高于表层.除采样点1和3, 其余点位表层水体的SiO44-浓度高于深层.除采样点2和3, 其余点位深层水体的NH4+浓度均高于表层.
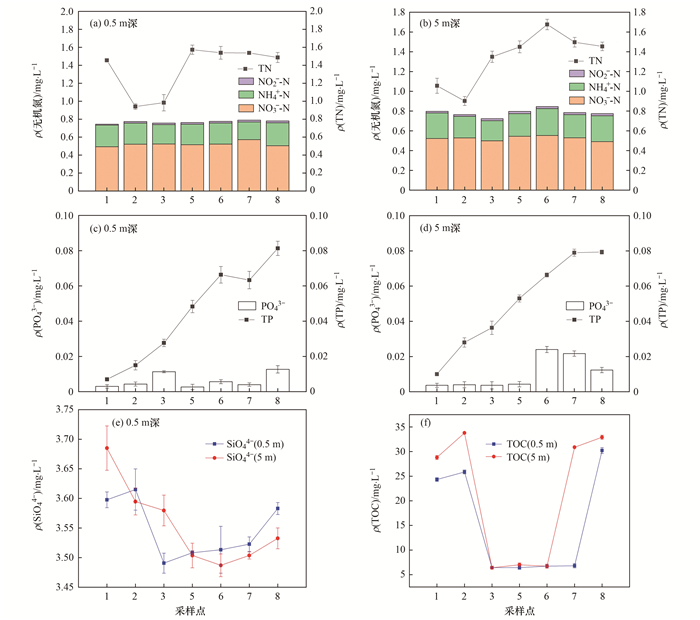
|
图 3 横山水库水体生源物质分布情况 Fig. 3 Distribution of biogenic substances in Hengshan Reservoir |
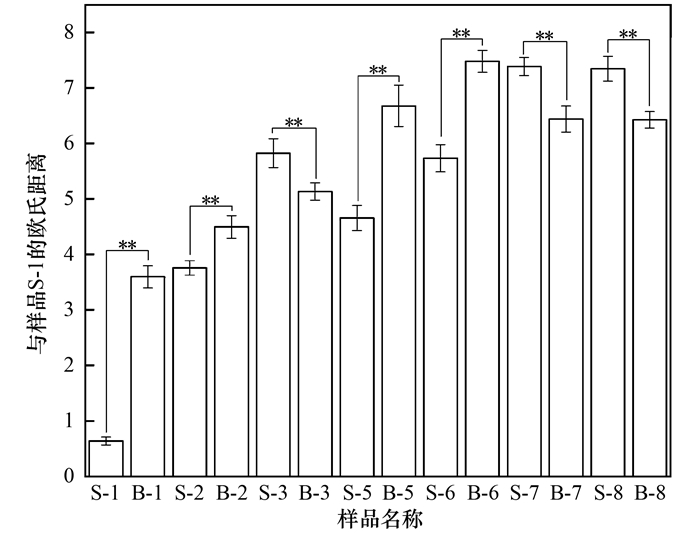
为探究不同水深对环境因子的综合影响, 本研究对表层和深层水体的理化及生源物质指标进行PCA分析.由图 4可知, PC1轴解释了环境因子构成变化的33.4%, PC2轴解释了26.03%.同时, 计算各样品与1号样点表层水样的欧氏距离, 比较环境因子在同一个采样点不同水深间的差异, 并对各采样点的表层和深层进行差异显著性检验, 结果如图 5所示.各采样点不同水深间欧氏距离的差异具有显著性(P<0.01).这说明环境因子构成在表层和深层水体间差异具有显著性, 进一步表明水深会影响水体环境异质性, 同时也证明本研究深度设置合理.
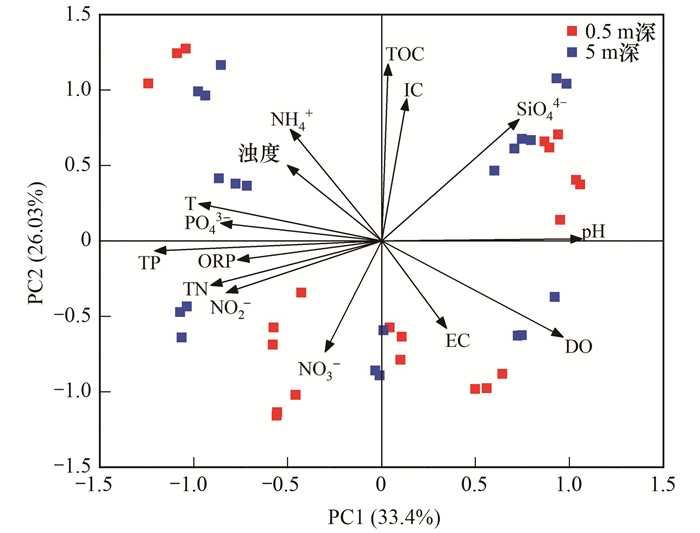
|
图 4 不同水深处理化因素差异分析 Fig. 4 Difference analysis of chemical factors in different water depths |
|
**表示P < 0.01. 图 5 各样品与S-1间的欧氏距离 Fig. 5 Euclidean distance between each sample and S-1 |
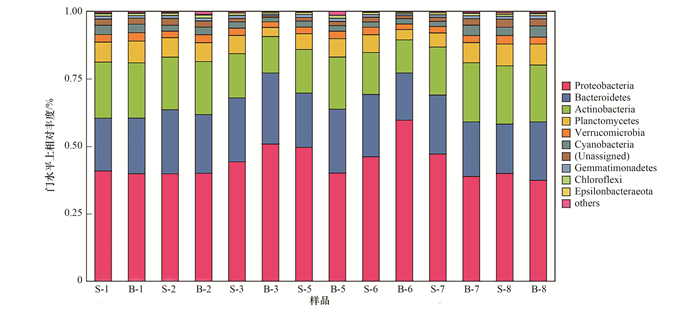
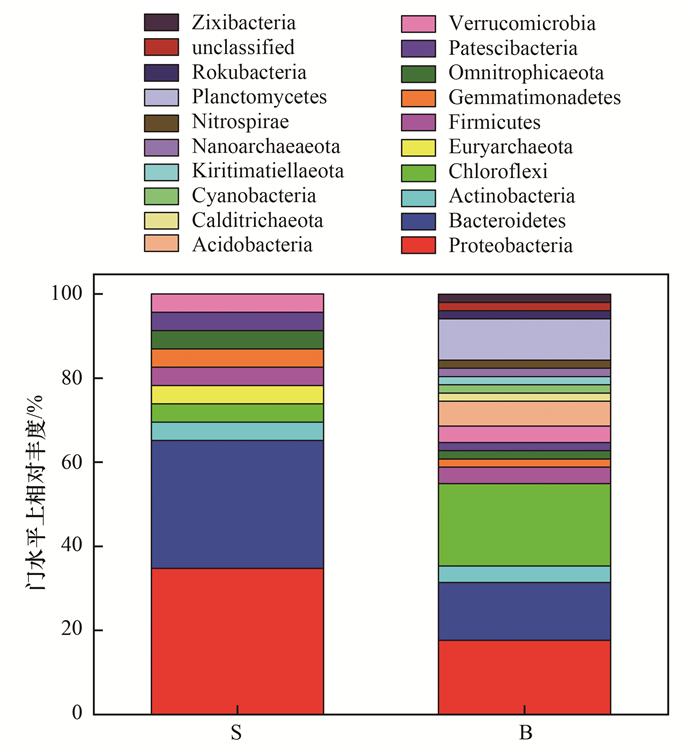
通过分类学分析可得门水平上浮游微生物物种组成以及相对丰度变化, 结果如图 6所示.各采样点的优势浮游微生物共有9个门, 其中变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)和浮霉菌门(Planctomycetes)相对丰度最高.同时, 人类活动影响较少的西北部采样点, 其表层和深层水体优势浮游微生物门相对丰度变化较小.例如Proteobacteria和Bacteroidetes在采样点1和2的相对丰度相近.
|
图 6 门水平上不同水深处浮游微生物群落物种相对丰度 Fig. 6 Species relative abundance of planktonic microbial community in different water depths at the phylum level |
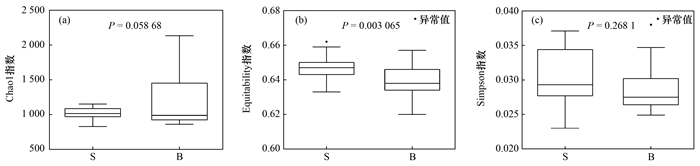
本研究选择3个指数表征浮游微生物群落α多样性, 其中Chao1指数表征微生物丰富度, Equitability指数表征微生物均匀性, Simpson指数是表征微生物丰富度和均匀度的综合性指标.由图 7(a)可知, 横山水库浮游微生物在表层水体中丰富度更高, 且Chao1指数均衡性较高; 由图 7(b)可知, 水库浮游微生物在表层水体中均匀度显著高于深层.同时, 图 7(c)结果也进一步表明, 表层水体微生物群落α多样性显著高于深层.
|
图 7 α多样性指数组间差异分析箱型图 Fig. 7 Box chart of α diversity index inter group difference analysis |
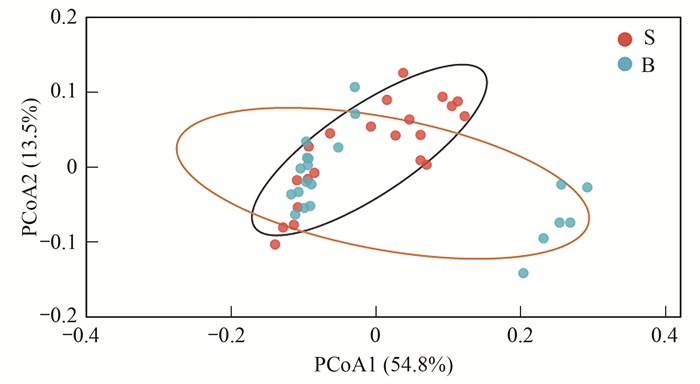
本研究采用主坐标分析(PCoA)对各采样点浮游微生物群落结构进行聚类.由图 8可知, PCoA1轴对浮游微生物群落结构变化的解释率为54.8%, PCoA2轴的解释率为13.5%.其中, 表层水体采样点较为集中, 表明各采样点表层浮游微生物群落结构较为相似.进一步对比表层及深层样点聚类情况发现, 表层浮游微生物群落结构与深层存在显著差异(ANOSIM, P < 0.05), 表明水深会显著改变浮游微生物群落结构.
|
图 8 不同水深处浮游微生物群落的PCoA图 Fig. 8 PCoA diagram of planktonic microbial community in different water depths |
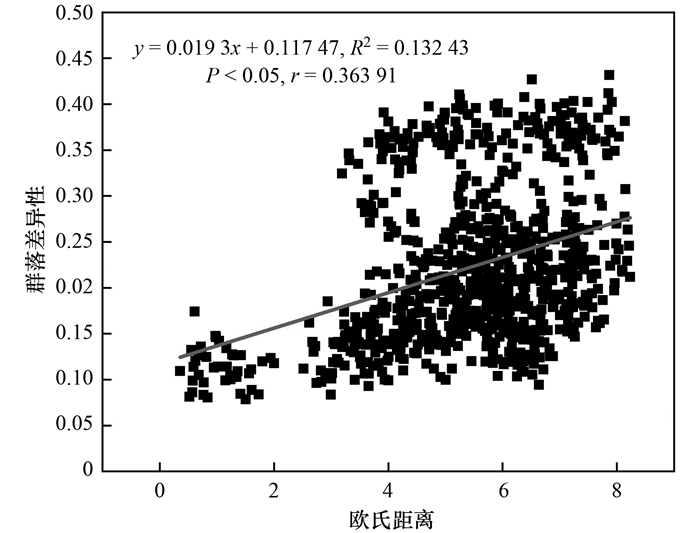
为探究影响浮游微生物群落变化的重要因子, 本研究对浮游微生物群落结构变化及环境异质性进行了相关性分析.结果如图 9所示, 浮游微生物群落结构变化与环境异质性变化呈显著正相关(r=0.36391, P < 0.05), 即随着环境异质性的增大, 群落差异性逐渐增大, 表明环境异质性会影响浮游微生物群落.
|
图 9 浮游微生物群落与环境差异性的相关性分析 Fig. 9 Correlation analysis between planktonic microbial community and environmental differences |
为确定影响浮游微生物群落结构变化的显著环境因子, 本研究利用Mantel和膨胀系数分析对环境因子进行初筛.结果如表 2所示, 当P < 0.05且vif < 20时, NH4+、NO3-、PO43-、TOC和pH这5个环境因子会显著影响浮游微生物群落结构.为进一步从上述5个因子中筛选出重要环境影响因子, 本研究采用DCA(detrended correspondence analysis)分析判定, 选择RDA分析微生物群落结构与初筛获得的5个环境因子的相关性(DCA分析中前4轴的最大梯度长度为1.145 7<3.0[9]).
|
|
表 2 Mantel分析和膨胀系数 Table 2 Mantel analysis and expansion coefficient table |
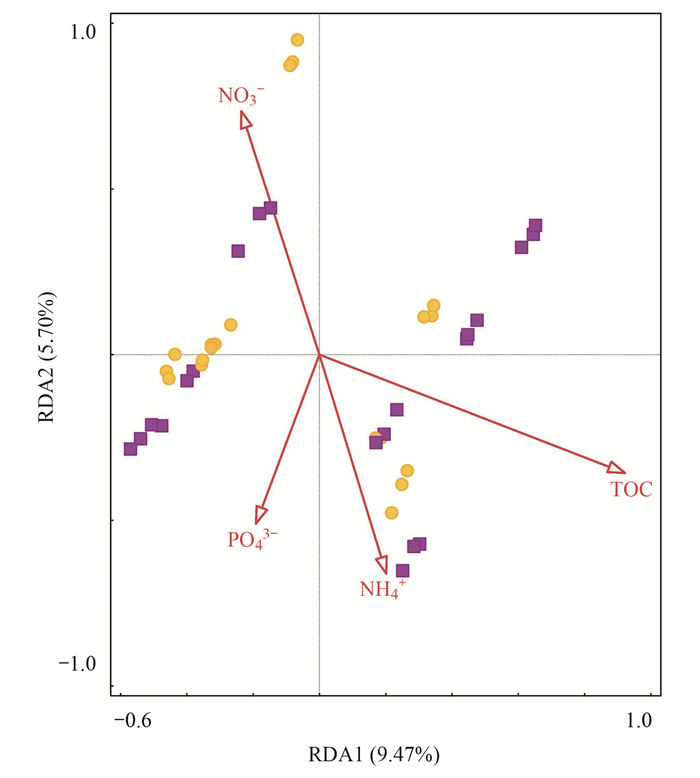
由图 10可知, RDA1轴的解释率为9.47%, RDA2轴的解释率为5.70%.且结果表明, 横山水库浮游微生物群落结构主要受到TOC、NO3-、NH4+和PO43-的影响, 其中TOC的影响最为显著. 因此, 生源物质变化是横山水库浮游微生物群落结构变化的显著影响因素.
|
黄色圆形表示表层微生物样品, 紫色方块表示深层微生物样品 图 10 水体环境因子与浮游微生物群落的RDA分析 Fig. 10 RDA analysis of water environmental factors and planktonic microbial community |
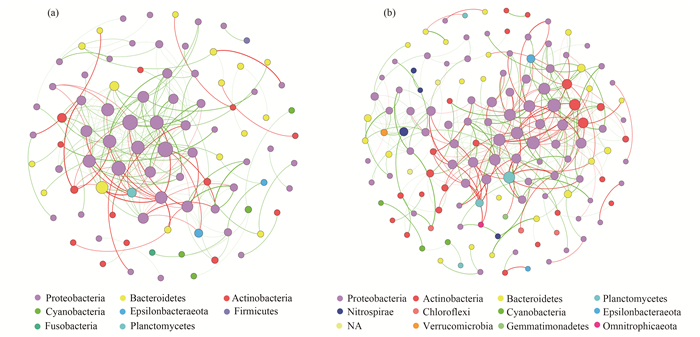
为探究群落种间互作关系, 本研究在门分类水平上构建横山水库不同水深的浮游微生物群落生态网络, 结果如图 11所示.由图 11(a)可知表层网络中共发现生物种有: 变形菌门、拟杆菌门、放线菌门、蓝细菌门和厚壁菌门等共8种, 由图 11(b)可知深层有: 变形菌门、浮霉菌门、硝化螺旋菌门、绿弯菌门和疣微菌门等共12种, 表明深层水体中浮游微生物群落中共发现的物种数目更多.节点的大小与连接数(即度)有关, 连接越多则点越大.结果表明, 表层和深层浮游微生物群落网络中连接关系最多的物种均为变形菌门.
|
(a)横山水库0.5 m深处的浮游微生物生态网络, (b)5 m深处的浮游微生物生态网络; 不同颜色的节点代表不同的门, 节点的大小按度设置; 绿线表示正相关关系, 红线表示负相关关系; NA表示未识别物种 图 11 门水平上横山水库不同水深处的浮游微生物生态网络 Fig. 11 Ecological network of planktonic microorganisms in different water depths of Hengshan Reservoir at the phylum level |
为进一步探究不同深度浮游微生物网络特性, 本研究对生态网络拓扑特性进行计算.由表 3可知, 深层浮游微生物分子生态网络的节点数和边数均高于表层, 表明其网络规模更大.同时, 表层和深层水体浮游微生物间相互作用以正相关为主, 即以协同合作为主, 竞争关系较弱, 其中表层水体微生物协同合作关系比例更高.此外, 由表 3还可知, 表层与深层网络模块化指数均大于0.4, 表明两网络均具有模块结构, 同一个模块的微生物群落可看作有相似的生态位[16].本研究发现深层网络模块化指数大于表层网络, 表明深层网络形成更大的节点模块用以维持网络的结构和功能.
|
|
表 3 横山水库分子生态网络拓扑属性 Table 3 Topological attribute table of molecular ecological network of Hengshan Reservoir |
此外, 表层网络的平均度、图密度和平均聚类系数均较高, 表明表层水体中浮游微生物生态网络联系更紧密、聚集程度更高.同时, 由表 3可知, 表层网络图平均路径长度更短, 进一步表明表层水体中浮游微生物群落的物种分离程度低、连通度高.因此, 表层浮游微生物间传递物质、能量和信息的效率更高, 对外界变化响应速度更快, 而深层浮游微生物群落抗环境干扰能力更强.
2.7 不同水深对浮游微生物群落关键物种的影响模块内连通度(Zi)反映一个节点在模块内的作用, 模块间连通度(Pi)反映该节点参与其他模块的程度.一般可将节点分为4个角色, 分别是外围节点(peripherals)、连接器(connectors)、模块枢纽(module hubs)和网络枢纽(network hubs).外围节点(Zi<2.5, Pi<0.62)表示只有少数连接且基本与模块内节点相连, 连接器(Zi<2.5, Pi≥0.62)表示与其他模块的连接度高, 模块枢纽(Zi≥2.5, Pi<0.62)表示与模块内节点有较为密切的连接, 网络枢纽(Zi≥2.5, Pi≥0.62)表示既与模块内节点高度连接又与其他模块的节点连通度高.一般将Zi≥2.5或Pi≥0.62的节点视为关键物种[7], 即除外围节点外的其他3类.
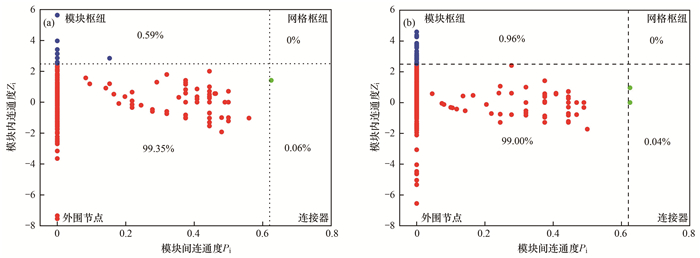
由图 12可知, 表层和深层浮游微生物网络中绝大部分节点为外围节点, 且两网络均未出现网络枢纽, 表明两个生态网络都没有同时与模块内和模块间高度连通的节点.两个网络中连接器的比例均较低, 表明网络所划分的模块之间连通性较低, 但由占比(表层网络中连接器占0.06%, 深层网络中占0.04%)可知0.5 m深的网络模块之间相互作用更紧密.深层网络中的模块枢纽显著高于表层, 表明深层网络的模块内物种之间的联系更紧密.综合结果表明, 不同水深处的网络结构是不同的.
|
(a)表层(0.5 m深), (b)深层(5 m深), 图中数字表示4种节点的占比 图 12 表层和深层水体浮游微生物Zi-Pi图 Fig. 12 The Zi-Pi diagram of planktonic microorganisms in surface and deep water |
从关键物种的占比来看, 从表层至深层水体, 关键物种由0.65%上升为1.00%.本研究在表层浮游微生物网络中筛选出23个物种为关键物种, 其中两个物种为连接器, 分别属于拟杆菌门(Bacteroidetes)和绿弯菌门(Chloroflexi); 深层浮游微生物网络中共筛选出51个关键物种, 其中两个物种为连接器, 分别属于浮霉菌门(Planctomycetes)和变形菌门(Proteobacteria).
图 13为表层及深层浮游微生物群落在门水平的关键物种组成.从中可知, 深层浮游微生物群落的关键物种数显著高于表层, 表明深层群落功能更复杂.其中, 表层网络中模块枢纽和连接器大部分属于变形菌门和拟杆菌门; 深层网络中大部分属于绿弯菌门、变形菌门和拟杆菌门.此外, 由2.2节图 6可知, 变形菌门和拟杆菌门在横山水库丰度最高, 表明其对于维持群落稳定性具有重要意义.此外, 虽然绿弯菌门在群落中相对丰度较低, 但其在关键物种中占比显著, 在深层浮游微生物种间互作方面发挥着重要作用.
|
图 13 关键物种组成 Fig. 13 Composition diagram of keystone species |
本研究发现, 深层水体的各生源物质浓度均高于表层, 这是由于蓄水后库区流速减缓, 水体颗粒物沉降作用增强, 其上吸附的生源物质随颗粒物沉降进入深层[18], 从而导致深层生源物质浓度升高.此外, 本研究还发现生源物质浓度在人类活动较多的采样点5~8显著高于人类活动较少的采样点1~3, 表明人类活动也会显著改变水库生源物质分布.同样, Wang等[17]的研究发现澜沧江中下游人口密集, 人为排放进一步促进了梯级水库养分的增加, 导致水库氮、磷浓度均显著增加.
本研究发现水深会显著改变水体环境异质性, 而环境异质性与浮游微生物群落变化具有显著相关性, 因此水深会驱动浮游微生物群落多样性和结构变化.商潘路等[19]研究金盆水库发现垂向分布上真菌种群的优势菌群和多样性存在显著差异, 种群结构受水深影响.本研究中表层水体浮游微生物多样性高于深层水体浮游微生物, 且不同水深间微生物群落存在差异, 体现了水库分层水位对微生物群落结构的重要性, 这与密云水库的研究结果一致[20].这是由于微生物的垂直分布受光照影响, 深层水体接收的光照较表层少, 微生物间竞争关系强, 最终导致对光照需求少的微生物成为优势物种, 使群落多样性降低.曾有研究发现大龙洞水库在垂直方向上细菌丰度表现为: 表层>中层>底层, 并认为这与浮游细菌的光合作用主要集中在真光层有关[21].另一方面, 已有研究表明, 随着营养盐水平的增加, 细菌群落多样性呈下降趋势[22], 与本研究中的深层生源物质浓度高于表层结果一致.此外, 本研究中浮游微生物的分布格局与TOC、NO3-、NH4+和PO43-等生源物质指标联系紧密, 以上营养盐在微生物生长发育过程中发挥重要作用.例如, 有研究表明在全球范围内, 水库是碳的净排放源[23]; TOC可以在生物、地质和化学过程发挥重要作用[24]; 在各种水生态环境中, 溶解无机氮是异养细菌重要的氮源[10]; 磷酸盐是影响南海南部海域浮游细菌群落丰度和多样性的主要因素[25].综上, 水环境中微生物的群落会随着位置而发生不同, 这是群落适应性、环境因子和人类活动变化共同导致的结果[26].
已有研究表明, 微生物群落网络拓扑结构的平均度和平均聚类系数越高, 平均路径长度越低, 物种间的相互作用越复杂[27].本研究发现表层浮游微生物群落生态网络更为复杂, 且共享生态位发展得更多.有研究发现, 网络连通性较高, 可能与微生物对环境扰动的快速响应有关[7, 28, 29], 因此, 本研究中表层微生物群落网络连通性较高表明, 表层微生物群落更易受环境扰动的影响[30].本研究表层浮游微生物协同合作关系更强的原因: 一方面是表层水体中光照更充足且水温更高, 已有研究表明资源更丰富的区域负相关关系少[31, 32]; 另一方面, 表层水体的生源物质浓度低于深层水体, 当表层细菌群落将可生化性强的物质消耗后, 可能会协同合作来分解或合成环境中难以利用的物质, 这样会增加物种间彼此之间的协同作用[33].此外, 环境因素对浮游细菌模块有正向或负向影响[34], 其中一些模块更容易受到养分变化的影响, 有助于整个群落的响应[35].值得注意的是, 不同种类微生物所需营养盐的适宜浓度不同.严重的营养污染会导致共生网络变得简单和脆弱, 并且过量的营养物质会影响微生物群落之间的潜在相互作用, 从而进一步损害生态系统的功能[27].微生物间相互作用是动态的, 依据所处的环境作出响应[36, 37].综上所述, 水深不仅会影响微生物的群落结构, 还会通过环境因子驱动微生物间种间互作模式发生变化.
由连接器和模块枢纽占比可知, 不同水深处拓扑作用不同, 模块性质也有显著变化.表层水体的关键种数目和种类均低于深层水体, 这一模式表明如果关键种受环境影响而消失, 表层微生物群落可能更容易分裂, 更容易受到整个群落崩溃的影响[38].关键种在维持群落和功能方面发挥重要作用[39, 40].变形菌门既作为优势种又作为关键种, 表明它们积极参与了水库生态系统的功能和过程[41].在将复杂分子转化为简单化合物的过程中拟杆菌门发挥着重要作用[20].曾有学者发现拟杆菌门已进化出一种独特的降解机制, 可以介导大部分拟杆菌藻类多糖的降解[42].有研究表明拟杆菌门与高浓度的溶解有机碳具有很强的相关性, 说明在高浓度的有机碳下, 拟杆菌门具有较高的活性[43], 该水库部分点位TOC浓度高, 这可能是拟杆菌成为关键种的原因.作为深层网络中关键种占比最高的绿弯菌门, 具有多种营养方式, 能涵盖已知的所有类型[44], 参与多种物质循环, 包括3-羟基丙酸循环固碳[45]和通过亚硝酸盐氧化菌参与硝化作用[46]等, 是生态学过程的重要参与者.
4 结论(1) 水深通过改变环境异质性对浮游微生物群落产生影响, 其中表层水体浮游微生物群落的α多样性显著高于深层, 且表层和深层浮游微生物群落结构存在显著差异.
(2) 共现网络分析结果表明, 表层浮游微生物间正相关关系占比79.91%, 显著高于深层微生物间正相关关系(67.78%), 说明表层微生物生态网络联系更紧密, 传递物质、能量和信息的效率更高, 对外界变化的响应速度更快.
(3) 表层浮游微生物网络中模块枢纽和连接器大部分属于拟杆菌门和变形菌门; 深层网络中大部分属于绿弯菌门、变形菌门和拟杆菌门.从表层至深层水体关键物种占比由0.65%上升为1.00%, 且深层水体浮游微生物中关键物种的种类和数目更多.
| [1] | 中华人民共和国水利部. 2019年全国水利发展统计公报[M]. 北京: 中国水利水电出版社, 2020. |
| [2] |
曾明正, 黄廷林, 邱晓鹏, 等. 我国北方温带水库——周村水库季节性热分层现象及其水质响应特性[J]. 环境科学, 2016, 37(4): 1337-1344. Zeng M Z, Huang T L, Qiu X P, et al. Seasonal stratification and the response of water quality of a temperate reservoir-Zhoucun Reservoir in north of China[J]. Environmental Science, 2016, 37(4): 1337-1344. |
| [3] |
王禹冰, 王晓燕, 庞树江, 等. 水库水体热分层的水质及细菌群落分布特征[J]. 环境科学, 2019, 40(6): 2745-2752. Wang Y B, Wang X Y, Pang S J, et al. Water quality characteristics and distribution of bacterial communities during thermal stratification in the Miyun Reservoir[J]. Environmental Science, 2019, 40(6): 2745-2752. |
| [4] | Zhao W Q, Hu A, Ni Z Y, et al. Biodiversity patterns across taxonomic groups along a lake water-depth gradient: effects of abiotic and biotic drivers[J]. Science of the Total Environment, 2019, 686: 1262-1271. DOI:10.1016/j.scitotenv.2019.05.381 |
| [5] | Tran P Q, Bachand S C, McIntyre P B, et al. Depth-discrete metagenomics reveals the roles of microbes in biogeochemical cycling in the tropical freshwater Lake Tanganyika[J]. The ISME Journal, 2021, 15(7): 1971-1986. DOI:10.1038/s41396-021-00898-x |
| [6] |
王雪, 刘晋仙, 柴宝峰, 等. 宁武亚高山湖泊细菌群落的时空格局及驱动机制[J]. 环境科学, 2019, 40(7): 3285-3294. Wang X, Liu J X, Chai B F, et al. Spatio-temporal patterns of microbial communities and their driving mechanisms in subalpine lakes, Ningwu, Shanxi[J]. Environmental Science, 2019, 40(7): 3285-3294. |
| [7] | Zhou J Z, Deng Y, Luo F, et al. Functional molecular ecological networks[J]. mBio, 2010, 1(4). DOI:10.1128/mBio.00169-10 |
| [8] | Barberán A, Bates S T, Casamayor E O, et al. Using network analysis to explore co-occurrence patterns in soil microbial communities[J]. The ISME Journal, 2012, 6(2): 343-351. DOI:10.1038/ismej.2011.119 |
| [9] |
秦宇, 郑望, 张曦, 等. 三峡库区中段水体微生物群落结构与环境因子相关性研究[J]. 长江流域资源与环境, 2021, 30(5): 1184-1193. Qin Y, Zheng W, Zhang X, et al. Correlation between microbial community structure and environmental factors in the middle section of the Three Gorges Reservoir Area[J]. Resources and Environment in the Yangtze Basin, 2021, 30(5): 1184-1193. |
| [10] |
王欢, 赵文, 谢在刚, 等. 碧流河水库细菌群落结构特征及其关键驱动因子[J]. 环境科学, 2018, 39(8): 3660-3669. Wang H, Zhao W, Xie Z G, et al. Bacterial community structure characteristics in the Biliuhe Reservoir and its key driving factors[J]. Environmental Science, 2018, 39(8): 3660-3669. |
| [11] | Wang J T, Fan H B, He X J, et al. Response of bacterial communities to variation in water quality and physicochemical conditions in a river-reservoir system[J]. Global Ecology and Conservation, 2021, 27. DOI:10.1016/j.gecco.2021.e01541 |
| [12] | Fuhrman J A, Steele J A. Community structure of marine bacterioplankton: patterns, networks, and relationships to function[J]. Aquatic Microbial Ecology, 2008, 53(1): 69-81. |
| [13] | Mo Y Y, Peng F, Gao X F, et al. Low shifts in salinity determined assembly processes and network stability of microeukaryotic plankton communities in a subtropical urban reservoir[J]. Microbiome, 2021, 9(1). DOI:10.1186/s40168-021-01079-w |
| [14] | Zeng Y H, Baumbach J, Barbosa E G V, et al. Metagenomic evidence for the presence of phototrophic Gemmatimonadetes bacteria in diverse environments[J]. Environmental Microbiology Reports, 2016, 8(1): 139-149. DOI:10.1111/1758-2229.12363 |
| [15] | Fan K K, Weisenhorn P, Gilbert J A, et al. Wheat rhizosphere harbors a less complex and more stable microbial co-occurrence pattern than bulk soil[J]. Soil Biology and Biochemistry, 2018, 125: 251-260. DOI:10.1016/j.soilbio.2018.07.022 |
| [16] |
熊俊茗, 马丽媛, 黄珊珊, 等. 分子生态网络揭示冶金微生物对能源底物的响应[J]. 生物工程学报, 2020, 36(12): 2674-2684. Xiong J M, Ma L Y, Huang S S, et al. Molecular ecological network reveals the response of metallurgical microorganisms to energy substrates[J]. Chinese Journal of Biotechnology, 2020, 36(12): 2674-2684. |
| [17] | Wang X, Wang C, Wang P F, et al. How bacterioplankton community can go with cascade damming in the highly regulated Lancang-Mekong River Basin[J]. Molecular Ecology, 2018, 27(22): 4444-4458. DOI:10.1111/mec.14870 |
| [18] |
史鹏程, 朱广伟, 杨文斌, 等. 新安江水库悬浮颗粒物时空分布、沉降通量及其营养盐效应[J]. 环境科学, 2020, 41(5): 2137-2148. Shi P C, Zhu G W, Yang W B, et al. Spatial-temporal distribution of suspended solids and its sedimentation flux and nutrients effects in Xin'anjiang Reservoir, China[J]. Environmental Science, 2020, 41(5): 2137-2148. |
| [19] |
商潘路, 陈胜男, 黄廷林, 等. 深水型水库热分层诱导水质及真菌种群结构垂向演替[J]. 环境科学, 2018, 39(3): 1141-1150. Shang P L, Chen S N, Huang T L, et al. Vertical distribution of fungal community composition and water quality during the deep reservoir thermal stratification[J]. Environmental Science, 2018, 39(3): 1141-1150. |
| [20] | Chen Y J, Liu Y, Wang X Y. Spatiotemporal variation of bacterial and archaeal communities in sediments of a drinking reservoir, Beijing, China[J]. Applied Microbiology and Biotechnology, 2017, 101(8): 3379-3391. DOI:10.1007/s00253-016-8019-1 |
| [21] |
辛胜林, 梁月明, 彭文杰, 等. 亚热带典型岩溶水库细菌丰度和细菌生产力及其与环境因子的关系[J]. 环境科学, 2018, 39(12): 5647-5656. Xin S L, Liang Y M, Peng W J, et al. Relationship between the bacterial abundance and production with environmental factors in a subtropical karst reservoir[J]. Environmental Science, 2018, 39(12): 5647-5656. |
| [22] |
薛银刚, 刘菲, 孙萌, 等. 太湖竺山湾春季浮游细菌群落结构及影响因素[J]. 环境科学, 2018, 39(3): 1151-1158. Xue Y G, Liu F, Sun M, et al. Community structure and influencing factors of bacterioplankton in spring in Zhushan Bay, Lake Taihu[J]. Environmental Science, 2018, 39(3): 1151-1158. |
| [23] | Keller P S, Marcé R, Obrador B, et al. Global carbon budget of reservoirs is overturned by the quantification of drawdown areas[J]. Nature Geoscience, 2021, 14(6): 402-408. DOI:10.1038/s41561-021-00734-z |
| [24] |
王从锋, 胡子龙, 杨正健, 等. 基于碳氮同位素的澜沧江水库TOC来源差异性分析[J]. 环境科学, 2019, 40(7): 3039-3048. Wang C F, Hu Z L, Yang Z J, et al. Analysis of total organic carbon source differences between new and old cascade reservoirs using carbon and nitrogen isotopes[J]. Environmental Science, 2019, 40(7): 3039-3048. |
| [25] |
白洁, 刘小沙, 侯瑞, 等. 南海南部海域浮游细菌群落特征及影响因素研究[J]. 中国环境科学, 2014, 34(11): 2950-2957. Bai J, Liu X S, Hou R, et al. Community structure and influencing factors of bacterioplankton in the southern South China Sea[J]. China Environmental Science, 2014, 34(11): 2950-2957. |
| [26] | Ung P, Peng C, Yuk S, et al. Dynamics of bacterial community in Tonle Sap Lake, a large tropical flood-pulse system in Southeast Asia[J]. Science of the Total Environment, 2019, 664: 414-423. DOI:10.1016/j.scitotenv.2019.01.351 |
| [27] | Yang Y Z, Gao Y C, Huang X N, et al. Adaptive shifts of bacterioplankton communities in response to nitrogen enrichment in a highly polluted river[J]. Environmental Pollution, 2019, 245: 290-299. DOI:10.1016/j.envpol.2018.11.002 |
| [28] | Chen J, Wang P F, Wang C, et al. Fungal community demonstrates stronger dispersal limitation and less network connectivity than bacterial community in sediments along a large river[J]. Environmental Microbiology, 2020, 22(3): 832-849. DOI:10.1111/1462-2920.14795 |
| [29] | Zhang B G, Zhang J, Liu Y, et al. Co-occurrence patterns of soybean rhizosphere microbiome at a continental scale[J]. Soil Biology and Biochemistry, 2018, 118: 178-186. DOI:10.1016/j.soilbio.2017.12.011 |
| [30] |
李冰, 李玉双, 魏建兵, 等. 不同土地利用方式对土壤细菌分子生态网络的影响[J]. 环境科学, 2020, 41(3): 1456-1465. Li B, Li Y S, Wei J B, et al. Effects of different land use typess on the molecular ecological network of soil bacteria[J]. Environmental Science, 2020, 41(3): 1456-1465. |
| [31] | De Menezes A B, Richardson A E, Thrall P H. Linking fungal-bacterial co-occurrences to soil ecosystem function[J]. Current Opinion in Microbiology, 2017, 37: 135-141. DOI:10.1016/j.mib.2017.06.006 |
| [32] | Banerjee S, Kirkby C A, Schmutter D, et al. Network analysis reveals functional redundancy and keystone taxa amongst bacterial and fungal communities during organic matter decomposition in an arable soil[J]. Soil Biology and Biochemistry, 2016, 97: 188-198. DOI:10.1016/j.soilbio.2016.03.017 |
| [33] |
陈光哲, 姜星宇, 胡洋. 不同营养程度湖泊中细菌群落组成及其分子生态网络特征[J]. 环境科学研究, 2020, 33(2): 375-384. Chen G Z, Jiang X Y, Hu Y. Characteristics of bacterial communities and their co-occurrence networks in lake ecosystem with different trophic states[J]. Research of Environmental Sciences, 2020, 33(2): 375-384. |
| [34] | Cao X Y, Zhao D Y, Xu H M, et al. Heterogeneity of interactions of microbial communities in regions of Taihu Lake with different nutrient loadings: a network analysis[J]. Scientific Reports, 2018, 8. DOI:10.1038/s41598-018-27172-z |
| [35] | Ren Z, Qu X D, Peng W Q, et al. Nutrients drive the structures of bacterial communities in sediments and surface waters in the river-lake system of Poyang Lake[J]. Water, 2019, 11(5). DOI:10.3390/w11050930 |
| [36] | Liu A, Archer A M, Biggs M B, et al. Growth-altering microbial interactions are responsive to chemical context[J]. PLoS One, 2017, 12(3). DOI:10.1371/journal.pone.0164919 |
| [37] |
朱瑞芬, 刘杰淋, 王建丽, 等. 基于分子生态学网络分析松嫩退化草地土壤微生物群落对施氮的响应[J]. 中国农业科学, 2020, 53(13): 2637-2646. Zhu R F, Liu J L, Wang J L, et al. Molecular ecological network analyses revealing the effects of nitrogen application on soil microbial community in the degraded grasslands[J]. Scientia Agricultura Sinica, 2020, 53(13): 2637-2646. DOI:10.3864/j.issn.0578-1752.2020.13.012 |
| [38] | Olesen J M, Bascompte J, Dupont Y L, et al. The modularity of pollination networks[J]. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(50): 19891-19896. DOI:10.1073/pnas.0706375104 |
| [39] | Xue Y Y, Chen H H, Yang J R, et al. Distinct patterns and processes of abundant and rare eukaryotic plankton communities following a reservoir cyanobacterial bloom[J]. The ISME Journal, 2018, 12(9): 2263-2277. DOI:10.1038/s41396-018-0159-0 |
| [40] |
李前, 李忠武, 聂小东, 等. 水土流失防治措施对马尾松林土壤微生物群落分子生态网络的影响[J]. 土壤学报, 2022, 59(3): 819-832. Li Q, Li Z W, Nie X D, et al. Effects of prevention and control measures of soil erosion on molecular ecological network of soil microbial community in pinus massoniana plantation[J]. Acta Pedologica Sinica, 2022, 59(3): 819-832. |
| [41] | Zhang J X, Yang Y Y, Zhao L, et al. Distribution of sediment bacterial and archaeal communities in plateau freshwater lakes[J]. Applied Microbiology and Biotechnology, 2015, 99(7): 3291-3302. DOI:10.1007/s00253-014-6262-x |
| [42] | Krüger K, Chafee M, Ben Francis T, et al. In marine Bacteroidetes the bulk of glycan degradation during algae blooms is mediated by few clades using a restricted set of genes[J]. The ISME Journal, 2019, 13(11): 2800-2816. DOI:10.1038/s41396-019-0476-y |
| [43] | Newton R J, Jones S E, Eiler A, et al. A guide to the natural history of freshwater lake bacteria[J]. Microbiology and Molecular Biology Reviews, 2011, 75(1): 14-49. DOI:10.1128/MMBR.00028-10 |
| [44] |
鲜文东, 张潇橦, 李文均. 绿弯菌的研究现状及展望[J]. 微生物学报, 2020, 60(9): 1801-1820. Xian W D, Zhang X T, Li W J. Research status and prospect on bacterial phylum Chloroflexi[J]. Acta Microbiologica Sinica, 2020, 60(9): 1801-1820. |
| [45] | Zarzycki J, Brecht V, Müller M, et al. Identifying the missing steps of the autotrophic 3-hydroxypropionate CO2 fixation cycle in Chloroflexus aurantiacus[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(50): 21317-21322. DOI:10.1073/pnas.0908356106 |
| [46] | Sorokin D Y, Lücker S, Vejmelkova D, et al. Nitrification expanded: discovery, physiology and genomics of a nitrite-oxidizing bacterium from the phylum Chloroflexi[J]. The ISME Journal, 2012, 6(12): 2245-2256. DOI:10.1038/ismej.2012.70 |
 2022, Vol. 43
2022, Vol. 43

