2. 江苏省环境科学研究院, 江苏省环境工程重点实验室, 南京 210036
2. Jiangsu Provincial Key Laboratory of Environmental Engineering, Jiangsu Provincial Academy of Environmental Science, Nanjing 210036, China
四氯乙烯(tetrachloroethene, PCE)和三氯乙烯(trichloroethylene, TCE)是氯原子取代乙烯分子中氢原子形成的化合物, 被广泛用作干洗剂、金属脱脂溶剂、萃取剂和化工中间体等[1 ~ 4].由于生产、利用、储存或者处理方式不当等一些原因, 氯代有机化合物产品的挥发、泄漏、污水排放和燃烧进入大气、土壤和地下水, 从而造成严重的环境污染[5 ~ 7].在美国环保署(EPA)的超级基金国家优先项目清单中, PCE和TCE作为常见污染物, 其中至少有60%的地点发现了TCE[2], 有9% ~ 34%的供水被认为存在TCE污染问题[8].PCE和TCE是地下水中第一和第三最常检出的挥发性有机化合物[9].同时, PCE和TCE严重危害着人类健康, 是33种优先有害物质之一[8].有研究发现, PCE和TCE及其还原脱氯产物二氯乙烯(DCEs)、氯乙烯(vinyl chloride, VC)都会对人体健康造成严重危害.触摸、食入和吸入等途径都可以使PCE和TCE进入体内[10, 11].PCE蒸气的吸入可能造成中枢神经系统急性功能障碍、头晕、嗜睡和其他形式的抑郁症, 而长期过量接触会导致神经行为功能缺陷[12, 13].接触TCE会对成人的内分泌系统、免疫系统和中枢神经系统造成严重影响, 也会造成听说障碍、肝脏疾病、皮疹、肾脏疾病和血液疾病等[2, 13 ~ 16].接触高浓度的DCEs可能会导致肝脏异常, VC具有破坏DNA的特性, 可能导致接触者患癌[15, 17].
由于氯代烯烃在地下水中普遍存在并会严重危害人类健康, 治理和解决其污染问题近年来已引起了国内外的普遍关注.在大多数含水层中, 污染物都能被存在的微生物利用转化.微生物修复与其他修复方式相比最大的优势在于容易原位修复、无次生污染和处置费用低.国内外研究已经证明[18 ~ 23], 在厌氧条件下, PCE和TCE等氯代乙烯可以作为电子受体并利用微生物进行还原脱氯.而且已经分离出许多能还原氯代烯烃的功能微生物, 如Dehalococcoides [24 ~ 26]、Dehalobacter [27, 28]、Dehalogenimonas [29]和Sulfurospirillum [30]等多种菌属.其电子受体是有机卤化合物, 电子供体是小分子有机酸, 如H2和甲酸等, 经电子传递链还原脱卤并同时能够得到能量供自己生长, 称为有机卤呼吸细菌(organohalide-respiring bacteria, OHRB)[31, 32].然而, 它们对PCE和TCE的脱氯能力不同, 只有部分脱卤细菌能够完全将其还原脱氯至无毒乙烯(ethylene, ETH), 很多土著微生物只能还原脱氯至顺-1,2-二氯乙烯(cis-1,2-dichloroethene, cis-1,2-DCE)和VC, 其它中间产物1, 1-二氯乙烯(vinylidene chloride, 1, 1-DCE)和反-1,2-二氯乙烯(trans-1,2-dichloroethylene, trans-1,2-DCE)极为少见, cis-1,2-DCE、VC和ETH为主要的脱氯产物[33, 34].1997年, Maymó-Gatell等[25]首次在厌氧微生物菌群中分离出D. mccartyi strain 195, 它是唯一能够使PCE还原脱氯为TCE并能够完全还原脱氯为ETH的菌株[24].然而, PCE脱氯为cis-1,2-DCE比其再进一步脱氯更快, 在热力学上更有利, 最慢的脱氯步骤是VC最终转化为ETH[35, 36].因此, 经常在受污染的地点观察到毒性更大的低氯代烯烃(cis-1,2-DCE和VC)的积累[37, 38].剩下的其他脱卤菌只能脱氯至中间产物cis-1,2-DCE.例如Suyama等[39]分离得到Desulfitobacterium sp. strain Y51, Scholz-Muramatsu等[40]分离得到的Sulfurospirillum multivorans和Holliger等[27]分离得到的Dehalobacter restrictus strain PER-K23都只能把PCE和TCE脱氯成cis-1,2-DCE.目前, 富含Dehalococcoides的菌群(如KB-1)的生物强化已广泛用于北美或欧洲受氯代溶剂污染场地的原位修复[41].然而, 我国高效厌氧脱卤菌剂的缺乏, 严重制约了TCE等卤代有机污染场地原位微生物修复技术的应用.因此, 获得一份高效的PCE/TCE厌氧微生物降解脱毒菌株是进行污染场地微生物修复的前提和基础, 是今后修复污染场地的重要研究方向.
本研究在实验室条件下从受氯代烯烃污染的地下水中富集到一个氯代乙烯厌氧还原脱氯的富集菌群W-1, 结合16S rRNA基因扩增子测序及荧光定量PCR(qPCR)技术阐明微生物群落结构组成以及脱氯功能菌, 并探究菌剂在受TCE污染的地下水中的脱氯潜能, 以期为地下水污染地原位修复提供菌株资源和理论指导.
1 材料与方法 1.1 供试试剂PCE、TCE、甲酸钠和乙酸钠纯度均 > 99%, VC(2 000 μg·mL-1, 可溶于甲醇)购自上海阿拉丁生化科技股份有限公司.cis-1,2-DCE纯度 > 98%, 购自安耐吉化学.ETH购自南京特种气体厂有限公司.甲醇和乙醇纯度均 > 99.8%, 购自国药集团化学试剂有限公司.乳酸钠为分析纯AR, 购自成都市科龙化工试剂厂.土壤DNA提取试剂盒购自德国凯杰QIAGEN.含重组质粒的Escherichia coli trans5α由中国科学院沈阳应用生态研究所严俊博士提供[42].质粒小量制备试剂盒GK2004-50购自上海捷瑞生物工程有限公司.本实验中厌氧培养基配方参照文献[43].
1.2 供试菌群来源建立微生物实验所用的地下水采自江苏省苏州市某污染场地, 在厌氧手套箱内, 地下水经0.22 μm滤膜抽滤, 将滤膜转移至厌氧培养基中, 以底物PCE和TCE为电子受体, 以甲醇、乙醇、甲酸钠、乙酸钠和乳酸钠的混合物为碳源和电子供体进行富集培养, 获得供试厌氧微生物富集培养液, 定期转接保持菌液活性, 并将此厌氧微生物菌群命名为W-1.
1.3 氯代乙烯还原脱氯菌群富集培养 1.3.1 富集培养体系构建以1%(体积分数)转接量从富集培养液中取1.6 mL转接至含有158.4 mL厌氧基础培养基的250 mL密闭厌氧瓶中.分别添加PCE和TCE作为电子受体进行富集培养, 监测其还原脱氯效果.实验组1添加电子受体PCE的量约为98.3 μmol(水相浓度理论值616.4 μmol·L-1), 混合电子供体中各物质的终浓度均为1 mmol·L-1, 此实验瓶编号为PCE-T1.实验组2添加电子受体TCE的量约为111.4 μmol(水相浓度理论值696.3 μmol·L-1), 混合电子供体中各物质的终浓度均为1 mmol·L-1, 设置3个重复即TCE-T1-1、TCE-T1-2和TCE-T1-3.所有菌液均放于30℃恒温厌氧(内含体积比为8∶1∶1的N2/CO2/H2混合气)培养箱内(Coy, USA), 于黑暗条件下静置培养.
1.3.2 微生物总DNA提取与16S rRNA基因扩增子测序在富集培养液初始及底物还原脱氯完全阶段, 即PCE-T1实验瓶第0、36、58、72和79 d, TCE-T1-1实验瓶第0、36和40 d, TCE-T1-2实验瓶第0、34和40 d, TCE-T1-3实验瓶第0、34和86 d, 无菌状态下抽取4 mL富集培养液至2 mL无菌离心管, 在12 000 r·min-1条件下离心10 min, 移去上清液留沉淀.之后按照QIANGEN DNeasy® PowerSoil® Pro Kit Handbook流程进行DNA提取, 最后用50 μL灭菌超纯水洗脱DNA至1.5 mL离心管内, 于-20℃保存备用.部分DNA样品(20 μL)利用高通量测序平台Illumina MiSeq PE300(Illumina, San Diego)进行16S rRNA基因扩增子测序(上海美吉生物医药科技有限公司).使用通用引物515F(5'-GTGCCAGCMGCCGCGGTAA-3')和907R(5'-CCGTCAATTCCTTTGAGTTT-3')扩增细菌和古生菌的16S rRNA基因序列V4和V5区, 随后在美吉生物公司并利用其在线平台(www.majorbio.com)分析测序数据.将MiSeq测序得到的PE reads(paired-end reads)进行样本拆分, 然后根据测序质量对双端Reads进行质控和过滤, 拼接得到优化数据, 之后使用QIIME2中序列降噪方法(DADA2)处理优化数据[44, 45], 从而得到ASV(Amplicon Sequence Variant)代表序列和丰度信息, 使用物种注释数据库silva 138/16s对ASV进行物种分类[46].
1.3.3 荧光定量PCR采用实时定量聚合酶链反应(qPCR)方法, 利用16S rRNA基因特异性引物(引物信息见表 1)追踪实验瓶(PCE-T1实验瓶第0、36、58、72和79 d, TCE-T1-1实验瓶第0、36和40 d, TCE-T1-2实验瓶第0、34和40 d, TCE-T1-3实验瓶第0、34和86 d)中Dehalococcoides和Dehalobacter的基因拷贝数.进行定量的标品为质粒DNA, 以10倍梯度稀释(基因拷贝数在101 ~ 108 copies·μL-1), 该质粒包含克隆的Dehalococcoides和Dehalobacter 16S rRNA基因片段.qPCR反应体系中有2 × ChamQ Universal SYBR qPCR Master Mix(Vazyme #Q711)10 μL、10 μmol·L-1上游和下游引物各0.5 μL及DNA模板2 μL(空白对照和稀释DNA), 剩余用ddH2O补充至20 μL.qPCR反应在QuantStudio 6 Flex(Applied Biosystems, Waltham, MA)进行, qPCR程序为两步法即50℃下维持2 min, 95℃ 10 min开始预变性, 然后40次循环, 每个循环下95℃变性15 s, 60℃退火和延伸34 s, 熔解曲线程序为仪器默认设置.每个质粒标品和DNA模板样品至少有2个技术重复.
|
|
表 1 引物信息 Table 1 Primer sequences used for qPCR and PCR |
1.3.4 Sanger法测序获得脱卤菌完整的16S rRNA基因序列
利用两组引物A和B(引物信息见表 1)扩增出Dehalococcoides 16S rRNA基因片段, 通过Sanger测序, 拼接获得接近其16S rRNA基因全长的核酸序列.A组引物27F-16S与Dhc1350R-16S, B组引物Dhc730F-16S与1492R-16S.其中27F-16S与1492R-16S是常用细菌16S rRNA的一对通用引物, Dhc730F-16S与Dhc1350R-16S是He等[47]报道过的Dehalococcoides特异性引物.PCR体系包括25 μL 2 × Phanta Max Master Mix(Vazyme #P525)、10 μmol·L-1上游和下游引物各2 μL, 总DNA模板2 μL(未稀释), ddH2O 19 μL, 共50 μL.PCR反应使用基因扩增仪(BIOER)进行, A组引物扩增程序如下:95℃预变性3 min;95℃变性15 s, 随后55℃退火30 s和72℃延伸40 s, 此反应35个循环;最后在72℃下再延伸5 min.B组引物扩增程序中72℃延伸时间设置为30 s, 其余与A组程序一致.在1%的琼脂糖凝胶上运行检查PCR产物扩增的特异性与片段大小, 所得产物交由擎科生物科技有限公司(北京)测序, 最后将测序得到的两段核酸序列拼接, 之后在EzBioCloud数据库(https://www.ezbiocloud.net/)进行16S rRNA核酸序列比对, 初步确定与本研究菌群中脱卤菌Dehalococcoides相似性最高的菌株, 并利用已报道的部分Dehalococcoides、Dehalogenimonas和Dehalobacter的16S rRNA基因序列, 在MEGA软件上使用邻接法(neighbor-joining method)构建系统发育树.
1.4 菌群W-1在地下水环境污染的应用采集300 mL受TCE污染的地下水, 因TCE的强挥发性和为保证样品初始条件一致性, 因此在实验前, 3个平行样均外源添加TCE为唯一电子受体, 使其水相浓度为418.7 μmol·L-1, 以模拟受TCE污染的地下水环境.从富集培养液中取150 mL分3份, 在12 000 r·min-1条件下离心20 min, 移去上清液留菌体沉淀.每250 mL密闭厌氧瓶分装100 mL地下水, 50 mL菌体沉淀, 外加混合电子供体中各物质的终浓度均为1 mmol·L-1, 设置3个重复即Y-1、Y-2和Y-3.放于30℃恒温厌氧培养箱内, 于黑暗条件下静置培养, 定期取样测量其中的污染物降解转化情况.
1.5 分析测试方法应用气相色谱GC-2010 Plus(SHIMADZU, 日本)火焰电离(GC-FID)检测器对PCE和TCE及其目标产物cis-1,2-DCE、VC和ETH进行定性定量检测.校准标样在超纯水中制备, 外标法定量, 取1 mL培养液样品加入到含有5 mL盐酸酸化超纯水(pH < 2)的20 mL顶空瓶中, 密封测样.检测方法如下:样品在顶空进样器HS-10(SHIMADZU, 日本)中于80℃恒温22 min;加压平衡后, 顶空气体样品通过气化室注入气相色谱柱(Agilent HP-PLOT/Q+PT, 30 m × 0.53 mm, 40 μm);柱温箱升温程序:初始温度150℃, 保持6 min, 以20℃·min -1升至200℃, 保持8 min;载气为氮气, 柱流速5 mL·min-1.
2 结果与讨论 2.1 氯代乙烯厌氧微生物的富集及还原脱氯特性研究 2.1.1 氯代乙烯的厌氧微生物还原脱氯在实验组1中PCE-T1添加98.3 μmol(水相浓度理论值为616.4 μmol·L-1)PCE, PCE被菌液依次还原脱氯为TCE、cis-1,2-DCE和VC, 并最终脱氯为无毒ETH, 如图 1(a).在第0 d加入PCE后, 其完全还原脱氯为无毒ETH的速率为8.7 µmol·(L·d)-1, 在第72 d和79 d加入等量PCE后还原脱氯速率显著加快, 达到(120.1 ± 4.9)µmol·(L·d)-1.
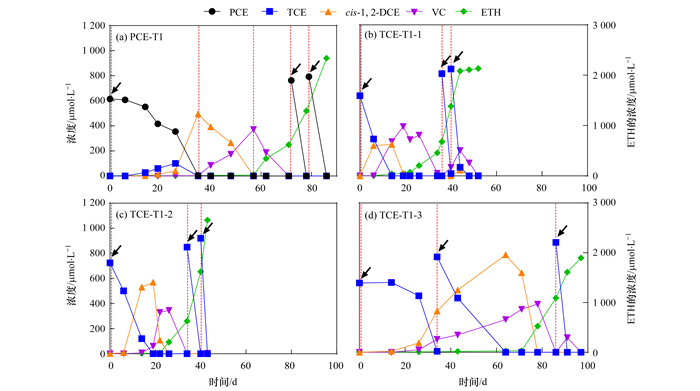
|
(a)富集培养液中PCE的脱氯动态;(b)~(d)富集培养液中TCE的脱氯动态;箭头表示补加98.3 μmol(水相浓度理论值为616.4 μmol·L-1)PCE或111.4 μmol(水相浓度理论值为696.3 μmol·L-1)TCE, 竖直虚线表示提取DNA的时间 图 1 富集培养液中氯代乙烯的脱氯动态 Fig. 1 Dechlorination dynamics of chlorinated ethylene in the enrichment |
实验组2中添加111.4 μmol(水相浓度理论值为696.3 μmol·L-1)TCE, 各实验瓶中TCE逐渐减少同时中间产物cis-1,2-DCE和VC积累增加, 并很快被进一步还原脱氯成无毒ETH, 如图1(b) ~ 1(d).TCE-T1-1和TCE-T1-2在第0 d加入TCE后35 d左右完全还原脱氯成无毒ETH, 还原脱氯速率为(19.5 ± 1.3)µmol·(L·d)-1, 在第36 d和40 d加入等量TCE后还原脱氯速率逐渐加快, 在5 d左右即可完全还原脱氯成无毒ETH, 还原脱氯速率为(172.4 ± 21.8)µmol·(L·d)-1.TCE-T1-3在第0 d添加TCE后34 d还原脱氯成cis-1,2-DCE, 第34 d再次加入等量TCE后86 d完全还原脱氯成无毒ETH, 还原脱氯速率为16.2 µmol·(L·d)-1, 同样, 第86 d再次加入TCE后还原脱氯速率加快, 在11 d即可完全还原脱氯成无毒ETH, 还原脱氯速率为80.3 µmol·(L·d)-1.
Hood等[50]进行的实验室研究中, 包含Dehalococcoides的土著微生物KB-1TM可以使197.9 μmol·L-1的TCE经过31 d完全还原脱氯为ETH, 而563.2 μmol·L-1的TCE经过52 d只能有97%能被脱氯为ETH, 会造成大量的VC的积累.Schaefer等[51]利用SDC-9™菌群使80 μmol·L-1的TCE完全还原脱氯为ETH.本研究中的厌氧微生物菌群W-1同样能够将PCE依次转化为TCE、cis-1,2-DCE和VC, 并最终转化为无毒ETH, 实现氯代乙烯的彻底还原脱氯.616.4 μmol·L-1的PCE最快经过6 d可以完全还原脱氯为ETH, 696.3 μmol·L-1的TCE最快只需4 d可以完全还原脱氯为ETH.W-1与KB-1TM和SDC-9TM两种国际上知名商业化菌剂相比(见表 2), 其还原脱氯的效率远远高于KB-1TM, 而相对于SDC-9TM虽然效率比较低但能还原更高浓度的TCE.
|
|
表 2 与知名菌剂在实验室条件下的脱氯效果比较1) Table 2 Comparison of dechlorination effects of well-known microbial inoculum under laboratory conditions |
2.1.2 氯代乙烯厌氧脱氯富集菌液的微生物种群结构
通过16S rRNA基因扩增子测序, 分析PCE-T1实验瓶中污染物依次脱去氯原子后富集菌液的微生物群落组成变化, 以及TCE-T1-1、TCE-T1-2和TCE-T1-3这3个富集菌液中与初始时刻相比在连续添加TCE后, 其脱去1个氯原子为cis-1,2-DCE及脱去3个氯原子为ETH污染物转化过程相应的微生物群落组成变化.
在PCE-T1富集菌液中的微生物群落结构中有16个属至少在1个样品中相对丰度大于1%, 如图 2(a).此富集菌液中Dehalococcoides和Dehalobacter的相对丰度较高, 在PCE脱去2个氯原子形成cis-1,2-DCE这一过程即Day0 ~ Day36, Dehalococcoides丰度从28.9%降低至1.1%, Dehalobacter丰度从1.9%增长至57.1%, 而随着cis-1,2-DCE脱去氯原子至ETH即Day36 ~ Day72, Dehalococcoides从1.1%增长至53.8%, Dehalobacter从57.1%降低至20.1%.当第二次添加的PCE完全还原脱氯至ETH时即Day79, Dehalococcoides(67.5%)和Dehalobacter(21.4%)两者占比高达88.9%.综上结果表明, 负责PCE还原脱氯的主要菌株为Dehalococcoides和Dehalobacter, 其中Dehalobacter参与PCE至cis-1,2-DCE这一过程.
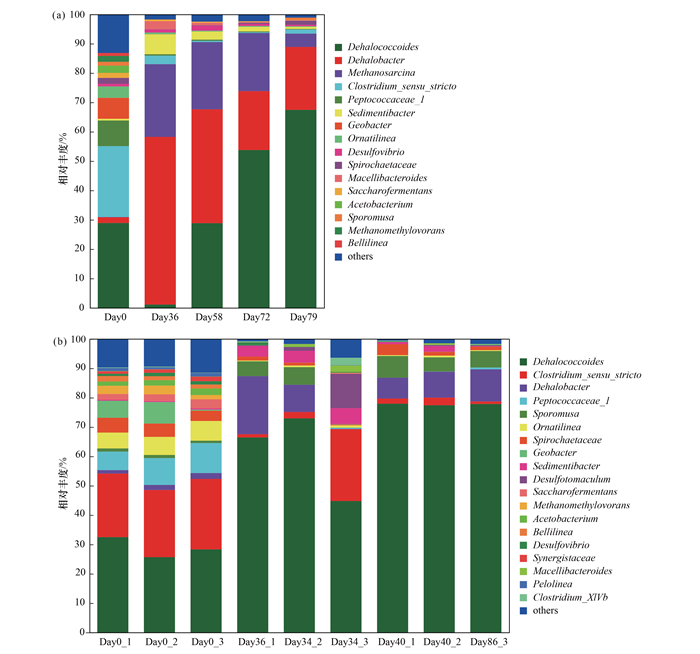
|
(a)PCE为电子受体的富集菌液中微生物群落结构变化;Day0、Day36、Day58和Day72表示PCE-T1初始时刻及依次脱去氯原子后的群落结构;Day79表示第二次喂养PCE完全脱氯为ETH时的群落结构组成;(b)TCE为电子受体的富集菌液中微生物群落结构变化;Day0_1、Day0_2和Day0_3表示TCE-T1-1、TCE-T1-2和TCE-T1-3这3个重复初始时刻的群落结构组成;Day34_1和Day34_2表示TCE-T1-1和TCE-T1-2第一次完全脱氯为ETH时的群落结构组成;Day34_3表示TCE-T1-3第一次完全脱氯为cis-1,2-DCE时的群落结构组成;Day40_1、Day40_2和Day86_3表示3个重复经过两轮喂养TCE完全脱氯为ETH时的群落结构组成 图 2 PCE/TCE厌氧微生物群落结构 Fig. 2 Microbial community structure of PCE and TCE anaerobic microorganisms |
在以TCE作为电子受体的实验组2中的微生物群落结构[图 2(b)]与以PCE作为电子受体的微生物群落结构相似, Dehalococcoides的相对丰度最高.在TCE-T1-1和TCE-T1-2菌液进行两轮TCE完全还原脱氯为ETH这一过程即Day0_1 ~ Day40_1和Day0_2 ~ Day40_2, Dehalococcoides从(29.1 ± 2.4)%增长至(77.7 ± 0.2)%, Dehalobacter从(1.5 ± 0.2)%增长至(8.0 ± 0.6)%.在TCE-T1-3菌液脱去1个氯原子成cis-1,2-DCE即Day0_3 ~ Day34_3, Dehalococcoides与初始时刻相比从28.3%增长至44.8%, Dehalobacter从2.0%降低至0.1%, 第34 d补加111.4 μmol的TCE全部脱氯为ETH这一过程即Day34_3 ~ Day86_3, Dehalococcoides从44.8%增长至77.9%, Dehalobacter从0.1%增长至11.0%.总之, 以TCE为电子受体的富集菌液经过两轮还原脱氯后即Day40_1、Day40_2和Day86_3, Dehalococcoides和Dehalobacter两者占比达(86.7 ± 0.9)%.
无论是以PCE为电子受体还是以TCE为电子受体, 富集菌液经过两轮还原脱氯后都获得具有较高丰度脱卤菌的菌液, 其中Dehalobacter主要参与从PCE至cis-1,2-DCE这一过程的还原脱氯, 而Dehalococcoides负责PCE至ETH整个过程的还原脱氯, 是此过程的主要脱卤菌.有研究表明[52, 53]氯代乙烯的还原脱氯与Dehalococcoides的丰度呈正相关, 虽然在很多地方都发现了Dehalococcoides菌种, 但是它们并不是普遍存在的, 它们的生长量和生长率比较低而且需要驯化的时间比较久, 而获得的W-1菌群中Dehalococcoides和Dehalobacter两者占比最高达88.9%.因此高丰度脱卤菌富集菌群W-1可以高效地完成氯代乙烯的还原脱氯, 能够解决具有更大毒性的低氯代烯烃在环境中积累赋存的问题, 为氯代乙烯彻底还原脱氯为无毒ETH提供了微生物资源, 对受氯代乙烯污染地下水的环境修复具有现实意义.
2.1.3 功能脱卤菌Dehalococcoides和Dehalobacter生长率计算基于16S rRNA基因扩增子测序结果, 为了研究Dehalococcoides和Dehalobacter伴随PCE和TCE脱氯过程的生长情况, 本研究利用qPCR技术对实验瓶(PCE-T1实验瓶第0、36、58、72和79 d;TCE-T1-1实验瓶第0、36和40 d;TCE-T1-2实验瓶第0、34和40 d;TCE-T1-3实验瓶第0、34和86 d)的功能脱卤菌在样品中的基因拷贝数进行绝对定量.
不同培养天数Dehalococcoides的基因拷贝数结果显示(图 3), 第0 ~ 79 d即经过两轮投喂, 共196.6 μmol PCE完全脱氯至ETH这一过程, PCE-T1实验瓶中Dehalococcoides的16S rRNA基因拷贝数从7.3 × 104 copies·mL-1显著增长到1.7 × 109 copies·mL-1, 每释放1 µmol Cl-增加1.7 × 108 copies.同样以TCE为电子受体的3个实验瓶第0 ~ 55 d即经过两轮投喂, 共222.8 μmol TCE完全脱氯至ETH这一过程, Dehalococcoides的16S rRNA基因拷贝数从(3.2 ± 0.5)× 105 copies·mL-1显著增长到(1.6 ± 0.4)× 109 copies·mL-1, 每释放1 µmol Cl-增加(1.9 ± 0.4)× 108 copies.上述结果进一步证明, Dehalococcoides是PCE至ETH整个过程的还原脱卤菌.
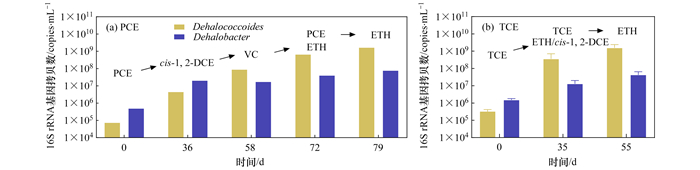
|
(a)PCE-T1富集菌液中Dehalococcoides和Dehalobacter微生物基因拷贝数变化, (b)TCE-T1-1、TCE-T1-2和TCE-T1-3富集菌液中Dehalococcoides和Dehalobacter微生物基因拷贝数变化;横坐标35 d和55 d为3个实验瓶取样时间中间值, ETH/cis-1,2-DCE表示TCE-T1-1和TCE-T1-2实验瓶中111.4 μmol TCE第一次完全脱氯为ETH, TCE-T1-3实验瓶中111.4 μmol TCE第一次完全脱氯为cis-1,2-DCE;箭头表示相应的污染物转化过程 图 3 不同培养天数Dehalococcoides和Dehalobacter的基因拷贝数 Fig. 3 Gene copy numbers of Dehalococcoides and Dehalobacter on different culture days |
对于不同培养天数Dehalobacter的基因拷贝数结果(图 3), PCE-T1实验瓶中, 第0 ~ 36 d即98.3 μmol PCE完全脱去2个氯原子成cis-1,2-DCE时, Dehalobacter的16S rRNA基因拷贝数5.0 × 105 copies·mL-1增长至2.1 × 107 copies·mL-1, 每释放1 µmol Cl-增加1.7 × 107 copies.而第36 ~ 72 d即cis-1,2-DCE再脱去氯原子至ETH时, Dehalobacter增加缓慢, 这一结果表明Dehalobacter只参与从TCE至cis-1,2-DCE这一过程的还原脱氯.以TCE为电子受体的3个实验瓶经过两轮投喂共222.8 μmol TCE即第0 ~ 55 d, Dehalobacter的16S rRNA基因拷贝数从(1.5 ± 0.1)× 106 copies·mL-1增长到(4.2 ± 1.1)× 107 copies·mL-1, 每释放1 µmol Cl-增加(4.9 ± 0.1)× 107 copies.
2.1.4 Dehalococcoides菌株鉴定利用核酸电泳扩增与Sanger测序本研究获得了两段大小分别为1 223 bp与734 bp长度的核酸序列, 最终拼接得到1 398 bp的片段, 接近16S rRNA基因序列全长, 即获取了Dehalococcoides较完整的16S rRNA基因序列.EzBioCloud数据库中Blast结果和系统发育树(图 4)显示, 本富集液中菌株Dehalococcoides sp. LWT1 16S rRNA与Maymó-Gatell等[25]在厌氧条件下分离出的脱卤拟球菌D. mccartyi strain 195 16S rRNA基因相似度达100%.
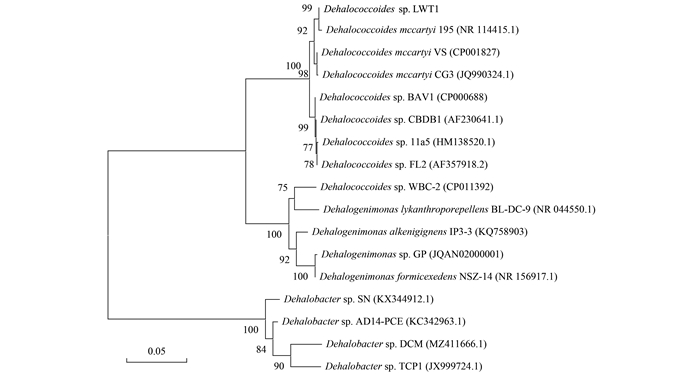
|
图 4 基于TCE还原脱氯微生物16S rRNA序列的系统发育树 Fig. 4 The 16S rRNA phylogenetic tree of TCE reductive dechlorination bacteria |
微生物群落是微生物菌种之间采用多种方式相互作用从而形成复杂的网络生态系统.有研究证明, OHRB的共存和相互作用能够使得有机卤化物以更快的速率逐步转化为无毒状态.例如, Ziv-El等[54]研究中Geobacter和Dehalococcoides混菌及Rouzeau-Szynalski等[55]研究中Desulfitobacterium和Dehalococcoides混菌可彻底使PCE和TCE还原脱氯为无毒ETH[48].本研究结果表明, Dehalococcoides和Dehalobacter是PCE至ETH这一过程主要的脱卤菌, 可以使PCE和TCE完全还原脱氯为无毒ETH, 其中Dehalobacter只参与PCE至cis-1,2-DCE这一过程的还原脱氯, 随着Dehalococcoides的生长量逐渐增加, Dehalococcoides成为主要的优势菌种, 因此Dehalobacter在后期会受到抑制从而使Dehalococcoides成为PCE至ETH整个过程主要的脱卤菌, Lai等[56]和Chen等[57]的研究中也证明了这一点.Dehalococcoides和Dehalobacter两者存在竞争机制, 并且在某种程度上这种竞争机制能够促进有机卤呼吸代谢, 从而使Dehalococcoides和Dehalobacter共同生长, 氯代乙烯快速脱氯.通过分析并阐明物种间的相互作用关系, 对于利用生物修复污染场地、实现有机卤化物完全解毒的终极目标具有重大意义.
2.2 菌群W-1在地下水污染环境的应用添加41.9 μmol(水相浓度理论值为418.7 μmol·L-1)TCE至实验瓶Y-1、Y-2和Y-3中, 各实验瓶中TCE逐渐减少同时中间产物cis-1,2-DCE、VC和无毒ETH动态变化如图 5.经过28 d TCE还原脱氯成无毒ETH, 产ETH量为(69.2 ± 9.8)%, 完全还原脱氯速率为(10.3 ± 1.5)µmol·(L·d)-1.
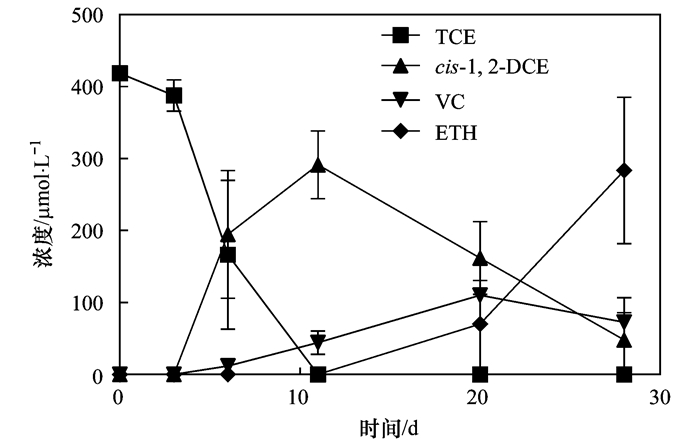
|
图 5 菌群W-1在地下水污染环境中的脱氯动态 Fig. 5 Dechlorination dynamics of bacteriophage W-1 in groundwater contaminated environments |
Hood等[50]进行的应用实验中, 包含Dehalococcoides 的土著微生物KB-1TM 使1 200 μmol·L-1的TCE迅速脱氯至VC, 然而经过5个月停滞后VC脱氯至ETH.Schaefer等[51]利用SDC-9TM菌群进行的土柱实验中120 μmol·L-1的DCE经过62 d左右完全脱氯至ETH.而本研究中的厌氧微生物菌群W-1进行的应用实验中, 418.7 μmol·L-1的TCE经过28 d产生大量ETH, 脱氯速率相较于菌剂KB-1TM和SDC-9TM是比较高的.依据前期实验室富集培养可得, 初期主要脱氯菌群较少, 生长速率较慢, 后期随脱氯菌群的增加, Dehalococcoides sp. LWT1会逐渐成为优势菌种, 脱氯速率会大大加快至最终实现TCE的彻底脱毒.研究和发掘TCE还原脱氯菌剂并应用于实际受污染的地下水环境, 为地下水污染地原位修复提供重要菌株资源具有重大意义.
3 结论(1)通过厌氧富集培养, 获得了一份将PCE / TCE厌氧还原脱氯至无毒ETH的高效厌氧菌剂W-1, PCE、TCE的脱氯速率分别是(120.1 ± 4.9)µmol·(L·d)-1和(172.4 ± 21.8)µmol·(L·d)-1.
(2)基于16S rRNA基因扩增子测序、qPCR技术及Sanger测序, 刻画了菌剂W-1的厌氧微生物菌群结构, 明确了Dehalococcoides和Dehalobacter是主要脱卤功能菌.
(3)将菌剂W-1应用于受TCE污染的地下水, 可实现TCE厌氧脱氯至无毒ETH的过程, 可为受氯代乙烯污染地下水开展原位厌氧微生物修复提供重要的微生物资源.
致谢: 感谢中国科学院沈阳应用生态研究所严俊博士为qPCR提供质粒.
| [1] | Deng J, Wu F, Gao S X, et al. Self-activated Ni(OH)2 cathode for complete electrochemical reduction of trichloroethylene to ethane in low-conductivity groundwater[J]. Applied Catalysis B: Environmental, 2022, 309. DOI:10.1016/j.apcatb.2022.121258 |
| [2] | Beamer P I, Luik C E, Abrell L, et al. Concentration of trichloroethylene in breast milk and household water from nogales, Arizona[J]. Environmental Science & Technology, 2012, 46(16): 9055-9061. |
| [3] | Zhang X F, Li H Q, Liu L F, et al. Exergetic and exergoeconomic assessment of a novel CHP system integrating biomass partial gasification with ground source heat pump[J]. Energy Conversion and Management, 2018, 156: 666-679. DOI:10.1016/j.enconman.2017.11.075 |
| [4] | Xie H B, Peng J L, Zhang X S, et al. Effects of mitochondrial reactive oxygen species-induced NLRP3 inflammasome activation on trichloroethylene-mediated kidney immune injury[J]. Ecotoxicology and Environmental Safety, 2022, 244. DOI:10.1016/j.ecoenv.2022.114067 |
| [5] | Mahato J K, Gupta S K. Advanced oxidation of trihalomethane (THMs) precursors and season-wise multi-pathway human carcinogenic risk assessment in Indian drinking water supplies[J]. Process Safety and Environmental Protection, 2022, 159: 996-1007. DOI:10.1016/j.psep.2022.01.066 |
| [6] | Mitra S, Gupta S K. Pilot-scale treatment of a trichloethylene rich synthetic wastewater in anaerobic hybrid reactor, with morphological study of the sludge granules[J]. Clean Technologies and Environmental Policy, 2014, 16(5): 947-956. DOI:10.1007/s10098-013-0695-2 |
| [7] |
高秋生, 赵永辉, 焦立新, 等. 白洋淀水体挥发性有机物污染特征与风险评价[J]. 环境科学, 2018, 39(5): 2048-2055. Gao Q S, Zhao Y H, Jiao L X, et al. Pollution characteristics and health risk assessment of volatile organic compounds in Baiyangdian Lake[J]. Environmental Science, 2018, 39(5): 2048-2055. |
| [8] | Huang B B, Lei C, Wei C H, et al. Chlorinated volatile organic compounds (Cl-VOCs) in environment—sources, potential human health impacts, and current remediation technologies[J]. Environment International, 2014, 71: 118-138. DOI:10.1016/j.envint.2014.06.013 |
| [9] | Moran M J, Zogorski J S, Squillace P J. Chlorinated solvents in groundwater of the United States[J]. Environmental Science & Technology, 2007, 41(1): 74-81. |
| [10] | Fang L, Norris C, Johnson K, et al. Toxic volatile organic compounds in 20 homes in Shanghai: concentrations, inhalation health risks, and the impacts of household air cleaning[J]. Building and Environment, 2019, 157: 309-318. DOI:10.1016/j.buildenv.2019.04.047 |
| [11] | Mo Z W, Lu S H, Shao M. Volatile organic compound (VOC) emissions and health risk assessment in paint and coatings industry in the Yangtze River Delta, China[J]. Environmental Pollution, 2021, 269. DOI:10.1016/j.envpol.2020.115740 |
| [12] | Boyes W K, Bercegeay M, Oshiro W M, et al. Acute perchloroethylene exposure alters rat visual-evoked potentials in relation to brain concentrations[J]. Toxicological Sciences, 2009, 108(1): 159-172. DOI:10.1093/toxsci/kfn265 |
| [13] | Ghobakhloo S, Khoshakhlagh A H, Morais S, et al. Exposure to volatile organic compounds in paint production plants: levels and potential human health risks[J]. Toxics, 2023, 11(2). DOI:10.3390/toxics11020111 |
| [14] | Hu R Y, Liu G J, Zhang H, et al. Levels, characteristics and health risk assessment of VOCs in different functional zones of Hefei[J]. Ecotoxicology and Environmental Safety, 2018, 160: 301-307. DOI:10.1016/j.ecoenv.2018.05.056 |
| [15] | Tsai W T. A survey on toxic volatile organic compounds (VOCs): toxicological profiles, health exposure risks, and regulatory strategies for mitigating emissions from stationary sources in Taiwan[J]. Atmosphere, 2023, 14(2). DOI:10.3390/atmos14020242 |
| [16] | Wang H, Banerjee N, Wang G D, et al. Autophagy dysregulation in trichloroethene-mediated inflammation and autoimmune response[J]. Toxicology, 2023, 487. DOI:10.1016/j.tox.2023.153468 |
| [17] | Yuan T H, Chen J L, Shie R H, et al. Liver fibrosis associated with potential vinyl chloride and ethylene dichloride exposure from the petrochemical industry[J]. Science of the Total Environment, 2020, 739. DOI:10.1016/j.scitotenv.2020.139920 |
| [18] | Niño De Guzmán G T, Hapeman C J, Millner P D, et al. Presence of organohalide-respiring bacteria in and around a permeable reactive barrier at a trichloroethylene-contaminated superfund site[J]. Environmental Pollution, 2018, 243: 766-776. DOI:10.1016/j.envpol.2018.08.095 |
| [19] | Gafni A, Siebner H, Bernstein A. Potential for co-metabolic oxidation of TCE and evidence for its occurrence in a large-scale aquifer survey[J]. Water Research, 2020, 171. DOI:10.1016/j.watres.2019.115431 |
| [20] |
卢晓霞, 李广贺, 张旭, 等. 不同氧化还原条件下氯乙烯的微生物脱氯[J]. 环境科学, 2002, 23(2): 29-33. Lu X X, Li G H, Zhang X, et al. Dechlorination of chlorinated ethenes under different redox conditions[J]. Environmental Science, 2002, 23(2): 29-33. |
| [21] |
李惠娣, 杨琦, 尚海涛. 甲醇为共代谢基质时四氯乙烯的厌氧生物降解[J]. 环境科学, 2004, 25(3): 84-88. Li H D, Yang Q, Shang H T. Anaerobic biodegradation of tetrachlorothylene with methanol as co-metabolism substrate[J]. Environmental Science, 2004, 25(3): 84-88. DOI:10.3321/j.issn:0250-3301.2004.03.018 |
| [22] | Ebrahimbabaie P, Pichtel J. Biotechnology and nanotechnology for remediation of chlorinated volatile organic compounds: current perspectives[J]. Environmental Science and Pollution Research, 2021, 28(7): 7710-7741. DOI:10.1007/s11356-020-11598-y |
| [23] |
何江涛, 李烨, 刘石, 等. 浅层地下水氯代烃污染的天然生物降解[J]. 环境科学, 2005, 26(2): 121-125. He J T, Li Y, Liu S, et al. Chlorinate solvents natural biodegradation in shallow groundwater[J]. Environmental Science, 2005, 26(2): 121-125. DOI:10.3321/j.issn:0250-3301.2005.02.025 |
| [24] | Cheng Y H, Chang S C, Lai Y L, et al. Microbiome reengineering by four environmental factors for the rapid biodegradation of trichloroethylene[J]. Journal of Environmental Management, 2023, 326. DOI:10.1016/j.jenvman.2022.116658 |
| [25] | Maymó-Gatell X, Chien Y T, Gossett J M, et al. Isolation of a bacterium that reductively dechlorinates tetrachloroethene to ethene[J]. Science, 1997, 276(5318): 1568-1571. DOI:10.1126/science.276.5318.1568 |
| [26] | He J Z, Ritalahti K M, Yang K L, et al. Detoxification of vinyl chloride to ethene coupled to growth of an anaerobic bacterium[J]. Nature, 2003, 424(6944): 62-65. DOI:10.1038/nature01717 |
| [27] | Holliger C, Hahn D, Harmsen H, et al. Dehalobacter restrictus gen. nov. and sp. nov., a strictly anaerobic bacterium that reductively dechlorinates tetra- and trichloroethene in an anaerobic respiration[J]. Archives of Microbiology, 1998, 169(4): 313-321. DOI:10.1007/s002030050577 |
| [28] | Rupakula A, Lu Y, Kruse T, et al. Functional genomics of corrinoid starvation in the organohalide-respiring bacterium Dehalobacter restrictus strain PER-K23[J]. Frontiers in Microbiology, 2014, 5. DOI:10.3389/fmicb.2014.00751 |
| [29] | Chen G, Kara Murdoch F, Xie Y C, et al. Dehalogenation of chlorinated ethenes to ethene by a novel isolate, "Candidatus Dehalogenimonas etheniformans"[J]. Applied and Environmental Microbiology, 2022, 88(12). DOI:10.1128/aem.00443-22 |
| [30] | Goris T, Schubert T, Gadkari J, et al. Insights into organohalide respiration and the versatile catabolism of Sulfurospirillum multivorans gained from comparative genomics and physiological studies[J]. Environmental Microbiology, 2014, 16(11): 3562-3580. DOI:10.1111/1462-2920.12589 |
| [31] |
李晓翠, 李秀颖, 宋玉芳, 等. 有机卤呼吸微生物菌群营养交互的作用机制[J]. 微生物学报, 2022, 62(6): 2226-2248. Li X C, Li X Y, Song Y F, et al. Mechanisms of metabolic interactions in microbial communities harboring organohalide-respiring bacteria[J]. Acta Microbiologica Sinica, 2022, 62(6): 2226-2248. |
| [32] | Chen W Y, Wu J H. Microbiome composition resulting from different substrates influences trichloroethene dechlorination performance[J]. Journal of Environmental Management, 2022, 303. DOI:10.1016/j.jenvman.2021.114145 |
| [33] | Underwood J C, Akob D M, Lorah M M, et al. Microbial community response to a bioaugmentation test to degrade trichloroethylene in a fractured rock aquifer, Trenton, N.J[J]. FEMS Microbiology Ecology, 2022, 98(7). DOI:10.1093/femsec/fiac077 |
| [34] |
李姜维, 杨晓永, 胡安谊, 等. 苯甲酸盐厌氧驯化体系中三氯乙烯的还原脱氯特性[J]. 环境科学, 2015, 36(10): 3756-3763. Li W J, Yang X Y, Hu A Y, et al. Reductive dechlorination of trichloroethylene by benzoate-enriched anaerobic cultures[J]. Environmental Science, 2015, 36(10): 3756-3763. |
| [35] | Futagami T, Goto M, Furukawa K. Biochemical and genetic bases of dehalorespiration[J]. The Chemical Record, 2008, 8(1): 1-12. DOI:10.1002/tcr.20134 |
| [36] | Li Y Q, Cai M J, Ji B B, et al. Pyridinic nitrogen enables dechlorination of trichloroethylene to acetylene by green rust: performance, mechanism and applications[J]. Science of the Total Environment, 2022, 824. DOI:10.1016/j.scitotenv.2022.153825 |
| [37] | Tiehm A, Schmidt K R. Sequential anaerobic/aerobic biodegradation of chloroethenes—aspects of field application[J]. Current Opinion in Biotechnology, 2011, 22(3): 415-421. DOI:10.1016/j.copbio.2011.02.003 |
| [38] | Yang X M, Zhang C, Liu F, et al. Groundwater geochemical constituents controlling the reductive dechlorination of TCE by nZVI: evidence from diverse anaerobic corrosion mechanisms of nZVI[J]. Chemosphere, 2021, 262. DOI:10.1016/j.chemosphere.2020.127707 |
| [39] | Suyama A, Iwakiri R, Kai K, et al. Isolation and characterization of Desulfitobacterium sp. strain Y51 capable of efficient dehalogenation of tetrachloroethene and polychloroethanes[J]. Bioscience, Biotechnology, and Biochemistry, 2001, 65(7): 1474-1481. DOI:10.1271/bbb.65.1474 |
| [40] | Scholz-Muramatsu H, Neumann A, Meßmer M, et al. Isolation and characterization of Dehalospirillum multivorans gen. nov., sp. nov., a tetrachloroethene-utilizing, strictly anaerobic bacterium[J]. Archives of Microbiology, 1995, 163(1): 48-56. DOI:10.1007/BF00262203 |
| [41] | Bælum J, Scheutz C, Chambon J C, et al. The impact of bioaugmentation on dechlorination kinetics and on microbial dechlorinating communities in subsurface clay till[J]. Environmental Pollution, 2014, 186: 149-157. DOI:10.1016/j.envpol.2013.11.013 |
| [42] | Yang Y, Higgins S A, Yan J, et al. Grape pomace compost harbors organohalide-respiring Dehalogenimonas species with novel reductive dehalogenase genes[J]. The ISME Journal, 2017, 11(12): 2767-2780. DOI:10.1038/ismej.2017.127 |
| [43] | Edwards E A, Grbić-Galić D. Anaerobic degradation of toluene and o-xylene by a methanogenic consortium[J]. Applied and Environmental Microbiology, 1994, 60(1): 313-322. DOI:10.1128/aem.60.1.313-322.1994 |
| [44] | Callahan B J, McMurdie P J, Rosen M J, et al. DADA2: high-resolution sample inference from illumina amplicon data[J]. Nature Methods, 2016, 13(7): 581-583. DOI:10.1038/nmeth.3869 |
| [45] | Thuronyi B W, Koblan L W, Levy J M, et al. Publisher correction: continuous evolution of base editors with expanded target compatibility and improved activity[J]. Nature Biotechnology, 2019, 37(9): 1091. |
| [46] | Quast C, Pruesse E, Yilmaz P, et al. The SILVA ribosomal RNA gene database project: improved data processing and web-based tools[J]. Nucleic Acids Research, 2012, 41(D1): D590-D596. DOI:10.1093/nar/gks1219 |
| [47] | He J Z, Ritalahti K M, Aiello M R, et al. Complete detoxification of vinyl chloride by an anaerobic enrichment culture and identification of the reductively dechlorinating population as a Dehalococcoides species[J]. Applied and Environmental Microbiology, 2003, 69(5): 996-1003. |
| [48] | Ritalahti K M, Amos B K, Sung Y, et al. Quantitative PCR targeting 16S rRNA and reductive dehalogenase genes simultaneously monitors multiple Dehalococcoides strains[J]. Applied and Environmental Microbiology, 2006, 72(4): 2765-2774. DOI:10.1128/AEM.72.4.2765-2774.2006 |
| [49] | Yu J, Zhou X F, Yang S J, et al. Design and application of specific 16S rDNA-targeted primers for assessing endophytic diversity in Dendrobium officinale using nested PCR-DGGE[J]. Applied Microbiology and Biotechnology, 2013, 97(22): 9825-9836. DOI:10.1007/s00253-013-5294-y |
| [50] | Hood E D, Major D W, Quinn J W, et al. Demonstration of enhanced bioremediation in a TCE source area at Launch Complex 34, Cape Canaveral Air Force Station[J]. Groundwater Monitoring & Remediation, 2008, 28(2): 98-107. |
| [51] | Schaefer C E, Condee C W, Vainberg S, et al. Bioaugmentation for chlorinated ethenes using Dehalococcoides sp.: comparison between batch and column experiments[J]. Chemosphere, 2009, 75(2): 141-148. |
| [52] | Pham H, Boon N, Marzorati M, et al. Enhanced removal of 1, 2-dichloroethane by anodophilic microbial consortia[J]. Water Research, 2009, 43(11): 2936-2946. |
| [53] | Grostern A, Edwards E A. Characterization of a Dehalobacter coculture that dechlorinates 1, 2-dichloroethane to ethene and identification of the putative reductive dehalogenase gene[J]. Applied and Environmental Microbiology, 2009, 75(9): 2684-2693. DOI:10.1128/AEM.02037-08 |
| [54] | Ziv-El M, Popat S C, Parameswaran P, et al. Using electron balances and molecular techniques to assess trichoroethene-induced shifts to a dechlorinating microbial community[J]. Biotechnology and Bioengineering, 2012, 109(9): 2230-2239. DOI:10.1002/bit.24504 |
| [55] | Rouzeau-Szynalski K, Maillard J, Holliger C. Frequent concomitant presence of Desulfitobacterium spp. and "Dehalococcoides" spp. in chloroethene-dechlorinating microbial communities[J]. Applied Microbiology and Biotechnology, 2011, 90(1): 361-368. |
| [56] | Lai Y, Becker J G. Compounded effects of chlorinated ethene inhibition on ecological interactions and population abundance in a Dehalococcoides - Dehalobacter coculture[J]. Environmental Science & Technology, 2013, 47(3): 1518-1525. |
| [57] | Chen K Z, Liu Z F, Wang X M, et al. Enhancement of perchloroethene dechlorination by a mixed dechlorinating culture via magnetic nanoparticle-mediated isolation method[J]. Science of the Total Environment, 2021, 786. DOI:10.1016/j.scitotenv.2021.147421 |
 2024, Vol. 45
2024, Vol. 45


