微塑料(microplastics, MPs)指环境中尺寸在5 mm以下的塑料微粒[1, 2].在水环境中MPs可能更容易被生物摄食[3], 也更容易成为各类污染物富集载体, 如重金属、有机污染物和抗生素等[4~8].同时, MPs还可以成为微生物的定植载体, 从而形成“塑料际”(plastisphere)这一特殊生态位[9, 10].有研究发现MPs能加快微生物在水相与沉积物相中的交换, 改变水环境中微生物组成结构[11].
由于抗生素的广泛使用, 抗生素抗性基因(antibiotic resistance genes, ARGs)污染也日益引起关注.MPs在富集微生物的过程中, 一些携带ARGs的致病菌也可能定殖在其表面, 使得ARGs在MPs表面富集[12~14].有研究表明, MPs对于环境中的致病菌以及ARGs可能存在选择性富集, 相对于天然基质, MPs上往往检测到更多种类以及更高丰度的ARGs.Wu等[11]通过实验室培养发现, 相对于表层水和沉积物, 假单胞菌(Pseudomonas)和芽孢杆菌(Bacillus)等机会致病菌在微塑料上占比更高.Wang等[15]将聚乙烯微塑料(PE)室内培养于采集的河流、河口和海洋表层水中, 并在水中添加不同种抗生素, 结果发现培养在含抗生素水体中的微塑料表面各类ARGs丰度明显升高, 并且还出现了sulA/folP-01和tetA等新的ARGs.这可能是由于MPs上细菌群落之间的距离相对于环境介质中更小, 有利于水平基因转移的发生, 细菌之间基因交换频繁促进了新的ARGs产生.而周昕原等[16]研究发现MPs的存在也会显著增加河水中ARGs绝对丰度, 并通过水平基因转移机制影响河水中ARGs的赋存与演变.由于小粒径微塑料更容易被水生生物摄食, 因此增加了ARGs与耐药菌进入食物链的风险, 从而威胁人类健康[17].
受人类活动影响, 河口环境是MPs和ARGs的重灾区, 我国以及世界范围的河口区都检测到了高丰度的MPs和ARGs污染[18~22], 以上污染物往往经由河口输送至海洋, 威胁着海洋生物和人类生存, 因此河口区MPs和ARGs复合污染问题值得关注.本文以福建省九龙江口为研究区域, 调查了该河口MPs和ARGs丰度的分布现状, 并对二者分布的相关性进行分析, 以期为河口以及近海MPs和ARGs的复合污染评估提供科学依据.
1 材料与方法 1.1 现场样品采集九龙江是福建省内第二大河流, 流域涵盖12个县(市、区)以及全省近20%的人口.干流和支流两岸兴建的工厂、禽畜和水产养殖业较为发达, 存在着多种点源和面源污染.2021年1月, 本研究采集九龙江河口区7个采样点的表层水、亚表层水和表层沉积物.各采样点详细信息见表 1.
|
|
表 1 各采样点坐标及样品采集信息1) Table 1 Coordinates of each sampling point and sample collection information |
用于测定MPs的样品采集:利用卡盖式采集器采集水样, 每个采样点取10 L, 现场用孔径10 μm的聚碳酸酯滤膜(Millipore, 美国)过滤, 滤膜保存于离心管中; 用箱式采泥器采集表层沉积物, 每个采样点采集约3 kg, 沉积物混匀后按1 kg分装3份, 作为平行样.
用于测定ARGs的样品采集:利用卡盖式采集器采集水样各1 L, 现场用0.22 μm的聚碳酸酯滤膜(Millipore, 美国)过滤.滤膜用无菌离心管装取, 干冰保存下运回实验室; 利用箱式采泥法采集沉积物, 置于50 mL无菌离心管, 干冰保存下运回实验室.实验室内, 所有样品于-80℃保存至分析.
1.2 微塑料分离和测定为减少样品受外来微塑料污染, 实验过程全程穿戴棉质实验服和丁腈手套.分离过程用到的容器尽量采用玻璃材质, 且实验前均用反渗透水和超纯水(18.2 MΩ ·cm)冲洗2~3次.所有实验溶液都经0.22 μm PC滤膜过滤后使用.样品过滤时用锡箔纸密封住过滤装置的开口.
水样中MPs的分离:将各采样点水样过滤后的多张滤膜分成3份.滤膜加入30%过氧化氢, 60℃水浴恒温消解48 h.消解完全后, 用孔径为10 μm的聚碳酸酯滤膜过滤收集微塑料.
沉积物样中MPs分离:沉积物样品在烘箱中40℃烘干至恒重.取100 g干燥的沉积物于洁净的玻璃烧杯中, 加入NaCl饱和盐水搅拌均匀, 静置12 h, 取上清液用10 μm滤膜过滤, 滤液倒回玻璃烧杯, 搅拌均匀后继续静置浮选.重复该过程2~3次.全部滤膜置于玻璃烧杯中, 加入30%过氧化氢至完全没过滤膜, 60℃水浴恒温消解48 h.用10 μm的聚碳酸酯滤膜过滤, 滤膜在玻璃培养皿中自然干燥.
使用体视显微镜(SMZ-1500, Nikon, 日本)观察挑选疑似MPs的颗粒样品.利用傅里叶红外光谱仪(Nicolet iN10TM, Thermo Fisher Scientific, 美国)的透射模式分析颗粒材质.样品的扫描谱图结果与HR Spectra Polymers and Plastics标准谱库进行比对, 只有匹配度在70%以上才能确定MPs的聚合物类型.对已经确认为MPs的样品, 再次在体视显微镜下观察, 测定其长度或直径和记录颜色等特征.本研究采集到的MPs形状主要为颗粒、纤维和薄膜这3种.在粒径测定中, 纤维状塑料测定实际长度, 颗粒或者薄膜状测定最大直径.
1.3 抗生素抗性基因测定利用Dneasy Powersoil DNA Isolation Kit试剂盒提取样品DNA, 提取步骤按照试剂盒说明书进行.提取的DNA样品通过微量核酸蛋白分析仪(Nanodrop2000, Thermo Scientific)对其纯度与浓度进行测定与检验.
测定的抗生素抗性基因包括:2种磺胺类抗性基因 sul1 和 sul2 , 3种四环素类抗性基因tetA、tetC和tetG, 1种喹诺酮类抗性基因qnrA, 1种大环内酯类抗性基因ermB, 1种氯霉素类抗性基因cmlA, 一类整合子基因 intI1 , 以16S rRNA基因为内参.本文引物序列及退火温度的设计主要参考文献[23~25], 引物合成均由上海生工生物工程有限公司完成.
利用实时荧光定量q-PCR仪(CFX96 Touch, Bio-Rad, 美国)进行定量.q-PCR定量所用试剂盒为AG SYBR Green Pro Taq HS预混型试剂盒, 反应体系的配置按照说明书进行.q-PCR反应程序设置:先95℃预变性加热30 s, 再95℃变性加热5 s, 退火30 s后72℃延伸30 s, 每次扩增循环40次.
ARGs丰度计算方法:绝对丰度指的是单位样品中ARGs的基因拷贝数(水样中为copies ·L-1, 沉积物中为copies ·g-1), 由式(1)计算得来; 相对丰度指的是所有细菌中抗生素耐药菌所占比例, 是ARGs绝对丰度和16S rRNA绝对丰度的比值, 由式(2)计算得来.
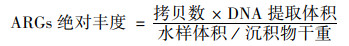
|
(1) |

|
(2) |
原始数据通过Excel 2019初步处理, 后续相关性、显著性分析、柱状图和相关性可视化等通过Origin和R-4.0软件完成.
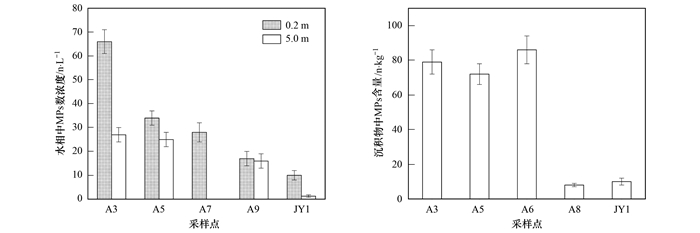
2 结果与讨论 2.1 九龙江河口MPs污染分布特征 2.1.1 MPs含量分布特征九龙江口各采样点水相与沉积物中MPs含量分布如图 1所示.沉积物样品中各采样点MPs含量范围为8~85 n ·kg-1, 水样中数浓度范围为2~66 n ·L-1.水相中MPs数浓度水平与我国其他河口相当[长江口(4.137±2.461 n ·L-1), 珠江口(7.85~10.95 n ·L-1)][18, 26], 但沉积物MPs含量要明显低于珠江口(80~9 579 n ·kg-1)[18].水相中, MPs数浓度的分布呈现沿河流向入海口位置递减的趋势.Haddout等[27]认为河口区MPs污染的分布受人类活动影响显著, 靠近排污口及其下游位置数浓度较高.而本次采样各采样点附近没有发现明显的排污点, 因而MPs数浓度的分布可能主要受海水稀释作用的影响.沉积物中MPs主要集中在上游采样点A3、A5和A6, 而靠近出海口的A8和JY1采样点MPs数浓度显著下降.可能是由于上游水流平缓, 塑料颗粒在水力传输过程中逐渐沉降到沉积物中, 而入海口位置风浪强、水力扰动较大且海域较为宽广, 所以采集到的MPs样品数量偏低.此外, A8采样点的沉积物为沙质, 赋存微塑料MPs的能力相对较差.
|
图 1 九龙江河口水相和沉积物中MPs含量 Fig. 1 Microplastics in the water and sediment of the Jiulong River estuary |
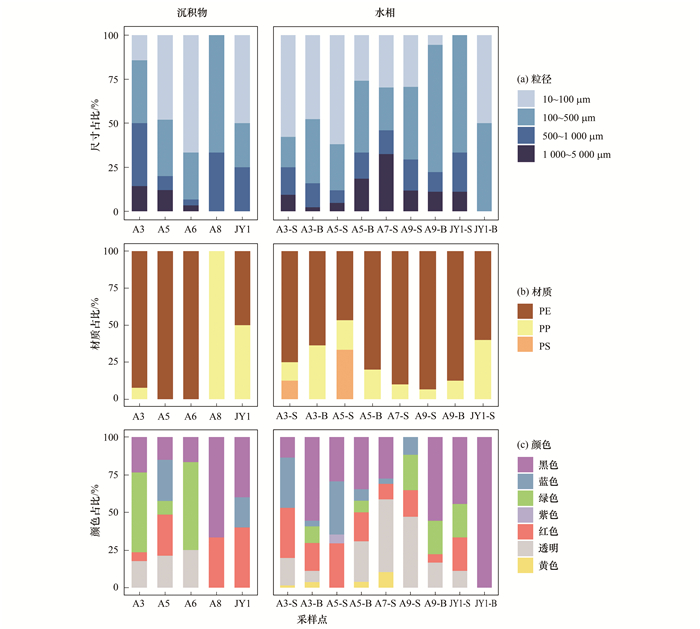
九龙江口的MPs性质分布特征如图 2所示.图 2(a)为样品中MPs的粒径分布.本研究检测的MPs粒径范围为10~5 000 μm, 可分成10~100、100~500、500~1 000和1 000~5 000 μm这4个等级.由结果可知, 测定的MPs粒径分布主要集中在10~1 000 μm范围, 水体中占比达到88.1%, 沉积物中占比达到92%.其他研究也发现我国流域水体中检测到的MPs以小粒径为主, 如在长江口、东海和太湖的表层水中的MPs粒径50~1 000 μm占主要部分[28, 29].总体来看, 九龙江河口沉积物中小粒径MPs占比高于水样.沉积物中大粒径MPs数量较少, 可能是因为大粒径MPs在传输过程中更容易受到风浪冲击, 使其加速破碎为小颗粒的MPs, 以及在沉积物浮选过程中可能存在一定的损耗.
|
图 2 MPs粒径、材质和颜色的分布特征 Fig. 2 Distribution characteristics of particle size, material, and color of microplastics |
图 2(b)为样品中MPs的材质分布.本次样品浮选收集到的样品中, MPs的材质主要是PP、PE和PS, 其中PP和PE的检出率最高, 水体中PP为6% ~36%, PE为60% ~93%; 沉积物中PE检出率为94%.
图 2(c)为样品中MPs的颜色分布.观察到的颜色有红、蓝、黑、黄、绿、紫色和透明.所有MPs样品中, 黑色和透明占比最高, 分别达到30.5%和22.3%.水样中MPs的颜色种类较沉积物中丰富, 而沉积物中主要为黑色和透明色.Zuo等[28]发现在珠江口红树林沉积物中黑色和透明色的MPs占主要部分, 这可能是由于沉降到沉积物中的MPs大多都经历过长期的日光照射、水力和风力的作用, 年久褪色所致[30].
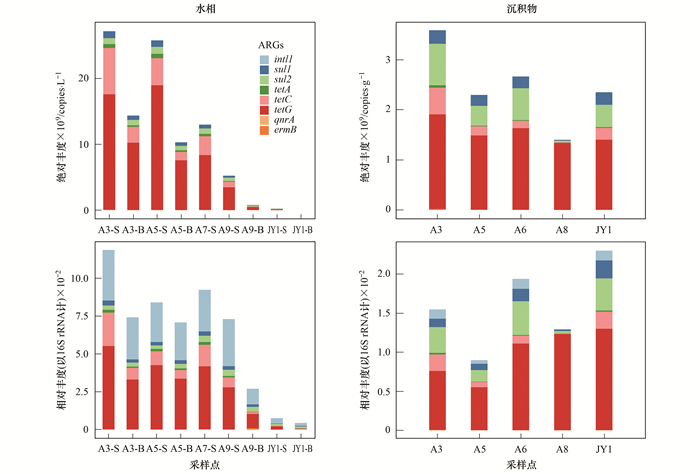
2.2 九龙江河口ARGs污染分布特征九龙江口水样与沉积物中ARGs绝对丰度与相对丰度分布如图 3所示.总体来看, 九龙江口优势ARGs主要为四环素类抗性基因tetC和tetG. tetC和tetG在水样中的绝对丰度分别为3.52×104~7.02×109copies ·L-1和3.28×104~1.89×1010 copies ·L-1, 沉积物中绝对丰度分别为1.02×107~5.34×108和1.34×109~1.89×109 copies ·g-1, 而 sul1、sul2、qnrA、cmlA和ermB基因丰度都较低.这与朱永官等[31]在九龙江流域沉积物中测得的抗性基因种类结果相一致, 四环素类抗性基因为优势基因.四环素是目前水产养殖业中普遍使用的抗生素, 这可能是四环素类ARGs普遍较高的重要原因.
|
图 3 九龙江口水相和沉积物中ARGs丰度 Fig. 3 Abundance of ARGs in water and sediment samples |
水体中检测到ARGs的相对丰度范围在4.46×10-6~1.19×10-4之间, 沉积物中ARGs相对丰度范围在8.98×10-3~2.29×10-2之间.其中, A8采样点由于沉积物为砂质而其他采样点为黏质沉积物, 因而测得的ARGs丰度较其他采样点低, 这是由于砂质沉积物结构松散且营养物含量较低, 不适宜于微生物生存, 而黏质的沉积物对于ARGs有较强的吸附作用.沉积物中ARGs相对丰度总体上比水体中显著高出1~3个数量级.何基兵等[32]检测了九龙江口15个表层水样品和6个沉积物样品的ARGs, 也发现沉积物中ARGs的检出率显著高于表层水.沉积物环境较稳定, 营养较水体丰富, 因此微生物种类和数量可能也更丰富, 携带ARGs的细菌占比也更高.沉积物中, 除优势基因tetC和tetG外, sul1 和 sul2 的相对丰度相对于水体中也显著升高, 相对丰度分别为3.28×10-3~3.7×10-2和2.49×10-3~3.89×10-2.另外, 水样中检测到了较高丰度的一类整合子基因 intI1 , 相对丰度为1.97×10-3~3.34×10-2, 其对水体中ARGs的水平传播方面起着重要作用.
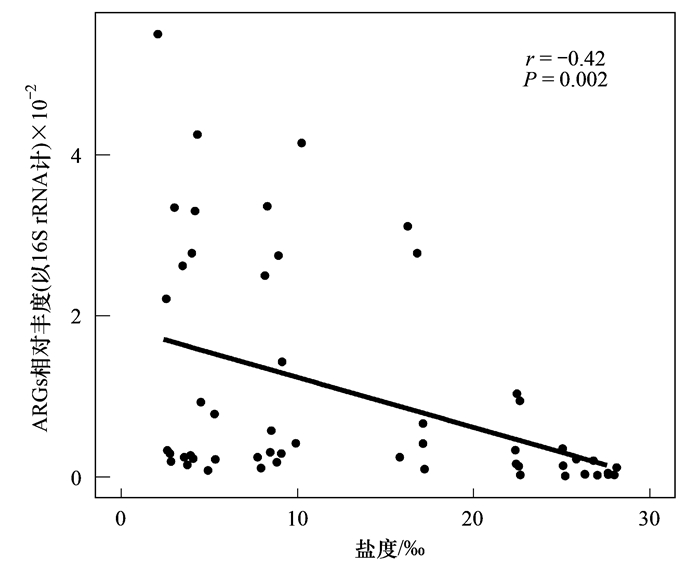
水样中, ARGs的分布总体上随水体盐度(表 1)的增加逐步递减, 将ARGs相对丰度和各采样点盐度进行Spearman相关性分析发现, 二者显著负相关(P=0.002, 图 4).何基兵等[32]的研究也发现九龙江河口表层水中部分磺胺类ARGs检出率随着盐度增加而减小.河口区域存在海水稀释作用, 一定程度上会促进ARGs丰度下降, 但最根本的原因在于内陆河流中携带ARGs的耐药菌不适应近海高盐度生长环境, 从而阻断了ARGs垂直传播, 导致丰度下降[33].
|
图 4 水样中ARGs和盐度的相关性 Fig. 4 Correlation between ARGs and salinity in water samples |
沉积物中, 各采样点ARGs的相对丰度并未呈现出水样中的趋势, 受盐度影响较小, 可能是由于沉积物环境受到的扰动小, 较为稳定.
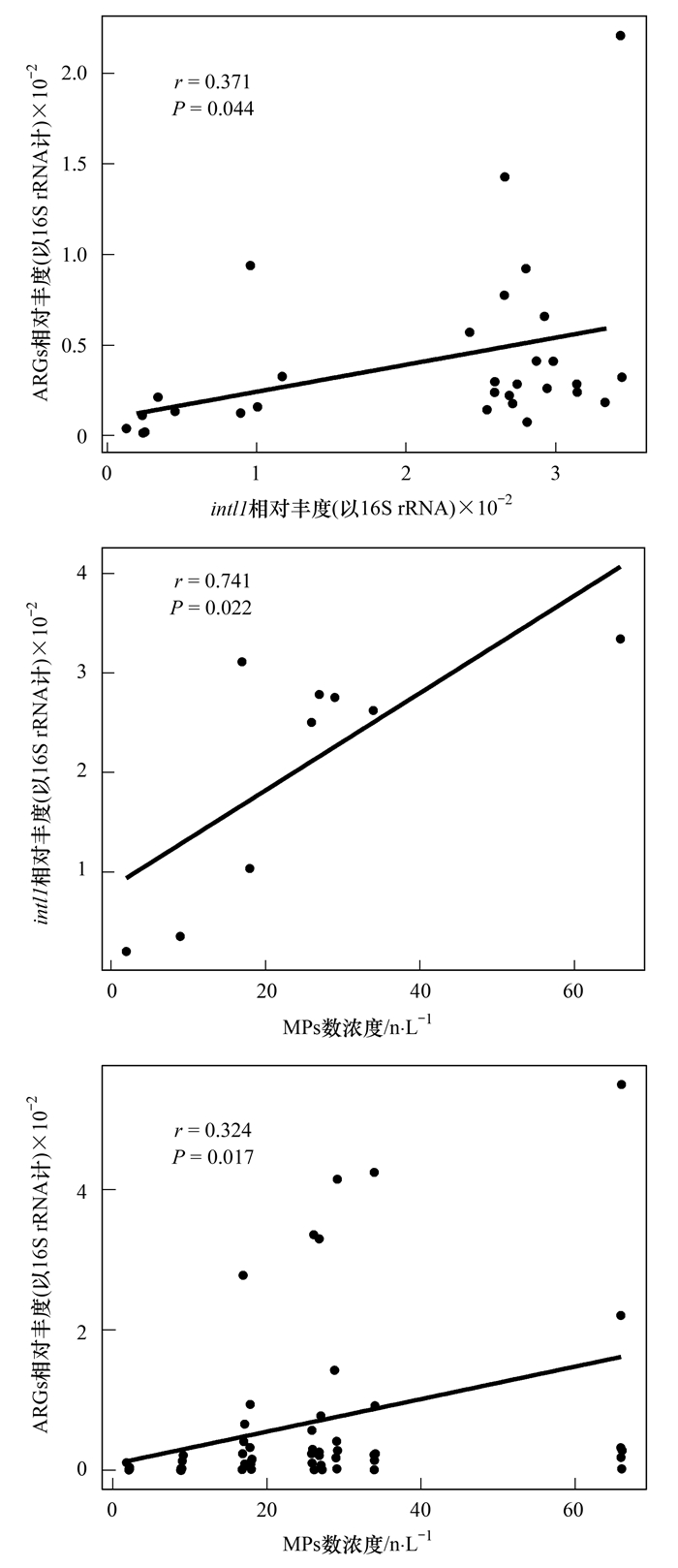
2.3 MPs污染和ARGs污染分布相关性将从各采样点水样中测得的ARGs相对丰度与一类整合子基因 intI1 的相对丰度、MPs数浓度进行两两相关性分析, 结果如图 5所示.
|
图 5 水样中ARGs、intI1 和MPs间相关性 Fig. 5 Correlation among ARGs, intI1 , and microplastics in water samples |
MPs浓度、ARGs和 intI1 基因两两之间均呈正相关关系(P < 0.05), 表明MPs具有富集耐药菌与ARGs的能力, 同时MPs上水平基因转移较为频繁.周曙仡聃等[34]采集海水并添加MPs进行49 d曝气培养实验, 发现MPs显著增加了海水中ARGs的丰度, ARGs与可移动遗传元件有着显著正相关关系, 该结果与本文的一致. intI1 是可移动遗传元件之一, 在一定程度能促进ARGs的水平转移, 从而增加ARGs的宿主菌种类, 扩大传播范围.河口环境中经过长期老化的MPs更容易成为微生物的载体, 同时由于比表面积小, MPs也能增加细菌种间接触和基因交流机会, 从而促进水平基因转移.因此, MPs在一定程度上会提高ARGs在不同环境中的传播风险, 两者的复合污染问题不容忽视.
3 结论(1) 九龙江口ARGs以四环素类和磺胺类为主, 水中高盐度会显著抑制ARGs丰度, 呈现沿河向入海口递减趋势.
(2) 水相与沉积物中的MPs以小粒径为主, 河口上游污染较为严重, MPs颜色和种类都较为丰富.
(3) 水相中, MPs、ARGs和 intI1 三者呈两两正相关, MPs对于河口环境中ARGs传播起着促进作用.
| [1] | Amelia T S M, Khalik W M A W M, Ong M C, et al. Marine microplastics as vectors of major ocean pollutants and its hazards to the marine ecosystem and humans[J]. Progress in Earth and Planetary Science, 2021, 8(1). DOI:10.1186/s40645-020-00405-4 |
| [2] | Gong J, Xie P. Research progress in sources, analytical methods, eco-environmental effects, and control measures of microplastics[J]. Chemosphere, 2020, 254. DOI:10.1016/j.chemosphere.2020.126790 |
| [3] | Klangnurak W, Chunniyom S. Screening for microplastics in marine fish of Thailand: the accumulation of microplastics in the gastrointestinal tract of different foraging preferences[J]. Environmental Science and Pollution Research, 2020, 27(21): 27161-27168. DOI:10.1007/s11356-020-09147-8 |
| [4] | Xie M W, Huang J L, Lin Z, et al. Field to laboratory comparison of metal accumulation on aged microplastics in coastal waters[J]. Science of the Total Environment, 2021, 797. DOI:10.1016/j.scitotenv.2021.149108 |
| [5] | Guo X, Wang J L. Sorption of antibiotics onto aged microplastics in freshwater and seawater[J]. Marine Pollution Bulletin, 2019, 149. DOI:10.1016/j.marpolbul.2019.110511 |
| [6] | Yu F, Yang C F, Zhu Z L, et al. Adsorption behavior of organic pollutants and metals on micro/nanoplastics in the aquatic environment[J]. Science of the Total Environment, 2019, 694. DOI:10.1016/j.scitotenv.2019.133643 |
| [7] | Liu G Z, Zhu Z L, Yang Y X, et al. Sorption behavior and mechanism of hydrophilic organic chemicals to virgin and aged microplastics in freshwater and seawater[J]. Environmental Pollution, 2019, 246: 26-33. DOI:10.1016/j.envpol.2018.11.100 |
| [8] |
范秀磊, 邹晔锋, 刘加强, 等. 老化前后轮胎磨损微粒与聚氯乙烯微粒对抗生素的吸附-解吸行为[J]. 环境科学, 2021, 42(4): 1901-1912. Fan X L, Zou Y F, Liu J Q, et al. Adsorption and desorption behaviors of antibiotics on TWP and PVC particles before and after aging[J]. Environmental Science, 2021, 42(4): 1901-1912. |
| [9] | Du Y H, Liu X B, Dong X S, et al. A review on marine plastisphere: biodiversity, formation, and role in degradation[J]. Computational and Structural Biotechnology Journal, 2022, 20: 975-988. DOI:10.1016/j.csbj.2022.02.008 |
| [10] | Zettler E R, Mincer T J, Amaral-Zettler L A. Life in the "Plastisphere": microbial communities on plastic marine debris[J]. Environmental Science & Technology, 2013, 47(13): 7137-7146. |
| [11] | Wu N, Zhang Y, Zhao Z, et al. Colonization characteristics of bacterial communities on microplastics compared with ambient environments (water and sediment) in Haihe Estuary[J]. Science of the Total Environment, 2020, 708. DOI:10.1016/j.scitotenv.2019.134876 |
| [12] | Liu Y, Liu W Z, Yang X M, et al. Microplastics are a hotspot for antibiotic resistance genes: Progress and perspective[J]. Science of the Total Environment, 2021, 773. DOI:10.1016/j.scitotenv.2021.145643 |
| [13] | Dong H, Chen Y L, Wang J, et al. Interactions of microplastics and antibiotic resistance genes and their effects on the aquaculture environments[J]. Journal of Hazardous Materials, 2021, 403. DOI:10.1016/j.jhazmat.2020.123961 |
| [14] | Feng G Q, Huang H N, Chen Y G. Effects of emerging pollutants on the occurrence and transfer of antibiotic resistance genes: a review[J]. Journal of Hazardous Materials, 2021, 420. DOI:10.1016/j.jhazmat.2021.126602 |
| [15] | Wang S S, Xue N N, Li W F, et al. Selectively enrichment of antibiotics and ARGs by microplastics in river, estuary and marine waters[J]. Science of the Total Environment, 2020, 708. DOI:10.1016/j.scitotenv.2019.134594 |
| [16] |
周昕原, 王言仔, 苏建强, 等. 微塑料对河水抗生素抗性基因的影响[J]. 环境科学, 2020, 41(9): 4076-4080. Zhou X Y, Wang Y Z, Su J Q, et al. Microplastics-induced shifts of diversity and abundance of antibiotic resistance genes in river water[J]. Environmental Science, 2020, 41(9): 4076-4080. |
| [17] | Rong D L, Wu Q P, Xu M F, et al. Prevalence, virulence genes, antimicrobial susceptibility, and genetic diversity of Staphylococcus aureus from retail aquatic products in China[J]. Frontiers in Microbiology, 2017, 8. DOI:10.3389/fmicb.2017.00714 |
| [18] | Yan M T, Nie H Y, Xu K H, et al. Microplastic abundance, distribution and composition in the Pearl River along Guangzhou city and Pearl River estuary, China[J]. Chemosphere, 2019, 217: 879-886. DOI:10.1016/j.chemosphere.2018.11.093 |
| [19] | Wu N, Zhang Y, Zhang X H, et al. Occurrence and distribution of microplastics in the surface water and sediment of two typical estuaries in Bohai Bay, China[J]. Environmental Science: Processes & Impacts, 2019, 21(7): 1143-1152. |
| [20] |
张丹丹, 郭亚平, 任红云, 等. 福建省敖江下游抗生素抗性基因分布特征[J]. 环境科学, 2018, 39(6): 2600-2606. Zhang D D, Guo Y P, Ren H Y, et al. Characteristics of antibiotic resistance genes in downstream areas of the Aojiang river, Fujian Province[J]. Environmental Science, 2018, 39(6): 2600-2606. |
| [21] | Chen Y R, Guo X P, Niu Z S, et al. Antibiotic resistance genes (ARGs) and their associated environmental factors in the Yangtze Estuary, China: from inlet to outlet[J]. Marine Pollution Bulletin, 2020, 158. DOI:10.1016/j.marpolbul.2020.111360 |
| [22] | Lu X M, Lu P Z. Seasonal variations in antibiotic resistance genes in estuarine sediments and the driving mechanisms[J]. Journal of Hazardous Materials, 2020, 383. DOI:10.1016/j.jhazmat.2019.121164 |
| [23] | Ji X L, Shen Q H, Liu F, et al. Antibiotic resistance gene abundances associated with antibiotics and heavy metals in animal manures and agricultural soils adjacent to feedlots in Shanghai; China[J]. Journal of Hazardous Materials, 2012, 235-236: 178-185. DOI:10.1016/j.jhazmat.2012.07.040 |
| [24] | Peak N, Knapp C W, Yang R K, et al. Abundance of six tetracycline resistance genes in wastewater lagoons at cattle feedlots with different antibiotic use strategies[J]. Environmental Microbiology, 2007, 9(1): 143-151. DOI:10.1111/j.1462-2920.2006.01123.x |
| [25] | Su H C, Liu Y S, Pan C G, et al. Persistence of antibiotic resistance genes and bacterial community changes in drinking water treatment system: from drinking water source to tap water[J]. Science of the Total Environment, 2018, 616-617: 453-461. DOI:10.1016/j.scitotenv.2017.10.318 |
| [26] | Zhao S Y, Zhu L X, Wang T, et al. Suspended microplastics in the surface water of the Yangtze Estuary System, China: first observations on occurrence, distribution[J]. Marine Pollution Bulletin, 2014, 86(1-2): 562-568. |
| [27] | Haddout S, Gimiliani G T, Priya K L, et al. Microplastics in surface waters and sediments in the Sebou estuary and Atlantic coast, Morocco[J]. Analytical Letters, 2022, 55(2): 256-268. |
| [28] | Zuo L Z, Sun Y X, Li H X, et al. Microplastics in mangrove sediments of the Pearl River estuary, south China: correlation with halogenated flame retardants' levels[J]. Science of the Total Environment, 2020, 725. DOI:10.1016/j.scitotenv.2020.138344 |
| [29] | Wang J D, Tan Z, Peng J P, et al. The behaviors of microplastics in the marine environment[J]. Marine Environmental Research, 2016, 113: 7-17. |
| [30] | Firdaus M, Trihadiningrum Y, Lestari P. Microplastic pollution in the sediment of Jagir Estuary, Surabaya City, Indonesia[J]. Marine Pollution Bulletin, 2020, 150. DOI:10.1016/j.marpolbul.2019.110790 |
| [31] | Zhu Y G, Zhao Y, Li B, et al. Continental-scale pollution of estuaries with antibiotic resistance genes[J]. Nature Microbiology, 2017, 2(4). DOI:10.1038/nmicrobiol.2016.270 |
| [32] | 何基兵, 胡安谊, 陈猛, 等. 九龙江河口及厦门污水处理设施抗生素抗性基因污染分析[J]. 微生物学通报, 2012, 39(5): 683-695. |
| [33] | Lu Z H, Na G S, Gao H, et al. Fate of sulfonamide resistance genes in estuary environment and effect of anthropogenic activities[J]. Science of the Total Environment, 2015, 527-528: 429-438. |
| [34] |
周曙仡聃, 朱永官, 黄福义. 微塑料对海水抗生素抗性基因的影响[J]. 环境科学, 2021, 42(8): 3785-3790. Zhou S Y D, Zhu Y G, Huang F Y. Microplastic-induced alterations to antibiotic resistance genes in seawater[J]. Environmental Science, 2021, 42(8): 3785-3790. |
 2022, Vol. 43
2022, Vol. 43


