2. 中机国际工程设计研究院有限责任公司华东分院, 南京 210049
2. China Machinery International Engineering Design & Research Institute Co., Ltd., (CMIE) East China Branch, Nanjing 210049, China
微塑料是指粒径小于5 mm的塑料碎片或颗粒, 主要来源于大塑料的裂解[1]和小塑料的直接排放等[2].近年来, 微塑料作为一种新污染物, 已在全球范围内引发了广泛关注.有研究发现, 微塑料在海洋、湖泊、水库和河流等水环境中分布广泛[3, 4], 并可通过其自身的物理、化学和载体等多重作用威胁生物体的生长、发育和繁殖[5], 甚至会随着食物链由低营养级向高营养级传递[6, 7], 最终威胁人类健康和生态安全.因此, 水环境中的微塑料污染又被称为“水中PM2.5”污染.
环境中的微塑料由于具有比表面积大、表面坚硬、疏水性强和官能团丰富等特点, 可为环境微生物提供理想的生态位[8~10], 极易被微生物快速附着并定殖, 并形成与周围环境显著不同的“塑料圈(plastisphere)”.有研究显示, 微塑料表面附着的细菌群落结构和功能与塑料聚合物的类型、营养盐状况、浊度和温度等环境因素密切有关[11, 12].定殖于微塑料表面的细菌一方面会改变微塑料的密度, 加快沉降, 使其免受风化和紫外辐射等影响[13, 14], 进而减缓微塑料的非生物降解; 另一方面, 微塑料表面生物膜的定殖也能导致微塑料降解菌的富集, 进而加快微塑料的生物降解[15].因此, 附着的细菌群落与微塑料之间存在紧密的相互作用, 探讨微塑料表面附着的细菌群落结构和功能对掌握两者在环境中的迁移与归趋具有重要的生态意义.
当前, 已有研究分析了附着于微塑料表面的细菌群落结构[16]、多样性[17]和功能[18]等变化, 初步阐述了其与环境因素的交互作用过程.但在自然水体中, 微塑料往往因密度和老化程度等因素差异而呈现垂向分布特征[19, 20], 附着于其上的细菌群落是否会因微塑料垂向分层而产生差异目前仍然无法确定.本研究以我国水环境中常见的两种微塑料: 聚对苯二甲酸乙二醇酯(polyethylene terephthalate, PET)和聚氯乙烯(polyvinyl chloride, PVC)为供细菌附着的载体, 在天然水体中进行微塑料垂向分布暴露实验, 通过分析不同分层微塑料表面附着细菌群落结构、生物多样性、功能响应和变化, 阐述微塑料垂向分布下的细菌群落响应规律, 研究结果对评估实际环境中微塑料污染风险具有重要指导意义.
1 材料与方法 1.1 实验材料以环境中常见的两种微塑料类型(PET和PVC)为基质, 探索附着生物膜的响应.两种塑料均来自于日常生活用品, 其中PET为矿泉水瓶, PVC为包装袋.将两种塑料制品分别剪裁成5 mm×5 mm的正方形塑料片, 用酒精擦拭后超声处理5 min, 以去除微塑料上携带的原始微生物.
1.2 生物膜培养水体微塑料垂向暴露采用圆柱形不锈钢丝网进行(直径约20 cm, 高约25 cm, 孔径>1 mm).首先, 分别将PET和PVC微塑料片等量地(45个)放入不锈钢滤网中(共18个滤网), 随之将滤网分3层固定在不锈钢绳上, 每层间隔30 cm.为了避免底层沉积物再悬浮的干扰效应, 将上述装置垂向等距地放入暴露水体中固定, 距离湖底30 cm, 距离水面分别为30、60和90 cm.此外, 钢丝下端坠有重物以维持该装置在水中的稳定.每种微塑料设置3个平行.
微塑料的暴露选择在河海大学东湖(118°46′55.25″E, 31°54′59.03″N), 水深与长江下游典型城市河湖水体的深度相一致, 约1.2 m, 其不同于海洋和深大水库, 无天然分层现象.微塑料暴露时间为2019年11月5日至12月5日, 实验期间每隔10 d检查生物膜的生长情况.该时间段内, 无明显降水汇入, 不会强烈扰动湖水和底泥, 使其处于相对稳定状态, 且水温适宜有利于微生物的生长和稳定.实验期间, 利用Hach探头原位检测东湖3种深度下的pH、溶解氧(DO)、氧化还原电位(ORP)、浊度和水温, 并采集水样测定水体中的氨氮(NH4+-N)和总磷(TP).总体上, pH、DO、ORP、NH4+-N、TP和温度在水体垂向上没有明显差异, 浊度具有一定的垂向差异性.水体理化性质具体数值如表 1所示.
|
|
表 1 东湖不同水深处水质参数均值 Table 1 Mean value of water quality parameters in different depths of East Lake |
1.3 生物膜提取
生物膜的提取根据Wang等[21]的方法进行.将暴露装置中的微塑料取出后, 迅速置于冰上并带回实验室.附着在微塑料上的生物膜用无菌去离子水仔细清洗, 随后刮片收集并采用0.22 μm的无菌纤维膜进行过滤, 收集附着的微生物.收集到的生物膜置于液氮保存, 以待后续DNA提取和细菌群落分析.
1.4 DNA提取、PCR扩增和高通量测序分析生物膜DNA的提取采用HiPure Bacterial DNA Kits(基迪奥生物, 广州)进行, 而后使用ABI StepOnePlus Real Time PCR System(Life Technologies, 美国)进行扩增.将样本中提取的基因组DNA作为PCR扩增模板, 用带有barcode的特异引物扩增16S rRNA的V3和V4区域进行高通量测序, 测序平台为Illumina MiSeq(基迪奥生物, 广州).其中, PCR引物序列为341F: CCTACGGGNGGC WGCAG; 806R: GGACTACHVGGGTATCTAAT.第一轮PCR扩增条件: 94℃预变性5 min, 之后98℃变性10 s, 62~66℃退火30 s(55℃ for 16s V4), 68℃延伸30 s, 30个循环, 最后在68℃保持5 min; 第二轮扩增条件: 94℃预变性2 min, 之后98℃变性10 s, 65℃退火30 s, 68℃延伸30 s, 12个循环, 最后在68℃保持5 min.两轮扩增产物均使用AMPure XP Beads进行纯化.扩增测序产生的原始数据通过FASTP软件对低质量序列进行过滤, 然后通过FLASH软件对序列进行组装和合并.使用USEARCH软件核对剩余的嵌合序列.
1.5 数据处理与分析生物信息在Omicsmart实时交互平台(http://www.omicsmart.com)进行分析.对物种组成多样性信息, 用Uparse软件对所有样品的全部Effective Tags序列聚类, 以97%的一致性将序列聚类成为OTU(operational taxonomic unit).随后进行OTU分析、物种组成分析、α多样性分析、β多样性分析和功能分析.根据OTU的物种注释和丰度信息, 使用Tax4Fun软件进行KEGG Pathway的功能代谢通路预测.不同处理之间的多重比较采用单因素方差分析中的Turkey统计检验法, P < 0.05为统计显著性.
2 结果与分析 2.1 细菌群落多样性分析原始数据在进行低质量reads过滤后再进行组装和再过滤, 共获得1 491 600条有效序列, 并将其分配给51 260个OTU.根据OTU聚类结果, 对每个OTU序列做代表注释, 得到对应的物种信息和基于物种的丰度分布情况.
基于OTU进行α多样性分析, 包括Chao1指数、ACE指数、Simpson指数、Shannon指数和覆盖率, 从而得到物种丰富度和多样性.各样本α多样性指数如表 2所示.所有样品覆盖率均在98.8%以上, 表明该测序深度下, 有效数据量足够大, 基本能反映样品中绝大多数细菌群落的物种信息.
|
|
表 2 不同微塑料附着细菌群落α多样性指数1) Table 2 The α diversity index of bacterial communities attached to different microplastics |
在暴露期内, 微塑料附着细菌群落的丰富度(Chao1指数、ACE指数)和多样性(Shannon指数、Simpson指数)均存在显著差异.就物种多样性而言, PVC在90 cm深度上附着的细菌Shannon指数是其在30 cm处的1.2倍, PET则是1.4倍.相同水深处, PET附着的细菌Shannon指数明显高于PVC, 90 cm处PET的Shannon指数是PVC的1.24倍.
在物种丰富度方面, PVC在90 cm深度上附着的细菌Chao1和ACE指数分别是30 cm处的1.7倍和1.72倍, 且PET在90 cm深度下的Chao1和ACE指数分别是30 cm处的2.02倍和2.08倍.此外, 在相同深度下附着于PET的细菌Chao1和ACE指数也明显高于PVC.这表明, 微塑料上附着的细菌总数随水深增加而明显增加, 且PET上附着的细菌总数高于PVC.
2.2 细菌群落结构和组成分析不同微塑料表面附着细菌在门和纲水平上的优势物种组成如图 1所示.在门(phylum)水平上[图 1(a)], 丰度前10的细菌占总量的79.66%~95.08%, 蓝藻门(Cyanobacteria)和变形菌门(Proteobacteria)为绝对的优势菌门, 其次为浮霉菌门(Planctomycetes)、疣微菌门(Verrucomicrobia)、拟杆菌门(Bacteroidetes). 90 cm处PET附着的蓝藻门丰度(18.85%)明显下降, 疣微菌门(Verrucomicrobia)相对丰度(13.35%)明显高于其他样本.其中, 蓝藻门(Cyanobacteria)主要是通过光合作用获得能力的细菌.拟杆菌门则是水中常见的细菌, 拟杆菌门的分布会受到微塑料类型和水深深度的影响.变形菌门为30、60和90 cm的塑料上的优势菌群, 常常作为初始定殖菌, 其可对水环境中的各种营养资源做出迅速反应以促进生物膜的形成[22].垂向上随着水深增加, 不同门类的细菌分布变得更加均衡, 细菌种类增多, 多样性提高.
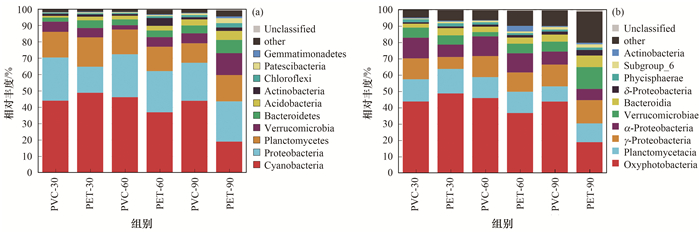
|
(a)门水平; (b)纲水平 图 1 不同水深细菌群落物种组成和相对丰度分布 Fig. 1 Species composition and relative abundance distribution of bacterial communities in different water depths |
在纲(class)水平上[图 1(b)], 3个不同水深微塑料附着细菌主要由生氧光细菌纲(Oxyphotobacteria)、浮霉菌纲(Planctomycetacia)、γ-变形菌纲(γ-Proteobacteria)、α-变形菌纲(α-Proteobacteria)、疣微菌纲(Verrucomicrobiae)、拟杆菌纲(Bacteroidia)、δ-变形菌纲(δ-Proteobacteria)、Phycisphaerae、Subgroup_6和放线菌纲(Actinobacteria)组成.其中, PET附着的绝对优势物种光氧细菌纲和浮霉菌纲均呈现出垂向变化的特征, 即30 cm处丰度大于60 cm, 均大于90 cm处.微塑料类型和水深可能共同影响附着细菌在纲水平上的物种组成.
为了深入理解不同微塑料附着细菌群落结构的垂向分布差异, 通过Indicator分析对具有统计学意义的指示物种(biomarker)进行分析(P < 0.05).在门和纲水平上, 90 cm处附着于PVC和PET的物种丰度均显著高于30 cm和60 cm, 且附着于PET的细菌物种多样性均高于PVC.
图 2展示了门水平下PVC和PET样本丰度差异显著的物种类别.结果显示, 在门水平下共获得16类具有显著性差异的菌门.其中, PET和PVC样本中共有菌门为11种, 说明细菌群落具有一定的相似性. PVC特有的差异菌门为衣原体门(Chlamydiae), 附着于PET微塑料特征菌群为拟杆菌门(Bacteroidetes)、广古菌门(Euryarchaeota)、Rokubacteria和黏胶球形菌门(Latescibacteria).此外, 特征物种具有一定的垂向分布差异性.在30 cm水深处, 异常球菌-栖热菌门(Deinococcus-Thermus)是PET和PVC仅有的特征菌门.在60 cm水深处, 衣原体门是PVC样本唯一的指示物种; PET无显著性差异物种.在水深90 cm处, 附着于PVC和PET的差异物种类别分别为10和14, 这与上述α多样性指数规律一致.90 cm处附着于PVC主要的差异菌门为Omnitrophicaeota、硝化螺旋菌门(Nitrospirae)、硝化刺菌门(Nitrospinae)、Kiritimatiellaeota和螺旋体门(Spirochaetes); PET在90 cm处主要的差异物种为Omnitrophicaeota、Patescibacteria、黏胶球形菌门、螺旋体门和广古菌门.
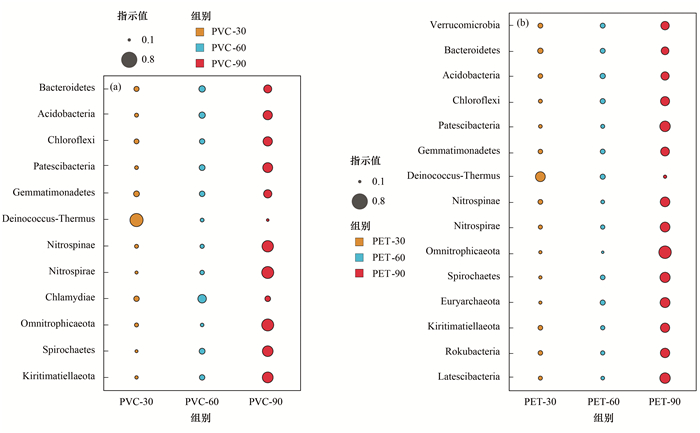
|
(a)PVC微塑料; (b)PET微塑料 图 2 附着于两种微塑料细菌群落在门水平下的指示物种分析 Fig. 2 Indicator analysis of bacterial communities attached to two typical plastics at the phylum level |
对所有微生物样本基于unweighted unifrac距离算法进行降维分析, 通过NMDS分析3种深度下细菌群落的差异性.如图 3所示, stress值为0.042, 表明NMDS分析具有可靠性.结果显示, 在PET生物膜中, PET-30和PET-60距离最近, 这表明两处细菌群落较为相似; 而PET-90与PET-30和PET-60距离变远, 表明在湖水深处, PET附着细菌群落发生了明显的改变; 对PVC而言, 3种深度下的距离都较远, PVC附着菌群随着水深变化明显, 相似性明显降低.
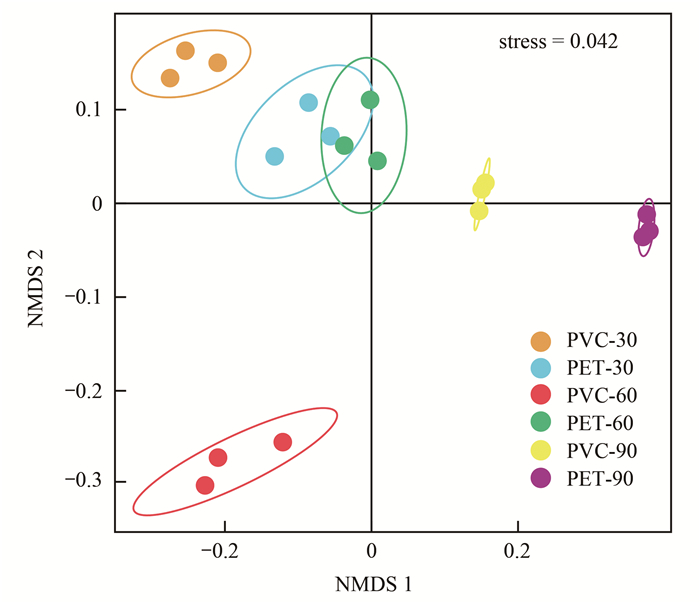
|
图 3 OTU水平下基于未加权细菌群落NMDS排序 Fig. 3 Bacterial community NMDS sequence based on unweighted UniFrac at OTU level |
为了调查塑料类型和水深深度对生物膜群落功能多样性的影响, 从16S获得的数据序列注释到KEGG数据库中, 分析塑料生物膜菌群的代谢通路.通过Tax4Fun对附着于生物膜上的细菌功能进行预测分析[23], 发现了代谢、环境信息处理、遗传信息处理、细胞转化、疾病和有机系统共6类一级功能层生物代谢通路, 且上述代谢通路相对丰度依次减小.进一步对二级功能层生物代谢进行分析, 发现共有34个子功能组成.如图 4(a)所示, 二级功能主要由碳水化合物代谢、辅助因子和维生素、氨基酸代谢、多酮类化合物和萜类化合物代谢、其他氨基酸代谢组成.
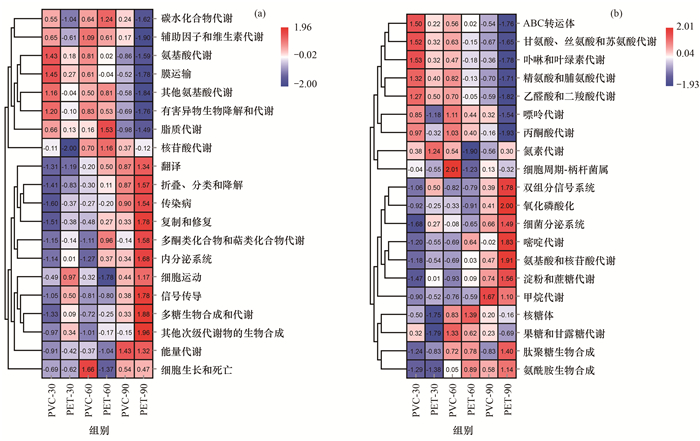
|
(a)二级功能; (b)三级功能 图 4 细菌群落功能丰度热图 Fig. 4 Abundance heat map of bacterial community |
三级功能垂向组成依二级功能改变而改变, 不同基质上附着细菌功能具有一定的差异性.如图 4(b)所示, ABC转运体、甘氨酸、丝氨酸和苏氨酸代谢、卟啉和叶绿素代谢、精氨酸和脯氨酸代谢功能表达与水深的深度呈负相关; 且在同一深度下, 附着于PVC的细菌功能表达强于PET.此外, 嘧啶代谢、氨基酸和核苷酸的代谢、淀粉和蔗糖代谢和氨酰胺生物合成代谢功能表达与水深深度呈正相关, 其在90 cm处的功能表达丰度显著高于30 cm和60 cm处的样本.整体而言, 三级微生物代谢通路具有一定的垂向规律: ABC转运体、甘氨酸、丝氨酸和苏氨酸代谢、卟啉和叶绿素代谢、精氨酸和脯氨酸代谢和丙酮酸代谢在水深30 cm和60 cm处的功能表达相对丰富; 双组分信号系统、氧化磷酸化、细菌分泌系统、嘧啶代谢、氨基糖和核苷酸代谢、淀粉和蔗糖代谢、甲烷代谢和氨酰胺生物合成等功能易于在深水处表达.
3 讨论水体微塑料可作为微生物附着的载体.本研究将两种微塑料在水体中进行垂向暴露后发现, 微塑料表面附着的细菌群落多样性随着水深的增加而明显增加, PET表面附着细菌群落多样性明显高于PVC, 且各样本功能具有一定的垂向分布规律.
3.1 微塑料类型对细菌群落结构的影响微生物群落结构与微塑料类型密切相关, 不同类型的微塑料由于其内在性质不同而影响微生物的群落结构.微塑料的粗糙度[24]、亲水性[25]、比表面积[11]和表面基团[26]等均会影响微生物的定殖.本研究中, 同一深度下PET附着细菌群落的α多样性指数均高于PVC微塑料, 这可能是因为PVC作为一种“有毒”微塑料[27], 抑制了细菌的生存和繁殖.已有研究发现, PVC会抑制nirS基因丰度[28]和反硝化细菌数量[29], 进而影响氮的转化.此外, 塑料生产过程中的各种化学添加剂(如增塑剂、阻燃剂和稳定剂等)的释放也会对附着微生物产生毒性抑制效应, 进而干扰其正常生长.附着于微塑料表面的微生物会改变微塑料自身的密度等特性[19], 反作用于微塑料本身, 从而影响微塑料在淡水环境中的迁移行为和分布特征.然而, 目前关于微塑料在水体中垂向迁移规律的相关研究仍旧匮乏, 需要开展进一步研究.
3.2 水深深度对微生物群落结构的影响水深深度也会影响细菌的群落结构, 无论是在门还是纲水平上, 深水环境都在一定程度上刺激了细菌的生长代谢.在门水平上, 蓝藻门在PET塑料上的丰度随着水深深度的增加而显著降低, 而PVC则基本无变化; 90 cm处PVC塑料附着的疣微菌门丰度与其在30 cm处相近, PET塑料上的附着物显著增加.在纲水平上, 不同水深PVC附着的生氧光细菌纲相对丰度基本一致, 其附着在PET塑料上的相对丰度随着水深的增加而显著减小.对比同一深度下不同底物上的细菌丰度变化, 发现浅水处附着在PVC和PET塑料上的生氧光细菌纲丰度相似, 深水处两者相对丰度差异较大, 说明相较于微塑料类型, 水深深度对细菌群落结构影响更大, 这一结果与已有的研究相一致[11].
生氧光细菌是一种利用光能进行自身同化代谢的细菌, 其在PET-30处大量检出, 而在深水处由于阳光稀少无法进行光合作用, PET-90处生氧光细菌数量明显减少.蓝藻门和生氧光细菌纲在PVC上无垂向变化, 这可能是因为PVC更易释放添加剂, 对光产生遮蔽效应抑制光合作用造成的[30].疣微菌是一种在富营养环境下利用硫酸盐降解多糖, 在寡营养条件下化能自养代谢获取能量的微生物[31], 其在90 cm处的丰度显著增加, 可能和深水环境有利于其生长繁殖有关.Helgason等[32]的研究发现疣微菌更易在深水环境处生存. γ变形菌纲为变形菌门下的纲, 其在90 cm处的丰度相较于浅水明显上升, 表明γ变形菌纲适合在深水中生存.垂向分布上, 随着水深的增加优势物种发生明显改变, 但多样性水平随着水深增加而提高, 这可能和深水环境距离沉积物更近, 更易与底部沉积物进行营养物质和微生物群落交换有关[33].
微塑料类型和水深深度不仅会影响物种多样性, 还会对细菌群落结构产生一定的差异性.通过对门水平上的差异物种进行研究, 发现附着于微塑料的细菌丰度在垂向暴露下具有显著差异性.附着于PVC和PET的异常球菌-栖热菌门是水深30 cm处的指示物种, 其在30 cm处的物种丰度均显著高于60 cm和90 cm, 这可能和浅水环境辐射和水温相对较高, 该菌门耐辐射和高温易于在浅水环境繁殖有关.拟杆菌门可降解聚合物[34], 常作为次级生产者定殖在微塑料表面[35].其在PVC-90处大量附着, 表明深水环境更有利于拟杆菌门以PVC为碳源而生长繁殖.硝化螺旋菌门和硝化刺菌门是一类亚硝酸盐氧化菌, 其能在氮转化过程中固定部分无机碳进而调控碳氮循环[36], Chen等[37]的研究发现微塑料生物膜具有促进亚硝酸盐氧化的作用.螺旋体门属于致病菌, 其主要营腐生或寄生生活[38].其在PVC-90和PET-90处大量繁殖, 这可能和表层沉积物再悬浮有关, 使得深水环境浊度相对较高且部分沉积物附着在深水环境微塑料生物膜上, 易于致病菌生长繁殖.已有研究表明沉积物可作为病原微生物库使其长期生存[39].
3.3 垂向分布下微塑料生物膜功能注释水深深度和微塑料类型主要通过影响细菌群落组成进而影响细菌的功能.在不同的水深处, 一级功能层丰度基本一致, 且主要功能为代谢和遗传信息处理, 说明代谢和遗传信息处理在细菌定殖过程中起着重要的功能作用.在垂向分布下, 各样本二级功能层丰度存在差异.在水深30 cm和60 cm处, PET和PVC细菌功能表达主要体现为碳水化合物代谢、辅助因子和维生素代谢、氨基酸代谢、膜运输、其他氨基酸代谢、有害异物生物降解和代谢、脂质代谢和核苷酸代谢这8种功能组成.其中, 碳水化合物代谢和卟啉和叶绿素代谢在30 cm和60 cm处功能丰度相对较高, 这可能和蓝藻细菌在浅水表面大量进行光合作用有关, 通过碳水化合物代谢为其提供生长发育所需的能量和代谢产物[40].氨基酸代谢可以促使细菌对氨基酸的吸收, 其对细菌的生存繁衍至关重要[41], 该功能在水深30 cm和60 cm处表达相对丰富, 说明深水环境在一定程度上抑制了该功能的表达.Bhagwat等[42]的研究发现生物膜可显著增强微塑料对污染物的载体潜力, 且附着于微塑料表面的细菌群落可以促进微塑料的分解[43], 这可能会诱发有害异物降解和代谢功能的表达.深水环境中, 两种微塑料生物群落功能多样性明显增加, 90 cm处PET附着细菌的功能丰度和多样性显著高于PVC, 进一步说明PVC塑料对附着细菌具有抑制效应.90 cm处的细菌群落功能主要体现为翻译、传染病、复制和修复、细胞运动、信号传导、多糖生物合成和代谢等12个方面.其中, 细胞运动和信号传导等过程主要由微塑料编码[44], 隶属于膜运输功能的ABC转运体可参与调节多糖的合成和生物代谢相关基因[45].上述功能均是生物膜形成和成熟过程中必不可少的功能, 这可能和深水环境细菌大量生长繁殖有关.已有研究指出, 生物膜上的细菌由于附着于微塑料将导致其代谢周转率升高[46], 故深水环境下细菌丰度和物种多样性的显著增加将会使得细胞运动、信号传导、多糖生物合成和代谢等功能高度表达.此外, 水深相同处的细菌表现出相近的功能, 进一步说明相较于基质类型, 水深深度对细菌群落结构和功能的影响更加显著.
4 结论(1) 在垂向分布下, 附着于PVC和PET微塑料的细菌物种丰富度均随着水深深度的增加而增加(30 cm < 60 cm < 90 cm).同一水深处, 附着于PET微塑料的细菌物种丰富度和多样性均高于PVC塑料, 更易附着细菌.
(2) 微塑料类型和水深深度均会影响细菌的群落结构, 相较于底物基质, 水深深度对细菌群落结构影响更大.3种深度下的优势菌门主要为: 蓝藻门(Cyanobacteria)、变形菌门(Proteobacteria)、浮霉菌门(Planctmoycetes)、疣微菌门(Verrucomicrobia)和拟杆菌门(Bacteroidetes).
(3) 附着于两种微塑料的细菌功能具有一定的垂向分布特征.附着于PET和PVC塑料上的细菌在30 cm和60 cm处的细菌功能相近, 但其在90 cm处的功能明显增加, 可能和细菌群落结构变化密切相关, 且PET上的细菌功能明显高于PVC.
| [1] | Van Cauwenberghe L, Vanreusel A, Mees J, et al. Microplastic pollution in deep-sea sediments[J]. Environmental Pollution, 2013, 182: 495-499. DOI:10.1016/j.envpol.2013.08.013 |
| [2] | Waldschläger K, Lechthaler S, Stauch G, et al. The way of microplastic through the environment-application of the source-pathway-receptor model (review)[J]. Science of the Total Environment, 2020, 713. DOI:10.1016/j.scitotenv.2020.136584 |
| [3] |
骆永明, 施华宏, 涂晨, 等. 环境中微塑料研究进展与展望[J]. 科学通报, 2021, 66(13): 1544-1562. Luo Y M, Shi H H, Tu C, et al. Research progresses and prospects of microplastics in the environment[J]. Chinese Science Bulletin, 2021, 66(13): 1544-1562. |
| [4] | Dris R, Imhof H, Sanchez W, et al. Beyond the ocean: contamination of freshwater ecosystems with (micro-)plastic particles[J]. Environmental Chemistry, 2015, 12(5): 539-550. DOI:10.1071/EN14172 |
| [5] | Huang D L, Tao J X, Cheng M, et al. Microplastics and nanoplastics in the environment: macroscopic transport and effects on creatures[J]. Journal of Hazardous Materials, 2021, 407. DOI:10.1016/j.jhazmat.2020.124399 |
| [6] | Yan Z H, Chen Y F, Bao X H, et al. Microplastic pollution in an urbanized river affected by water diversion: combining with active biomonitoring[J]. Journal of Hazardous Materials, 2021, 417. DOI:10.1016/j.jhazmat.2021.126058 |
| [7] | Huang W, Song B, Liang J, et al. Microplastics and associated contaminants in the aquatic environment: a review on their ecotoxicological effects, trophic transfer, and potential impacts to human health[J]. Journal of Hazardous Materials, 2021, 405. DOI:10.1016/j.jhazmat.2020.124187 |
| [8] |
季梦如, 马旖旎, 季荣. 微塑料圈: 环境微塑料对微生物的载体作用[J]. 环境保护, 2020, 48(23): 19-27. Ji M R, Ma Y N, Ji R. Plastisphere: the vector effects of microplastics on microbial communities[J]. Environmental Protection, 2020, 48(23): 19-27. |
| [9] | Miao L Z, Wang P F, Hou J, et al. Distinct community structure and microbial functions of biofilms colonizing microplastics[J]. Science of the Total Environment, 2019, 650: 2395-2402. DOI:10.1016/j.scitotenv.2018.09.378 |
| [10] | Wright R J, Erni-Cassola G, Zadjelovic V, et al. Marine plastic debris: a new surface for microbial colonization[J]. Environmental Science & Technology, 2020, 54(19): 11657-11672. |
| [11] | Miao L Z, Yu Y, Adyel T M, et al. Distinct microbial metabolic activities of biofilms colonizing microplastics in three freshwater ecosystems[J]. Journal of Hazardous Materials, 2021, 403. DOI:10.1016/j.jhazmat.2020.123577 |
| [12] | Chen X C, Xiong X, Jiang X M, et al. Sinking of floating plastic debris caused by biofilm development in a freshwater lake[J]. Chemosphere, 2019, 222: 856-864. DOI:10.1016/j.chemosphere.2019.02.015 |
| [13] |
周倩, 涂晨, 张晨捷, 等. 滨海湿地环境中微塑料表面性质及形貌变化[J]. 科学通报, 2021, 66(13): 1580-1591. Zhou Q, Tu C, Zhang C J, et al. Surface properties and changes in morphology of microplastics exposed in-situ to Chinese coastal wetlands[J]. Chinese Science Bulletin, 2021, 66(13): 1580-1591. |
| [14] | Rummel C D, Jahnke A, Gorokhova E, et al. Impacts of biofilm formation on the fate and potential effects of microplastic in the aquatic environment[J]. Environmental Science & Technology Letters, 2017, 4(7): 258-267. |
| [15] | Yuan J H, Ma J, Sun Y R, et al. Microbial degradation and other environmental aspects of microplastics/plastics[J]. Science of the Total Environment, 2020, 715. DOI:10.1016/j.scitotenv.2020.136968 |
| [16] | Li C C, Gan Y D, Dong J Y, et al. Impact of microplastics on microbial community in sediments of the Huangjinxia Reservoir—water source of a water diversion project in western China[J]. Chemosphere, 2020, 253. DOI:10.1016/j.chemosphere.2020.126740 |
| [17] | Wu N, Zhang Y, Zhao Z, et al. Colonization characteristics of bacterial communities on microplastics compared with ambient environments (water and sediment) in Haihe Estuary[J]. Science of the Total Environment, 2020, 708. DOI:10.1016/j.scitotenv.2019.134876 |
| [18] | Hu H, Jin D F, Yang Y Y, et al. Distinct profile of bacterial community and antibiotic resistance genes on microplastics in Ganjiang River at the watershed level[J]. Environmental Research, 2021, 200. DOI:10.1016/j.envres.2021.111363 |
| [19] | Miao L Z, Gao Y X, Adyel T M, et al. Effects of biofilm colonization on the sinking of microplastics in three freshwater environments[J]. Journal of Hazardous Materials, 2021, 413. DOI:10.1016/j.jhazmat.2021.125370 |
| [20] | Kowalski N, Reichardt A M, Waniek J J. Sinking rates of microplastics and potential implications of their alteration by physical, biological, and chemical factors[J]. Marine Pollution Bulletin, 2016, 109(1): 310-319. DOI:10.1016/j.marpolbul.2016.05.064 |
| [21] | Wang L F, Tong J X, Li Y, et al. Bacterial and fungal assemblages and functions associated with biofilms differ between diverse types of plastic debris in a freshwater system[J]. Environmental Research, 2021, 196. DOI:10.1016/j.envres.2020.110371 |
| [22] | Bhagwat G, Zhu Q Y, O'Connor W, et al. Exploring the composition and functions of plastic microbiome using whole-genome sequencing[J]. Environmental Science & Technology, 2021, 55(8): 4899-4913. |
| [23] | Aßhauer K P, Wemheuer B, Daniel R, et al. Tax4Fun: predicting functional profiles from metagenomic 16S rRNA data[J]. Bioinformatics, 2015, 31(17): 2882-2884. DOI:10.1093/bioinformatics/btv287 |
| [24] | Hossain M R, Jiang M, Wei Q H, et al. Microplastic surface properties affect bacterial colonization in freshwater[J]. Journal of Basic Microbiology, 2019, 59(1): 54-61. DOI:10.1002/jobm.201800174 |
| [25] | Pinto M, Langer T M, Hüffer T, et al. The composition of bacterial communities associated with plastic biofilms differs between different polymers and stages of biofilm succession[J]. PLoS One, 2019, 14(6). DOI:10.1371/journal.pone.0217165 |
| [26] | Xie H F, Chen J J, Feng L M, et al. Chemotaxis-selective colonization of mangrove rhizosphere microbes on nine different microplastics[J]. Science of the Total Environment, 2021, 752. DOI:10.1016/j.scitotenv.2020.142223 |
| [27] | Wang Q J, Wangjin X X, Zhang Y, et al. The toxicity of virgin and UV-aged PVC microplastics on the growth of freshwater algae Chlamydomonas reinhardtii[J]. Science of the Total Environment, 2020, 749. DOI:10.1016/j.scitotenv.2020.141603 |
| [28] | Seeley M E, Song B, Passie R, et al. Microplastics affect sedimentary microbial communities and nitrogen cycling[J]. Nature Communications, 2020, 11(1). DOI:10.1038/s41467-020-16235-3 |
| [29] | Dai H H, Gao J F, Wang Z Q, et al. Behavior of nitrogen, phosphorus and antibiotic resistance genes under polyvinyl chloride microplastics pressures in an aerobic granular sludge system[J]. Journal of Cleaner Production, 2020, 256. DOI:10.1016/j.jclepro.2020.120402 |
| [30] |
王琳, 马旖旎, 季荣. PS和PVC微塑料对小球藻的生长抑制效应及其影响途径[J]. 环境科学学报, 2021, 41(4): 1538-1544. Wang L, Ma Y N, Ji R. Effects of PS and PVC microplastics on the growth of Chlorella sp.[J]. Acta Scientiae Circumstantiae, 2021, 41(4): 1538-1544. |
| [31] |
王泽铭, 李传虹, 马巧丽, 等. 湿度盐度pH协同驱动锡林河景观疣微菌群空间异质性[J]. 微生物学报, 2021, 61(6): 1728-1742. Wang Z M, Li C H, Ma Q L, et al. Moisture, salinity and pH co-driving spatial heterogeneity of Verrucomicrobial populations in Xilin River landscape[J]. Acta Microbiologica Sinica, 2021, 61(6): 1728-1742. |
| [32] | Helgason B L, Konschuh H J, Bedard-Haughn A, et al. Microbial distribution in an eroded landscape: buried A horizons support abundant and unique communities[J]. Agriculture, Ecosystems & Environment, 2014, 196: 94-102. |
| [33] | Tu C, Chen T, Zhou Q, et al. Biofilm formation and its influences on the properties of microplastics as affected by exposure time and depth in the seawater[J]. Science of the Total Environment, 2020, 734. DOI:10.1016/j.scitotenv.2020.139237 |
| [34] | Zhang M J, Zhao Y R, Qin X, et al. Microplastics from mulching film is a distinct habitat for bacteria in farmland soil[J]. Science of the Total Environment, 2019, 688: 470-478. DOI:10.1016/j.scitotenv.2019.06.108 |
| [35] | Xu X Y, Wang S, Gao F L, et al. Marine microplastic-associated bacterial community succession in response to geography, exposure time, and plastic type in China's coastal seawaters[J]. Marine Pollution Bulletin, 2019, 145: 278-286. DOI:10.1016/j.marpolbul.2019.05.036 |
| [36] | Wang J, Qin X, Guo J B, et al. Evidence of selective enrichment of bacterial assemblages and antibiotic resistant genes by microplastics in urban rivers[J]. Water Research, 2020, 183. DOI:10.1016/j.watres.2020.116113 |
| [37] | Chen X C, Chen X F, Zhao Y H, et al. Effects of microplastic biofilms on nutrient cycling in simulated freshwater systems[J]. Science of the Total Environment, 2020, 719. DOI:10.1016/j.scitotenv.2020.137276 |
| [38] |
郭芝芝, 苏振华, 邸琰茗, 等. 北运河京津冀段河道浮游微生物群落多样性变化分析[J]. 环境科学, 2022, 43(2): 803-812. Guo Z Z, Su Z H, Di Y M, et al. Analysis on diversity of plankton microbial community in the Beijing-Tianjin-Hebei section of the North Canal River[J]. Environmental Science, 2022, 43(2): 803-812. |
| [39] | Mwanamoki P M, Devarajan N, Thevenon F, et al. Assessment of pathogenic bacteria in water and sediment from a water reservoir under tropical conditions (Lake Ma Vallée), Kinshasa democratic republic of Congo[J]. Environmental Monitoring and Assessment, 2014, 186(10): 6821-6830. DOI:10.1007/s10661-014-3891-6 |
| [40] |
张瑞海, 宋振, 付卫东, 等. 植被恢复对刺萼龙葵根际土壤细菌群落结构与功能的影响[J]. 环境科学, 2021, 42(1): 433-442. Zhang R H, Song Z, Fu W D, et al. Effects of vegetation restoration on the structure and function of the rhizosphere soil bacterial community of Solanum rostratum[J]. Environmental Science, 2021, 42(1): 433-442. |
| [41] |
刘坤和, 薛玉琴, 竹兰萍, 等. 嘉陵江滨岸带不同土地利用类型对土壤细菌群落多样性的影响[J]. 环境科学, 2022, 43(3). Liu K H, Xue Y Q, Zhu L P, et al. Effect of different land use types on the diversity of soil bacterial community in the coastal zone of Jialing River[J]. Environmental Science, 2022, 43(3). DOI:10.13227/j.hjkx.202106174 |
| [42] | Bhagwat G, Tran T K A, Lamb D, et al. Biofilms enhance the adsorption of toxic contaminants on plastic microfibers under environmentally relevant conditions[J]. Environmental Science & Technology, 2021, 55(13): 8877-8887. |
| [43] | Zhu L X, Zhao S Y, Bittar T B, et al. Photochemical dissolution of buoyant microplastics to dissolved organic carbon: rates and microbial impacts[J]. Journal of Hazardous Materials, 2020, 383. DOI:10.1016/j.jhazmat.2019.121065 |
| [44] | Wu X J, Pan J, Li M, et al. Selective enrichment of bacterial pathogens by microplastic biofilm[J]. Water Research, 2019, 165. DOI:10.1016/j.watres.2019.114979 |
| [45] | Han J, Gao Q X, Zhang Y G, et al. Transcriptomic and ectoine analysis of halotolerant Nocardiopsis gilva YIM 90087T under salt stress[J]. Frontiers in Microbiology, 2018, 9. DOI:10.3389/fmicb.2018.00618 |
| [46] | Lyons B P, Cowie W J, Maes T, et al. Marine plastic litter in the ROPME Sea Area: current knowledge and recommendations[J]. Ecotoxicology and Environmental Safety, 2020, 187. DOI:10.1016/j.ecoenv.2019.109839 |
 2022, Vol. 43
2022, Vol. 43


