2. 中国科学院大学, 北京 100049
2. University of Chinese Academy of Sciences, Beijing 100049, China
抗生素抗性基因(antibiotic resistance genes, ARGs)被认为是一种新型的环境污染物[1, 2], 近年来逐渐引起了世界各国科学家和各种环境健康组织的关注.与传统的化学污染物不同, 抗生素抗性基因由于其生物学特性, 可在不同细菌之间转移和传播甚至是自我扩增, 表现出独特的环境行为特征[3].虽然抗生素及抗性基因在自然环境中一直存在[4], 但是在城市污水[5]、施用有机肥的水稻土[6]、城市河水[7]、河口沉积物[8]和塑料污染土壤[9]等受人类活动影响的环境中检测出了高丰度的各类抗生素抗性基因.因此, 人类活动显著改变了环境微生物抗性基因的种类和分布, 形成了人为干扰下的微生物大迁徙[10].在医疗保健领域和畜牧养殖行业, 存在着抗生素过度和使用不当现象[11], 并可能导致人类无药可用的境地, 造成了极大的健康风险[12].越来越多的研究表明, 致病菌耐药性与环境微生物抗性和抗生素抗性基因有关[13, 14].世界卫生组织(WHO)在2018年11月12~18日开展了世界提高抗生素认识周(World Antibiotic Awareness Week)相关活动, 并把本次活动周主题定为:急需作出改变, 人们很快就没有可用的抗生素了.
生活垃圾填埋场是城市生活垃圾、废弃塑料制品、污水厂的处置污泥、医疗处理废弃物和垃圾焚烧厂飞灰残渣等固体废弃物的主要的最终堆放场所[15].受人类生产生活的影响, 这些固体垃圾中往往有抗生素残留以及含有抗生素抗性的微生物[16, 17].此外, 垃圾填埋场从开始垃圾填埋到填埋场封场后一直不停地产生渗滤液[18].垃圾渗滤液是伴随城市生活垃圾填埋场整个运营生命周期的二次污染物.填埋场垃圾渗滤液具有污染物成分复杂、生化降解性差和污染物浓度高等特点[19], 是一种人为制造的“极端环境”, 同时也是垃圾填埋场以及垃圾渗滤液中环境微生物生存进化的一种选择压力.垃圾渗滤液处理过程中微生物抗生素抗性的研究还处于起步阶段.对垃圾渗滤液处理过程中抗生素抗性基因种类、富集情况和演变机制等还不是十分清楚, 开展垃圾渗滤液抗生素抗性基因污染研究显得十分必要.
本研究采用高通量定量PCR(high through-put qPCR, HT-qPCR)技术, 对东南沿海某城市生活垃圾填埋场渗滤液在处理过程中的环境微生物抗生素抗性进行了探讨, 从环境抗生素抗性整体集合的角度出发, 对垃圾渗滤液中抗生素抗性基因的多样性、丰度、演变格局和影响因子进行了分析, 通过增加对垃圾渗滤液抗生素抗性污染的认识, 以期为进一步提高垃圾渗滤液处置管理水平和垃圾填埋场渗滤液环境健康风险评价提供理论支撑.
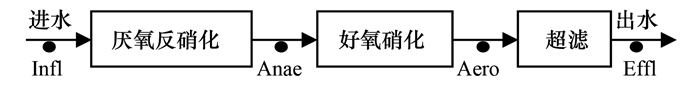
1 材料与方法 1.1 城市生活垃圾填埋场概况及垃圾渗滤液处理工艺流程该填埋场位于福建省沿海地区, 填埋场容量715万m3(含扩容), 2015年正式停止垃圾填埋, 暂未完成封场措施.该城市生活垃圾填埋场建造年代较早, 配建的垃圾渗滤液收集池和调节池为同一个池子.垃圾填埋场渗滤液处理的主要工艺如图 1所示.与近年来的渗滤液主流处理工艺相比, 该渗滤液处理工艺显得相对落后, 垃圾渗滤液日均处理量约为800 m3·d-1.渗滤液超滤出水最终直接排入市政污水收集管网.
|
图 1 渗滤液处理工艺流程示意 Fig. 1 Flow chart of the leachate treatment |
根据渗滤液处理工艺特点, 在图 1中黑点所示的工艺段, 分别采集了渗滤液进水(Infl)、厌氧反硝化(Anae)、好氧硝化(Aero)和超滤出水(Effl), 采样体积分别为20、1、1和1 000 mL, 每个工艺段进行3个采样重复(1 h内完成).采用六联抽滤设备对所采集的总计12个样品进行抽滤, 所用的滤膜为直径50 mm的0.22 μm灭菌过滤膜(ADVANTEC, 日本), 滤膜避光冷冻保存.滤膜用手术剪剪成约2 mm×2 mm的小块, 分别添加到FastDNA® Spin Kit for Soil试剂盒中的Lysing Matrix E tube, 根据试剂盒说明书提供的标准流程, 获得样品总DNA, 然后统一用100 μL DES进行洗脱, 对所获得的DNA样品进行电泳验证(1%的琼脂糖凝胶). DNA样品用QuantiFluor® dsDNA (Promega Corporation, 美国)试剂盒测定DNA浓度, 用灭菌的超纯水将DNA样品统一稀释至25 ng·μL-1, 所得到的DNA样品置于-20℃冰箱保存.
1.3 普通qPCR与超高通量PCR根据标准质粒外标法[20], 采用普通qPCR方法测定渗滤液样品16S rDNA的绝对丰度, 所用引物参照Su等[21]的研究. SmartChip Real-Time PCR Systems(WaferGen Inc., 美国)超高通量PCR系统能够同时进行5 184个PCR反应, 并且PCR体积仅为常规的1/200, 还具有DNA样品需求量少、节约试剂、自动化程度高等特点.实验中所用的296对引物简要说明如下:1对16S rDNA引物、8对转座子引物、2对整合子引物和285对抗生素抗性基因引物[21].这些抗性基因引物具有典型的代表性, 涵盖了大部分已知的抗生素抗性基因类型, 整合子和转座子引物则用于研究微生物潜在的水平基因转移情况.
超高通量PCR扩增反应体系为100 nL. PCR体系中各个试剂的最终浓度为:1×的LightCycler 480 SYBR® Green ⅠMaster Mix(Roche, 美国), 1 μg·μL-1的BSA, 3 ng·μL-1的DNA模板, 1 μmol·L-1的上下游引物.具体的反应条件为:初始温度95℃预变性10 min; 95℃变性30 s, 60℃退火延伸30 s后收集相应的荧光信号, 总计40个循环; 仪器可以自动进行熔解曲线分析, 确保PCR质量.每个SmartChip芯片都有包含了灭菌超纯水作为阴性对照样品.每个样品须进行3次技术重复PCR实验.最终PCR反应得到的数据通过Cycler预设的过滤筛选条件(PCR扩增效率, 单峰或多峰等)进行导出.
1.4 重金属元素检测与抗性基因数据分析重金属元素采用ICP-MS方法测定.超高通量定量PCR的数据采用Excel 2013进行计算整理.在对渗滤液样品进行16S rDNA常规普通qPCR定量的基础上, 参考已有的抗生素抗性的研究方法[22, 23], 计算沉积物样品抗性基因、可移动遗传元件(mobile genetic elememts, MGEs)的绝对拷贝数.根据rRNA数据库, 每个细菌细胞约含有4.1个16S rRNA基因[24], 计算归一化的抗生素抗性基因和可移动遗传元件丰度.抗生素抗性基因组成和结构差异分析采用adonis和anosim检验分析方法.抗性基因绝对丰度采用t检验方法分析组间差异.分析采用OriginPro 9.0进行常规普通作图, 采用R 3.3.3进行pheatmap热图分析, 采用SPSS Statistics 22进行相关性分析.
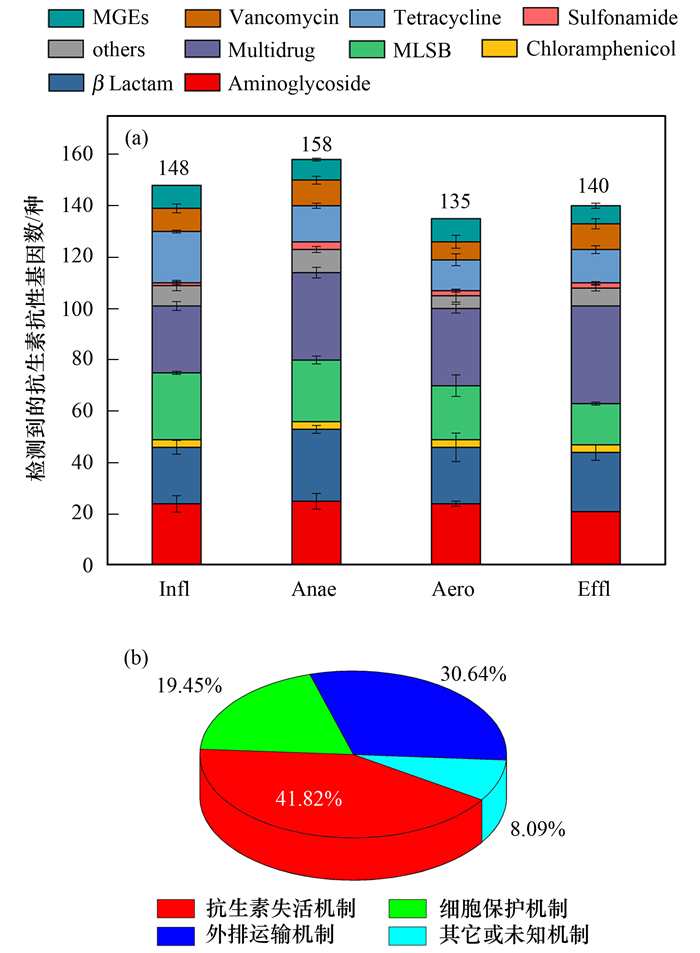
2 结果与讨论 2.1 渗滤液处理过程中抗生素抗性基因的种类渗滤液中总共检测出8大类, 192种抗生素抗性基因, 9种可移动遗传元件(MGEs), 具体结果见图 2(a).渗滤液进水(Infl)和渗滤液出水(Effl)分别检测出148和140种抗生素抗性基因, 两者抗性基因种类分布和组成没有显著差异.抗性基因定性数据表明, 渗滤液经过处理后, 仍然维持种类丰富的抗性基因, 检出率仍达46.7%. 图 2的结果表明, 厌氧反硝化阶段的抗性基因种类数显著高于好氧硝化阶段(P<0.05), 抗性基因的组成(抗性谱)也有显著差异.有研究表明, 抗生素抗性基因和厌氧微生物有比较密切的关系[25, 26], 因此厌氧阶段抗性基因种类数最高可能与厌氧微生物有关.抗生素抗性基因的种类数始终处于较高水平, 表明该渗滤液处理工艺对抗生素抗性基因种类数目消减效果相对有限, 可能对接收渗滤液出水的城市污水厂和最终的受纳水体有潜在的环境健康风险.
|
图 2 抗生素抗性基因的种类数和抗性机制 Fig. 2 Number of ARGs detected and their mechanism of resistance |
图 2(b)的结果表明, 抗生素本体失活(antibiotic deactivation)、抗生素外排泵(efllux pumps)和细菌个体保护(cellular protection)是渗滤液微生物抗生素抗性的最主要的3种机制, 比例分别为41.82%、30.64%和19.45%, 这个结果与Ouyang等[7]关于河水的研究相似, 表明环境微生物抗生素抗性机制比较接近.
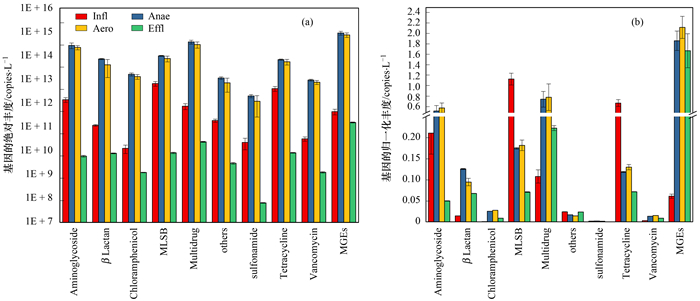
2.2 渗滤液处理过程中抗性基因的丰度由图 3可知, 渗滤液处理过程中, 各类抗生素抗性基因和可移动遗传元件在绝对丰度的水平上都呈现先上升后下降的趋势.除了出水样品, 渗滤液中各种抗生素抗性基因绝对丰度达到了1010~1015的数量级.虽然渗滤液处理后的出水中抗生素抗性基因种类数没有显著下降[图 2(a)], 但是氨基糖苷类抗生素(Aminoglycoside)、β内酰胺类抗生素(β Lactam)、氯霉素类(Chloramphenicol)、大环内酯类-林肯酰胺类-链阳性菌素B类抗生素(MLSB)、多重抗药类(Multidrug)、磺胺类抗生素(Sulfonamide)、四环素类抗生素(Tetracycline)和万古霉素类抗生素(Vancomycin)这8大类抗生素抗性基因绝对丰度都是极显著下降的(P<0.01), 表明渗滤液处理能够有效消减抗生素抗性基因.但是渗滤液处理后的出水中总的抗性基因丰度仍然高达9.2×1010 copies·L-1, 接近城市河流或者高于自然环境水体中的抗生素抗性基因[7, 27], 显示出水中的抗生素抗性基因的环境健康风险不可忽视.
|
图 3 抗生素抗性基因的丰度 Fig. 3 Abundance of antibiotic resistance genes |
有研究统计表明, 单个细菌细胞平均含有4.1个拷贝的16S rRNA基因[24], 据此计算出单个细菌携带的抗生素抗性基因的拷贝数[图 3(b)].总体上, 渗滤液进水(Infl)原样单个细菌细胞携带的总的各类抗性基因拷贝数最高.但是处理后的渗滤液出水中单个细菌的可移动遗传元件(MGEs)高达2.1个, 显著高于渗滤液进水, 表明渗滤液处理过程中可移动遗传元件发生了强烈变化.此外, 抗生素抗性基因丰度和可移动遗传元件的细胞水平拷贝数都显著高于河水[7]和污泥堆肥[21]等样品, 表明在填埋场堆体中形成的渗滤液微生物中抗生素抗性可能有自己独特的环境行为特征.
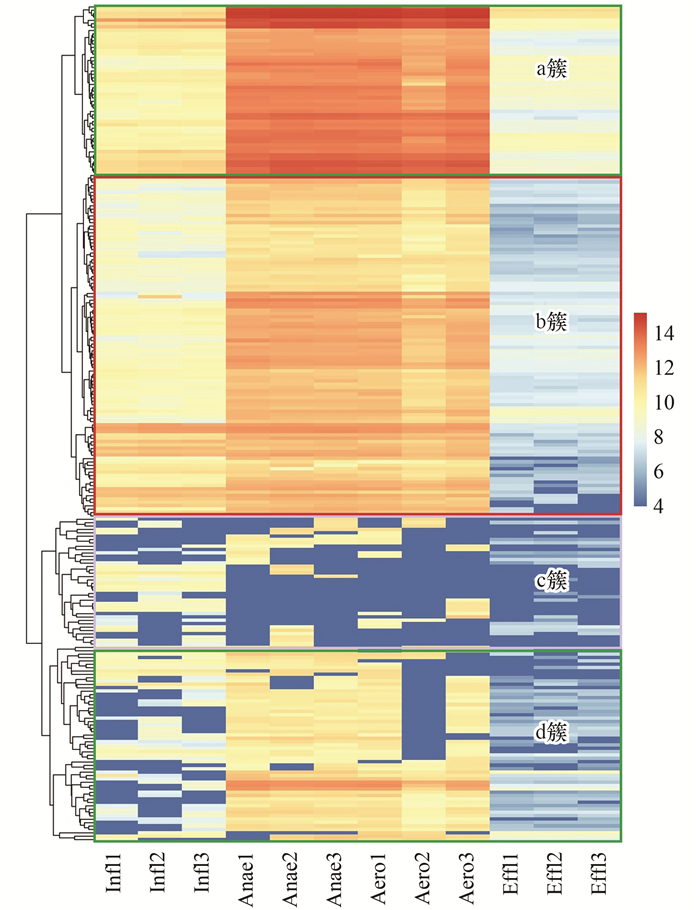
2.3 渗滤液处理过程中抗性基因分布格局聚类热图分析表明(图 4), 渗滤液处理过程中抗性基因分布格局发生了显著变化.数据结果显示, 渗滤液中的抗生素抗性基因聚类成a、b、c和d簇. a簇的基因主要有tnpA、mexF、blaTEM、blaOXY、vanC和aacC等基因, a簇的基因在所有样品中的丰度水平都处于总体较高水平. b簇是4个聚类基因簇中抗性基因种类最多的, 在渗滤液进水(Infl)、厌氧反硝化(Anae)和好氧硝化(Aero)的处理阶段, 抗生素抗性基因丰度较高, 但是在出水阶段(Effl), 抗性基因水平显著下降, 这些基因主要包含ampC、emK、tetV、oleC和ceoA等. c簇是指在整个渗滤液处理阶段, 抗生素抗性基因的检出率和绝对丰度水平都处于较低的抗性基因簇. d簇则是在厌氧反硝化(Anae)和好氧硝化(Aero)阶段, 抗性基因种类和丰度相对较高, 高于渗滤液进水; 在渗滤液出水阶段, 个别抗生素抗性基因仍然维持较高丰度.渗滤液处理过程中抗生素抗性基因分布格局的变化表明, 渗滤液处理有效改变并消减了渗滤液中的抗性基因丰度, 但是渗滤液出水中的抗生素抗性基因去向、宿主和风险等仍需开展进一步研究.
|
图 4 抗生素抗性基因分布格局 Fig. 4 Pattern of antibiotic resistance genes |
Cr、Cd、Ni和As等重金属元素的检测结果如图 5所示.针对相关重金属元素检测结果, 结合可移动遗传元件(MGEs)的绝对丰度, 进行了Pearson相关关系分析.由表 1结果可知, 各类抗生素抗性基因与Cr、Cd、Ni、As、MGEs、整合子(integron)和转座子(transposon)都呈现极显著的强相关关系, 表明重金属元素和可移动遗传元件与抗生素抗性基因可能存在着明显的共选择作用.以往的一些研究表明, 某些类型的抗生素抗性基因与某些重金属、可移动遗传元件存着的一定关系, 但是有些没有统计上的相关关系[28], 而本研究表明渗滤液抗生素抗性基因与重金属、可移动遗传元件存着显著的强相关关系, 因此渗滤液中抗性基因的传播扩散机制可能比较独特, 需采取多种手段进一步研究渗滤液的微生物学机制.
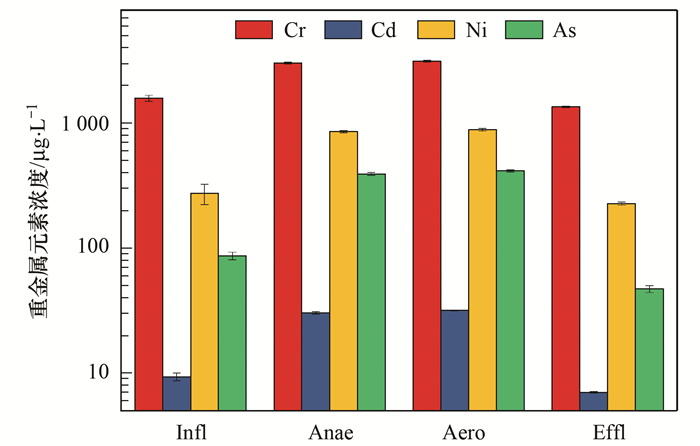
|
图 5 渗滤液中重金属元素检测结果 Fig. 5 Concentration of heavy metals in leachate |
|
|
表 1 抗生素抗性基因与重金属元素和可移动遗传元件的相关关系1) Table 1 Pearson's correlation between ARGs, metals, and MGEs |
3 结论
(1) 渗滤液处理工艺使得渗滤液出水中抗生素抗性基因丰度减至9.2×1010 copies·L-1, 降低了5个数量级, 该工艺能有效消减渗滤液中的抗生素抗性基因的丰度.
(2) 相较于自然环境, 渗滤液出水的抗生素抗性基因种类有140种, 检出率高达46.7%, 种类和丰度仍然维持在较高水平, 存在潜在的生态环境健康风险.
(3) 渗滤液中抗生素抗性基因与Cr、Cd、Ni、As、MGEs、整合子(integron)和转座子(transposon)呈现极显著的强相关关系, 抗生素抗性基因的迁移传播可能受重金属元素和可移动遗传元件的共同影响.
| [1] | Zhu Y G, Zhao Y, Li B, et al. Continental-scale pollution of estuaries with antibiotic resistance genes[J]. Nature Microbiology, 2017, 2: 16270. DOI:10.1038/nmicrobiol.2016.270 |
| [2] | Pruden A, Pei R T, Storteboom H, et al. Antibiotic resistance genes as emerging contaminants:studies in northern Colorado[J]. Environmental Science & Technology, 2006, 40(23): 7445-7450. DOI:10.1021/es060413l |
| [3] |
苏建强, 黄福义, 朱永官. 环境抗生素抗性基因研究进展[J]. 生物多样性, 2013, 21(4): 481-487. Su J Q, Huang F Y, Zhu Y G. Antibiotic resistance genes in the environment[J]. Biodiversity Science, 2013, 21(4): 481-487. |
| [4] | Martínez J L. Antibiotics and antibiotic resistance genes in natural environments[J]. Science, 2008, 321(5887): 365-367. DOI:10.1126/science.1159483 |
| [5] | Rizzo L, Manaia C, Merlin C, et al. Urban wastewater treatment plants as hotspots for antibiotic resistant bacteria and genes spread into the environment:a review[J]. Science of the Total Environment, 2013, 447: 345-360. DOI:10.1016/j.scitotenv.2013.01.032 |
| [6] |
黄福义, 李虎, 韦蓓, 等. 长期施用猪粪水稻土抗生素抗性基因污染研究[J]. 环境科学, 2014, 35(10): 3869-3873. Huang F Y, Li H, Wei B, et al. Long-term manure application induced shift of diversity and abundance of antibiotic resistance genes in paddy soil[J]. Environmental Science, 2014, 35(10): 3869-3873. |
| [7] | Ouyang W Y, Huang F Y, Zhao Y, et al. Increased levels of antibiotic resistance in urban stream of Jiulongjiang River, China[J]. Applied Microbiology and Biotechnology, 2015, 99(13): 5697-5707. DOI:10.1007/s00253-015-6416-5 |
| [8] | Chen B W, Yang Y, Liang X M, et al. Metagenomic profiles of antibiotic resistance genes (ARGs) between human impacted estuary and deep ocean sediments[J]. Environmental Science & Technology, 2013, 47(22): 12753-12760. DOI:10.1021/es403818e |
| [9] |
黄福义, 杨凯, 张子兴, 等. 微塑料对河口沉积物抗生素抗性基因的影响[J]. 环境科学, 2019, 40(5): 2234-2239. Huang F Y, Yang K, Zhang Z X, et al. Effects of microplastics on antibiotic resistance genes in estuarine sediments[J]. Environmental Science, 2019, 40(5): 2234-2239. |
| [10] | Zhu Y G, Gillings M, Simonet P, et al. Microbial mass movements[J]. Science, 2017, 357(6356): 1099-1100. DOI:10.1126/science.aao3007 |
| [11] | Cully M. Public health:the politics of antibiotics[J]. Nature, 2014, 509(7498): S16-S17. DOI:10.1038/509S16a |
| [12] | Smith R A, M'Ikanatha N M, Read A F. Antibiotic resistance:a primer and call to action[J]. Health Communication, 2015, 30(3): 309-314. DOI:10.1080/10410236.2014.943634 |
| [13] |
庄芳芳, 苏建强, 陈辉煌, 等. 基于高通量定量PCR研究城市化小流域微生物污染特征[J]. 生态毒理学报, 2017, 12(5): 141-152. Zhuang F F, Su J Q, Chen H H, et al. Characterization of microbial contaminants using high-throughput quantitative PCR in a small urbanizing catchment[J]. Asian Journal of Ecotoxicology, 2017, 12(5): 141-152. |
| [14] | Fischbach M A, Walsh C T. Antibiotics for emerging pathogens[J]. Science, 2009, 325(5944): 1089-1093. DOI:10.1126/science.1176667 |
| [15] |
黄福义, 安新丽, 李力, 等. 生活垃圾填埋场对河流抗生素抗性基因的影响[J]. 中国环境科学, 2017, 37(1): 203-209. Huang F Y, An X L, Li L, et al. High-throughput profiling of antibiotic resistance genes in river in the vicinity of a landfill[J]. China Environmental Science, 2017, 37(1): 203-209. |
| [16] | Chen Q L, Li H, Zhou X Y, et al. An underappreciated hotspot of antibiotic resistance:the groundwater near the municipal solid waste landfill[J]. Science of the Total Environment, 2017, 609: 966-973. DOI:10.1016/j.scitotenv.2017.07.164 |
| [17] | Song L Y, Li L, Yang S, et al. Sulfamethoxazole, tetracycline and oxytetracycline and related antibiotic resistance genes in a large-scale landfill, China[J]. Science of the Total Environment, 2016, 551-552: 9-15. DOI:10.1016/j.scitotenv.2016.02.007 |
| [18] |
熊建英, 郑正. 垃圾填埋场渗滤液溶解性有机质特性及其去除技术综述[J]. 环境化学, 2015, 34(1): 44-53. Xiong J Y, Zheng Z. Characteristics of the dissolved organic matter in landfill leachate and their removal technology:a review[J]. Environmental Chemistry, 2015, 34(1): 44-53. |
| [19] |
王鹤茹, 宋永伟, 徐峙辉, 等. 均相/异相催化剂光降解垃圾渗滤液效果的研究[J]. 环境科学学报, 2016, 36(9): 3243-3250. Wang H R, Song Y W, Xu Z H, et al. Photochemical degradation of landfill leachate by homogeneous/heterogeneous catalyst combinations[J]. Acta Scientiae Circumstantiae, 2016, 36(9): 3243-3250. |
| [20] |
李丽, 赵成萍, 李宏, 等. 质粒制备绝对定量PCR标准曲线方法的建立[J]. 农业生物技术学报, 2011, 19(6): 1157-1162. Li L, Zhao C P, Li H, et al. Establishment of the plasmid standard curve generation method for absolute quantification PCR[J]. Journal of Agricultural Biotechnology, 2011, 19(6): 1157-1162. DOI:10.3969/j.issn.1674-7968.2011.06.024 |
| [21] | Su J Q, Wei B, Ouyang W Y, et al. Antibiotic resistome and its association with bacterial communities during sewage sludge composting[J]. Environmental Science & Technology, 2015, 49(12): 7356-7363. |
| [22] | Chen Y H, Su J Q, Zhang J Y, et al. High-throughput profiling of antibiotic resistance gene dynamic in a drinking water river-reservoir system[J]. Water Research, 2019, 149: 179-189. DOI:10.1016/j.watres.2018.11.007 |
| [23] | Chen Q L, An X L, Zhu Y G, et al. Application of Struvite Alters the Antibiotic Resistome in Soil, Rhizosphere, and Phyllosphere[J]. Environmental Science & Technology, 2017, 51(14): 8149-8157. DOI:10.1021/acs.est.7b01420 |
| [24] | Klappenbach J A, Saxman P R, Cole J R, et al. rrndb:the ribosomal RNA operon copy number database[J]. Nucleic Acids Research, 2001, 29(1): 181-184. DOI:10.1093/nar/29.1.181 |
| [25] | Aydin S, Ince B, Ince O. Assessment of anaerobic bacterial diversity and its effects on anaerobic system stability and the occurrence of antibiotic resistance genes[J]. Bioresource Technology, 2016, 207: 332-338. DOI:10.1016/j.biortech.2016.01.080 |
| [26] | Aydin S, Ince B, Ince O. Development of antibiotic resistance genes in microbial communities during long-term operation of anaerobic reactors in the treatment of pharmaceutical wastewater[J]. Water Research, 2015, 83: 337-344. DOI:10.1016/j.watres.2015.07.007 |
| [27] |
张丹丹, 郭亚平, 任红云, 等. 福建省敖江下游抗生素抗性基因分布特征[J]. 环境科学, 2018, 39(6): 2600-2606. Zhang D D, Guo Y P, Ren H Y, et al. Characteristics of antibiotic resistance genes in downstream areas of the Aojiang River, Fujian Province[J]. Environmental Science, 2018, 39(6): 2600-2606. |
| [28] | Zhao Y, Su J Q, An X L, et al. Feed additives shift gut microbiota and enrich antibiotic resistance in swine gut[J]. Science of the Total Environment, 2018, 621: 1224-1232. DOI:10.1016/j.scitotenv.2017.10.106 |
 2019, Vol. 40
2019, Vol. 40


