抗生素抗性基因(antibiotic resistance genes, ARGs)作为一种新型环境污染物在环境研究领域受关注的程度日益增加[1, 2], 它们的主要传播方式是水平基因转移(horizontal gene trasfer, HGT).ARGs可以整合到一些可移动基因元件(mobile gene elements, MGEs; 比如质粒、整合子和转座子等)上, 进而在菌株之间进行传播[3~5].由于兽用抗生素具有防治动物疫病和促进生长的双重功效, 在养殖业中以亚治疗剂量长期添加于动物饲料中[6].有研究对施用了多年猪粪、鸡粪和牛粪的农业土壤进行了各种微观及田间实验, 发现施用粪肥后土壤ARGs的水平显著提高[7~9].此外, 也有研究证实, 畜禽粪便促进了ARGs在土壤中的横向转移.畜禽粪便中的ARGs可进一步通过食物链, 从环境向人体转移, 从而增加人类治愈细菌感染性疾病的风险, 严重威胁到人类健康[10].
堆肥不仅能够让畜禽粪便资源利用更加合理化, 还能利用堆肥过程中产生的高温有效去除ARGs, 并迅速降解粪便中残留的抗生素, 防止堆肥过程中再次诱导产生ARGs, 最终降低畜禽粪便中ARGs对生态环境造成的危害[11].在猪粪堆肥过程中, Cui等[12]研究了四环素类、磺胺类和氟喹诺酮类抗性基因, 并发现在堆肥56 d后, 被选中的目标ARGs, 除了parC, 在堆肥结束后均没有被检测到.王晓慧[13]发现在鸡粪堆肥之后, 相比于新鲜鸡粪tetA和tetC基因的含量都有所降低, 去除率分别为98.8%和69.6%.郑宁国等[14]进行了猪粪与锯末共堆肥的实验, 在堆肥结束时blaTEM去除率达99.9%, qnrS的去除率达99.7%.Cui等[12]将水稻秸秆与蘑菇渣作为堆肥填充料添加到鸡粪堆肥过程中, 在实验结束时, ARGs的平均去除率为86%, 比不添加填充料的对照组有更高的去除率.
随着我国医药行业的发展, 中药厂在生产过程中产生大量中药废渣.中药渣的传统处理方式如焚烧、填埋、固定区域堆放等不仅要投入大量资金, 而且也造成了资源浪费以及环境污染[15, 16].有研究发现中药渣成分复杂, 存在一定的活性物质, 是一种无公害有机肥料[17, 18].而堆肥化处理成本较低、无害化程度较高, 利用好氧高温堆肥技术处理中药渣是一种可行途径[16, 19].鸡粪在畜禽粪便中占有较大比例, 且鸡粪中ARGs的相对丰度高于猪粪[20], 目前针对鸡粪堆肥中ARGs变化的研究相对较少, 因此, 本实验将鸡粪与中药渣进行高温好氧共堆肥, 采用实时荧光定量PCR法检测堆肥过程中不同堆肥阶段样品中ARGs和MGEs的丰度, 探究添加中药渣的高温好氧堆肥对不同ARGs的影响, 寻求一种更加高效的畜禽粪便中ARGs的去除方法.
1 材料与方法 1.1 实验设计本实验所用鸡粪取自山西省太原市畜牧养殖场, 中药渣来自制药厂废渣.堆肥实验在矩形泡沫箱中进行(53 cm×37 cm×27 cm), 箱体四壁上分别留一个小孔(d=2 cm), 用于通风.鸡粪与中药渣按照体积比堆置, 设置3个重复, 调节碳氮比为25, 含水量控制在60%左右, 定期进行翻堆通风, 自然堆置46 d.每天于上午08:00使用温度计测量堆体中心的温度, 同时记录当天气温.
1.2 样品采集以第0 d收集的样品作为对照, 在堆肥的第3、7、14、28和46 d进行取样(取样时间对应堆肥的不同阶段, 即升温阶段、高温阶段、降温阶段和腐熟阶段), 取样点位于箱体四周和中间, 将样品混匀后存放于-80℃为后续实验用.
1.3 DNA提取样品的基因组提取使用E. Z. N. A® Soil DNA Kit(Omega Bio-Tek, USA), 按照生产商的说明操作, 用多功能读版机Infinite 200 PRO(TECAN, Sweden)测定DNA浓度和纯度.每个样品提取3份, 然后将3份DNA提取物合并在一起于-20℃保存待用.
1.4 实时荧光定量PCR在检测的100种ARGs中共检测出21种, 其中包括6种常见的四环素类抗性基因(tetG、tetH、tetM、tetQ、tetR和tetW), 4种氨基糖苷类抗性基因(aacA/aphD、aacC 2、aadE和aphA3 ), 4种大环内酯-林可酰胺类抗性基因(lnuA-01 、lnuB-01 、lnuB-02 和mel), 4种β-内酰胺类抗性基因(blaPSE、blaTEM、blaOXA1 和blaOXA1/blaOXA30 ), 1种喹诺酮类抗性基因(qnrD), 2种磺胺类抗性基因(sulⅠ 和sulⅢ )以及2种整合酶基因(intI1和intI2)和3种转座酶基因(tnpA-01 、tnpA-02 和tnpA-03 ).同时对细菌16S rRNA基因进行qPCR检测.这些ARGs在qPCR检测中所用引物序列、目标基因片段大小及退火温度主要来源于已发表的文献.含有这些特定基因的质粒由浙江天科生物技术有限公司(浙江, 中国)生产, 作为qPCR标准曲线10倍稀释的标准品, 并以无菌水作为阴性对照.将标准品与待测样品一起在StepOnePlusTM实时荧光定量PCR仪(Life Technologies, USA)上进行qPCR检测.所有样品3个平行, 最终计算平均值.
qPCR反应体系采用10 μL体系, 包括TB Green Premix Ex Taq Ⅱ(Tli RNaseH Plus)(2×)5 μL, 上下游引物各0.4 μL, ROX Reference Dye(50×)0.2μL, DNA Sample, 1μL, ddH2O, 3μL.反应热循环步骤如下: ①95℃, 30s, 1 cycle; ②95℃, 5s, 40cycles; ③60℃, 30s, 40cycles.CT检测限为40个循环, 通过熔解曲线和凝胶电泳验证引物特异性.
1.5 数据分析数据采用Microsoft Excel 2013和SPSS 13.0进行处理和分析, 作图采用Microsoft Excel 2013、SigmaPlot 14.0、R 3.5.1及Gephi等完成.用Pearson线性相关分析确定MGEs和ARGs丰度之间是否具有显著相关性, 用单因素方差分析(one-way ANOVA)对MGEs不同天数之间的差异性进行分析, 用线性方程模拟堆肥过程中ARGs丰度随着堆肥天数的变化情况.
2 结果与分析 2.1 堆肥过程中温度变化图 1为堆肥过程中堆体及环境温度变化.在堆置前4 d, 堆体处于升温阶段, 堆体连续8 d的温度达到55℃以上, 达到无害化腐熟标准.堆体从第12 d开始温度低于50℃, 33 d之后堆体温度趋于稳定在16℃左右, 略高于环境温度.
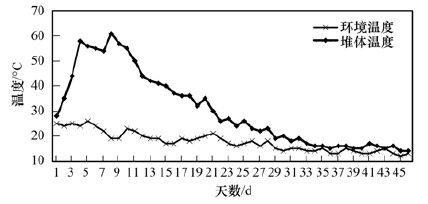
|
图 1 堆肥过程中温度变化 Fig. 1 Change of temperature during composting |
堆肥过程中MGEs的相对丰度变化及差异性比较如图 2所示.在初始样品中整合酶基因intⅠ2的相对丰度高于intⅠ1, 转座酶基因中tnpA-03 的相对丰度最高.堆肥结束后整合酶基因(intⅠ1和intⅠ2)与转座酶基因(tnpA-01 、tnpA-02 和tnpA-03 )的相对丰度都得到有效降低, 其中tnpA-01 减少了两个数量级.intⅠ1和tnpA-03 的第0、3 d和第7、14、28、46 d具有显著性差异(P<0.05).除tnpA-01 外, 在堆肥过程中第7、14、28 d和46 d之间没有显著性差异.intⅠ1和tnpA-01 在堆肥过程中表现出相似的趋势, 在第3 d其相对丰度值升高, 随后其相对丰度值逐渐下降.转座酶基因tnpA-01 和tnpA-02 分别在堆肥第28 d和第14 d降到最低.
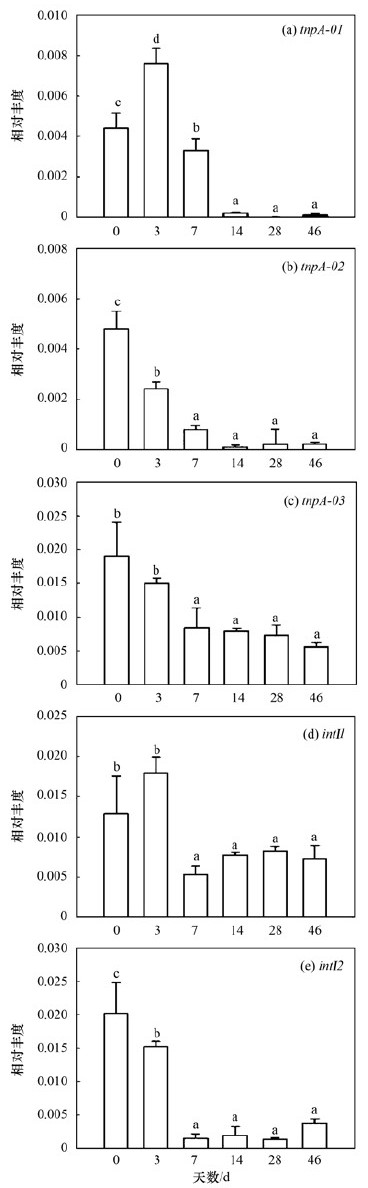
|
数值为3个重复的平均值, 竖线显示的是标准偏差, 相同字母表明处理之间差异不显著(P<0.05) 图 2 堆肥过程中MGEs相对丰度的变化 Fig. 2 Variations of the relative abundances of MGEs during co-composting |
在对照鸡粪鲜样中, 检测到的ARGs相对丰度如图 3所示.其中相对丰度最高的ARGs是tetM(0.148 4), 丰度最低的为sulⅢ (0.001 8).这些ARGs按照分子机制主要分为三大类, 包括细胞保护机制、泵外排机制和抗生素失活机制, 三类机制中抗生素失活类机制占比最高.
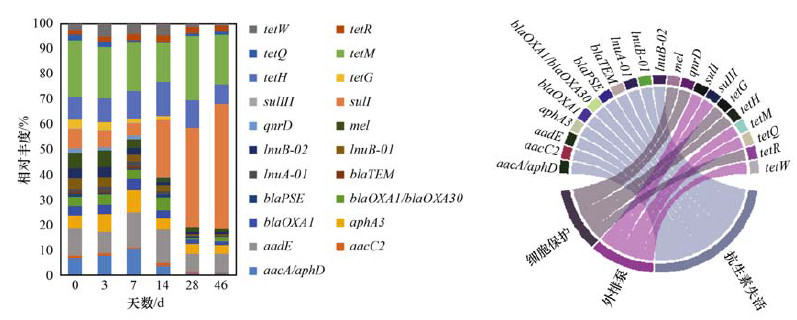
|
图 3 堆肥过程中ARGs相对丰度和作用机制 Fig. 3 Relative abundance and the mechanism of ARGs during co-composting |
在堆肥结束后添加中药渣的各类ARGs的相对丰度都有明显降低, 并且在堆肥过程中整体上是随着堆肥天数呈线性式降低(除sulⅠ ), 见图 4~8.氨基糖苷类抗性基因整体呈现先增长后降低的趋势(图 4), aphA3基因在第3 d出现突然增长, 第14 d又急剧下降.其中aacA/aphD的去除率最高, 达到93.83%.aacA/aphD和aadE在堆肥天数内呈显著性线性减少(R2=0.698 3, P=0.038, R2=0.792 0, P=0.018).
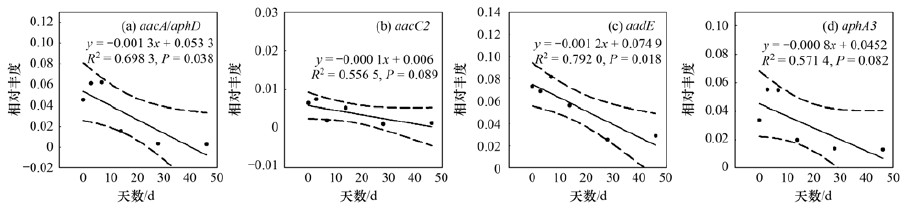
|
虚线表示95%置信区间, 下同 图 4 堆肥过程中氨基糖苷类抗性基因相对丰度的变化 Fig. 4 Variations in the relative abundances of aminoglycosides during co-composting |
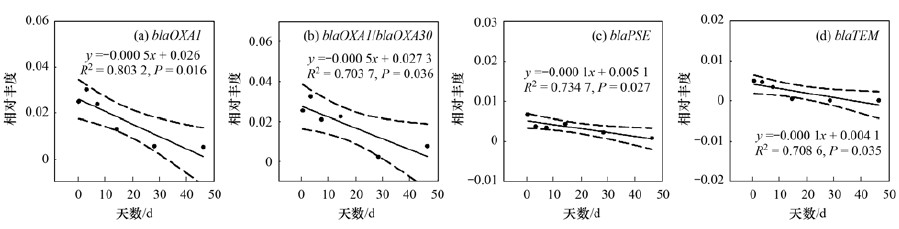
|
图 5 堆肥过程中β-内酰胺类抗性基因相对丰度的变化 Fig. 5 Variations in the relative abundances of beta lactamases during co-composting |
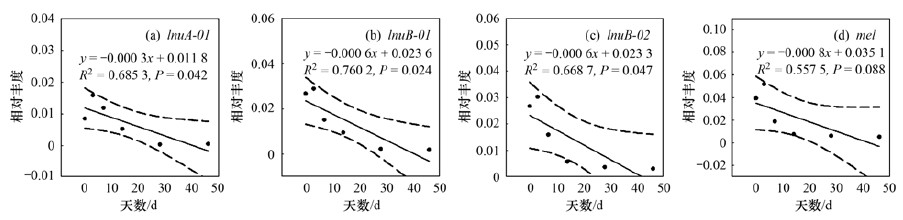
|
图 6 堆肥过程中大环内酯-林可酰胺类抗性基因相对丰度的变化 Fig. 6 Variations in the relative abundances of macrolide-lincomamides during co-composting |
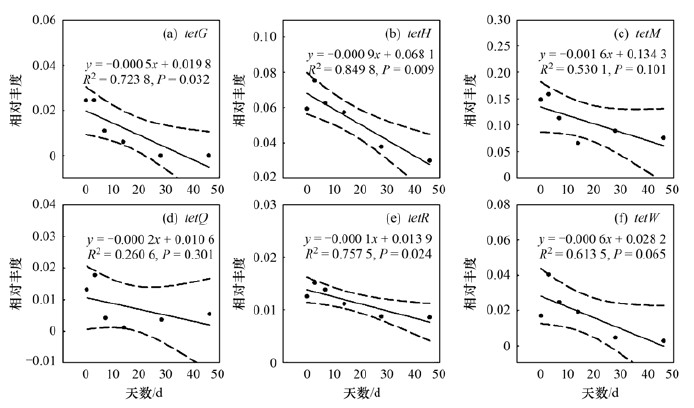
|
图 7 堆肥过程中四环素类抗性基因相对丰度的变化 Fig. 7 Variations in the relative abundances of tetracyclines during co-composting |
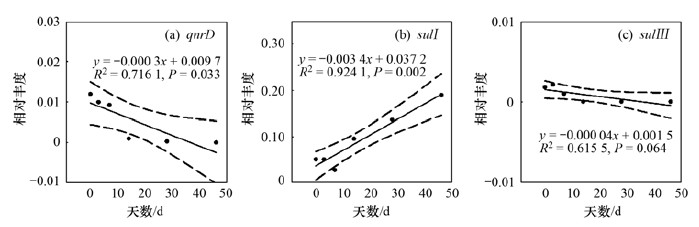
|
图 8 堆肥过程中喹诺酮类和磺胺类抗性基因相对丰度的变化 Fig. 8 Variations in the relative abundances of quinolones and sulfonamides during co-composting |
β-内酰胺类抗性基因整体呈线性减少(图 5).就增减趋势而言, blaOXA1 和blaOXA1/blaOXA30 基因的增长趋势相同, 都是在第3 d出现增长, 随后逐渐降低.ARGs随堆肥天数变化拟合程度最佳的是blaOXA1 (R2=0.803 2, P=0.016), 去除率最高的是blaTEM, 达到97.02%.
大环内酯-林可酰胺类抗性基因在堆肥结束后整体减少, 去除率最高的是lnuA-01 , 达到94.48%.mel基因在堆肥结束后相对丰度减少了一个数量级(图 6), 但是并没有表现出显著性的线性下降趋势(R2=0.557 5, P=0.088).
四环素类抗性基因在堆肥天数内其相对丰度随时间的线性关系如图 7所示.在检出的6种四环素类抗性基因中tetG、tetH、tetR和tetW在堆肥过程中表现出相似的变化, 在第3 d短暂的增长后又逐渐降低.而tetM和tetQ在第28 d再次出现了增长, 其中tetH下降的线性拟合程度最优(R2=0.849 8, P=0.009), 但其去除率仅为49.60%.tetM检出时其相对丰度最高(0.148 4), 但去除率最低仅为49.11%, tetG的去除率最高, 达到99.77%.
堆肥过程中喹诺酮类和磺胺类抗性基因相对丰度随堆肥天数的变化如图 8所示.样品中检出的sulⅠ 的相对丰度值(0.051 3)高于sulⅢ (0.001 8), 随着堆肥天数的增加其相对丰度呈线性增加的趋势(R2=0.924 1, P=0.002), 堆肥结束后其相对丰度值达到最大(0.189 5), 而sulⅢ 在堆肥结束时得到有效去除, 去除率为99.88%.喹诺酮类抗性基因(qnrD)在堆肥期间呈线性下降(R2=0.716 1, P=0.033), 其相对丰度降低了3个数量级, 去除率达99.89%, 是检测到的ARGs中去除率最高的.
2.4 ARGs与MGEs的相关性分析抗生素抗性基因和可移动基因元件之间的相关关系如表 1所示.在结果检测到的6类ARGs中, tnpA-01 与氨基糖苷类、β-内酰胺类和喹诺酮类抗性基因具有显著正相关(P < 0.05), 与大环内酯-林可酰胺类和四环素类抗性基因具有极显著正相关(P < 0.01).tnpA-02 只与喹诺酮类具有显著正相关(P < 0.05).intⅠ1和tnpA-03 与几类抗性基因之间不具有相关性.氨基糖苷类和β-内酰胺类与其他类别的抗性基因之间具有显著相关性.β-内酰胺类与大环内酯-林可酰胺类及四环素类抗性基因之间呈现极显著正相关(P < 0.01), 与磺胺类呈显著负相关(P < 0.05).
|
|
表 1 抗生素抗性基因和可移动基因元件之间的相关关系1) Table 1 Correlations among the abundance of antibiotic resistant genes and mobile gene elements |
抗生素抗性基因之间共选择模式网络分析的结果包括了20个节点, 44条边(图 9).同时表明不仅相同种类ARGs之间存在显著相关性, 不同种类之间也存在显著相关性.β-内酰胺类抗性基因不仅自身存在相关性, 如blaOXA1/blaOXA30 与blaTEM具有显著相关性(P < 0.05), 它也与大环内酯-林可酰胺类、磺胺类和四环素类抗性基因具有相关性, 如blaOXA1 与tetG、tetR、sulⅢ 及lnuB-02 都具有显著相关性(P < 0.05).
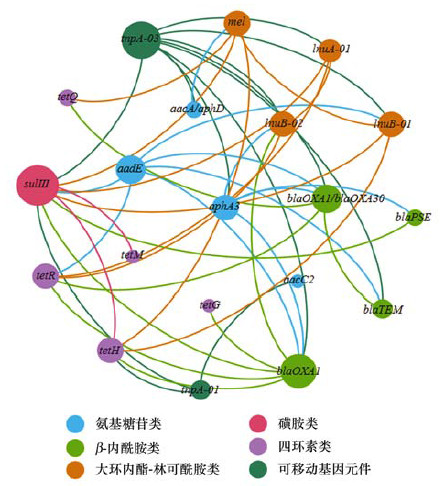
|
不同颜色的节点代表不同类型的抗性基因, 而边缘表示相邻节点之间显著性的相互关系; 每个节点的大小与节点间显著相关性的数量成正比 图 9 抗生素抗性基因之间共选择模式的网络分析 Fig. 9 Network analysis showing the co-occurrence patterns among the ARGs |
本实验表明在添加了中药渣的鸡粪堆肥后, ARGs与MGEs(除sulⅠ )的相对丰度都减少, 这可能是因为中药渣的添加消除了一些ARGs的潜在宿主细菌.相关研究表明加入中药渣的污泥堆肥, 在堆肥结束时, 堆体中的微生物群落发生了很大变化[21], 也有研究证实细菌群落的变化与ARGs的变化密切相关[22].有研究发现高温对可移动基因元件整合子等有消减作用[23, 24].堆肥过程中部分MGEs的丰度降低可能是因为堆肥过程产生的高温对其产生了影响.Zhang等[25]的研究发现intI1在污泥堆肥中占主导地位, 并且在堆肥的最后阶段, intI1的丰度显著增加.Zhang等[26]在添加不同表面活性剂的鸡粪堆肥后intI1的丰度降低了两倍.在本实验中, intI1的丰度在堆肥结束时也减少, 但是没有转座酶基因减少得多.
Wright[27]认为ARGs的变化与其不同的机制和不同的环境压力有关, 这可以用来解释不同种类ARGs在不同堆肥阶段的变化趋势不同.高温有效地减少了ARGs的丰度.在本实验中, 喹诺酮类抗性基因的去除率高达99.89%, 是检测到的ARGs中去除率最高的.有研究表明高温堆肥对鸡粪中喹诺酮类抗性基因去除效果最好, 对部分抗性基因的去除效果有待提高[28].Selvam等[29]研究了高温堆肥过程对于四环素类及磺胺类抗性基因的影响, 发现四环素类抗性基因tetW及intI1在堆肥过程中出现了增殖, 而本实验tetW的去除率达到83.95%, 这可能是在添加了中药渣之后, 对tetW的宿主菌产生了影响.在畜禽养殖中不同的饲料等因素也会对ARGs产生影响, Li等[30]的研究表明ARGs的丰度与土壤中残留的抗生素浓度具有强烈的相关性.崔二苹[31]在鸡粪肥堆肥过程中, 发现抗生素抗性基因的增减与致病菌的增减顺序一致, 这些致病菌上可能携带着某些特定的抗生素抗性基因.在堆肥过程中的高温阶段能杀死一些ARGs低温宿主菌, 很多病原菌都是ARGs的携带者, 但是他们都不能在高温下生存[22, 32], 而环境温度也会对ARGs的消除产生影响.鸡粪与中药渣共堆肥后ARGs丰度降低的另一个原因可能是在堆肥过程中抗生素大量降解, 之前有研究表明, 残留抗生素对于ARGs的多样性及丰度的提高具有潜在的促进作用[33].sulⅠ 的含量高于四环素类抗性基因, 这与Cui等[12]的研究一致.也有其他研究表明, 磺胺类抗性基因在鸡粪中的含量高于其他粪便, 说明鸡粪可能是磺胺类抗性基因的一个储存库.sulⅠ 的丰度显著高于sulⅢ , 这是因为sulⅠ 的宿主范围广, 并且sulⅠ 基因位于intⅠ1的3′端保守区域内, 这种联系可能有利于sulⅠ 基因在自然环境中的存在[34, 35].
ARGs之间的相关性表明, 这些基因可能携带某些特定的微生物种群或者某些特定的MGEs[36, 37].本实验结果显示氨基糖苷类与磺胺类呈显著负相关(P<0.01), 这可能跟他们的宿主菌间存在竞争或抑制作用有关[38].tnpA-01 与四环素类抗性基因之间的显著相关性在Zhu等[39]的研究结果中也出现.在堆肥结束后, 转座酶基因tnpA-01 的丰度降低了两个数量级, tnpA-02 降低了一个数量级, 由此可见, 转座子可能是影响堆肥前后ARGs差异的一个重要因素.intI1广泛存在于革兰氏阳性细菌中, 有研究发现在没有抗生素的选择压力下intI1的检出率很低[40].Wang等[41]的研究表明, intI1与所研究的抗生素抗性基因(tetG、tetW、sulⅠ 和sulⅡ )显著相关, 说明intI1在ARGs横向转移起到非常重要的作用, 也进一步证明了Ⅰ类整合子是在ARGs传播中的一个重要途径[42].但是, 本实验中Ⅰ类整合子和ARGs之间并没有表现出显著相关性, 而tnpA-01 与ARGs表现出显著相关性.也有研究发现ARGs与Ⅰ类整合子之间并没有相关性, 这个结果表明ARGs的横向转移潜能是非常低的.所以探究在粪肥中两者的关系以及进一步了解ARGs的转移机制, 是今后研究的一个方向.有研究发现ARGs与非同源ARGs之间存在一定的相关性, 一个整合子可以通过位点特异性重组捕获多个外源基因盒, 从而使多种抗性基因表现出共现现象[43].Li等[44]发现氯霉素抗性基因catB3 与磺胺类抗性基因dfrA1 共现概率达100%.共选择网络分析表明, 多种ARGs(同源或非同源)在环境中可能常常同时出现, blaOXA1/blaOXA30 与其他ARGs的关系最密切, 它们可能存在于相同的整合子-基因盒或宿主菌中, 这为将其中某种ARGs作为指示基因来预测其他相关基因的丰度提供了可能性.
4 结论(1) 鸡粪与中药渣共堆肥结束后, 有效降低了整合酶基因intI1和intI2, 转座酶基因tnp-01 、tnpA-02 和tnpA-03 的相对丰度.
(2) 堆肥结束后检测到的抗生素抗性基因(除sulⅠ )的相对丰度普遍减少, 其中blaTEM、qnrD、sulⅢ 和tetG的去除率在95%以上, aadE、tetH、tetM和tetR的去除率较低.
(3) 转座酶基因tnpA-01 与抗生素抗性基因具有显著相关性, 不同抗生素抗性基因之间也具有相关性.
| [1] | Pruden A, Pei R T, Storteboom H, et al. Antibiotic resistance genes as emerging contaminants:studies in northern Colorado[J]. Environmental Science & Technology, 2006, 40(23): 7445-7450. |
| [2] | Knapp C W, Engemann C A, Hanson M L, et al. Indirect evidence of transposon-mediated selection of antibiotic resistance genes in aquatic systems at low-level oxytetracycline exposures[J]. Environmental Science & Technology, 2008, 42(14): 5348-5353. |
| [3] | Ash R J, Mauck B, Morgan M. Antibiotic resistance of gram-negative bacteria in rivers, United States[J]. Emerging Infectious Diseases, 2002, 8(7): 713-716. DOI:10.3201/eid0807.010264 |
| [4] | Gogarten J P, Townsend J P. Horizontal gene transfer, genome innovation and evolution[J]. Nature Reviews Microbiology, 2005, 3(9): 679-687. DOI:10.1038/nrmicro1204 |
| [5] | Fouace J. Transfer of resistance plasmids in staphylococcus aureus[J]. Annales de Microbiologie, 1974, 125(4): 517-520. |
| [6] |
罗义, 周启星. 抗生素抗性基因(ARGs)——一种新型环境污染物[J]. 环境科学学报, 2008, 28(8): 1499-1505. Luo Y, Zhou Q X. Antibiotic resistance genes (ARGs) as emerging pollutants[J]. Acta Scientiae Circumstantiae, 2008, 28(8): 1499-1505. |
| [7] | Marti R, Scott A, Tien Y C, et al. Impact of manure fertilization on the abundance of antibiotic-resistant bacteria and frequency of detection of antibiotic resistance genes in soil and on vegetables at harvest[J]. Applied and Environmental Microbiology, 2013, 79(18): 5701-5709. DOI:10.1128/AEM.01682-13 |
| [8] | Udikovic-Kolic N, Wichmann F, Broderick N A, et al. Bloom of resident antibiotic-resistant bacteria in soil following manure fertilization[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(42): 15202-15207. DOI:10.1073/pnas.1409836111 |
| [9] | Seyfried E E, Newton R J, Rubert K F, et al. Occurrence of tetracycline resistance genes in aquaculture facilities with varying use of oxytetracycline[J]. Microbial Ecology, 2010, 59(4): 799-807. DOI:10.1007/s00248-009-9624-7 |
| [10] |
周启星, 罗义, 王美娥. 抗生素的环境残留、生态毒性及抗性基因污染[J]. 生态毒理学报, 2007, 2(3): 243-251. Zhou Q X, Luo Y, Wang M E. Environmental residues and ecotoxicity of antibiotics and their resistance gene pollution:a review[J]. Asian Journal of Ecotoxicology, 2007, 2(3): 243-251. |
| [11] |
邹威, 罗义, 周启星. 畜禽粪便中抗生素抗性基因(ARGs)污染问题及环境调控[J]. 农业环境科学学报, 2014, 33(12): 2281-2287. Zou W, Luo Y, Zhou Q X. Pollution and environmental regulation of antibiotic resistance genes(ARGs) in livestock manure[J]. Journal of Agro-Environment Science, 2014, 33(12): 2281-2287. |
| [12] | Cui E P, Wu Y, Zuo Y R, et al. Effect of different biochars on antibiotic resistance genes and bacterial community during chicken manure composting[J]. Bioresource Technology, 2016, 203: 11-17. DOI:10.1016/j.biortech.2015.12.030 |
| [13] |
王晓慧.禽畜粪便堆肥过程中耐药基因变化的研究[D].上海: 华东师范大学, 2017.8-12. Wang X H. Study on the change of ARGs in composting of livestock and poultry manure[D]. Shanghai: East China Normal University, 2017.8-12. http://cdmd.cnki.com.cn/Article/CDMD-10269-1017075897.htm |
| [14] |
郑宁国, 黄南, 王卫卫, 等. 高温堆肥过程对猪粪来源抗生素抗性基因的影响[J]. 环境科学, 2016, 37(5): 1986-1992. Zheng N G, Huang N, Wang W W, et al. Effects of thermophilic composting on antibiotic resistance genes (ARGs) of swine manure source[J]. Environmental Science, 2016, 37(5): 1986-1992. |
| [15] |
杨冰, 丁斐, 李伟东, 等. 中药渣综合利用研究进展及生态化综合利用模式[J]. 中草药, 2017, 48(2): 377-383. Yang B, Ding F, Li W D, et al. Research progress on comprehensive utilization of Chinese medicine residue and ecological comprehensive utilization pattern[J]. Chinese Traditional and Herbal Drugs, 2017, 48(2): 377-383. |
| [16] |
王引权, 吴小琴, 朱田田, 等. 中药固渣堆肥利用研究[J]. 中药材, 2008, 31(11): 1622-1624. Wang Y Q, Wu X Q, Zhu T T, et al. Study on utilization of solid slag compost of Chinese medicinal herbal[J]. Journal of Chinese Medicinal Materials, 2008, 31(11): 1622-1624. |
| [17] |
张红刚, 滕婧, 李顺祥, 等. 资源节约-中药渣高效综合利用研究进展[J]. 广州化工, 2013, 41(12): 16-18. Zhang H G, Teng J, Li S X, et al. Research progress of comprehensive utilization of Chinese medicine residue for resources conservation[J]. Guangzhou Chemical Industry, 2013, 41(12): 16-18. |
| [18] |
肖瑛琼, 叶发兵. 中药渣生物有机肥的制备及检测[J]. 湖北师范大学学报:自然科学版, 2017, 37(3): 37-42. Xiao Y Q, Ye F B. Preparation and analysis of biological organic fertilizer with the reside of Chinese medicine[J]. Journal of Hubei Normal University (Natural Science), 2017, 37(3): 37-42. |
| [19] |
王引权, Schuchardt F, 陈晖, 等. 翻堆频率对中药渣堆肥过程及其理化性质的影响[J]. 中国农学通报, 2012, 28(29): 247-252. Wang Y Q, Schuchardt F, Chen H, et al. Effects of turning frequency on chemical and physical properties in windrow composting of spent Chinese herbal medicine[J]. Chinese Agricultural Science Bulletin, 2012, 28(29): 247-252. |
| [20] | Han X M, Hu H W, Chen Q L, et al. Antibiotic resistance genes and associated bacterial communities in agricultural soils amended with different sources of animal manures[J]. Soil Biology and Biochemistry, 2018, 126: 91-102. DOI:10.1016/j.soilbio.2018.08.018 |
| [21] |
宿程远, 郑鹏, 阮祁华, 等. 中药渣与城市污泥好氧共堆肥的效能[J]. 环境科学, 2016, 37(10): 4062-4068. Su C Y, Zhen P, Ruan Q H, et al. Efficiency of aerobic co-composting of urban sludge and Chinese medicinal herbal residues[J]. Environmental Science, 2016, 37(10): 4062-4068. |
| [22] | Qian X, Sun W, Gu J, et al. Reducing antibiotic resistance genes, integrons, and pathogens in dairy manure by continuous thermophilic composting[J]. Bioresource Technology, 2016, 220: 425-432. DOI:10.1016/j.biortech.2016.08.101 |
| [23] | Ghosh S, Ramsden S J, LaPara T M. The role of anaerobic digestion in controlling the release of tetracycline resistance genes and class 1 integrons from municipal wastewater treatment plants[J]. Applied Microbiology and Biotechnology, 2009, 84(4): 791-796. DOI:10.1007/s00253-009-2125-2 |
| [24] | Ma Y J, Wilson C A, Novak J T, et al. Effect of various sludge digestion conditions on sulfonamide, macrolide, and tetracycline resistance genes and class I integrons[J]. Environmental Science & Technology, 2011, 45(18): 7855-7861. |
| [25] | Zhang J Y, Chen M X, Sui Q W, et al. Impacts of addition of natural zeolite or a nitrification inhibitor on antibiotic resistance genes during sludge composting[J]. Water Research, 2016, 91: 339-349. DOI:10.1016/j.watres.2016.01.010 |
| [26] | Zhang L, Gu J, Wang X J, et al. Behavior of antibiotic resistance genes during co-composting of swine manure with Chinese medicinal herbal residues[J]. Bioresource Technology, 2017, 244: 252-260. DOI:10.1016/j.biortech.2017.07.035 |
| [27] | Wright G D. The antibiotic resistome:the nexus of chemical and genetic diversity[J]. Nature Reviews Microbiology, 2007, 5(3): 175-186. DOI:10.1038/nrmicro1614 |
| [28] | Yang B, Meng L, Xue N D. Removal of five fluoroquinolone antibiotics during broiler manure composting[J]. Environmental Technology, 2018, 39(3): 373-381. DOI:10.1080/09593330.2017.1301568 |
| [29] | Selvam A, Xu D L, Zhao Z Y, et al. Fate of tetracycline, sulfonamide and fluoroquinolone resistance genes and the changes in bacterial diversity during composting of swine manure[J]. Bioresource Technology, 2012, 126: 383-390. DOI:10.1016/j.biortech.2012.03.045 |
| [30] | Li J J, Xin Z H, Zhang Y Z, et al. Long-term manure application increased the levels of antibiotics and antibiotic resistance genes in a greenhouse soil[J]. Applied Soil Ecology, 2017, 121: 193-200. DOI:10.1016/j.apsoil.2017.10.007 |
| [31] |
崔二苹.生物质炭对粪肥堆肥过程中抗生素抗性基因行为特征的影响[D].杭州: 浙江大学, 2016.38-43. Cui E P. The effect of biochar addition on antibiotic resistance genes during manure composting[D]. Hangzhou: Zhejiang University, 2016.38-43. http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=Y2980895 |
| [32] | Erickson M C, Liao J, Boyhan G, et al. Fate of manure-borne pathogen surrogates in static composting piles of chicken litter and peanut hulls[J]. Bioresource Technology, 2010, 101(3): 1014-1020. DOI:10.1016/j.biortech.2009.08.105 |
| [33] | Fang H, Wang H F, Cai L, et al. Prevalence of antibiotic resistance genes and bacterial pathogens in long-term manured greenhouse soils as revealed by metagenomic survey[J]. Environmental Science & Technology, 2015, 49(2): 1095-1104. |
| [34] | Sköld O. Sulfonamide resistance:mechanisms and trends[J]. Drug Resistance Updates, 2000, 3(3): 155-160. DOI:10.1054/drup.2000.0146 |
| [35] | Partridge S R, Tsafnat G, Coiera E, et al. Gene cassettes and cassette arrays in mobile resistance integrons[J]. FEMS Microbiology Reviews, 2009, 33(4): 757-784. DOI:10.1111/j.1574-6976.2009.00175.x |
| [36] | Hu H W, Wang J T, Li J, et al. Field-based evidence for copper contamination induced changes of antibiotic resistance in agricultural soils[J]. Environmental Microbiology, 2016, 18(11): 3896-3909. DOI:10.1111/1462-2920.13370 |
| [37] | Han X M, Hu H W, Shi X Z, et al. Impacts of reclaimed water irrigation on soil antibiotic resistome in urban parks of Victoria, Australia[J]. Environmental Pollution, 2016, 211: 48-57. DOI:10.1016/j.envpol.2015.12.033 |
| [38] | Wang F H, Qiao M, Chen Z, et al. Antibiotic resistance genes in manure-amended soil and vegetables at harvest[J]. Journal of Hazardous Materials, 2015, 299: 215-221. DOI:10.1016/j.jhazmat.2015.05.028 |
| [39] | Zhu Y G, Johnson T A, Su J Q, et al. Diverse and abundant antibiotic resistance genes in Chinese swine farms[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(9): 3435-3440. DOI:10.1073/pnas.1222743110 |
| [40] | Rosser S J, Young H K. Identification and characterization of class 1 integrons in bacteria from an aquatic environment[J]. Journal of Antimicrobial Chemotherapy, 1999, 44(1): 11-18. |
| [41] | Wang F H, Qiao M, Lv Z E, et al. Impact of reclaimed water irrigation on antibiotic resistance in public parks, Beijing, China[J]. Environmental Pollution, 2014, 184: 247-253. DOI:10.1016/j.envpol.2013.08.038 |
| [42] | Gillings M R, Krishnan S, Worden P J, et al. Recovery of diverse genes for class 1 integron-integrases from environmental DNA samples[J]. FEMS Microbiology Letters, 2008, 287(1): 56-62. DOI:10.1111/fml.2008.287.issue-1 |
| [43] | He L Y, Liu Y S, Su H C, et al. Dissemination of antibiotic resistance genes in representative broiler feedlots environments:Identification of indicator ARGs and correlations with environmental variables[J]. Environmental Science & Technology, 2014, 48(22): 13120-13129. |
| [44] | Li X L, Shi L, Yang W Q, et al. New array of aacA4-catB3-dfrA1 gene cassettes and a noncoding cassette from a class-1-integron-positive clinical strain of Pseudomonas aeruginosa[J]. Antimicrobial Agents and Chemotherapy, 2006, 50(6): 2278-2279. DOI:10.1128/AAC.01378-05 |
 2019, Vol. 40
2019, Vol. 40


