萘(naphthalene)是一种含两个苯环的、在自然界广泛分布的典型低分子量多环芳烃(polycyclic aromatic hydrocarbons, PAHs), 长期接触萘会引起人体内血液成分的变化, 肝转氨酶活性升高, 出现黄疸, 严重威胁人体健康, 因而被美国环保署(USEPA)列为优先控制的PAHs之一[1].由于萘结构简单且容易从自然界分离得到其降解菌株, 常被用作研究PAHs生物降解的模式化合物[2].
当前的研究表明萘在有氧及厌氧条件下均能够被微生物降解, 其中其有氧降解的机制早已被阐明[3~5].然而, 包括萘在内的一些典型PAHs的厌氧降解被认为在土壤、沉积物等富含厌氧微环境的生境中起到不可忽视的作用[6].萘的厌氧降解可能存在两种不同的起始路径, 即羧化作用和甲基化作用, 并且长期以来一直存在争议[7].当前, 被多数人接受的萘降解过程是通过羧化作用形成2-萘甲酸[8, 9].尤其是Mouttaki等[9]对N47富集培养物的体外研究表明, 13C标记的重碳酸盐与2-萘甲酸进行了碳(C)的交换, 这有力地证明了萘的确进行了酶催化的羧化作用.萘经过羧化反应转化为2-萘甲酸, 随后被未被证实的ATP酶激活转化为2-萘甲酰-CoA, 然后由2-萘甲酰-CoA还原酶催化2-萘甲酰-CoA形成5, 6, 7, 8-四氢萘甲酰-CoA[7, 10], 5, 6, 7, 8-四氢萘甲酰-CoA可进一步还原成为六氢萘甲酰-CoA产物.最后, 经过羧化开环作用, 进而逐步转变为较易被微生物厌氧代谢的化合物.
一般说来, 萘的厌氧代谢需要以具备高氧化还原电位的无机化合物作为其降解过程中的电子受体, 常见的电子受体有NO3-、Fe3+、CO2及SO42-等[11].其中硝酸盐在土壤、沉积物及水体等含有较多微氧及厌氧的环境中大量存在, 因此以其为电子受体的反硝化过程被认为可能在上述环境萘的去除中起到不可忽视的作用[12, 13].早在1988年, Mihelcic等[14]就首次报道了萘可以以硝酸根作为电子受体进行氧化分解, 开启了萘及其它PAHs厌氧代谢研究的大门.随后, 一些典型的萘反硝化降解菌群及纯培养菌株也被富集或分离, 从而极大地推进了萘厌氧代谢机制的研究[11, 15, 16].但关于萘的矿化是与完整的反硝化过程偶联或是只能与硝酸盐的还原(产生亚硝酸盐)相偶联的问题, 仍不明确[15].可见, 具备反硝化降解萘的微生物在土壤、沉积物等环境中普遍存在, 且反硝化过程与萘厌氧代谢的偶联机制还不确定.然而, 当前的研究多集中于萘反硝化相关微生物的获取、降解效率及萘代谢的相关分子生物学等方面, 关于反硝化与萘降解过程相互关系的报道还不多见.因此, 本研究从油田区域的农田土壤中富集萘反硝化降解菌群, 分析该菌群代谢萘过程中反硝化相关物质(N2O、N2、NO3-、NO2-)浓度及反硝化相关的功能基因(narG: periplasmic nitrate reductase gene; nirS: cd1-nitrite reductase gene)丰度的动态变化, 探讨反硝化过程与萘降解之间的偶联关系, 以期为进一步深入探究萘的反硝化降解机制提供一定的理论依据.
1 材料与方法 1.1 土壤萘反硝化降解菌群的富集及培养富集用土壤样品采集于湖北省潜江市境内江汉油田区的农田0~20 cm表层(112°29′E, 30°49′N).主要理化性质如下:pH 8.18, 有机质24.95 g·kg-1, 硝态氮21.84 mg·kg-1, 铵态氮4.16 mg·kg-1, 有效磷15.94 mg·kg-1, 总PAHs 0.80 mg·kg-1.
本研究中, 通过以萘为单一碳源的无机盐培养基来富集土壤中的萘反硝化降解菌群.富集用的无机盐培养基参照Lu等[11]的方法进行配制, 并灭菌备用.富集过程简述如下:吸取50 μL萘的丙酮溶液(20 mg·L-1)于100 mL的血清瓶中, 室温下在通风橱中挥发20 min, 然后依次加入45 mL培养基和5 g鲜土, 用丁基胶塞和铝盖封口后, 连续通入高纯氮气(每瓶5 min), 再用加水的注射器调节瓶内的气压至大气压.最后, 于25℃摇床(160 r·min-1)避光培养.在培养的第5 d通入高纯氮气进行换气, 9 d后, 抽取5 mL菌液转接至含新鲜培养基的血清瓶内, 进行下一轮富集.初次的加土富集处理设置了4次重复, 同时以只接种5 mL无菌水的处理为对照(CK).进行4轮富集后, 获取到的富集菌群对萘的降解率在50%左右.提取混合富集菌液的DNA用于Illumina MiSeq测序, 剩余菌液保藏于4℃用于后续的再培养实验.
1.2 培养物基因组DNA的提取及Illumina MiSeq测序取富集培养液(20 mL)于20℃, 8000 r·min-1离心10 min收集菌体(上清液回收用于萘的提取), 用Fast DNA® SPIN Kit for Soil(MP Biomedicals, 美国)试剂盒参照说明书提取各样品中的DNA, 并用1%的琼脂糖凝胶电泳进行检测, 最后保存于-20℃备用.
本研究中, 提取4轮富集后的菌液总DNA, 交上海美吉生物医药科技有限公司进行Illumina MiSeq测序.简要流程如下:以样品DNA为模板用引物对338F(ACHYCTACGGGAGGCHGC)和806R(GGACTACHVGGGTWTCTAAT)对细菌16S rDNA基因序列的V3~V4高变区进行PCR扩增, 用2%琼脂糖凝胶电泳检测后进行切胶回收; 参照电泳初步定量结果, 用QuantiFluorTM-ST蓝色荧光定量系统(Promega, 美国)对PCR产物进行定量, 按照测序量要求进行相应比例的混合, 并构建Illumina平台文库测序.使用mothur(version v.1.30.1)软件处理所得序列, 按照97%相似性对非重复序列(不含单序列)进行OTU(Operational Taxonomic Unit)聚类, 在聚类过程中去除嵌合体, 得到OTU的代表序列, 进行OTU聚类分析[17]. OTU聚类后, 获取的代表性OTU在Silva数据库(www.arb-silva.de)对进行比对, 进行物种注释, 最后在门和属水平上进行群落结构的分析.
1.3 富集菌群的再培养及萘的提取和测定按1.1节的方法将原始菌液再次转接至新鲜培养基中, 密封后, 设置了充入10%(体积比)的乙炔(抑制N2O还原为N2)和不充入乙炔的两组处理, 同时以只接种5 mL无菌水的处理为对照处理(CK), 各处理均设置3次重复.按上述方法进行培养, 期间不进行换气, 并于培养的第1、3、7、9 d取样测定培养液中的萘浓度、硝酸盐、亚硝酸浓度、瓶内气体浓度, 并参照1.2节的方法提取各时间段DNA用于后续的定量PCR.
萘的提取参照国家标准(HJ 478-2009)[18], 采用整瓶萃取的方法进行.主要步骤如下, ①萃取:摇匀血清瓶中的富集液, 将45 mL的样品全部倒入250 mL的分液漏斗中, 加入1.5 g氯化钠, 用50 mL正己烷分3次洗涤血清瓶后加入分液漏斗, 振摇5 min, 静置分层, 收集有机相, 水相再用正己烷重复萃取2遍, 弃去水相, 合并有机相, 加适量无水硫酸钠至萃取液澄清, 轻摇静置; ②浓缩:将萃取液转移至250 mL浓缩瓶中, 用正己烷洗涤所盛有机相瓶3次, 每次5~10 mL, 洗涤液也并入同一浓缩瓶中, 于-46 kPa, 45℃下旋蒸浓缩至3~5 mL; ③置换:加入25 mL甲醇(液相色谱纯)进行溶剂置换, 再于50~55℃旋蒸浓缩至3~5 mL, 并转移到比色管中, 用甲醇定容至5 mL; ④上样:过0.45 μm滤膜, 以萘的标准品(Dr. Ehrenstorfer, 德国)配制标准曲线, 用高效液相色谱HITACHI L-7100(Hitachi High-Technologies Corporation, 日本)测定各样品中的萘浓度, 并通过公式(1)和公式(2)分别计算萘的降解率(%)及降解速率[mg·(L·d)-1].

|
(1) |
式中, ctck为各取样天CK中测得的萘浓度(mg·L-1); ct为各取样天接种富集菌群中萘的浓度(mg·L-1), 20为添加的萘初始浓度(mg·L-1).
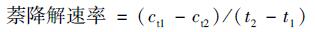
|
(2) |
式中, ct1和ct2分别为前后两个连续的采样时间点测得的血清瓶中萘浓度(mg·L-1), t1和t2分别为前后两个连续取样测定的时间点(d).
1.4 硝酸根、亚硝酸根及气体浓度的测定在上述各取样时间点, 用注射器抽取5 mL富集培养液, 过0.45 μm滤膜于离心管中, 取1.25 mL定容至25 mL, 随后用离子色谱仪ICS-1100(Dionex Corporation, 美国)测定NO3-及NO2-的浓度.
在上述各取样天抽取瓶内气体, 用气相色谱(Agilent Technologies 7890A, 美国)测定血清瓶中的N2O浓度, 并按下列公式分别计算N2O[公式(3)]及N2[公式(4)]的产生速率[μg·(L·h)-1].
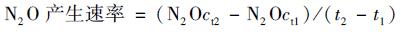
|
(3) |
式中, N2Oct1和N2Oct2分别为前后两个连续的采样时间点测得的血清瓶中N2O浓度(μg·L-1), t1和t2分别为前后两个连续取样测定的时间点(h).
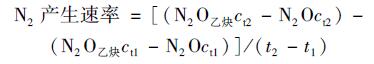
|
(4) |
式中, N2O乙炔ct1和N2O乙炔ct2分别为前后两个连续的采样时间点测得的充入乙炔处理的血清瓶中N2O浓度(μg·L-1), N2Oct1、N2Oct2、t1和t2同公式(3).
1.5 反硝化相关的narG及nirS的定量PCR反硝化相关的narG及nirS基因丰度的检测用SYBR GREEN法, 在荧光定量PCR仪(ABI7500, Applied Biosystems, 美国)上进行.引物对1960F(TYAGTSGGSCARGARAA)和2650R(TYTCRTAC CABGTBGC)[19], Cd3aF(GTSAACGTSAAGGARA CSGG)和R3cd(GASTTCGGRTGSGTCTTGA)[20]分别用于narG及nirS基因的扩增.定量PCR按照Premix Ex TaqTM(TaKaRa Biotechnology, 日本)的说明书进行, 反应采用25 μL体系, 其中含上下游引物各1 μL, DNA模板1 μL. narG基因的反应条件为:94℃预变性5 min; 94℃变性30 s, 58℃退火1 min, 72℃延伸45 s, 共40个循环; 72℃延伸10 min. nirS基因的反应条件为:94℃预变性3 min; 94℃变性30 s, 52℃退火40 s, 72℃延伸1 min, 共40个循环; 72℃延伸10 min.实验中的标准曲线参照He等[21]的方法进行, 在各反应完成后, 设置了溶解曲线程序(60~90℃), 用来检测real-time PCR产物的特异性, 最后通过ABI 7500 Software(version 2.0.6)来分析所得的数据并计算各样品中相关基因的拷贝数.
1.6 数据处理与分析实验数据的统计分析在SPSS 17.0(SPSS, 美国)软件中, 用单因素方差分析的S-N-K(Student-Newman-Keuls test)法检验不同样品之间的差异显著性(P < 0.05表示差异显著).用SPSS中的Pearson相关检验来完成各指标之间的相关性分析.
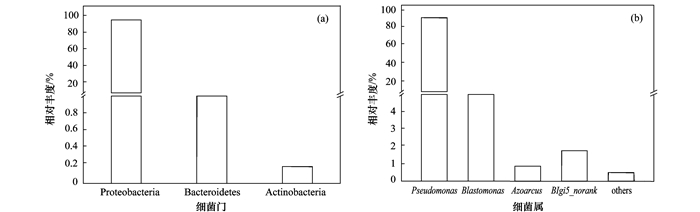
2 结果与分析 2.1 富集菌群的群落组成通过Illumina MiSeq测序从该萘反硝化降解菌群中共获取了30454条有效序列, 在97%的相似度下可分为17个OTUs, 它们分别属于变形菌门(9)、拟杆菌门(2)、放线菌门(3)和厚壁菌门(3).其在门和属水平上的细菌群落结构见图 1.可见, 该富集物中的细菌由变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)和放线菌门(Actinobacteria)组成, 且Proteobacteria(98.02%)是最明显的优势菌门[图 1(a)].该富集物中可检测的优势已知菌属(相对丰度>1%)则为变形菌门中的假单胞菌属(Pseudomonas)、芽单胞菌门中的芽单胞菌属(Blastomonas)和BIgi5_norank[图 1(b)].其中Pseudomonas是富集菌群中的优势菌属, 也是一种具备反硝化能力的菌属, 其相对丰度为90.89%.
|
图 1 富集菌群在细菌门和属水平上的群落组成 Fig. 1 Bacterial community compositions based on phylum and genus in the enriched consortium |
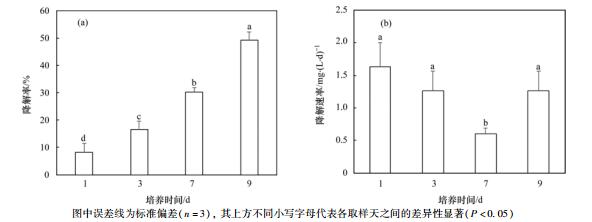
培养的第1、3、7及9 d, 富集培养液中萘降解率及萘降解速率的动态变化如图 2所示.可见, 富集菌群中萘的降解率在培养期间不断增加, 从第1 d的8.16%增加至第9 d的49.11%[图 2(a)].第1、3及9 d时萘的降解速率显著高于第7 d的萘降解速率(P < 0.05), 其中第1 d的最高[1.63 mg·(L·d)-1], 而第3及9 d的降解速率与第1 d无显著差异[图 2(b)].
|
图 2 富集液中萘降解率及降解速率的动态变化 Fig. 2 Dynamic changes of the naphthalene degradation rate and degradation speed during incubation |
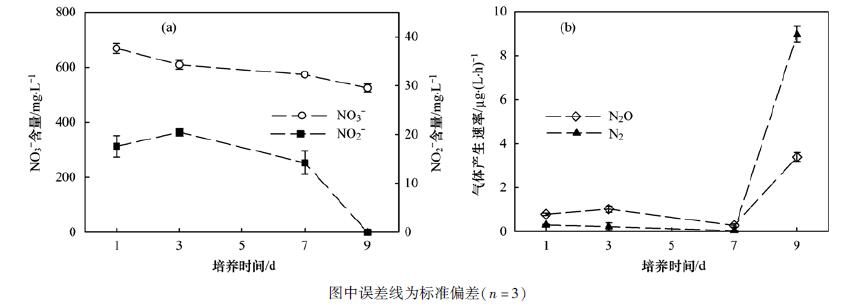
富集培养液中NO3-、NO2-浓度及气体产生速率的动态变化如图 3所示.可知, 培养期间培养液中NO3-的浓度不断减少, 而NO2-在前3 d有所累积, 之后迅速下降, 第9 d培养液中已经检测不到NO2-[图 3(a)].培养期间富集培养血清瓶中N2O和N2产生速率的动态变化如图 3(b)所示.培养期间, 富集培养液中不论是N2O还是N2的产生速率在培养的第1、3及7 d差别均较小, 但是在培养的第9 d它们的产生速率均显著高于前3个取样天(P < 0.05).对两种气体的产生速率进行比较, 培养的1、3及7 d, N2O的产生速率均高于N2, 但第9 d后者[8.97 μg·(L·h)-1]显著高于前者[3.39 μg·(L·h)-1](P < 0.05).
|
图 3 富集液中NO3-、NO2-浓度及N2O、N2产生速率的动态变化 Fig. 3 Dynamic changes of NO3- and NO2- concentrations and production rates of N2O and N2 during incubation |
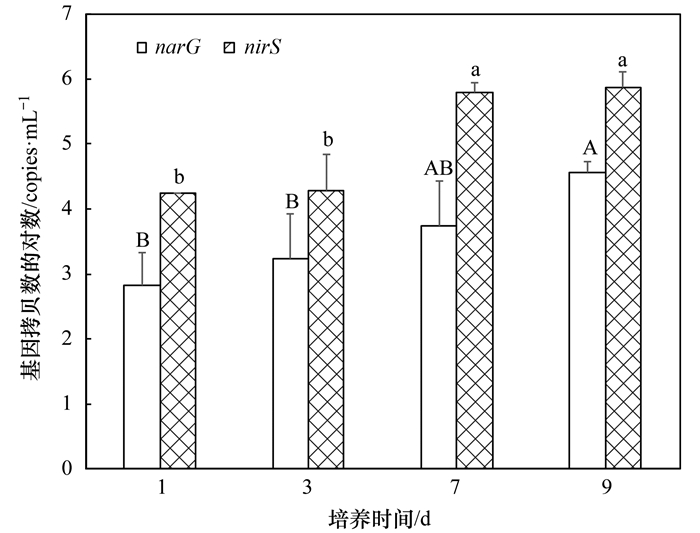
图 4为培养过程中narG及nirS丰度的动态变化.可见, 在培养期间, narG基因丰度低于nirS丰度, 且它们均呈现随时间的延续而增加的趋势.其中, narG基因丰度在培养的第9 d达到最大(3.80×104 copies·mL-1), 显著高于1、3 d的narG丰度.而后两次取样的nirS基因丰度(第7及9 d)显著高于第1及3 d(P < 0.05).
|
图中误差线为标准偏差(n=3), 其上方不同大写和小写字母分别指示narG及nirS基因在不同取样天的差异显著(P < 0.05) 图 4 培养期间富集培养液中narG及nirS基因的丰度 Fig. 4 Abundances of narG and nirS genes in the enriched consortium during incubation |
Illumina MiSeq测序的结果表明, 假单胞菌属(Pseudomonas)是富集菌群中的最大优势微生物.该菌是变形菌门的重要属, 在土壤、水、空气等潮湿的环境中广泛存在, 且可以以多数有机污染物作为碳源和能源进行生长[22], 并具备较强的反硝化能力[23].同时, 反硝化条件下假单胞菌属对典型PAHs(萘、蒽)的降解也已经为人们所证实[24~26]. Liang等[26]从汕头近海分离得到1株假单胞菌能够在缺氧反硝化条件下降解菲、荧蒽等PAHs.此外, 本研究的富集菌群中还检测到芽单胞菌属(Blastomonas)和固氮弓菌属(Azoarcus), 其中有关Blastomonas对污染物的降解目前还鲜有报道, 其是否具备反硝化能力还有待进一步研究.而Azoarcus被证实具备较强的复杂有机物降解能力[27, 28], 可见它们可能也在萘的反硝化代谢中起到了一定的协同作用.
本研究通过以萘为单一碳源的培养基, 厌氧条件下富集萘的反硝化降解菌群, 并初步探究其对萘的降解与反硝化过程的偶联关系.结果表明, 该富集菌群9 d内对萘的降解率可达49.11%, 且对萘的平均降解速率为1.189 mg·(L·d)-1.已有的研究表明, 富集的反硝化菌群及纯培养菌株对萘的降解率在20.2%~96.3%[11, 15, 16]. Dou等[16]通过在厌氧条件下, 从土壤中分离得到的纯培养菌对萘的降解速率达到1.8 mg·(L·d)-1, 而且当萘起始浓度低于30 mg·L-1时, 几乎所有的萘在25 d内被降解.可见, 本研究获取的反硝化菌群具备较强的萘代谢能力.该菌群代谢萘的过程中, 培养液中NO3-浓度逐渐下降, 表明NO3-的利用与萘的代谢直接相关.其原因在于, 首先NO3-作为该富集菌群反硝化作用的电子受体被还原, 其次NO3-还可作为微生物的氮源, 通过同化型硝酸盐还原过程被微生物同化[13].与NO3-不同, NO2-浓度在培养初期有明显的积累, 表明培养初期(1~3 d)由NO3-还原形成NO2-的过程(反硝化的第一步)占主导作用, 而此时也具备较高的萘降解速率.这表明, 萘的降解可能与反硝化过程的第一步具有密切的关系.当前, PAHs反硝化降解过程中的亚硝酸积累现象在富集菌群及土壤复杂环境下均有报道[16, 29, 30].如Dou等[16]发现萘在反硝化条件下的降解过程中, 主要为亚硝酸盐的积累.而Chang等[29]在研究台湾龟山等地区土壤对PAHs的降解时发现, 土壤中的菲、芘、蒽、芴、萘降解过程中伴随有大量的亚硝酸盐累积.可见, 硝酸盐还原为亚硝酸盐的过程在萘的反硝化降解中具有重要的地位.本研究中, 3 d后培养液中NO2-浓度的急剧下降及后期(第9 d)气体生成速率的急剧上升, 均表明此后NO2-还原过程(反硝化过程中气态产物产生步骤)开始发生.与此同时, 之后的5、7及9 d同样伴随了萘的降解, 可见反硝化过程中的NO2-还原及气态产物产生过程也可能与萘的代谢相偶联.翟晓峰等报道[31], 无论以NO3-还是NO2-作为电子受体, 反硝化菌均可产生N2O, 但以NO2-为电子受体时释放的N2O更多.在反硝化阶段, 各种还原酶对电子的竞争能力不同[32], 且NO2-可能对N2O还原酶具有较强的抑制作用[33], 因此本研究在NO2-浓度急剧下降的后期, 检测到大量的N2产生.不同的菌群对萘的代谢与反硝化过程偶联的机制也可能不同.如Rockne等[15]从海洋沉积物中分离到两株能够利用硝酸盐为电子受体降解萘的细菌, 它们分别与Pseudomonas stutzeri及Vibrio pelagius有较近的亲缘关系.这两种菌株代谢过程存在明显的差别, 前者能够将萘的部分矿化与完整的反硝化过程偶联, 而后者只能将萘的矿化与硝酸盐的还原(产生亚硝酸盐)相偶联.
本研究中, 反硝化相关的硝酸盐还原酶基因(narG)和亚硝酸盐还原酶基因(nirS)的丰度均随培养时间而增加, 表明富集菌群中的反硝化细菌数目随培养时间延长而快速增加.值得注意的是narG基因的丰度与硝酸根(r=-0.862, P < 0.01)的浓度呈显著负相关, 而nirS基因丰度则与亚硝酸根(r=-0.725, P < 0.01)的浓度呈显著负相关, 表明上述基因的丰度并不与其对应底物浓度成正比, 而是随底物的消耗而增长.虽然培养后期, 相关基因丰度增加的同时也检测到较高气体生产速率, 但仅在narG基因丰度与N2O的产生速率之间有显著正相关(r=0.632, P < 0.05), 表明反硝化基因丰度与反硝化活性存在一定的联系, 但基于DNA水平的定量PCR还不足以反映环境中反硝化微生物的真实活性[34, 35].因此, 要充分考察基因丰度与反硝化活性的关系, 基于mRNA的定量PCR可能是未来进一步深入研究PAHs厌氧代谢与反硝化偶联机制的有效手段.
4 结论(1) 变形菌门中的Pseudomonas是该富集菌群中最大优势微生物, 同时Blastomonas和Azoarcus可能在萘降解过程中起到一定的协助作用.
(2) 富集获取到萘的反硝化降解菌群, 平均降解速率为1.189 mg·(L·d)-1, 其9 d对萘的降解率可达49.11%.
(3) 该菌群代谢萘的过程不仅与反硝化的第一步反应硝酸盐还原过程有密切的偶联关系, 还可能与亚硝酸盐还原的含氮气体产生过程相关.
| [1] |
王艳, 辛嘉英, 宋昊, 等. 生物降解萘的研究进展[J]. 中国生物工程杂志, 2009, 29(9): 119-124. Wang Y, Xin J Y, Song H, et al. Advances in biodegradation of naphthalene[J]. China Biotechnology, 2009, 29(9): 119-124. |
| [2] | Seo J S, Keum Y S, Li Q X. Bacterial degradation of aromatic compounds[J]. International Journal of Environmental Research and Public Health, 2009, 6(1): 278-309. DOI:10.3390/ijerph6010278 |
| [3] | Harayama S, Rekik M, Wasserfallen A, et al. Evolutionary relationships between catabolic pathways for aromatics:conservation of gene order and nucleotide sequences of catechol oxidation genes of pWW0 and NAH7 plasmids[J]. Molecular and General Genetics MGG, 1987, 210(2): 241-247. DOI:10.1007/BF00325689 |
| [4] | Baboshin M, Akimov V, Baskunov B, et al. Conversion of polycyclic aromatic hydrocarbons by Sphingomonas sp. VKM B-2434[J]. Biodegradation, 2008, 19(4): 567-576. DOI:10.1007/s10532-007-9162-2 |
| [5] | Lee H J, Kim J M, Lee S H, et al. Gentisate 1, 2-dioxygenase, in the third naphthalene catabolic gene cluster of Polaromonas naphthalenivorans CJ2, has a role in naphthalene degradation[J]. Microbiology, 2011, 157(10): 2891-2903. DOI:10.1099/mic.0.049387-0 |
| [6] |
姚炎红, 王明霞, 左小虎, 等. 典型油田多环芳烃污染对土壤反硝化微生物群落结构的影响[J]. 环境科学, 2016, 37(12): 4750-4759. Yao Y H, Wang M X, Zou X H, et al. Effects of PAHs pollution on the community structure of denitrifiers in a typical oilfield[J]. Environmental Science, 2016, 37(12): 4750-4759. |
| [7] | Meckenstock R U, Mouttaki H. Anaerobic degradation of non-substituted aromatic hydrocarbons[J]. Current Opinion in Biotechnology, 2011, 22(3): 406-414. DOI:10.1016/j.copbio.2011.02.009 |
| [8] | Musat F, Galushko A, Jacob J, et al. Anaerobic degradation of naphthalene and 2-methylnaphthalene by strains of marine sulfate-reducing bacteria[J]. Environmental Microbiology, 2009, 11(1): 209-219. DOI:10.1111/emi.2009.11.issue-1 |
| [9] | Mouttaki H, Johannes J, Meckenstock R U. Identification of naphthalene carboxylase as a prototype for the anaerobic activation of non-substituted aromatic hydrocarbons[J]. Environmental Microbiology, 2012, 14(10): 2770-2774. DOI:10.1111/emi.2012.14.issue-10 |
| [10] | Eberlein C, Johannes J, Mouttaki H, et al. ATP-dependent/-independent enzymatic ring reductions involved in the anaerobic catabolism of naphthalene[J]. Environmental Microbiology, 2013, 15(6): 1832-1841. DOI:10.1111/emi.2013.15.issue-6 |
| [11] | Lu X Y, Zhang T, Fang H H P, et al. Biodegradation of naphthalene by enriched marine denitrifying bacteria[J]. International Biodeterioration & Biodegradation, 2011, 65(1): 204-211. |
| [12] | Burgin A J, Hamilton S K. Have we overemphasized the role of denitrification in aquatic ecosystems? A review of nitrate removal pathways[J]. Frontiers in Ecology and the Environment, 2007, 5(2): 89-96. DOI:10.1890/1540-9295(2007)5[89:HWOTRO]2.0.CO;2 |
| [13] |
江利梅, 陈杏娟, 马连营, 等. 硝酸盐还原促进毒害性有机污染物降解的研究进展[J]. 微生物学通报, 2014, 41(8): 1661-1666. Jiang L M, Chen X J, Ma L Y, et al. Perspectives on the nitrate reduction coupling biodegradation of toxic organic contaminants[J]. Microbiology China, 2014, 41(8): 1661-1666. |
| [14] | Mihelcic J R, Luthy R G. Microbial degradation of acenaphthene and naphthalene under denitrification conditions in soil-water systems[J]. Applied and Environmental Microbiology, 1988, 54(5): 1188-1198. |
| [15] | Rockne K J, Chee-Sanford J C, Sanford R A, et al. Anaerobic naphthalene degradation by microbial pure cultures under nitrate-reducing conditions[J]. Applied and Environmental Microbiology, 2000, 66(4): 1595-1601. DOI:10.1128/AEM.66.4.1595-1601.2000 |
| [16] | Dou J F, Liu X, Ding A Z. Anaerobic degradation of naphthalene by the mixed bacteria under nitrate reducing conditions[J]. Journal of Hazardous Materials, 2009, 165(1-3): 325-331. DOI:10.1016/j.jhazmat.2008.10.002 |
| [17] | Schloss P D, Gevers D, Westcott S L. Reducing the effects of PCR amplification and sequencing artifacts on 16S rRNA-based studies[J]. PLoS One, 2011, 6(12): e27310. DOI:10.1371/journal.pone.0027310 |
| [18] | 中华人民共和国环境保护部. HJ 478-2009, 水质多环芳烃的测定液液萃取和固相萃取高效液相色谱法[S]. 北京: 中国环境科学出版社, 2009. |
| [19] | Philippot L, Piutti S, Martin-Laurent F, et al. Molecular analysis of the nitrate-reducing community from unplanted and maize-planted soils[J]. Applied and Environmental Microbiology, 2002, 68(12): 6121-6128. DOI:10.1128/AEM.68.12.6121-6128.2002 |
| [20] | Michotey V, Méjean V, Bonin P. Comparison of methods for quantification of cytochrome cd1-denitrifying bacteria in environmental marine samples[J]. Applied and Environmental Microbiology, 2000, 66(4): 1564-1571. DOI:10.1128/AEM.66.4.1564-1571.2000 |
| [21] | He J Z, Shen J P, Zhang L M, et al. Quantitative analyses of the abundance and composition of ammonia-oxidizing bacteria and ammonia-oxidizing archaea of a Chinese upland red soil under long-term fertilization practices[J]. Environmental Microbiology, 2007, 9(9): 2364-2374. DOI:10.1111/emi.2007.9.issue-9 |
| [22] | Ridgway H F, Safarik J, Phipps D, et al. Identification and catabolic activity of well-derived gasoline-degrading bacteria from a contaminated aquifer[J]. Applied and Environmental Microbiology, 1990, 56(11): 3565-3575. |
| [23] |
孟婷, 杨宏. 高效反硝化细菌的快速培养及群落结构多样性分析[J]. 环境科学, 2017, 38(9): 3816-3822. Meng T, Yang H. Rapid culture, microbial community structure, and diversity of high-efficiency denitrifying bacteria[J]. Environmental Science, 2017, 38(9): 3816-3822. |
| [24] | Lee K, Park J W, Ahn I S. Effect of additional carbon source on naphthalene biodegradation by Pseudomonas putida G7[J]. Journal of Hazardous Materials, 2003, 105(1-3): 157-167. DOI:10.1016/j.jhazmat.2003.08.005 |
| [25] | Jacques R J S, Santos E C, Bento F M, et al. Anthracene biodegradation by Pseudomonas sp. isolated from a petrochemical sludge landfarming site[J]. International Biodeterioration & Biodegradation, 2005, 56(3): 143-150. |
| [26] | Liang L, Song X H, Kong J, et al. Anaerobic biodegradation of high-molecular-weight polycyclic aromatic hydrocarbons by a facultative anaerobe Pseudomonas sp. JP1[J]. Biodegradation, 2014, 25(6): 825-833. DOI:10.1007/s10532-014-9702-5 |
| [27] | Rhee S K, Lee G M, Yoon J H, et al. Anaerobic and aerobic degradation of pyridine by a newly isolated denitrifying bacterium[J]. Applied and Environmental Microbiology, 1997, 63(7): 2578-2585. |
| [28] | Ma Q, Qu Y Y, Shen W L, et al. Bacterial community compositions of coking wastewater treatment plants in steel industry revealed by Illumina high-throughput sequencing[J]. Bioresource Technology, 2015, 179: 436-443. DOI:10.1016/j.biortech.2014.12.041 |
| [29] | Chang B V, Shiung L C, Yuan S Y. Anaerobic biodegradation of polycyclic aromatic hydrocarbon in soil[J]. Chemosphere, 2002, 48(7): 717-724. DOI:10.1016/S0045-6535(02)00151-0 |
| [30] | Rockne K J, Strand S E. Anaerobic biodegradation of naphthalene, phenanthrene, and biphenyl by a denitrifying enrichment culture[J]. Water Research, 2001, 35(1): 291-299. DOI:10.1016/S0043-1354(00)00246-3 |
| [31] |
翟晓峰, 蒋成爱, 吴光学, 等. 以甲醇为碳源生物反硝化过程释放一氧化二氮的试验研究[J]. 环境科学, 2013, 34(4): 1421-1427. Zhai X F, Jiang C A, Wu G X, et al. Nitrous oxide emission during denitrification for activated sludge acclimated with methanol as the organic carbon[J]. Environmental Science, 2013, 34(4): 1421-1427. |
| [32] | Schalk-Otte S, Seviour R J, Kuenen J G, et al. Nitrous oxide (N2O) production by Alcaligenes faecalis during feast and famine regimes[J]. Water Research, 2000, 34(7): 2080-2088. DOI:10.1016/S0043-1354(99)00374-7 |
| [33] |
王莎莎, 彭永臻, 巩有奎, 等. 不同电子受体低氧条件下生物反硝化过程中氧化亚氮产量[J]. 水处理技术, 2011, 37(8): 58-60, 67. Wang S S, Peng Y Z, Gong Y K, et al. Production of N2O in different electron acceptors during denitrification process under low dissolved oxygen concentration[J]. Technology of Water Treatment, 2011, 37(8): 58-60, 67. |
| [34] | Boyle S A, Rich J J, Bottomley P J, et al. Reciprocal transfer effects on denitrifying community composition and activity at forest and meadow sites in the Cascade Mountains of Oregon[J]. Soil Biology and Biochemistry, 2006, 38(5): 870-878. DOI:10.1016/j.soilbio.2005.08.003 |
| [35] | Zhou Z F, Zheng Y M, Shen J P, et al. Responses of activities, abundances and community structures of soil denitrifiers to short-term mercury stress[J]. Journal of Environmental Sciences, 2012, 24(3): 369-375. DOI:10.1016/S1001-0742(11)60747-X |
 2018, Vol. 39
2018, Vol. 39


