2. 河南师范大学生命科学学院, 新乡 453007;
3. 资源微生物与功能分子河南省高校重点实验室培育基地(河南师范大学), 新乡 453007;
4. 新乡市环境保护监测站, 新乡 453007
2. College of Life Sciences, Henan Normal University, Xinxiang 453007, China;
3. Key Laboratory for Microorganisms and Functional Molecules (Henan Normal University), University of Henan Province, Xinxiang 453007, China;
4. Environmental Monitoring Station of Xinxiang City, Xinxiang 453007, China
改革开放以来, 畜禽养殖业在我国得到了迅速的发展.现如今, 中国已经成为世界上畜禽养殖规模最大的国家之一, 但随之而来是每年超过20亿t畜禽粪便的产生和排放, 且这一数字仍在逐年递增[1, 2].在规模化的养殖行业中, 兽药抗生素通常被用作疾病治疗剂和生长促进剂而频繁使用[3], 由此造成了动物肠道中抗生素的残留和耐药细菌的积累, 并伴随粪便排泄排出体外[4].因此, 携带抗生素、耐药细菌的畜禽粪便如果未经处理直接作为有机肥料施用于农田, 会给生态环境带来风险[5].
众多研究证实, 畜禽粪便已经成为了自然环境中耐药细菌和耐药基因的储“库”[6].例如, Yang等[7]在鸡粪样品中检测到包括大肠埃希氏菌(Escherichia coli)、粪产碱杆菌(Alcaligenes faecalis)和肠球菌(Enterococcus spp.)在内的多种抗生素耐药的人致病菌. Brooks等[8]从养猪场的储粪池中检测到高于105 cfu·mL-1的致病性沙门氏菌(Salmonella spp.)和弯曲杆菌(Campylobacter spp.), 并且它们大多具有耐药性. Chee-Sanford等[9]在两个猪粪化粪池中均检测到7种四环素类抗生素的耐药基因tetO、tetQ、tetW、tetM、tetB(P)、tetS和tetT.其中, tetO和tetW基因在牛粪、鸡粪和猪粪中均显示较高丰度[10]. Mu等[11]也发现鸡粪中喹诺酮类耐药基因oqxB和磺胺类耐药基因sul1、sul2的丰度分别达到了3.48×1010、1.39×1010和7.53×109 copies·g-1.
耐药基因的存在是细菌表现耐药性的根本原因.有研究表明, 耐药细菌随着畜禽粪便排出动物体外后, 其携带的耐药基因可以通过水平转移的方式, 在质粒、转座子、整合子等可移动元件的作用下传递给其它环境微生物, 从而导致耐药性的传播和扩散[12~14]. Qu等[15]利用比较基因组学证实了可移动的DNA元件是鸡肠道微生物中主要的功能性成分. Stokes等[16]也发现猪源E. coli中检测到的intI1(Ⅰ类整合子)携带的耐药基因岛上同时存在多种耐药基因.而一个捕获多个基因盒的整合子, 已经被证实能够完成多个耐药基因的转移[17].因此, 当这样的畜禽粪便排入自然环境后, 在高丰度的耐药基因、可移动元件及其它环境压力共存的情况下, 环境中的敏感细菌可能会因此产生甚至是多重的抗生素耐药性[18], 而一旦人类致病菌通过这种方式获得多重耐药, 则会对人类健康造成巨大威胁.
目前, 关于畜禽粪便中耐药细菌和耐药基因的污染问题已经引起了国内外学者的广泛关注, 但是已报道的研究工作多集中于某一种或几种典型抗生素或耐药基因的污染现象, 涉及多重耐药细菌和耐药基因多样性的系统研究并不深入.本文选择不同来源的鸡粪和猪粪为研究对象, 通过对其中可培养多重耐药菌的群落结构以及176种耐药基因和2种可移动元件的存在、富集情况进行解析, 以期为畜禽粪肥的合理处置、安全利用提供参考依据.
1 材料与方法 1.1 样品采集选择新乡市郊区饲用抗生素的3家养鸡场(养殖规模>10 000只)和3家养猪场(存栏量>300头), 在各养殖场的储粪池中采用5点混合取样法[19], 分别采集3份粪便样品, 各自混匀后置于冰盒中带回实验室. 24 h内进行细菌培养实验, 其余样品于-20℃保存.
1.2 总可培养异养细菌和多重耐药细菌的培养与计数将粪便样品制成悬液, 梯度稀释后分别涂布于灭菌的Luria-Bertani(LB)平板和同时含有四环素(16 μg·mL-1)、环丙沙星(4 μg·mL-1)、庆大霉素(16 μg·mL-1)的LB平板上, 37℃培养72 h, 计数LB平板上的总可培养细菌数和含抗生素平板上的多重耐药细菌数[20], 并按照以下公式[19]计算多重耐药细菌的比例.含抗生素平板上抗生素浓度的设定参考CLSI(2016)标准[21].

|
从长有多重耐药菌的平板上随机挑选单菌落, 分离纯化后分别接种到新鲜灭菌LB液体培养基中, 37℃、150 r·min-1振荡培养12 h, 采用菌液PCR的方法扩增细菌16S rDNA[22].引物选用27F(AGAGTTTGATCCTGGCTCAG)和1492R(GGTTACC TTGTTACGACTT)[23]. 25 μL反应体系包括:12.5 μL 2×PCR Taq MasterMix, 0.8 μL引物, 1 μL菌液, 9.9 μL ddH2O.反应条件[24]:95℃预变性10 min; 35个循环包括:94℃变性30 s, 57℃退火30 s, 72℃延伸90 s; 最后72℃延伸10 min. PCR产物用1%琼脂糖凝胶电泳检测合格后送交生工生物工程(上海)股份有限公司进行16S rDNA测序, 测序结果在NCBI数据库中进行BLAST比对, 最后利用MEGA 6.0软件构建多重耐药菌的系统进化树.
对已鉴定的多重耐药菌株, 使用含有四环素、环丙沙星和庆大霉素的Kirby Bauer药敏纸片(Thermo Fisher Oxoid, 英国)进行抗生素敏感性试验[25].即将活化的单菌株用0.9%无菌生理盐水制成0.5麦氏标准悬液, 均匀涂布于MHA营养琼脂平板上, 再将药敏纸片置于其上, 37℃培养18 h, 测量抑菌圈直径并判定耐药结果[21].实验同时选用E. coli ATCC 25922菌株作为质控菌株.
1.4 多重耐药细菌的群落结构分析用无菌生理盐水收集生长在含抗生素平板上的所有多重耐药菌落, 再用Bacterial DNA试剂盒(Omega, 美国)对收集到的菌液提取DNA, 并利用超微量分光光度计(NanoDrop 2000, Thermo Fisher Scientific)和琼脂糖凝胶电泳检测DNA质量.合格的DNA样品送至Novogene公司(北京)利用HiSeq 2500测序平台进行16S rDNA高通量测序分析. PCR引物采用515F(GTGCCAGCMGCCGCGGTAA)和806R(GGACTACHVGGGTWTCTAAT)[26], 数据分析参考文献[27].
1.5 耐药基因的高通量定量分析称取1.0 g粪便样品, 利用Stool DNA试剂盒(Omega, 美国)进行总DNA提取.检测合格的DNA送至Wcgene公司(上海), 利用高通量定量PCR检测包括磺胺类、四环素类、β-内酰胺类、氨基糖苷类、万古霉素类、FCA(氟喹诺酮、喹诺酮、氟苯尼考、氯霉素和酰胺醇类)和MLSB(大环内酯、林可酰胺和链阳性菌素B类)在内的七大类176种耐药基因、2个可移动元件(Ⅰ类整合子和转座子Tn916/1545)和16S rDNA的存在情况[28, 29], 每个样品3次重复.进一步根据2-ΔΔCt方法, 以一个无抗生素饲喂的家养猪粪为对照, 选取Ct检测值≤30的耐药基因, 按如下公式计算饲用抗生素的猪粪样品中各类耐药基因的平均富集倍数[30]:

|
利用软件Microsoft Excel 2016进行实验数据的处理及图表制作, 采用SPSS V22.0软件进行ANOVA方差分析.
2 结果与分析 2.1 畜禽粪便中多重耐药细菌的分布根据样品采集时的调研结果, 本研究选择当地养殖场普遍使用的四环素、环丙沙星和庆大霉素作为多重耐药细菌筛选的抗生素类型, 对不同来源的鸡粪、猪粪中总可培养异养细菌和同时对上述3种抗生素具有耐药性的多重耐药细菌的数量进行了统计.结果显示, 被检测的3个鸡粪样品中, 总可培养细菌数量为6.59×108~7.10×109 cfu·g-1, 多重耐药细菌数量为8.17×107~6.90×108 cfu·g-1.来自猪场的3个粪便样品中, 总菌数量和多重耐药菌数量分别为2.03×109~1.37×1010 cfu·g-1和1.63×108~1.09×109 cfu·g-1, 其丰度均高于鸡粪样品.进一步比较不同畜禽粪便中多重耐药菌占总细菌的比例, 结果显示在图 1中.
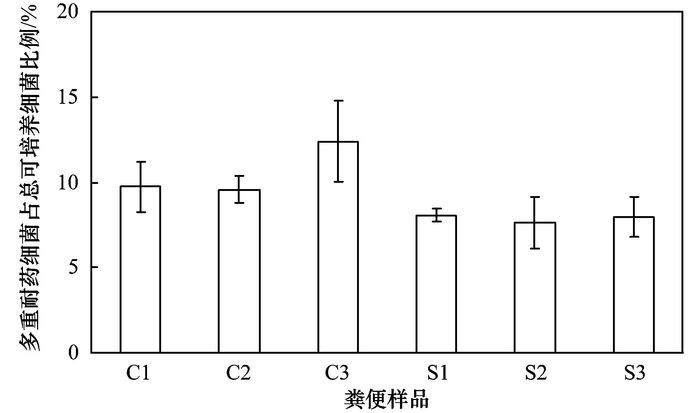
|
C1~C3:3个鸡粪样品, S1~S3:3个猪粪样品; 下同 图 1 畜禽粪便中可培养多重耐药细菌占总可培养细菌的比例 Fig. 1 Rates of multidrug-resistant bacteria to total cultural heterotrophic bacteria in livestock manures |
由图 1可知, 鸡粪中多重耐药菌比例为9.73%~12.40%, 而猪粪中该比例为7.96%~8.04%, 低于鸡粪样品.同时单因素方差分析结果表明, 3个鸡粪样品中多重耐药细菌的平均比例显著高于猪粪样品(P<0.05).
2.2 多重耐药细菌的鉴定及其耐药性随机挑取来自鸡粪和猪粪的多重耐药细菌菌落, 分离纯化后经16S rDNA测序和序列比对, 构建系统进化树的结果如图 2所示.从中可知, 有19株菌株测序成功, 分别属于大肠埃希氏菌(E. coli)、奇异变形杆菌(Proteus mirabilis)、普通变形杆菌(Proteus vulgaris)、粪产碱杆菌(Alcaligenes faecalis)和摩氏摩根菌(Morganella morganii).进一步利用药敏试验对这19株多重耐药菌的耐药性进行验证, 结果显示, 所有菌株对供试的四环素、环丙沙星和庆大霉素这3种抗生素均表现出耐药性.
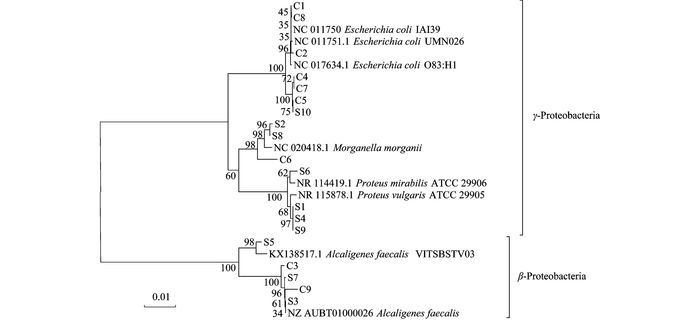
|
图 2 畜禽粪便中部分多重耐药细菌的系统进化树 Fig. 2 Phylogenetic tree of the partial multidrug-resistant bacteria in livestock manures |
采用高通量测序技术对畜禽粪便中所有可培养多重耐药细菌的群落组成进行了分析, 从中筛选出相对丰度Top10的优势属, 分属于埃希氏菌属(Escherichia)、香味菌属(Myroides)、不动杆菌属(Acinetobacter)、变形杆菌属(Proteus)、依格纳季氏菌属(Ignatzschineria)、嗜冷杆菌属(Psychrobacter)、普罗维登斯菌属(Providencia)、肠球菌属(Enterococcus)、产碱杆菌属(Alcaligenes)和费克蓝姆菌属(Facklamia), 各属细菌在粪便样品中的分布情况显示于图 3.从中可知, 鸡粪中Escherichia(6.09%~58.95%)、Proteus(3.64%~19.44%)、Ignatzschineria(1.36%~21.24%)和Acinetobacter(3.12%~4.98%)比例较高, 猪粪中则是Escherichia(3.54%~18.34%)、Acinetobacter(7.12%~51.75%)、Facklamia(9.94%~48.72%)和Proteus(1.68%~3.56%)呈显著优势.由此可见, 虽然多重耐药菌在不同来源的畜禽粪便中的丰度有差异, 但是Escherichia、Acinetobacter和Proteus细菌在所有样品中均普遍存在且保持较高丰度, 该结果与2.2节中部分多重耐药菌的16S rDNA鉴定结果相符.
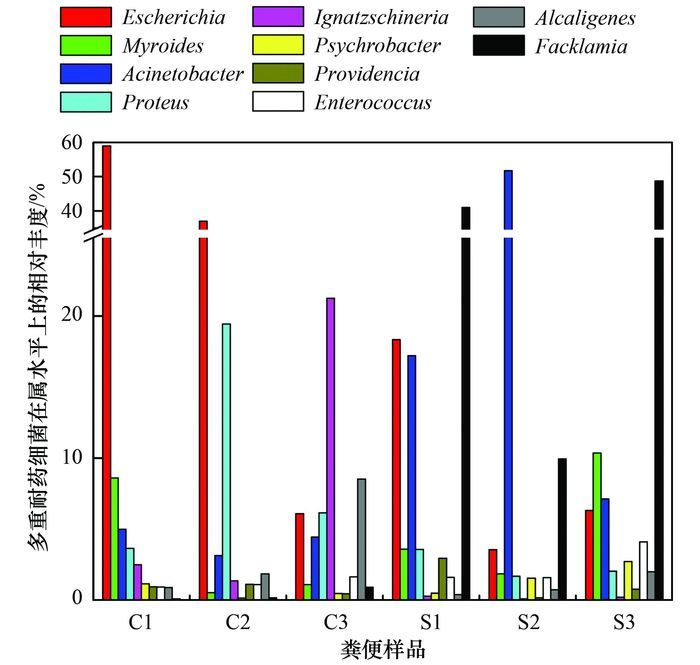
|
图 3 畜禽粪便中具代表性多重耐药细菌在属水平上的相对丰度 Fig. 3 Relative abundance of representative multidrug-resistant bacteria at the genus level in livestock manures |
利用高通量定量PCR检测了畜禽粪便中176种耐药基因和2个可移动元件的存在情况, 各样品的基因检出数量显示于图 4.从中可知, 鸡粪中检出耐药基因131~147种, 猪粪中检出136~146种, 供检测的7类耐药基因和2个可移动元件在所有粪便样品中均被100%检出.
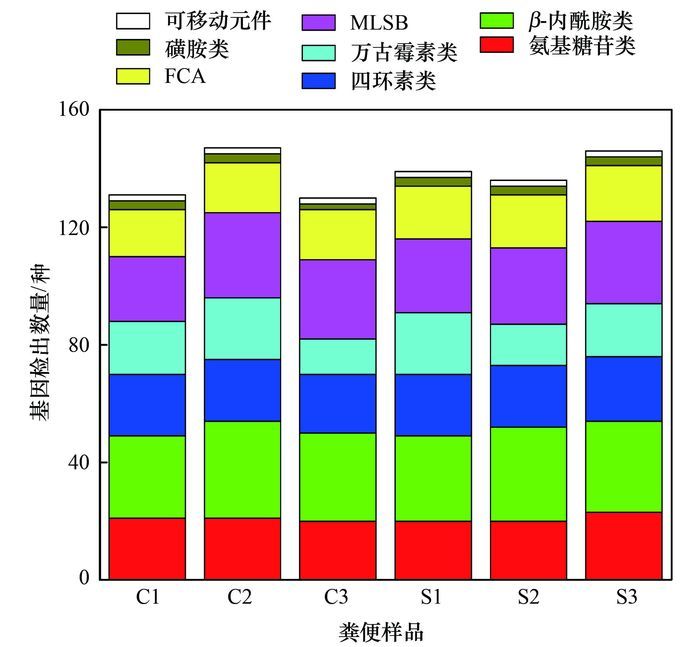
|
图 4 畜禽粪便中耐药基因的检出数量 Fig. 4 Number of antibiotic-resistant genes in livestock manures |
进一步利用未饲用过抗生素的猪粪为对照, 计算猪粪样品中耐药基因的富集情况, 结果显示于表 1.从中可知, 饲喂抗生素的3个猪粪样品中各类耐药基因的富集倍数均明显增加, 总富集倍数达到1.96×104~1.54×105倍.从基因类型来看, 各类耐药基因的平均富集倍数依次为:四环素类>β-内酰胺类>MLSB>氨基糖苷类>FCA>磺胺类>万古霉素类.
|
|
表 1 猪粪样品中耐药基因的富集倍数 Table 1 Enrichment multiple of antibiotic-resistant genes in swine manure samples |
3 讨论
多项研究已证实, 畜禽粪便中普遍存在抗生素耐药细菌[7].其原因一方面是因为粪便中残留的抗生素对微生物群落进行筛选, “淘汰”了一些抗生素敏感菌株; 另一方面, 动物在长期饲喂抗生素的过程中, 肠道微生物在抗生素的选择压力下通过突变产生了耐药性[31].本研究中, 来自不同养殖场的鸡粪、猪粪中都存在高丰度的多重耐药细菌, 这与养殖过程中长期饲喂抗生素有关.同时, 鸡粪中多重耐药细菌的比例显著高于猪粪样品(P<0.05), 该结果与本课题组前期报道的结果类似[19].造成该现象的原因可能与动物体对药物的代谢能力、抗生素饲喂剂量、饲喂方式等有关[32].据调查, 养鸡业中的饲养密度、抗生素使用量、使用频次通常比生猪养殖业要大, 因而导致鸡粪中的抗生素残留水平、耐药细菌和耐药基因丰度相对较高[33].另外, 与哺乳动物相比, 鸡的消化系统不够完善[34], 造成了鸡粪中未消化营养物质的残留, 也为耐药细菌的生长和繁殖提供了良好的生存条件.
单菌株鉴定和群落结构分析的结果均表明, 畜禽粪便中检出的优势多重耐药细菌主要集中在Escherichia、Acinetobacter和Proteus中.其中, 高丰度的Escherichia spp.虽然多为肠道共生菌, 但也可能成为引发尿路感染和胃肠道疾病的条件致病菌[35].同时, 大量具有抗生素耐药性的E. coli在畜禽粪便中被频繁报道, 被视为普遍的粪源性耐药微生物[7, 36], 其在环境中的传播风险应当引起足够的重视. Acinetobacter spp.细菌广泛分布在水体、土壤等自然环境中[37], 其中的某些种在临床上被频繁分离.例如典型的Acinetobacter baumannii被认为是一种医院获得性肺炎的“罪魁祸首”, 可以引起皮肤感染、心内膜炎、败血症和尿路感染等其他疾病[38], 且往往表现出多种耐药性[39].本研究中, 药敏试验的结果进一步验证了随机挑取的多重耐药细菌, 确实表现出对四环素、环丙沙星和庆大霉素的耐药性, 并且这其中可能还包括未检测的更广泛的耐药性或其它种属的耐药菌株.目前, 多重耐药细菌感染人类的病例数量在全球范围内日益增加, 甚至有的“超级细菌”感染已经造成了“无药可治”的局面.据统计, 美国每年有两百万人遭受多重耐药菌的感染, 由此造成的死亡人数达23 000人; 欧洲每年也有25 000人死于多重耐药菌感染[40, 41].因此, 多重耐药细菌在环境中的存在和传播已经成为全球范围内的棘手问题, 通过畜禽粪便进入到自然环境的多重耐药菌, 尤其是耐药性的人类致病菌的存在应该引起足够的关注.
耐药基因的qPCR检测表明, 不论鸡粪还是猪粪, 氨基糖苷类、四环素类、磺胺类和β-内酰胺类抗生素的耐药基因都在其中普遍存在, 同时它们也是在河流、湖泊、污水和农田等自然环境中检出频率较高的耐药基因类型[42, 43].近年来, 作为新型污染物的耐药基因在环境中的污染问题已经越来越多地引发人们的关注.也有学者认为, 耐药基因在环境中的传播主要是由于畜禽粪肥在农业领域中的应用[44].值得注意的是, 本研究检测的两种可移动元件在所有粪便样品中均被100%检出.而已有研究证实, 可移动元件是造成细菌之间甚至不同种属之间基因转移的重要因素——耐药基因可以与可移动元件共存而实现自身的转移和传递, 也有证据表明携带耐药基因的可移动元件能够转移到人体共生菌和致病菌中[45]. Zhu等[46]在研究我国商业养猪场的猪粪、堆肥和施加粪肥的土壤时也发现, 耐药基因与转座子之间呈现很高的正相关性.因此, 本研究中所有粪便样品中可移动元件的检出也提示了畜禽粪便传播耐药基因的风险.进一步比较耐药基因的富集情况也发现, 饲喂抗生素的猪粪与未饲用抗生素的猪粪相比各类耐药基因的富集倍数明显上升, 说明了抗生素的使用对促进细菌耐药性发展的积极影响.实际上, 由于养殖过程中通常是多类型抗生素交替使用, 因此造成了动物粪便中耐药基因类型的多样性, 也为细菌多重耐药性的产生提供了可能.同时, 就抗生素种类而言, 由于四环素类抗生素是传统养殖业中用量最大的类型[47], 因此在饲喂抗生素的猪粪中该类耐药基因的富集倍数也明显最高.
总之, 畜禽粪便已经被认为是耐药细菌和耐药基因的重要富集位点, 这样的粪便施用于农田, 会导致耐药细菌和耐药基因在土壤中的富集, 也存在耐药基因传播扩散甚至促进细菌发展多重耐药性的风险.因此, 控制畜禽粪便中耐药基因的污染, 采用新型有效的处理技术消减粪肥中的抗生素耐药性势在必行.
4 结论(1) 畜禽粪便中普遍存在同时对多种抗生素耐药的多重耐药细菌, 其中包括人类致病菌.
(2) 畜禽粪便中分布有多种耐药基因, 且与非抗生素饲喂的粪便相比, 各类耐药基因的富集倍数明显增加.
| [1] |
朱建春, 张增强, 樊志民, 等. 中国畜禽粪便的能源潜力与氮磷耕地负荷及总量控制[J]. 农业环境科学学报, 2014, 33(3): 435-445. Zhu J C, Zhang Z Q, Fan Z M, et al. Biogas potential, cropland load and total amount control of animal manure in China[J]. Journal of Agro-Environment Science, 2014, 33(3): 435-445. DOI:10.11654/jaes.2014.03.005 |
| [2] |
朱宁, 马骥. 中国畜禽粪便产生量的变动特征及未来发展展望[J]. 农业展望, 2014, 10(1): 46-48. Zhu N, Ma J. Changes and outlook about production amount of livestock and poultry manure in China[J]. Agricultural Outlook, 2014, 10(1): 46-48. |
| [3] | Sun W, Qian X, Gu J, et al. Mechanisms and effects of arsanilic acid on antibiotic resistance genes and microbial communities during pig manure digestion[J]. Bioresource Technology, 2017, 234: 217-223. DOI:10.1016/j.biortech.2017.03.025 |
| [4] | Sarmah A K, Meyer M T, Boxall A B A. A global perspective on the use, sales, exposure pathways, occurrence, fate and effects of veterinary antibiotics (VAs) in the environment[J]. Chemosphere, 2006, 65(5): 725-759. DOI:10.1016/j.chemosphere.2006.03.026 |
| [5] | Hamscher G, Sczesny S, Höper H, et al. Determination of persistent tetracycline residues in soil fertilized with liquid manure by high-performance liquid chromatography with electrospray ionization tandem mass spectrometry[J]. Analytical Chemistry, 2002, 74(7): 1509-1518. DOI:10.1021/ac015588m |
| [6] |
孙刚, 袁守军, 计峰, 等. 畜禽粪便中抗生素残留危害及其研究进展[J]. 环境与健康杂志, 2009, 26(3): 277-279. Sun G, Yuan S J, Ji F, et al. Environmental impact of antibiotics contamination from livestock and poultry dejecta:a review of recent researches[J]. Journal of Environment and Health, 2009, 26(3): 277-279. |
| [7] | Yang Q X, Ren S W, Niu T Q, et al. Distribution of antibiotic-resistant bacteria in chicken manure and manure-fertilized vegetables[J]. Environmental Science and Pollution Research, 2014, 21(2): 1231-1241. DOI:10.1007/s11356-013-1994-1 |
| [8] | Brooks J P, Adeli A, McLaughlin M R. Microbial ecology, bacterial pathogens, and antibiotic resistant genes in swine manure wastewater as influenced by three swine management systems[J]. Water Research, 2014, 57: 96-103. DOI:10.1016/j.watres.2014.03.017 |
| [9] | Chee-Sanford J C, Aminov R I, Krapac I J, et al. Occurrence and diversity of tetracycline resistance genes in lagoons and groundwater underlying two swine production facilities[J]. Applied and Environmental Microbiology, 2001, 67(4): 1494-1502. DOI:10.1128/AEM.67.4.1494-1502.2001 |
| [10] | McKinney C W, Loftin K A, Meyer M T, et al. Tet and sul antibiotic resistance genes in livestock lagoons of various operation type, configuration, and antibiotic occurrence[J]. Environmental Science & Technology, 2010, 44(16): 6102-6109. |
| [11] | Mu Q H, Li J, Sun Y X, et al. Occurrence of sulfonamide-, tetracycline-, plasmid-mediated quinolone-and macrolide-resistance genes in livestock feedlots in northern China[J]. Environmental Science and Pollution Research, 2015, 22(9): 6932-6940. DOI:10.1007/s11356-014-3905-5 |
| [12] |
杨凤霞, 毛大庆, 罗义, 等. 环境中抗生素抗性基因的水平传播扩散[J]. 应用生态学报, 2013, 24(10): 2993-3002. Yang F X, Mao D Q, Luo Y, et al. Horizontal transfer of antibiotic resistance genes in the environment[J]. Chinese Journal of Applied Ecology, 2013, 24(10): 2993-3002. |
| [13] | Blair J M A, Webber M A, Baylay A J, et al. Molecular mechanisms of antibiotic resistance[J]. Nature Reviews Microbiology, 2015, 13(1): 42-51. |
| [14] | Yang Q X, Zhang H, Guo Y H, et al. Influence of chicken manure fertilization on antibiotic-resistant bacteria in soil and the endophytic bacteria of pakchoi[J]. International Journal of Environmental Research and Public Health, 2016, 13(7): 662. DOI:10.3390/ijerph13070662 |
| [15] | Qu A N, Brulc J M, Wilson M K, et al. Comparative metagenomics reveals host specific metavirulomes and horizontal gene transfer elements in the chicken cecum microbiome[J]. PLoS One, 2008, 3(8): e2945. DOI:10.1371/journal.pone.0002945 |
| [16] | Stokes H W, Gillings M R. Gene flow, mobile genetic elements and the recruitment of antibiotic resistance genes into Gram-negative pathogens[J]. FEMS Microbiology Reviews, 2011, 35(5): 790-819. DOI:10.1111/j.1574-6976.2011.00273.x |
| [17] | Hall R M, Collis C M. Mobile gene cassettes and integrons:capture and spread of genes by site-specific recombination[J]. Molecular Microbiology, 1995, 15(4): 593-600. |
| [18] | Willetts N, Wilkins B. Processing of plasmid DNA during bacterial conjugation[J]. Microbiological Reviews, 1984, 48(1): 24-41. |
| [19] |
祁诗月, 任四伟, 李雪玲, 等. 禽畜养殖粪便中多重抗生素抗性细菌研究[J]. 生态学报, 2013, 33(13): 3970-3977. Qi S Y, Ren S W, Li X L, et al. Multidrug-resistant bacteria in livestock feces[J]. Acta Ecologica Sinica, 2013, 33(13): 3970-3977. |
| [20] | 陈天寿. 微生物培养基的制造与应用[M]. 北京: 中国农业出版社, 1995: 179-184. |
| [21] | Clinical and Laboratory Standards Institute. Performance standards for antimicrobial susceptibility testing:twenty-sixth edition, M100S[S]. Wayne, PA:CLSI, 2016. |
| [22] |
唐晔盛, 李英, 朱静洁, 等. 菌落PCR在大规模基因组测序中的应用[J]. 生物化学与生物物理进展, 2002, 29(2): 316-318. Tang Y S, Li Y, Zhu J J, et al. Colony PCR apply to the rice genome sequencing[J]. Progress in Biochemistry and Biophysics, 2002, 29(2): 316-318. |
| [23] | Patel J B. 16S rRNA gene sequencing for bacterial pathogen identification in the clinical laboratory[J]. Molecular Diagnosis, 2001, 6(4): 313-321. DOI:10.2165/00066982-200106040-00012 |
| [24] | Kim H C, Kim Y T, Kim H, et al. Development of broad-range and specific 16S rRNA PCR for use in routine diagnostic clinical microbiology[J]. Journal of Life Science, 2014, 24(4): 361-369. DOI:10.5352/JLS.2014.24.4.361 |
| [25] | Bauer A W, Kirby W M M, Sherris J C, et al. Antibiotic susceptibility testing by a standardized single disk method[J]. American Journal of Clinical Pathology, 1966, 45(4): 493-496. |
| [26] | Caporaso J G, Lauber C L, Walters W A, et al. Global patterns of 16S rRNA diversity at a depth of millions of sequences per sample[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(Suppl 1): 4516-4522. |
| [27] |
温崇庆, 何瑶瑶, 薛明, 等. 高通量测序分析DNA提取引起的对虾肠道菌群结构偏差[J]. 微生物学报, 2016, 56(1): 130-142. Wen C Q, He Y Y, Xue M, et al. Biases on community structure during DNA extraction of shrimp intestinal microbiota revealed by high-throughput sequencing[J]. Acta Microbiologica Sinica, 2016, 56(1): 130-142. |
| [28] | Goldstein C, Lee M D, Sanchez S, et al. Incidence of Class 1 and 2 integrases in clinical and commensal bacteria from livestock, companion animals, and exotics[J]. Antimicrobial Agents and Chemotherapy, 2001, 45(3): 723-726. DOI:10.1128/AAC.45.3.723-726.2001 |
| [29] | Liu M M, Ding R, Zhang Y, et al. Abundance and distribution of Macrolide-Lincosamide-Streptogramin resistance genes in an anaerobic-aerobic system treating spiramycin production wastewater[J]. Water Research, 2014, 63: 33-41. DOI:10.1016/j.watres.2014.05.045 |
| [30] |
黄福义, 李虎, 韦蓓, 等. 长期施用猪粪水稻土抗生素抗性基因污染研究[J]. 环境科学, 2014, 35(10): 3869-3873. Huang F Y, Li H, Wei B, et al. Long-term manure application induced shift of diversity and abundance of antibiotic resistance genes in paddy soil[J]. Environmental Science, 2014, 35(10): 3869-3873. |
| [31] | Heuer H, Schmitt H, Smalla K. Antibiotic resistance gene spread due to manure application on agricultural fields[J]. Current Opinion in Microbiology, 2011, 14(3): 236-243. DOI:10.1016/j.mib.2011.04.009 |
| [32] | Zhao L, Dong Y H, Wang H. Residues of veterinary antibiotics in manures from feedlot livestock in eight provinces of China[J]. Science of the Total Environment, 2010, 408(5): 1069-1075. DOI:10.1016/j.scitotenv.2009.11.014 |
| [33] | Van Houweling C D, Gainer J H. Public health concerns relative to the use of subtherapeutic levels of antibiotics in animal feeds[J]. Journal of Animal Science, 1978, 46(5): 1413-1424. DOI:10.2527/jas1978.4651413x |
| [34] | Lanyasunya T P, Rong W H, Abdulrazak S A, et al. Factors limiting use of poultry manure as protein supplement for dairy cattle on smallholder farms in Kenya[J]. International Journal of Poultry Science, 2006, 5(1): 75-80. DOI:10.3923/ijps.2006.75.80 |
| [35] | Ronald A. The etiology of urinary tract infection:traditional and emerging pathogens[J]. Disease-a-Moutu, 2003, 49(2): 71-82. |
| [36] | Jiang H X, Lü D H, Chen Z L, et al. High prevalence and widespread distribution of multi-resistant Escherichia coli isolates in pigs and poultry in China[J]. The Veterinary Journal, 2011, 187(1): 99-103. DOI:10.1016/j.tvjl.2009.10.017 |
| [37] | Doughari H J, Ndakidemi P A, Human I S, et al. The ecology, biology and pathogenesis of Acinetobacter spp.:an overview[J]. Microbes and Environments, 2011, 26(2): 101-112. DOI:10.1264/jsme2.ME10179 |
| [38] | Dent L L, Marshall D R, Pratap S, et al. Multidrug resistant Acinetobacter baumannii:a descriptive study in a city hospital[J]. BMC Infectious Diseases, 2010, 10: 196. DOI:10.1186/1471-2334-10-196 |
| [39] | Fournier P E, Richet H, Weinstein R A. The epidemiology and control of Acinetobacter baumannii in health care facilities[J]. Clinical Infectious Diseases, 2006, 42(5): 692-699. DOI:10.1086/500202 |
| [40] | Hampton T. Report reveals scope of US antibiotic resistance threat[J]. JAMA, 2013, 310(16): 1661-1663. DOI:10.1001/jama.2013.280695 |
| [41] | Laxminarayan R, Duse A, Wattal C, et al. Antibiotic resistance-the need for global solutions[J]. The Lancet Infectious Diseases, 2013, 13(12): 1057-1098. DOI:10.1016/S1473-3099(13)70318-9 |
| [42] | Lu Z H, Na G S, Gao H, et al. Fate of sulfonamide resistance genes in estuary environment and effect of anthropogenic activities[J]. Science of the Total Environment, 2015, 527-528: 429-438. DOI:10.1016/j.scitotenv.2015.04.101 |
| [43] | Wang F H, Qiao M, Lv Z E, et al. Impact of reclaimed water irrigation on antibiotic resistance in public parks, Beijing, China[J]. Environmental Pollution, 2014, 184: 247-253. DOI:10.1016/j.envpol.2013.08.038 |
| [44] | Byrnebailey K G, Gaze W H, Kay P, et al. Prevalence of sulfonamide resistance genes in bacterial isolates from manured agricultural soils and pig slurry in the United Kingdom[J]. Antimicrobial Agents and Chemotherapy, 2009, 53(2): 696-702. DOI:10.1128/AAC.00652-07 |
| [45] | Allen H K, Donato J, Wang H H, et al. Call of the wild:antibiotic resistance genes in natural environments[J]. Nature Reviews Microbiology, 2010, 8(4): 251-259. DOI:10.1038/nrmicro2312 |
| [46] | Zhu Y G, Johnson T A, Su J Q, et al. Diverse and abundant antibiotic resistance genes in Chinese swine farms[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(9): 3435-3440. DOI:10.1073/pnas.1222743110 |
| [47] | Li Y W, Wu X L, Mo C H, et al. Investigation of sulfonamide, tetracycline, and quinolone antibiotics in vegetable farmland soil in the Pearl River Delta area, southern China[J]. Journal of Agricultural and Food Chemistry, 2011, 59(13): 7268-7276. DOI:10.1021/jf1047578 |
 2018, Vol. 39
2018, Vol. 39


