2. 浙江省农业科学院环境资源与土壤肥料研究所, 杭州 310021
2. Institute of Environment, Resource, Soil and Fertilizer, Zhejiang Academy of Agricultural Sciences, Hangzhou 310021, China
由于畜禽养殖业广泛使用抗生素,特别是以促进畜禽生长为目的的抗生素滥用,导致了大量抗生素抗性微生物(antibiotic resistance bacteria,ARB)和抗生素抗性基因(antibiotic resistance genes,ARGs)的出现[1]. 有研究表明转座子、 整合子、 质粒等[2, 3, 4]介导细菌ARGs的水平传递和扩散会进一步增加环境和人体健康的风险.
畜禽粪便是环境中ARGs的重要来源之一,对于畜禽粪便的安全施用受到广泛关注. 在我国畜禽粪便常作为有机肥的主要来源施用于农田,这可能会造成抗生素抗性基因扩散与传播[5, 6]. 黄福义等[7]报道称长期施用猪粪水稻土壤中抗生素抗性基因的种类和含量均显著高于未施用猪粪有机肥土壤,其中喹诺酮类/氯霉素类抗性基因mexF的丰度是未施用猪粪水稻田的1 791倍. 因此畜禽粪便的安全施用日益受到广泛关注. 好氧堆肥是畜禽粪便无害化、 资源化利用的主要手段. 已有报道称堆肥技术可以降解畜禽粪便中抗生素[8]、 致病微生物[9]和ARGs[10]. 目前,对于ARGs的堆肥法降解大多针对于四环素类和磺胺类抗性基因,而大环内酯类、 β-内酰胺类和喹诺酮类抗性基因的研究较为缺乏. 在我国畜产和水产养殖业,大环内酯类、 β-内酰胺类和喹诺酮类抗生素也大量地使用. Zhang等[11]报道称我国养猪业中常用的36种抗生素使用总量为48 400 t,其中大环内酯类为5 840 t、 β-内酰胺类为10 600 t、 喹诺酮类13 900 t. 生猪养殖废弃物通常会有erm(A、 B、 C、 E 和 F)、 bla(TEM、 SHV、 OXA 和 CTX)和qnr(A、 B 和 S)的存在[12, 13, 14, 15]. 因此有必要对高温堆肥过程对这三类基因的影响进行深入研究.
本文选取杭州市萧山某猪粪有机肥厂的猪粪堆肥为研究对象,采用普通PCR和荧光定量PCR法对堆肥过程不同时段堆肥样品中的大环内酯类、 β-内酰胺类和喹诺酮类ARGs进行定性和绝对定量检测,探究高温堆肥过程对不同类型ARGs的影响,以期为猪粪有机肥安全施用提供理论依据.
1 材料与方法 1.1 供试材料堆肥原料采用猪粪和锯末,取自杭州萧山汇仁有机肥厂. 堆肥初期样品理化性质见表 1.
| 表 1 猪粪堆肥原料理化性质 Table 1 Characteristics of raw materials for swine manure composting |
将辅料(0.7-1 cm的锯末)和猪粪按质量比2 ∶8的比例均匀混合后,堆置成条垛(长5-7 m,宽2.0-2.5 m,高1.2-1.5 m). 猪粪堆肥实验设置3个重复,堆置60 d左右.
1.3 样品采集猪粪样品:采集堆肥第0 d作为空白对照.
堆肥样品:在堆置的第0、 7、 14、 22、 30、 35、 41、 48、 55 和62 d采集样品. 样品为堆体的5个点的上部、 中部和下部取样并制成混合样约1 kg,对200 g样品进行真空冷冻干燥(LABCONCO FreeZone 12 L,美国)以备总基因组DNA提取,200 g鲜样进行理化指标检测,剩余样品保存于-80℃为后续实验用.
总碳和总氮按照中华人民共和国农业行业标准进行检测[16],含水率按质量法测定,pH值按堆肥样品和去离子水1 ∶10的比例稀释,用pH计(PE20,METTLER TOLEDO,Switzerland)测定.
1.4 猪粪堆肥样品DNA提取取真空冷冻干燥样品0.100 0 g,采用DNA提取试剂盒PowerSoil DNA Isolation Kit(MoBio,USA),按照试剂盒说明书进行操作,提取的DNA用超微量紫外-可见分光光度计(NanoDrop2000)检测含量及纯度(A260/A280值在1.8-2.0之间)表明DNA纯度较高.
1.5 普通PCR检测及标准质粒的制备 1.5.1 普通PCR检测实验选择5种常见的大环内酯类抗性基因(ermA、 ermB、 ermC、 ermE和ermF)、 3种β-内酰胺类抗性基因(blaTEM、 blaSHV和blaCTX)及2种喹诺酮类抗性基因(qnrA和qnrS)对样品进行PCR检测,引物设计见表 2. 25 μL的PCR反应体系包含:12.5 μL KAPA2G Fast Multiplex Mix(KAPA Biosystems,USA),1 μL的DNA模板,浓度为10 μmol ·L-1的上下游引物各0.5 μL和10.5 μL的ddH2O. 反应程序:94℃预变性5 min,35个循环为94℃变性30 s,退火30 s(退火温度见表 2),72℃延伸1 min,最终72℃延伸5 min. 引物及退火温度见表 2.
1.5.2 标准质粒的制备将PCR产物点至制备好的1%琼脂糖凝胶点样孔内,经电泳凝胶成像仪观测结果并拍照记录后,切下目的条带并用琼脂糖凝胶回收试剂盒(QIAGEN,Germany)回收纯化. 纯化产物连接到pMD18 T-vector(TaKaRa,Japan)载体上,然后转化感受态大肠杆菌细胞DH5α. 挑取单菌落,用2 mL氨苄抗性的LB培养过夜,用PCR法进行鉴定. 选择阳性菌进行测序,利用BLASTn对比目的序列与测序序列. 用质粒小量提取试剂盒(QIAGEN,Germany)提取重组菌质粒,使用NanoDrop测定质粒浓度. 以所提取的质粒10倍稀释系列(拷贝数从n×1010系列稀释到n×102),以拷贝数为横坐标,Ct值为纵坐标制作标准曲线. 标准质粒拷贝数计算公式如下:
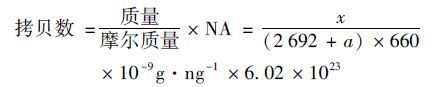
本实验对4种大环内酯类抗性基因(ermA、 ermB、 ermC和ermF)、 3种β-内酰胺类抗性基因(blaTEM、 blaSHV和blaCTX)及2种喹诺酮类抗性基因(qnrA和qnrS)进行定量检测. 荧光定量反应体系为10 μL体系,包括5 μL的2×QuantiFast SYBRGreen PCR Master Mix(QIAGEN,Germany),引物(10 μmol ·L-1)各1 μL,DNA模板1 μL RNase-free water 2 μL. 定量PCR反应程序: 95℃预变性2 min,40个循环包括95℃变性10 s,退火10 s(退火温度见表 2),72℃延伸40s,仪器自动添加溶解曲线程序. 用灭菌超纯水代替样品做为阴性对照,每个样品3次重复.
| 表 2 PCR和Q-PCR引物序列及退火温度 Table 2 Primer sequences and annealing temperature of PCR and Q-PCR assays |
数据采用Excel 2007和SPSS 20.0进行分析,显著性检验采用单因素方差分析.
2 结果与讨论 2.1 猪粪堆肥过程中温度的变化在理化指标中,温度对堆肥的影响最为突出,报道称温度可以促进抗生素和抗生素抗性基因的降解[21]. 本实验记录了猪粪堆肥过程中温度的动态变化,如图 1所示. 在整个堆肥过程中,升温阶段从开始(22℃)到第18 d(53℃);高温期从19 d持续到49 d(55℃);降温期从第50 d开始直至堆肥实验结束(29℃). 这与实验室规模的堆肥温度变化曲线明显不同[18],本实验猪粪堆肥温度的升温阶段缓慢,这是由于C/N比低的缘故. 另外,堆肥条垛比较大,微生物的种类丰富,条件无法如实验室规模那样精细,也可能会导致温度与小规模实验有比较大的差异.
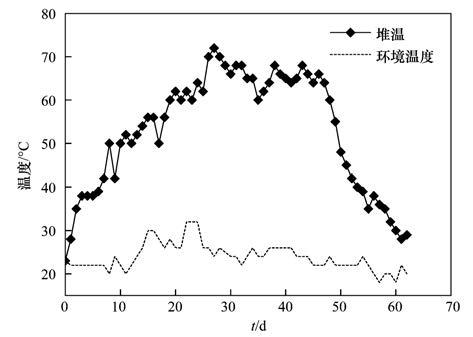 | 图 1 猪粪堆肥过程中温度的变化 Fig. 1 Evolution of temperature during swine manure composting |
本实验从猪粪堆肥样品中共检测到4种大环内酯类抗性基因(ermA、 ermB、 ermC和ermF)、 3种β-内酰胺类抗性基因(blaTEM、 blaSHV和blaCTX)和2种喹诺酮类抗性基因(qnrA和qnrS),并对这9种ARGs进行定量检测. 为保证荧光定量PCR检测结果的可靠性,将目的基因标准品质粒进行测序. 用Blastn对测序结果进行比对,如表 3所示,所得比对结果相似度达99%以上. 扩增效率(Efficiency)在98.181%-119.425%之间,相关系数R2均高于0.990,所有基因溶解曲线只有唯一特异峰,实验选取的9对引物具有较高特异性.
根据标准曲线得到堆肥初始样品(第0 d)中ARGs的丰度和分布情况(表 4)和三类ARGs丰度变化曲线(图 2). 在堆肥初始样品中,大环内酯类抗性基因的丰度比β-内酰胺类抗性基因和喹诺酮类抗性基因高1-2个数量级,其中ermB丰度最高为9.88×108 copies ·g-1.
ARGs普遍存在于环境中,大环内酯类、 β-内酰胺类和喹诺酮类抗生素抗性基因在环境中常常被检出. Marti等[20]在检测了一条污水处理厂附近河流上下游水体和沉积物的qnrA、 qnrB、 qnrS、 blaTEM、 blaCTX、 blaSHV和ermB. Knapp等[12]在养猪废水泻湖处理中检测到ermB、 ermF、 blaTEM和blaSHV,其中ermB丰度最高为108.3 copies ·mL-1. Roberts[19]报道称ermB基因有33个基因型,其是erm类基因型最多的大环内酯类抗生素抗性基因. 由于其具有丰富的基因型,这可能是导致其在环境中更易传播和增殖. 这些原因可能导致堆肥样品中ermB基因的丰度比较高.
| 表 3 9种ARGs的Blastn结果 Table 3 Blastn results of nine ARGs |
| 表 4 猪粪堆肥初始样品中9种ARGs的丰度和分布 /copies ·g-1Table 4 Abundance and distribution of nine ARGs in the intial swine manure composting samples/copies ·g-1 |
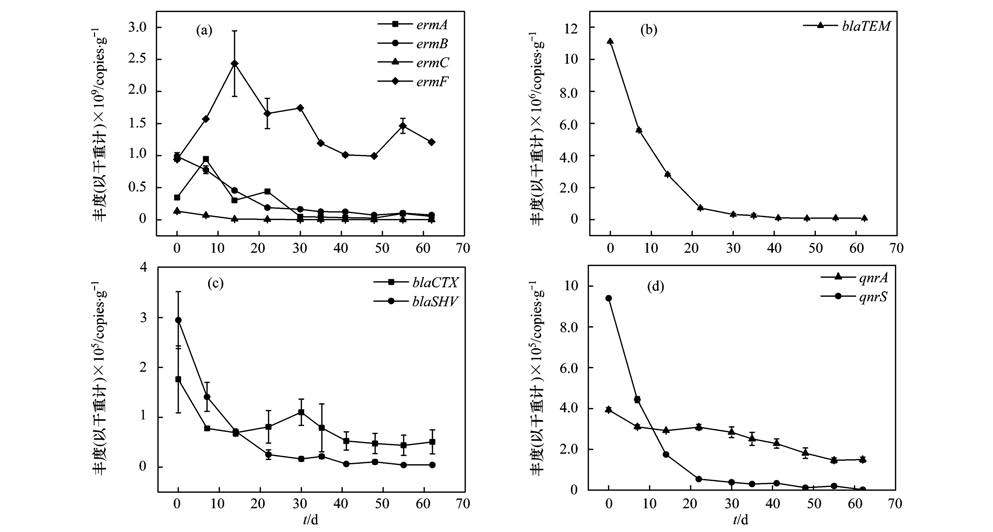 | (a)大环内酯类抗性基因丰度的变化;(b) blaTEM基因丰度的变化; (c)blaCTX和blaSHV基因丰度的变化;(d)喹诺酮类抗性基因丰度的变化 图 2 猪粪堆肥过程中抗生素抗性基因丰度的变化 Fig. 2 Abundance change of ARGs during swine manure composting |
本实验不同的erm基因堆肥中呈现出不同的变化归趋[图 2(a)]. 其中在堆肥升温阶段的第0-7 d,ermA和ermF分别从3.47×108 copies ·g-1和9.41×108 copies ·g-1增加到9.41×108 copies ·g-1和1.57×109 copies ·g-1,之后ermA开始降低,到30 d之后基本维持在107 copies ·g-1,去除率为84.9%;而ermF继续增殖到第14 d,第15-50 d开始降低,但在之后又有上升的趋势,最终比初始堆肥样品高(P>0.05). 在堆肥过程中,ermB和ermC含量从开始就呈现下降趋势,堆肥结束就下降至1.24×106 copies ·g-1和7.47×107 copies ·g-1,去除率分别达到92.4%和99.1%. ermC在堆肥过程中仅仅降低了1个数量级,且保持在107 copies ·g-1左右. 同一ARGs在不同堆肥阶段有增加和下降趋势可能由于不同堆肥阶段微生物群落结构会有不同[20, 22],这可能会造成携带不同类型抗生素抗性基因的变化趋势会有不同. 与bla基因相比,erm基因则以质粒和转座子在革兰氏阳性菌和革兰氏阴性菌均有发现[19];Liu等[3]在四环素废水处理系统中的抗生素抗性基因与转座子intI 1 的分布特征和丰度变化研究中发现,intI 1 与抗性基因的增殖有关; Su等[23]报道称可动遗传因子(mobile genetic elements,MEG)和群落结构会影响抗生素抗性基因的分布和归趋. 因此,erm基因较难去除且环境扩散性比较强.
2.4 猪粪堆肥过程中β-内酰胺类抗性基因丰度的变化从堆肥开始之初,β-内酰胺类抗性基因丰度[图 2(b)和图 2(c)]含量开始就呈现下降趋势,在堆肥到第22 d后bla基因降解速率开始降低. 堆肥结束时blaTEM含量为8.71×104 copies ·g-1,去除率达到了99.9%,比初始堆肥样品降低了3个数量级;blaCTX堆肥结束后含量为2.42×104 copies ·g-1,去除率为63.8%,降低了1个数量级. blaSHV堆肥结束后含量为4.42×103 copies ·g-1,去除率为98.5%,降低了2个数量级. blaTEM和blaSHV经常伴随质粒在肠杆菌中发现[24],肠杆菌是一类革兰氏阴性菌、 兼性厌氧微生物. 因此,在高温好氧堆肥作用下肠杆菌大多会被杀灭,其携带的抗生素抗性也随之降解. 在堆肥进行完成之后,这3种β-内酰胺类抗生素抗性基因依然保持在较低含量. 这可能由于在猪粪堆肥翻堆时,部分在条垛的底层温度比较低且是局部厌氧环境.
2.5 猪粪堆肥过程中喹诺酮类抗性基因丰度的变化整个堆肥过程中,喹诺酮类抗性基因丰度的变化[图 2(d)]. 本实验堆肥初始时qnrA和qnrS的丰度都比erm和bla抗性基因丰度低,分别为3.9×105 copies ·g-1和9.4×105 copies ·g-1. 堆肥过程中qnrS呈现急剧降低趋势,在堆肥结束时含量已降至2.6×103 copies ·g-1,去除率达到99.7%. 与qnrS相比,qnrA含量随着堆肥的进行降解速率较为缓慢,堆肥结束时丰度为1.5×105 copies ·g-1,去除率达到61.8%.
Marti等[20]在研究废水处理系统和排入河流的上下游的水体和污泥里ARGs的分布和丰度结果显示,qnrA在河流下游检测不到,而qnrS在下游河床沉积物中含量要高,并且不同类型的抗生素抗性基因表现出不同的丰度和归趋. 这可能由于qnrS宿主菌大多是嗜温菌,因此,在堆肥结束后检测丰度比较低[22, 25]. 而qnrA的基因型比qnrS要多且大多是通过质粒介导基因的水平传递[26, 27],这样导致前者比后者更难从堆肥产品中去除.
3 结论(1)在堆肥初始红霉素类抗性基因拷贝数明显高于β-内酰胺类和喹诺酮类抗性基因.
(2)不同类型的抗生素抗性基因具有不同变化趋势,blaTEM去除率最高,为99.9%;而ermF却在堆肥结束后增加了28.7%;同一类型ARGs 中不同种类的ARGs去除效率也有较大差异.
| [1] | Jechalke S, Heuer H, Siemens J, et al. Fate and effects of veterinary antibiotics in soil[J]. Trends in Microbiology, 2014, 22 (9): 536-545. |
| [2] | Stokes H W, Hall R M. A novel family of potentially mobile DNA elements encoding site-specific gene-integration functions: integrons[J]. Molecular Microbiology, 1989, 3 (12): 1669-1683. |
| [3] | Liu M M, Zhang Y, Yang M, et al. Abundance and distribution of tetracycline resistance genes and mobile elements in an oxytetracycline production wastewater treatment system[J]. Environmental Science & Technology, 2012, 46 (14): 7551-7557. |
| [4] | Kocsis B, Kocsis E, Fontana R, et al. Identification of blaLAP-2 and qnrS1 genes in the internationally successful Klebsiella pneumoniae ST147 clone[J]. Journal of Medical Microbiology, 2013, 62 (Pt 2): 269-273. |
| [5] | Udikovic-Kolic N, Wichmann F, Broderick N A, et al. Bloom of resident antibiotic-resistant bacteria in soil following manure fertilization[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111 (42): 15202-15207. |
| [6] | Hsu J T, Chen C Y, Young C W, et al. Prevalence of sulfonamide-resistant bacteria, resistance genes and integron-associated horizontal gene transfer in natural water bodies and soils adjacent to a swine feedlot in northern Taiwan[J]. Journal of Hazardous Materials, 2014, 277: 34-43. |
| [7] | 黄福义, 李虎, 韦蓓, 等. 长期施用猪粪水稻土抗生素抗性基因污染研究[J]. 环境科学, 2014, 35 (10): 3869-3873. |
| [8] | Ho Y B, Zakaria M P, Latif P A, et al. Degradation of veterinary antibiotics and hormone during broiler manure composting[J]. Bioresource Technology, 2013, 131: 476-484. |
| [9] | Hanajima D, Kuroda K, Fukumoto Y, et al. Effect of addition of organic waste on reduction of Escherichia coli during cattle feces composting under high-moisture condition[J]. Bioresource Technology, 2006, 97 (14): 1626-1630. |
| [10] | Wang J, Ben W W, Zhang Y, et al. Effects of thermophilic composting on oxytetracycline, sulfamethazine, and their corresponding resistance genes in swine manure[J]. Environmental Science: Processes & Impacts, 2015, 17 (9): 1654-1660. |
| [11] | Zhang Q Q, Ying G G, Pan C G, et al. A comprehensive evaluation of antibiotics emission and fate in the river basins of China: source analysis, multimedia modeling, and linkage to bacterial resistance[J]. Environmental Science & Technology, 2015, 49 (11): 6772-6782. |
| [12] | Knapp C W, Zhang W, Sturm B S M, et al. Differential fate of erythromycin and beta-lactam resistance genes from swine lagoon waste under different aquatic conditions[J]. Environmental Pollution, 2010, 158 (5): 1506-1512. |
| [13] | Garofalo C, Vignaroli C, Zandri G, et al. Direct detection of antibiotic resistance genes in specimens of chicken and pork meat[J]. International Journal of Food Microbiology, 2007, 113 (1): 75-83. |
| [14] | Bibbal D, Dupouy V, Ferré J P, et al. Impact of three ampicillin dosage regimens on selection of ampicillin resistance in Enterobacteriaceae and excretion of blaTEM genes in swine feces[J]. Applied and Environmental Microbiology 2007, 73 (15): 4785-4790. |
| [15] | Zhu Y G, Johnson T A, Su J Q, et al. Diverse and abundant antibiotic resistance genes in Chinese swine farms[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110 (9): 3435-3440. |
| [16] | NY 525-2012, 有机肥料[S]. |
| [17] | Strommenger B, Kettlitz C, Werner G, et al. Multiplex PCR assay for simultaneous detection of nine clinically relevant antibiotic resistance genes in Staphylococcus aureus[J]. Journal of Clinical Microbiology, 2003, 41 (9): 4089-4094. |
| [18] | 韦蓓, 黄福义, 李虎, 等. 污泥堆肥过程中磺胺类和大环内酯类抗性基因的残留[J]. 应用与环境生物学报, 2014, 20 (3): 395-400. |
| [19] | Roberts M C. Update on macrolide-lincosamide-streptogramin, ketolide, and oxazolidinone resistance genes[J]. FEMS Microbiology Letters, 2008, 282 (2): 147-159. |
| [20] | Marti E, Jofre J, Balcazar J L. Prevalence of antibiotic resistance genes and bacterial community composition in a river influenced by a wastewater treatment plant[J]. PLoS One, 2013, 8 (10): e78906. |
| [21] | Selvam A, Xu D L, Zhao Z Y, et al. Fate of tetracycline, sulfonamide and fluoroquinolone resistance genes and the changes in bacterial diversity during composting of swine manure[J]. Bioresource Technology, 2012, 126: 383-390. |
| [22] | 王伟东, 王小芬, 朴哲, 等. 堆肥化过程中微生物群落的动态[J]. 环境科学, 2007, 28 (11): 2591-2597. |
| [23] | Su J Q, Wei B, Ouyang W Y, et al. Antibiotic resistome and its association with bacterial communities during sewage sludge composting[J]. Environmental Science & Technology, 2015, 49 (12): 7356-7363. |
| [24] | Bradford P A. Extended-spectrum β-lactamases in the 21st century: characterization, epidemiology, and detection of this important resistance threat[J]. Clinical Microbiology Reviews, 2001, 14 (4): 933-951. |
| [25] | Strahilevitz J, Jacoby G A, Hooper D C, et al. Plasmid-mediated quinolone resistance: a multifaceted threat[J]. Clinical Microbiology Reviews, 2009, 22 (4): 664-689. |
| [26] | Jacoby G, Cattoir V, Hooper D, et al. qnr Gene nomenclature[J]. Antimicrobial Agents and Chemotherapy, 2008, 52 (7): 2297-2299. |
| [27] | Robicsek A, Strahilevitz J, Sahm D F, et al. qnr Prevalence in ceftazidime-resistant Enterobacteriaceae isolates from the United States[J]. Antimicrobial Agents and Chemotherapy, 2006, 50 (8): 2872-2874. |
 2016, Vol. 37
2016, Vol. 37


