四环素类抗生素(tetracyclines, TCs)是一类广谱药物, 具有较好地抗菌性能和较低的副作用, 已被广泛用于细菌感染的治疗和控制, 并作为饲料添加剂以促进动物生长.据统计, 我国2013年抗生素使用量达16.2万t, 其中TCs约1.2万t, 占7.4%[1].由于TCs进入人体和动物体内后只有部分能被代谢吸收, 大部分未被吸收的抗生素母体及其代谢产物通过尿液和粪便被排入污水, 导致相当数量的TCs进入生态环境中.
近年来, 越来越多的研究报道在不同的环境介质中检出不同浓度水平的TCs[2~4], 同时也检出较高丰度的四环素抗性细菌(TRB)和抗性基因(TC-ARGs)[5].TCs在环境中的长期残留对细菌微生物具有选择性效应, 能诱导其产生抗药性, 并促进TC-ARGs的转移和散播[6~9].前期研究发现, 污水中TCs浓度与TRB丰度具有显著相关性, 但与tetO和tetW基因丰度之间相关性不显著, 表明TCs暴露对TRB的演变可能具有促进作用[10].有研究表明, 即使较低浓度的TCs仍可诱导释放TC-ARGs[11].由此可见, 关于TCs对TC-ARGs演变行为的过程影响仍尚不明晰[12].虽然目前已有较多针对环境中TC-ARGs的丰度水平及其分布归趋的研究报道[13~15], 但关于其抗性表达水平及影响因子方面的研究还很少, TC-ARGs丰度的高低并不能准确地反映其实际风险水平, 相比之下, TC-ARGs抗性表达的强弱更能体现其功能活性和环境风险[16].
基于此, 本文以从污水处理活性污泥中筛选分离的TRB作为研究对象, 采用荧光定量PCR(qPCR)和逆转录PCR(RT-PCR)方法对TC-ARGs丰度及转录表达水平进行定量检测分析, 考察了不同浓度四环素(TC)抗生素暴露对TC-ARGs丰度变化及其表达水平的作用影响, 探讨了TC与TC-ARGs丰度及其表达水平之间的相关关系, 以期为环境中ARGs的实际风险评估提供数据支持.
1 材料与方法 1.1 TRB筛选污泥样品取自实验室厌氧-好氧活性污泥系统沉淀池[7], 量取10 mL泥水混合物[VSS=(9.96±0.12)g ·L-1], 在4℃和10 000 r ·min-1条件下离心10 min(Heraeus Multifuge X1R, Thermo Scientific), 弃去上清液, 收集沉淀物.称取0.1 g沉淀物分散至100 mL含TC的LB培养基中, 然后置于(37±2)℃恒温振荡培养箱(SPH-1102C, 上海世平实验设备有限公司), 在150 r ·min-1转速下培养20 h后获得培养混合液;量取1 mL培养混合液于100 mL含TC的LB培养基中再次培养筛选, 按此操作反复培养7 d, 以获得不含固体颗粒的TRB混合菌.LB培养基组成如下:10 mg ·L-1 TC, 10 g ·L-1 NaCl, 10 g ·L-1胰蛋白胨, 5 g ·L-1酵母提取物[生工生物工程(上海)股份有限公司], 其中TC筛选浓度参照CLSI(Clinical Laboratory Standards Institute)中针对TC对细菌微生物的致死浓度进行确定[17].
1.2 不同TC浓度下TRB培养条件取100 μL在1.1节筛选所得的TRB样品D600=(0.8±0.02)于100 mL LB培养基中, TC添加浓度分别为10 μg ·L-1、100 μg ·L-1、1 mg ·L-1、10 mg ·L-1和100 mg ·L-1, 另外设置不添加TC的LB培养基作为空白对照, 所有操作均设置3个平行组.为考察TC对TRB的长期胁迫效应, 每20 h作为一个周期, 每周期培养结束后, 将培养菌液在4℃和12 000 r ·min-1条件下离心5 min, 收集菌体沉淀, 使用磷酸盐缓冲溶液进行菌体重悬、漂洗和富集, 随后作为下一周期的接种菌液, 反复连续培养7 d.
1.3 TRB菌体基因组、RNA提取及单链cDNA的合成将TRB培养后所得培养基在4℃和12 000 r ·min-1条件下离心, 收集菌体沉淀, 使用磷酸盐缓冲溶液反复清洗以除去残留培养基, 然后分别采用细菌基因组DNA试剂盒和RNA prep Pure试剂盒(TIANGEN)提取TRB菌体的DNA和RNA.将RNA样品与FastKing cDNA第一链合成试剂盒(TIANGEN)中的预混试剂混合均匀, 利用高效反转录酶(FastKing)的强RNA亲和性和热稳定性, 在42℃下反应15 min, 完成cDNA第一链的合成.此外, 预混试剂中含有的热敏gDNase能够高效去除基因组DNA, 以消除基因组DNA对RNA的干扰影响.cDNA合成结束后, 再于95℃下反应3 min, 以获得灵敏度高且稳定性好的单链cDNA样品.采用Qubit® 2.0核酸蛋白测定仪(Invitrogen)分别测定DNA、RNA和cDNA的浓度, 所有样品均保存于-80℃冰箱中.
1.4 TC-ARGs丰度变化及其表达水平采用qPCR(LightCycler® 96, Roche)对TC-ARGs丰度及其表达水平进行定量检测, 目标基因所用的扩增引物序列、扩增子大小、退火温度, 以及热循环反应程序等详见文献[18, 19].qPCR反应采用20 μL体系, 包括Essential DNA Green Master 10 μL, 上下游引物各1.5 μL, DNA或cDNA模板1 μL, ddH2O 6 μL.每组样品均设置3个平行样, 并使用无菌水作为阴性对照.利用熔解曲线(65~95℃, 每0.5℃读数)分析扩增产物特异性, 优化qPCR反应条件和引物浓度, 以消除引物二聚体和非特异性扩增对结果的干扰影响.
根据Lin等[20]的研究报道, 采用2-ΔΔCt法计算TC-ARGs的转录表达水平, 方法如下:
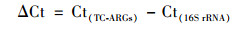
|
(1) |
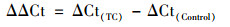
|
(2) |

|
(3) |
式中, Ct为循环阈值, 空白对照为不含TC的LB培养基.
1.5 数据分析采用qPCR自带的SW 1.1软件计算基因丰度;运用Microsoft Excel 2017分析TC-ARGs丰度变化及其转录表达水平;采用SPSS 22.0进行数据统计学分析, 设置统计检验显著性水平P < 0.05具有显著相关性.所有图表均采用Origin 8.5软件绘制.
2 结果与讨论 2.1 不同浓度TC对TC-ARGs丰度变化的作用影响图 1为TC-ARGs在不同浓度TC暴露下的丰度变化情况.分析可知, 在未添加TC的空白培养基中, TC-ARGs丰度水平在整个培养周期内变化相对稳定, 表明TC并非是影响TC-ARGs基因丰度变化的决定性因素.Wan等[21]的研究发现, 有机碳含量对饮用水中ARGs丰度具有重要影响, 这也可能是本研究中TC-ARGs丰度在整个培养周期维持在稳定水平的潜在原因之一, 某种程度上也说明禁止或减少抗生素的使用可能对ARGs丰度的削减或消失影响较小[22], 然而目前关于抗生素对ARGs的直接影响机制还尚不明晰[12].有研究报道, 大肠杆菌在TC胁迫下能够启动节能存活模式, 通过自我保护机制抵御TC的细胞毒性[23], 且适宜的TC浓度会诱导ARGs产生, 并增大细菌突变的发生几率[24].细菌ARGs常由抗药基因组中的编码ARGs演变而来, 长期抗生素暴露可以强化细菌抗药特性, 导致此类编码抗药性的前体基因演变形成基因组ARGs[25].此外, 抗生素胁迫还会促进抗生素抗性变异体在细菌体之间的散播[26], 这可能是导致tetW基因丰度上升的原因(图 1).tetM和tetW基因丰度均于第3 d发生上浮, 随后出现小幅度下跌并逐渐趋于平稳.Aires等[27]在针对最小抑制浓度(MIC)TC存在条件下筛选获得的人体肠道双歧杆菌(Bifidobacterium)染色体DNA杂交实验中发现, tetM和tetW基因能于同一条染色体下进行复制, 这可能是因为混合菌体系中发生了同源DNA基因交换, 从而出现tetM和tetW基因丰度波动类似的情况.

|
图 1 不同浓度TC胁迫下TC-ARGs的丰度变化 Fig. 1 Gene abundances of TC-ARGs under stresses of different TC concentrations |
tetO基因丰度在整个培养周期内变化不显著(P>0.05), 表明不同浓度TC(10 μg ·L-1~100 mg ·L-1)胁迫对其丰度变化影响较小.相比之下, 其它TC-ARGs在不同浓度TC胁迫下的响应行为差别较大, 其中tetA、tetG和tetW基因丰度随TC浓度的升高基本呈现出逐渐上升的趋势, 且tetA和tetW基因丰度与TC浓度显著正相关(P < 0.05).当TC浓度小于100 μg ·L-1时, tetC基因丰度变化较小, 而当TC浓度为1 mg ·L-1时, tetC基因丰度显著升高, 且随着TC浓度的继续升高呈现显著降低的趋势(P < 0.05).tetM基因丰度总体上随着TC浓度的升高而降低.由此可见, 不同浓度TC胁迫对不同类型TC-ARGs的作用影响有所不同, 且TC浓度与TC-ARGs基因丰度之间并未呈现线性对应变化趋势, 这可能与目标TRB体系有关.由于本研究所使用的是从污水污泥中筛选获得的TRB混合菌体系, 不同类别的TRB对TC胁迫的耐受程度和响应行为具有差异性, 一些只能够耐受相对较低浓度TC(如10 μg ·L-1和100 μg ·L-1)的TRB在高浓度TC(100 mg ·L-1)胁迫下反而可能会受到抑制甚至死亡, 从而导致其携带的TC-ARGs丰度发生变化.此外, 低浓度TC胁迫可能是导致TC-ARGs在环境介质中持续稳定存在的原因之一, 如tetA、tetC、tetG和tetX, 其基因丰度在整个培养周期内变化不显著(P>0.05).
2.2 不同浓度TC对TC-ARGs表达水平的作用影响图 2为不同浓度TC对TC-ARGs表达水平的影响过程.分析可知, 在短期(1 d)TC胁迫下, TC-ARGs转录表达水平整体上随TC浓度的升高呈现逐渐上调的变化趋势, 表明TC暴露对其ARGs的转录表达具有促进作用, 这可能与TRB在应对TC胁迫时所作出的应急响应有关.Versluis等[28]的研究表明, 抗生素胁迫作用能够促使ARGs表达更加活跃.在TC选择性压力下, TC能够影响TRB中TC-ARGs启动子活性, 而启动子可调控外排泵蛋白、核糖体蛋白或酶修饰蛋白等的表达, 从而影响TC与其作用位点之间的有效结合[29].

|
图 2 不同浓度TC胁迫下TC-ARGs的转录表达水平 Fig. 2 Transcriptional expression levels of TC-ARGs under stresses of different TC concentrations |
tetA基因在整个培养周期内(7 d)表现出较为稳定的抗性表达水平, 且当TC浓度为100 mg ·L-1, tetA基因转录表达水平显著上调(P < 0.05), 上调倍数最高达5.3倍, 这可能是因为载有tetA基因的TRB本身能够耐受较高浓度的TC(如100 mg ·L-1).此外, 长期暴露在亚致死浓度的抗生素条件下能够刺激产生活性氧物质[24], 从而导致基因发生变异, 诱导ARB耐受更高浓度的抗生素[26].Goh等[30]的研究发现, 大肠杆菌在MIC浓度抗生素胁迫条件下, 对应ARGs的转录表达水平平均上调2~10倍.Ramos等[31]的研究报道, tetA基因对TC具有一定的选择性, 其表达水平受tetR基因负调控, tetR基因对TC浓度变化较为敏感, 进而会影响tetA基因的表达水平.有研究报道, 基因变异或遗传因子的转移同样会导致ARGs表达水平的上调[32].tetX基因表达水平在培养周期内整体呈现逐渐下调的变化趋势, 由于tetX为酶修饰基因, 其编码的蛋白具有化学修饰和降解TC的功能[33], 因此能够通过修饰或分解TC分子以削弱TC对TRB的胁迫影响, 从而降低其转录表达水平.
除tetA和tetX基因外, 其余TC-ARGs的表达水平在整个培养周期内未表现出明显的变化规律, 波动较大.由于本研究所采用的为TRB混合菌体系, 不同类别的TRB对TC胁迫的耐受程度具有差异性, 从而导致其所携带的TC-ARGs表达水平的不同.在长期(7 d)培养基培养过程中, TRB对TC可能会发生部分降解, 从而削弱TC对TRB的胁迫作用.此外, 与TC相比, TC降解产物可能毒性更高, 对TRB的胁迫或抑制作用更为显著, 进而影响TRB的表达水平.在短期(1 d)培养基培养过程中, TRB对TC的降解影响很小, TC是对TRB形成胁迫效应的主要因素, 因此TC浓度梯度对TC-ARGs表达水平的影响作用较为显著(图 2).
2.3 相关性分析由表 1分析可知, 除tetO和tetX基因外, 其余TC-ARGs丰度与TC浓度之间存在显著相关性(P < 0.05), 表明TC浓度变化对TC-ARGs丰度的演变具有较大影响, 其中TC浓度与tetA、tetG和tetW基因丰度显著正相关, 但与tetC和tetM基因丰度显著负相关.Huang等[34]的研究同样发现, 污泥中tetA和tetG基因丰度随着TC暴露浓度的升高而发生显著增殖.由此可见, TC胁迫对不同类型TC-ARGs的作用影响程度不同, 这可能与载有TC-ARGs的TRB种类及其对TC的耐受程度有关.此外, TC浓度与tetA、tetW和tetX基因转录表达水平之间存在显著正相关性(P < 0.01), 表明TC胁迫能够刺激携带有tetA、tetW和tetX基因的TRB作出应急响应, 使其抗性表达水平显著上调.相比之下, tetC、tetG、tetM和tetO基因转录表达水平与TC浓度之间并未发现具有显著相关性, 这可能是因为携带有这些TC-ARGs的TRB对TC胁迫的响应差异所造成的, 也有可能TRB通过其它方式抵御TC的胁迫作用[35].
|
|
表 1 TC胁迫过程中与TC-ARGs丰度和表达水平相关性分析1) Table 1 Correlations between TC concentrations and gene abundances and expression levels of TC-ARGs |
从图 3可以看出, tetA和tetW基因丰度与其转录表达水平之间具有显著的线性相关关系(P < 0.001), 某种程度上表明tetA和tetW基因表达的强弱可以采用其基因丰度的高低进行衡量和评价, 同时结合表 1可以发现, TC胁迫可能是引起tetA和tetW基因丰度和表达水平升高的主要原因.除此之外, 其它TC-ARGs基因丰度与其转录表达水平之间线性相关关系不显著(P>0.05), 表明TC-ARGs基因丰度并不能代表其转录表达水平, 而ARGs表达水平的高低直接关系着其功能活性和环境风险[14].

|
图 3 TC-ARGs丰度与其表达水平相关性分析 Fig. 3 Correlations between gene abundances of TC-ARGs and their expression levels |
(1) 在不同浓度TC胁迫条件下, TC-ARGs基因丰度变化差异较大, 其中tetA、tetG和tetW基因丰度随着TC浓度的升高呈现上升趋势.
(2) 短期TC胁迫对TC-ARGs转录表达具有促进作用, 但长期TC暴露对TC-ARGs表达水平的作用影响差别较大.其中, tetA基因表达水平相对较为稳定, 当TC浓度为100 mg ·L-1时, tetA基因转录表达水平显著上调, 上调倍数最高达5.3倍.相比之下, tetX基因转录表达水平呈现逐渐下调趋势, 其余TC-ARGs表达水平在培养周期内变化波动较大.
(3) 由相关性分析结果可知, TC与TC-ARGs (除tetO和tetX基因外)之间存在显著相关性, 且与tetA、tetW和tetX基因转录表达水平之间存在显著正相关性, 表明TC胁迫对TC-ARGs的丰度变化及表达水平具有重要影响.此外, tetA和tetW基因丰度与其转录表达水平之间具有显著线性相关性, 表明一定程度上可采用其基因丰度变化反映其表达水平的变化.
| [1] | Zhang Q Q, Ying G G, Pan C G, et al. Comprehensive evaluation of antibiotics emission and fate in the river basins of China:source analysis, multimedia modeling, and linkage to bacterial resistance[J]. Environmental Science & Technology, 2015, 49(11): 6772-6782. |
| [2] | Daghrir R, Drogui P. Tetracycline antibiotics in the environment:a review[J]. Environmental Chemistry Letters, 2013, 11(3): 209-227. DOI:10.1007/s10311-013-0404-8 |
| [3] | Carvalho I T, Santos L. Antibiotics in the aquatic environments:a review of the European scenario[J]. Environment International, 2016, 94: 736-757. DOI:10.1016/j.envint.2016.06.025 |
| [4] |
武旭跃, 邹华, 朱荣, 等. 太湖贡湖湾水域抗生素污染特征分析与生态风险评价[J]. 环境科学, 2016, 37(12): 4596-4604. Wu X Y, Zou H, Zhu R, et al. Occurrence, distribution and ecological risk of antibiotics in surface water of the Gonghu Bay, Taihu Lake[J]. Environmental Science, 2016, 37(12): 4596-4604. |
| [5] | Suzuki S, Pruden A, Virta M, et al. Editorial:antibiotic resistance in aquatic systems[J]. Frontiers in Microbiology, 2017, 8: 14. |
| [6] | Song J X, Rensing C, Holm P E, et al. Comparison of metals and tetracycline as selective agents for development of tetracycline resistant bacterial communities in agricultural soil[J]. Environmental Science & Technology, 2017, 51(5): 3040-3047. |
| [7] | Gao P, Xu W L, Ruan X H, et al. Long-term impact of a tetracycline concentration gradient on the bacterial resistance in anaerobic-aerobic sequential bioreactors[J]. Chemosphere, 2018, 205: 308-316. DOI:10.1016/j.chemosphere.2018.04.135 |
| [8] |
张俊, 罗方园, 熊浩徽, 等. 环境因素对土壤中几种典型四环素抗性基因形成的影响[J]. 环境科学, 2014, 35(11): 4267-4274. Zhang J, Luo F Y, Xiong H H, et al. Effect of environmental factors on the formation of several typical tetracycline resistance genes in soil[J]. Environmental Science, 2014, 35(11): 4267-4274. |
| [9] |
张俊, 杨晓洪, 葛峰, 等. 长期施用四环素残留猪粪对土壤中耐药菌及抗性基因形成的影响[J]. 环境科学, 2014, 35(6): 2374-2380. Zhang J, Yang X H, Ge F, et al. Effects of long-term application of pig manure containing residual tetracycline on the formation of drug-resistant bacteria and resistance genes[J]. Environmental Science, 2014, 35(6): 2374-2380. |
| [10] | Gao P, Munir M, Xagoraraki I. Correlation of tetracycline and sulfonamide antibiotics with corresponding resistance genes and resistant bacteria in a conventional municipal wastewater treatment plant[J]. Science of the Total Environment, 2012, 421-422: 173-183. DOI:10.1016/j.scitotenv.2012.01.061 |
| [11] | Pruden A, Larsson D G J, Amézquita A, et al. Management options for reducing the release of antibiotics and antibiotic resistance genes to the environment[J]. Environmental Health Perspectives, 2013, 121(8): 878-885. DOI:10.1289/ehp.1206446 |
| [12] | Sengupta S, Chattopadhyay M K, Grossart H P. The multifaceted roles of antibiotics and antibiotic resistance in nature[J]. Frontiers in Microbiology, 2013, 4: 47. |
| [13] | Li B, Yang Y, Ma L P, et al. Metagenomic and network analysis reveal wide distribution and co-occurrence of environmental antibiotic resistance genes[J]. The ISME Journal, 2015, 9(11): 2490-2502. DOI:10.1038/ismej.2015.59 |
| [14] | Zhou Y T, Niu L L, Zhu S Y, et al. Occurrence, abundance, and distribution of sulfonamide and tetracycline resistance genes in agricultural soils across China[J]. Science of the Total Environment, 2017, 599-600: 1977-1983. DOI:10.1016/j.scitotenv.2017.05.152 |
| [15] |
黄圣琳, 何势, 魏欣, 等. 污水处理厂中四环素类抗生素残留及其抗性基因污染特征研究进展[J]. 化工进展, 2015, 34(6): 1779-1785. Huang S L, He S, Wei X, et al. Pollution characteristics of tetracycline residues and tetracycline resistance genes in sewage treatment plants:a review[J]. Chemical Industry and Engineering Progress, 2015, 34(6): 1779-1785. |
| [16] | Sorek R, Cossart P. Prokaryotic transcriptomics:a new view on regulation, physiology and pathogenicity[J]. Nature Reviews Genetics, 2010, 11(1): 9-16. DOI:10.1038/nrg2695 |
| [17] | Reller L B, Weinstein M, Jorgensen J H, et al. Antimicrobial susceptibility testing:a review of general principles and contemporary practices[J]. Clinical Infectious Diseases, 2009, 49(11): 1749-1755. DOI:10.1086/647952 |
| [18] |
魏欣, 薛顺利, 杨帆, 等. 零价铁对污泥高温厌氧消化过程中四环素抗性基因及第一类整合子的消减影响[J]. 环境科学, 2017, 38(2): 697-702. Wei X, Xue S L, Yang F, et al. Effect of zero valent iron on the decline of tetracycline resistance genes and class 1 integrons during thermophilic anaerobic digestion of sludge[J]. Environmental Science, 2017, 38(2): 697-702. |
| [19] |
杨帆, 徐雯丽, 钱雅洁, 等. 零价铁对污泥厌氧消化过程中四环素抗性基因水平转移的作用影响[J]. 环境科学, 2018, 39(4): 1748-1755. Yang F, Xu W L, Qian Y J, et al. Effect of zero valent iron on the horizontal gene transfer of tetracycline resistance genes during anaerobic sludge digestion process[J]. Environmental Science, 2018, 39(4): 1748-1755. |
| [20] | Lin W F, Li S, Zhang S T, et al. Reduction in horizontal transfer of conjugative plasmid by UV irradiation and low-level chlorination[J]. Water Research, 2016, 91: 331-338. DOI:10.1016/j.watres.2016.01.020 |
| [21] | Wan K, Zhang M L, Ye C S, et al. Organic carbon:an overlooked factor that determines the antibiotic resistome in drinking water sand filter biofilm[J]. Environment International, 2019, 125: 117-124. DOI:10.1016/j.envint.2019.01.054 |
| [22] | Tamminen M, Karkman A, Lõhmus A, et al. Tetracycline resistance genes persist at aquaculture farms in the absence of selection pressure[J]. Environmental Science & Technology, 2011, 45(2): 386-391. |
| [23] |
赖晓琳, 吴平霄, 阮博. 四环素对大肠杆菌抗生素抗性基因进化的影响[J]. 环境科学学报, 2019, 39(8): 2475-2482. Lai X L, Wu P X, Ruan B. Evolution of antibiotic resistance genes in E. coli by tetracycline[J]. Acta Scientiae Circumstantiae, 2019, 39(8): 2475-2482. |
| [24] | Kohanski M A, DePristo M A, Collins J J. Sublethal antibiotic treatment leads to multidrug resistance via radical-induced mutagenesis[J]. Molecular Cell, 2010, 37(3): 311-320. DOI:10.1016/j.molcel.2010.01.003 |
| [25] | Wright G D. The antibiotic resistome:the nexus of chemical and genetic diversity[J]. Nature Reviews Microbiology, 2007, 5(3): 175-186. DOI:10.1038/nrmicro1614 |
| [26] | Paiva M C, Reis M P, Costa P S, et al. Identification of new bacteria harboring qnrS and aac(6')-Ib/cr and mutations possibly involved in fluoroquinolone resistance in raw sewage and activated sludge samples from a full-scale WWTP[J]. Water Research, 2017, 110: 27-37. DOI:10.1016/j.watres.2016.11.056 |
| [27] | Aires J, Doucet-Populaire F, Butel M J. Tetracycline resistance mediated by tet(W), tet(M), and tet(O) genes of Bifidobacterium isolates from humans[J]. Applied and Environmental Microbiology, 2007, 73(8): 2751-2754. DOI:10.1128/AEM.02459-06 |
| [28] | Versluis D, D'Andrea M M, Garcia J R, et al. Mining microbial metatranscriptomes for expression of antibiotic resistance genes under natural conditions[J]. Scientific Reports, 2015, 5: 11981. DOI:10.1038/srep11981 |
| [29] | Roberts M C. Tetracycline resistance determinants:mechanisms of action, regulation of expression, genetic mobility, and distribution[J]. FEMS Microbiology Reviews, 1996, 19(1): 1-24. DOI:10.1111/j.1574-6976.1996.tb00251.x |
| [30] | Goh E B, Yim G, Tsui W, et al. Transcriptional modulation of bacterial gene expression by subinhibitory concentrations of antibiotics[J]. Proceedings of the National Academy of Sciences of the United States of America, 2002, 99(26): 17025-17030. DOI:10.1073/pnas.252607699 |
| [31] | Ramos J L, Martínez-Bueno M, Molina-Henares A J, et al. The tetR family of transcriptional repressors[J]. Microbiology and Molecular Biology Reviews, 2005, 69(2): 326-356. DOI:10.1128/MMBR.69.2.326-356.2005 |
| [32] | Depardieu F, Podglajen I, Leclercq R, et al. Modes and modulations of antibiotic resistance gene expression[J]. Clinical Microbiology Reviews, 2007, 20(1): 79-114. DOI:10.1128/CMR.00015-06 |
| [33] | Ghosh S, Sadowsky M J, Roberts M C, et al. Sphingobacterium sp. strain PM2-P1-29 harbours a functional tet(X) gene encoding for the degradation of tetracycline[J]. Journal of Applied Microbiology, 2009, 106(4): 1336-1342. DOI:10.1111/j.1365-2672.2008.04101.x |
| [34] | Huang K L, Tang J Y, Zhang X X, et al. A comprehensive insight into tetracycline resistant bacteria and antibiotic resistance genes in activated sludge using next-generation sequencing[J]. International Journal of Molecular Sciences, 2014, 15(6): 10083-10100. DOI:10.3390/ijms150610083 |
| [35] | Kaplan E, Marano R B M, Jurkevitch E, et al. Enhanced bacterial fitness under residual fluoroquinolone concentrations is associated with increased gene expression in wastewater-derived qnr plasmid-harboring strains[J]. Frontiers in Microbiology, 2018, 9: 1176. DOI:10.3389/fmicb.2018.01176 |
 2020, Vol. 41
2020, Vol. 41


