2. 林业生态工程教育部工程研究中心,北京 100083;
3. 宁夏盐池毛乌素沙地生态系统国家定位观测研究站,盐池 751500;
4. 林木资源高效生产全国重点实验室,北京 100083;
5. 水土保持国家林业和草原局重点实验室,北京 100083
2. Engineering Research Center of Forestry Ecological Engineering, Ministry of Education, Beijing 100083, China;
3. Yanchi Ecology Research Station of the Mu Us Desert, Yanchi 751500, China;
4. State Key Laboratory of Efficient Production of Forest Resources, Beijing 00083, China;
5. Key Laboratory of State Forestry and Grassland Administration on Soil and Water Conservation, Beijing 100083, China
真菌作为土壤微生物群落的重要组成部分,驱动着有机物分解、营养循环和生态系统生产力等多个生态系统过程[1, 2],并在维持生态系统功能中发挥重要作用[3]. 土壤真菌通过执行不同营养策略发挥多种生态功能[4]:共生营养型真菌能够促进有机磷矿化率,增强植物对磷的获取[5];腐生营养型真菌的存在有效促进了枯落物中难降解物质等成分的快速分解,提高土壤中碳和营养物质周转效率[6];病理营养型真菌的大量存在可能会引发植物病害,抑制植物生长[7]. 土壤真菌功能群的多样性和分布决定了土壤真菌群落的生态效应,对生态系统养分循环和植物生长都有重要意义[8].
环境选择真菌群落,真菌适应环境变化[9, 10]. 气候因子,尤其是温度,在影响土壤真菌生存和生物化学代谢活动的同时,通过影响植被生长和土壤元素的生物地球化学循环间接影响土壤真菌群落[11, 12]. 植被可释放根系分泌物,并与菌根真菌的相互作用直接影响土壤真菌群落组成. 同时,土壤真菌群落参与到地上和地下凋落物分解的食物网中,其结构和功能在物质循环过程中发生改变[13]. 土壤pH直接影响土壤真菌群落物种丰度[14],也通过生物地球化学元素循环对土壤真菌产生间接持久影响[15]. 高氮环境能够抑制外生菌根真菌的共生,进而影响真菌群落组成[16]. 因此,阐明土壤真菌群落结构与功能群分布及其影响因素对深入理解土壤真菌群落具有重要意义.
樟子松(P. sylvestris var. mongolica)是我国北方风沙区引种范围最广的防风固沙造林树种,也是典型的外生菌根真菌依赖树种[17]. 从樟子松的天然分布区到各主要引种地,经纬度差异较大,跨越不同的生物气候区,气候条件巨变. 目前,同一气候区樟子松土壤真菌群落组成和功能群已有广泛研究,但是不同气候区不同林龄樟子松的土壤真菌群落组成和功能群差异及其影响因素尚不明确. 本研究以半干旱-亚湿润干旱区不同林龄樟子松为研究对象,采用高通量测序技术,深入分析地区尺度和局域尺度上,不同气候区不同林龄樟子松林土壤真菌群落结构与功能群特征,并探究其与气候因子、土壤理化性质等环境因素的关系,并提出两个假设:①不同气候区樟子松土壤真菌群落多样性随引种时间增加而增加;②不同气候区樟子松土壤真菌功能群受气候影响呈现显著差异,随着引种时间增加,人工林土壤真菌功能群组成逐渐与天然林土壤真菌功能群趋同. 本研究结果以期为不同气候区樟子松人工林科学管理提供理论依据.
1 材料与方法 1.1 研究区概况研究区分别位于内蒙古红花尔基樟子松国家森林公园、内蒙古海拉尔国家森林公园、辽宁章古台沙地国家森林公园和陕西榆林红石峡沙地植物园. 研究区概况详见表 1.
|
|
表 1 研究区概况 Table 1 General introduction of study area |
1.2 样本采集与处理
以呼伦贝尔樟子松天然林(NF)为对照,每个研究区选择3个典型林龄:23~24 a林龄(中林龄)、33~36 a林龄(近熟林)和44~45 a林龄(成熟林)作为研究对象,共有1处天然林样地和9处人工林样地(3个沙地×3个林龄),共10个样地.
在土壤微生物活动频繁的7~8月,进行样地调查与样品采集. 每个样地随机设置5个样方(20 m×20 m). 每个样方中随机选取间距10 m以上的3棵长势均一的樟子松标准木作为采样对象. 去除地表枯落物后,收集标准木林下土(0~20 cm). 每棵标准木取3个重复,并将3个重复混合成一个样品装入塑封袋中并编号,共50份样品立刻置于-4 ℃便携式保温箱保存,并于72 h内带回实验室.
1.3 土壤真菌分子鉴定和功能群划分使用PowerSoil® DNA Kit(Mo Bio Laboratotries,美国)试剂盒对土壤真菌进行DNA提取,采用真菌通用引物ITS1F(5'-CTTGGTCATTTAGAGGAAGTAA-3')和ITS2(5'-GCTGCGTTCTTCATCGATGC-3'),对真菌rDNA ITS区段进行PCR扩增. 对PCR产物进行凝胶电泳检测,使用Illumina MiSep(Illumina,美国)进行测序. 测序结果对97%的相似水平上的OTU单元进行聚类并进行生物信息统计分析(UPARSE:highly accurate OTU sequences from microbial amplicon reads微生物扩增子读取的高精度OTU序列).
基于FUNGuild(V1.0)软件划分真菌功能群[4],对置信度为“极可能”(highly probable)和“很可能”(probable)的划分结果进行保留. 同时,对比文献研究结果,确定置信度为“可能”(possible)的划分结果的功能类型. 将具有复合营养型(tropic mode)或复合功能群(guild)的群落分别归纳为“复合营养型”.
1.4 气象数据来源与土壤理化性质测定气象数据来源于中国气象科学数据中心(https://data.cma.cn/),时间范围为2009~2018年,包括年平均日照时数(annual sunshine time,St)、年平均气温(annual temperature,Ta)和年平均降水量(annual precipitation,Pa).
采用容重法测定土壤含水量(soil water content,SWC),重铬酸钾稀释热法测定土壤有机质(soil organic matter,SOM),靛酚蓝比色法测定土壤全氮(total nitrogen,TN),钼锑抗比色法测定土壤全磷(total phosphorus,TP),火焰光度法测定土壤全钾(total potassium,TK).
1.5 数据分析采用Origin 2024、R(4.3.2)、SPSS20和Canoco 4.5软件进行数据处理和绘图. 其中,群落组成、功能群组成相对丰度堆积图和功能群热图使用Origin 2024完成,真菌群落多样性、弦图、群落组成热图、主成分分析(principal co-ordinates analysis,PCoA)、功能群与环境因子分析热图和方差分解分析(variance partitioning analysis,VPA)均使用R 4.3.2完成,单因素方差分析(one-way ANOVA)和双因素方差分析(two-way ANOVA)使用SPSS 20完成,典范对应分析(canonical correspondence analysis,CCA)使用Canoco 4.5完成.
2 结果与分析 2.1 樟子松林地土壤真菌物种多样性半干旱-亚湿润干旱区樟子松土壤真菌OTU可以分为17门、50纲、136目、295科和612属. 不同气候区不同林龄土壤真菌Chao1指数无显著性差异,天然林和人工林土壤真菌Shannon和Simpson指数呈现显著差异(P<0.05). 共生营养型和腐生营养型真菌的Shannon指数较病理营养型波动更大(图 1).
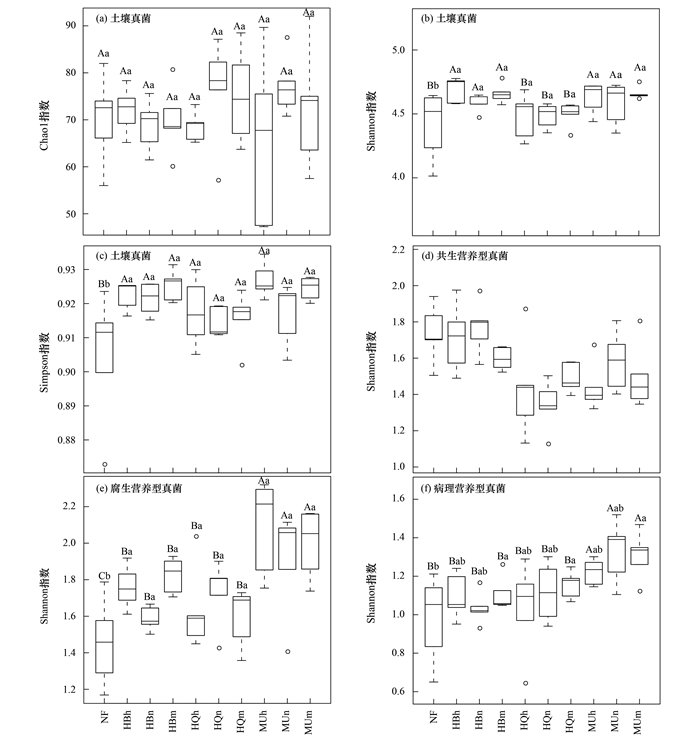
|
NF:呼伦贝尔天然林,HB:呼伦贝尔沙地;HQ:科尔沁沙地;MU:毛乌素沙地;h:中林龄,n:近熟林,m:成熟林;不同大写字母表示不同沙地土壤真菌多样性存在显著差异,不同小写字母表示不同林龄下土壤真菌多样性存在显著差异 图 1 土壤真菌α多样性指数和不同真菌功能群Shannon多样性指数 Fig. 1 Soil fungal α diversity index and Shannon diversity index of different fungal functional groups |
基于Bray-curtis距离进行属水平的主坐标分析,3个气候区不同沙地的土壤真菌群落分布在不同象限,不同沙地的土壤真菌群落距离较远,相互之间无重叠(图 2). 在物种水平上均有显著差异,而同一沙地不同林龄的土壤真菌群落结构组成相似度较高,随着林龄增长,呼伦贝尔沙地樟子松人工林土壤真菌群落与天然林土壤真菌群落重叠度增大. 毛乌素沙地不同林龄樟子松土壤真菌群落组成相似度高,而科尔沁沙地的土壤真菌群落组成相似度较低.
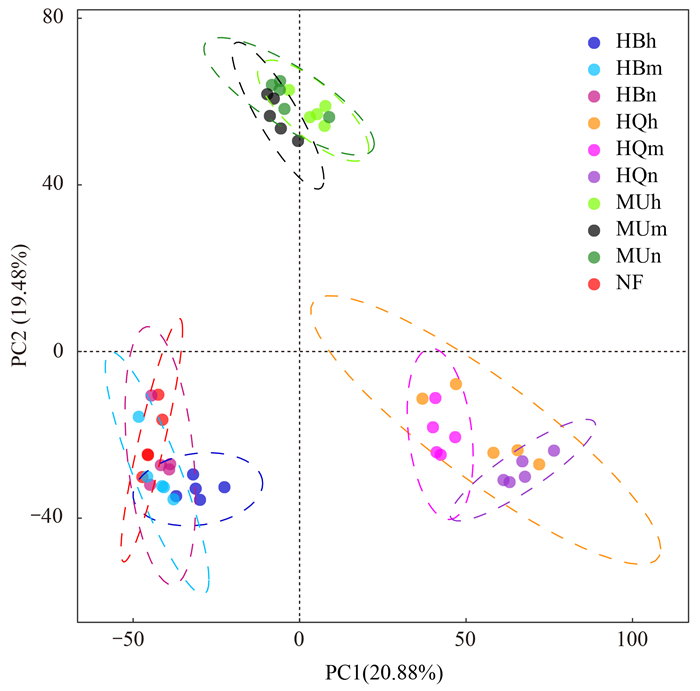
|
不同颜色的圆点表示不同样点,不同颜色虚线表示不同样地 图 2 半干旱-亚湿润干旱区樟子松土壤真菌群落β多样性 Fig. 2 The β diversity of soil fungal communities in semi-arid and dry sub-humid climates of P. sylvestris forests |
门水平上子囊菌门(Ascomycota)和担子菌门(Basidiomycota)占比最高,在所有样地中占比超过85%[图 3(a)多样性指数和不同真菌功能群Shannon多样性指数]. 属水平上青霉属(Penicillium)、美口菌属(Calostoma)和被孢霉属(Mortierella)丰度较高[图 3(b)]. 从图 3(b)可以看出,除了科尔沁中林龄和近熟林两个样地真菌占比最高的是美口菌属,其他样地都是青霉属占比最高. 青霉属在呼伦比尔沙地和毛乌素沙地的分布基本在20%左右,而在科尔沁沙地分布相对较少,仅占8%左右. 美口菌属在3个沙地中变化趋势不一,但是在各个沙地的成熟林中占比均最低(< 1%). 被孢霉属的占比在各个沙地的不同林龄土壤真菌中含量相对稳定. 乳牛杆菌属(Suillus)在各个沙地的中林龄土壤真菌中占比均高于近熟林和成熟林. 不管是门水平还是属水平上,不同气候区樟子松天然林和各林龄人工林无明显差异.
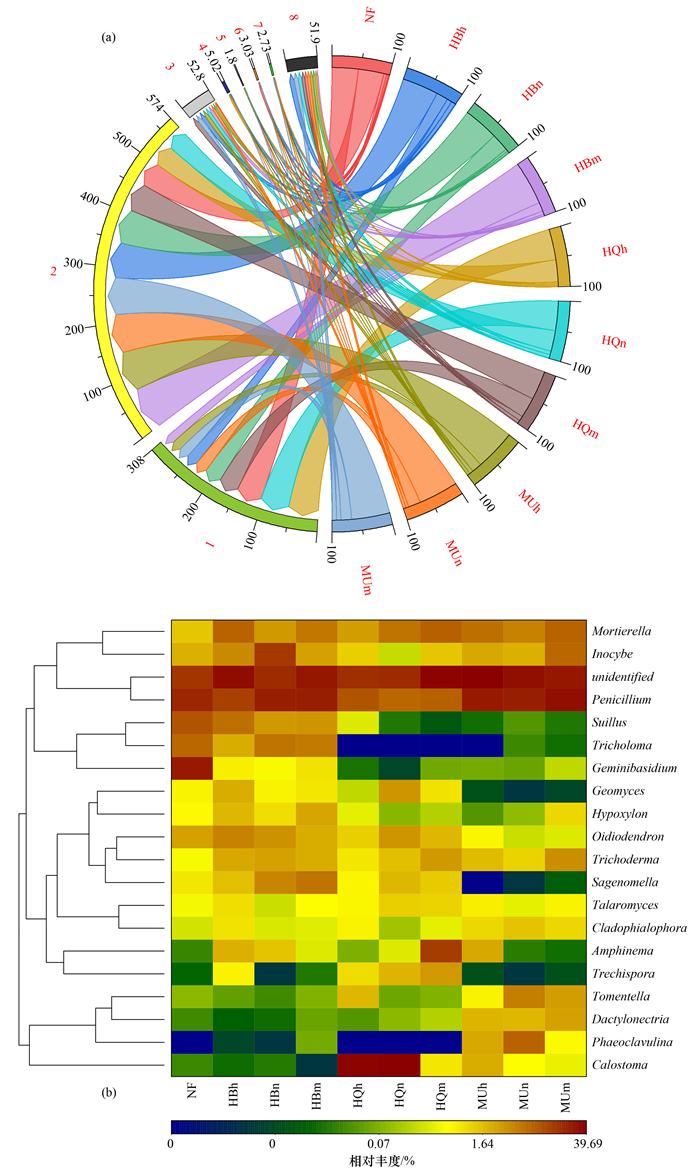
|
图中仅列出了各个样地不同林龄的樟子松土壤真菌中含量较高的属(前20);1. Basidiomycota,2.Ascomycota,3. Mortierellomycota,4. Glomeromycota,5. Rozellomycota,6. Mucoromycota,7. 其他门,8.未鉴定门 图 3 半干旱-亚湿润干旱区樟子松土壤真菌群落组成分析 Fig. 3 Soil fungal community composition of P. sylvestris forests in semi-arid and dry sub-humid climates |
用FUNGuild数据库对半干旱-亚湿润干旱区樟子松土壤真菌群落的营养类型进行比对,半干旱-亚湿润干旱区樟子松土壤真菌群落均以共生营养型(symbiotroph)、病理营养型(pathotroph)、腐生营养型(saprotroph)和复合营养型(multiple trophic mode)为主要营养类型(图 4). 这4种营养型的相对丰度均大于2%,不同营养型在不同样地占比差异较大. 天然林中腐生营养型占比最大(52.46%),呼伦贝尔沙地人工林和毛乌素沙地人工林中复合营养型占比最大(分别为37.64%~44.34%和39.98%~46.19%),而科尔沁沙地人工林中中林龄和近熟林共生营养型占比最大(56.1%~71.37%),在成熟林中复合营养型占比最大(39.75%). 病理营养型一直是占比最低的,尤其是在天然林中占比仅为2.1%. 就共生营养型真菌来看,在科尔沁沙地中,中林龄占比较大,超过70%. 随着林龄增长,共生营养型真菌占比逐渐降低. 腐生营养型在天然林中占比最高,在人工林中占比优势并不明显.
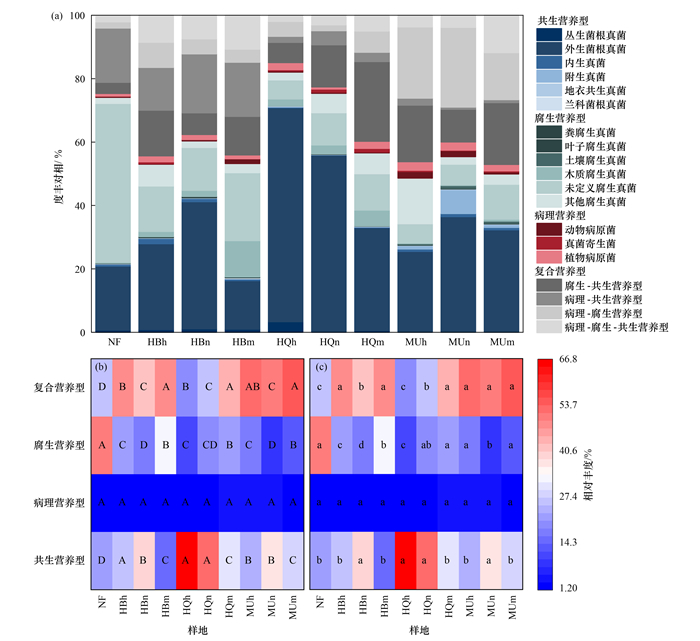
|
(a)半干旱-亚湿润干旱区樟子松土壤真菌功能群特征,(b)不同沙地同林龄功能热图,不同大写字母表示同林龄状况下不同沙地呈显著差异,(c)同一沙地不同林龄功能热图;不同小写字母表示同一沙地状况下不同林龄呈显著差异 图 4 半干旱-亚湿润干旱区樟子松土壤真菌群落功能群分析 Fig. 4 Functional groups of soil fungal communities of P. sylvestris forests in semi-arid and dry sub-humid climates |
不同气候区不同林龄樟子松土壤真菌功能群变化趋势不同. 同一沙地不同林龄下土壤真菌在共生营养型、腐生营养型的分布有着明显差异[图 4(b)],不同沙地的变化趋势也不同. 共生营养型在呼伦贝尔沙地和毛乌素沙地都是先明显增长后明显降低,在科尔沁沙地明显降低(P<0.05). 不同气候区的成熟林中共生营养型无明显差异,但与天然林有明显差异(P<0.05). 各个沙地中林龄和近熟林同林龄的3个营养型有明显差异(P<0.05),而成熟林中3个营养型无明显差异. 这说明随着林龄增长,不同气候区同一林龄的微生物真菌功能群组成(复合型营养型、腐生营养型和共生营养型)从存在显著性差异到无显著差异,但与天然林仍有显著差异(P<0.05)[图 4(c)]. 不同气候区不同林龄的樟子松土壤各个真菌功能群受到地区因素(地理位置)的极显著影响(P<0.01)(表 2),林龄对腐生真菌、共生营养真菌和复合营养真菌有显著影响(P<0.05),而对病理营养型真菌无显著影响. 不同地区不同林龄的交互作用对腐生真菌和共生营养型真菌影响显著(P<0.05).
|
|
表 2 地区和林龄对土壤真菌功能群的方差分析 Table 2 ANOVA analysis of region and stand age on functional groups of soil fungi |
2.3 樟子松林地土壤真菌群落组成与结构的影响因素
樟子松真菌群落和功能群主要影响因子存在差异,气候因子、土壤理化和植被(人工林/天然林、人工林林龄),樟子松真菌群落和功能群差异的总解释量为50.1%[图 5(c)]. 从气候区来看,CCA分析两轴总解释量为77.7%[图 5(a)],所选环境因子能够很好地表征其对土壤真菌功能群的影响. 其中,呼伦贝尔沙地土壤真菌功能群主要驱动因子为总氮,科尔沁沙地土壤真菌群主要环境驱动因子为年均温,毛乌素沙地土壤真菌功能群主要驱动因子为总磷. 而对土壤腐生营养型和病理营养型真菌有极显著影响的是土壤养分(特别是土壤全钾、土壤有机质和土壤总氮)和气候因子(年均降雨量和年均日照时数),共生营养型受环境因子的影响较少,仅年均温对其有一定的影响[图 5(b)].
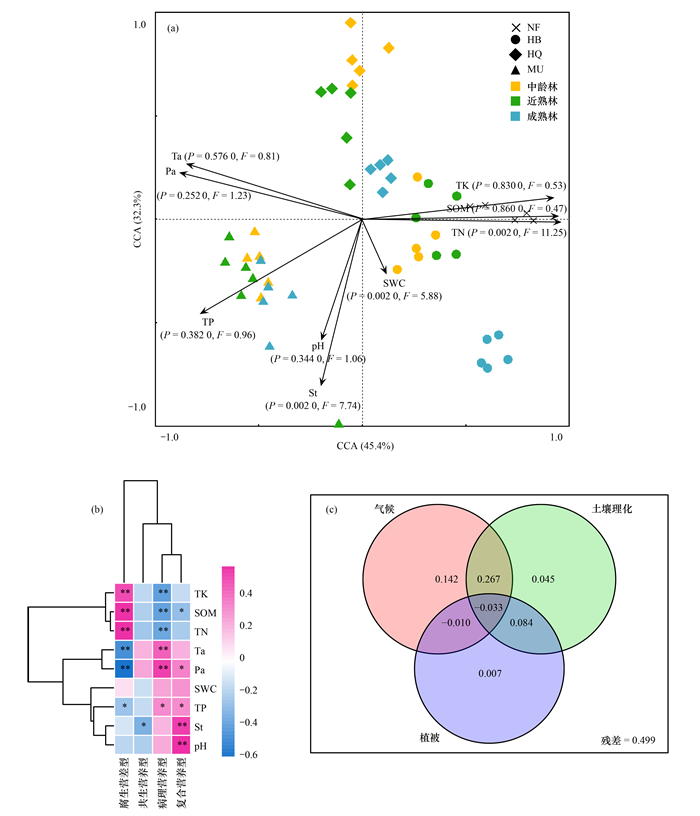
|
(a)土壤真菌群落与环境因子的典范对应分析;(b)土壤真菌功能群与环境因子的相关性分析;(c)气候因子(Ta、Pa、St)、土壤理化性质(SWC、pH、SOM、TN、TP、TK)和不同沙地不同林龄樟子松对土壤真菌功能群群落变化的方差分解分析;St:日照时数;Ta:年均温;Pa:年均降雨量;SWC:土壤含水量;SOM:土壤有机质;TP:土壤全磷;TN:土壤总氮;TK:土壤全钾;不同形状表示不同样地,不同颜色表示同一样地不同林龄,向量表示与环境变量显著相关的方向和大小. *:P<0.05,**:P<0.01. 图 5 半干旱-亚湿润干旱区樟子松土壤真菌群落组成与环境因子的相关性分析 Fig. 5 Correlation analysis between soil fungi and environmental factors of P. sylvestris forests in semi-arid and dry sub-humid climates |
不同气候区不同林龄樟子松土壤真菌群落α多样性指数无显著差异,但β多样性差异显著. 这是由于土壤真菌群落α多样性更多地受土壤性质和植物类型等因素的影响[18],其中土壤pH是土壤真菌α多样性最重要的影响因素[19]. 在本研究中3个沙地土壤pH在6.68~7.42之间,无显著性差异,对土壤共生营养型、腐生营养型和病理营养真菌也无显著影响. 植被类型对土壤真菌α多样性影响显著,尤其是对植被类型有依赖关系的共生营养型真菌[20]. 而土壤真菌β多样性则受环境差异和地理距离影响[21]. 本研究中不同气候区3个沙地植被均为樟子松且地理距离差异较大. 因此不同气候区樟子松林土壤真菌群落β多样性呈现明显差异性,同一沙地不同林龄则出现高度相似性.
不同气候区樟子松林土壤真菌群落的优势菌门为子囊菌门和担子菌门. 大多数子囊菌门具有富含黑色素的细胞壁,可以抵抗沙漠环境中的多种非生物辐射胁迫[22, 23]. 同时,子囊菌门和担子菌门是土壤真菌主要活跃的分解者,能够相互配合并促进樟子松林有机质的分解和养分循环[11, 24]. 在属水平上,植物病原菌青霉属比例较高,可能跟取样时间有关系. 有研究表明,在樟子松生长季中,土壤真菌种青霉属明显增长[25]. 美口菌属与松科植物形成外生菌根真菌,属共生营养型[26],能够促进引种后的樟子松定植,在成熟林含量最低,可能是与樟子松衰退有一定关系[27]. 被孢霉属作为腐生真菌,参与土壤养分转化和促进植物生长,可能对沙漠地区的植被恢复产生积极影响[28, 29]. 乳牛肝菌属是松科专性的外生菌根真菌,能够与树木形成共生关系,在引种早期对樟子松定植有重要作用[30],随着林龄增长,外生菌根真菌群落发生变化,乳牛肝菌属含量降低[17].
真菌功能群划分结果表明,腐生营养型在天然林中最占优势,共生营养在人工林中相对占优势. 樟子松是典型的外生菌根依赖性树种[31],而外生菌根真菌多为共生营养型和复合营养型土壤真菌,能够协助植物完成对土壤中氮磷钾等养分的获取,提高植株生长速度,缓解干旱胁迫[32],在引种物种适应不利的土壤环境过程中起到重要作用[33]. 而在天然林中,腐生营养型真菌占比更高,可能是因为它可以分解枯落物和动物粪便等有机质,促进碳磷元素循环进而影响贫瘠地区的生物地球化学循环和植物生长[34]. 同一沙地的不同林龄下土壤真菌在共生营养型、腐生营养型的分布有着明显差异. 这反映真菌对地上植被变化的适应,樟子松引种以后改变了草本层和枯落物层,进而对土壤真菌产生直接影响. 这也与之前的研究一致,土壤真菌的群落组成会受到特定树种改变的影响[35]. 随着林龄增长,共生营养型降低,腐生营养型逐渐增多. 这可能是由于共生营养型尤其是外生菌根真菌,如乳牛肝菌属(Suillus spp.)具有高度的宿主特异性[31],群落随生存环境和寄主生长周期变化[11]. 而腐生真菌能够借助其腐养能力在低碳营养环境中获得优势[36]. 随着林龄增长,不同气候区微生物真菌功能群组成从显著性差异到无显著差异,但与天然林仍有显著差异. 这可能是由于树种是造林后土壤真菌群落组成的主要调节因子[13]. 在引种树种都是樟子松的情况下,随着林龄增长,土壤真菌受到树种调节,群落组成从之前的异质性逐渐趋同.
3.2 环境因子对土壤真菌群落组成与结构的影响不同气候区土壤真菌群落功能群主要驱动因子不同. 科尔沁沙地土壤真菌群落主要环境驱动因子为日照时数,这可能是科尔沁沙地土壤菌根真菌占比远高于其他两个沙地的原因,同时,菌根真菌生长与植物生长密切相关[37],进而体现在真菌群落功能群的环境驱动因子是年均日照时数. 而呼伦贝尔沙地和毛乌素沙地的土壤真菌功能群的主要驱动因子分别是总氮和总磷. 这可能是由于呼伦贝尔沙地处于半干旱气候区,水热条件较其他两个沙地更好,根系生长代谢和凋落物输入的有机碳、氮分解更为充分,这也体现在土壤腐生真菌(尤其是近熟林和成熟林)占比相对更高上[38]. 而毛乌素沙地土壤总磷含量明显高于其他两个地区,这可能是跟成土母质有关,也有可能是沙地水热条件较差,影响土壤的快速固定、矿化和风化,限制磷素流失,使土壤全磷含量较高.
在区域尺度上,气候因子(如温度和降水等)与真菌群落组成有密切关系[39];在局域尺度范围内,同一个沙地外部气候条件相对一致,植被类型和土壤性质塑造着土壤真菌的物种多样性和群落组成[10]. 这与本文研究结论一致,日照时数、土壤含水量和总氮这3个环境因子是对不同气候区下樟子松林土壤功能群有极显著影响. 年均日照时数作为太阳辐射的表征指标,直接影响土壤温度和植物生长等与土壤真菌代谢密切相关的因素[40]. 同时,本研究区在荒漠生态系统,土壤水分和养分匮乏,这也成为影响不同气候区樟子松土壤真菌功能群分布的极显著因素. 低含水量会影响限制土壤溶液中养分的迁移速率与化学反应过程,会抑制微生物群落功能和活性的表达[41]. 氮元素的增加可能会强烈影响外生菌根的定殖,进而影响土壤真菌群落组成[42].
土壤真菌群落对环境异质性的反应更为积极,尤其是腐生真菌[38]. 腐生菌群落是参与凋落物降解最丰富的真菌群,并随演替呈上升趋势 [43]. 腐生真菌的生长高度依赖于可获得的营养物质的可用性和基质的质量[44]. 腐生真菌的活动尤其是参与分解的酶活性受日照时长影响较大. 年均降雨量有效改变了土壤含水量,进而改变了腐生真菌参与枯落物分解的效率[45]. 但对共生营养型真菌来说,宿主物种之间的偏好可能是外生菌根真菌本地谱系多样化的重要进化驱动因素[46],因此在土壤共生营养型真菌以外生菌根真菌为主的樟子松林中,环境因子对土壤共生营养型真菌影响极少.
4 结论(1)不同气候区不同林龄樟子松土壤真菌群落α多样性指数无显著差异,但β多样性差异显著.
(2)在土壤真菌群落组成上,不同气候区樟子松天然林和各林龄人工林的菌门和菌属无明显差异. 不同气候区土壤腐生营养型、共生营养型和复合型营养型真菌在中林龄和近熟林有显著性差异(P<0.05),在成熟林无显著差异(P > 0.05),但与天然林仍有显著差异(P<0.05).
(3)土壤真菌功能群在不同尺度上受到的影响因素不同. 半干旱-亚湿润干旱区樟子松土壤真菌群落和功能群的主要驱动因子是日照时长、土壤含水量和总氮. 而不同气候区樟子松土壤真菌群落功能群的主要驱动因子不同. 对土壤腐生营养型和病理营养型真菌有极显著影响的是土壤养分(特别是土壤全钾、土壤有机质和土壤总氮)和气候因子(年均降雨量和年均日照时数),共生营养型受环境因子的影响较少,仅年均温对其有一定的影响
| [1] | Hug L A, Thomas B C, Sharon I, et al. Critical biogeochemical functions in the subsurface are associated with bacteria from new phyla and little studied lineages[J]. Environmental Microbiology, 2016, 18(1): 159-173. DOI:10.1111/1462-2920.12930 |
| [2] | Ai C, Zhang S Q, Zhang X, et al. Distinct responses of soil bacterial and fungal communities to changes in fertilization regime and crop rotation[J]. Geoderma, 2018, 319: 156-166. DOI:10.1016/j.geoderma.2018.01.010 |
| [3] | Liu D, Wang H L, An S S, et al. Geographic distance and soil microbial biomass carbon drive biogeographical distribution of fungal communities in Chinese Loess Plateau soils[J]. Science of the Total Environment, 2019, 660: 1058-1069. DOI:10.1016/j.scitotenv.2019.01.097 |
| [4] | Nguyen N H, Song Z W, Bates S T, et al. FUNGuild: an open annotation tool for parsing fungal community datasets by ecological guild[J]. Fungal Ecology, 2016, 20: 241-248. DOI:10.1016/j.funeco.2015.06.006 |
| [5] | Cao M A, Liu R C, Xiao Z Y, et al. Symbiotic fungi alter the acquisition of phosphorus in Camellia oleifera through regulating root architecture, plant phosphate transporter gene expressions and soil phosphatase activities[J]. Journal of Fungi, 2022, 8(8). DOI:10.3390/jof8080800 |
| [6] | Moore J C, Berlow E L, Coleman D C, et al. Detritus, trophic dynamics and biodiversity[J]. Ecology Letters, 2004, 7(7): 584-600. DOI:10.1111/j.1461-0248.2004.00606.x |
| [7] | Yuan M M, Kakouridis A, Starr E, et al. Fungal-bacterial cooccurrence patterns differ between arbuscular mycorrhizal fungi and nonmycorrhizal fungi across soil niches[J]. mBio, 2021, 12(2). DOI:10.1128/MBIO.03509-20 |
| [8] | Detheridge A P, Comont D, Callaghan T M, et al. Vegetation and edaphic factors influence rapid establishment of distinct fungal communities on former coal-spoil sites[J]. Fungal Ecology, 2018, 33: 92-103. DOI:10.1016/j.funeco.2018.02.002 |
| [9] | Crowther T W, van den Hoogen J, Wan J, et al. The global soil community and its influence on biogeochemistry[J]. Science, 2019, 365(6455). DOI:10.1126/science.aav0550 |
| [10] | Zeng Q C, Jia P L, Wang Y, et al. The local environment regulates biogeographic patterns of soil fungal communities on the Loess Plateau[J]. CATENA, 2019, 183. DOI:10.1016/j.catena.2019.104220 |
| [11] | Priyashantha A K H, Dai D Q, Bhat D J, et al. Plant–fungi interactions: where it goes?[J]. Biology, 2023, 12(6). DOI:10.3390/biology12060809 |
| [12] | Soudzilovskaia N A, Douma J C, Akhmetzhanova A A, et al. Global patterns of plant root colonization intensity by mycorrhizal fungi explained by climate and soil chemistry[J]. Global Ecology and Biogeography, 2015, 24(3): 371-382. DOI:10.1111/geb.12272 |
| [13] | Liu J L, Le T H, Zhu H N, et al. Afforestation of cropland fundamentally alters the soil fungal community[J]. Plant and Soil, 2020, 457(1): 279-292. |
| [14] | Jiang Y J, Liang Y T, Li C M, et al. Crop rotations alter bacterial and fungal diversity in paddy soils across East Asia[J]. Soil Biology and Biochemistry, 2016, 95: 250-261. DOI:10.1016/j.soilbio.2016.01.007 |
| [15] | Sridhar B, Lawrence G B, Debenport S J, et al. Watershed-scale liming reveals the short- and long-term effects of pH on the forest soil microbiome and carbon cycling[J]. Environmental Microbiology, 2022, 24(12): 6184-6199. DOI:10.1111/1462-2920.16119 |
| [16] | Corrales A, Turner B L, Tedersoo L, et al. Nitrogen addition alters ectomycorrhizal fungal communities and soil enzyme activities in a tropical montane forest[J]. Fungal Ecology, 2017, 27: 14-23. DOI:10.1016/j.funeco.2017.02.004 |
| [17] | Ning C, Mueller G M, Egerton-Warburton L M, et al. Host phylogenetic relatedness and soil nutrients shape ectomycorrhizal community composition in native and exotic pine plantations[J]. Forests, 2019, 10(3). DOI:10.3390/f10030263 |
| [18] | Geml J, Arnold A E, Semenova-Nelsen T A, et al. Community dynamics of soil-borne fungal communities along elevation gradients in neotropical and palaeotropical forests[J]. Molecular Ecology, 2022, 31(7): 2044-2060. DOI:10.1111/mec.16368 |
| [19] | Wang J T, Zheng Y M, Hu H W, et al. Soil pH determines the alpha diversity but not beta diversity of soil fungal community along altitude in a typical Tibetan forest ecosystem[J]. Journal of Soils and Sediments, 2015, 15(5): 1224-1232. DOI:10.1007/s11368-015-1070-1 |
| [20] | Hendershot J N, Read Q D, Henning J A, et al. Consistently inconsistent drivers of microbial diversity and abundance at macroecological scales[J]. Ecology, 2017, 98(7): 1757-1763. DOI:10.1002/ecy.1829 |
| [21] | Egidi E, Delgado-Baquerizo M, Plett J M, et al. A few Ascomycota taxa dominate soil fungal communities worldwide[J]. Nature Communacation, 2019, 10(1). DOI:10.1038/s41467-019-10373-z |
| [22] | Geng X Z, Zuo J C, Meng Y H, et al. Changes in nitrogen and phosphorus availability driven by secondary succession in temperate forests shape soil fungal communities and function[J]. Ecology and Evolution, 2023, 13(10). DOI:10.1002/ece3.10593 |
| [23] | Zhou H, Gao Y, Jia X H, et al. Network analysis reveals the strengthening of microbial interaction in biological soil crust development in the Mu Us Sandy Land, northwestern China[J]. Soil Biology and Biochemistry, 2020, 144. DOI:10.1016/j.soilbio.2020.107782 |
| [24] | Lundell T K, Mäkelä M R, Hildén K. Lignin-modifying enzymes in filamentous basidiomycetes-ecological, functional and phylogenetic review[J]. Journal of Basic Microbiology, 2010, 50(1): 5-20. DOI:10.1002/jobm.200900338 |
| [25] |
任悦, 高广磊, 丁国栋, 等. 毛乌素沙地樟子松人工林根内真菌群落结构与功能时间动态特征[J]. 环境科学, 2024, 45(7): 4241-4250. Ren Y, Gao G L, Ding G D, et al. Temporal variations in community structure and function of root-associated fungi associated with Pinus sylvestris var. mongholica plantations in the Mu Us sandy land[J]. Environmental Science, 2024, 45(7): 4241-4250. |
| [26] | Wilson A W, Hobbie E A, Hibbett D S. The ectomycorrhizal status of Calostoma cinnabarinum determined using isotopic, molecular, and morphological methods[J]. Canadian Journal of Botany, 2007, 85(4): 385-393. DOI:10.1139/B07-026 |
| [27] | Guo M S, Gao G L, Ding G D, et al. Drivers of ectomycorrhizal fungal community structure associated with Pinus sylvestris var. mongolica differ at regional vs. local spatial scales in northern China[J]. Forests, 2020, 11(3). DOI:10.3390/F11030323 |
| [28] | Zhang Y, Cao H Y, Zhao P S, et al. Vegetation restoration alters fungal community composition and functional groups in a desert ecosystem[J]. Frontiers in Environmental Science, 2021, 9. DOI:10.3389/fenvs.2021.589068 |
| [29] | Zhang H S, Wu X H, Li G, et al. Interactions between arbuscular mycorrhizal fungi and phosphate-solubilizing fungus (Mortierella sp.) and their effects on Kostelelzkya virginica growth and enzyme activities of rhizosphere and bulk soils at different salinities[J]. Biology and Fertility of Soils, 2011, 47(5): 543-554. DOI:10.1007/s00374-011-0563-3 |
| [30] | Kretzer A, Li Y N, Szaro T, et al. Internal transcribed spacer sequences from 38 recognized species of Suillus sensu lato: phylogenetic and taxonomic implications[J]. Mycologia, 1996, 88(5): 776-785. DOI:10.1080/00275514.1996.12026715 |
| [31] | Bâ A M, Diédhiou A G, Prin Y, et al. Management of ectomycorrhizal symbionts associated to useful exotic tree species to improve reforestation performances in tropical Africa[J]. Annals of Forest Science, 2010, 67(3). DOI:10.1051/forest/2009108 |
| [32] | Shi Z Y, Mickan B, Feng G, et al. Arbuscular mycorrhizal fungi improved plant growth and nutrient acquisition of desert ephemeral Plantago minuta under variable soil water conditions[J]. Journal of Arid Land, 2015, 7(3): 414-420. DOI:10.1007/s40333-014-0046-0 |
| [33] |
李丹丹, 李佳文, 高广磊, 等. 科尔沁沙地樟子松(Pinus sylvestris var. mongolia)人工林土壤真菌群落结构和功能特征[J]. 中国沙漠, 2023, 43(4): 241-251. Li D D, Li J W, Gao G L, et al. Soil fungal community structure and functional characteristics associated with Pinus sylvestris var. mongolica plantations in the Horqin sandy land[J]. Journal of Desert Research, 2023, 43(4): 241-251. |
| [34] | Li Y H, Han X, Li B, et al. Soil addition improves multifunctionality of degraded grasslands through increasing fungal richness and network complexity[J]. Geoderma, 2023, 437. DOI:10.1016/j.geoderma.2023.116607 |
| [35] | Asplund J, Kauserud H, Ohlson M, et al. Spruce and beech as local determinants of forest fungal community structure in litter, humus and mineral soil[J]. FEMS Microbiology Ecology, 2019, 95(2). DOI:10.1093/femsec/fiy232 |
| [36] | Guo Z M, Zhang X Y, Dungait J A J, et al. Contribution of soil microbial necromass to SOC stocks during vegetation recovery in a subtropical karst ecosystem[J]. Science of the Total Environment, 2021, 761. DOI:10.1016/j.scitotenv.2020.143945 |
| [37] | Bennett J A, Maherali H, Reinhart K O, et al. Plant-soil feedbacks and mycorrhizal type influence temperate forest population dynamics[J]. Science, 2017, 355(6321): 181-184. DOI:10.1126/science.aai8212 |
| [38] | Kang P, Pan Y Q, Ran Y C, et al. Soil saprophytic fungi could be used as an important ecological indicator for land management in desert steppe[J]. Ecological Indicators, 2023, 150. DOI:10.1016/j.ecolind.2023.110224 |
| [39] | Dunthorn M, Kauserud M, Bass D, et al. Yeasts dominate soil fungal communities in three lowland Neotropical rainforests[J]. Environmental Microbiology Reports, 2017, 9(5): 668-675. DOI:10.1111/1758-2229.12575 |
| [40] | Zhang H, Yin Q, Nakajima T, et al. Influence of changes in solar radiation on changes of surface temperature in China[J]. Acta Meteorologica Sinica, 2013, 27(1): 87-97. DOI:10.1007/s13351-013-0109-8 |
| [41] | Gong X Q, Jarvie S, Zhang Q, et al. Community assembly of plant, soil bacteria, and fungi vary during the restoration of an ecosystem threatened by desertification[J]. Journal of Soils and Sediments, 2023, 23(1): 459-472. DOI:10.1007/s11368-022-03329-2 |
| [42] | Suz L M, Barsoum N, Benham S, et al. Environmental drivers of ectomycorrhizal communities in Europe's temperate oak forests[J]. Molecular Ecology, 2014, 23(22): 5628-5644. DOI:10.1111/mec.12947 |
| [43] | Meng W J, Li Y L, Qu Z L, et al. Fungal community structure shifts in litter degradation along forest succession induced by pine wilt disease[J]. Microbiological Research, 2024, 280: 127588. DOI:10.1016/j.micres.2023.127588 |
| [44] |
李德会, 韩周林, 吴庆贵, 等. 影响森林细根分解的腐生真菌功能特性研究综述[J]. 世界林业研究, 2021, 34(3): 25-31. Li D H, Han Z L, Wu Q G, et al. Review of functional characteristics of saprophytic fungi affecting the decomposition of fine roots in forest ecosystem[J]. World Forestry Research, 2021, 34(3): 25-31. |
| [45] | Castaño C, Bonet J A, Oliva J, et al. Rainfall homogenizes while fruiting increases diversity of spore deposition in Mediterranean conditions[J]. Fungal Ecology, 2019, 41: 279-288. DOI:10.1016/j.funeco.2019.07.007 |
| [46] | van Galen L G, Orlovich D A, Lord J M, et al. Correlated evolution in an ectomycorrhizal host–symbiont system[J]. New Phytologist, 2023, 238(3): 1215-1229. DOI:10.1111/nph.18802 |
 2025, Vol. 46
2025, Vol. 46


