2. 中国科学院大学, 北京 100049;
3. 江西省红壤及种质资源研究所, 南昌 330000
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. Institute of Red Soil and Germplasm Resources, Nanchang 330000, China
红壤性水稻土是我国南方重要的农田土壤, 在保障粮食安全方面发挥着至关重要的作用. 近年来, 由于片面追求高产而采用过量施用化肥和高强度种植等传统管理模式, 导致了土壤酸化[1]、肥力水平下降和微生物多样性的降低[2], 进而制约了土壤肥力与作物产量的可持续发展[3]. 有机物料还田已被证明是改善土壤肥力的一项有效措施[4, 5]. 水稻秸秆、紫云英、畜禽粪便是南方稻田最主要的有机肥源[6, 7], 在冬绿肥还田的基础上, 合理配置秸秆和畜禽粪便等有机肥源是土壤地力和水稻产量协同提升的关键[8, 9]. 但不同有机物料配置对水稻产量的长期效应(> 30 a)及其微生物作用机制仍有待研究.
在植物-土壤-微生物系统中, 微生物(特别是细菌)是驱动土壤养分循环从而促进植物生长的关键因子[10]. 有研究表明, 在农田生态系统中, 有机物料还田不仅影响了微生物群落组成[11], 而且可以协同提升土壤养分循环和作物产量[12, 13]. 首先, 有机物料为细菌提供了新鲜碳源和氮磷等养分, 不仅促进了细菌群落的生长和代谢活性[14], 并且显著改变了细菌群落的组成, 特别是增加了富营养型细菌[15], 如革兰氏阴性菌的丰度[16]. 细菌的群落组成与作物产量息息相关[17], 前期研究表明, 有机物料还田处理提高了革兰氏阴性菌与革兰氏阳性菌的比例, 增加土壤酶活性和促进土壤养分循环, 提高了水稻产量[7].
近年来, 随着扩增子测序功能预测和宏基因组功能注释等研究方法的发展, 学术界开展了有机物料还田影响细菌群落功能的研究[18]. 例如, 酸性农业土壤中有机肥的施用调节了氮循环相关功能基因的丰度, 提高了微生物对氮的吸收和转化能力[19]. 但也有研究表明, 秸秆还田降低了稻田土壤中氮循环的相关基因[20]. 因此, 不同有机物料还田对土壤微生物功能的影响仍存在争论. 此外, 碳、氮等养分转化功能基因的丰度是影响作物产量的关键[21]. 最新研究表明, 长期施用有机物料(包括牛粪、麦秸和绿肥)可同时提高了土壤有机碳的矿化、氮的固定和磷转运的能力, 从而提高了作物产量[22]. 然而, 鲜见有研究探讨微生物群落中哪些种群的变化影响了碳、氮转化功能基因的丰度.
尽管大量研究已经证实有机物料还田改变了土壤微生物群落的组成和功能, 并提高了作物产量, 但长期不同有机物料还田下, 土壤微生物群落中种群的变化如何通过影响碳氮转化的功能基因, 进而调控水稻产量的机制仍不完全清楚. 基于以上研究背景与现状, 本研究针对南方典型红壤性水稻土, 依托红壤性水稻土有机肥长期试验平台, 围绕有机物料还田对细菌群落和功能以及水稻产量的影响机制, 采用基于高通量测序的细菌群落组成分析和功能预测, 重点研究:①不同有机物料还田下, 水稻产量、土壤性质、细菌群落组成和功能基因相对丰度;②不同有机物料还田下, 细菌群落组成通过功能基因影响水稻产量的机制. 本研究结果可为红壤性水稻土的持续管理和产量提升提供科学指导.
1 材料与方法 1.1 试验地概况依托江西省红壤及种质资源研究所设置在进贤县的红壤性水稻土有机肥定位试验站(28°37′N, 116°26′E). 该研究区域位于典型的亚热带季风气候区, 其年平均气温和年降水量分别为18.1℃和1 727 mm. 试验地所用土壤为第四纪红粘土发育的红壤性水稻土, 属于潴育性水稻土类型. 本试验自1981年起持续至今, 采用早稻-早稻-紫云英(Astragalus sinicus L.)的轮作模式:早稻与晚稻的种植周期为每年4月中旬至10月中旬, 10月中旬至次年3月种植紫云英作为冬季覆盖作物. 试验小区规格为60 m2, 每个处理3次重复, 并按照随机区组排列. 本研究选择其中5种施肥处理:①不施肥(CK)、②施化肥(NPK)、③化肥+紫云英(NG)、④化肥+紫云英+猪粪(NGM)和⑤化肥+紫云英+稻草(NGS). 肥料施用量见表 1.
|
|
表 1 红壤性水稻土有机肥定位试验不同处理施肥量/kg·hm-2 Table 1 Fertilizer application rate of different treatments/kg·hm-2 |
1.2 土壤样品采集与理化性质测定
2021年晚稻成熟期(插秧后90d), 按S型取样法从每小区表层(0~20 cm)采集9个土样, 并混合均匀. 采集的新鲜土样立即置于冰上运回实验室. 取回的土壤样品分为3份:一份冷冻于-80℃, 用于微生物分析;一份冷藏于4℃, 用于测定土壤速效养分和可溶性碳氮;余下样本风干后用于全量土壤养分和pH分析. 土壤pH采用玻璃电极法(土水比1∶2.5)测定[23]. 土壤有机碳(SOC)通过重铬酸钾氧化法测定. 土壤中的可利用磷(AP)与总磷(TP)采用钼蓝法测量[23]. 硝态氮(NO3-)与铵态氮(NH4+)用2 mol·L-1 KCl提取后, 通过连续流动分析仪测定[23]. 土壤可溶性有机碳(DOC)和氮(DON)的提取及测定:将5 g土样与25 mL蒸馏水(比例1∶5)混合, 振荡8 h(180 r·min-1), 离心(2 800 r·min-1, 10 min), 过滤(0.45 µm滤膜), 并用元素分析仪测定[24]. 水稻产量测定:晚稻成熟期进行人工收割, 自然风干, 人工脱粒并称取籽粒干重, 计算每个试验小区水稻产量.
1.3 土壤DNA提取与高通量测序称取0.5 g土壤新鲜样品, 按MagPure Soil DNA KF Kit操作指南提取DNA, 提取后的DNA用NanoDrop 2000和琼脂糖凝胶电泳检测浓度与纯度. 以提取的DNA为模板, 采用细菌通用引物(343F与798R)[25]和Takara Ex Taq高保真酶进行16S rRNA基因的PCR扩增. PCR产物经纯化、量化后, 利用Illumina NovaSeq 6000平台测序. 测序获得的原始数据后, 首先使用Cutadapt软件, 将raw data序列剪切掉引物序列. 然后使用DADA2[26]将上一步合格的双端raw data按照QIIME 2默认参数进行质量过滤, 降噪, 拼接及去嵌合体等质控分析, 最终得到43 208个OTU. 功能预测采用PICRUSt 2软件进行分析, 利用QIIME[27]获得的closed OTU-table与KEGG(Kyoto Encyclopedia of Genes and Genomes)数据库进行比对, 获得不同的数据库功能预测信息.
1.4 数据分析采用SPSS 24.0软件进行方差分析(ANOVA), 检验多组数据间差异, 差异显著性水平设定为P < 0.05, 并采用Tukey多重比较方法检验不同处理组间显著性差异. 采用R语言(3.6.3版本)中的“vegan”程序包, 计算微生物群落α多样性指数和基于Bray-Curtis距离的主坐标分析(PCoA). 利用R语言(3.6.3版本)中“randomForest”程序包及其“rfUtilities”扩展包构建随机森林模型, 并借助“vegan”包实施置换多元方差分析(PERMANOVA). 功能基因差异分析在Stamp软件中通过Welch's t-test计算. 在Smartpls软件中构建偏最小二乘法(PLS)路径模型, 并用拟合优度评估模型.
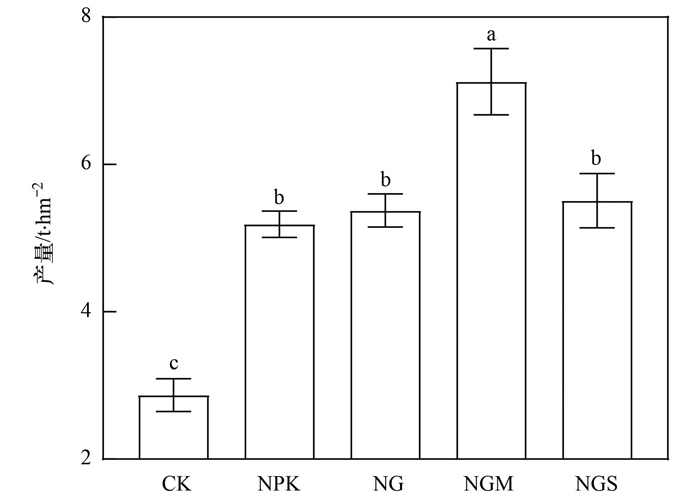
2 结果与分析 2.1 有机物料还田对土壤化学性质与水稻产量的影响不同有机物料还田均显著影响了土壤化学性质(P < 0.05, 表 2). 其中, NGM处理中SOC含量最高, 是CK处理的1.67倍, 其次为NGS和NG处理, 分别为CK处理的1.51倍和1.40倍, 而NPK处理与CK处理没有显著差异. 土壤TN和TP含量在各处理中的变化趋势与有机碳相似, 即:NGM > NGS、NG > NPK、CK(P < 0.05). NGM处理的AP、NO3-、NH4+和DOC含量显著高于NG和NGS处理(P < 0.05). DON的含量在NG中最高, 分别是NGS和NGM处理的1.14倍和1.35倍. 施用有机肥和化肥均显著提高了水稻产量, 其中NGM处理的水稻产量最高(P < 0.05, 图 1), 分别是NG和NGS处理的1.33倍和1.29倍, 而NG和NGS处理与NPK处理没有显著差异.
|
|
表 2 不同施肥处理对土壤化学性质的影响1) Table 2 Soil chemical properties under different treatments |
|
不同小写字母表示不同处理的差异显著(P < 0.05) 图 1 不同施肥处理水稻产量 Fig. 1 Grain yield of rice of different treatments |
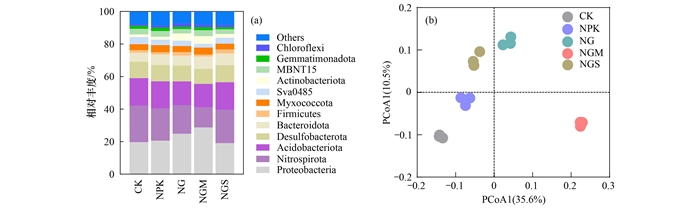
与CK相比, NPK处理显著增加了土壤细菌群落的α多样性指数(P < 0.05). 有机物料还田处理中只有NGM处理显著提高了Shannon指数, 而其他处理的α多样性指数与CK处理没有显著差异(表 3). 在门水平上分析细菌群落组成, 其中优势微生物为:变形菌门(Proteobacteria, 22.57%, 平均丰度, 下同)、硝化螺旋菌门(Nitrospirota, 18.56%)和酸杆菌门(Acidobacteriota, 15.87%), 而脱硫杆菌门(Desulfobacterota, 9.90%)、拟杆菌门(Bacteroidota, 6.60%)和厚壁菌门(Firmicutes, 1.80%)的相对丰度较低[图 2(a)]. 有机物料还田显著增加了Proteobacteria、放线菌(Actinobacteriota)和Bacteroidota的相对丰度, 与CK(5.83%)相比, NG(10.08%)、NGM(10.16%)和NGS(6.75%)处理显著增加了α-Proteobacteria的相对丰度(P < 0.05), 但降低了Nitrospirota的相对丰度, 其中NGM处理的影响最大. NGS处理(16.94%)的Acidobacteriota丰度高于NG(14.62%)和NGM处理(14.37%). PCoA分析也表明, 有机物料还田显著影响细菌群落组成(r2 = 0.63, P < 0.01), 特别是NGM处理, 与其他处理存在明显差异[图 2(b)].
|
|
表 3 细菌群落多样性指数 Table 3 The α diversity indexes of the bacterial communities |
|
图 2 不同施肥处理对门水平土壤细菌群落组成和结构影响 Fig. 2 Effects of different treatments on the relative abundance of the major soil bacteria and the community structure at the phylum level |
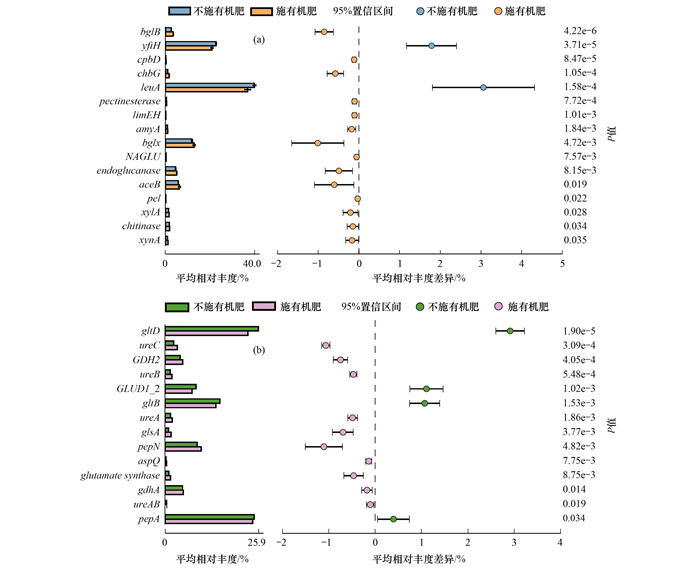
基于KEGG数据库预测的结果表明, 针对有机碳降解功能基因, 有机物料还田显著提升了编码淀粉酶(amyA)、纤维素酶(bglX、bglB、endoglucanase)、半纤维素酶(xylA、xynA)、几丁质酶(NAGLU、cpbD、chbG、chitinase)和果胶酶(pectinesterase、pel)基因的相对丰度(P < 0.05). 而芳香族化合物(leuA)和木质素(yfiH)分解相关基因的相对丰度在CK和NPK处理中较高[图 3(a), P < 0.05]. 针对有机氮矿化功能基因, 施用有机物料提高了脲酶(ureA、ureB、ureC)、谷氨酸脱氢酶(gdhA)、谷氨酰胺酶(aspQ)和氨基肽酶N(pepN)基因的丰度(P < 0.05).
|
(a)有机碳降解功能基因;(b)有机氮矿化功能基因 图 3 不同施肥处理对有机碳降解和有机氮矿化的功能基因的影响 Fig. 3 Functional genes related to C-decomposition and N-mineralization in the different treatments |
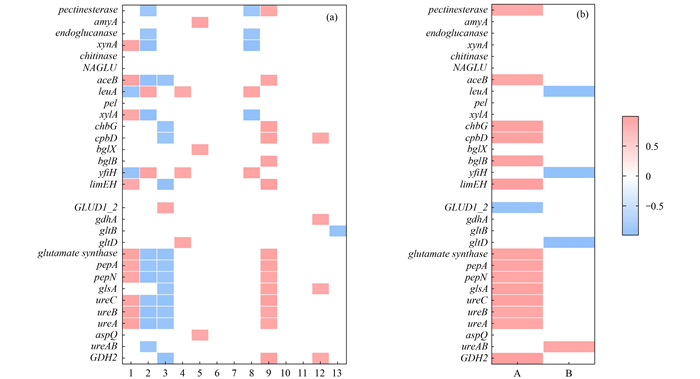
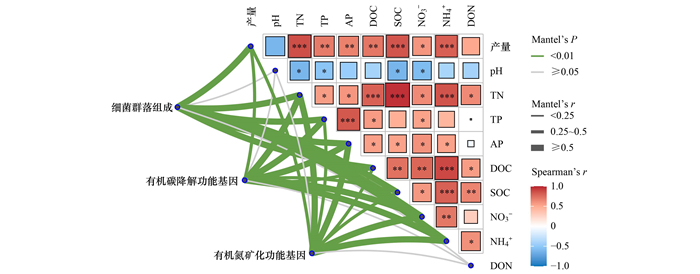
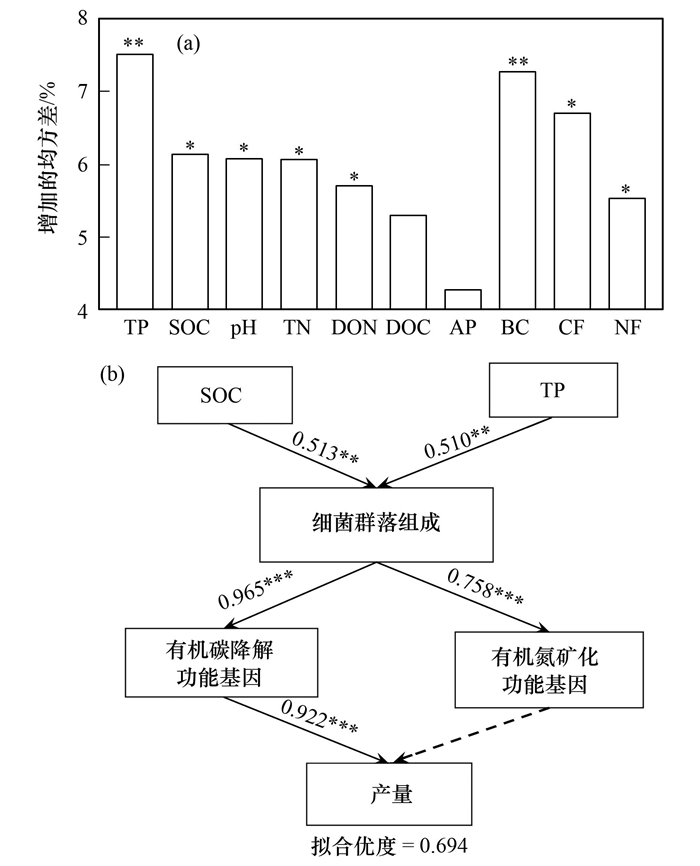
相关性分析表明, Proteobacteria和Actinobacteriota门以及α-Proteobacteria纲与有机碳降解与有机氮矿化功能基因显著正相关性(图 4), 并且细菌群落组成及碳、氮矿化功能基因相对丰度与土壤AP、NH4+、NO3-、TP、DOC、TN和SOC等理化性质以及水稻产量显著相关(图 5). 采用随机森林分析影响水稻产量潜在因子的重要性, 结果显示, TP(7.52%)、SOC(6.14%)、pH(6.09%)、TN(6.08%)、DON(5.72%)、细菌群落组成(7.27%)、有机碳降解功能基因(6.70%)以及有机氮矿化功能基因(5.54%)均对水稻产量的变异有显著影响(图 6, P < 0.05). 基于此, 构建土壤理化性质(TP和SOC)、细菌群落组成、有机碳降解与有机氮矿化功能基因及水稻产量的偏最小二乘路径模型(图 5). 模型结果显示, SOC(路径系数=0.513, P < 0.01)和TP(路径系数=0.510, P < 0.01)直接影响细菌群落组成. 细菌群落组成(路径系数=0.965, P < 0.001)通过调控有机碳降解相关功能基因显著影响水稻产量(路径系数=0.922, P < 0.001).
|
1.Proteobacteria, 2.Nitrospirota, 3.Acidobacteriota, 4.Desulfobacterota, 5.Bacteroidota, 6.Firmicutes, 7.Myxococcota, 8.Sva0485, 9.Actinobacteriota, 10.MBNT, 11.Gemmatimonadota, 12.Chloroflexi, 13.Others; A.α-Proteobacteria, B. γ-Proteobacteria 图 4 细菌门/纲与功能基因的相关性 Fig. 4 Correlation of bacterial phylum and class abundance with functional genes |
|
*表示不同的显著性水平, *表示P < 0.05, **表示P < 0.01, ***表示P < 0.001 图 5 细菌群落组成和功能与土壤理化性质以及水稻产量之间的关系 Fig. 5 Relationship between bacterial community composition and function with soil physicochemical properties and rice yield |
|
BC:细菌群落组成, CF:有机碳降解功能基因, NF:有机氮矿化功能基因;箭头旁边的数字表示路径系数, 线条粗细表示相关性大小, 实线表示显著相关, 虚线表示不显著;*表示P < 0.05, **表示P < 0.01, ***表示P < 0.001 图 6 土壤化学性质与细菌群落及功能对水稻产量影响的随机森林模型和偏最小二乘路径模型 Fig. 6 Effects of soil chemical properties, bacterial community and functions on rice yield based on random forest model and PLS path model |
有研究表明有机物料在还田后可以提高土壤有机养分库容, 并通过微生物作用下养分的周转, 持久和平衡地提供养分, 促进水稻增产[28, 29]. 本研究中, 与NPK相比, 有机物料还田(NG、NGM和NGS)增加了土壤养分含量和产量, 这与前人的研究结果一致[30]. 并且, NGM处理的土壤养分含量和水稻产量显著高于NG和NGS处理. 这是由于, 与植物残体相比, 畜禽粪便碳氮比低, 且腐植化程度高, 因此更容易形成土壤腐植质并续存在土壤中[31]. 全球范围内的荟萃分析结果也证实, 施用粪肥对SOC和TN含量增加的比例为27%和33%, 高于施用秸秆(26%和28%)和绿肥(7.3%和29%)[32, 33].
3.2 有机物料还田对土壤细菌群落结构和功能的影响有机物料还田对细菌α多样性的影响尚无定论, 有研究发现与单施化肥相比, 有机物料投入可以增加细菌多样性和丰富度[8 ~ 34]. 但也有荟萃分析表明, 有机肥投入对细菌α多样性没有显著影响[35];这与本研究的结果一致. 大量研究证明有机物料还田显著改变了细菌群落结构[15 ~ 36]. 本研究中, 有机物料还田, 特别是NGM处理, 显著增加了Proteobacteria, 特别是α-Proteobacteria以及放线菌的相对丰度. α-Proteobacteria和放线菌属于快速生长(r-型)的富营养型细菌[37];因此, NGM处理通过提高土壤易分解碳源和养分, 可能促进了这些富营养型细菌的增长[35 ~ 38]. 同时, 有机物料还田后, 以碳为能源的微生物得到增长, 加剧了对氮资源的竞争, 进而抑制了如Nitrospirota等慢速生长(K-型)的寡营养型微生物的增长[39]. 酸杆菌门被认为是寡营养型微生物, 有降解贫乏和难分解碳底物的潜能[40], 因此本研究中, 秸秆的施用(NGS处理)可能由于提高了难降解的木质素含量, 从而增加了酸杆菌门的丰度.
有机物料还田也影响了有机碳分解和氮矿化相关功能基因的相对丰度. 在有机碳分解的功能基因中, 有机物料还田提高了编码降解半纤维素、纤维素等易分解碳的功能基因的相对丰度, 但降低了编码木质素等难分解碳的功能基因的相对丰度, 这与Li等[41]和Asiloglu等[42]的研究结果一致. 这可能与有机物料的还田提高了易分解碳含量, 促进了偏好利用易分解碳的α-Proteobacteria和放线菌等富营养型微生物的生长相关. 本研究中α-Proteobacteria和放线菌与降解易分解碳的各功能基因丰度成显著正相关(图 4). 前期宏基因组功能注释的研究表明, 编码易分解碳(如碳水化合物、半纤维素等)降解的功能基因在放线菌和α-Proteobacteria中均有较高表达量[43, 44]. 同时, 氮矿化的功能基因也与α-Proteobacteria和放线菌显著正相关, 这可能是由于这两种细菌在利用碳源的同时, 也促进了有机氮的矿化, 为自身生长提供氮源.
3.3 有机物料还田与土壤理化特性、微生物群落、功能基因和产量的关系有研究发现施用有机物料可以改变土壤理化性质, 从而有利于微生物生长和功能基因表达[45]. 前期研究表明, 稻田土壤中磷和SOC含量是塑造细菌群落结构差异的决定因子[46];这与本文的研究结果一致. 并且如前文讨论中所述, 本研究连续施用有机物料通过增加富营养型细菌(α-Proteobacteria和放线菌)的相对丰度, 从而增强了易分解碳降解和有机氮矿化的功能基因的丰度. 并且, 碳、氮转化相关功能基因丰度的增加, 通过促进养分周转, 增加了土壤中速效养分, 特别是NH4+、NO3-和DON的含量(图 5), 从而提高了水稻产量. 与Li等[47]的研究结果一致, 该研究发现连续施用有机物料显著提高了参与碳循环和氮循环的功能基因的丰度, 并平均提高了13.2%的年粮食产量和38.0%的土壤有机碳含量, 但化学肥料的施用对菌群及功能酶基因丰度的影响较小. 综上所述, 本研究通过长期定位试验研究表明, 施用有机物料提供新鲜易分解碳源, 促进了富营养型细菌的生长, 增强了易分解碳和有机氮矿化基因的表达, 从而提高了水稻产量.
4 结论有机物料还田显著增加了红壤性水稻土SOC、TN、TP、和AP等养分含量, 其中以处理(NGM)对土壤肥力和水稻产量的提升效果最好. 不同有机物料还田处理显著改变了土壤细菌群落结构, 特别是增加了α-Proteobacteria和放线菌等富营养型细菌的相对丰度, 从而增强了与碳氮矿化相关的功能基因的丰度, 进一步影响水稻产量.
| [1] | Cai Z J, Wang B R, Xu M G, et al. Intensified soil acidification from chemical N fertilization and prevention by manure in an 18-year field experiment in the red soil of southern China[J]. Journal of Soils and Sediments, 2015, 15(2): 260-270. DOI:10.1007/s11368-014-0989-y |
| [2] |
袁红朝, 秦红灵, 刘守龙, 等. 长期施肥对红壤性水稻土细菌群落结构和数量的影响[J]. 中国农业科学, 2011, 44(22): 4610-4617. Yuan H Z, Qin H L, Liu S L, et al. Response of abundance and composition of the bacterial community to long-term fertilization in paddy soils[J]. Scientia Agricultura Sinica, 2011, 44(22): 4610-4617. |
| [3] | Zhu Q C, Liu X J, Hao T X, et al. Modeling soil acidification in typical Chinese cropping systems[J]. Science of the Total Environment, 2018, 613-614: 1339-1348. DOI:10.1016/j.scitotenv.2017.06.257 |
| [4] | Dai X L, Song D L, Zhou W, et al. Partial substitution of chemical nitrogen with organic nitrogen improves rice yield, soil biochemical indictors and microbial composition in a double rice cropping system in South China[J]. Soil and Tillage Research, 2021, 205. DOI:10.1016/j.still.2020.104753 |
| [5] | Wang H, He X, Zhang Z F, et al. Eight years of manure fertilization favor copiotrophic traits in paddy soil microbiomes[J]. European Journal of Soil Biology, 2021, 106. DOI:10.1016/j.ejsobi.2021.103352 |
| [6] | Dai X L, Zhou W, Liu G R, et al. Soil C/N and ph together as a comprehensive indicator for evaluating the effects of organic substitution management in subtropical paddy fields after application of high-quality amendments[J]. Geoderma, 2019, 337: 1116-1125. DOI:10.1016/j.geoderma.2018.11.023 |
| [7] | Zhou G P, Gao S J, Lu Y H, et al. Co-incorporation of green manure and rice straw improves rice production, soil chemical, biochemical and microbiological properties in a typical paddy field in Southern China[J]. Soil and Tillage Research, 2020, 197. DOI:10.1016/j.still.2019.104499 |
| [8] | Luo G W, Li L, Friman V P, et al. Organic amendments increase crop yields by improving microbe-mediated soil functioning of agroecosystems: a meta-analysis[J]. Soil Biology and Biochemistry, 2018, 124: 105-115. DOI:10.1016/j.soilbio.2018.06.002 |
| [9] | Yang L, Zhou X, Liao Y L, et al. Co-incorporation of rice straw and green manure benefits rice yield and nutrient uptake[J]. Crop Science, 2019, 59(2): 749-759. DOI:10.2135/cropsci2018.07.0427 |
| [10] | Durrer A, Gumiere T, Zagatto M R G, et al. Organic farming practices change the soil bacteria community, improving soil quality and maize crop yields[J]. Peerj, 2021, 9. DOI:10.7717/peerj.11985 |
| [11] | Zhao S C, Li K J, Zhou W, et al. Changes in soil microbial community, enzyme activities and organic matter fractions under long-term straw return in north-central China[J]. Agriculture, 2016, 216: 82-88. |
| [12] |
孔培君, 郑洁, 栾璐, 等. 不同秸秆还田方式对旱地红壤细菌群落、有机碳矿化及玉米产量的影响[J]. 环境科学, 2021, 42(12): 6047-6057. Kong P J, Zheng J, Luan L, et al. Effects of different types of straw returning on the bacterial community, organic carbon mineralization and maize yield in upland red soil[J]. Environmental Science, 2021, 42(12): 6047-6057. |
| [13] | Zhu J, Peng H, Ji X H, et al. Effects of reduced inorganic fertilization and rice straw recovery on soil enzyme activities and bacterial community in double-rice paddy soils[J]. European Journal of Soil Biology, 2019, 94. DOI:10.1016/j.ejsobi.2019.103116 |
| [14] | Li F, Chen L, Zhang J B, et al. Bacterial community structure after long-term organic and inorganic fertilization reveals important associations between soil nutrients and specific taxa involved in nutrient transformations[J]. Frontiers in Microbiology, 2017, 8. DOI:10.3389/fmicb.2017.00187 |
| [15] | Bian Q, Wang X Y, Bao X G, et al. Exogenous substrate quality determines the dominant keystone taxa linked to carbon mineralization: evidence from a 30-year experiment[J]. Soil Biology and Biochemistry, 2022, 169. DOI:10.1016/j.soilbio.2022.108683 |
| [16] | Wang X Y, Bian Q, Jiang Y J, et al. Organic amendments drive shifts in microbial community structure and keystone taxa which increase C mineralization across aggregate size classes[J]. Soil Biology and Biochemistry, 2021, 153. DOI:10.1016/j.soilbio.2020.108062 |
| [17] |
林婷婷, 郑洁, 朱国繁, 等. 有机肥处理对旱地红壤细菌群落及玉米生产力的影响[J]. 环境科学, 2023, 44(12): 6965-6972. Lin T T, Zheng J, Zhu G F, et al. Effects of organic fertilization on bacterial community and maize productivity in dryland red soil[J]. Environmental Science, 2023, 44(12): 6965-6972. |
| [18] | Zhang Y J, Gao W, Ma L, et al. Long-term partial substitution of chemical fertilizer by organic amendments influences soil microbial functional diversity of phosphorus cycling and improves phosphorus availability in greenhouse vegetable production[J]. Agriculture, 2023, 341. DOI:10.1016/j.agee.2022.108193 |
| [19] | Hu X J, Gu H D, Liu J J, et al. Metagenomics reveals divergent functional profiles of soil carbon and nitrogen cycling under long-term addition of chemical and organic fertilizers in the black soil region[J]. Geoderma, 2022, 418. DOI:10.1016/j.geoderma.2022.115846 |
| [20] | Lin J T, Xu Z Y, Xue Y H, et al. N2O emissions from soils under short-term straw return in a wheat-corn rotation system are associated with changes in the abundance of functional microbes[J]. Agriculture, 2023, 341. DOI:10.1016/j.agee.2022.108217 |
| [21] | Wu X J, Peng J J, Liu P F, et al. Metagenomic insights into nitrogen and phosphorus cycling at the soil aggregate scale driven by organic material amendments[J]. Science of the Total Environment, 2021, 785. DOI:10.1016/j.scitotenv.2021.147329 |
| [22] | Zhou G P, Fan K K, Gao S J, et al. Green manuring relocates microbiomes in driving the soil functionality of nitrogen cycling to obtain preferable grain yields in thirty years[J]. Science China Life Sciences, 2024, 67(3): 596-610. DOI:10.1007/s11427-023-2432-9 |
| [23] | 鲁如坤. 土壤农业化学分析方法[M]. 北京: 中国农业科技出版社, 2000. |
| [24] | Li X M, Sun G X, Chen S C, et al. Molecular chemodiversity of dissolved organic matter in paddy soils[J]. Environmental Science & Technology, 2018, 52(3): 963-971. |
| [25] | Nossa C W, Oberdorf W E, Yang L Y, et al. Design of 16S rRNA gene primers for 454 pyrosequencing of the human foregut microbiome[J]. World Journal of Gastroenterology, 2010, 16(33): 4135-4144. DOI:10.3748/wjg.v16.i33.4135 |
| [26] | Callahan B J, McMurdie P J, Rosen M J, et al. DADA2: high-resolution sample inference from illumina amplicon data[J]. Nature Methods, 2016, 13(7): 581-583. DOI:10.1038/nmeth.3869 |
| [27] | Bolyen E, Rideout J R, Dillon M R, et al. Reproducible, interactive, scalable and extensible microbiome data science using QIIME 2[J]. Nature Biotechnology, 2019, 37(8): 852-857. DOI:10.1038/s41587-019-0209-9 |
| [28] | Song W F, Shu A P, Liu J A, et al. Effects of long-term fertilization with different substitution ratios of organic fertilizer on paddy soil[J]. Pedosphere, 2022, 32(4): 637-648. DOI:10.1016/S1002-0160(21)60047-4 |
| [29] |
颜慧, 钟文辉, 李忠佩, 等. 长期施肥对红壤水稻土磷脂脂肪酸特性和酶活性的影响[J]. 应用生态学报, 2008, 19(1): 71-75. Yan H, Zhong W H, Li Z P, et al. Effects of long-term fertilization on phospholipid fatty acids and enzyme activities in paddy red soil[J]. Chinese Journal of Applied Ecology, 2008, 19(1): 71-75. |
| [30] | Liu H Y, Xu W, Li J Y, et al. Short-term effect of manure and straw application on bacterial and fungal community compositions and abundances in an acidic paddy soil[J]. Journal of Soils and Sediments, 2021, 21(9): 3057-3071. DOI:10.1007/s11368-021-03005-x |
| [31] | Ye G P, Lin Y X, Liu D Y, et al. Long-term application of manure over plant residues mitigates acidification, builds soil organic carbon and shifts prokaryotic diversity in acidic ultisols[J]. Applied Soil Ecology, 2019, 133: 24-33. DOI:10.1016/j.apsoil.2018.09.008 |
| [32] | Liu S B, Wang J Y, Pu S Y, et al. Impact of manure on soil biochemical properties: a global synthesis[J]. Science of the Total Environment, 2020, 745. DOI:10.1016/j.scitotenv.2020.141003 |
| [33] | Ma D K, Yin L N, Ju W L, et al. Meta-analysis of green manure effects on soil properties and crop yield in northern China[J]. Field Crops Research, 2021, 266. DOI:10.1016/j.fcr.2021.108146 |
| [34] | Luo J P, Liao G C, Banerjee S, et al. Long-term organic fertilization promotes the resilience of soil multifunctionality driven by bacterial communities[J]. Soil Biology and Biochemistry, 2023, 177. DOI:10.1016/j.soilbio.2022.108922 |
| [35] | Shu X Y, Liu W J, Huang H, et al. Meta-analysis of organic fertilization effects on soil bacterial diversity and community composition in agroecosystems[J]. Plants, 2023, 12(22). DOI:10.3390/plants12223801 |
| [36] | Li W T, Liu M, Wu M, et al. Bacterial community succession in paddy soil depending on rice fertilization[J]. Applied Soil Ecology, 2019, 144: 92-97. DOI:10.1016/j.apsoil.2019.07.014 |
| [37] | Fierer N, Bradford M A, Jackson R B. Toward an ecological classification of soil bacteria[J]. Ecology, 2007, 88(6): 1354-1364. DOI:10.1890/05-1839 |
| [38] | Francioli D, Schulz E, Lentendu G, et al. Mineral vs. Organic amendments: microbial community structure, activity and abundance of agriculturally relevant microbes are driven by long-term fertilization strategies[J]. Frontiers in Microbiology, 2016, 7. DOI:10.3389/fmicb.2016.01446 |
| [39] | Han S, Huang Q Y, Chen W L. Partitioning nitrospira community structure and co-occurrence patterns in a long-term inorganic and organic fertilization soil[J]. Journal of Soils and Sediments, 2021, 21(2): 1099-1108. DOI:10.1007/s11368-020-02813-x |
| [40] | Fierer N. Embracing the unknown: disentangling the complexities of the soil microbiome[J]. Nature Reviews Microbiology, 2017, 15(10): 579-590. DOI:10.1038/nrmicro.2017.87 |
| [41] | Li H, Zhang Y Y, Yang S, et al. Variations in soil bacterial taxonomic profiles and putative functions in response to straw incorporation combined with N fertilization during the maize growing season[J]. Agriculture, 2019, 283. DOI:10.1016/j.agee.2019.106578 |
| [42] | Asiloglu R, Sevilir B, Samuel S O, et al. Effect of protists on rhizobacterial community composition and rice plant growth in a biochar amended soil[J]. Biology and Fertility of Soils, 2021, 57(2): 293-304. DOI:10.1007/s00374-020-01525-1 |
| [43] | Bao Y Y, Dolfing J, Guo Z Y, et al. Important ecophysiological roles of non-dominant Actinobacteria in plant residue decomposition, especially in less fertile soils[J]. Microbiome, 2021, 9(1). DOI:10.1186/s40168-021-01032-x |
| [44] | Wang X Y, Liang C, Mao J D, et al. Microbial keystone taxa drive succession of plant residue chemistry[J]. The ISME Journal, 2023, 17(5): 748-757. DOI:10.1038/s41396-023-01384-2 |
| [45] | Tang S, Ma Q X, Marsden K A, et al. Microbial community succession in soil is mainly driven by carbon and nitrogen contents rather than phosphorus and sulphur contents[J]. Soil Biology and Biochemistry, 2023, 180. DOI:10.1016/j.soilbio.2023.109019 |
| [46] | Su J Q, Ding L J, Xue K, et al. Long-term balanced fertilization increases the soil microbial functional diversity in a phosphorus-limited paddy soil[J]. Molecular Ecology, 2015, 24(1): 136-150. DOI:10.1111/mec.13010 |
| [47] | Li J, Yang Y, Wen J L, et al. Continuous manure application strengthens the associations between soil microbial function and crop production: evidence from a 7-year multisite field experiment on the Guanzhong Plain[J]. Agriculture, 2022, 338. DOI:10.1016/j.agee.2022.108082 |
 2025, Vol. 46
2025, Vol. 46


