2. 东北农业大学资源与环境学院, 哈尔滨 150036
2. College of Resources and Environment, Northeast Agricultural University, Harbin 150036, China
随着抗生素在人类和动物疾病治疗中的广泛应用, 其带来的抗生素耐药基因(antibiotic resistance genes, ARGs)威胁成为了全球关注的焦点[1, 2].目前, 联合国环境规划署已将ARGs污染归类为世界六大新兴环境问题之一[3, 4].ARGs通过赋予宿主细菌耐药性而使抗生素无效, 降低疾病治疗的疗效[5].有研究表明, ARGs在环境中的繁殖和传播会加剧环境细菌和人类病原体之间有害细菌的共享和交换, 导致人类耐药性病原体数量逐年增加[6].英国政府报告指出, 若不对环境中的ARGs加以控制, 2050年全球将有1 000万人死于抗生素耐药性病原体引起的疾病[7], 这凸显了ARGs污染的严重性[4].全球抗生素使用量中约有2/3来自畜禽养殖业, 其中高达90%的抗生素可能通过粪便排出, 因此畜禽粪污被认为是ARGs的重要储存库[8]. 而畜禽粪污作为肥料被广泛施用于农田, 导致ARGs在农田土壤中富集[9], 土壤中的ARGs可能会通过细菌的定殖和迁移等途径向土壤-植物系统中扩散[10]. 植物微生物可以增加环境微生物与人体微生物的相互作用, 这被认为是环境ARGs进入人体的重要途径[11].而蔬菜作为一种食用类植物, 其可食用部分积累的ARB和ARGs可通过食物链进入人体, 对人类健康的构成了严重威胁[12].因此, 了解ARGs在土壤-蔬菜系统中的赋存特征、迁移规律以及影响因素对于推进畜禽粪污的资源化、无害化利用, 实现农业绿色循环可持续发展至关重要.
本文在简要论述土壤-蔬菜系统中ARGs来源及危害的基础上, 聚焦土壤-蔬菜系统中ARGs的环境行为, 梳理总结了土壤-蔬菜系统中ARGs的赋存特征、迁移规律及关键影响因素, 并提出适宜于土壤-蔬菜系统中ARGs污染防控的建议及展望, 以期为我国的农业绿色发展提供理论支撑.
1 土壤-蔬菜系统中ARGs的来源与危害 1.1 土壤-蔬菜系统中ARGs的主要来源 1.1.1 固体畜禽粪肥土壤-植物系统中ARGs的来源多样, 其中畜禽粪便的资源化还田利用被认为是促进农业生态系统中ARGs增殖和传播的主要来源之一(图 1).据报道, 我国畜禽养殖业每年约产生38亿t畜禽粪污, 畜禽粪便存在大量抗生素和ARGs残留, 已经被认为是农业环境中重要的ARGs储存池[8].有研究发现, 家庭畜禽粪便中存在多种常见ARGs以及高风险ARGs(blaampC、blaOXA-1和blaTEM-1), 鸡场的ARGs水平高于猪场和牛场[13].然而, 以上粪污作为有机肥料还田提高土壤肥力会增加土壤中ARGs的水平(增加至106~108 copies·g-1)[11]. Mu等[4]针对猪粪还田的研究发现, 粪肥的施用促进了ARGs在土壤底层的富集, 增加了1~2个数量级.值得注意的是, 即便是施用生态友好型粪肥(牛粪源蚯蚓粪), 仍然存在增加土壤中ARGs水平的情况[14].此外, 施用畜禽粪肥也能显著增加蔬菜(根和叶)中的ARGs丰度(表 1). Fan等[15]针对施用畜禽粪便的沙拉类蔬菜的调查结果显示, ARGs潜在病原菌在沙拉蔬菜的叶片中的相对丰度显著高于土壤中, 存在更高的风险.由此可见, 畜禽粪肥的施用会将大量畜禽源ARGs引入农田生态系统, 导致ARGs进一步在土壤-蔬菜系统中传播.
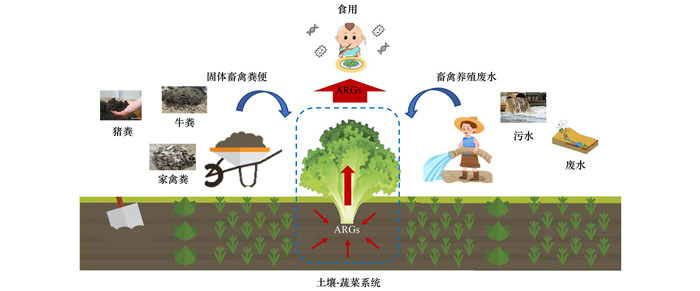
|
图 1 土壤-蔬菜系统中ARGs的来源及危害 Fig. 1 Sources and harm of ARGs in soil-vegetable systems |
|
|
表 1 土壤-蔬菜系统中不同来源的ARGs丰度 Table 1 ARGs abundance from different sources in soil-vegetable systems |
1.1.2 畜禽养殖废水
畜禽废水主要是以畜禽养殖过程中产生的污水、沼液为主.将畜禽养殖废水经处理后(再生水)进行灌溉是缓解农业缺水和干旱的有效解决方案.然而经处理后的再生水中仍存在大量抗生素、ARB以及ARGs的残留.Yang等[16]的研究发现, 1类整合子(intI1)、2类整合子(intI2)和共轭质粒(traA)在猪场废水的各个处理单元中普遍存在, 高达101~107 copies·mL-1, 而ARGs丰度达103~108 copies·mL-1. 再生水的灌溉会将以上残留的抗生素、ARB以及ARGs等污染物带入土壤环境中, 进而促进抗生素耐药性在土壤-蔬菜系统中发展和传播[17, 18]. Zhang等[19]收集了猪场废水灌溉的栽培作物(白菜、生菜、玉米)和野生植物(稗草、海棠、狗尾草), 发现所有栽培作物和野生植物的根、叶和豆中普遍存在ARGs.另外, 在土壤-蔬菜系统中长期利用再生水灌溉后, 与ARGs密切相关的变形杆菌(Proteus)、拟杆菌(Bacteroides)和不动杆菌(Acinetobacter)等相对丰度也发生了显著变化[20].由此可见, 再生水的灌溉会通过引入抗生素以及微生物群落进而影响农业生态系统中的抗生素耐药性, 对土壤原有的耐药性施加了强大的选择压力, 成为了土壤-植物系统中抗生素耐药性传播的重要来源之一.
1.2 土壤-蔬菜系统中ARGs的潜在风险与危害土壤中的ARGs会随土壤微生物在根系的定殖转移到植物细菌群落(包括内生菌和根茎层细菌), 以上ARGs以及ARB会随着食物链进入人体, 这对人类健康构成潜在威胁(图 1).有研究报道, 受耐药菌污染的叶菜中可分离出会引起人类肠道发热和肠胃炎的韦氏沙门氏菌和乙型副伤寒沙门氏菌[25];而食用被肺炎克雷伯菌等耐多药病原体污染的生蔬菜会导致免疫功能低下的人患急性支气管肺炎和分娩肺炎, 并通过水平基因转移增加与肠道中的常驻微生物共享ARGs(如ESBL/AmpC基因)的风险[26].由此可见, 食用受耐药菌污染的生食蔬果可能会通过直接引入ARGs或ARB定殖人体的方式造成人体肠道疾病甚至引发其他器官病变.另外, 定殖在人体内的ARGs或ARB也会通过使抗生素失效从而降低疾病治疗效果的方式对人类构成威胁.例如, 在2003年的“非典”事件中, 由于SARS病毒对大环内酯类、氟喹诺酮类、β-内酰胺类和四环素类等抗生素的抗性, 最终导致全球919人死亡, 超过8 442人感染, 涉及32个国家和地区[27].因此, 在合理进行畜禽废弃物资源化利用, 实现农业绿色可持续发展的同时, 重点关注畜禽源ARGs在土壤-蔬菜系统中的环境行为及影响因素, 对降低人类健康风险具有重要意义.
2 土壤-蔬菜系统中ARGs的污染特征与行为归趋 2.1 ARGs的赋存分布特征土壤-蔬菜系统被认为是ARGs从环境向人类传播的重要媒介.迄今为止, 土壤与蔬菜中已检出大量不同种类的ARGs, 如氨基糖苷类(aadA、aacC4等)、β-内酰胺类(AmpC、OXA-1、IMP-1等)、大环内酯类(MLSB)(ermB、ermF、ermX等)、多药类(ttgA、mexA等)和四环素类(tetM、tetC、tetPA等)等ARGs[28, 29].在土壤-蔬菜系统中, 土壤的ARGs水平最高, 以氨基糖苷类(aminoglycoside)和多药类(multidrug)ARGs为主, 其次是大环内酯类ARGs[macrolide-lincosamide-streptogramin B resistance(MLSB)]和四环素类ARGs(tetracycline), 丰度范围在105~109 copies·g-1.与非根际土壤相比, 根际土壤约高出1~2个数量级(表 2), 具有更大的ARGs风险.
|
|
表 2 不同土壤-蔬菜系统中ARGs丰度水平1) Table 2 Abundance of ARGs in soil-vegetable systems |
此外, 动物粪便和再生水的还田利用还对植物体中ARGs的多样性与丰度产生影响[30], 有研究表明, 施用粪肥种植的有机蔬菜中ARGs丰度高达普通蔬菜的8倍[28];同时粪肥施加亦可显著提高水稻叶部aadA1、tnpA、blaSHV和qacE-delta1等ARGs的丰度(P < 0.05)[31].与土壤中分布规律不同的是, 四环素类ARGs(tetracycline)和多药类ARGs(multidrug)在植物体内中丰度最高, 氨基糖苷类ARGs(aminoglycoside)和大环内酯类ARGs[macrolide-lincosamide-streptogramin B resistance(MLSB)]的污染水平次之.另外, 蔬菜不同部位中ARGs的累积水平也存在差异, 呈现出茎部 > 根部 > 叶片的整体分布模式.具体来说, 叶菜类蔬菜可食用部分(叶片)的ARGs丰度约为105~107 copies·g-1, 而在非可食用部分中丰度约为106~109 copies·g-1, 茎部的ARGs丰度较根部高出1~2个数量级(表 2).土壤-蔬菜系统中存在的畜禽源ARGs可能会伴随食物链向人体迁移, 对环境和人类造成双重威胁, 加剧ARGs的环境风险.因此, 本研究进一步对土壤-蔬菜系统中ARGs的迁移行为与机制进行了分析.
2.2 土壤-蔬菜系统中ARGs的迁移行为与扩散机制 2.2.1 ARGs的迁移传播行为抗生素耐药性在土壤中的传播与土壤微生物群落的演替有关, 土壤细菌群落显著影响ARB和ARGs的流行和传播[35, 36]. Sun等[37]的实验表明, 施用粪肥会在土壤环境中引入粪肥微生物组分, 增加土壤中的ARGs丰度.而土壤ARGs可经内生菌的土壤-根系跨界传输或通过根表面气孔和机械损伤处进入植物内生系统, 极大威胁农产品质量和安全[38]. Xu等[39]直观地追踪了ARGs向植物内生菌的传播, 发现土壤细菌, 特别是变形杆菌可以从大肠杆菌中捕获RP4质粒, 并促进其向植物内生菌的传播. 在植物体内, 许多携带ARGs的植物细菌群落通常来自附着在植物根部汲取渗出营养物质的土壤致病菌[40]. Zhang等[19]通过研究ARGs在土壤-植物系统的转移发现, 小白菜和谷仓草叶层中ARGs主要来源于根部及土壤.植物组织和根际土壤中ARGs的重叠与共现, 表明植物根系是ARGs从环境向植物体内传播的重要途径之一.另外, 粪肥源ARGs和ARB也可以借助作物运输营养物质(即水、无机盐和有机营养物质)及蒸腾作用或者通过作物根系而进入植物组织内部[41].先前有研究通过标记内生细菌发现, 内生菌可以通过维管束迁移到植物叶片部[42].携带ARGs的细菌也可能附着在蔬菜的叶表面成为叶际微生物, 作为叶内生菌在叶组织中定殖[30].植物也可以从土壤中吸收抗生素残留物, 对植物微生物产生长期选择压力, 从而导致植物抗性组分的出现[12].此外, 也有研究发现植物微生物组中存在周围环境(包括土壤)中未检出的ARGs, 这表明植物微生物组也具有特有的ARGs[43].由此可见, 粪肥源ARGs和ARB通过细菌在植物与土壤之间、植物与粪肥之间、土壤与粪肥之间的传播以及通过复杂的地上和地下营养转移, 在土壤-蔬菜系统中构成了一个广泛的畜禽源耐药性扩散网络(图 2).
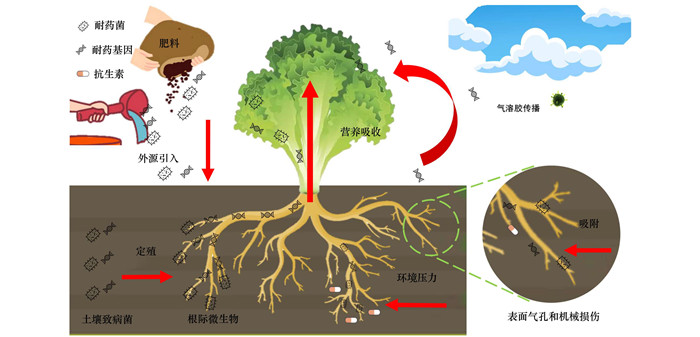
|
图 2 土壤-蔬菜系统中ARGs的迁移扩散途径 Fig. 2 Migration pathway of ARGs in soil-vegetable system |
粪肥源ARGs成功定殖在作物组织后, 与植物内生菌之间的相互作用还会涉及基因层面的交互过程[39]. 外源菌可利用其在植物中形成的共生生物膜(复杂的细菌聚集体)促进它们之间的基因交流, 进而帮助外源菌更好地适应植物内部生境, 并通过与植物内生菌相互作用, 使得植物内生环境逐渐利于粪肥源ARGs/耐药菌的定殖与生长, 最终形成稳定的共生关系[44]. Zhang等[19]的研究表明, 生菜根内生菌中ARGs的丰度远远高于其叶内生菌的丰度, 这证实了ARGs存在从地下部分向地上部分转移的趋势.另外, 根系微生物群落中仅12个细菌OTUs(Group Ⅱ)可能进一步传播到叶片内生菌, 这说明粪肥源ARGs从根系向地上部传输也存在梯度空间分布.植物组织内微生物的传播能力可直接影响ARGs的传播, 如厚壁菌门(Firmicutes)作为ARGs的潜在宿主菌, 在生物炭的作用下无法由根系向茎叶组织迁移, 导致tetW无法被迁移至茎叶部位[45].而且, 不同土壤生境会影响土壤作物间ARGs的传播, 且同种土壤条件下, 不同作物吸收积累ARGs的多样性和丰度亦不相同, 如生菜和小白菜在其体内相关微生物菌群中积累的ARGs比芫荽和莴苣多[28].
综上所述, 微生物动态变化以及营养转移可能是粪肥源ARGs和ARB在土壤-蔬菜系统中传播的主要原因, ARGs在土壤-植物系统中的定殖则主要归因于植物内生环境以及内生菌的变化.然而, 粪肥源ARGs在土壤-植物系统中的迁移和传播也会受到一些环境因素的影响, 包括蔬菜的种植模式、种植的蔬菜种类以及土壤性质.因此, 仍需要进一步分析环境因素与粪肥源ARGs迁移和传播之间的潜在关联.
3 土壤-蔬菜系统中ARGs迁移富集的影响因素 3.1 种植模式有研究表明, 与单作相比, 间作种植方式可以显著降低土壤-作物系统中的抗生素和重金属污染[46].由此可见, 种植模式(单作、间作以及轮作等)的变化也可以改变土壤-作物系统中ARGs的传播潜力.一项盆栽试验表明, 印度芥菜和黑麦草间作可减少土壤中11.40%~23.22%的ARGs积累[47].关于种植模式之间ARGs去除效果的差异, 主要归因于细菌群落组成的变化.在间作模式中, ARGs进行基因水平转移(horizontal gene transfer, HGT)的重要载体(主要是变形杆菌、放线菌、拟杆菌)比单作模式更为丰富;而在轮作模式中, 土壤优势菌群会随着作物的更替在绿弹性菌、变形杆菌、酸性菌和放线菌、厚壁菌间交互演替[23]. ARGs宿主细菌会受到土壤微生物群落的影响, 而采用不同的种植模式会通过改变细菌群落结构, 从而影响ARGs在土壤-植物系统中的转移和分布.除了种植模式外, 植物类型和土壤类型也可能对ARGs在土壤-植物系统中的传播产生重大影响.
3.2 蔬菜种类不同蔬菜种类(苦苣、生菜、油菜和芫荽等)的叶片特性(气孔、水合孔、叶脉沟槽和表皮细胞连接处等), 决定了叶片中ARGs与微生物的携带能力[48].Chen等[49]利用HT-qPCR证明了植物物种改变了植物叶片部内的ARGs分布;Guo等[28]在施用鸡粪种植蔬菜的研究中发现, 生菜和小白菜的叶子比芫荽和苦苣含有更多的ARGs.此外, 植物品种也决定了植物释放的挥发性有机化合物(如丙酮、醛类、醇类和倍半萜类)[50], 以上化合物的促发育和抗菌作用也影响着土壤细菌群落的结构和组成, 从而影响了ARB和ARGs在土壤-蔬菜系统中的扩散[51].Zhang等[52]发现高分子量(≥10 000 g·mol-1)的根渗出物通过与抗生素的络合作用促进四环素(TCH)在根际的沉积, 从而加强了四环素耐药菌的生长和抗性基因的丰度.Wang等[11]的研究发现, 种植生菜的土壤中ARGs的相对丰度高于种植苦苣的土壤.除蔬菜外, 在葡萄、大麻、甘蔗和其他经济作物中都发现了ARGs和微生物变异对于品种的依赖性[53].但目前关于蔬菜种类对土壤-蔬菜系统中ARGs影响的报道中考虑的条件较为单一, 机制解析尚不明确, 有待进一步加强研究的广度和深度.
3.3 土壤类型土壤类型是农业生态系统的重要因素, 对土壤中的理化性质、保水能力和微生物群落结构有显著影响[54].不同的土壤类型具有不同的特征, 如质地、排水和有机质, 以上特征是ARGs在土壤中传播的关键影响因子[55].例如, 较粗的土壤颗粒可以为细菌活动提供较大的比表面积和必要的营养素, 并进一步导致ARGs的HGT增强[56].此外, pH值、含水量、总氮和总磷等土壤特性也与ARGs的赋存和迁移有关[55, 57].有研究发现, 与褐土相比, 潮土和盐碱土中ARGs的相对丰度更高[54].而一项关于pH对ARGs影响的研究表明, 酸性环境有利于ARGs的繁殖, 但碱性环境会导致ARGs衰减[58]. Song等[59]在不同土壤类型中施用猪粪发现, 有机肥在红壤中的传播风险高于其他土壤.除了土壤性质外, 微生物群落是土壤生态系统的核心组成部分, 其组成和功能受土壤类型的影响[55].不同土壤类型间的差异微生物群落的组成和功能也在调节ARGs在土壤中的传播中起着重要作用[60].
3.4 污染物残留除种植模式、蔬菜类型和土壤类型外, 抗生素、重金属等污染物残留也是影响ARGs在土壤-植物系统中传播的重要因素.有研究表明, 高含量的抗生素会通过产生新的ARB增加环境中的选择性压力[61], 但低含量的抗生素则会促进ARB的生长[62].苗荪等[63]的研究发现, 低剂量抗生素能够选择性地抑制敏感细菌的生长, 有利于ARB的生存和繁殖.在土壤-蔬菜系统中, 植物吸收、土壤微生物代谢以及根际分泌物的降解可以降低土壤中抗生素的含量[64, 65], 进而促进了ARGs宿主菌的生长[66]. 另一方面, 抗生素的暴露持续时间也影响了土壤-蔬菜系统中ARGs的传播和扩散.有研究发现, 土壤中含有抗生素的肥料施用的持续时间和频率对于维持和传播土壤-植物系统中的抗生素耐药性非常重要[67, 68].例如, Liu等[67]研究了施用时间(沼液施用1、3和5 a)对ARGs变化的影响, 结果表明长期施用沼液会不断向土壤环境中引入抗生素, 导致持续的选择压力, 进而促进ARGs的扩散.
除了抗生素外, 重金属和有机污染物以及混合污染也影响着土壤-蔬菜系统中ARGs的繁殖和传播[69].有研究表明, 一定范围内重金属含量带来的共选择压力可以促进ARGs宿主细菌的进化[70]. 例如, Qi等[70]发现长期暴露于一定范围内的镍(Ni)污染(≤400 mg·kg-1)会显著增加ARGs的多样性和丰度.但对于植物, 在添加锌和铜的污水污泥改良土壤中收获的萝卜和生菜中没有观察到ARGs丰度的显著变化[21].土壤-蔬菜系统中的重金属可以通过拮抗剂效应、金属络合和共选择等机制与抗生素相互作用, 进而影响ARGs的扩散和传播.
4 土壤-蔬菜系统中ARGs污染防控措施随着畜禽粪污在农业中的广泛施用, 抗生素耐药性在土壤-植物系统中日益传播, 对粮食安全和公众健康构成重大威胁[71, 72].先前的大量研究发现, 蔬菜(特别是即食沙拉类)可食用部分中检测出大量具有抗生素耐药性的细菌, 以上耐药菌及相应的ARGs将通过直接接触或食物摄入在人类体内富集, 导致一些人类疾病的抗菌治疗失效[24].因此, 对畜禽源ARGs和ARB在土壤-蔬菜系统中的传播和扩散进行防控是十分必要的, 最大程度地减少向食物链中ARGs的输入, 降低向人体传播的风险.
目前, 对土壤-蔬菜系统中畜禽源ARGs和ARB的管控策略应首先以控制输入为主, 包括:①防止在畜禽养殖场及畜禽诊所中过度使用或滥用抗生素, 减少抗生素消费;②加强或优化畜禽废弃物中ARGs去除技术, 积极探索其他替代传统废弃物利用的方案.其次, 应当加强蔬菜在种植过程中的管控措施, 包括:①制定畜禽废弃物(动物粪便和污水污泥)还田的应用准则;②增加对农业生产中畜禽废弃物资源化利用技术的培训, 合理应用畜禽废弃物, 以控制和减轻畜禽源抗生素耐药性在土壤-蔬菜系统中的传播.最后, 还应当对土壤-蔬菜中畜禽源ARGs和ARB进入食物链以及对人类造成的风险进行阻控和监测, 包括:①增加蔬菜在最后一次施肥与收获之间的延迟期, 以防止抗生素残留和抗生素耐药性在植物中的传播;②加强对畜禽源ARGs和ARB在土壤-蔬菜系统中的基础监测, 全面评估畜禽源ARGs和ARB对食品安全和人类健康的风险.
5 展望目前的研究主要集中在明确传统畜禽粪肥的土壤-蔬菜系统中ARGs污染赋存情况及驱动因素, 但针对传统畜禽粪肥施用土壤-蔬菜系统中ARGs的传播机制解析以及非传统畜禽粪肥施用的土壤-蔬菜系统中ARGs的污染情况仍缺乏系统研究.因此, 未来的研究应进一步关注以下4点。
(1)土壤-蔬菜系统中ARGs污染不容忽视, 尽管已有大量研究报道了土壤-蔬菜系统中ARGs的污染特征, 但进一步明晰畜禽源ARGs在土壤-蔬菜系统中的传播与迁移机制是控制ARGs传播和降低其健康风险的关键, 故开展畜禽源ARGs在土壤-蔬菜中的机制解析研究非常必要;
(2)肥料种类繁多, 不同肥料间存在显著差异, 虽已有研究对牛粪、猪粪以及鸡粪开展了相关工作, 但若要全面了解和系统评估土壤-蔬菜系统中ARGs污染的情况, 还需进一步加强对不同畜禽废弃物(牛粪、羊粪等)以及生态友好型粪肥代替物(蚯蚓粪等)等在土壤-蔬菜系统中施用的监测研究;
(3)土壤-蔬菜系统中ARGs迁移富集的影响因素探究尚不完整, 土壤-蔬菜系统是多重污染物共存的复杂体系, 而ARGs的迁移富集与其他共存污染物(重金属、抗生素及微塑料等)的交互作用及其对蔬菜耐药性的影响协同机制至今尚未明晰, 亟需进一步探究;
(4)为真正实现畜禽废弃物资源化、无害化利用, 推动畜禽养殖产业的绿色可持续发展, 需要进一步明确施用畜禽粪肥种植的蔬菜中ARB和ARGs的食用安全风险, 对土壤-蔬菜系统中的耐药性风险进行量化分析, 建立风险评估标准及预警体系, 以预防与食用含有ARB的蔬菜相关的人类健康风险.
6 结论本文对土壤-蔬菜系统中畜禽源ARGs的来源危害、环境行为以及管控措施进行了综述.土壤-蔬菜系统中ARGs和ARB的出现主要归因于固体畜禽粪便还田以及液体畜禽废水灌溉等农业措施带来的ARGs和抗生素残留.ARGs在土壤-蔬菜系统中呈现出土壤 > 非可食用部分 > 可食用部分的赋存特征, 细菌群落的动态变化和植物的营养吸收对ARGs在土壤和蔬菜间的传播起着至关重要的作用.此外, 种植模式、蔬菜种类以及土壤类型也对ARGs的迁移产生影响.
| [1] | Han M Z, Zhang L, Zhang N, et al. Antibiotic resistome in a large urban-lake drinking water source in middle China: dissemination mechanisms and risk assessment[J]. Journal of Hazardous Materials, 2022, 424. DOI:10.1016/j.jhazmat.2021.127745 |
| [2] | Lin Z B, Yuan T, Zhou L, et al. Impact factors of the accumulation, migration and spread of antibiotic resistance in the environment[J]. Environmental Geochemistry and Health, 2021, 43(5): 1741-1758. DOI:10.1007/s10653-020-00759-0 |
| [3] | Wang F, Fu Y H, Sheng H J, et al. Antibiotic resistance in the soil ecosystem: a one health perspective[J]. Current Opinion in Environmental Science & Health, 2021, 20. DOI:10.1016/j.coesh.2021.100230 |
| [4] | Mu M R, Yang F X, Han B J, et al. Manure application: a trigger for vertical accumulation of antibiotic resistance genes in cropland soils[J]. Ecotoxicology and Environmental Safety, 2022, 237. DOI:10.1016/j.ecoenv.2022.113555 |
| [5] | Martínez J L, Coque T M, Baquero F. What is a resistance gene? Ranking risk in resistomes[J]. Nature Reviews Microbiology, 2015, 13(2): 116-123. DOI:10.1038/nrmicro3399 |
| [6] | Zhu D, Delgado-Baquerizo M, Su J Q, et al. Deciphering potential roles of earthworms in mitigation of antibiotic resistance in the soils from diverse ecosystems[J]. Environmental Science & Technology, 2021, 55(11): 7445-7455. |
| [7] | Van den Meersche T, Rasschaert G, Vanden Nest T, et al. Longitudinal screening of antibiotic residues, antibiotic resistance genes and zoonotic bacteria in soils fertilized with pig manure[J]. Environmental Science and Pollution Research, 2020, 27(22): 28016-28029. DOI:10.1007/s11356-020-09119-y |
| [8] | Wang H, Qi J F, Qin R, et al. Intensified livestock farming increases antibiotic resistance genotypes and phenotypes in animal feces[J]. Communications Earth & Environment, 2023, 4(1). DOI:10.1038/s43247-023-00790-w |
| [9] | Laconi A, Mughini-Gras L, Tolosi R, et al. Microbial community composition and antimicrobial resistance in agricultural soils fertilized with livestock manure from conventional farming in northern Italy[J]. Science of the Total Environment, 2021, 760. DOI:10.1016/j.scitotenv.2020.143404 |
| [10] |
张红娜, 董梦洁, 周玉法, 等. 动物粪便施肥措施促进耐药基因在粪便-土壤-蔬菜之间的散播[J]. 环境科学, 2021, 42(4): 2080-2088. Zhang H N, Dong M J, Zhou Y F, et al. Animal manure fertilization promotes antibiotic resistance gene dissemination among manure, soil, and vegetables[J]. Environmental Science, 2021, 42(4): 2080-2088. |
| [11] | Wang F H, Sun R B, Hu H W, et al. The overlap of soil and vegetable microbes drives the transfer of antibiotic resistance genes from manure-amended soil to vegetables[J]. Science of the Total Environment, 2022, 828. DOI:10.1016/j.scitotenv.2022.154463 |
| [12] | Chen P, Yu K F, He Y L. The dynamics and transmission of antibiotic resistance associated with plant microbiomes[J]. Environment International, 2023, 176. DOI:10.1016/j.envint.2023.107986 |
| [13] | Gu Y R, Shen S Z, Han B J, et al. Family livestock waste: an ignored pollutant resource of antibiotic resistance genes[J]. Ecotoxicology and Environmental Safety, 2020, 197. DOI:10.1016/j.ecoenv.2020.110567 |
| [14] | Mu M R, Yang F X, Han B J, et al. Insights into the panorama of antibiotic resistome in cropland soils amended with vermicompost in China[J]. Science of the Total Environment, 2023, 868. DOI:10.1016/j.scitotenv.2023.161658 |
| [15] | Fan X T, Su J Q, Zhou S Y D, et al. Plant cultivar determined bacterial community and potential risk of antibiotic resistance gene spread in the phyllosphere[J]. Journal of Environmental Sciences, 2023, 127: 508-518. DOI:10.1016/j.jes.2022.06.006 |
| [16] | Yang F X, Han B J, Gu Y R, et al. Swine liquid manure: a hotspot of mobile genetic elements and antibiotic resistance genes[J]. Scientific Reports, 2020, 10(1). DOI:10.1038/s41598-020-72149-6 |
| [17] | Buta M, Hubeny J, Zieliński W, et al. Sewage sludge in agriculture-the effects of selected chemical pollutants and emerging genetic resistance determinants on the quality of soil and crops-a review[J]. Ecotoxicology and Environmental Safety, 2021, 214. DOI:10.1016/j.ecoenv.2021.112070 |
| [18] | Kampouris I D, Agrawal S, Orschler L, et al. Antibiotic resistance gene load and irrigation intensity determine the impact of wastewater irrigation on antimicrobial resistance in the soil microbiome[J]. Water Research, 2021, 193. DOI:10.1016/j.watres.2021.116818 |
| [19] | Zhang Y, Zhou J, Wu J, et al. Distribution and transfer of antibiotic resistance genes in different soil-plant systems[J]. Environmental Science and Pollution Research, 2022, 29(39): 59159-59172. DOI:10.1007/s11356-021-17465-8 |
| [20] | Liu Y, Neal A L, Zhang X X, et al. Cropping system exerts stronger influence on antibiotic resistance gene assemblages in greenhouse soils than reclaimed wastewater irrigation[J]. Journal of Hazardous Materials, 2022, 425. DOI:10.1016/j.jhazmat.2021.128046 |
| [21] | You R, Margenat A, Lanzas C S, et al. Dose effect of Zn and Cu in sludge-amended soils on vegetable uptake of trace elements, antibiotics, and antibiotic resistance genes: human health implications[J]. Environmental Research, 2020, 191. DOI:10.1016/j.envres.2020.109879 |
| [22] | Wen X, Xu J J, Xiang G F, et al. Multiple driving factors contribute to the variations of typical antibiotic resistance genes in different parts of soil-lettuce system[J]. Ecotoxicology and Environmental Safety, 2021, 225. DOI:10.1016/j.ecoenv.2021.112815 |
| [23] | Han B J, Yang F X, Shen S Z, et al. Effects of soil habitat changes on antibiotic resistance genes and related microbiomes in paddy fields[J]. Science of the Total Environment, 2023, 895. DOI:10.1016/j.scitotenv.2023.165109 |
| [24] | Shen Y K, Ryser E T, Li H, et al. Bacterial community assembly and antibiotic resistance genes in the lettuce-soil system upon antibiotic exposure[J]. Science of the Total Environment, 2021, 778. DOI:10.1016/j.scitotenv.2021.146255 |
| [25] | Chen Z Q, Bai J, Wang S J, et al. Prevalence, antimicrobial resistance, virulence genes and genetic diversity of Salmonella isolated from retail duck meat in Southern China[J]. Microorganisms, 2020, 8(3). DOI:10.3390/microorganisms8030444 |
| [26] | Zekar F M, Granier S A, Touati A, et al. Occurrence of third-generation cephalosporins-resistant Klebsiella pneumoniae in fresh fruits and vegetables purchased at markets in Algeria[J]. Microbial Drug Resistance, 2020, 26(4): 353-359. DOI:10.1089/mdr.2019.0249 |
| [27] | Stadler K, Masignani V, Eickmann M, et al. SARS—beginning to understand a new virus[J]. Nature Reviews Microbiology, 2003, 1(3): 209-218. DOI:10.1038/nrmicro775 |
| [28] | Guo Y J, Qiu T L, Gao M, et al. Diversity and abundance of antibiotic resistance genes in rhizosphere soil and endophytes of leafy vegetables: focusing on the effect of the vegetable species[J]. Journal of Hazardous Materials, 2021, 415. DOI:10.1016/j.jhazmat.2021.125595 |
| [29] | Wang Y Z, Zhou S Y D, Zhou X Y, et al. Manure and biochar have limited effect on lettuce leaf endophyte resistome[J]. Science of the Total Environment, 2023, 860. DOI:10.1016/j.scitotenv.2022.160515 |
| [30] | Zhang Y J, Hu H W, Chen Q L, et al. Transfer of antibiotic resistance from manure-amended soils to vegetable microbiomes[J]. Environment International, 2019, 130. DOI:10.1016/j.envint.2019.104912 |
| [31] | Zhang Y, Zhao J Y, Chen M L, et al. Fecal antibiotic resistance genes were transferred through the distribution of soil-lettuce-snail food chain[J]. Environmental Science and Pollution Research, 2023, 30(37): 87793-87809. DOI:10.1007/s11356-023-28606-6 |
| [32] | Zhang Y, Cheng D M, Xie J, et al. Long-term field application of manure induces deep selection of antibiotic resistomes in leaf endophytes of Chinese cabbage[J]. Science of the Total Environment, 2023, 882. DOI:10.1016/j.scitotenv.2023.163334 |
| [33] | Gao Y J, Luo W, Zhang H F, et al. Enrichment of antibiotic resistance genes in roots is related to specific bacterial hosts and soil properties in two soil-plant systems[J]. Science of the Total Environment, 2023, 886. DOI:10.1016/j.scitotenv.2023.163933 |
| [34] | Yue Z F, Zhang J, Ding C F, et al. Transfer and distribution of antibiotic resistance genes in the soil-peanut system receiving manure for years[J]. Science of the Total Environment, 2023, 869. DOI:10.1016/j.scitotenv.2023.161742 |
| [35] | Wang L Q, Li Y, Zhao Z, et al. Tidal flat aquaculture pollution governs sedimentary antibiotic resistance gene profiles but not bacterial community based on metagenomic data[J]. Science of the Total Environment, 2022, 833. DOI:10.1016/j.scitotenv.2022.155206 |
| [36] | Zhu D, Ma J, Li G, et al. Soil plastispheres as hotspots of antibiotic resistance genes and potential pathogens[J]. The ISME Journal, 2022, 16(2): 521-532. DOI:10.1038/s41396-021-01103-9 |
| [37] | Sun Y P, Snow D, Walia H, et al. Transmission routes of the microbiome and resistome from manure to soil and lettuce[J]. Environmental Science & Technology, 2021, 55(16): 11102-11112. |
| [38] | Yu Y T, Zhang Q, Zhang Z Y, et al. Plants select antibiotic resistome in rhizosphere in early stage[J]. Science of the Total Environment, 2023, 858. DOI:10.1016/j.scitotenv.2022.159847 |
| [39] | Xu H, Chen Z Y, Huang R Y, et al. Antibiotic resistance gene-carrying plasmid spreads into the plant endophytic bacteria using soil bacteria as carriers[J]. Environmental Science & Technology, 2021, 55(15): 10462-10470. |
| [40] |
生弘杰, 王芳, 相雷雷, 等. 土壤中抗生素抗性基因的环境行为与阻控研究进展[J]. 土壤学报, 2023, 60(1): 39-49. Sheng H J, Wang F, Xiang L L, et al. Environmental behavior and control of antibiotic resistance genes in soil — a review[J]. Acta Pedologica Sinica, 2023, 60(1): 39-49. |
| [41] | In't Zandt D, Kolaříková Z, Cajthaml T, et al. Plant community stability is associated with a decoupling of prokaryote and fungal soil networks[J]. Nature Communications, 2023, 14(1). DOI:10.1038/s41467-023-39464-8 |
| [42] | Hou S J, Thiergart T, Vannier N, et al. A microbiota-root-shoot circuit favours Arabidopsis growth over defence under suboptimal light[J]. Nature Plants, 2021, 7(8): 1078-1092. DOI:10.1038/s41477-021-00956-4 |
| [43] | Huang R L, Ding J X, Guo Y W, et al. Habitat determines the relationships among bacteria, resistance genes and mobile genetic elements in the soil–plant system[J]. European Journal of Soil Science, 2022, 73(1). DOI:10.1111/ejss.13132 |
| [44] | Li L G, Dechesne A, Madsen J S, et al. Plasmids persist in a microbial community by providing fitness benefit to multiple phylotypes[J]. The ISME Journal, 2020, 14(5): 1170-1181. DOI:10.1038/s41396-020-0596-4 |
| [45] | Duan M L, Li Z J, Yan R P, et al. Mechanism for combined application of biochar and Bacillus cereus to reduce antibiotic resistance genes in copper contaminated soil and lettuce[J]. Science of the Total Environment, 2023, 884. DOI:10.1016/j.scitotenv.2023.163422 |
| [46] | Cao X R, Luo J P, Wang X Z, et al. Responses of soil bacterial community and Cd phytoextraction to a Sedum alfredii-oilseed rape (Brassica napus L. and Brassica juncea L.) intercropping system[J]. Science of the Total Environment, 2020, 723. DOI:10.1016/j.scitotenv.2020.138152 |
| [47] | Cui E P, Cui B J, Fan X Y, et al. Ryegrass (Lolium multiflorum L.) and Indian mustard (Brassica juncea L.) intercropping can improve the phytoremediation of antibiotics and antibiotic resistance genes but not heavy metals[J]. Science of the Total Environment, 2021, 784. DOI:10.1016/j.scitotenv.2021.147093 |
| [48] | Li J, Daniell T J, Jin M K, et al. Phyllosphere antibiotic resistome in a natural primary vegetation across a successional sequence after glacier retreat[J]. Environment International, 2023, 174. DOI:10.1016/j.envint.2023.107903 |
| [49] | Chen Q L, Hu H W, Zhu D, et al. Host identity determines plant associated resistomes[J]. Environmental Pollution, 2020, 258. DOI:10.1016/j.envpol.2019.113709 |
| [50] | Liu Z X, Wang M Y, Wu M, et al. Volatile organic compounds (VOCs) from plants: from release to detection[J]. TrAC Trends in Analytical Chemistry, 2023, 158. DOI:10.1016/j.trac.2022.116872 |
| [51] | Zhao M L, Zhao J, Yuan J, et al. Root exudates drive soil-microbe-nutrient feedbacks in response to plant growth[J]. Plant, Cell & Environment, 2021, 44(2): 613-628. |
| [52] | Zhang M, Cai Z X, Zhang G F, et al. Abiotic mechanism changing tetracycline resistance in root mucus layer of floating plant: the role of antibiotic-exudate complexation[J]. Journal of Hazardous Materials, 2021, 416. DOI:10.1016/j.jhazmat.2021.125728 |
| [53] | Comeau D, Novinscak A, Joly D L, et al. Spatio-temporal and cultivar-dependent variations in the cannabis microbiome[J]. Frontiers in Microbiology, 2020, 11. DOI:10.3389/fmicb.2020.00491 |
| [54] | Wang L J, Wang J H, Wang J, et al. Soil types influence the characteristic of antibiotic resistance genes in greenhouse soil with long-term manure application[J]. Journal of Hazardous Materials, 2020, 392. DOI:10.1016/j.jhazmat.2020.122334 |
| [55] |
陈瑞, 程建华, 唐翔宇. 高垦殖丘陵区不同类型农用地土壤中抗生素抗性基因分布特征[J]. 环境科学, 2023, 44(12): 6947-6954. Chen R, Cheng J H, Tang X Y. Characteristics of antibiotic resistance genes distribution in different types of agricultural land soils in highly cultivated hilly areas[J]. Environmental Science, 2023, 44(12): 6947-6954. |
| [56] |
李冰, 李玉双, 魏建兵, 等. 不同土地利用方式对土壤细菌分子生态网络的影响[J]. 环境科学, 2020, 41(3): 1456-1465. Li B, Li Y S, Wei J B, et al. Effects of different land use typess on the molecular ecological network of soil bacteria[J]. Environmental Science, 2020, 41(3): 1456-1465. |
| [57] | Qiu L L, Wu J J, Qian Y, et al. Impact of biochar-induced vertical mobilization of dissolved organic matter, sulfamethazine and antibiotic resistance genes variation in a soil-plant system[J]. Journal of Hazardous Materials, 2021, 417. DOI:10.1016/j.jhazmat.2021.126022 |
| [58] | Liu Z S, Zhao Y X, Zhang B F, et al. Deterministic effect of pH on shaping soil resistome revealed by metagenomic analysis[J]. Environmental Science & Technology, 2023, 57(2): 985-996. |
| [59] | Song T T, Zhu C X, Li B X, et al. Manure application led to higher antibiotic resistance risk in red soil compared with black soil and fluvo-aquic soil[J]. Journal of Hazardous Materials Advances, 2023, 9. DOI:10.1016/j.hazadv.2022.100209 |
| [60] | Li H Y, Cheng W M, Li B H, et al. The fate of antibiotic resistance genes during co-composting of swine manure with cauliflower and corn straw[J]. Bioresource Technology, 2020, 300. DOI:10.1016/j.biortech.2019.122669 |
| [61] | Zhao R X, Yu K, Zhang J Y, et al. Deciphering the mobility and bacterial hosts of antibiotic resistance genes under antibiotic selection pressure by metagenomic assembly and binning approaches[J]. Water Research, 2020, 186. DOI:10.1016/j.watres.2020.116318 |
| [62] | Xiong Q, Hu L X, Liu Y S, et al. Microalgae-based technology for antibiotics removal: from mechanisms to application of innovational hybrid systems[J]. Environment International, 2021, 155. DOI:10.1016/j.envint.2021.106594 |
| [63] |
苗荪, 陈磊, 左剑恶. 环境中抗生素抗性基因丰度与抗生素和重金属含量的相关性分析: 基于Web of Science数据库检索[J]. 环境科学, 2021, 42(10): 4925-4932. Miao S, Chen L, Zuo J E. Correlation analysis among environmental antibiotic resistance genes abundance, antibiotics concentrations, and heavy metals concentrations based on web of science searches[J]. Environmental Science, 2021, 42(10): 4925-4932. |
| [64] | Huang D L, Xiao R H, Du L, et al. Phytoremediation of poly- and perfluoroalkyl substances: a review on aquatic plants, influencing factors, and phytotoxicity[J]. Journal of Hazardous Materials, 2021, 418. DOI:10.1016/j.jhazmat.2021.126314 |
| [65] | Kurade M B, Ha Y H, Xiong J Q, et al. Phytoremediation as a green biotechnology tool for emerging environmental pollution: a step forward towards sustainable rehabilitation of the environment[J]. Chemical Engineering Journal, 2021, 415. DOI:10.1016/j.cej.2021.129040 |
| [66] | Song M K, Peng K, Jiang L F, et al. Alleviated antibiotic-resistant genes in the rhizosphere of agricultural soils with low antibiotic concentration[J]. Journal of Agricultural and Food Chemistry, 2020, 68(8): 2457-2466. DOI:10.1021/acs.jafc.9b06634 |
| [67] | Liu C, Chen Y X, Li X H, et al. Temporal effects of repeated application of biogas slurry on soil antibiotic resistance genes and their potential bacterial hosts[J]. Environmental Pollution, 2020, 258. DOI:10.1016/j.envpol.2019.113652 |
| [68] | Zheng F, Bi Q F, Giles M, et al. Fates of antibiotic resistance genes in the gut microbiome from different soil fauna under long-term fertilization[J]. Environmental Science & Technology, 2021, 55(1): 423-432. |
| [69] | Fu Y H, Jia M Y, Wang F, et al. Strategy for mitigating antibiotic resistance by biochar and hyperaccumulators in cadmium and oxytetracycline co-contaminated soil[J]. Environmental Science & Technology, 2021, 55(24): 16369-16378. |
| [70] | Qi Z, Qi Y, Le Z W, et al. The Interactions between antibiotic resistance genes and heavy metal pollution under co-selective pressure influenced the bio-enzyme activity[J]. Frontiers in Chemistry, 2021, 9. DOI:10.3389/fchem.2021.691565 |
| [71] | Sorinolu A J, Tyagi N, Kumar A, et al. Antibiotic resistance development and human health risks during wastewater reuse and biosolids application in agriculture[J]. Chemosphere, 2021, 265. DOI:10.1016/j.chemosphere.2020.129032 |
| [72] | Zainab S M, Junaid M, Xu N, et al. Antibiotics and antibiotic resistant genes (ARGs) in groundwater: a global review on dissemination, sources, interactions, environmental and human health risks[J]. Water Research, 2020, 187. DOI:10.1016/j.watres.2020.116455 |
 2024, Vol. 45
2024, Vol. 45


