2. 河北省生态环境科学研究院, 石家庄 050037;
3. 河北省污染防治生物技术实验室, 石家庄 050000
2. Hebei Provincial Academy of Ecological Environmental Science, Shijiazhuang 050037, China;
3. Hebei Key Laboratory of Pollution Prevention Biotechnology, Shijiazhuang 050000, China
近年来, 抗生素已被广泛用于人类和动物疾病的治疗[1]. 根据其化学结构, 可分为喹诺酮类(quinolones, QNs)、β-内酰胺类(β-lactam)、大环内酯类(macrolides, MLs)和磺胺类(sulfonamides, SAs)抗生素等[2]. 其中, QNs抗生素因具有价格低廉、抗菌性强和毒副作用小等特点, 被广泛作为人类用药和兽用药[3, 4]. 据统计, 2020年全球抗生素消费量已达22.5万t, 且呈现逐年增长的趋势[5]. 我国作为全球最大的抗生素生产和使用国, 年使用量约占全球消费量的45%[6]. 然而, 70%以上的抗生素不能被生物体完全吸收, 多数随尿液和粪便通过污水处理系统运输进入环境中[7, 8]. 虽然水环境中的抗生素浓度较低, 但长期接触仍会对生物体造成潜在风险, 并对微生物群落产生影响[9, 10].
目前, 已在我国多种水环境中检出各类抗生素, 包括湖泊[11]、河流[12]、海湾[13]和河口[14]等. 其中, 湖泊作为重要的水生生态系统, 与其他水环境相比, 其污染物停留时间长. 此外, 沉积物作为水生态系统中主要的微生物聚集地, 与水体具有良好的交换作用且连通性好, 是水环境中抗生素理想的储库[15 ~ 17]. 与其他污染物相比, 抗生素不仅会抑制某些微生物的生长, 还会刺激携带抗生素耐药基因(antibiotic resistance genes, ARGs), 导致微生物群落结构和生物地球化学功能发生变化[18, 19]. 加之, 微生物作为物质循环的主要参与者, 对湖泊生态系统的稳定性具有重要作用. 目前, 抗生素对微生物群落的影响主要表现在丰度、多样性、组成和功能这4个方面. 如:高含量的磺胺嘧啶导致土壤中细菌群落的相对丰度显著降低[20];磺胺甲唑可作为碳源促进微生物的繁殖, 使其群落丰度上升[17];土霉素通过抑制土壤中的氨氧化微生物, 使其微生物群落结构多样性发生改变[21];且随土霉素含量的增加, 沉积物中微生物群落的多样性显著降低[22];恩诺沙星等抗生素的存在显著降低了群落的多样性, 并使菌群结构发生改变[23];左旋氟沙星和土霉素能在属水平上显著改变原核微生物群落结构[24];氧氟沙星等抗生素能增加微生物群落的反硝化功能基因的丰度[25];高含量的磺胺噻唑通过抑制氨氧化等功能细菌的生长来改变群落功能[26]. 然而, 以上研究较少关注自然湖泊中低浓度抗生素干扰下微生物群落结构和功能基因的时空变化, 以及抗生素对其功能基因的影响. 因此, 本研究拟通过阐明自然湖泊中微生物群落结构和功能基因的时空变化规律, 建立抗生素与微生物群落的相关关系, 并进一步识别长期低浓度抗生素干扰下湖泊微生物群落结构和功能的主要影响因素.
白洋淀作为华北平原最大的浅水草型湖泊, 也是雄安新区的重要生态支撑, 具有众多生态功能[27]. 此前研究表明白洋淀中抗生素含量较高, 但研究多集中于其污染特征、生态风险和源解析等方面, 而关于抗生素对其微生物群落结构和功能基因影响的研究仍未见报道[28 ~ 30]. 因此, 本文选择白洋淀为研究区域, 分析典型抗生素——QNs含量及其时空分布特征, 并利用16S rRNA高通量测序技术对沉积物中微生物群落组成、功能和多样性进行研究, 探讨微生物群落的时空变化特征及其影响因素, 以期为白洋淀生态修复和抗生素风险管控提供科学的理论依据和数据支撑.
1 材料与方法 1.1 样品采集通过文献和现场调研, 于2018年8月和2019年4月共选取16个采样点采集沉积物样品. 并根据地理位置将其划分为7个区, 分别为藻苲淀区(S1~S3)、南刘庄区(S4~S6)、烧车淀区(S7)、枣林庄区(S8~S10)、采蒲台区(S11、S12)、圈头区(S13、S14)和端村区(S15、S16)(图 1). 使用干净的彼得森采泥器在各样点0~10cm处采集沉积物样品并去除其中异物, 用聚乙烯塑料袋密封并进行标记, 低温保存运送至实验室, 在-20℃的黑暗环境中冷冻保存备用[31]. 各区分别取均值作为该区的指标值.
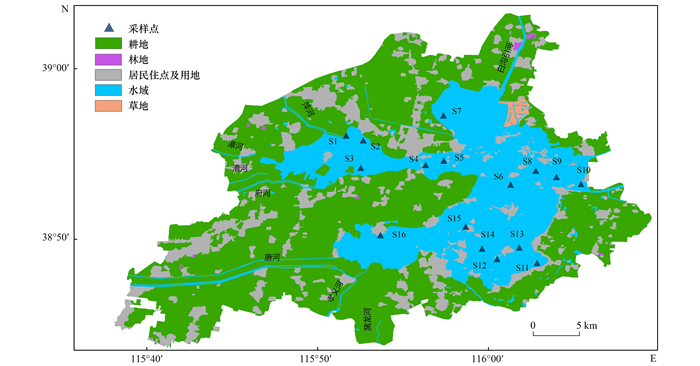
|
图 1 白洋淀采样点分布示意 Fig. 1 Distribution of sampling sites in Baiyangdian Lake |
用于测定抗生素的沉积物样品经冷冻干燥后粉碎研磨, 过40目筛, 称取4.0 g过筛后的样品和适量的硅藻土(Na2EDTA处理过的), 将其混合均匀装入(34 mL)萃取池, 以乙腈-磷酸盐缓冲液(pH = 3)为萃取液. 使用ASE 350快速溶剂萃取仪(Thermo, Germany)进行萃取, 萃取液用平行浓缩蒸发仪(Buchi, Switzerland)浓缩至1mL以下, 转移至锥形瓶中, 用超纯水稀释至200 mL待测[32].
1.3 抗生素的测定样品中的抗生素含量采用高效液相色谱-三重四极杆质谱联用仪(HPLC-MS/MS)进行测定, 仪器具体操作参考文献[28]. 用1 mol·L-1 H2SO4溶液调节pH为3, 将InertSep HLB固相萃取柱依次用6 mL甲醇和6 mL超纯水进行活化. 萃取后在负压下抽真空干燥30 min, 依次用6 mL氨水甲醇溶液(体积比为2%)和6 mL纯甲醇溶液洗脱, 洗脱液经氮吹至近干后, 用甲醇水溶液(1∶1, 体积比)定容至1 mL.
本研究选取的目标抗生素为QNs, 分别为氟甲喹(flumequine, FLU)、氧氟沙星(ofloxacin, OFL)、氟罗沙星(fleroxacin, FLE)、马波沙星(marbofloxacin, MAR)、环丙沙星(ciprofloxacin, CIP)、恩诺沙星(enrofloxacin, ENR)、诺氟沙星(norfloxacin, NOR)、依诺沙星(enoxacin, ENO)、沙氟沙星(sarafloxacin, SAR)、奥比沙星(orbifloxacin, ORB)、喹酸(oxolinic acid, OXO)、吡哌酸(pipemidic Acid, PIP)、双氟沙星(difloxacin, DIF)和司帕沙星(sparfloxacin, SPA). 标准品均购自Sigma-Aldrich(steinheim, Germany), 试剂均属于分析纯(纯度 > 95%).
1.4 DNA提取与测序取适量新鲜的沉积物样品, 将其置于50 mL离心管内(V沉积物样品 > 30 mL)做好标记, 送至上海美吉生物医药科技有限公司进行16S rRNA基因高通量测序. 根据制造商的协议使用DNeasy PowerSoil Kit(QI AGEN, Hilden, Germany)试剂盒提取样品中的DNA. 用引物338F(5′-ACTCCTACGGGAGGCAGCAG-3′)和806R(5′-GGACTACHVGGGTWTCTAAT-3′)进行PCR扩增, 利用2%的琼脂糖凝胶电泳检测扩增产物, 使用Illumina MiSeq平台进行基因测序. 分别用Trimmomatic和FLASH软件对原始DNA序列进行质控和拼接以得到优质序列, 根据97%的相似度对序列进行OTU聚类[18, 33].
1.5 质量控制采用内标法进行定量. 用甲醇水溶液(1∶1, 体积比)将标准储备液逐级稀释成浓度依次为0.1、0.5、1.0、5.0、10.0、50.0和100.0 ng·mL-1的标准系列, 并设空白组. 经HPLC-MS分析获得质量浓度与峰面积的标准曲线(相关系数均 > 0.99), 各QNs的回收率为71.3%~105.2%.
1.6 数据统计与分析采样点图基于ArcGIS 10.7绘制, 使用SPSS Statistics 26进行Pearson相关性分析, 用单因素方差进行组间差异性分析. 采用Kruskal-Wallis检验分析白洋淀沉积物中门和属分类上微生物群落的差异性. 为避免共现性的问题, 通过VIF方差膨胀因子分析将自相关性较高(VIF > 10)的环境变量进行剔除, 再用Canoco 5.0进行RDA分析以判断环境变量对微生物群落的影响. 通过美吉生信云分析平台进行微生物群落α多样性分析、群落组成分析和PICRUSt功能基因预测, 使用Origin pro 2021绘制微生物多样性指数的柱状分布图和功能基因丰度的条形图等.
2 结果与分析 2.1 白洋淀沉积物中QNs抗生素的时空分异特征白洋淀沉积物中QNs抗生素的检出情况如表 1所示. 就检出率而言, 2018年8月共检出11种QNs, 其中PIP(71.43%)、FLE(71.43%)、SPA(71.43%)、OXO(100%)和FLU(100%)的检出率较高;而2019年4月仅检出5种, OFL(57.14%)和FLU(85.71%)的检出率较高. 就检出含量而言, 8月ω(CIP)平均值最高(1.04 ng·g-1), ω(SPA)平均值最低(0.12 ng·g-1);4月ω(OFL)平均值最高(2.97 ng·g-1), ω(OXO)平均值最低(0.10 ng·g-1). 总体上, 从2018年8月至2019年4月, ω(QNs)平均值由3.91 ng·g-1升高为6.34 ng·g-1.
|
|
表 1 白洋淀沉积物中QNs的检出率和检出含量1) Table 1 Detection rate and content of QNs in sediment of Baiyangdian Lake |
本研究中, 选取两月份均检出且检出率较高的QNs类抗生素以研究其时空分布特征, 如图 2所示. 其中, 2018年8月CIP、OFL、OXO、FLU和总QNs在圈头和烧车淀的含量平均值最高;而2019年4月则在南刘庄、采蒲台和圈头的含量平均值最高, 其他区域含量较低. 加之, 除OXO外, 从2018年8月至2019年4月, ω(OFL)、ω(CIP)、ω(FLU)和ω(QNs)平均值均呈升高趋势, 分别由0.34、1.04、0.43和3.91 ng·g-1升高为2.97、2.36、0.70和6.34 ng·g-1. QNs作为人畜共用抗生素, 并作为生长促进剂在水产养殖中使用广泛, 而淀区养殖业较多[34]. 因此, 其含量的升高可能与当地春季饲料和诱饵的投放量增加有关. 此外, 单因素方差分析结果表明OXO和总QNs含量季节差异显著(P < 0.05).

|
图 2 白洋淀沉积物总QNs含量的时空分布特征 Fig. 2 Spatiotemporal distribution of total QNs content in sediment of Baiyangdian Lake |
就微生物群落组成而言, 2018年8月共检出56门564属, 共计978种;而2019年4月则检出50门602属, 共计1 092种. 在门水平上, 2018年8月至2019年4月主要菌门并无显著变化, 均为变形菌门(Proteobacteria)和绿弯菌门(Chloroflexi), 变形菌门的平均相对丰度由37.7%降为34.8%, 而绿弯菌门的平均相对丰度由18.5%升为19.8%[图 3(a)和图 3(b)]. 在属水平上, 前5种相对丰度较高的菌属由norank_ f__Anaerolineaceae、硫杆菌属(Thiobacillus)、norank_c__Acidobacteria、norank_c__Bacteroidetes_vadinHA17和脱氯单胞菌属(Dechloromonas)变为不动杆菌属(Acinetobacter)、norank_ f__Anaerolineaceae、norank_o__SBR1031、铜绿假单胞菌属(Paenisporosarcina)和硫杆菌属(Thiobacillus)[图 3(c)和3(d)]. 其中, 2018年8月norank_ f__Anaerolineaceae和硫杆菌属为主要优势菌属, 相对丰度分别为5.17%(采蒲台)~12.6%(枣林庄)和1.52%(端村)~7.63%(圈头);2019年4月的优势菌属分别为不动杆菌属和norank_ f_Anaerolineaceae, 其相对丰度分别为1.56%(端村)~1.33%(南刘庄)和3.14%(圈头)~8.99%(端村). 根据Kruskal-Wallis秩和检验, 属水平微生物群落组成空间差异显著, 而门水平微生物群落组成并无显著空间差异(P ≥ 0.05).

|
(a)和(b)分别为8月和4月沉积物中门水平微生物群落组成;(c)和(d)分别为8月和4月沉积物中属水平微生物群落组成 图 3 白洋淀沉积物中微生物群落时空分布 Fig. 3 Spatiotemporal distribution of microbial communities in sediment of Baiyangdian Lake |
两个季节的Coverage指数平均值均大于0.95, 表明此次测序结果可以较好代表样品中微生物的真实情况, 数据的可靠性较高. OTU聚类和α多样性分析结果表明(图 4), 2018年8月至2019年4月, Simpson指数、Chao指数和Ace指数的平均值分别由0.004 9、3 668和3 623升高为0.013、4 320和4 427;加之, OTU数量的平均值由3 463升高为4 438, 表明白洋淀沉积物中微生物群落多样性和丰度均呈升高趋势.
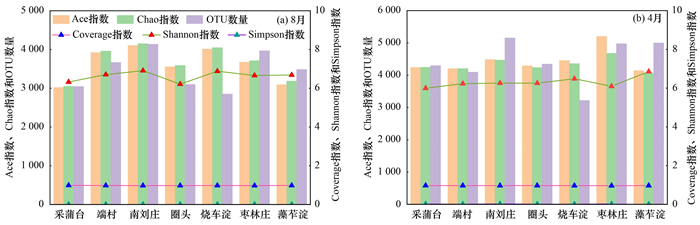
|
图 4 多样性指数和OTU数量的时空分布 Fig. 4 Spatiotemporal distribution of diversity index and OTU number |
多样性指数分析结果表明(图 4), 2018年8月和2019年4月Simpson指数的数值范围分别为0.003~0.009和0.003~0.023, 最大值均在南刘庄, 最小值分别在藻苲淀和圈头;Shannon指数的数值范围分别为6.21~6.91和6.00~6.86, 最大值在南刘庄和藻苲淀, 最小值在圈头和采蒲台;Chao指数和Ace指数的数值范围分别为3 047~4 147、3 018~4 103和4 060~4 676、4 138~5 200, 最大值均在南刘庄和枣林庄, 最小值分别在采蒲台和藻苲淀. 此外, 2018年8月和2019年4月的Shannon指数、Simpson指数、Ace指数和Chao指数均无显著空间差异;而Simpson指数、Ace指数、Chao指数和OTU数量的季节差异显著(P < 0.05和P < 0.01).
2.4 微生物群落COG功能基因的时空分异特征COG功能基因相对丰度如图 5所示, 2018年8月至2019年4月其相对丰度并无显著变化, 均为氨基酸转运和代谢类的相对丰度最高(14.33%~13.68%变为14.11%~13.48%), 其次为能源生产和转换类(13.58%~13.04%变为13.13%~12.74%)、信号转导机制类(13.05%~11.70%变为12.20%~11.49%)、细胞壁/膜/包膜生物发生类(12.69%~11.85%变为12.34%~11.91%)、无机离子的转运与代谢类(10.10%~9.82%变为10.30%~9.72%)、复制、重组和修复类(10.23%~9.80%变为10.18%~9.77%)、翻译、核糖体结构和发生类(9.97%~9.37%变为9.76%~9.31%)、碳水化合物运输与代谢类(9.85%~9.23%变为10.79%~9.77%)和转录类功能基因(9.42%~8.72%变为9.94%~9.34%). 其中, 能源生产和转换类、碳水化合物运输与代谢类、转录类、细胞壁/膜/包膜生物发生类和信号传导机制类功能基因季节差异显著(P < 0.01和P < 0.05), 而8月和4月各区功能基因的相对丰度无显著空间差异.
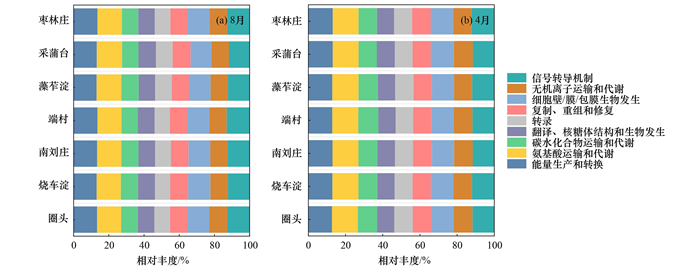
|
图 5 COG功能基因相对丰度 Fig. 5 Relative abundance of COG functional genes |
为研究白洋淀沉积物中环境因子对微生物群落多样性和COG功能基因的影响, 将筛选后的环境因子与其进行Pearson相关性分析, 结果表明(图 6), 环境因子中QNs与微生物群落多样性和功能基因存在显著相关性. 其中, 2018年8月MAR与Shannon指数呈负相关性(P < 0.05, r = -0.824);Coverage指数分别与FLE(P < 0.05, r = 0.758)、OFL(P < 0.05, r = 0.756)和CIP(P < 0.05, r = 0.758)呈正相关性[图 6(a)]. 2019年4月Simpson指数分别与CIP和QNs呈正相关性(P < 0.05, r = 0.838和P < 0.01, r = 0.948);Chao指数分别与SAR(P < 0.05, r = 0.778)和CIP(P < 0.05, r = 0.811)呈正相关性;SAR与Coverage指数呈正相关性(P < 0.01, r = 0.937)[图 6(b)]. 此外, 2018年8月至2019年4月, QNs与功能基因的相关性显著减弱. 如图 7所示, RDA分析结果表明2018年8月与2019年4月环境因子对微生物群落多样性与功能基因的总解释量分别为94.48%与98.59%, 主要影响因子分别为MAR(F = 2.8, P = 0.08)、OXO(F = 1.4, P = 0.36)和FLU(F = 1.0, P = 0.43)与CIP(F = 3.9, P = 0.06)和SAR(F = 2.3, P = 0.26).

|
(a)8月环境因子与多样性指数、OTU数量和功能基因相关性;(b)4月环境因子与多样性指数、OTU数量和功能基因相关性;C、E、G、J、K、L、M、P和T分别为:能源生产和转换类、氨基酸运输和代谢类、碳水化合物运输和代谢类、翻译、核糖体结构和生物发生类、转录类、复制、重组和修复类、细胞壁/膜/包膜生物发生类、无机离子运输和代谢类和信号转导机制类;1、2、3、4、5和6分别为:OTU数量、Shannon指数、Simpson指数、Chao指数、Coverage指数和Ace指数;*表示P < 0.05, **表示P < 0.01 图 6 白洋淀沉积物中环境因子与多样性指数、OTU数量和功能基因相关性分析 Fig. 6 Correlation analysis between environmental factors and diversity index, OTU number and functional genes in sediment of Baiyangdian Lake |
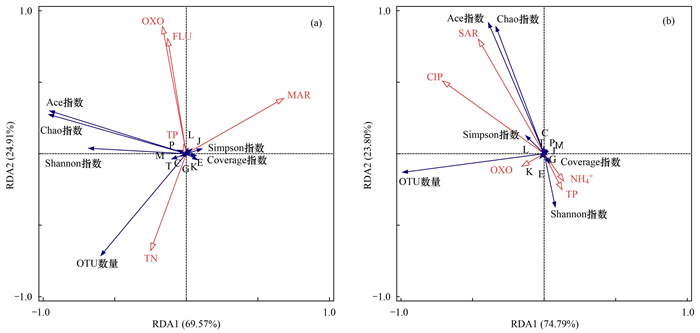
|
(a)8月环境因子与微生物群落多样性指数、OTU数量和功能基因的RDA分析;(b)4月环境因子与微生物群落多样性指数、OTU数量和功能基因的RDA分析;C、E、G、J、K、L、M、P和T分别为:能源生产和转换类、氨基酸运输和代谢类、碳水化合物运输和代谢类、翻译、核糖体结构和生物发生类、转录类、复制、重组和修复类、细胞壁/膜/包膜生物发生类、无机离子运输和代谢类和信号转导机制类;红色箭头表示环境因子, 蓝色箭头表示白洋淀沉积物中微生物群落多样性指数、OTU数量和功能基因;红色箭头长度表示影响微生物群落相关性大小, 红蓝箭头夹角余弦值表示因子对其影响程度 图 7 白洋淀沉积物中环境因子与多样性指数、OTU数量和功能基因的RDA分析 Fig. 7 RDA analysis of diversity index, OTU number, and functional genes with environmental factors in sediment of Baiyangdian Lake |
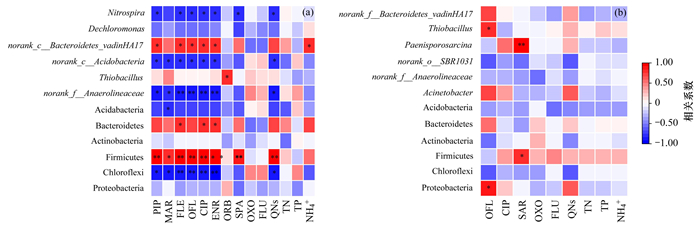
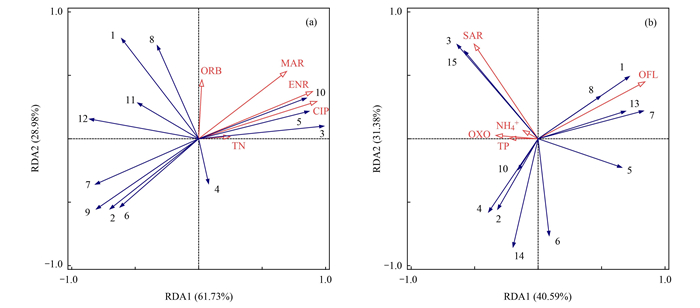
将进行VIF方差膨胀系数筛选后的环境因子与微生物群落进行Pearson相关性分析, 结果表明:2018年8月Chloroflexi、norank_ f__Anaerolineaceae、norank_c__Acidobacteria和Nitrospira丰度与CIP、ENR和MAR显著负相关(P < 0.01和P < 0.05), Firmicutes和norank_c__Bacteroidetes_vadinHA17丰度与CIP、ENR和MAR显著正相关(P < 0.01和P < 0.05), Bacteroidetes丰度与CIP和ENR显著正相关(P < 0.05)[图 8(a)];而2019年4月OFL与Proteobacteria和Thiobacillus丰度显著正相关(P < 0.05), SAR与Firmicutes和Paenisporosarcina丰度显著正相关(P < 0.05)[图 8(b)]. 加之, RDA分析结果表明(图 9), 2018年8月与2019年4月环境因子对微生物群落的总解释量分别为90.71%与71.97%, CIP(F = 6.4, P = 0.07)、ENR(F = 5.8, P = 0.05)和MAR(F = 3.1, P = 0.07)与OFL(F = 2.8, P = 0.02)和SAR(F = 3.1, P = 0.22)是影响其结构变化的主要因子.
|
(a)8月环境因子与门、属水平微生物群落相关性;(b)4月环境因子与门、属水平微生物群落相关性;*表示P < 0.05, **表示P < 0.01 图 8 白洋淀沉积物中环境因子与微生物群落组成相关性分析 Fig. 8 Correlation analysis between environmental factors and microbial community composition in sediment of Baiyangdian Lake |
|
(a)8月环境因子与微生物群落优势门、属的RDA分析;(b)4月环境因子与微生物群落优势门、属的RDA分析;1. Proteobacteria,2. Chloroflexi,3. Firmicutes,4. Actinobacteria,5. Bacteroidetes,6. Acidobacteria,7. norank_f__Anaerolineaceae,8. Thiobacillus,9. norank_c__Acidobacteria,10. norank_c__Bacteroidetes _vadinHA17,11. Dechloromonas,12. Nitrospira,13. Acinetobacter,14. norank_o__SBR1031,15. Paenisporosarcina;红色箭头为环境因子,蓝色箭头表示白洋淀沉积物中微生物群落优势门、属;红色箭头长度表示影响微生物群落相关性大小,红蓝箭头夹角余弦值表示因子对各门、属的影响程度 图 9 白洋淀沉积物中环境因子与微生物群落优势门、属的RDA分析 Fig. 9 RDA analysis of dominant phyla and genera microbial communities with environmental factors in sediment of Baiyangdian Lake |
不同湖泊沉积物中优势菌门组成如表 2所示, 不同湖泊沉积物中的微生物群落组成类型具有差异性, 这与污染物类型、水质状况和季节等多种因素有关[35]. 本研究中, 沉积物中优势菌门主要为变形菌、绿弯菌、厚壁菌、放线菌和拟杆菌, 以上沉积物细菌在有机污染物降解、碳循环和水生生态系统生物地球化学循环过程中起到重要作用[36, 37]. 此前, 研究结果表明艾比湖中变形菌为绝对优势门, 其相对丰度超过40%[38], 南海湖[39]、巴木错湖[40]、衡水湖[41]、大通湖[42]、岱海湖[43]、太湖[44]和巢湖[45]中均为变形菌占比最高, 分别为49.72%、54.00%、39.40%、39.68%、82.60%、19.07%和37.61%. 加之, 变形菌作为细菌最大的一门, 在湖泊沉积物中广泛存在, 其包括大量的病原体, 如造成胃肠道感染的幽门螺旋杆菌和大肠杆菌[46]. 其中, 大肠杆菌则会携带抗生素抗性基因, 因此废水处理设施性能有待提高以减少污水和废水排放到水生环境中[47]. 此外, 变形菌门中的根瘤菌、硫酸盐还原菌及亚硝化单胞菌等对环境中碳、硫及氮等元素代谢发挥重要作用[47].
|
|
表 2 不同湖泊沉积物中优势菌门组成 Table 2 Composition of dominant bacteria in sediment of different lakes |
就时间变化而言, 2018年8月至2019年4月, 本研究中绿弯菌和厚壁菌的相对丰度均升高. 已有研究表明, 绿弯菌作为光能自养型细菌, 不仅能在各种条件下较好生存, 其多数种类是关于有机污染物降解的细菌, 进而改变沉积物中的水溶性有机物组成, 如抗生素[39, 48]. 此外, 绿弯菌还会引起沉积物中营养物质的释放, 陈楠[49]研究表明藻华暴发前太湖中的绿弯菌相对丰度快速上升. 因此, 白洋淀中绿弯菌的相对丰度升高可能表示湖水的富营养化程度有所加剧. 厚壁菌亦作为白洋淀沉积物中的优势菌门之一, 前人的研究发现厚壁菌门具有较高的耐污能力, 成为抗生素污染下的潜在宿主, 则其丰度的升高可能与QNs等污染物的含量升高有关[50]. 此外, 从2018年8月至2019年4月, 微生物群落的丰富度和多样性整体呈升高的趋势, 这可能与8月降雨径流汇入, 导致水温升高以及氮磷等物质含量的改变有关, 使得真菌群落发生改变, 优势物种大量繁殖, 而其余物种随着种间关系以及外界因素的变化, 导致其α多样性升高[51];优势菌属发生了显著变化, 这与魏念鹏[52]的研究结果一致, 这可能与季节以及其他环境因子的变化有关.
3.2 沉积物中微生物群落的结构及功能基因的影响因素微生物群落的结构和功能不仅会受到沉积物特性的影响, 还与污染物类型等多种因素有关[53]. Shang等[54]的研究表明, 温度、pH和DO是影响呼伦湖中季节性细菌多样性的主要因素;而本研究中环境因子中的QNs则为其主要影响因素. 就微生物群落多样性和结构组成而言, 2018年8月影响微生物群落多样性和结构变化的主要因素是MAR、OXO、FLU、ENR和CIP;而2019年4月影响其变化的主要因素则为CIP、OFL和SAR. 此前, Tang等[55]的研究亦表明抗生素会影响微生物群落的多样性和结构. Li等[56]的研究发现变形菌的丰度随着磺胺甲唑含量的升高而显著降低, 而绿弯菌和厚壁菌丰度则呈现升高的趋势;QNs的添加会使群落的丰富度下降, 结构发生显著变化[57];在特定的浓度范围内, 随着土霉素浓度的增加, Shannon多样性指数显著降低[58].
就微生物群落功能而言, QNs对微生物群落功能基因的影响呈现季节性动态变化, 这一现象可能与功能基因对抗生素等污染胁迫的降解反应及毒性响应差异有关[59]. 本研究中, 2018年8月影响微生物群落功能变化的包括MAR、OXO和FLU, 而2019年4月影响其变化的是OFL和SAR. 这与之前的研究一致, 如OFL等抗生素能增加沉积物中微生物群落的反硝化功能基因的丰度[25]. 其中, 信号转导机制类基因作为微生物应对环境变化的重要基因, 与几种抗生素均具有显著相关性, 这与Zhang等[18]的研究结果一致. 这一现象可能与抗生素可以作为信号分子刺激生物膜形成、诱变和致毒, 进而使它们对微生物群落在信号转导中的功能有所贡献有关[60]. 此外, 参与复制、重组和修复类发生的功能基因与大多数抗生素呈显著正相关, 这些功能基因对细菌抵抗抗生素等抗菌剂的有害影响至关重要[61]. 加之, 重组和修复机制也会促进诱变以获得抗生素耐药性[62]. 而参与无机离子、核苷酸和氨基酸转化的转运和代谢功能基因与几种抗生素呈正相关, 表明抗生素含量的增加可能会促进微生物对无机离子、核苷酸和氨基酸转化的转运和代谢. 总体而言, QNs是影响白洋淀沉积物中微生物群落结构及功能变化的主要因素.
4 结论(1)白洋淀沉积物中总喹诺酮类(QNs)抗生素含量呈现显著的季节差异.
(2)白洋淀沉积物中微生物群落优势菌属季节变化显著, 且空间变化差异显著.
(3)白洋淀微生物群落多样性指数和功能基因的季节差异显著.
(4)白洋淀沉积物中抗生素含量会影响微生物群落的结构和功能基因. 因此, 需关注湖泊抗生素污染对微生物群落的影响.
| [1] | Ju H Y, Liu L, Liu X M, et al. A comprehensive study of the source, occurrence, and spatio-seasonal dynamics of 12 target antibiotics and their potential risks in a cold semi-arid catchment[J]. Water Research, 2023, 229. DOI:10.1016/j.watres.2022.119433 |
| [2] |
刘高燕. 抗生素残留对环境影响的调查研究[J]. 化工中间体, 2015, 11(1): 13-15. Liu G Y. Investigations on the impact of antibiotic residues on the environment[J]. Chemical Intermediates, 2015, 11(1): 13-15. |
| [3] | Wang X L, Jiao Y N, Wang G L, et al. Occurrence of quinolones in cultured fish from Shandong Province, China and their health risk assessment[J]. Marine Pollution Bulletin, 2022, 180. DOI:10.1016/j.marpolbul.2022.113777 |
| [4] | Zou M Y, Tian W J, Zhao J, et al. Quinolone antibiotics in sewage treatment plants with activated sludge treatment processes: a review on source, concentration and removal[J]. Process Safety and Environmental Protection, 2022, 160: 116-129. DOI:10.1016/j.psep.2022.02.013 |
| [5] | Chen Y L, Jiang C X, Wang Y L, et al. Sources, environmental fate, and ecological risks of antibiotics in sediments of Asia's longest river: a whole-basin investigation[J]. Environmental Science & Technology, 2022, 56(20): 14439-14451. |
| [6] | Tiseo K, Huber L, Gilbert M, et al. Global trends in antimicrobial use in food animals from 2017 to 2030[J]. Antibiotics, 2020, 9(12). DOI:10.3390/antibiotics9120918 |
| [7] | Carvalho I T, Santos L. Antibiotics in the aquatic environments: a review of the European scenario[J]. Environment International, 2016, 94: 736-757. DOI:10.1016/j.envint.2016.06.025 |
| [8] | Wang Z, Yang Y Y, Gomes M P, et al. Editorial: antibiotics and antibiotic resistance genes in waters: pollution, risks, and control[J]. Frontiers in Environmental Science, 2022, 10. DOI:10.3389/fenvs.2022.967118 |
| [9] | Danner M C, Robertson A, Behrends V, et al. Antibiotic pollution in surface fresh waters: occurrence and effects[J]. Science of the Total Environment, 2019, 664: 793-804. DOI:10.1016/j.scitotenv.2019.01.406 |
| [10] | Ben Y J, Fu C X, Hu M, et al. Human health risk assessment of antibiotic resistance associated with antibiotic residues in the environment: a review[J]. Environmental Research, 2019, 169: 483-493. DOI:10.1016/j.envres.2018.11.040 |
| [11] | Zhou Q Q, Liu G J, Arif M, et al. Occurrence and risk assessment of antibiotics in the surface water of Chaohu Lake and its tributaries in China[J]. Science of the Total Environment, 2022, 807. DOI:10.1016/j.scitotenv.2021.151040 |
| [12] |
吴天宇, 李江, 杨爱江, 等. 赤水河流域水体抗生素污染特征及风险评价[J]. 环境科学, 2022, 43(1): 210-219. Wu T Y, Li J, Yang A J, et al. Characteristics and risk assessment of antibiotic contamination in Chishui River Basin, Guizhou Province, China[J]. Environmental Science, 2022, 43(1): 210-219. |
| [13] | Liu D, Xu Y Y, Junaid M, et al. Distribution, transfer, ecological and human health risks of antibiotics in bay ecosystems[J]. Environment International, 2022, 158. DOI:10.1016/j.envint.2021.106949 |
| [14] | Li F F, Chen L J, Chen W D, et al. Antibiotics in coastal water and sediments of the East China Sea: distribution, ecological risk assessment and indicators screening[J]. Marine Pollution Bulletin, 2020, 151. DOI:10.1016/j.marpolbul.2019.110810 |
| [15] | Yang Y Y, Song W J, Lin H, et al. Antibiotics and antibiotic resistance genes in global lakes: a review and meta-analysis[J]. Environment International, 2018, 116: 60-73. DOI:10.1016/j.envint.2018.04.011 |
| [16] | Jamieson A J, Malkocs T, Piertney S B, et al. Bioaccumulation of persistent organic pollutants in the deepest ocean fauna[J]. Nature Ecology & Evolution, 2017, 1(3). DOI:10.1038/s41559-016-0051 |
| [17] |
王永强. 艾比湖流域典型抗生素、抗性基因及微生物群落的分布特征[D]. 济南: 山东师范大学, 2020. Wang Y Q. Distribution characteristics of typical antibiotics, antibiotic resistance genes and microbial community in Ebinur Lake Basin[D]. Ji'nan: Shandong Normal University, 2020. |
| [18] | Zhang L L, Zhang C, Lian K T, et al. Effects of chronic exposure of antibiotics on microbial community structure and functions in hyporheic zone sediments[J]. Journal of Hazardous Materials, 2021, 416. DOI:10.1016/j.jhazmat.2021.126141 |
| [19] | Labbate M, Seymour J R, Lauro F, et al. Editorial: anthropogenic impacts on the microbial ecology and function of aquatic environments[J]. Frontiers in Microbiology, 2016, 7. DOI:10.3389/fmicb.2016.01044 |
| [20] | Qiu L L, Daniell T J, Banwart S A, et al. Insights into the mechanism of the interference of sulfadiazine on soil microbial community and function[J]. Journal of Hazardous Materials, 2021, 419. DOI:10.1016/j.jhazmat.2021.126388 |
| [21] |
喻娇, 冯乃宪, 喻乐意, 等. 土壤环境中典型抗生素残留及其与微生物互作效应研究进展[J]. 微生物学杂志, 2017, 37(6): 105-113. Yu J, Feng N X, Yu L Y, et al. Advances in typical antibiotics residues and their mutual effects with microorganisms in soil environment[J]. Journal of Microbiology, 2017, 37(6): 105-113. |
| [22] | Feng Y, Hu J C, Chen Y S, et al. Ecological effects of antibiotics on aquaculture ecosystems based on microbial community in sediments[J]. Ocean & Coastal Management, 2022, 224. DOI:10.1016/j.ocecoaman.2022.106173 |
| [23] | Alexandrino D A M, Mucha A P, Almeida C M R, et al. Biodegradation of the veterinary antibiotics enrofloxacin and ceftiofur and associated microbial community dynamics[J]. Science of the Total Environment, 2017, 581: 359-368. |
| [24] | Zhou Z G, Zhang Z Y, Feng L, et al. Adverse effects of levofloxacin and oxytetracycline on aquatic microbial communities[J]. Science of the Total Environment, 2020, 734. DOI:10.1016/j.scitotenv.2020.139499 |
| [25] | Zhang R J, Xu X M, Jia D T, et al. Sediments alleviate the inhibition effects of antibiotics on denitrification: functional gene, microbial community, and antibiotic resistance gene analysis[J]. Science of the Total Environment, 2022, 804. DOI:10.1016/j.scitotenv.2021.150092 |
| [26] | Zhang L L, Zhang A T, Yang Y T, et al. Structure and function response of bacterial communities towards antibiotic contamination in hyporheic zone sediments[J]. Chemosphere, 2022, 309. DOI:10.1016/j.chemosphere.2022.136606 |
| [27] | Bai J H, Guan Y, Liu P P, et al. Assessing the safe operating space of aquatic macrophyte biomass to control the terrestrialization of a grass-type shallow lake in China[J]. Journal of Environmental Management, 2020, 266. DOI:10.1016/j.jenvman.2020.110479 |
| [28] |
申立娜, 张璐璐, 秦珊, 等. 白洋淀喹诺酮类抗生素污染特征及其与环境因子相关性研究[J]. 环境科学学报, 2019, 39(11): 3888-3897. Shen L N, Zhang L L, Qin S, et al. The occurrence and distribution of quinolones (QNs) and correlation analysis between QNs and physical-chemical parameters in Baiyangdian lake, North China[J]. Acta Scientiae Circumstantiae, 2019, 39(11): 3888-3897. |
| [29] | Yang L, Wang T Y, Zhou Y Q, et al. Contamination, source and potential risks of pharmaceuticals and personal products (PPCPs) in Baiyangdian Basin, an intensive human intervention area, China[J]. Science of the Total Environment, 2021, 760. DOI:10.1016/j.scitotenv.2020.144080 |
| [30] | Zhang L, Bai J H, Zhang K G, et al. Occurrence, bioaccumulation and ecological risks of antibiotics in the water-plant-sediment systems in different functional areas of the largest shallow lake in North China: impacts of river input and historical agricultural activities[J]. Science of the Total Environment, 2023, 857. DOI:10.1016/j.scitotenv.2022.159260 |
| [31] |
童帮会. 淀山湖典型抗生素污染特征、来源及风险评价[D]. 上海: 华东师范大学, 2019. Tong B H. Pollution characteristics, sources and risk assessment of typical antibiotics in Dianshan Lake of Shanghai[D]. Shanghai: East China Normal University, 2019. |
| [32] |
剧泽佳, 付雨, 赵鑫宇, 等. 喹诺酮类抗生素在城市典型水环境中的分配系数及其主要环境影响因子[J]. 环境科学, 2022, 43(9): 4543-4555. Ju Z J, Fu Y, Zhao X Y, et al. Distribution coefficient of QNs in urban typical water and its main environmental influencing factors[J]. Environmental Science, 2022, 43(9): 4543-4555. |
| [33] |
赵鑫宇, 剧泽佳, 陈慧, 等. 石家庄市土壤中喹诺酮类抗生素空间分布特征及其与微生物群落相关性[J]. 环境科学, 2022, 43(9): 4684-4696. Zhao X Y, Ju Z J, Chen H, et al. Spatial distribution of quinolone antibiotics and its correlation relationship with microbial community in soil of Shijiazhuang City[J]. Environmental Science, 2022, 43(9): 4684-4696. |
| [34] | Liu Y Y, Hua Z L, Lu Y, et al. Quinolone distribution, trophodynamics, and human exposure risk in a transit-station lake for water diversion in east China[J]. Environmental Pollution, 2022, 311. DOI:10.1016/j.envpol.2022.119985 |
| [35] |
朱婷婷, 田从魁. 水库底泥中微生物多样性及其与环境因子相关性分析[J]. 北京大学学报(自然科学版), 2018, 54(3): 625-632. Zhu T T, Tian C K. Analysis on microbial diversity in the sediments and its relationship with environmental factors in a reservoir[J]. Acta Scientiarum Naturalium Universitatis Pekinensis, 2018, 54(3): 625-632. |
| [36] |
阴星望, 田伟, 丁一, 等. 丹江口库区表层沉积物细菌多样性及功能预测分析[J]. 湖泊科学, 2018, 30(4): 1052-1063. Yin X W, Tian W, Ding Y, et al. Composition and predictive functional analysis of bacterial communities in surface sediments of the Danjiangkou Reservoir[J]. Journal of Lake Sciences, 2018, 30(4): 1052-1063. |
| [37] |
牛凤霞, 吉芳英, 赵艮, 等. 龙景湖沉积物的细菌群落垂向分布特征[J]. 中国环境科学, 2017, 37(6): 2322-2331. Niu F X, Ji F Y, Zhao G, et al. Vertical distribution of bacterial communities in sediments of Longjing Lake[J]. China Environmental Science, 2017, 37(6): 2322-2331. |
| [38] | Wang Y Q, Lu S Y, Liu X H, et al. Profiles of antibiotic resistance genes in an inland salt-lake Ebinur Lake, Xinjiang, China: the relationship with antibiotics, environmental factors, and microbial communities[J]. Ecotoxicology and Environmental Safety, 2021, 221. DOI:10.1016/j.ecoenv.2021.112427 |
| [39] |
赵忠, 滕飞, 李卫平, 等. 包头南海湖不同湖区春季沉积物细菌群落结构[J]. 灌溉排水学报, 2019, 38(6): 99-104. Zhao Z, Teng F, Li W P, et al. Spatial variation of the sediment microbial communities in Baotou Nanhai Lake in spring[J]. Journal of Irrigation and Drainage, 2019, 38(6): 99-104. |
| [40] | Wei C, Sun D, Yuan W L, et al. Metagenomics revealing molecular profiles of microbial community structure and metabolic capacity in Bamucuo lake, Tibet[J]. Environmental Research, 2023, 217. DOI:10.1016/j.envres.2022.114847 |
| [41] |
曹洋, 孙鹤铭, 刘利, 等. 冬季衡水湖沉积物微生物群落结构特征及影响因素[J]. 环境工程技术学报, 2023, 13(1): 154-163. Cao Y, Sun H M, Liu L, et al. Microbial community structure characteristics and influencing factors in sediments of Hengshui Lake in winter[J]. Journal of Environmental Engineering Technology, 2023, 13(1): 154-163. |
| [42] | Chao C X, Wang L G, Li Y, et al. Response of sediment and water microbial communities to submerged vegetations restoration in a shallow eutrophic lake[J]. Science of the Total Environment, 2021, 801. DOI:10.1016/j.scitotenv.2021.149701 |
| [43] | Du C L, Yang F, Li X G, et al. Metagenomic analysis of microbial community structure and distribution of resistance genes in Daihai Lake, China[J]. Environmental Pollution, 2022, 302. DOI:10.1016/j.envpol.2022.119065 |
| [44] | Li L, Li F B, Deng M, et al. Microplastics distribution characteristics in typical inflow rivers of Taihu lake: linking to nitrous oxide emission and microbial analysis[J]. Water Research, 2022, 225. DOI:10.1016/j.watres.2022.119117 |
| [45] | Yang C M, Sun J L, Chen Y Y, et al. Linkage between water soluble organic matter and bacterial community in sediment from a shallow, eutrophic lake, Lake Chaohu, China[J]. Journal of Environmental Sciences, 2020, 98: 39-46. |
| [46] | Divya S P, Hatha A A M. Screening of tropical estuarine water in south-west coast of India reveals emergence of ARGs-harboring hypervirulent Escherichia coli of global significance[J]. International Journal of Hygiene and Environmental Health, 2019, 222(2): 235-248. |
| [47] |
李依微. 练江流域抗生素时空分布及其与微生物群落相关性[D]. 汕头: 汕头大学, 2021. Li Y W. Temporal and spatial distribution of antibiotics and their correlation with microbial communities in Lianjiang River Basin[D]. Shantou: Shantou University, 2021. |
| [48] | de Moraes P C, Franco D C, Pellizari V H, et al. Effect of plankton-derived organic matter on the microbial community of coastal marine sediments[J]. Journal of Experimental Marine Biology and Ecology, 2014, 461: 257-266. |
| [49] |
陈楠. 太湖沉积物微生物群落组成与物质循环及藻华爆发的相关性[D]. 北京: 中国农业大学, 2015. Chen N. Relationship between microbial community structure and nutrient cycles and algal bloom in Taihu Lake[D]. Beijing: China Agricultural University, 2015. |
| [50] |
李轶, 胡童, 王琳琼, 等. 环丙沙星和铜复合污染下河流底质微生物群落与抗生素抗性基因的交互关系[J]. 河海大学学报(自然科学版), 2022, 50(6): 75-84. Li Y, Hu T, Wang L Q, et al. Interaction relationship between microbial communities and antibiotic resistance genes under ciprofloxacin and copper co-contamination in river sediments[J]. Journal of Hohai University (Natural Sciences), 2022, 50(6): 75-84. |
| [51] |
方康. 无定河沉积泥沙中微生物群落特征及其影响因素研究[D]. 西安: 西安理工大学, 2022. Fang K. Study on the characteristics of microbial community and its influencing factors in sedimentary sediments of Wuding River Major: soil and water conservation and desertification control[D]. Xi'an: Xi'an University of Technology, 2022. |
| [52] |
魏念鹏. 镜泊湖水体中微生物多样性与环境因子的响应关系研究[D]. 哈尔滨: 哈尔滨商业大学, 2022. Wei N P. Study on the relationship between microbial diversity and environmental factors in JingPo Lake[D]. Harbin: Harbin University of Commerce, 2022. |
| [53] | Bardgett R D. The biology of soil: a community and ecosystem approach[M]. New York: Oxford University Press, 2005. |
| [54] | Shang Y Q, Wu X Y, Wang X B, et al. Factors affecting seasonal variation of microbial community structure in Hulun Lake, China[J]. Science of the Total Environment, 2022, 805. DOI:10.1016/j.scitotenv.2021.150294 |
| [55] | Tang T T, Liu M, Du Y, et al. Mechanism of action of single and mixed antibiotics during anaerobic digestion of swine wastewater: microbial functional diversity and gene expression analysis[J]. Environmental Research, 2023, 219. DOI:10.1016/j.envres.2022.115119 |
| [56] | Li Z L, Cheng R, Chen F, et al. Selective stress of antibiotics on microbial denitrification: inhibitory effects, dynamics of microbial community structure and function[J]. Journal of Hazardous Materials, 2021, 405. DOI:10.1016/j.jhazmat.2020.124366 |
| [57] | Meng F G, Gao G H, Yang T T, et al. Effects of fluoroquinolone antibiotics on reactor performance and microbial community structure of a membrane bioreactor[J]. Chemical Engineering Journal, 2015, 280: 448-458. |
| [58] | Kong W D, Zhu Y G, Fu B J, et al. The veterinary antibiotic oxytetracycline and Cu influence functional diversity of the soil microbial community[J]. Environmental Pollution, 2006, 143(1): 129-137. |
| [59] |
余诗航, 刘珈佑, 李占杰, 等. 高通量测序揭示某石化固废填埋场污染对土壤微生物群落结构和功能的影响[J]. 北京师范大学学报(自然科学版), 2022, 58(2): 277-285. Yu S H, Liu J Y, Li Z J, et al. Influence of pollutants on soil microbial community structure and function at a petrochemical landfill site: a high-throughput sequencing study[J]. Journal of Beijing Normal University (Natural Science), 2022, 58(2): 277-285. |
| [60] | Bleich R, Watrous J D, Dorrestein P C, et al. Thiopeptide antibiotics stimulate biofilm formation in Bacillus subtilis[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(10): 3086-3091. |
| [61] | Eloe E A, Fadrosh D W, Novotny M, et al. Going deeper: metagenome of a hadopelagic microbial community[J]. PLoS One, 2011, 6(5). DOI:10.1371/journal.pone.0020388 |
| [62] | Dos Vultos T, Mestre O, Tonjum T, et al. DNA repair in Mycobacterium tuberculosis revisited[J]. FEMS Microbiology Reviews, 2009, 33(3): 471-487. |
 2024, Vol. 45
2024, Vol. 45


