2. 南水北调中线水源区流域生态安全高等学校学科创新引智基地, 南阳 473061
2. Overseas Expertise Introduction Center for Discipline Innovation of Watershed Ecological Security in the Water Source Area of the Middle Route of South-to-North Water Diversion, Nanyang 473061, China
丹江口水库作为南水北调中线工程水源地, 截至2023年3月30日, 已累计向受水区调水550亿m3, 直接受益人口超8 500万, 取得了显著的生态和经济效应. 丹江口水库水质状况直接关系到南水北调中线工程安全运行, 水库水质持续稳定在Ⅱ类标准及以上. 但其面临着较大的氮、磷负荷, 其中总氮浓度相对较高, 是影响水质的最为主要因素[1, 2].
浮游细菌作为水生生态系统的重要组成, 通过固氮作用、硝化作用、反硝化作用、氮同化还原及异化还原作用等以及磷活化、磷吸收等驱动水体中氮、磷生物化学循环[3 ~ 5]. 针对丹江口水库浮游细菌群落组成及其驱动的氮磷元素循环, 本课题组基于扩增子测序的PICRUSt功能预测进行了研究, 发现主要涉及K02588 nifH、K10535 hao、K00368 nirK等氮循环功能基因和K00655 plsC、K01507 ppa、K02036 pstB等磷循环功能基因[6 ~ 9]. 基于扩增子测序的功能预算成本较小, 但它在准确预测微生物组功能方面存在着较大的局限性. 宏基因组学能从基因组的水平和微生物群落的层面上分析微生物的组成和功能, 在水库、湖泊、海洋和河流等不同水体生境得到广泛的应用, 为揭示浮游细菌驱动的水体氮、磷元素循环提供了有效方法[10 ~ 15].
丹江口水库作为人工加高的深水型水库, 平均水深30 m, 水库垂直方向上水温、光照、溶解氧、氮、磷营养盐浓度以及其流速、流量等具有显著差异, 对浮游细菌群落组成和功能可能产生显著影响. 目前, 对丹江口水库表层浮游细菌群落组成和功能已经开展相关研究[7, 12, 16], 但垂向环境因子变化对细菌群落结构以及氮和磷元素循环功能基因的影响过程尚不明确, 有必要开展进一步研究加以阐明.
本研究选取丹江口库区库心和渠首等5个生态点位, 采用宏基因组学研究表层、中层和底层垂直分布浮游细菌群落组成, 分析预测浮游细菌氮和磷循环相关基因表达情况, 通过揭示丹江口水库浮游细菌垂直分布状况及其驱动的氮磷元素循环, 以期为丹江口水库水环境保护提供参考依据.
1 材料与方法 1.1 样品采集根据丹江口水库所处的地理位置、人类活动和调水工程影响, 共选取5个生态位点:丹库库区的库心(KX)、南水北调中线工程的取水口上游100 m处的渠首(QS)、丹江支流和灌河支流在丹库交汇处的黑鸡嘴(HJZ)、轮渡及渔船停靠的码头宋岗(SG)及丹江口水库丹库和汉库的交界处台子山(TZS), 具体位置信息见图 1[7, 9, 16]. 库心、渠首、黑鸡嘴、宋岗和台子山实际水深分别为53、19、43、27和26 m, 将不同采样点划分为表层(B)、中层(Z)和底层(D)这3层. 于2022年3月进行样品采集, 采用柱状采水器采集水样.
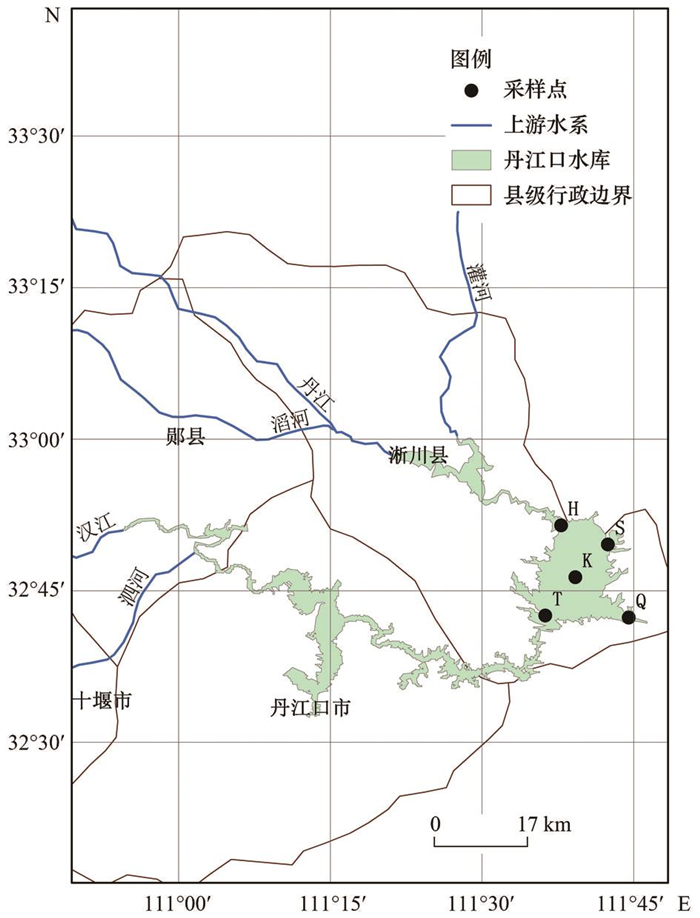
|
图 1 采样点分布示意 Fig. 1 Location map of the sample sites of Danjiangkou Reservoir |
参照《水和废水监测分析方法》(第四版)[17]测定水样pH值、水温(T)、总氮(TN)、氨氮(NH4+-N)、总磷(TP)、电导率(EC)、溶解氧(DO)、高锰酸盐指数以及叶绿素a含量(Chla)等理化指标.
1.3 浮游细菌总DNA提取1.5 L表层、中层和底层水样经0.22 µm无菌微孔滤膜过滤, 放置于无菌离心管中, 采用Omega Water DNA Kit(Omega, USA)提取浮游细菌总DNA. 将提取得到的浮游细菌总DNA通过微量紫外分光光度计测定DNA浓度和纯度.
1.4 宏基因组测序分析采用HiSeq2000测序平台对水样进行宏基因组学测序. 将测序平台上得到的原始序列用Fastp(v0.20.0)软件对其进行质控, 剪切掉数据中的低质量reads, 获得高质量序列, 采用MEGAHIT v1.1.2软件进行序列组装. 对拼接结果进行ORF基因预测, 对预测结果进行聚类, 建立非冗余基因集. 使用DIAMOND(https://github.com/bbuchfink/diamond)将非冗余基因集序列与egg NOG数据库进行比对(参数:blastp;E-value ≤1e-5), 获得基因对应的直系同源蛋白簇(clusters of orthologous groups of proteins, COG), 使用COG对应的基因丰度总和计算其丰度. 将非冗余基因集与KEGG基因(kyoto encyclopedia of genes and genomes, http://www.genome.jp/kegg/)数据库进行比对, 得出各处理组在各层级中相对丰度的分析结果. 使用Diamond软件将非冗余基因集的氨基酸序列与NR数据库进行比对后获得对应物种注释, 最后将每一物种所对应的基因丰度总和用于计算该物种的丰度. 使用CAZy数据库的对应工具hmmscan将非冗余基因集与CAZy数据库进行比对, 比对参数设置期望值为1e-5, 获得基因对应的碳水化合物活性酶注释信息, 然后使用碳水化合物活性酶对应的基因丰度总和计算该碳水化合物活性酶的丰度. 宏基因组高通量测序数据基于KEGG数据库功能注释结果中的氮、磷标记基因(KEGG Ontology, KO)信息提取氮循环和磷循环标记基因集, 基于RPKM丰度计算方法, 进行功能组成分析, 差异分析和相关性分析.
2 结果与分析 2.1 水体理化因子垂直分布特征丹江口水库不同分层水体理化性质指标测定结果如表 1所示, T、ORP、DO、EC、总溶解性固体(TDS)和Chla等指标表现出明显的垂直分布特征. T在不同样点间的范围为10.5~18.3℃, ρ(DO)和ρ(Chla)在不同样点间的范围分别为8.9~12.4 mg·L-1和0.89~8.58 mg·m-3, 以上指标随着水深的增加而降低. ORP在不同样点间的范围为212~281 mV, EC在不同样点间的范围为251~271 μS·cm-1, ρ(TDS)在不同样点间的范围为169~176 mg·L-1, 以上指标随着水深的增加而增加. 丹江口水库ρ(TN)相对较高, 在不同样点间的范围为0.81~2.82 mg·L-1, ρ(NH4+-N)、ρ(NO3--N)、ρ(TP)和ρ(PO43--P)范围分别为0.06~1.46、0.65~1.64、0.01~0.06和0.01~0.02 mg·L-1.
|
|
表 1 各样品主要理化参数 Table 1 Main physical and chemical properties of different samples |
2.2 群落组成及其影响因素分析
宏基因组分析表明, 丹江口水库不同分层样品细菌门水平上有:变形菌门(Proteobacteria, 31.28%~55.25%, 相对丰度, 下同)、放线菌门(Actinobacteria, 14.93%~20.19%)、浮霉菌门(Planctomycetes, 2.43%~7.05%)、拟杆菌门(Bacteroidetes, 3.03%~7.00%)、疣微菌门(Verrucomicrobia, 1.72%~7.75%)和绿弯菌门(Chloroflexi, 1.16%~11.40%), 以上优势种群的序列总和占全部序列的75.35%~84.58%(图 2).
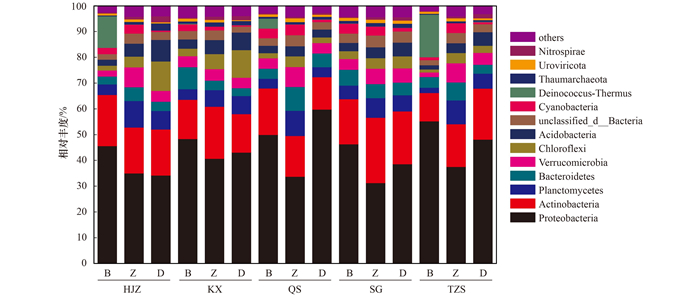
|
图 2 不同样品浮游细菌门水平上物种相对丰度分布 Fig. 2 Relative read abundance of bacterioplankton community structures at phylum level in different samples |
属水平上主要由Limnohabitans(1.00%~8.95%)、奇异球菌属(Deinococcus, 0.02%~12.22%)、Candidatus_Fonsibacter(0.94%~2.35%)、棍状杆菌属(Clavibacter, 0.56%~1.94%)、莱茵海默氏菌属(Rheinheimera, 0.07%~1.39%)、不动杆菌属(Acinetobacter, 0.03%~4.88%)、疣微菌属(Pedosphaera, 0.18%~1.00%)、Nitrospira(0.24%~1.74%)和多核杆菌属(Polynucleobacter, 0.29%~1.90%)等优势属组成(图 3).
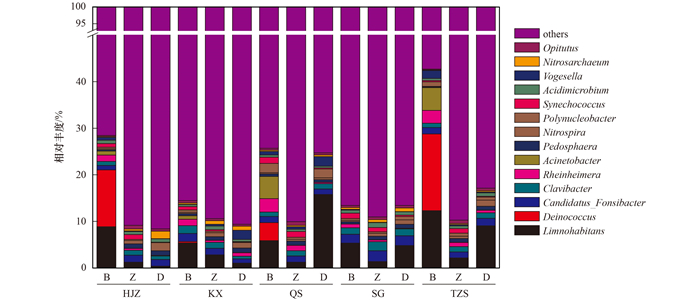
|
图 3 不同样品浮游细菌属水平上物种相对丰度分布 Fig. 3 Relative read abundance of bacterioplankton community structures at genus level in different samples |
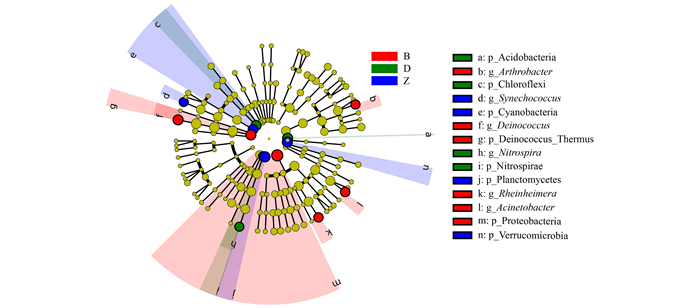
浮游细菌群落的NMDS分析结果如图 4所示, 丹江口水库表层样品分布于NMDS图左侧, 中层样品分布NMDS图中部, 底层样品分布NMDS图中右侧. 以上分析表明样品不同深度来源的浮游细菌群落结构具有差异. 后续ANOSIM分析结果表明, 表层与中层和底层, 中层和底层的差异P值分别为0.004、0.016和0.011, 以上差异达到显著水平. LEfSe分析表明, 在LDA > 3.5的水平上, 变形菌门和异常球菌-栖热菌门(Deinococcus-Thermus)在表层, 浮霉菌门、疣微菌门和蓝细菌门(Cyanobacteria)在中层, 绿弯菌门、酸杆菌门(Acidobacteria)和Nitrospirae在底层样品中存在显著差异. 不动杆菌属、节杆菌属(Arthrobacter)、奇异球菌属和莱茵海默氏菌属在表层, 聚球藻属(Synechococcus)在中层, Nitrospira在底层样品中存在显著差异(图 5). 将方差膨胀因子(variance inflation factor, VIF) > 10的变量剔除后与属水平上浮游细菌群落进行RDA分析, 结果表明T(P = 0.028)、ORP(P = 0.006)、DO(P = 0.035)和Chla(P = 0.001)是影响浮游细菌群落组成的主要因素.
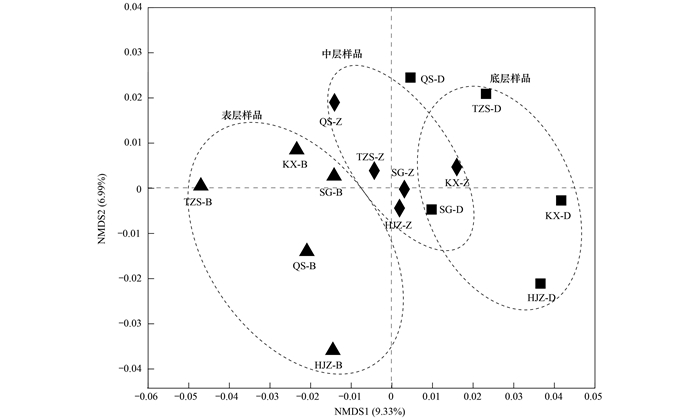
|
图 4 浮游细菌群落的NMDS分析 Fig. 4 NMDS analysis of bacterioplankton community |
|
图 5 不同样品差异细菌分布 Fig. 5 Taxon with statistical differences between different bacterial communities |
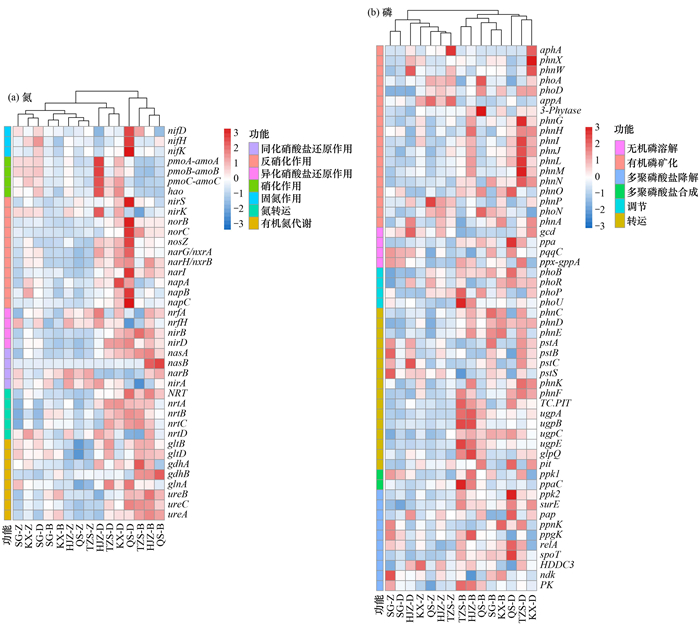
丹江口水库不同深度浮游细菌主要参与:氮循环生物固氮过程(nitrogen fixation), 以及生物脱氮过程中的硝化作用(nitrification)、反硝化作用(denitrification)、异化硝酸盐还原作用(dissimilatory nitrogen reduction, DNRA)、同化硝酸盐还原作用(assimilatory nitrogen reduction, ANRA)、氮转运(nitrogen transport)和有机氮代谢(organic N metabolism)这7个主要途径, 涉及gltB、glnA、gltD、gdhA、NRT、ureC、narG/nxrA、nirB、nasA nrtA、nrtC和nrtB等39个氮循环功能基因[图 6(a)]. 宏基因组数据中筛选到的氮循环标记基因集, 基于绝对丰度数据使用z-score进行丰度标准化处理[图 6(a)]. 通过聚类可以发现[图 6(a)], 主要分为3大类, 表层样品的QS-B、HJZ-B、TZS-B和底层样品的QS-D聚集为一类, 底层样品的HJZ-D、TZS-D和KX-D聚集为一类, 其余样品聚为一类. 从热图 6(a)可知, 表层样品QS-B、HJZ-B、TZS-B和底层样品QS-D、HJZ-D、TZS-D、KX-D基因丰度高于另外一大组, 整体趋势为表层和底层氮循环功能基因丰度高于中层样品. 其中表层样品QS-B、HJZ-B和TZS-B在有机氮代谢中gltB、gltD、gdhA、gdhB、glnA、ureB、ureC和ureA, 同化硝酸盐还原作用中nasA和nasB, 异化硝酸盐还原作用nirB和nirD基因丰度高于其他样品, 硝化作用pmoA-amoA、pmoB-amoB、pmoC-amoC和hao基因丰度低于其他样品. 底层样品QS-D、HJZ-D、TZS-D和KX-D在硝化作用中pmoA-amoA、pmoB-amoB、pmoC-amoC和hao, 反硝化作用中nirS、nirK、norB、norC、nosZ和narG/nxrA等, 氮转运中NRT、nrtA、nrtB、nrtC和nrtD基因丰度高于中层样品, 其中QS-D样品生物固氮过程中nifD、nifH和nifK, 反硝化作用nirS、norB、norC、nosZ、narG/nxrA、narH/nxrB、narI、napB和napC基因丰度为所有样品中最高;TZS-D样品硝化作用pmoA-amoA、pmoB-amoB、pmoC-amoC和hao, 反硝化作用nirK基因丰度为所有样品中最高. 以上结果表明不同深度是影响氮循环功能基因组成和丰度的主要因素, 表层和底层氮循环功能基因组成有所差异, 整体丰度高于中层样品.
|
图 6 氮、磷循环功能基因Heatmap图 Fig. 6 Heatmap showing the predicted functional genes related to the nitrogen and phosphorus cycles |
丹江口水库不同深度浮游细菌主要参与磷循环有机磷矿化(organic P mineralization)、无机磷溶解(inorganic P solubilization)、调节(regulatory)、转运(transporters)、多聚磷酸盐合成(polyphosphate synthesis)和多聚磷酸盐降解(polyphosphate degradation)等6个主要途径, 涉及pstS、ppx-gppA、glpQ、ppk1、ppnK、relA、PK、pstA、pstC、pstB和phoU等54个磷循环功能基因[图 6(b)]. 聚类分析结果与氮循环结果类似, 聚集为3大类, 底层样品QS-D、TZS-D、KX-D聚集为一类, 表层样品QS-B、TZS-B、HJZ-B、KX-B和SG-B聚集为一类, 其余样品聚为一类. 从热图 6(b)可知, 表层所有样品和底层样品QS-D、TZS-D和KX-D基因丰度高于另外一大组, 整体趋势为表层和底层磷循环功能基因丰度高于中层样品. 其中底层样品QS-D、TZS-D和KX-D在有机磷矿化中phnG、phnH、phnI、phnJ、phnL、phnM和phnN, 转运中phnK和phnF, 多聚磷酸盐降解中ppk2、surE和pap基因丰度高于其他样品. 表层样品在有机磷矿化中phnG、phnH、phnI、phnJ、phnL、phnM、phnN和phnO, 转运中phnC、phnD、phnE、phnF、TC.PIT、ugpA、ugpB、ugpC、ugpE和glpQ, 调节中phoB, 多聚磷酸盐降解中ppk2、surE、relA和spoT基因丰度高于中层样品. 以上结果表明, 不同深度是影响磷循环功能基因组成和丰度的主要因素, 表层和底层磷循环功能基因组成有所差异, 整体丰度高于中层样品.
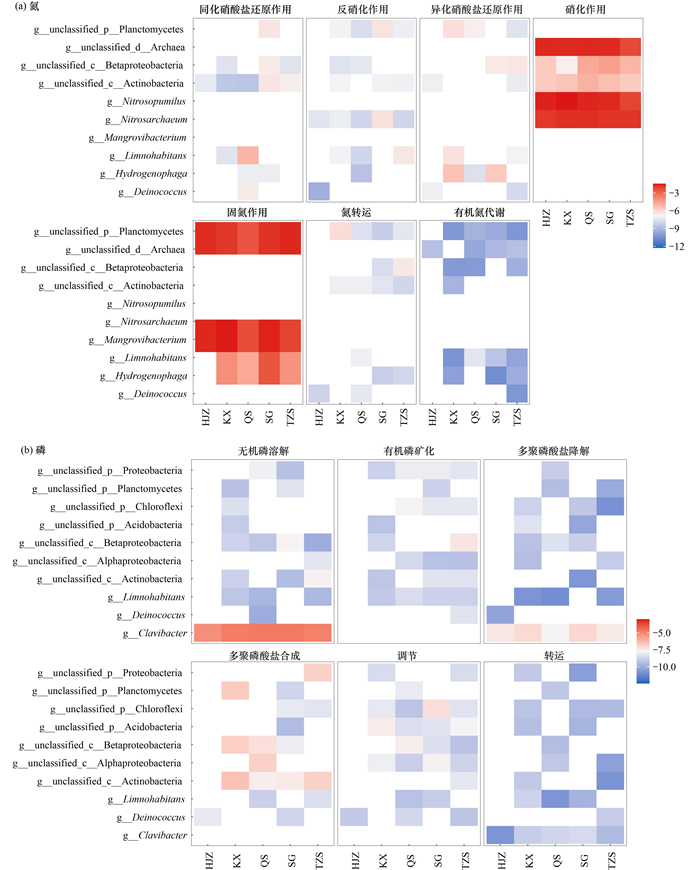
2.4 与氮、磷循环相关的关键细菌物种组成物种与氮、磷循环功能贡献度分析结果如图 7所示. 在属水平上共筛选出奇异球菌属(Deinococcus)、嗜氢菌属(Hydrogenophaga)、Limnohabitans、Nitrosarchaeum和亚硝化侏儒菌属(Nitrosopumilus)等10种主要功能菌群在氮循环过程中贡献比例高[图 7(a)]. 其中Nitrosarchaeum、亚硝化侏儒菌属参与硝化作用;Nitrosarchaeum和红树林杆菌属(Mangrovibacterium)参与固氮过程, 嗜氢菌属(Hydrogenophaga)、Limnohabitans参与除黑鸡嘴样点外的固氮过程;Nitrosarchaeum参与反硝化作用;Limnohabitans、嗜氢菌属和奇异球菌属参与部分样点的固氮过程、异化硝酸盐还原作用、同化硝酸盐还原作用、氮转运和有机氮代谢.
|
图 7 物种与氮、磷循环功能贡献度分析 Fig. 7 Contribution of core functional microbes to the nitrogen and phosphorus cycle |
在属水平上共筛选出Limnohabitans、奇异球菌属(Deinococcus)和棍状杆菌属(Clavibacter)等10种主要功能菌群在磷循环过程中贡献比例高[图 7(b)]. 其中棍状杆菌属参与无机磷溶解、多聚磷酸盐降解和转运等磷代谢过程, Limnohabitans参与除黑鸡嘴位点外的有机磷矿化和无机磷溶解等磷代谢过程, 奇异球菌属参与黑鸡嘴样点多聚磷酸盐合成、多聚磷酸盐降解和转运等磷代谢过程.
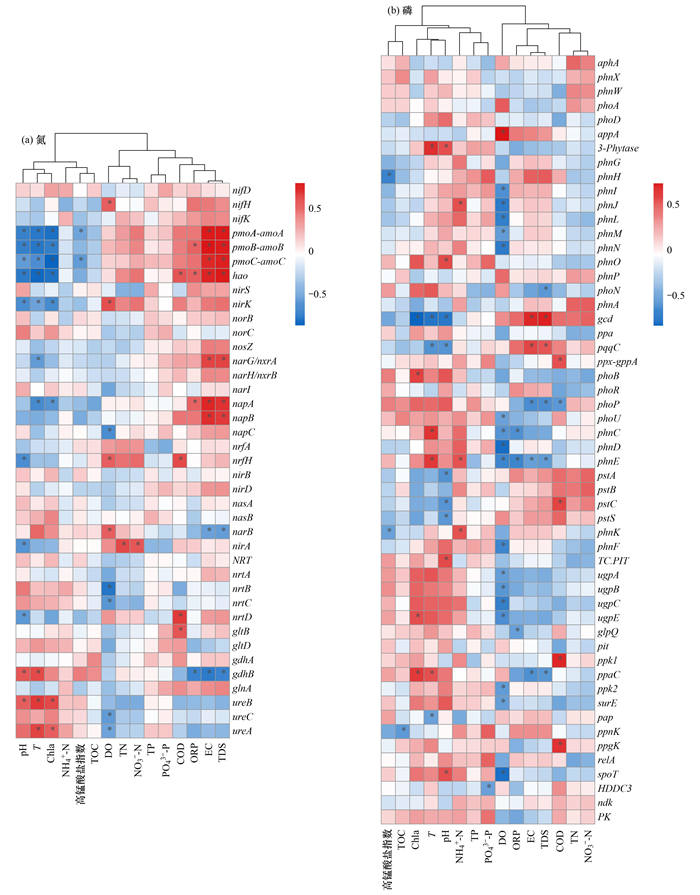
2.5 氮磷循环功能基因驱动因素分析将氮循环功能基因与水质理化指标进行相关性分析, 结果如图 8(a)所示. 硝化作用pmoA-amoA、pmoB-amoB、pmoC-amoC和hao主要与TDS、EC呈显著正相关, 与pH、T和Chla呈显著负相关. 反硝化作用narG/nxrA、narH/nxrB、napA和napB与TDS、EC呈显著正相关. T和Chla与ureB和ureA显著正相关, 与napA显著负相关. DO与nifH、nirK和nrfH呈显著正相关, 与nap、nrtB、nrtC、ureC和ureA呈显著负相关. TN和NO3--N与nirA显著正相关, NH4+-N与pmoA-amoA和pmoC-amoC显著负相关.
|
图 8 氮、磷循环功能基因与环境因子相关性分析热图 Fig. 8 Heat map of Spearman correlation analysis of nitrogen and phosphorus cycle genes and physicochemical parameters |
磷循环功能基因与水质理化指标相关性分析结果如图 8(b)所示, 有机磷矿化中3-Phytase与pH和T显著正相关, phnH与高锰酸盐指数显著负相关, phnJ与NH4+-N, phnO与pH均显著正相关, DO与phnI、hnJ、phnL、phnM和phnN显著负相关的同时, 与appA显著正相关. 无机磷溶解中gcd和pqqC与pH、T显著负相关的同时, 与TDS、EC显著正相关. 调节循环中phoR与TDS、EC和COD显著负相关. Chla、T、pH、NH4+-N、CODcr与转运循环中phnC、phnE、pstC、phnK、TC.PIT和ugpE等呈显著正相关, DO与phoU、phnC、phnD、phnE、phnF、ugpA、ugpB、ugpC和ugpE均显著负相关. 多聚磷酸盐合成中ppaC与Chla和T显著正相关, 与TDS、EC显著负相关. 多聚磷酸盐降解中ppgK和spoT与COD和pH显著正相关, ppk2、surE、pap、ppnK、spoT和HDDC3与TOC、T、PO43--P、DO、EC和TDS显著负相关.
3 讨论 3.1 丹江口水库浮游细菌垂向分布及其影响因素已有学者采用16S rRNA扩增子测序的方法对丹江口库区不同年份和季节的表层样品浮游细菌进行了研究, 发现其主要由放线菌门、变形菌门、Bacteroidetes和Firmicutes等优势门, hgcI_clade、Fluviicola、CL500-29_marine_group和Limnohabitans等优势属组成[6, 7, 16]. 对比宏基因组分析的浮游细菌群落组成, 在优势门, 特别是优势属的组成上有显著差异, 分析原因可能是采样时间上细菌群落的差异, 但更大可能是测序方法的不同. 相较16S rRNA扩增子测序, 宏基因组测序对微生物基因组随机进行打断, 并通过组装的方式, 将小片段拼接成较长的序列, 在物种鉴定过程中, 能更准确地进行注释, 可以更深入地表征微生物多样性[18].
南水北调中线工程调水系统自2014年启动, 丹江口水库作为南水北调中线工程的水源区, 水库正常蓄水位由157 m提升至170 m, 深度的增加会引起环境因子的变化, 从而影响浮游细菌的组成. 与Yue等[19]乌江渡水库和洪家渡水库浮游细菌垂向分布特征一致, NMDS结合ANOSIM分析表明丹江口水库浮游细菌组成在垂直方向上表现出显著的差异. 差异细菌分析表明主要为不动杆菌属、节杆菌属、奇异球菌属、莱茵海默氏菌属、聚球藻属和Nitrospira. 与其他湖泊、水库垂向环境因子的变化结果类似[19, 20], 丹江口水库随着水深的增加, 阳光变弱, T、DO和氧化还原电位降低, 浮游植物的生长繁殖受到限制, Chla也逐渐降低. T是影响浮游细菌生长繁殖的重要环境因子, 细菌内部酶的活性决定了其新陈代谢的能力, 在一定T范围内酶的活性与T成正比[21]. Li等[22]研究发现当T低于14 ℃时, T对浮游细菌的调控作用超过了其他影响因素, 是影响细菌分布的主要因素. 丹江口水库表层水样T高于14 ℃, 中层和底层T均低于14 ℃, 浮游细菌组成受T影响显著. 溶解氧是水生生物生命活动不可缺少的物质, 水库中的溶解氧主要来源于大气中的游离氧以及浮游植物和水生植物的光合作用, 蓝细菌和一些光合细菌也起一定作用[23]. 同时浮游植物光合作用过程中释放的DOM是浮游细菌的主要营养源. 丹江口水库溶解氧和Chla含量变化趋势一致, 随深度增加浓度降低, 是影响浮游细菌群落垂直分布的主要因素. 总之, 丹江口水库水深变化引起的理化性质中T、氧化还原电位、DO和Chla垂向差异, 导致浮游细菌群落呈明显的垂直分布模式.
3.2 丹江口水库氮磷循环基因垂向分布及其驱动因素微生物可以通过氨化作用、硝化作用、反硝化作用和固氮作用等将N2、无机氮化合物、有机氮化合物在自然界中相互转化, 从而驱动地球氮元素的生物地球化学循环[4, 5]. 丹江口库区总氮介于0.81~2.88 mg·L-1, 相对浓度较高, 是影响水质的主要风险因子. 之前的研究者采用荧光定量PCR和高通量测序技术表明, 丹江口水库表层水样和沉积物中具有丰富的氮循环微生物和功能基因的存在[9, 24]. 课题组对丹江口水库不同深度氮循环相关功能基因进行分析, 发现涉及生物固氮、硝化作用和反硝化作用等7个主要途径的39个氮循环功能基因, 与Zhu等[25]预测的河流中氮循环功能基因组成类似. 丹江口水库水深变化引起的理化性质驱动浮游细菌群落呈明显的垂直分布模式, 氮循环功能基因组成和丰度也呈现此趋势. 氮循环基因拷贝数聚类分析发现, 不同深度是影响氮循环功能基因组成和丰度的主要因素, 表层和底层氮循环功能基因组成有所差异, 整体丰度高于中层样品[图 6(a)]. 分析原因可能为表层为地表径流和大气氮沉降的主要汇入层, 底层距离沉积物较近, 沉积物中氮相对较高[7, 9, 24]. 不同深度样品在氮循环功能基因的丰度上有所差异, 表层样品有机氮代谢、异化硝酸盐还原作用中部分基因丰度高于其他样品, 底层样品在硝化作用、反硝化作用部分基因丰度高于其他样品. 分析表明驱动的因素主要为DO、pH、T、TDS、EC和Chla, 以上环境因子随深度增加浓度降低或升高, 表明丹江口水库水深变化引起的理化性质垂向差异, 导致浮游细菌功能呈明显的垂直分布特征. 有研究表明Nitrosopumilus和Nitrosarchaeum是长江流域中氨氧化古菌(AOA)的主要组成, 在氨氧化中起到重要作用[26]. 红树林杆菌属(Mangrovibacterium)是已报道的具有固氮能力的微生物, 参与丹江口水库固氮过程[27]. 嗜氢菌属(Hydrogenophaga)除了有脱氮功能外, 还对多种有机物具有良好的降解作用[28]. Limnohabitans作为淡水生态系统中常见的属, 可以存在于各种环境中, 可以捕获藻类来源的有机物质和参与氮磷循环[29]. 分析表明奇异球菌属(Deinococcus)、嗜氢菌属(Hydrogenophaga)、Limnohabitans、Nitrosarchaeum和亚硝化侏儒菌属(Nitrosopumilus)在丹江口水库氮循环过程中贡献比例高, 起到主要作用[图 7(a)].
磷元素作为我国大部分水库的限制性要素, 是水库富营养化控制的关键因子[30]. 课题组对丹江口水库不同深度磷循环功能基因预测表明, 其涉及有机磷矿化、无机磷溶解等6个主要途径的54个氮循环功能基因, 表现出磷循环功能基因的多样性[31]. 与之前的PICRUSt2预测的41个磷功能基因相比, 数量更多, 更为准确和全面地分析了丹江口水库不同深度磷循环功能基因的组成[6]. 与氮循环基因随水深变化的趋势一致, 表层、中层和底层磷循环功能基因有所差异, 这与Llorens-Marès等[15]研究的高山深湖磷循环基因随深度呈现差异一致. 分析表明DO、pH、T、TDS、EC和Chla与循环功能基因显著相关, 以上环境因子随丹江口水库深度增加浓度降低或升高, 导致浮游细菌磷循环功能基因呈明显的垂直分布特征. 磷循环功能基因整体趋势为表层和底层基因丰度高于中层样品, 该趋势与氮循环功能基因的趋势一致. 有研究表明Limnohabitans、奇异球菌属(Deinococcus)和棍状杆菌属(Clavibacter)是已报道的磷循环微生物, 分析表明以上微生物在丹江口水库磷循环过程中贡献比例高, 起到主要作用[6].
4 结论(1)丹江口水库水深变化引起的理化性质T、氧化还原电位、DO和Chla垂向差异, 导致浮游细菌群落呈明显的垂直分布模式.
(2)宏基因组分析表明, 丹江口水库浮游细菌参与生物固氮、硝化作用、反硝化作用和异化硝酸盐还原作用等氮循环过程, 涉及gltB、glnA、gltD、gdhA、NRT和ureC等39个氮循环功能基因.
(3)宏基因组分析表明丹江口水库浮游细菌参与有机磷矿化、无机磷溶解、调节和多聚磷酸盐合成等磷循环过程, 涉及pstS、ppx-gppA、glpQ、ppk1和ppnK等54个氮循环功能基因.
(4)垂直分布是影响氮、磷循环功能基因组成和丰度的主要因素, 表层和底层氮、磷循环功能基因组成有所差异, 整体丰度高于中层样品.
| [1] |
朱媛媛, 田进军, 李红亮, 等. 丹江口水库水质评价及水污染特征[J]. 农业环境科学学报, 2016, 35(1): 139-147. Zhu Y Y, Tian J J, Li H L, et al. Water qaulity assessment and pollution profile identification of Danjiangkou Reservoir, China[J]. Journal of Agro-Environment Science, 2016, 35(1): 139-147. |
| [2] | Wei M J, Duan P F, Gao P C, et al. Exploration and application of hydrochemical characteristics method for quantification of pollution sources in the Danjiangkou Reservoir area[J]. Journal of Hydrology, 2020, 590. DOI:10.1016/j.jhydrol.2020.125291 |
| [3] |
任丽娟, 何聃, 邢鹏, 等. 湖泊水体细菌多样性及其生态功能研究进展[J]. 生物多样性, 2013, 21(4): 421-432. Ren L J, He D, Xing P, et al. Bacterial diversity and ecological function in lake water bodies[J]. Biodiversity Science, 2013, 21(4): 421-432. |
| [4] | Kuypers M M M, Marchant H K, Kartal B. The microbial nitrogen-cycling network[J]. Nature Reviews Microbiology, 2018, 16(5): 263-276. DOI:10.1038/nrmicro.2018.9 |
| [5] |
杨雪琴, 连英丽, 颜庆云, 等. 滨海湿地生态系统微生物驱动的氮循环研究进展[J]. 微生物学报, 2018, 58(4): 633-648. Yang X Q, Lian Y L, Yan Q Y, et al. Microbially-driven nitrogen cycling in coastal ecosystems[J]. Acta Microbiologica Sinica, 2018, 58(4): 633-648. |
| [6] | Chen Z J, Liu Y Q, Li Y Y, et al. The seasonal patterns, ecological function and assembly processes of bacterioplankton communities in the Danjiangkou Reservoir, China[J]. Frontiers in Microbiology, 2022, 13. DOI:10.3389/fmicb.2022.884765 |
| [7] |
张菲, 田伟, 孙峰, 等. 丹江口库区表层浮游细菌群落组成与PICRUSt功能预测分析[J]. 环境科学, 2019, 40(3): 1252-1260. Zhang F, Tian W, Sun F, et al. Community structure and predictive functional analysis of surface water bacterioplankton in the Danjiangkou Reservoir[J]. Environmental Science, 2019, 40(3): 1252-1260. |
| [8] |
孙峰, 田伟, 张菲, 等. 丹江口库区库滨带植被土壤细菌群落多样性及PICRUSt功能预测分析[J]. 环境科学, 2019, 40(1): 421-429. Sun F, Tian W, Zhang F, et al. Composition and predictive functional analysis of rhizosphere bacterial communities in riparian buffer strips in the Danjiangkou Reservoir, China[J]. Environmental Science, 2019, 40(1): 421-429. |
| [9] |
阴星望, 田伟, 丁一, 等. 丹江口库区表层沉积物细菌多样性及功能预测分析[J]. 湖泊科学, 2018, 30(4): 1052-1063. Yin X W, Tian W, Ding Y, et al. Composition and predictive functional analysis of bacterial communities in surface sediments of the Danjiangkou Reservoir[J]. Journal of Lake Sciences, 2018, 30(4): 1052-1063. |
| [10] | Ren Z, Ma K, Jia X, et al. Metagenomics unveils microbial diversity and their biogeochemical roles in water and sediment of thermokarst lakes in the Yellow River source area[J]. Microbial Ecology, 2023, 85(3): 904-915. DOI:10.1007/s00248-022-02053-1 |
| [11] | Kuang B, Xiao R, Hu Y P, et al. Metagenomics reveals biogeochemical processes carried out by sediment microbial communities in a shallow eutrophic freshwater lake[J]. Frontiers in Microbiology, 2023, 13. DOI:10.3389/fmicb.2022.1112669 |
| [12] | Dang C Y, Xia Y, Zheng M S, et al. Metagenomic insights into the profile of antibiotic resistomes in a large drinking water reservoir[J]. Environment International, 2020, 136. DOI:10.1016/j.envint.2019.105449 |
| [13] |
罗建桦, 陶晔, 邢鹏, 等. 湖泊微生物宏基因组学研究进展[J]. 湖泊科学, 2020, 32(1): 271-280. Luo J H, Tao Y, Xing P, et al. Mini-review: advances of metagenomics research for lake microbiomes[J]. Journal of Lake Sciences, 2020, 32(1): 271-280. |
| [14] |
王朱珺, 王尚, 刘洋荧, 等. 宏基因组技术在氮循环功能微生物分子检测研究中的应用[J]. 生物技术通报, 2018, 34(1): 1-14. Wang Z J, Wang S, Liu Y Y, et al. The applications of metagenomics in the detection of environmental microbes involving in nitrogen cycle[J]. Biotechnology Bulletin, 2018, 34(1): 1-14. |
| [15] | Llorens-Marès T, Catalan J, Casamayor E O. Taxonomy and functional interactions in upper and bottom waters of an oligotrophic high-mountain deep lake (Redon, Pyrenees) unveiled by microbial metagenomics[J]. Science of the Total Environment, 2020, 707. DOI:10.1016/j.scitotenv.2019.135929 |
| [16] | Chen Z J, Xu G, Ding C Y, et al. Illumina MiSeq sequencing and network analysis the distribution and co-occurrence of bacterioplankton in Danjiangkou Reservoir, China[J]. Archives of Microbiology, 2020, 202(4): 859-873. DOI:10.1007/s00203-019-01798-7 |
| [17] | 国家环境保护总局. 水和废水监测分析方法[M]. ((第四版)). 北京: 中国环境科学出版社, 2002. |
| [18] | Durazzi F, Sala C, Castellani G, et al. Comparison between 16S rRNA and shotgun sequencing data for the taxonomic characterization of the gut microbiota[J]. Scientific Reports, 2021, 11(1). DOI:10.1038/s41598-021-82726-y |
| [19] | Yue Y H, Cai L, Tang Y, et al. Vertical distribution of bacterial community in water columns of reservoirs with different trophic conditions during thermal stratification[J]. Frontiers in Environmental Science, 2021, 9. DOI:10.3389/fenvs.2021.632089 |
| [20] |
杜蕾, 李文宝, 杨旭, 等. 达里诺尔湖夏季水体浮游细菌群落垂向变化特征差异[J]. 地球科学, 2020, 45(5): 1818-1829. Du L, Li W B, Yang X, et al. Vertical Changes of planktonic bacteria community and predictive functional analysis in summer Dali-Nor Lake[J]. Earth Science, 2020, 45(5): 1818-1829. |
| [21] | Vrede K. Nutrient and temperature limitation of bacterioplankton growth in temperate lakes[J]. Microbial Ecology, 2005, 49(2): 245-256. DOI:10.1007/s00248-004-0259-4 |
| [22] | Li W K W. Annual average abundance of heterotrophic bacteria and Synechococcus in surface ocean waters[J]. Limnology and Oceanography, 1998, 43(7): 1746-1753. DOI:10.4319/lo.1998.43.7.1746 |
| [23] | Yang M L, Shi J, Wang B L, et al. Control of hydraulic load on bacterioplankton diversity in cascade hydropower reservoirs, southwest China[J]. Microbial Ecology, 2020, 80(3): 537-545. DOI:10.1007/s00248-020-01523-8 |
| [24] | Dang C Y, Liu W, Lin Y X, et al. Dominant role of ammonia-oxidizing bacteria in nitrification due to ammonia accumulation in sediments of Danjiangkou Reservoir, China[J]. Applied Microbiology and Biotechnology, 2018, 102(7): 3399-3410. DOI:10.1007/s00253-018-8865-0 |
| [25] | Zhu J M, Hong Y G, Zada S, et al. Spatial variability and co-acclimation of phytoplankton and bacterioplankton communities in the Pearl River Estuary, China[J]. Frontiers in Microbiology, 2018, 9. DOI:10.3389/fmicb.2018.02503 |
| [26] | Liu S F, Wang H Y, Chen L M, et al. Comammox Nitrospira within the Yangtze River continuum: community, biogeography, and ecological driversd[J]. the ISME Journal, 2020, 14(10): 2488-2504. DOI:10.1038/s41396-020-0701-8 |
| [27] | Huang X F, Liu Y J, Dong J D, et al. Mangrovibacterium diazotrophicum gen. nov., sp. nov., a nitrogen-fixing bacterium isolated from a mangrove sediment, and proposal of Prolixibacteraceae fam. nov[J]. International Journal of Systematic and Evolutionary Microbiology, 2014, 64: 875-881. DOI:10.1099/ijs.0.052779-0 |
| [28] |
杨浩, 张国珍, 杨晓妮, 等. 16S rRNA高通量测序研究集雨窖水中微生物群落结构及多样性[J]. 环境科学, 2017, 38(4): 1704-1716. Yang H, Zhang G Z, Yang X N, et al. Microbial community structure and diversity in cellar water by 16S rRNA high-throughput sequencing[J]. Environmental Science, 2017, 38(4): 1704-1716. |
| [29] | Zheng L, Xing Y Z, Ding A Z, et al. Brownification of freshwater promotes nitrogen-cycling microorganism growth following terrestrial material increase and ultraviolet radiation reduction[J]. Science of the total Environment, 2022, 853. DOI:10.1016/j.scitotenv.2022.158556 |
| [30] | Zhang H X, Cao X H, Huo S L, et al. Changes in China's river water quality since 1980: management implications from sustainable development[J]. npj Clean Water, 2023, 6(1). DOI:10.1038/s41545-023-00260-y |
| [31] | McMahon K D, Read E K. Microbial contributions to phosphorus cycling in eutrophic lakes and wastewater[J]. Annual Review of Microbiology, 2013, 67: 199-219. DOI:10.1146/annurev-micro-092412-155713 |
 2024, Vol. 45
2024, Vol. 45


