2. 南阳师范学院水资源与环境工程学院, 南阳 473061
2. School of Water Resources and Environmental Engineering, Nanyang Normal University, Nanyang 473061, China
自从20世纪40年代人类生活中出现第一种塑料以来, 全球塑料工业开始兴起.据估计, 到2050年, 大约有1.2亿t塑料废物将被埋在垃圾填埋场或存在自然环境中.塑料废物会通过太阳辐射、风、环境温度变化、水浸泡和砂砾研磨等过程进一步降解, 形成微塑料(直径5 mm以下)[1], 其种类主要包括聚乙烯(PE)、聚丙烯(PP)、聚苯乙烯(PS)和聚氯乙烯(PVC)等[2].微塑料作为一种新兴的全球范围的环境污染物, 在淡水、土壤、公海, 甚至极地地区的地表水和沉积物中均被检测到, 影响着全球的海洋、淡水和陆地生态系统[3, 4], 也受到科学界的广泛关注.陆地生态系统中的微塑料主要来源于农田塑料地膜的降解, 以聚乙烯(polyethylene, PE)为主要成分的塑料地膜在我国已经广泛应用40多年, 其产量和用量均居世界首位, 而高密度聚乙烯(high density polyethylene, HDPE)是农业地膜中最常使用的塑料.在我国农业生产中, 农户大量使用HDPE农用地膜以达到提高作物产量目的, 但由于环保意识淡薄以及农膜回收技术欠缺导致覆膜回收率较低, 风化分解而成的微塑料带来严重的环境污染问题[5].HDPE农用地膜残留会提高耐受性微生物群落丰度, 并改变土壤结构和理化性质, 损害作物生长.此外, 微塑料具有较大的比表面积和较强的疏水性, 可吸附多种农药, 影响其生态效应[6~8].
氯嘧磺隆是美国杜邦公司开发的磺酰脲类除草剂代表品种之一 [9], 因其高效、杀草谱广和针对性强等特点, 在20世纪90年代初期, 氯嘧磺隆及其复配制剂在我国得到广泛应用.但其不易挥发、不易光解且半衰期长, 土壤中较低残留便会对后茬敏感作物产生药害, 导致农作物产量减少, 并且氯嘧磺隆易随降水渗透至地表及地下, 引发地下水及局部饮用水源的污染, 从而造成潜在的环境和健康风险[10].此外, 氯嘧磺隆喷施以后进入土壤, 影响土壤微生物及其活性, 改变群落结构和土壤生物学功能, 且在短期内很难恢复[11].
土壤是储存和供应养分、进行碳周转和元素生物地球化学循环的介质.土壤微生物是土壤最活跃的组成, 是评价土壤健康的关键生物指标[12].在土壤系统中, 根际是土壤中植物根系和微生物相互作用的生物活性区, 而根际微生物是其中的重要组成成分, 可以活化土壤中难溶的营养物质, 促进植物的营养吸收, 合成或降解植物激素, 促进植物生长, 协助植物抵抗病害和非生物胁迫(干旱、重金属等), 而根际土壤微生物群落结构与功能多样性的改变均能反映土壤质量和健康状况[13, 14].目前关于单一污染物, 如微塑料、有机农药等对作物根际土壤微生物的影响已有报道[15, 16], 但关于两者的复合胁迫效应研究较少.
大豆是我国重要的粮食作物和油料作物, 也是我国重要的进口农产品.在我国的种植面积占主要作物播种面积的8%左右.大豆覆膜栽培是一项有效的高产栽培技术, 可增加作物干物质的积累, 提高产量[17].而氯嘧磺隆作为一种选择性芽前和芽后除草剂, 主要用于大豆田防除阔叶杂草[18].但随之而来的微塑料与氯嘧磺隆在农田土壤中的复合胁迫问题也不容小觑.因此, 本文以大豆作为供试植物, 以高密度聚乙烯微塑料(HDPE)和氯嘧磺隆为研究对象, 分析复合胁迫下对大豆生长的影响, 并通过高通量测序技术和网络分析, 探究HDPE和氯嘧磺隆复合胁迫对大豆根际土壤细菌群落多样性、结构组成和互作网络的影响, 结合PICRUSt2功能分析土壤功能的变化, 阐明HDPE和氯嘧磺隆对大豆的复合毒性, 以期为评价HDPE与氯嘧磺隆复合胁迫下生态环境风险以及为后续污染土壤的修复提供指导.
1 材料与方法 1.1 实验材料氯嘧磺隆购自南京格雷福斯生物科技有限公司, 纯度97%.高密度聚乙烯(HDPE, 500目)购自东莞鑫淼新材料有限公司, 无毒, 无味, 密度为0.941~0.960 g·cm-3, 结晶度为80%~90%, 软化点为125~135℃.实验土壤采集地点为河南省南阳市的大豆种植基地(32°59′N, 112°32′E), 土壤类型为黄棕壤, 自然风干后过2 mm筛, 待用.理化性质为:ω(有机质)19.6 g·kg-1, ω(总氮)1.07 g·kg-1, ω(总磷)0.89 g·kg-1, ω(总钾)15.16 g·kg-1, pH 7.2.
1.2 大豆种子处理大豆品种为中黄37, 购自南京雷克顿生物科技有限公司.将大豆种子置于无菌水中并浸泡12 h后, 转移至8%的NaClO中振荡消毒5 min, 无菌水清洗5次, 置于湿润的无菌纱布上, 25℃避光萌发2 d.挑选萌发良好的种子接种至霍格兰营养液中, 以25℃, 11 h光照, 18℃, 13 h黑暗为一周期, 培养3 d待用.
1.3 HDPE和氯嘧磺隆对大豆生长的影响取过筛后的供试土壤装入盆钵中(1.0 kg·盆-1), 并设置4个处理, 分别为:不添加HDPE和氯嘧磺隆(CK), 添加1% HDPE(质量分数, PE), 添加1 mg·kg-1氯嘧磺隆(L), 添加1% HDPE和1 mg·kg-1氯嘧磺隆(LP).选取长势一致的大豆幼苗转移至上述4种不同处理的土壤中生长, 设置培养箱条件为35℃, 16 h光照, 25℃, 8 h黑暗, 连续培养20 d后, 将各处理中的幼苗拔出, 测定茎叶鲜重、干重、根系鲜重和干重, 并利用抖落法收集根际土[19], 每组处理设置3个重复.
1.4 HDPE对土壤中氯嘧磺隆半衰期的影响本实验方法如1.3节, 其中针对1 mg·kg-1氯嘧磺隆(L)、1% HDPE和1 mg·kg-1氯嘧磺隆(LP)的处理, 分别于0、1、3、5、7、9、14、18和20 d取大豆根际土, 测定土壤中氯嘧磺隆残留含量, 每个处理设置3个重复.
1.5 大豆根际土壤细菌总DNA的提取、高通量测序和数据分析利用土壤DNA提取试剂盒(FastDNA® SPIN Kit for Soil, MP BiomedicalsTM)提取4个处理培养20 d的根际土壤总DNA.利用NanoDrop OneC超微量紫外分光光度计测定DNA浓度与纯度, 合格样品寄送至北京百迈客生物科技有限公司.根据16S rRNA基因V3-V4区序列设计引物, 在引物末端加上测序接头, 进行PCR扩增并对其产物进行纯化、定量和均一化, 形成测序文库, 同时进行文库质检, 质检合格后用Illumina HiSeq 2500进行测序, 每组处理设置3个重复.
高通量测序所得的原始数据(raw reads)首先通过Trimmomatic v0.33软件, 进行过滤, 然后利用cutadapt 1.9.1软件进行引物序列的识别与去除, 得到不包含引物序列的clean reads, 利用FLASH v1.2.7软件, 通过overlap对每个样品的reads进行拼接, 得到clean reads.再利用UCHIME v4.2软件鉴定并去除嵌合体序列, 得到最终有效数据(effective reads).为研究各样本的物种组成, 对所有样本的有效数据, 通过Usearch软件对OTUs(operational taxonomic units)进行聚类(97%的一致性), 并对OTUs的序列进行物种注释.根据物种注释结果, 选取不同样本在门(phylum)和属(genus)水平相对丰度较高的物种, 生成物种相对丰度柱形累加图, 以便直观查看各样本在不同分类水平上的物种及其比例.基于OTU进行Alpha多样性分析(丰富度指数Chao1、ACE和多样性指数Shannon和Simpson), 通过QIIME计算Beta多样性距离矩阵, 并利用R语言工具对样品进行层次聚类(UPGMA, unweighted pair-group method with arithmetic mean), 以判断各样品间物种组成的相似性.同时进行PCoA(principal co-ordinates analysis)统计分析, 可视化呈现不同样本群落组成的差异程度.通过R语言工具, 基于weighted unifrac距离算法得到样品间的距离矩阵, 绘制样品热图(heatmap), 可根据颜色梯度的变化判断样品间的差异性, 通过PERMANOVA/Anosim对样品差异进行显著性分析.利用LEfSe检验, 分析属水平下组间具有显著差异的物种(P < 0.05).利用PICRUSt2软件将待预测的特征序列与软件中已有的系统发育树进行物种注释, 使用IMG微生物基因组数据进行功能信息的输出, 进而推测样本中的功能基因组成, 从而分析不同样品之间在功能上的差异.根据不同物种在各个样品中的丰度以及变化情况, 进行斯皮尔曼(Spearman)秩相关分析并筛选相关性大于0.1且P值小于0.05的数据构建相关性网络.基于网络图的分析, 可以获得物种在环境样本中的共存关系, 推测物种在同一环境下的相互作用情况及重要的模式信息.
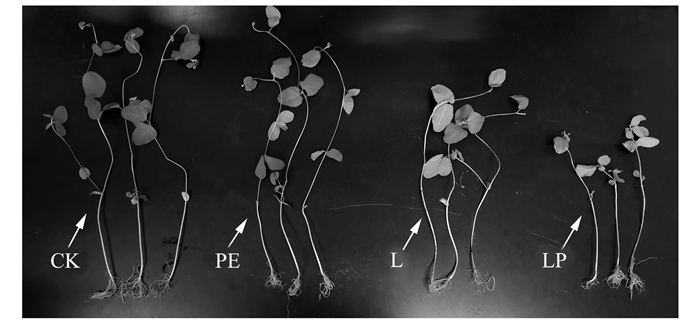
2 结果与分析 2.1 HDPE与氯嘧磺隆对大豆生长的影响与对照(CK)相比, HDPE(PE)、氯嘧磺隆(L)单一及两者复合处理(LP)均能抑制大豆的生长(表 1和图 1), 其中PE处理对茎叶鲜重、根鲜重和茎叶干重的抑制率分别为22.4%、20.0%和23.5%, L处理的抑制率分别为25.1%、26.7%和26.5%, LP处理的抑制率最高, 分别为61.0%、46.7%和61.8%, 对根干重的抑制率也达到33.3%, 均达到显著差异水平(P < 0.05).
|
|
表 1 大豆根茎叶第20 d生长情况1)/g Table 1 Growth of soybean roots, stems, and leaves on the 20th day/g |
|
1)CK:添加无菌水处理的土壤, PE:添加1% HDPE处理的土壤, L:添加1 mg·kg-1氯嘧磺隆处理的土壤, LP:添加1% HDPE和1 mg·kg-1氯嘧磺隆处理的土壤;同列不同小写字母表示在P < 0.05水平差异显著 图 1 大豆根茎叶生长情况 Fig. 1 Growth of soybean roots, stems, and leaves |
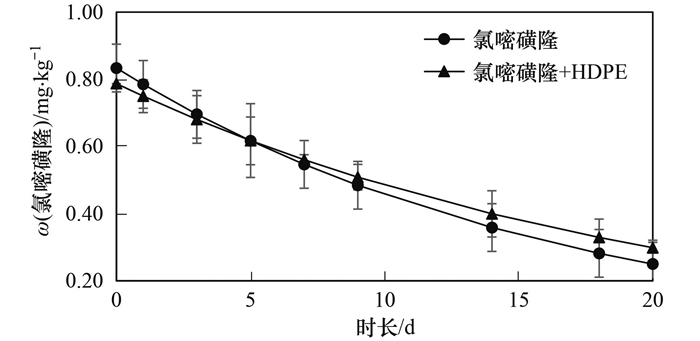
大豆根际土壤中氯嘧磺隆的残留动态如图 2所示, 氯嘧磺隆土壤中的降解规律符合化学一级动力学反应:
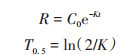
|
|
图 2 氯嘧磺隆在不同处理下的残留动态 Fig. 2 Degradation of chlorimuron-ethyl in different treatments |
式中, C0为土壤中的初始氯嘧磺隆含量(mg·kg-1), R为t时土壤中的氯嘧磺隆含量(mg·kg-1), t为培养时间(d), K为农药的降解速率常数, T0.5为氯嘧磺隆降解半衰期.未添加HDPE的处理中, 初始ω(氯嘧磺隆)为0.83 mg·kg-1, 20 d后降为0.25 mg·kg-1, 降解速率常数为0.060 3, 半衰期为11.5 d.加入HDPE后, 初始ω(氯嘧磺隆)为0.79 mg·kg-1, 20 d后降为0.30 mg·kg-1, 降解速率常数为0.048 5, 半衰期增加至14.3 d.由此可知, HDPE的存在降低土壤中氯嘧磺隆的降解速率, 延长其在土壤中的半衰期, 表明HDPE与氯嘧磺隆复合胁迫所造成的潜在危害更大, 与大豆生长结果一致.
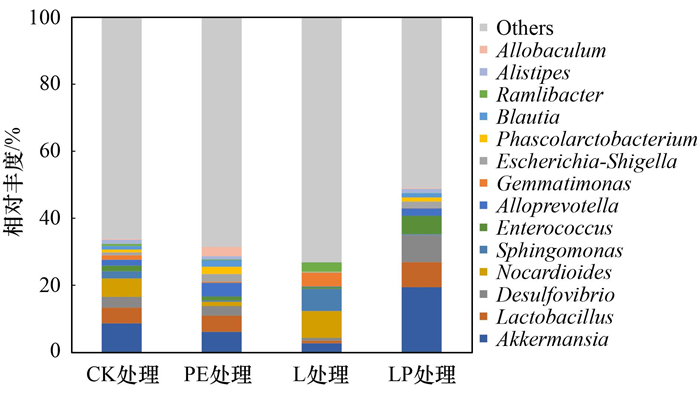
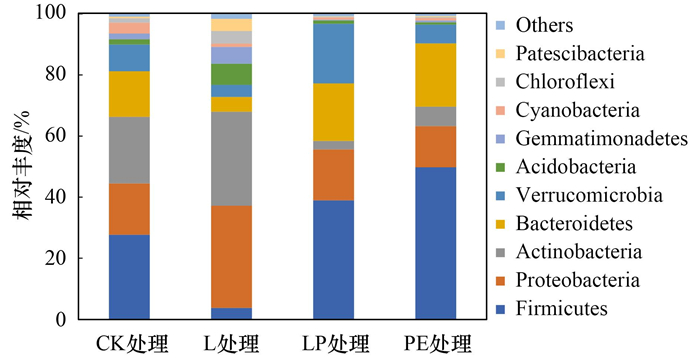
2.3 大豆根际土壤细菌群落物种组成和Alpha、Beta多样性分析大豆根际土壤细菌群落在门与属水平上的组成差异如图 3和图 4所示, CK条件下土壤细菌群落由23个门, 408个属组成, PE处理土壤由22个门, 389个属组成, L处理土壤由23个门, 390个属组成, 三者在门与属水平上的组成数量差别不大, 但LP处理土壤则由20个门, 312个属组成, 属水平组成数量上显著降低.各处理在门水平上(> 1%)主要由厚壁菌门(Firmicutes, CK:27.7%, L:3.7%, LP:39.0%, PE:49.7%)、变形菌门(Proteobacteria, CK:16.8%, L:33.5%, LP:16.7%, PE:13.5%)、放线菌门(Actinobacteria, CK:21.8%, L:30.8%, LP:2.8%, PE:6.4%)、拟杆菌门(Bacteroidetes, CK:14.9%, L:4.8%, LP:18.8%, PE:20.7%)、疣微菌门(Verrucomicrobia, CK:8.8%, L:3.9%, LP:19.5%, PE:6.1%)、酸杆菌门(Acidobacteria, CK:1.7%, L:7.0%, LP:1.1%, PE:0.8%)、芽单胞菌门(Gemmatimonadetes, CK:1.9%, L:5.5%, LP:0.2%, PE:0.5%)、蓝藻门(Cyanobacteria, CK:3.6%, L:1.1%, LP:0.7%, PE:0.8%)、绿弯菌门(Chloroflexi, CK:1.5%, L:4.1%, LP:0.2%, PE:0.3%)和髌骨细菌门(Patescibacteria, CK:0.5%, L:4.0%, LP:0.2%, PE:0.3%)等组成.厚壁菌门、变形菌门、变形菌门和拟杆菌门为各条件下的优势菌群, 但各物种的相对丰度存在差异.与CK相比, L处理显著降低厚壁菌门(由27.7%降至3.7%)和拟杆菌门的相对丰度(由14.9%降至4.8%), 但提高变形菌门(由16.8%升至33.5%)和放线菌门的相对丰度(由21.8%升至30.8%).PE处理降低变形菌门的相对丰度(由16.8%降至13.5%), 但显著提高厚壁菌门的相对丰度(由27.7%升至49.7%).LP则显著降低放线菌门的相对丰度(由21.8%降至2.8%), 提高厚壁菌门(由27.7%升至39.0%)、疣微菌门(由8.8%升至19.5%)和拟杆菌门(由14.9%升至18.8%)的相对丰度.此外, LP处理也降低变形菌门、酸杆菌门、芽单胞菌门、蓝藻门、绿弯菌门和髌骨细菌门的相对丰度.
|
图 3 不同处理土壤细菌属水平的相对丰度 Fig. 3 Relative abundance of bacterial community at genus level in soil with different treatments |
|
图 4 不同处理土壤细菌门水平的相对丰度 Fig. 4 Relative abundance of bacterial community at phylum level in soil with different treatments |
各处理在属水平上(> 0.5%)主要由阿克曼氏菌属(Akkermansia, CK:8.6%, PE:6.0%, L:2.7%, LP:19.3%)、乳杆菌属(Lactobacillus, CK:4.7%, PE:4.8%, L:0.8%, LP:7.4%)、脱硫弧菌属(Desulfovibrio, CK:3.2%, PE:2.8%, L:0.8%, LP:8.0%)、类诺卡氏菌属(Nocardioides, CK:5.4%, PE:1.3%, L:8.0%, LP, 0.2%)、鞘氨醇单胞菌属(Sphingomonas, CK:2.1%, PE:0.4%, L:6.6%, LP:0.2%)等组成.与CK相比, LP显著提高阿克曼氏菌属、乳杆菌属和脱硫弧菌属的相对丰度, PE和L处理则降低其相对丰度.LP处理显著降低类诺卡氏菌属和鞘氨醇单胞菌属的相对丰度, PE处理同样降低其相对丰度, 但抑制效果不如LP处理, L处理则提高类诺卡氏菌属和鞘氨醇单胞菌属的相对丰度.结果表明L和PE处理对上述菌群的相对丰度均有影响, 但LP处理的影响更为明显, 并且变化不一致.
土壤细菌群落Alpha多样性指数分析结果如表 2所示, CK条件下, 大豆根际细菌群落丰富度指数(ACE、Chao1)和多样性指数(Simpson、Shannon)最高, 而L、PE和LP处理均会降低细菌群落的丰富度指数和多样性指数.显著性分析表明PE和L处理土壤的丰富度与多样性指数与CK无显著差异, 而LP处理下土壤细菌群落的丰富度和多样性指数均显著低于CK以及另外两种处理(P < 0.05)
|
|
表 2 不同土壤样品细菌群落Alpha多样性指数分析1) Table 2 Alpha diversity analysis of bacterial community in different soil samples |
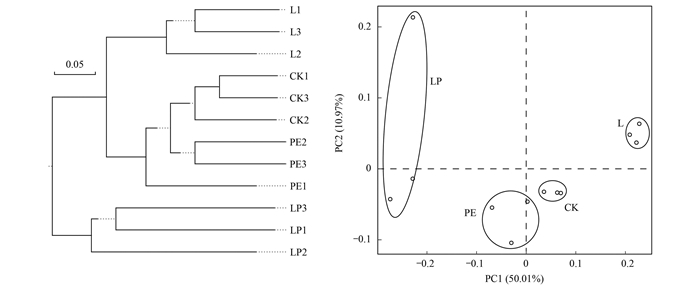
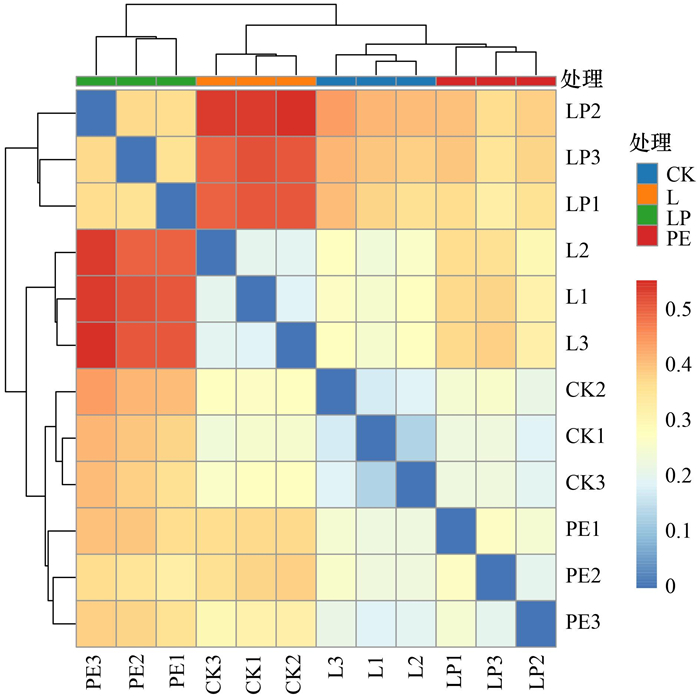
利用UPGMA和PCoA分析样品差异情况, 结果如图 5所示.UPGMA分析可知CK和L、PE处理的样品分布在同一个支上, 而LP处理样品独立在一个分支上且分支长度相对较长, 说明LP处理相较于其他处理在细菌群落结构上存在较大差异.类似结果同样出现在PCoA分析和样品聚类热图分析中(图 6).PERMANOVA/Anosim分析表明不同分组间细菌群落差异达到显著水平(P < 0.05).
|
图 5 不同处理土壤样品UPGMA和PCoA分析 Fig. 5 UPGMA and PCoA analysis in soil with different treatments |
|
图 6 不同处理土壤样品细菌根际群落差异性分析 Fig. 6 Difference analysis of bacterial rhizosphere community in different soil samples |
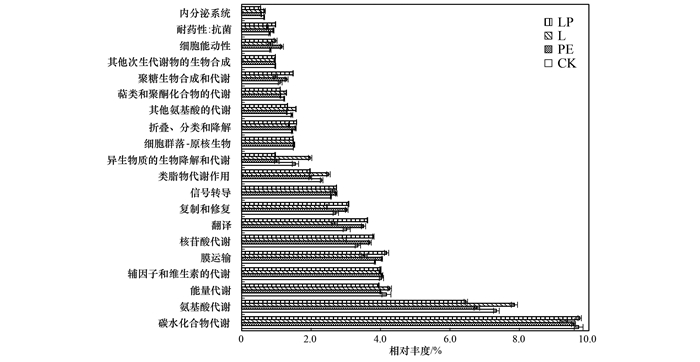
利用PICRUSt2对不同处理的细菌群落功能进行预测, 共得到代谢(metabolism)、环境信息处理(environmental information processing)、遗传信息处理(genetic information processing)、人类疾病(human diseases)、有机体系统(organismal systems)和细胞进程(cellular processes)6种一级功能生物代谢通路, 其中代谢的相对丰度显著高于其他5种功能.在6种一级功能生物代谢通路下, 可分为43种二级功能生物代谢通路.相对丰度前20的二级功能种类中, 11种归于代谢, 如碳水化合物代谢、氨基酸代谢和能量代谢等, 3种归于遗传信息处理, 如翻译、复制和修复等, 2种归于环境信息处理, 如信号转导等, 2种归于细胞进程, 如细胞运动等, 1种归于有机体系统(内分泌系统), 1种归于人类疾病(耐药性)(图 7).在此20种二级功能中, 与CK相比, LP处理提高大豆根际土壤细菌膜运输、核苷酸代谢、信号转导和翻译等功能的相对丰度, 降低氨基酸代谢、能量代谢和脂质代谢等功能的相对丰度, 对土壤细菌功能的影响幅度大于L和PE处理.
|
图 7 不同土壤样品细菌功能预测 Fig. 7 Prediction of bacterial function in different soil samples |
LEfSe [line discriminant analysis(LDA)effect size]分析并展示组间在丰度上有显著差异的物种.从图 8中可以看出, 在不同水平上共发现40个差异菌群(LDA > 4.0, P < 0.05), 其中L处理中有19个差异菌群, 属水平上的有类诺卡氏菌属(Nocardioides)、沙壤土杆菌属(Ramlibacter)和芽单胞菌属(Gemmatimonas)等.LP有11个, 属水平上的有脱硫弧菌属(Desulfovibrio)、阿克曼氏菌属(Akkermansia)、肠球菌属(Enterococcus)和乳杆菌属(Lactobacillus).PE有10个, 属水平上的有志贺氏菌(Escherichia_Shigella)和别样棒菌属(Allobaculum)等.CK条件下不存在显著差异的菌群.不同处理调控不同细菌群落的相对丰度, 导致物种组成与结构功能上有所差异.
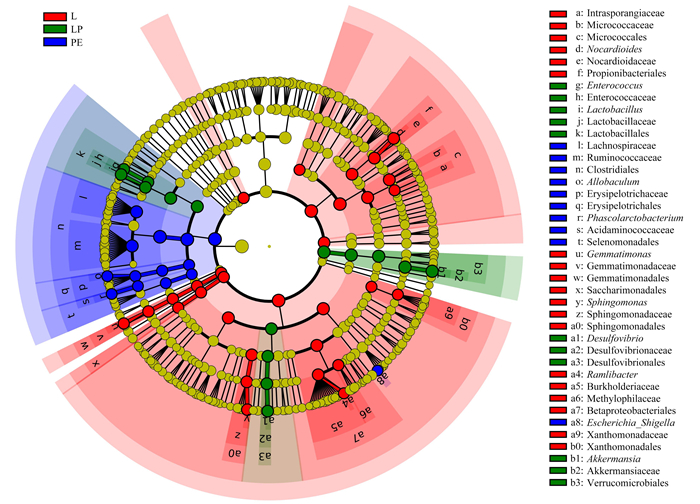
|
图 8 LEfSe分析不同处理土壤中的差异菌属 Fig. 8 LEfSe analysis evolutionary branching diagram |
通过斯皮尔曼(Spearman)秩相关分析并筛选相关性大于0.1且P值小于0.05的数据构建细菌相关性网络, 结果如图 9所示.CK条件下, 土壤细菌网络图中的总连接数(total links)为1 436个, 其中正相关(positive links)736个, 负相关(negative links)700个, PE处理的总连接数1 236个, 其中正相关727个, 负相关509个, L处理的总连接数1 230个, 其中正相关700个, 负相关530个, LP处理的总连接数1 031个, 其中正相关539个, 负相关492个.PE、L、LP处理下土壤细菌网络图中的总连接数、正相关、负相关相较于CK均呈现减少趋势, 其中LP处理细菌网络结构最为简单.在其他参数中, 4组不同处理的网络模块数量(number of communities, 3)、网络直径(network diameter, 1)、平均最短路径(average shortest path length, 1)和平均聚类系数(average clustering coefficient, 1)均一致.而LP处理的网络密度(network density, LP:0.326, CK:0.454, L:0.399, PE:0.391)最小, 模块性(modularity, LP:0.661, CK:0.261, L:0.522, PE:0.533)最大.
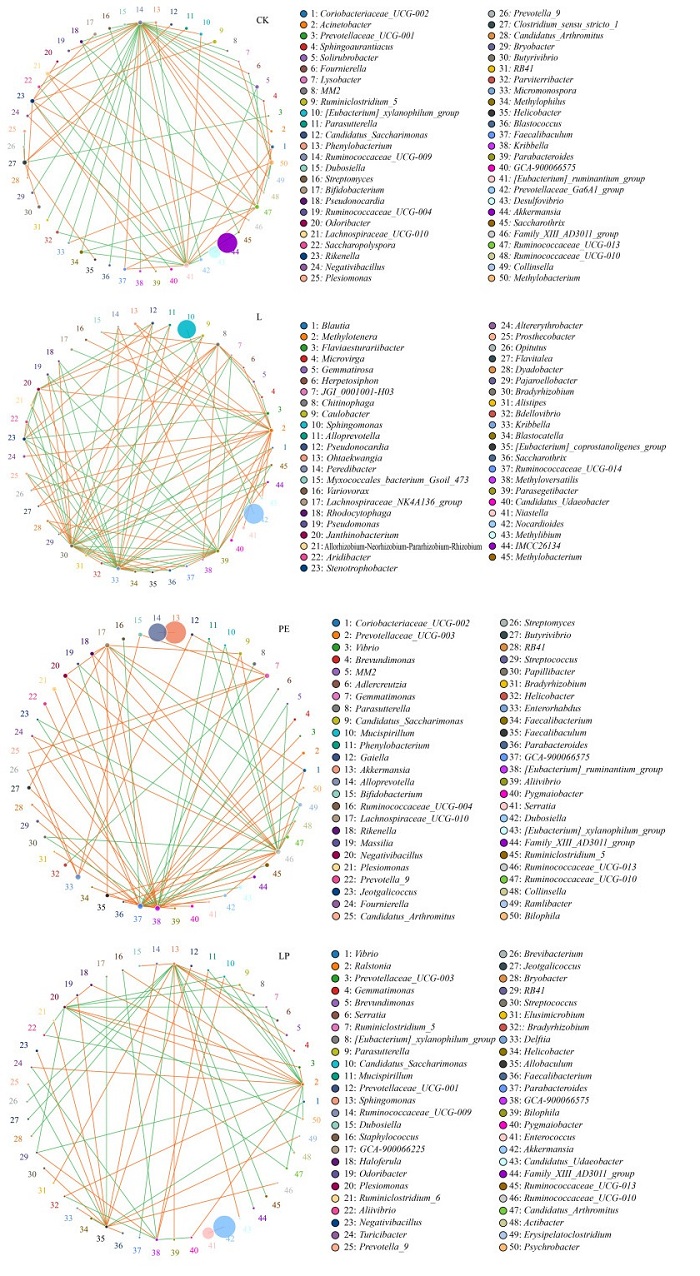
|
橙色表示正相关, 绿色表示负相关 图 9 不同处理土壤中的细菌相关性网络分析(相关性最高的前50个属) Fig. 9 Network analysis of bacteria in soil with different treatments (top 50 genera with the highest correlation) |
根据相关性网络中物种的总连接数, 推断不同处理条件下细菌网络中的关键菌群.如表 3所示, CK条件下的网络中, 连接数最高的有类诺卡氏菌属(Nocardioides)、异普雷沃菌属(Alloprevotella)、鞘氨醇单胞菌属(Sphingomonas)和芽单胞菌属(Gemmatimonas)等, 主要分布在厚壁菌门、拟杆菌门、变形菌门和芽单胞菌门.LP处理土壤细菌网络中, 连接数最高的有阿克曼氏菌属(Akkermansia)、脱硫弧菌属(Desulfovibrio)、乳杆菌属(Lactobacillus)和异普雷沃菌属(Alloprevotella)等, 主要分布在疣微菌门、变形菌门、厚壁菌门和拟杆菌门.L处理土壤细菌网络中连接数最高的有类诺卡氏菌属(Nocardioides)、鞘氨醇单胞菌属(Sphingomonas)、芽单胞菌属(Gemmatimonas)和嗜甲基菌属(Methylophilus)等, 主要分布在放线菌门、变形菌门和芽单胞菌门.PE处理土壤细菌网络中连接数最高的有阿克曼氏菌属(Akkermansia)、异普雷沃菌属(Alloprevotella)、脱硫弧菌属(Desulfovibrio)和别样棒菌属(Allobaculum)等, 主要分布在疣微菌门、厚壁菌门、变形菌门和拟杆菌门.由此推测, 不同条件下细菌网络中关键菌群种类与分布各不相同, 结合网络参数, HDPE与氯嘧磺隆复合胁迫(LP)较单一处理(L和PE)对网络结构的简化作用更为明显, 与此同时关键菌群也发生显著变化.
|
|
表 3 不同处理土壤样品细菌相关性网络结构分析 Table 3 Analysis of bacterial correlation network structure of soil samples with different treatments |
3 讨论 3.1 HDPE与氯嘧磺隆对大豆生长的影响
土壤在生态系统的可持续发展中起着非常重要的作用, 洁净土壤是农业生态系统健康和稳定的基础[20], 而土壤中氯嘧磺隆的残留会导致后茬敏感作物生长受阻[21], 如影响大豆的根系结瘤, 严重干扰土壤氮素转化, 影响大豆土壤生物质量和大豆生长[22, 23], 这与本研究的结果是一致的.微塑料被认为是环境污染物的重要载体, 对诸多物质具有很强的吸附能力[24, 25].有研究表明聚乙烯等微塑料能通过吸附农药而改变其半衰期[22].本实验结果表明HDPE存在的情况下, 氯嘧磺隆在供试土壤中的半衰期由11.5 d延长至14.3 d, 并且相较于HDPE与氯嘧磺隆单一处理, 两者复合胁迫显著降低大豆的茎叶鲜重和干重、根部鲜重和干重, 抑制效果更为强烈, 表明HDPE与氯嘧磺隆复合胁迫所造成的潜在危害更大.
3.2 HDPE与氯嘧磺隆对大豆根际细菌群落多样性和组成的影响土壤细菌群落是影响土壤质量的一个重要因素[26, 27].微塑料能通过改变土壤团聚体稳定性, 抑制土壤酶活性, 进而改变细菌群落的丰富度和多样性[28].重复施用氯嘧磺隆会降低土壤酶活性, 改变土壤微生物群落结构[29~31].微塑料与其他物质混施会降低土壤微生物群落多样性, 而单一微塑料影响较小[32].本实验通过高通量测序技术测得HDPE和氯嘧磺隆复合胁迫的土壤样品细菌由20个门, 312个属组成, 门和属水平上的组成数量显著低于对照组、氯嘧磺隆和HDPE单一处理组, 并且细菌群落结构丰富度指数(ACE、Chao1)和多样性指数(Simpson、Shannon)均显著低于其他3组处理.推测原因为与单一处理相比, HDPE和氯嘧磺隆复合胁迫使大豆根系在土壤中的分布更复杂, 导致土壤细菌与大豆根系分泌物互动更频繁, 从而改变土壤细菌的代谢活性, 打破物种组成和比例之间的动态平衡, 造成根际微生物种类和多样性的改变, 并影响群落的稳定性[33, 34].
此外, 对照条件下分析大豆根际土壤中相对丰度占比前20, 并且存在显著差异细菌的潜在功能, 发现:类诺卡氏菌属(Nocardioides)参与生物修复有机污染物[35]、鞘氨醇单胞菌属(Sphingomonas)参与生物防治[36]、芽单胞菌属(Gemmatimonas)通过还原N2O参与氮循环[37]、脱硫弧菌属(Desulfovibrio)参与生物修复重金属污染[38], 表明大豆根际土壤也是分离功能菌株的良好资源.
3.3 HDPE与氯嘧磺隆对大豆根际细菌群落功能的影响HDPE和氯嘧磺隆复合胁迫改变土壤细菌群落结构的同时, 也使土壤细菌功能代谢通路产生差异.不同处理下大豆根际细菌共有代谢、环境信息处理、遗传信息处理、细胞过程、人类疾病和有机体系统6种一级功能生物代谢通路, 表明大豆根际土壤细菌利用这6种功能维系生态系统的稳定[39, 40], 其中代谢是土壤细菌群落的核心功能.根际土壤细菌通过代谢活动来参与土壤物质循环与转化, 进而影响植物生长和作物产量.因此, 氨基酸代谢、碳水化合物代谢、脂质代谢和能量代谢等二级功能生物代谢通路相对丰度较高[41].与对照、HDPE和氯嘧磺隆单一胁迫相比, HDPE和氯嘧磺隆复合胁迫降低氨基酸代谢、脂质代谢和能量代谢等代谢功能的相对丰度, 从而抑制大豆生长.功能分析结果也表明大豆生长受到抑制, 可能原因之一为HDPE和氯嘧磺隆两种污染物的存在, 直接影响其生长, 另一方面HDPE和氯嘧磺隆会影响根际土壤细菌功能, 尤其是个别关键功能的相对丰度, 间接影响大豆生长.
3.4 HDPE与氯嘧磺隆对大豆根际细菌互作网络的影响细菌互作网络分析可以将微生物群落的相互作用关系可视化, 并揭示物种在微生境中的共现关系及主要影响因素.模块性用来衡量一个网络能否自然地划分成模块, 是生态系统结构的重要特征, 可识别高度连接的分类群, 反映复杂网络中的协同关系、竞争互动和生态位分化以及一些进化特性, 能够体现环境变化对生物群落组成的影响[42].HDPE和氯嘧磺隆复合胁迫模块性最大, 可推测该网络具有模块结构且对生物群落组成的影响最大.本研究中4种处理的网络直径和平均最短路径均相同, 说明4种处理的网络的传输性能和效率相差不大.脱硫弧菌属(Desulfovibrio)是对照条件下的关键菌属, 具有潜在生物修复重金属污染的功能[38].肠球菌属(Enterococcus)是HDPE和氯嘧磺隆复合胁迫下的关键菌属, 常做粪便指示菌[43], 关键菌群发生变化, 功能也发生显著变化.关键类群是微生物群落中高度连接的类群, 具有特殊而重要的作用, 其改变可引起群落结构和功能的急剧变化[44].
由此推测, HDPE和氯嘧磺隆复合胁迫降低大豆根际土壤细菌互作网络间的总连接数和密度, 使其结构更加简化, 增加模块性, 影响细菌群落组成与功能, 破坏植物-微生物生态体系的平衡, 抑制大豆的生长, 较单一胁迫的影响更为显著 [45, 46].
4 结论(1)HDPE处理提高氯嘧磺隆在土壤中的半衰期, 由11.5 d升至14.3 d, 并且HDPE与氯嘧磺隆复合胁迫显著降低大豆的茎叶鲜重、干重、根部鲜重和干重, 影响大豆生长.
(2)HDPE与氯嘧磺隆复合胁迫下, 大豆根际细菌群落主要由疣微菌门、变形菌门、厚壁菌门和拟杆菌门等20个门, 阿克曼氏菌属、脱硫弧菌属和乳杆菌属等312个属组成, 其在门和属水平上的组成数量显著降低.
(3)结合Alpha、Beta多样性分析可知, HDPE与氯嘧磺隆复合胁迫显著降低大豆根际细菌群落的丰富度和多样性, 改变群落结构.LEfSe物种差异分析表明HDPE与氯嘧磺隆复合胁迫的土壤中存在显著差异的物种有阿克曼氏菌属, 乳杆菌属, 脱硫弧菌, 类诺卡氏菌属和肠球菌属等, 主要分布在疣微菌门、变形菌门和厚壁菌门.
(4)PICRUSt2功能分析显示HDPE与氯嘧磺隆复合胁迫影响土壤细菌各功能的相对丰度.Network互作网络分析推测复合胁迫降低大豆根际土壤细菌间的总连接数和网络密度, 使网络结构更为简单, 并引起关键菌群种类的改变.
| [1] | Yu F, Li Y, Huang G Q, et al. Adsorption behavior of the antibiotic levofloxacin on microplastics in the presence of different heavy metals in an aqueous solution[J]. Chemosphere, 2020, 260. DOI:10.1016/j.chemosphere.2020.127650 |
| [2] | Ram B, Kumar M. Correlation appraisal of antibiotic resistance with fecal, metal and microplastic contamination in a tropical Indian river, lakes and sewage[J]. npj Clean Water, 2020, 3(1). DOI:10.1038/s41545-020-0050-1 |
| [3] | Cao Y R, Lin H J, Zhang K, et al. Microplastics: a major source of phthalate esters in aquatic environments[J]. Journal of Hazardous Materials, 2022, 432. DOI:10.1016/J.JHAZMAT.2022.128731 |
| [4] | Walker T R. (Micro) plastics and the UN sustainable development goals[J]. Current Opinion in Green and Sustainable Chemistry, 2021, 30. DOI:10.1016/J.COGSC.2021.100497 |
| [5] | Wu C C, Pan S D, Shan Y P, et al. Microplastics mulch film affects the environmental behavior of adsorption and degradation of pesticide residues in soil[J]. Environmental Research, 2022, 214. DOI:10.1016/J.ENVRES.2022.114133 |
| [6] |
白润昊, 崔吉晓, 范瑞琪, 等. 农田土壤地膜源微塑料分离检测方法优化[J]. 中国环境科学, 2023, 43(5): 2404-2412. Bai R H, Cui J X, Fan R Q, et al. Temporal and spatial variations of sedimentary nitrogen forms in the Lancang River and Nu River[J]. China Environmental Science, 2023, 43(5): 2404-2412. DOI:10.3969/j.issn.1000-6923.2023.05.032 |
| [7] |
严昌荣, 戚瑞敏, 薛颖昊, 等. 甘肃省中东部农户地膜应用及回收现状[J]. 农业工程学报, 2019, 35(15): 211-216. Yan C R, Qi R M, Xue Y H, et al. Application and recovery of plastic mulching film by farmers in central and eastern Gansu province[J]. Transactions of the Chinese Society of Agricultural Engineering, 2019, 35(15): 211-216. DOI:10.11975/j.issn.1002-6819.2019.15.026 |
| [8] | Lan T, Cao F, Cao L C, et al. A comparative study on the adsorption behavior and mechanism of pesticides on agricultural film microplastics and straw degradation products[J]. Chemosphere, 2022, 303. DOI:10.1016/J.CHEMOSPHERE.2022.135058 |
| [9] | Medo J, Hricáková N, Maková J, et al. Effects of sulfonylurea herbicides chlorsulfuron and sulfosulfuron on enzymatic activities and microbial communities in two agricultural soils[J]. Environmental Science and Pollution Research, 2020, 27(33): 41265-41278. DOI:10.1007/s11356-020-10063-0 |
| [10] |
杨峰山, 张瑞, 肖延臣, 等. 一株氯嘧磺隆降解菌分离鉴定及降解条件优化[J]. 生物工程学报, 2020, 36(3): 560-568. Yang F S, Zhang R, Xiao Y C, et al. Isolation and identification of a chlorimuron-ethyl-degrading bacterium and optimization of its degradation conditions[J]. Chinese Journal of Biotechnology, 2020, 36(3): 560-568. |
| [11] | Qin Z R, Zhao Z H, Xia L L, et al. Research trends and hotspots of aquatic biofilms in freshwater environment during the last three decades: a critical review and bibliometric analysis[J]. Environmental Science and Pollution Research, 2022, 29(32): 47915-47930. DOI:10.1007/s11356-022-20238-6 |
| [12] | Fierer N, Wood S A, Bueno de Mesquita C P. How microbes can, and cannot, be used to assess soil health[J]. Soil Biology and Biochemistry, 2021, 153. DOI:10.1016/j.soilbio.2020.108111 |
| [13] | Zhu Y G, Lin X Y, Chu H Y. Editorial: rhizosphere microbiome special issue[J]. Plant and Soil, 2022, 470(1-2): 1-3. DOI:10.1007/s11104-021-05280-6 |
| [14] | Yu Z X, Dai Y M, Li T T, et al. A novel pathway of chlorimuronethyl biodegradation by Chenggangzhangella methanolivorans strain CHL1 and its molecular mechanisms[J]. International Journal of Molecular Sciences, 2022, 23(17). DOI:10.3390/IJMS23179890 |
| [15] | Zhu J H, Liu S Q, Wang H Q, et al. Microplastic particles alter wheat rhizosphere soil microbial community composition and function[J]. Journal of Hazardous Materials, 2022, 436. DOI:10.1016/J.JHAZMAT.2022.129176 |
| [16] | Shahid M, Khan M S. Tolerance of pesticides and antibiotics among beneficial soil microbes recovered from contaminated rhizosphere of edible crops[J]. Current Research in Microbial Sciences, 2022, 3. DOI:10.1016/J.CRMICR.2021.100091 |
| [17] |
王艳丽, 王铭晨, 何芳, 等. 不同基因型大豆苗期与成熟期耐荫性探究[J]. 中国油料作物学报, 2023, 45(3): 558-566. Wang Y L, Wang M C, He F, et al. Shade tolerance at seedling and maturing stages of soybean with different genotypes[J]. Chinese Journal of Oil Crop Sciences, 2023, 45(3): 558-566. |
| [18] | Yu Z X, Gu W, Yang Y, et al. Whole-genome sequencing of a chlorimuron-ethyl-degrading strain: Chenggangzhangella methanolivorans CHL1 and its degrading enzymes[J]. Microbiology Spectrum, 2022, 10(4). DOI:10.1128/SPECTRUM.01822-22 |
| [19] | Kang J, Peng Y F, Xu W F. Crop root responses to drought stress: molecular mechanisms, nutrient regulations, and interactions with microorganisms in the rhizosphere[J]. International Journal of Molecular Sciences, 2022, 23(16). DOI:10.3390/IJMS23169310 |
| [20] | Pérez-Reverón R, Álvarez-Méndez S J, González-Sálamo J, et al. Nanoplastics in the soil environment: analytical methods, occurrence, fate and ecological implications[J]. Environmental Pollution, 2023, 317. DOI:10.1016/J.ENVPOL.2022.120788 |
| [21] | Ma Q Y, Han X Y, Song J L, et al. Characterization of a new chlorimuron-ethyl-degrading strain Cedecea sp. LAM2020 and biodegradation pathway revealed by multiomics analysis[J]. Journal of Hazardous Materials, 2023, 443. DOI:10.1016/J.JHAZMAT.2022.130197 |
| [22] |
纪红, 陈修文, 赵宏利, 等. 微塑料对高等植物生长发育影响研究进展[J]. 科学技术与工程, 2021, 21(18): 7415-7424. Ji H, Chen X W, Zhao H L, et al. Research advances of the influence of microplastics on the growth and development of higher plants[J]. Science Technology and Engineering, 2021, 21(18): 7415-7424. DOI:10.3969/j.issn.1671-1815.2021.18.003 |
| [23] | Wang L, Liu Y, Kaur M, et al. Phytotoxic effects of polyethylene microplastics on the growth of food crops soybean (Glycine max) and mung bean (Vigna radiata)[J]. International Journal of Environmental Research and Public Health, 2021, 18(20). DOI:10.3390/IJERPH182010629 |
| [24] | Wang J, Liu X H, Liu G N, et al. Size effect of polystyrene microplastics on sorption of phenanthrene and nitrobenzene[J]. Ecotoxicology and Environmental Safety, 2019, 173: 331-338. DOI:10.1016/j.ecoenv.2019.02.037 |
| [25] | Budhiraja V, Urh A, Horvat P, et al. Synergistic adsorption of organic pollutants on weathered polyethylene microplastics[J]. Polymers, 2022, 14(13). DOI:10.3390/polym14132674 |
| [26] | Mahnert A, Moissl-Eichinger C, Berg G. Microbiome interplay: plants alter microbial abundance and diversity within the built environment[J]. Frontiers in Microbiology, 2015, 6. DOI:10.3389/fmicb.2015.00887 |
| [27] | Barriuso J, Marín S, Mellado R P. Effect of the herbicide glyphosate on glyphosate-tolerant maize rhizobacterial communities: a comparison with pre-emergency applied herbicide consisting of a combination of acetochlor and terbuthylazine[J]. Environmental Microbiology, 2010, 12(4): 1021-1030. DOI:10.1111/j.1462-2920.2009.02146.x |
| [28] | Hou J H, Xu X J, Yu H, et al. Comparing the long-term responses of soil microbial structures and diversities to polyethylene microplastics in different aggregate fractions[J]. Environment International, 2021, 149. DOI:10.1016/J.ENVINT.2021.106398 |
| [29] | Zang H L, Liu W J, Cheng Y, et al. Bioremediation of historically chlorimuron-ethyl-contaminated soil by co-culture chlorimuronethyl-degrading bacteria combined with the spent mushroom substrate[J]. Microorganisms, 2020, 8(3). DOI:10.3390/microorganisms8030369 |
| [30] |
丁峰, 赖金龙, 季晓晖, 等. 聚乙烯微塑料对玉米根际土壤微生物群落结构的影响[J]. 中国生态农业学报(中英文), 2021, 29(6): 970-978. Ding F, Lai J L, Ji X H, et al. Effects of polyethylene microplastics on the microbial community structure of maize rhizosphere soil[J]. Chinese Journal of Eco-Agriculture, 2021, 29(6): 970-978. |
| [31] | Shi J H, Dang Q L, Zhang C Y, et al. Insight into effects of polyethylene microplastics in anaerobic digestion systems of waste activated sludge: interactions of digestion performance, microbial communities and antibiotic resistance genes[J]. Environmental Pollution, 2022, 310. DOI:10.1016/J.ENVPOL.2022.119859 |
| [32] | Liu Z Q, Li Y Z, Wang J, et al. Unraveling consequences of the co-exposure of polyethylene microplastics and acid rain on plantmicrobe-soil system[J]. Chemosphere, 2022, 307. DOI:10.1016/J.CHEMOSPHERE.2022.135941 |
| [33] | Li Q S, Wu L K, Chen J, et al. Biochemical and microbial properties of rhizospheres under maize/peanut intercropping[J]. Journal of Integrative Agriculture, 2016, 15(1): 101-110. DOI:10.1016/S2095-3119(15)61089-9 |
| [34] |
王新, 侯佳文, 宋磊, 等. 植物-微生物联合修复化学农药污染土壤的研究进展[J]. 生物安全学报, 2022, 31(3): 217-223. Wang X, Hou J W, Song L, et al. Research progress on plantmicrobe remediation of soils contaminated by chemical pesticides[J]. Journal of Biosafety, 2022, 31(3): 217-223. |
| [35] | Lee J M, Jin C Z, Kang M K, et al. Nocardioides humilatus sp. nov., isolated from farmland soil in the Republic of Korea[J]. International Journal of Systematic and Evolutionary Microbiology, 2022, 72(2). DOI:10.1099/IJSEM.0.004928 |
| [36] | Wachowska U, Irzykowski W, Jędryczka M, et al. Biological control of winter wheat pathogens with the use of antagonistic Sphingomonas bacteria under greenhouse conditions[J]. Biocontrol Science and Technology, 2013, 23(10): 1110-1122. DOI:10.1080/09583157.2013.812185 |
| [37] | Oshiki M, Toyama Y, Suenaga T, et al. N2O reduction by Gemmatimonas aurantiaca and potential involvement of Gemmatimonadetes bacteria in N2O reduction in agricultural soils[J]. Microbes and Environments, 2022, 37(2). DOI:10.1264/JSME2.ME21090 |
| [38] | Lu S H, Li X, Xi Y N, et al. Insight the roles of loosely-bound and tightly-bound extracellular polymeric substances on Cu2+, Zn2+ and Pb2+ biosorption process with Desulfovibrio vulgaris[J]. Journal of Colloid and Interface Science, 2021, 596: 408-419. DOI:10.1016/j.jcis.2021.03.152 |
| [39] |
刘柱, 南镇武, 林松明, 等. 黄河下游谷子花生间作农田土壤细菌群落结构与功能预测[J]. 环境科学, 2023, 44(8): 4575-4584. Liu Z, Nan Z W, Lin S M, et al. Soil bacterial community structure and function prediction of millet/peanut intercropping farmland in the lower yellow river[J]. Environmental Science, 2023, 44(8): 4575-4584. |
| [40] | Deng Y, Fu S D, Sarkodie E K, et al. Ecological responses of bacterial assembly and functions to steep Cd gradient in a typical Cd-contaminated farmland ecosystem[J]. Ecotoxicology and Environmental Safety, 2022, 229. DOI:10.1016/j.ecoenv.2021.113067 |
| [41] |
刘坤和, 薛玉琴, 竹兰萍, 等. 嘉陵江滨岸带不同土地利用类型对土壤细菌群落多样性的影响[J]. 环境科学, 2022, 43(3): 1620-1629. Liu K H, Xue Y Q, Zhu L P, et al. Effect of different land use types on the diversity of soil bacterial community in the coastal zone of Jialing river[J]. Environmental Science, 2022, 43(3): 1620-1629. |
| [42] |
万凌琳, 陈芷凡, 郭佳, 等. 生物共现网络原理及其在淡水生态系统评估中的应用[J]. 湖泊科学, 2022, 34(6): 1765-1787. Wan L L, Chen Z F, Guo J, et al. Principle and application of cooccurrence networks for freshwater ecosystem assessment[J]. Journal of Lake Sciences, 2022, 34(6): 1765-1787. |
| [43] | Di Cesare A, Frangipani E, Citterio B, et al. Class 1 integron and Enterococcus spp. abundances in swine farms from the"Suckling piglets" to the "Fatteners" production category[J]. Veterinary Microbiology, 2022, 274. DOI:10.1016/J.VETMIC.2022.109576 |
| [44] |
胡志娥, 肖谋良, 丁济娜, 等. 长期覆膜条件下农田土壤微生物群落的响应特征[J]. 环境科学, 2022, 43(10): 4745-4754. Hu Z E, Xiao M L, Ding J N, et al. Response characteristics of soil microbial community under long-term film mulching[J]. Environmental Science, 2022, 43(10): 4745-4754. |
| [45] |
张浩, 孙洁, 杨慧颖, 等. 高密度聚乙烯微塑料胁迫对棉花生长、枯萎病发生和根际细菌群落的影响[J]. 环境科学, 2023, 44(5): 2955-2964. Zhang H, Sun J, Yang H Y, et al. Effects of microplastic highdensity polyethylene on cotton growth, occurrence of Fusarium wilt, and rhizosphere soil bacterial community[J]. Environmental Science, 2023, 44(5): 2955-2964. |
| [46] | Zhou Y, Ma J, Yang J H, et al. Soybean rhizosphere microorganisms alleviate Mo nanomaterials induced stress by improving soil microbial community structure[J]. Chemosphere, 2023, 310. DOI:10.1016/J.CHEMOSPHERE.2022.136784 |
 2024, Vol. 45
2024, Vol. 45


