自1940年青霉素被用于临床医学以来, 抗生素在保障人类健康和促进畜牧业发展方面均发挥了不可替代的作用.然而近年来, 由于抗生素的乱用和滥用而导致的抗生素污染已引起了人们的持续关注.我国2023年3月1日起实施的《重点管控新污染物清单(2023年版)》中抗生素被列入重点管控范围, 被实施禁止、限制、限排等环境风险管控措施.此外, 由抗生素污染而导致的细菌耐药性问题也已经发展成为一个世界性的环境问题.据世界卫生组织报道, 预计到2050年, 抗生素耐药问题会导致高达1 000万人死亡.如果任其发展, 可累计造成100万亿美元的经济损失[1].调查发现, 进入环境中的抗生素会给环境中的微生物施加选择压力[2], 诱导ARBs的出现及ARGs水平基因转移(horizontal gene transfer, HGT)的发生[3], 从而对生态环境及人类健康产生威胁.基于此, 国家卫生健康委等13部门联合制定了《遏制微生物耐药国家行动计划(2022-2025年)》, 用来积极应对微生物耐药带来的挑战, 更好地保护生态环境和人民健康.
作为抗生素类药物的产地, 制药厂在抗生素生产过程中的污染问题已得到广泛关注.调查显示制药废水成分十分复杂, 除了有高浓度的抗生素残留, 大多数制药废水还含有高浓度的化学需氧量(chemical oxygen demand, COD)、含氮化合物与较高的盐度水平[4].这不仅会增加抗生素的赋存还会影响废水处理过程中ARBs与ARGs的去除效率.对于规模较大的综合性制药厂, 在生产抗生素的同时还可能会有镇痛药、抗组胺药和抗结核药等其他药物的生产, 以上药物也可能作为诱导因素促进抗生素抗性(antibiotic resistance, AR)的传播发展[5].值得注意的是, PWWTPs常被认为是制药企业控制耐药性污染向环境扩散的重要屏障, 但针对PWWTPs的调查发现, 在废水处理中各种消毒剂的使用反而有可能增加AR的传播[6].此外, 不同检测方法对相同PWWTPs中AR的检测结果也不尽相同.因此, 对制药废水中的抗生素、ARBs及ARGs的污染特征及检测手段进行调查和总结, 可提高人们对于抗生素和ARGs的赋存状态及环境风险的认知.基于此, 本文综述了近年来制药废水中的抗生素、ARBs及ARGs的污染现状, 并对诱导ARGs的环境因素进行了总结;概括了制药废水中AR评估方法的相关研究进展, 并对不同评估方法的特点及应用案例进行概述;对制药废水处理过程中抗生素及ARGs的相关去除工艺特别是深度处理工艺的实际效果进行了阐述, 以期为降低抗生素及AR的污染风险、制定抗生素及ARGs排放标准等提供理论依据.
1 制药废水中的抗生素抗性污染问题 1.1 制药废水中的抗生素残留及水质特点据国家统计局数据显示, 在2020年中国化学原料药产量已超270万t.作为制药和化工生产中的关键原材料, 水在包括生产、材料加工和冷却冲洗在内的一系列操作中均发挥着不可替代的作用, 因而生产药品的同时会产生大量的废水.据统计每生产1 t的抗生素将产生约500~6 500 m3的废水[7].作为以上废水收集处理的终端, PWWTPs得到了世界各地学者的关注, 有研究表明PWWTPs的出水中仍含有高浓度的抗生素(表 1)[8].
|
|
表 1 制药废水处理厂中抗生素浓度水平1) Table 1 Antibiotic concentration levels in pharmaceutical wastewater treatment plants |
制药工业中的废水通常来源于药物的合成和配制.以往对于制药废水中抗生素及ARGs的研究大多关注于PWWTPs的终端处理结果上, 但想要真正了解制药废水中抗生素的残留水平与AR污染情况, 必须熟悉抗生素的生产工艺及流程, 并对不同工艺流程的水质特点及影响因素进行考察.常用抗生素的生产方法主要有:微生物发酵法、化学合成法和半化学合成法, 其中以微生物发酵法居多[9].这3种抗生素生产过程产生的废水情况差异明显, 不仅如此, 在同一种药物生产方法的不同流程中水质变化也十分明显.Xue等[10]在调查中国河北某一使用发酵法生产头孢拉定的制药厂时发现, 其他车间废水中抗生素残留大都在μg·L-1水平, 然而合成车间的废水中ρ(头孢拉定)的最高值可达1 328 mg·L-1, 这可能与该工艺的提取效率相对较低有关.除了抗生素残留情况, 其他水质指标(pH、COD、BOD、TN、NH4+_N、TP和SO42-)在生产过程中也有明显的波动.例如, 制药过程中不同生产车间产生的原始废水中ρ(COD)可达4 410~40 000 mg·L-1[11].
综合来看, 常用的发酵工艺生产抗生素的废水水质主要有以下特点[4]:发酵废液因含有发酵残余基质及各种未消耗的原材料而导致其具有较高的COD、生物需氧量(biological oxygen demand, BOD)、总氮(total nitrogen, TN)和总悬浮固体(total suspended solids, TSS), 同时其中的溶解性有机氮(dissolved organic nitrogen, DON)成分复杂往往具有潜在的生物毒性[12];冲洗废水因含有抗生素降解中间产物及洗涤剂、消毒剂、表面活性剂等添加剂, 使其生物毒性较高且pH波动较大(一般在4~8)[5];此外, 母液的回收沉淀产生的高盐废水[13], 会对微生物细胞产生较大的渗透压, 使其脱水致死.以上水质指标参数对抗生素残留水平以及抗性污染产生均有重要影响, 具体内容将在本章第3节展开论述.除生产方法外, 制药废水中的抗生素残留还受许多其他因素的影响, 包括废水(进水)中抗生素本身的性质特点和废水处理系统的设计和运行特点等.Wang等[14]调查了5个大型PWWTPs中典型抗生素在出水中的残留情况.结果显示, 同一处理工艺对不同抗生素的处理效果不尽相同;不同处理工艺对相同抗生素的去除效果也各不一样.例如, 使用循环活性污泥(CASS)工艺污水厂出水中四环素(TC)和氧四环素(OTC)的去除效率分别为61.2%及98.5%.而使用膨胀颗粒污泥床+膜生物反应器工艺(EGSB + MBR)的制药污水处理厂在进水中OTC的浓度更低的情况下, 对OTC的去除率仅为90.7%.
1.2 制药废水中抗生素抗性细菌与抗性基因的污染现状自2006年, Pruden等[21]将ARGs定义为新兴污染物以来, ARBs和ARGs在各环境介质中的丰度及风险已引起了研究者的广泛关注.由于制药废水的特殊性质, 其含有的ARGs更是研究者们关注的热点.有研究将抗生素抗性细菌定义为[22]:如果某种抗生素对于菌株的最小抑菌浓度(minimum inhibitory concentration, MIC)高于其相应的亲本野生型菌株, 则认为该菌株对此抗生素具有耐药性.同样, 当一段基因的存在使细菌能够承受更高的抗生素浓度, 或当它的缺失增加了细菌对抗生素的敏感性时, 将此基因定义为耐药基因(或耐药因子), 这个定义还包括许多非移动染色体耐药基因[23].
以往的研究多关注市政污水处理厂(wastewater treatment plants, WWTPs)中抗生素的残留问题, 而对制药废水中抗生素的赋存及其引发的环境效应了解得相对欠缺[24].有研究表明, PWWTPs内高浓度的抗生素残留会对细菌的多重耐药指数(multiple antibiotic resistance, MAR, 评估分离菌株或菌群的抗生素耐药性水平的工具, 以判断环境中耐药性污染情况)产生重要影响, 使其成为多重耐药病原菌发展的“温床”[25].不仅如此, 调查显示PWWTPs中的ARGs检出率与检测浓度均高于市政污水[8], 其原因主要有以下两方面:首先, 过去制药废水与市政污水处理主要关注悬浮固体、营养物质和有机物等生化指标, 在处理工艺的设计阶段均未考虑对抗生素及耐药基因的去除[26].其次, 制药废水中高浓度的抗生素残留对ARBs有良好的筛选作用.特别是在以生物处理工艺为主体的制药废水处理中, 活性污泥或膜生物反应器提供了丰富的营养物质与高浓度的细菌密度[27], 加上持续的亚抑制浓度(低于相应药物的最小抑菌浓度)抗生素带来的选择压力, 使得ARBs逐渐成为优势菌群, 也为ARGs的水平转移创造了理想环境[28].
表 2汇总了国内外学者针对PWWTPs中AR污染情况的研究案例, 其中ARBs污染问题十分突出.不仅ARBs的检出频率很高, 同时具有多种抗生素耐药性的耐药菌也频繁出现.Zhang等[25]对中国北方某PWWTPs内及厂区周围的生物气溶胶样本的研究结果显示, 在分离菌株中45%的细菌对3种及以上抗生素具有耐药性, 并且分析出曝气装置对其附近空气中耐药病菌的检出率有重要影响, 证明了厂区内员工及附近居民感染多重耐药病菌的潜在风险.Li等[29]研究发现, 在中国辽宁省某PWWTPs的厌氧生物处理阶段, 四环素类耐药菌在总可培养菌中占比达到了89.1%.同时整体生物处理阶段出水中磺胺类抗生素耐药菌的检出丰度(5.01×104 CFU·mL-1)高于进水, 进而提出PWWTPs的生物处理可增加ARBs在环境中的富集.Obayiuwana等[30]对尼日利亚14家制药公司收集的废水混合样本使用抗生素药物的MIC对筛选出的耐药菌株进行测试, 结果显示磺胺类药物对所有菌株的MIC均大于或等于1 024 mg·L-1.更为严重的是, 由于当地缺乏有效的废水处理设施, 制药废水已成为耐药细菌的潜在“储存库”威胁当地的公众健康.
|
|
表 2 制药废水中的抗生素抗性污染情况 Table 2 Antibiotic resistance contamination in pharmaceutical wastewater |
与ARBs的污染问题相似, ARGs在制药废水中也广泛存在.Wang等[14]调查了中国北部5座PWWTPs处理废水过程中ARGs的丰度变化, 结果显示某些类型的ARGs(如:sul1、sul2、tetO、tetT、tetQ、tetW和tetM)的相对丰度在整个处理流程中不断升高, 且在最终出水和脱水污泥中仍然保持较高浓度.Guo等[26]对中国东南部的6座PWWTPs废水中的ARGs进行调查也得出了相似的结论, 不仅如此, 其中使用发酵工艺制药所产生废水中的ARGs浓度显著高于化学合成法制药废水中的ARGs.6座PWWTPs中共有5种不同的生物处理工艺, 而使用移动床生物膜反应器(moving bed biofilm reactor, MBBR)工艺的废水处理厂, 在其生物处理单元之后ARGs的浓度显著下降, 而在其余4种生物处理工艺(内循环厌氧反应器耦合序批式反应器、厌氧+好氧反应池、A/A/O和深井曝气池+活性污泥池)后ARGs的丰度并无显著变化.因此, 不同的制药流程及不同的废水处理工艺都可显著影响出水中ARGs的类型及丰度.更多相关的调查检测数据汇总于表 2, 综合不同案例的调查结果, PWWTPs对ARGs的处理效果在不同的研究对象中各有差异, 但均出现在最终脱水污泥中ARGs的丰度远高于水中丰度的情况, 由此说明经PWWTPs处理后制药废水中大多数的ARGs仅是从废水向污泥中的转移而未达到真正去除的目的, 这也使得PWWTPs成为ARBs及ARGs向环境传播的一个重要的点源.
值得注意的是, 根据所处环境不同, 耐药基因主要有两种存在形式, 即存在于细菌细胞内的胞内DNA(iDNA)和游离态的胞外DNA(eDNA)[35].iDNA携带的iARGs因为有细菌细胞壁和细胞膜的保护作用, 在污水处理厂的消毒处理中其结构较难被破坏[36].但是也有研究发现eARGs虽然裸露在环境中, 却仍具有环境持久性, 存在周期可达数月之久[37].因此, 在研究水环境中ARGs的种类与丰度时, 研究者往往使用0.22 μm的无菌滤膜对环境样本中的基因组进行分离, 以达到区分鉴定iARGs与eARGs的目地[38].上文所举几个案例的研究者大多未对此进行有意的区别, 但排放到环境中的eARGs可以再次被细菌直接吸收, 导致ARG在环境中进一步繁殖[39].因此, 今后的研究应更加注意区分两种形式ARGs, 特别是eARGs的去除方式及效率, 以降低潜在的风险.
1.3 诱导抗生素耐药基因的环境因素如前述, 制药废水往往成分较为复杂, 复杂的环境条件会对AR的产生、扩散等过程造成影响.截至目前, 针对AR在制药废水中传播扩散的环境影响因素的研究比较有限, 但是世界各地学者对WWTPs水体环境中诱导AR的环境因素(例如各种重金属离子、除抗生素以外的有机物和各种水质指标)已经做了充分的研究, 这可为进一步研究制药废水环境中耐药性的控制提供帮助.
1.3.1 抗生素的影响已有研究表明抗生素的过度使用和滥用是导致产生AR问题的关键.细菌通过染色体基因突变获得的ARGs往往取决于环境中的抗生素的存在及其浓度水平[40], 而PWWTPs中大量的抗生素残留自然为ARBs与ARGs的出现创造了条件.Guo等[26]对6座PWWTPs中极高浓度的抗生素(远高于相应药物的MIC)与ARGs丰度的相关性进行了分析, 结果证实了林可胺类、喹诺酮类、头孢菌素和氨基糖苷类抗生素与对应的耐药基因之间均具有显著的正相关.与此类似, Wang等[14]在选定的PWWTPs的进水和出水中检测到高浓度的抗生素残留, 通过偏相关分析显示出水ARGs的相对丰度与抗生素浓度显著相关.上述研究结果证明了含有抗生素残留的水体是抗生素抗性传播的重要途径, 特别是PWWTPs中的废水.虽然使用相关性分析不能直接证明抗生素与ARGs丰度间的因果关系, 但此类研究表明PWWTPs中的高浓度抗生素残留可能会影响相关ARGs的增殖和赋存.
1.3.2 重金属的影响除了抗生素浓度这一影响因素以外, 环境中重金属和其他选择压力源的存在也是导致ARGs数量增加的重要原因[41].重金属和抗生素复合污染已成为各环境介质中的一种普遍现象[42], 二者复合污染的健康效应以及它们对ARGs的丰度、多样性、传播扩散等过程的影响, 一直是大家关注的重点.与抗生素不同, 金属不会被降解, 并倾向于向环境微生物积累, 因此在环境中会导致长期的选择压力[43].Chen[44]观察到污泥中的重金属和抗生素均与ARGs之间存在显著的相关性, 特别是锌、铅和四环素.Gao等[45]调查一个WWTPs时也发现, 锌和铅的浓度与导致对红霉素耐药的ARGs(ereB、mefA、mefE和ermB)的拷贝数呈显著正相关.也有例外, Wang等[46]调查发现与单独使用Cu(Ⅱ)或四环素相比, 长期接触Cu(Ⅱ)和四环素会导致经序列间歇式活性污泥法(sequencing batch reactor, SBR)处理的废水中ARGs的丰度降低.但除去少数的研究个例, 重金属与抗生素对于细菌ARGs表达的协同作用已经得到了充分的实验论证[47, 48].因此, 鉴于市政污水中的大量研究结果, 在PWWTPs环境中可能存在的重金属与抗生素复合污染对于AR的污染问题, 需要引起更多学者的关注和进一步针对性的探究.
1.3.3 其他环境条件的影响环境条件同样会影响ARGs的产生.Li等[29]对中国辽宁省某PWWTPs中ARGs进行了动态追踪, 冗余分析(redundancy analysis, RDA)显示四环素ARGs(tetW)与水样中TN和DO呈正相关, 此外该作者还观察到了tetB、sul1、sul2和gyrA基因与pH和电导率(EC)呈正相关, 证实了制药废水环境不同理化性质对于ARGs的影响.鉴于PWWTPs水样的复杂性, 在不同的样本环境利用不同的分析考察方式以及不同的细菌耐药表型都会对调查结果产生重要影响, 因而还需要大量的研究针对PWWTPs的废水环境条件展开调查, 为更加全面地了解PWWTPs中AR问题提供参考.同样的, 各类相关性分析虽不能直接证明以上环境条件变化与AR之间的因果关系, 但以上研究可以说明, 一些水质参数可能作为驱动因素, 并在PWWTPs中ARGs的持久性和增殖中发挥重要作用.
2 制药废水中抗生素抗性的检测方法ARBs与ARGs作为新兴的环境污染物已为大家所共识[21], 但现阶段人们对于这类污染物健康风险的认知仍较为浅薄.探明制药废水中AR的污染水平及生态效应, 首先亟需确定ARBs与ARGs在制药废水环境中出现的频次及丰度.目前评估水环境中AR的方法主要有两大类, 一类是基于传统培养的方法, 另一类是基于分子生物学的方法[49].这两类方法各有优缺点, 前者主要用于水中抗生素抗性的分型表征, 后者主要用以研究ARGs和ARBs的种类与丰度.
基于培养的方法是针对ARBs进行选择性培养的一种较为传统的手段.该方法首先对目标菌株的抗性表型进行表征, 再进一步评价细菌对抗生素药物的耐药能力.现今关于细菌耐药性的培养研究大多参考美国的临床与实验室标准协会(Clinical and Laboratory Standards Institute, CLSI)或者欧盟的欧洲抗生素敏感性测试委员会(European Committee on Antibiotic Susceptibility Testing, EUCAST)所提供的标准方法.细菌药敏试验中使用最广泛的方法为纸片扩散法与微量肉汤稀释法, 前者主要用以表征分离得到的单一菌种抗性, 后者则主要用以测定目标菌株的具体耐药性能, 如抗生素MIC和最小杀菌浓度(minimum bactericidal concentration, MBC)的测定.虽然不依赖于传统培养的方法近年来发展迅速并在环境领域耐药性水平的检测中大量应用, 但世界各地的学者还是会选择基于培养的方法对环境样本中耐药性流行率和耐药性模式进行初步判定.前文在讨论制药废水中耐药细菌的污染现状时, 所列举的各研究案例中学者们都用到了筛选培养、纸片扩散法和微量肉汤稀释法等培养方法.基于培养的方法最大的优点是可以分离出具有抗生素耐药性的菌株, 并对其抗性表型与耐药能力进行表征, 但从环境样本中进行耐药菌株的筛选培养, 分离纯化与鉴定保存等工作繁琐耗时[50].此外, 有研究表明, 自然界中绝大多数微生物尚不能利用现有的培养技术分离获得, 目前利用传统的培养分离方法分离鉴定出的微生物只占环境中全部微生物种类的0.1%~10%[51], 并且即使是携带了ARGs的菌株在某些条件下也可能不表达抗性, 这就使得仅基于培养方法研究环境中的ARBs和ARGs存在有一定的偏差[49].
基于分子生物学的方法可避免传统培养方法的某些弊端, 可从分子水平上更加客观、准确地揭示环境中抗性基因的存在现状.基于培养的方法其研究的对象主要为ARBs, 而基于分子生物学的方法则主要是从ARGs的角度分析环境中抗生素抗性水平的高低.用于快速检测和鉴定环境样本或ARBs中ARGs的分子生物学技术大多是以聚合酶链式反应(polymerase chain reaction, PCR)方法为基础进行的, 一般认为, 基于PCR的方法分析抗性水平时, 是基于携带有ARGs即具有抗生素抗性这一基本假设的[49].主要检测方法包括:PCR、实时荧光定量PCR(real-time quantitative polymerase chain reaction, qPCR)、16S rRNA基因测序技术、多位点序列分型技术、基因外重复回文序列PCR、PCR-变性梯度凝胶电泳和限制性片段长度多态性聚合酶链反应等, 它们在ARBs菌种鉴定及ARGs丰度检测中均发挥了重要作用.
基于PCR检测技术直接检测制药废水中耐药性的方法主要依赖于已知ARGs的现有引物, 不适合发现环境中未被发现的或者新出现的ARGs.高通量测序技术(又称为下一代测序技术)的出现克服了基于PCR方法的这一缺点, 不仅如此, 它还是研究微生物群落结构多样性、基因组多样性和进行环境样本ARGs的广谱筛选的重要手段.此外, 近年来越来越多的学者运用三代测序技术进行了环境耐药性的测试研究, 三代测序技术主要的特点是:读长变长、程序流程简化、避免PCR扩增偏好和可直接测定碱基上的修饰情况[52].此外, 基于拉曼光谱技术发展起来的高通量染色体构象捕获技术、单细胞拉曼光谱、表面增强拉曼光谱和显微共聚焦拉曼技术等各种手段也在环境耐药性研究领域不断应用.表 3汇总了基于高通量测序技术和基于拉曼光谱技术的各种分子生物学方法在耐药性研究中的应用.
|
|
表 3 分子生物技术在环境耐药性研究领域的应用 Table 3 Applications of molecular biotechnology in the field of environmental drug resistance research |
3 制药废水处理中抗生素抗性的控制研究
如前述, 制药废水通常含有极高浓度的抗生素、ARBs和ARGs.然而, 传统的PWWTPs并不是专门为去除以上新兴污染物而设计的.因此, 目前的PWWTPs对抗生素、ARBs和ARGs的去除效率通常较低.同时, 越来越多的证据表明, 在WWTPs和PWWTPs中广泛应用的生物处理工艺可能促进了废水处理过程中ARBs和ARGs的增殖, 同时吸附在剩余污泥中的抗生素外排同样也伴随着环境风险[65], 从而促进了抗生素耐药性的传播扩散.要想减少在生物处理环节大量的ARBs的出现以及ARGs的污染, 在废水进入生物处理单元之前对抗生素的量进行控制十分关键[66], 如使用膜滤、人工湿地和絮凝等工艺在进入生物处理单元之前从废水中捕获/拦截ARBs与ARGs.此外, 现阶段在污水处理系统的终末阶段会补充各种高级氧化工艺(advanced oxidation processes, AOPs)等深度处理技术以增强对抗生素、ARBs和ARGs的去除效率.以往的研究表明, 在各种AOPs(如紫外线照射、臭氧氧化、氯消毒、Fenton和Fenton/UV处理)中产生的大量活性氧(reactive oxygen species, ROS)不仅可以有效地破坏抗生素, 还可以通过破坏ARBs的核酸和细胞膜等而使其失活(如图 1).
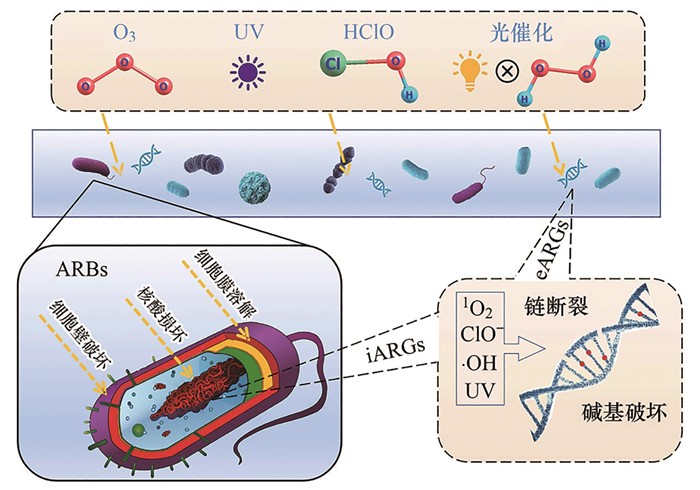
|
图 1 不同深度处理技术(臭氧化消毒、氯化消毒、紫外线照射和光催化氧化)去除抗生素抗性细菌以及抗生素抗性基因的机制 Fig. 1 Mechanisms of removal of antibiotic resistant bacteria and antibiotic resistance genes using different deep treatment technologies (ozonation disinfection, chlorination disinfection, UV irradiation, and photocatalytic oxidation) |
为了提高制药废水中抗生素、ARBs及ARGs的去除效率, 近年来研究者们又进一步研究出了不同的制药废水新处理技术.例如有报道称混合处理技术以及两种或两种以上技术的集成是未来强化处理抗生素废水技术发展的焦点, 对于废水中抗生素的去除具有良好的应用前景[67].
Hou等[68]设计了上流式厌氧污泥床(UASB)、缺氧-好氧池(A/O)与4个单独的AOPs(UV、臭氧化、Fenton和Fenton/UV)的联用体系, 可高效去除供试制药废水中所含的18种抗生素和10种ARGs.此外, 其研究结果证实该联用体系中的生物处理单元对废水中抗生素的去除贡献较大, 而ARGs主要由AOPs清除.生物处理单元是目前常用的去除抗生素的手段, 具有成本低、普适性高等特点.然而, 由于部分抗生素通常不可生物降解, 同时抗生素及其降解产物对细菌的生长均有抑制作用, 因此某些抗生素的生物去除是不稳定和不充分的.电芬顿(EF)工艺已被证明对不同类药物均有较好的降解效率[69, 70], 但相对较高的能源消耗限制了其进一步的发展和应用.将二者联合使用的生物-电芬顿(bio-EF)技术可以通过结合两者的优势来产生协同效应从而在提高抗生素去除效率的同时也可较好地控制运行成本[71].Valério等[72]研究了光催化与臭氧氧化的结合技术对四环素类抗生素的降解效率.结果显示, 与单独使用光催化或臭氧化处理抗生素相比, 将二者联用后, 废水中的四环素降解效率可达100%, 降解动力学也比二者单独处理过程的总和提高20%.
此外, 还有将传统膜技术与光芬顿反应结合[73]、催化剂与光芬顿技术结合[74]以及纳米零价铁(nZVI)与UV或H2O2[75]联用等技术的发明可使用.以上混合联用技术均可为提高制药废水中抗生素、ARBs及ARGs的去除提供新的思路及依据.
4 展望有研究显示PWWTPs的脱水污泥中含有数量惊人的ARGs, 由此可见现阶段PWWTPs对耐药性污染的处理往往限于从水中拦截ARGs, 并未达到真正去除的效果.此外, 不仅是PWWTPs的生物处理阶段有ARGs和ARBs的增殖现象, 在氯消毒的过程中大量ARGs的增殖问题也应引起重视, 因而需要进一步研究耐药性污染在PWWTPs中的传播扩散途径.
结合运用基于培养的方法与基于分子生物学技术的方法, 已经成为近年来国内外学者探究环境耐药性污染问题的重要手段.随着分子生物学技术的不断进步, 高通量测序的检测成本不断下降, 未来基于二代或者三代检测技术进行的宏基因组测序工作会对PWWTPs耐药性污染的风险评估、大尺度迁移传播和废水环境中的微生物群落结构与耐药基因的相关性等研究有重要的帮助.此外, 结合拉曼光谱技术对废水中耐药菌的快速分类鉴定未来也会有可观的应用场景.在进行ARGs的转化或结合转移等实验时, 也需要关注ARGs的不同类型对实验结果的差异性.例如对不同抗生素的ARGs或者拥有不同耐药机制的ARGs;同时, iARGs与eARGs也需要进一步区分实验, 因为一些结果表明未设置深度处理的传统处理工艺中eARGs往往更难被去除, 并可在自然环境中保持数月之久或被其他细菌吸收从而产生潜在生态风险.
对于ARBs及ARGs的处理工艺来说, 现有的深度处理工艺在PWWTPs控制耐药性污染中发挥着重要作用, 可以实现废水中ARGs和ARBs的有效去除, 但在氯化消毒与臭氧化消毒出水中ARGs和ARBs的丰度升高的问题仍然不可忽视.另外, 光催化氧化等工艺研究大多仍停留在实验室阶段, 未来还需要进一步推进实际应用的研究, 同时进行新型处理工艺的开发和应用.
此外, 尽管污水处理厂对于ARGs和ARBs具备一定的处理效果, 但是出水中抗生素残留仍然是诱导受纳水体AR传播扩散的关键所在, 因而降低ARBs的环境选择压力, 即减少抗生素的残留对于控制PWWTPs中耐药性问题的发展至关重要.过往针对PWWTPs的研究中大多仅关注了抗生素浓度的变化, 但是抗生素浓度的减少并不代表废水中抑菌活性等的比例减少, 其对抗生素耐药性的诱导作用可能依然存在, 抗生素与抗性基因之间剂量-效应关系目前也并不清楚.未来研究需要更多同时关注抗生素中间产物、前驱体以及生产副产物的去除效果等, 此外还应尽快明确抗生素与抗性基因之间剂量-效应的关系以期能更有效地评估抗生素及ARGs的生态风险.
进入排水系统前对制药废水进行生产源处理也可以有效清除抗生素、降低ARBs或ARGs的浓度.可采用生命周期评价方法, 定量研究制药全过程对环境影响的负荷, 以使大家更为彻底、全面、综合地了解制药生产全过程中对环境的不利影响, 从而可采取相应管理和处置措施现场减排, 以降低该过程的环境生态风险.此外, 在对全过程分析理解的基础上, 可以使用循环经济生产模式, 在抗生素产生的源头应用膜(超滤膜、纳滤膜和反渗透膜等)等回收技术, 将部分回收的物质加以利用, 以提高药物生产过程的中废物减量化的比例, 从而降低对环境的影响.
5 结论国家多部委在《重点管控新污染物清单(2023年版)》中将抗生素列为第一类需要管控的新污染物.抗性基因能够使细菌产生对多种抗生素药物的耐药性, 近些年来也受到广泛关注, 并被看作为一类新兴环境污染物.PWWTPs被认为是抗生素抗性污染向环境传播的点源之一, 对制药废水中抗生素及耐药情况进行探究可提高人们对抗生素及ARGs这类新污染物的认知.综述了PWWTPs环境中的抗生素残留、ARBs和ARGs的污染现状, 对抗生素抗性的检测方法和控制手段进行了概述, 同时对未来制药废水中抗生素抗性的研究内容和重点进行了展望, 可为抗生素及ARGs这类新污染的环境归趋研究及控制去除手段的选择提供一定理论参考.
| [1] | Roope L S, Smith R D, Pouwels K B, et al. The challenge of antimicrobial resistance: What economics can contribute[J]. Science, 2019, 364(6435). DOI:10.1126/science.aau4679 |
| [2] | Hu Y R, Jiang L, Sun X Y, et al. Risk assessment of antibiotic resistance genes in the drinking water system[J]. Science of the Total Environment, 2021, 800. DOI:10.1016/j.scitotenv.2021.149650 |
| [3] | Hu Y R, Jiang L, Zhang T Y, et al. Occurrence and removal of sulfonamide antibiotics and antibiotic resistance genes in conventional and advanced drinking water treatment processes[J]. Journal of Hazardous Materials, 2018, 360: 364-372. DOI:10.1016/j.jhazmat.2018.08.012 |
| [4] | Shi X Q, Leong K Y, Ng H Y. Anaerobic treatment of pharmaceutical wastewater: A critical review[J]. Bioresource Technology, 2017, 245: 1238-1244. DOI:10.1016/j.biortech.2017.08.150 |
| [5] | Gadipelly C, Pérez-González A, Yadav G D, et al. Pharmaceutical industry wastewater: Review of the technologies for water treatment and reuse[J]. Industrial & Engineering Chemistry Research, 2014, 53(29): 11571-11592. |
| [6] | Liu S S, Qu H M, Yang D, et al. Chlorine disinfection increases both intracellular and extracellular antibiotic resistance genes in a full-scale wastewater treatment plant[J]. Water Research, 2018, 136: 131-136. DOI:10.1016/j.watres.2018.02.036 |
| [7] | GB 21903-2008, 发酵类制药工业水污染物排放标准[S]. |
| [8] | Wang K, Zhuang T, Su ZX, et al. Antibiotic residues in wastewaters from sewage treatment plants and pharmaceutical industries: Occurrence, removal and environmental impacts[J]. Science of the Total Environment, 2021, 788. DOI:10.1016/j.scitotenv.2021.147811 |
| [9] |
杨军, 陆正禹, 胡纪萃, 等. 抗生素工业废水生物处理技术的现状与展望[J]. 环境科学, 1997, 18(3): 83-85. Yang J, Lu Z Y, Hu J C, et al. Advances in biological treatmemt processes of antibiotic production wastewater[J]. Environmental Science, 1997, 18(3): 83-85. DOI:10.3321/j.issn:0250-3301.1997.03.025 |
| [10] | Xue J J, Lei D D, Zhao X M, et al. Antibiotic residue and toxicity assessment of wastewater during the pharmaceutical production processes[J]. Chemosphere, 2022, 291. DOI:10.1016/j.chemosphere.2021.132837 |
| [11] | Cetecioglu Z, Ince B, Gros M, et al. Biodegradation and reversible inhibitory impact of sulfamethoxazole on the utilization of volatile fatty acids during anaerobic treatment of pharmaceutical industry wastewater[J]. Science of the Total Environment, 2015, 536: 667-674. DOI:10.1016/j.scitotenv.2015.07.139 |
| [12] | Hu H D, Jiang C, Ma H J, et al. Removal characteristics of don in pharmaceutical wastewater and its influence on the N-nitrosodimethylamine formation potential and acute toxicity of DOM[J]. Water Research, 2017, 109: 114-121. DOI:10.1016/j.watres.2016.10.010 |
| [13] | Shi X Q, Ng K K, Li X R, et al. Investigation of intertidal wetland sediment as a novel inoculation source for anaerobic saline wastewater treatment[J]. Environmental Science & Technology, 2015, 49(10): 6231-6239. |
| [14] | Wang J L, Mao D Q, Mu Q H, et al. Fate and proliferation of typical antibiotic resistance genes in five full-scale pharmaceutical wastewater treatment plants[J]. Science of the Total Environment, 2015, 526: 366-373. DOI:10.1016/j.scitotenv.2015.05.046 |
| [15] | Guo X Y, Yan Z, Zhang Y, et al. Removal mechanisms for extremely high-level fluoroquinolone antibiotics in pharmaceutical wastewater treatment plants[J]. Environmental Science and Pollution Research, 2017, 24(9): 8769-8777. DOI:10.1007/s11356-017-8587-3 |
| [16] | Larsson D G J, De Pedro C, Paxeus N. Effluent from drug manufactures contains extremely high levels of pharmaceuticals[J]. Journal of Hazardous Materials, 2007, 148(3): 751-755. DOI:10.1016/j.jhazmat.2007.07.008 |
| [17] | Hou J, Wang C, Mao D Q, et al. The occurrence and fate of tetracyclines in two pharmaceutical wastewater treatment plants of Northern China[J]. Environmental Science and Pollution Research, 2016, 23: 1722-1731. DOI:10.1007/s11356-015-5431-5 |
| [18] | Tahrani L, Van Loco J, Mansour H B, et al. Occurrence of antibiotics in pharmaceutical industrial wastewater, wastewater treatment plant and sea waters in Tunisia[J]. Journal of Water & Health, 2016, 14(2): 208-213. DOI:10.3969/j.issn.1005-8443.2016.02.027 |
| [19] | Pan C Y, Bao Y Y, Xu B T. Seasonal variation of antibiotics in surface water of Pudong new area of shanghai, China and the occurrence in typical wastewater sources[J]. Chemosphere, 2020, 239. DOI:10.1016/j.chemosphere.2019.124816 |
| [20] | Sim W J, Lee J W, Lee E S, et al. Occurrence and distribution of pharmaceuticals in wastewater from households, livestock farms, hospitals and pharmaceutical manufactures[J]. Chemosphere, 2011, 82(2): 179-186. DOI:10.1016/j.chemosphere.2010.10.026 |
| [21] | Pruden A, Pei R T, Storteboom H, et al. Antibiotic resistance genes as emerging contaminants: Studies in northern Colorado[J]. Environmental Science & Technology, 2006, 40(23): 7445-7450. |
| [22] | Alekshun M N, Levy S B. Molecular mechanisms of antibacterial multidrug resistance[J]. Cell, 2007, 128(6): 1037-1050. DOI:10.1016/j.cell.2007.03.004 |
| [23] | Martínez J L, Coque T M, Baquero F. What is a resistance gene? Ranking risk in resistomes[J]. Nature Reviews Microbiology, 2015, 13(2): 116-123. DOI:10.1038/nrmicro3399 |
| [24] | Wang G A, Zhou S H, Han X K, et al. Occurrence, distribution, and source track of antibiotics and antibiotic resistance genes in the main rivers of Chongqing city, southwest China[J]. Journal of Hazardous Materials, 2020, 389. DOI:10.1016/j.jhazmat.2020.122110 |
| [25] | Zhang M Y, Zuo J E, Yu X, et al. Quantification of multi-antibiotic resistant opportunistic pathogenic bacteria in bioaerosols in and around a pharmaceutical wastewater treatment plant[J]. Journal of Environmental Sciences, 2018, 72: 53-63. DOI:10.1016/j.jes.2017.12.011 |
| [26] | Guo X Y, Yan Z, Zhang Y, et al. Behavior of antibiotic resistance genes under extremely high-level antibiotic selection pressures in pharmaceutical wastewater treatment plants[J]. Science of the Total Environment, 2018, 612: 119-128. DOI:10.1016/j.scitotenv.2017.08.229 |
| [27] | Sørensen S J, Bailey M, Hansen L H, et al. Studying plasmid horizontal transfer in situ: A critical review[J]. Nature Reviews Microbiology, 2005, 3(9): 700-710. DOI:10.1038/nrmicro1232 |
| [28] | Auerbach E A, Seyfried E E, Mcmahon K D. Tetracycline resistance genes in activated sludge wastewater treatment plants[J]. Water Research, 2007, 41(5): 1143-1151. DOI:10.1016/j.watres.2006.11.045 |
| [29] | Li L X, Guo C S, Fan S S, et al. Dynamic transport of antibiotics and antibiotic resistance genes under different treatment processes in a typical pharmaceutical wastewater treatment plant[J]. Environmental Science and Pollution Research, 2018, 25(30): 30191-30198. DOI:10.1007/s11356-018-2913-2 |
| [30] | Obayiuwana A, Ogunjobi A, Yang M, et al. Characterization of bacterial communities and their antibiotic resistance profiles in wastewaters obtained from pharmaceutical facilities in Lagos and Ogun states, Nigeria[J]. International Journal of Environmental Research and Public Heal, 2018, 15(7). DOI:10.3390/ijerph15071365 |
| [31] | Tahrani L, Soufi L, Mehri I, et al. Isolation and characterization of antibiotic-resistant bacteria from pharmaceutical industrial wastewaters[J]. Microbial Pathogenesis, 2015, 89: 54-61. DOI:10.1016/j.micpath.2015.09.001 |
| [32] | Obayiuwana A, Ibekwe A M. Antibiotic resistance genes occurrence in wastewaters from selected pharmaceutical facilities in Nigeria[J]. Water, 2020, 12(7). DOI:10.3390/w12071897 |
| [33] | Zhai W C, Yang F X, Mao D Q, et al. Fate and removal of various antibiotic resistance genes in typical pharmaceutical wastewater treatment systems[J]. Environmental Science and Pollution Research International, 2016, 23(12): 12030-12038. DOI:10.1007/s11356-016-6350-9 |
| [34] | Tao W D, Zhang X X, Zhao F Z, et al. High levels of antibiotic resistance genes and their correlations with bacterial community and mobile genetic elements in pharmaceutical wastewater treatment bioreactors[J]. PLoS One, 2016, 11(6). DOI:10.1371/journal.pone.0156854 |
| [35] | Dong P Y, Wang H, Fang T T, et al. Assessment of extracellular antibiotic resistance genes (eARGs) in typical environmental samples and the transforming ability of eARG[J]. Environment International, 2019, 125: 90-96. DOI:10.1016/j.envint.2019.01.050 |
| [36] | Zhang Y N, Zhang T T, Liu H Y, et al. Simulated sunlight-induced inactivation of tetracycline resistant bacteria and effects of dissolved organic matter[J]. Water Research, 2020, 185. DOI:10.1016/j.watres.2020.116241 |
| [37] | Salyers A A, Amábile-Cuevas C F. Why are antibiotic resistance genes so resistant to elimination?[J]. Antimicrobial Agents and Chemotherapy, 1997, 41(11): 2321-2325. DOI:10.1128/AAC.41.11.2321 |
| [38] |
郭行磐. 长江口滨岸水环境中抗生素抗性基因的赋存特征[D]. 上海: 华东师范大学, 2019. Guo X P. Occurrence and abundance of antibiotic resistance genes in aquatic environments along the Yangtze Estuary[D]. Shanghai: East China Normal University, 2019. |
| [39] | Wang J L, Chen X Y. Removal of antibiotic resistance genes (ARGs) in various wastewater treatment processes: An overview[J]. Critical Reviews in Environmental Science and Technology, 2022, 52(4): 571-630. DOI:10.1080/10643389.2020.1835124 |
| [40] | Zhang Q C, Lambert G, Liao D, et al. Acceleration of emergence of bacterial antibiotic resistance in connected microenvironments[J]. Science, 2011, 333(6050): 1764-1767. DOI:10.1126/science.1208747 |
| [41] | Gullberg E, Albrecht L M, Karlsson C, et al. Selection of a multidrug resistance plasmid by sublethal levels of antibiotics and heavy metals[J]. mBio, 2014, 5(5). DOI:10.1128/mBio.01918-14 |
| [42] | Lindberg R H, Björklund K, Rendahl P, et al. Environmental risk assessment of antibiotics in the Swedish environment with emphasis on sewage treatment plants[J]. Water Research, 2007, 41(3): 613-619. DOI:10.1016/j.watres.2006.11.014 |
| [43] | Bondarczuk K, Markowicz A, Piotrowska-Seget Z. The urgent need for risk assessment on the antibiotic resistance spread via sewage sludge land application[J]. Environment International, 2016, 87: 49-55. DOI:10.1016/j.envint.2015.11.011 |
| [44] | Chen C Y. DNA polymerases drive DNA sequencing-by-synthesis technologies: Both past and present[J]. Frontiers in Microbiology, 2014, 5. DOI:10.3389/fmicb.2014.00305 |
| [45] | Gao P, He S, Huang S L, et al. Impacts of coexisting antibiotics, antibacterial residues, and heavy metals on the occurrence of erythromycin resistance genes in urban wastewater[J]. Applied Microbiology and Biotechnology, 2015, 99(9): 3971-3980. DOI:10.1007/s00253-015-6404-9 |
| [46] | Wang Z C, Xia P H, Gao M C, et al. Long-term effects of combined divalent copper and tetracycline on the performance, microbial activity and community in a sequencing batch reactor[J]. Bioresource Technology, 2018, 249: 916-923. DOI:10.1016/j.biortech.2017.11.006 |
| [47] | Pal C, Asiani K, Arya S, et al. Metal resistance and its association with antibiotic resistance[M]. Amsterdam: Elsevier, 2017. |
| [48] | Mazhar S H, Li X J, Rashid A, et al. Co-selection of antibiotic resistance genes, and mobile genetic elements in the presence of heavy metals in poultry farm environments[J]. Science of the Total Environment, 2021, 755. DOI:10.1016/j.scitotenv.2020.142702 |
| [49] | Rizzo L, Manaia C, Merlin C, et al. Urban wastewater treatment plants as hotspots for antibiotic resistant bacteria and genes spread into the environment: A review[J]. Science of the Total Environment, 2013, 447: 345-360. DOI:10.1016/j.scitotenv.2013.01.032 |
| [50] | Figueira V, Serra E, Manaia C M. Differential patterns of antimicrobial resistance in population subsets of Escherichia coli isolated from waste- and surface waters[J]. Science of the Total Environment, 2010, 409(6): 1017-1023. |
| [51] | Amann R I, Ludwig W, Schleifer K H. Phylogenetic identification and in situ detection of individual microbial cells without cultivation[J]. Microbiological Reviews, 1995, 59(1): 143-169. DOI:10.1128/mr.59.1.143-169.1995 |
| [52] |
王玉静, 陆梓涔, 陈俊煜, 等. 高通量测序技术的发展及其在临床检测中的应用[J]. 厦门大学学报(自然科学版), 2021, 60(5): 811-820. Wang Y J, Lu Z C, Chen J Y, et al. Development of the high-throughput sequencing technology and its application in clinical detection[J]. Journal of Xiamen University (Natural Science), 2021, 60(5): 811-820. |
| [53] | Schürch A C, Arredondo-Alonso S, Willems R J L, et al. Whole genome sequencing options for bacterial strain typing and epidemiologic analysis based on single nucleotide polymorphism versus gene-by-gene-based approaches[J]. Clinical Microbiology and Infection, 2018, 24(4): 350-354. DOI:10.1016/j.cmi.2017.12.016 |
| [54] | Kuroda M, Ohta T, Uchiyama I, et al. Whole genome sequencing of meticillin-resistant Staphylococcus aureus [J]. The Lancet, 2001, 357(9264): 1225-1240. DOI:10.1016/S0140-6736(00)04403-2 |
| [55] |
王卓, 申笑涵, 施奇惠. 单细胞基因组测序技术新进展及其在生物医学中的应用[J]. 遗传, 2021, 43(2): 108-117. Wang Z, Shen X H, Shi Q H. Advances in single-cell whole genome sequencing technology and its application in biomedicine[J]. Hereditas, 2021, 43(2): 108-117. DOI:10.3760/cma.j.cn511374-20200216-00083 |
| [56] |
黄志强, 邱景璇, 李杰, 等. 基于16s rRNA基因测序分析微生物群落多样性[J]. 微生物学报, 2021, 61(5): 1044-1063. Huang Z Q, Qiu J X, Li J, et al. Exploration of microbial diversity based on 16S rRNA gene sequence analysis[J]. Acta Microbiologica Sinica, 2021, 61(5): 1044-1063. |
| [57] | Dai D J, Brown C, Bürgmann H, et al. Long-read metagenomic sequencing reveals shifts in associations of antibiotic resistance genes with mobile genetic elements from sewage to activated sludge[J]. Microbiome, 2022, 10(1). DOI:10.1186/s40168-021-01216-5 |
| [58] | Jiang X W, Cui X J, Xu H, et al. Whole genome sequencing of extended-spectrum beta-lactamase (ESBL)-producing Escherichia coli isolated from a wastewater treatment plant in China[J]. Frontiers in Microbiology, 2019, 10. DOI:10.3389/fmicb.2019.01797 |
| [59] |
徐菲, 杨凯, 朱龙吉, 等. 土壤病原微生物检测技术研究进展[J]. 农业环境科学学报, 2022, 41(12): 2593-2603. Xu F, Yang K, Zhu L J, et al. Advances in the detection technology of pathogenic microorganisms in soil[J]. Journal of Agro-Environment Science, 2022, 41(12): 2593-2603. DOI:10.11654/jaes.2022-1012 |
| [60] | Yang K, Chen Q L, Chen M L, et al. Temporal dynamics of antibiotic resistome in the plastisphere during microbial colonization[J]. Environmental Science & Technology, 2020, 54(18): 11322-11332. |
| [61] | Li H Z, Zhang D D, Yang K, et al. Phenotypic tracking of antibiotic resistance spread via transformation from environment to clinic by reverse D2O single-cell Raman probing[J]. Analytical Chemistry, 2020, 92(23): 15472-15479. DOI:10.1021/acs.analchem.0c03218 |
| [62] | Knauer M, Ivleva N P, Niessner R, et al. A flow-through microarray cell for the online SERS detection of antibody-captured E. coli bacteria[J]. Analytical and Bioanalytical Chemistry, 2012, 402(8): 2663-2667. DOI:10.1007/s00216-011-5398-0 |
| [63] | Cui L, Zhang Y J, Huang W E, et al. Surface-enhanced Raman spectroscopy for identification of heavy metal arsenic(v)-mediated enhancing effect on antibiotic resistance[J]. Analytical Chemistry, 2016, 88(6): 3164-3170. DOI:10.1021/acs.analchem.5b04490 |
| [64] |
高鹏亚, 苏英会, 孙晖, 等. 显微共聚焦拉曼技术在细菌分类鉴定中的应用[J]. 疾病监测, 2021, 36(1): 74-81. Gao P Y, Su Y H, Sun H, et al. Application of micro confocal Raman technique in classification and identification of bacteria[J]. Disease Surveillance, 2021, 36(1): 74-81. |
| [65] | Simon N S. Loosely bound oxytetracycline in riverine sediments from two tributaries of the Chesapeake bay[J]. Environmental Science & Technology, 2005, 39(10): 3480-3487. |
| [66] |
张昱, 唐妹, 田哲, 等. 制药废水中抗生素的去除技术研究进展[J]. 环境工程学报, 2018, 12(1): 1-14. Zhang Y, Tang M, Tian Z, et al. Research progress of removal technology of antibiotics from antibiotic production wastewater[J]. Chinese Journal of Environmental Engineering, 2018, 12(1): 1-14. |
| [67] | Eniola J O, Kumar R, Barakat M A, et al. A review on conventional and advanced hybrid technologies for pharmaceutical wastewater treatment[J]. Journal of Cleaner Production, 2022, 356. DOI:10.1016/j.jclepro.2022.131826 |
| [68] | Hou J, Chen Z Y, Gao J, et al. Simultaneous removal of antibiotics and antibiotic resistance genes from pharmaceutical wastewater using the combinations of up-flow anaerobic sludge bed, anoxic-oxic tank, and advanced oxidation technologies[J]. Water Research, 2019, 159: 511-520. DOI:10.1016/j.watres.2019.05.034 |
| [69] | Dirany A, Aaron S E, Oturan N, et al. Study of the toxicity of sulfamethoxazole and its degradation products in water by a bioluminescence method during application of the electro-Fenton treatment[J]. Analytical and Bioanalytical Chemistry, 2011, 400(2): 353-360. DOI:10.1007/s00216-010-4441-x |
| [70] | El-Ghenymy A, Rodríguez R M, Brillas E, et al. Electro-Fenton degradation of the antibiotic sulfanilamide with Pt/carbon-felt and BDD/carbon-felt cells. Kinetics, reaction intermediates, and toxicity assessment[J]. Environmental Science and Pollution Research, 2014, 21(14): 8368-8378. DOI:10.1007/s11356-014-2773-3 |
| [71] | Ganzenko O, Trellu C, Papirio S, et al. Bioelectro-fenton: Evaluation of a combined biological—advanced oxidation treatment for pharmaceutical wastewater[J]. Environmental Science and Pollution Research, 2018, 25(21): 20283-20292. DOI:10.1007/s11356-017-8450-6 |
| [72] | Valério A, Wang J F, Tong S, et al. Synergetic effect of photocatalysis and ozonation for enhanced tetracycline degradation using highly macroporous photocatalytic supports[J]. Chemical Engineering and Processing-Process Intensification, 2020, 149. DOI:10.1016/j.cep.2020.107838 |
| [73] | Miralles-Cuevas S, Audino F, Oller I, et al. Pharmaceuticals removal from natural water by nanofiltration combined with advanced tertiary treatments (solar photo-Fenton, photo-Fenton-like Fe(Ⅲ)–EDDS complex and ozonation)[J]. Separation and Purification Technology, 2014, 122: 515-522. DOI:10.1016/j.seppur.2013.12.006 |
| [74] | Ho D P, Vigneswaran S, Ngo H H. Photocatalysis-membrane hybrid system for organic removal from biologically treated sewage effluent[J]. Separation and Purification Technology, 2009, 68(2): 145-152. DOI:10.1016/j.seppur.2009.04.019 |
| [75] | Mondal S K, Saha A K, Sinha A. Removal of ciprofloxacin using modified advanced oxidation processes: kinetics, pathways and process optimization[J]. Journal of Cleaner Production, 2018, 171: 1203-1214. DOI:10.1016/j.jclepro.2017.10.091 |
 2024, Vol. 45
2024, Vol. 45


