2. 中国科学院南京土壤研究所土壤与农业可持续发展国家重点实验室, 南京 210008
2. State Key Laboratory of Soil and Sustainable Agriculture, Institute of Soil Science, Chinese Academy of Sciences, Nanjing 210008, China
氮和磷是作物必需的两种大量矿质营养元素, 也是农业生产中广泛使用促进作物产量的肥料元素[1].其中氮是植物体蛋白质和核酸的重要元素, 磷是植物体内DNA、细胞膜和ATP等重要组成物质, 氮和磷是作物产量的关键限制因素.土壤中的氮和磷主要以有机态形式存在, 而无机态往往会被矿物或土壤颗粒固定, 不能被植物直接吸收利用, 因此, 土壤中养分周转和作物获取养分能力决定了作物的产量[2].
土壤微生物在氮、磷周转中发挥着重要作用[3, 4], 直接影响土壤养分含量和有效性[5].在自然界中, 微生物形成了复杂的氮转化网络, 微生物主要通过氮的固定、氨化、硝化、反硝化和异化性硝酸盐还原等过程调节氮的生物有效性, 影响土壤中植物可利用氮的形式[6].生物固氮是指固氮菌将大气中的氮气还原成氨的过程, 是自然界中规模最大的天然氮肥工厂, 也是作物氮素来源之一[7].如固氮微生物Bradyrhizobium sp.定殖于根瘤中, 并给豆科植物提供铵盐[8].土壤中的无机态氮, 主要包括铵态氮和硝态氮, 是作物氮素的重要来源.土壤中的大部分有机氮源无法直接被作物吸收利用, 必须在微生物(氨化细菌)的作用下被分解为氨或小分子氮才能被作物利用.土壤磷循环主要包括有机磷矿化、磷的微生物固定、沉淀与溶解、吸附和解吸等过程.在农业土壤中, 很大一部分磷被固定为相对不可获取的无机和有机形态[9].有机磷的分解主要通过微生物分泌胞外酶如磷酸酶、植酸酶和C-P裂解酶等[10]. Zhang等[11]研究发现溶磷菌产碱假单胞菌(Pseudomonas alcaligenes)可提高土壤中磷酸酶的活性, 从而使微生物和植物获取更多的无机磷.此外, 微生物也会通过分泌有机酸, 如柠檬酸、葡萄糖酸等溶解被固定的无机磷[10].有研究表明, 土壤性质、施肥和有机质含量等对氮、磷转化有显著影响[12~14], 如pH可能通过影响微生物群落组成和结构进而调节土壤中氮、磷的动态[15, 16].微生物介导的氮磷转化过程是由酶催化完成, 目前已经鉴定出编码催化氮、磷转化相关的酶基因, 如编码同化硝酸还原酶的nirA、编码脲酶的ureABC和编码碱性磷酸酶的phoD基因等[8].然而, 从基因水平揭示农业生态系统中土壤微生物调控土壤氮磷养分循环过程的环境影响因素还不清楚.
近年来, 宏基因组测序技术的快速发展为全面理解复杂的土壤微生物群落组成和结构, 探究土壤微生物基因参与调控土壤养分循环的关键过程提供了可能.本研究基于3种典型农田土壤(黑土、潮土和红壤), 利用宏基因组测序技术探究不同土壤类型中玉米根际微生物氮磷转化功能, 从基因的角度揭示玉米根际微生物氮磷转化的功能特征.
1 材料与方法 1.1 试验地概况本研究田间试验设置于中国科学院生态系统试验站中的封丘试验站.封丘农田生态系统试验站(114°24′E, 35°00′N)位于河南省封丘县潘店乡, 海拔67.5 m, 属半干旱、半湿润的暖温带季风气候, 年均温为13.9℃, 年降水量605 mm, 主要集中在6~9月.年日照时数2 300~2 500 h.无霜期220 d左右.
1.2 供试材料供试土壤选择了3种不同的农田土壤, 分别采自东北松嫩平原海伦的黄土母质发育而成的黑土、黄淮海平原封丘的黄河冲积物发育的潮土和南方丘陵区鹰潭的第四纪红黏土发育的红壤.供试玉米品种为郑单958.
1.3 试验设计在各地分层(20 cm每层)采集3种土壤的剖面(1.2 m宽×1.4 m长×1 m深), 运至封丘站后按原来的土层顺序填装试验小区, 试验小区为1.2 m宽×1.4 m长×1 m深的砌砖水泥池(微区面积1.2 m宽×1.4 m长=1.68 m2), 小区隔墙厚20 cm, 露出地表 20 cm, 底部铺石英沙, 内壁覆盖防水布.试验设置2个处理: ①不施肥处理(CK): 每年6月初种植1季玉米, 不施用任何肥料, 采用雨养, 不灌溉, 人工定期除草; ②常规施肥处理(NPK): 每年种植1季玉米, 施肥量为N 150 kg ·hm-2、P2 O5 75 kg ·hm-2和K2 O 60 kg ·hm-2, 肥料分别为尿素、(NH4)2 HPO4和KCl, 种植前条施底肥(1/2氮肥、全部磷肥和钾肥), 玉米大喇叭口期追施尿素(1/2氮肥), 其他管理条件同不施肥处理.每个处理设3个重复.
玉米成熟后, 采集土壤根际样品, 用土铲将玉米根部挖出, 将非根际土抖落掉, 收集部分非根际土用于理化性质分析.将粘附在根表面的土壤装入无菌袋中, 密封后立即放入生物样品采样箱中, 低温运至实验室.粘附在根表面的土壤即为根际土.在实验室无菌条件下, 放入50 mL无菌0.9% NaCl溶液搅动5 min, 7 888 r ·min-1离心10 min收集土壤, 保存于-80℃, 用于微生物测序分析.土壤的基本理化性质测定参照《土壤农业化学分析方法》[17], 其土壤基本理化性质如表 1所示.
|
|
表 1 供试土壤基本理化性质1) Table 1 Physical and chemical properties of the tested soils |
1.4 土壤总DNA提取及宏基因组测序
称取-80℃保存的根际土壤样品2 g, 采用美国MO BIO PowerSoil DNA Isolation Kit(12888-100, MO BIO)的DNA提取试剂盒并结合液氮冷冻研磨的方法提取土壤中微生物基因组DNA[18].提取后的DNA进行1%(质量浓度)琼脂糖凝胶电泳检测DNA片段大小, 采用NanoDrop 2000(Thermo Fisher, 美国)测定提取液中双链DNA的浓度和纯度.检测合格保存于-20℃.
宏基因组测序采用HiSeq2000(Illumina, 美国)高通量测序平台完成.利用基于De-Brujin graph原理的拼接软件SOAPdenovo(version 1.05)采用不同大小的k-mer(51、55、59和63)对过滤后的数据进行组装, 在scaffolds内部gaps处, 将scaffolds重新打断成新的scaftigs, 并对大于等于500 bp的scaftigs进行统计, 从不同k-mer的组装结果中选择N50最大的组装结果.利用软件MetaGeneMark对组装结果进行开放阅读框的预测, 然后根据得到的gff注释文件还原成.fna基因序列文件, 并从中筛选出长度大于100bp的基因序列, 并翻译成对应的氨基酸序列.利用CD-HIT将所有预测出来的基因聚类(一致性>95%, 覆盖度>90%), 选择每一类中最长的基因序列后去除其余冗余基因, 从而构建有关样品的非冗余基因集.通过SOAPaligner将Clean Reads比对非冗余基因集, 根据每个基因被比对上的reads条数与基因的长度, 计算得到每个基因在样品中的丰度, 并汇总成所有样品的基因丰度表.运用BLAST将基因集与KEGG的基因数据库(GENES)进行比对, 获得KEGG数据库的KO注释信息和对应的基因丰度, 进行代谢通路丰度的统计.
1.5 数据分析文中数据采用Excel 2010和IBM SPSS Statistics 26软件进行基本处理和分析, 通过one-way ANOVA和Tukey法对不同处理(施肥和土壤类型)的功能基因丰度进行方差分析和多重比较.采用随机森林法对生物和非生物因子和土壤微生物功能多样性及氮、磷循环相关基因的关系进行分析, 其中土壤微生物功能多样性是由KEGG数据库中的功能注释得到KO表征.随机森林分析用R(version 3.4.1)软件randomForest包中的randomForest函数对生物和非生物因子做重要性评价, 得到衡量生物和非生物因子重要性的指标均方差增加值.
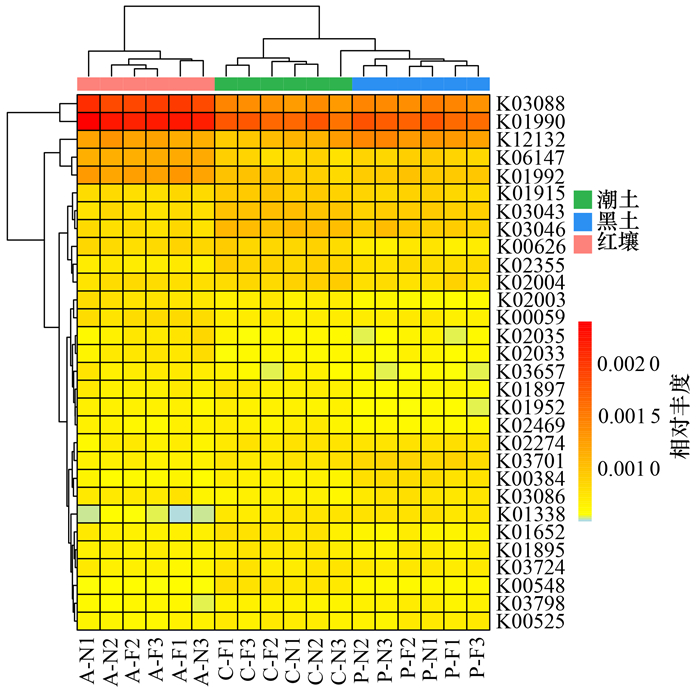
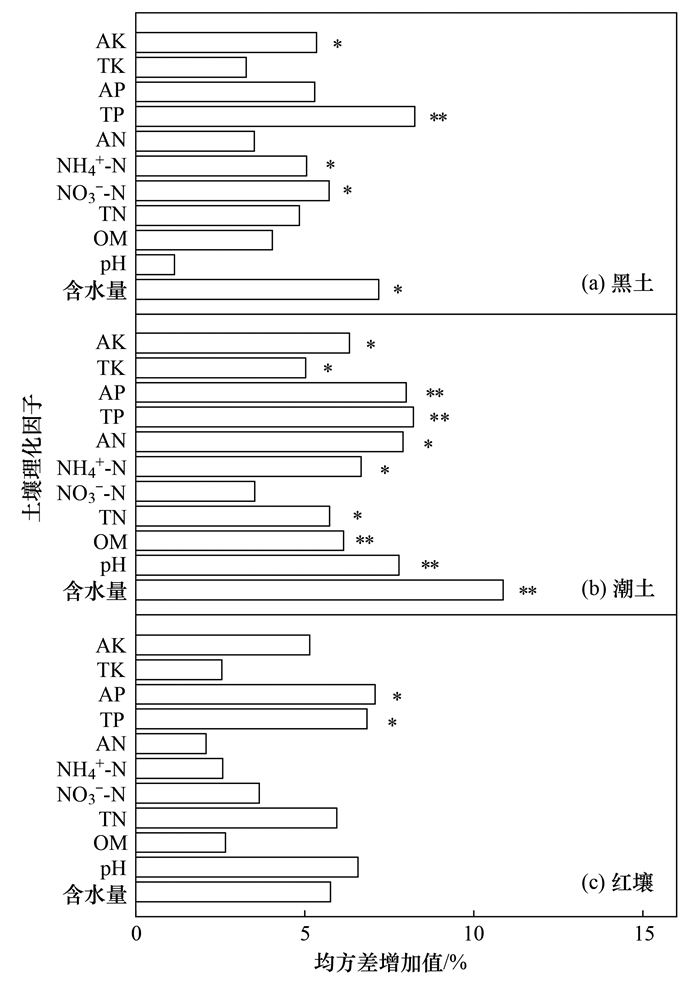
2 结果与分析 2.1 土壤微生物功能多样性及其影响因素为了探究不同土壤微生物功能多样性及其主导的环境影响因素, 对3种土壤样品进行宏基因组测序和功能注释分析.KO是蛋白质(酶)的一个分类体系, 其中序列高度相似、在同一条通路上有相似功能的蛋白质被归为一组.根据所有样品在KEGG数据库中的功能注释得到KO信息及其对应的基因丰度, 选取所有样品中丰度排名前30的KO及它们在每个样品中的丰度信息绘制热图(图 1).所有样品中均以遗传信息处理通路KO03088(RNA polymerase sigma-70 factor, ECF subfamily)和环境信息处理通路KO01990(ABC-2 type transport system ATP-binding protein)涉及的基因丰度最高.此外, KO丰度热图中的聚类结果表明, 3种土壤的KO信息聚类分异明显, 同一类型的土壤样品的KO信息更相近, 表明具有更为相似的功能多样性.随机森林分析结果表明, 不同土壤环境因子对3种土壤微生物功能多样性存在显著关联.黑土中KO组成结构主要受土壤AK(5.35%, 均方差增加值, 下同, P=0.050)、TP(8.25%, P=0.009 9)、NH4+-N(5.05%, P=0.030)、NO3--N(5.72%, P=0.040)和含水量(7.18%, P=0.020)的控制.土壤pH(7.78%, P=0.020)、含水量(10.87%, P=0.009 9)、TP(8.21%, P=0.009 9)、AP(8.00%, P=0.009 9)、TK(5.02%, P=0.050)、AK(6.32%, P=0.030)、NH4+-N(6.67%, P=0.030)、TN(5.73%, P=0.030)和OM(6.14%, P=0.030)是影响潮土KO组成结构的主要因素, 而红壤中KO组成结构与TP(6.84%, P=0.030)和AP(7.08%, P=0.020)含量关系较为密切(图 2).总体来说, 不同土壤类型微生物群落功能多样性及其主导的环境因子存在差异.
|
A、C和P分别表示红壤、潮土和黑土, F表示施NPK肥, N表示不施肥; 数字1、2和3表示重复 图 1 排名前30的KO热图 Fig. 1 Heatmap of top 30 KO |
|
OM: 有机质, TN: 全氮, NO3--N: 硝态氮, NH4+-N: 铵态氮, AN: 碱解氮, TP: 全磷, AP: 速效磷, TK: 全钾, AK: 速效钾; *表示P<0.05, **表示P<0.01 图 2 影响微生物功能多样性的土壤理化因子 Fig. 2 Effects of soil physico-chemical factors on microbial functional diversity |
土壤微生物是土壤氮磷等元素循环的驱动者, 可以从宏基因组中注释到各通路的基因信息中提取与氮磷循环的有关途径, 进一步从功能基因层面揭示微生物参与的氮磷转化的过程动态.
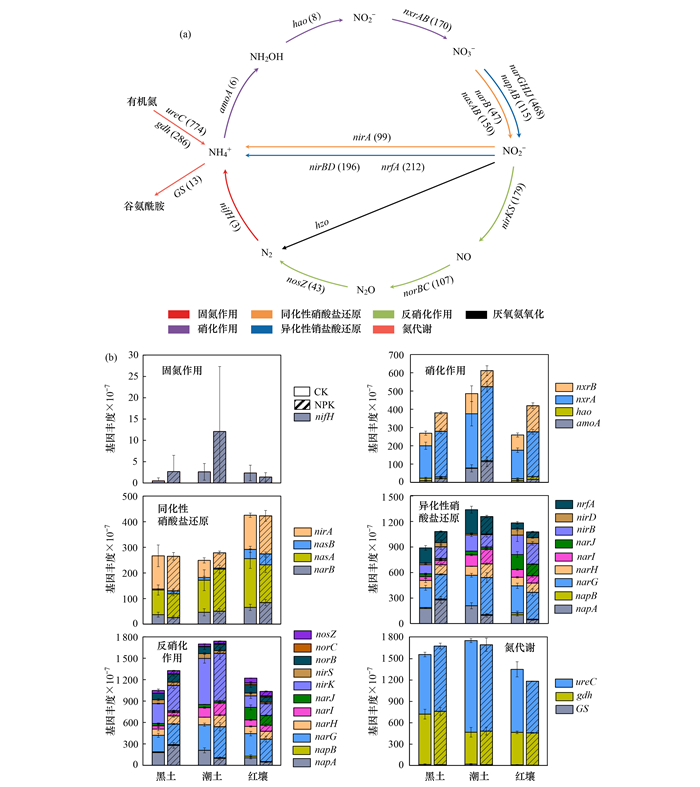
在农田生态系统中, 微生物参与的影响作物氮素吸收利用的氮循环过程主要包括固氮、氨化、谷氨酰胺合成、硝化、同化性硝酸盐还原、异化性硝酸盐还原、反硝化和厌氧氨氧化等过程[图 3(a)].微生物在固氮酶的催化作用下将大气中的氮气还原成氨而固定(EC: 1.18.6.1), 该过程注释到KO02588(nifH)的基因有3个; 微生物体内的脲酶可以分解土壤中的尿素产生氨供植物利用(EC: 3.5.1.5), 样品中注释到KO01428(ureC)的基因有774个; 谷氨酰胺合成(GS)是铵同化的主要途径(EC: 6.3.1.2).在土壤中, 铵态氮会在氨单加氧酶的作用下被氧化成羟胺(EC: 1.14.18.3 1.14.99.39), 然后由羟胺氧化还原酶氧化为亚硝酸盐(EC: 1.7.2.6), 最终由亚硝酸氧化还原酶氧化为硝酸盐(EC: 1.7.99.4)完成硝化作用, 注释到此过程中的KO10944(amoA)、KO10535(hao)、KO00370(nxrA)和KO00371(nxrB)的基因分别为6、8、140和30个.与此同时, 土壤中的硝酸盐会由硝酸盐还原酶还原成亚硝酸盐(EC: 1.7.99.4), 其中注释到同化型还原酶KO00367(narB)、KO00372(nasA)和KO00360(nasB)的基因分别有47、131和19个; 异化型还原酶: KO00370(narG)、KO00371(narH)、KO00374(narI)、KO00373(narJ)、KO02567(napA)和KO02568(napB)的基因分别有166、198、60、44、105和10个; 之后亚硝酸还原酶会进一步将亚硝酸盐转化为氨(EC: 1.7.7.1和EC: 1.7.1.15)或一氧化氮(EC: 1.7.2.1), 样品中注释到KO00366(nirA)、KO00362(nirB)、KO00363(nirD)、KO00368(nirK)和KO15864(nirS)的基因分别有99、161、35、145和34个.由于所有样品中没有注释到N2H4氧化还原酶基因hzo, 故暂不考虑微生物参与厌氧氨氧化过程.
|
(a)中括号中数字为在所有样本中注释到通路中相关酶的基因个数 图 3 微生物参与的氮循环过程及相关基因丰度 Fig. 3 Nitrogen cycle process and related gene abundance involved in microorganisms |
获得微生物参与氮循环有关的基因信息后, 进一步分析样品中编码各氮转化过程通路中相关酶的基因丰度[图 3(b)], 发现所有样品中均以脲酶基因(ureC)和葡萄糖脱氢酶基因(gdh)丰度最高, 分别达7.25×10-5~12.88×10-5和4.47×10-5~7.49×10-5, 说明土壤微生物具有极强的有机氮分解代谢的潜能.土壤中的有机氮化合物可被微生物分解用于满足微生物自身氮素营养需求或供作物吸收利用.其次是参与土壤反硝化过程的硝酸还原酶基因narG和亚硝酸还原酶基因nirK, 其丰度分别为2.31×10-5~4.35×10-5和1.61×10-5~6.67×10-5, 编码硝化过程中亚硝酸氧化还原酶的基因nxrA丰度也达1.55×10-5~4.05×10-5, 这两个过程将直接影响土壤作物可直接利用的NO3--N和NH4+-N的含量.与不同土壤类型中微生物功能多样性一致, 不同土壤类型的样品中调控氮循环的功能基因丰度各异, 其中编码生物固氮、硝化、异化性硝酸盐还原、反硝化和氮代谢过程相关酶的功能基因均以潮土中的总丰度最高, 而涉及同化性硝酸盐还原的功能基因在红壤中总丰度要显著高于黑土和潮土.
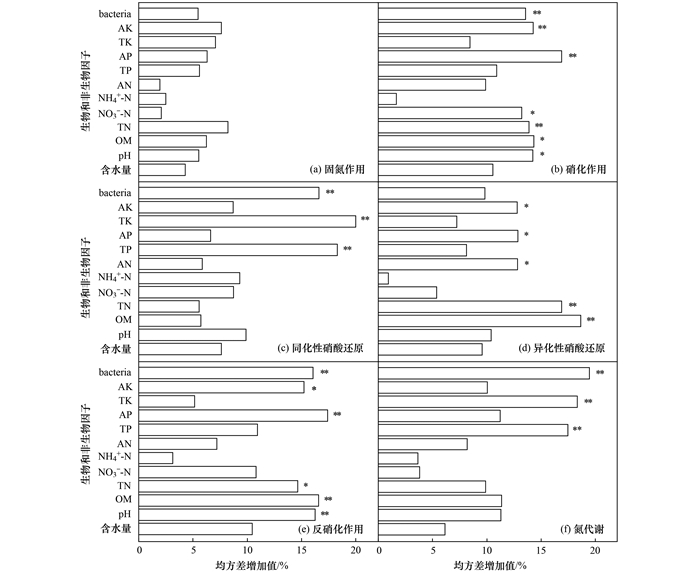
通过随机森林分析, 进一步揭示氮循环各过程功能基因丰度受土壤理化性质和微生物丰富度不同程度的影响(图 4).结果表明, 与氮代谢过程有关的基因丰度受不同的生物因素(如微生物丰富度)和非生物因素(如土壤理化性质)影响.举例来说, 固氮过程相关基因与生物和非生物因素不存在显著关联(P>0.05), 同化性硝酸盐还原过程相关基因主要受TK(19.88%, 均方差增加值, 下同, P=0.007 5)、TP(18.24%, P=0.008 7)和细菌丰富度(16.43%, P=0.009 7)显著影响, 而编码硝化和反硝化过程相关酶的功能基因均受土壤AP(16.61%, P=0.006 4; 17.31%, P=0.006 4)、OM(14.02%, P=0.007 9; 16.54%, P=0.008 9)、pH(13.95%, P=0.045 1; 16.08%, P=0.007 2)、AK(14.00%, P=0.008 9; 15.24%, P=0.019)、TN(13.55%, P=0.009 5; 14.62%, P=0.011)和细菌丰富度(13.34%, P=0.009 9; 16.03%, P=0.007 8)显著影响.
|
bacteria: 细菌丰富度, OM: 有机质, TN: 全氮, NO3--N: 硝态氮, NH4+-N: 铵态氮, AN: 碱解氮, TP: 全磷, AP: 速效磷, TK: 全钾, AK: 速效钾; *表示P<0.05, **表示P<0.01 图 4 影响氮循环功能基因丰度的生物和非生物因子 Fig. 4 Effects of biotic and abiotic factors on nitrogen cycle functional gene abundance |
土壤磷元素的代谢循环中, 有机磷的矿化主要是在微生物分泌的磷酸酶的作用下进行的, 包括碱性磷酸酶(EC: 3.1.3.1)和酸性磷酸酶(EC: 3.1.3.2).所有样品中注释到编码碱性磷酸酶KO01113(phoD)和酸性磷酸酶KO01078(PHO)的基因分别有1 093和42个.各样品中碱性磷酸酶基因丰度在数量级上远高于酸性磷酸酶基因丰度[图 5(a)和5(b)].其中, phoD基因丰度在3种土壤类型中没有显著差异, 分别为1.36×10-4~1.67×10-4、1.32×10-4~1.68×10-4和1.08×10-4~1.55×10-4, 而酸性磷酸酶基因在黑土中的丰度要略高于红壤和潮土, 分别为6.78×10-6~1.11×10-5、3.62×10-6~7.52×10-6和2.34×10-6~7.61×10-6.随机森林分析表明碱性磷酸酶基因丰度受土壤水分(14.16%, 均方差增加值, 下同, P=0.022)、OM(13.29%, P=0.039)和TN(12.59%, P=0.049)含量显著影响, 而酸性磷酸酶基因除了受土壤水分(13.79%, P=0.034)、OM(14.90%, P=0.0097)和TN(12.41%, P=0.042)含量的影响之外, 土壤AP(14.16%, P=0.022)含量对其影响更为显著[图 5(c)和5(d)].
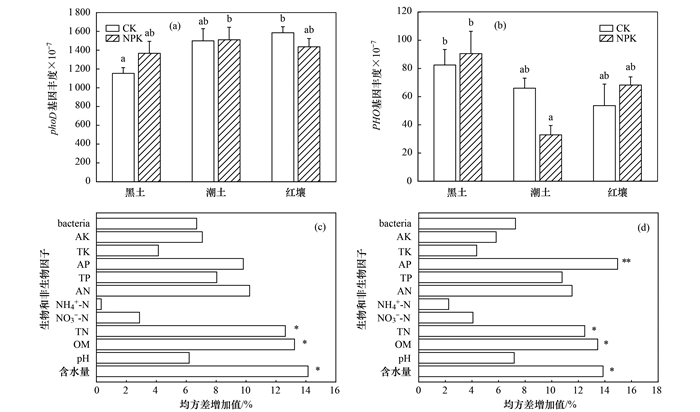
|
(a)和(c)为碱性磷酸酶, (b)和(d)为酸性磷酸酶; 不同小写字母表示土壤类型或施肥处理之间差异显著(P<0.05); bacteria: 细菌丰富度, OM: 有机质, TN: 全氮, NO3--N: 硝态氮, NH4+-N: 铵态氮, AN: 碱解氮, TP: 全磷, AP: 速效磷, TK: 全钾, AK: 速效钾; *表示P<0.05, **表示P<0.01 图 5 有机磷矿化过程中的功能基因丰度和影响因素 Fig. 5 Abundance of functional genes and influencing factors in the process of organic phosphorus mineralization |
了解土壤微生物功能多样性以及氮磷代谢过程相关功能基因多样性后, 进一步探究与磷转化相关的关键微生物类群在不同土壤类型中的分布信息.基于过往的研究发现类诺卡氏菌(Nocardioides sp.)和鞘氨醇单胞菌(Sphingomonas sp.)与磷转化密切相关[19].从宏基因组测序注释到的基因信息获得类诺卡氏菌(Nocardioides sp.)和鞘氨醇单胞菌(Sphingomonas sp.)在3种土壤类型中的分布, 发现其中含有丰富的促进土壤有机磷矿化分解的碱性磷酸酶基因KO01113(phoD).红壤中类诺卡氏菌(Nocardioides sp.)包含的phoD基因丰度显著高于潮土和黑土, 分别为6.90×10-6~2.18×10-5、2.86×10-6~1.04×10-5和8.47×10-8~3.26×10-7[图 6(a)].3种土壤中鞘氨醇单胞菌(Sphingomonas sp.)包含的phoD基因丰度十分丰富且差异不显著, 表明鞘氨醇单胞菌在土壤磷循环中可能是潜在的关键类群, 其在黑土、潮土和红壤中的丰度分别为2.47×10-7~2.74×10-6、3.23×10-7~2.31×10-6和2.90×10-7~2.55×10-6[图 6(b)].
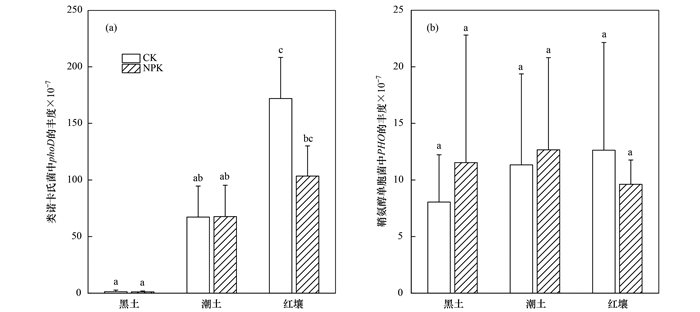
|
不同小写字母表示土壤类型或施肥处理之间差异显著(P<0.05) 图 6 类诺卡氏菌和鞘氨醇单胞菌中编码碱性磷酸酶的基因丰度 Fig. 6 Gene abundance of encoding alkaline phosphatase in Nocardioides sp. and Sphingomona sp. |
在农田生态系统中, 作物的氮磷利用率不仅与土壤中氮磷养分元素的含量有关, 更取决于氮磷养分元素的形态和可利用性.土壤微生物驱动的氮磷转化过程主要是由微生物体内存在的一系列代谢相关的酶(蛋白)起关键作用.探究氮磷转化的代谢途径和编码相关酶的功能基因是从根本上揭示微生物驱动氮磷转化的机制的必要途径.
本研究发现固氮酶基因nifH数量和丰度均相对较低.张淼等[20]和Mao等[21]对玉米作物的研究均发现nifH丰度较其它基因低, 这与本文结果一致.这可能是与玉米作物的固氮因子(气生根粘液等)比较低而导致其根际微生物群落中固氮菌群较少有关, 也可能因为在微生物体内或根际土壤中固氮酶基因nifH丰度相对较低[22].此外, 有研究报道宏基因组测序所能获得的nifH基因丰度要比扩增子测序所获得的丰度低[23, 24].随机森林分析发现, nifH丰度与细菌丰富度和土壤理化性质没有显著相关关系, 这可能也与nifH丰度均相对较低有关.土壤中的大部分有机氮源包括施入的尿素都无法被作物直接吸收利用, 脲酶催化脲素分解为NH4+的氨化作用是将土壤有机氮转化为氨, 是供作物吸收利用氮素的重要途径.本研究发现所有样品中均以编码脲酶的ureC基因丰度最高, 表明土壤微生物具有较强的分解脲素的能力.此外, 本研究发现ureC基因丰度与细菌丰富度、TP和TK显著相关, 这可从细菌丰度和养分平衡得到很好地理解[25].微生物对有机氮的矿化作用是微生物获得自身生长所需的能量和营养元素的重要途径, 为了养分平衡, 需要从土壤获取其它养分[26].在氮素不受限制的农田生态系统中, 微生物矿化的有机氮供作物吸收的部分可达作物从土壤中吸收的氮素的30%~50%[27].铵态氮和硝态氮是作物能直接吸收利用的主要的无机氮形态, 其含量直接受硝化和反硝化作用的影响, 硝化和反硝化过程是农田生态系统中氮素损失的主要途径之一[28, 29].本研究发现硝化和反硝化过程主要受细菌丰富度、pH和养分含量(OM、TN、AP和AK)的影响.pH是细菌群落组成变异最重要的影响因子, 直接影响了细菌丰富度、amoA、nirK和nirS丰度[30].OM、TN、AP和AK等养分含量直接或间接作为硝化和反硝化菌的代谢底物, 进而影响细菌群落[31].硝化反应的限速步骤在于氨氧化过程, 其速率主要受氨单加氧酶(AMO)控制, 编码氨单加氧酶的amoA基因丰度在潮土中最高, 这可能是因为潮土的pH较高.随机森林分析表明硝化作用与土壤pH显著相关(图 4).有研究表明酸性土壤会降低氨单加氧酶的底物NH3浓度, 从而抑制了amoA的表达[32]. nirK和nirS是催化亚硝酸盐还原的主要酶类, 本研究发现, 在测定的3种土壤中nirK基因丰度均显著高于nirS, 这与前人研究的结果一致[33~35], 但也有nirK基因丰度低于nirS的报道[36].产生这样现象的原因可能是采用的检测方法不同所致, 张淼等[20]发现对同一样点采用PCR和宏基因组测序两种不同技术所得到的结果截然相反.微生物参与的氮转化过程极其复杂, 涉及的功能基因种类和数量较多, 微生物携带的氮转化各过程的功能基因多样性和丰度与其具体功能之间的定量关系, 需要在之后进一步研究验证.
土壤磷代谢的限制性在于磷元素通常存在于矿物中而难以直接被作物吸收利用, 土壤中30%~50%的磷以有机磷的形式存在, 需要在磷酸酶的作用下矿化成无机磷后才能被作物和土壤微生物吸收和进一步利用, 磷素也因此成为农田生态系统的限制性养分元素之一[37].磷酸酶分为碱性磷酸酶和酸性磷酸酶, 碱性磷酸酶主要由细菌产生, 而酸性磷酸酶主要由植物根系和根际真菌分泌[38~40].注释到各土壤中编码碱性磷酸酶的phoD基因数目和丰度均要显著高于酸性磷酸酶PHO基因.在碱性磷酸酶家族中, phoD基因被认为是土壤中最重要的碱性磷酸酶基因[41], 土壤中phoD基因的丰度与碱性磷酸酶活性成正相关关系[42, 43].TN、OM和土壤含水量是影响phoD基因的主要因子, 这与前人研究结果一致[44, 45].含水量可通过影响携带phoD基因的微生物群落间接影响其丰度.笔者课题组之前研究发现了与磷转化相关的关键微生物类诺卡氏菌(Nocardioides sp.)和鞘氨醇单胞菌(Sphingomonas sp.), 本研究发现其功能基因中均含有丰富的phoD基因, 因此, 推测关键类群正是由其含有的phoD基因编码碱性磷酸酶, 通过分泌碱性磷酸酶矿化分解土壤中的有机磷来促进作物磷素的吸收利用.
4 结论(1) 根际微生物功能多样性受土壤类型影响, 黑土和潮土根际微生物功能多样性主要受含水量和养分含量的影响, 红壤受TP和AP调控.
(2) 在各个氮转化过程中, 3种土壤类型均以ureC、narG、nirK、nasA和nxrA的丰度较高.同化性硝酸盐还原的功能基因在红壤中总丰度要高于黑土和潮土, 其它过程相关酶的功能基因总丰度以潮土最高.编码硝化和反硝化过程相关酶的功能基因丰度主要受细菌丰富度、pH和养分含量的驱动.编码氮代谢过程相关酶的功能基因丰度主要受土壤细菌丰富度、TK和TP含量的驱动.
(3) 有机磷矿化过程中注释到的碱性磷酸酶phoD基因数目和丰度均显著高于酸性磷酸酶PHO基因. phoD基因数目为1 093个, PHO数目为42个. phoD和PHO基因在3种土壤类型中的丰度分别在1.08×10-4~1.68×10-4和2.34×10-6~1.11×10-5之间. phoD和PHO丰度均受土壤水分、OM和TN显著影响, 但AP含量对PHO丰度影响最大.类诺卡氏菌(Nocardioides sp.)和鞘氨醇单胞菌(Sphingomonas sp.)中发现了丰富的碱性磷酸酶基因phoD.
| [1] |
沈仁芳, 孙波, 施卫明, 等. 地上-地下生物协同调控与养分高效利用[J]. 中国科学院院刊, 2017, 32(6): 566-574. Shen R F, Sun B, Shi W M, et al. Interactions between above-and below-ground organisms for nutrient-efficient utilization[J]. Bulletin of Chinese Academy of Sciences, 2017, 32(6): 566-574. DOI:10.16418/j.issn.1000-3045.2017.06.003 |
| [2] |
储成才, 王毅, 王二涛. 植物氮磷钾养分高效利用研究现状与展望[J]. 中国科学: 生命科学, 2021, 51(10): 1415-1423. Chu C C, Wang Y, Wang E T. Improving the utilization efficiency of nitrogen, phosphorus and potassium: current situation and future perspectives[J]. Scientia Sinica Vitae, 2021, 51(10): 1415-1423. |
| [3] | Ollivier J, Töwe S, Bannert A, et al. Nitrogen turnover in soil and global change[J]. FEMS Microbiology Ecology, 2011, 78(1): 3-16. DOI:10.1111/j.1574-6941.2011.01165.x |
| [4] | Dai Z M, Liu G F, Chen H H, et al. Long-term nutrient inputs shift soil microbial functional profiles of phosphorus cycling in diverse agroecosystems[J]. The ISME Journal, 2020, 14(3): 757-770. DOI:10.1038/s41396-019-0567-9 |
| [5] | Berendsen R L, Pieterse C M J, Bakker P A H M. The rhizosphere microbiome and plant health[J]. Trends in Plant Science, 2012, 17(8): 478-486. DOI:10.1016/j.tplants.2012.04.001 |
| [6] | Nelson M B, Martiny A C, Martiny J B H. Global biogeography of microbial nitrogen-cycling traits in soil[J]. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(29): 8033-8040. |
| [7] | de Bruijin J. Biological nitrogen fixation[A]. In: Lugtenberg B(Ed.). Principles of Plant-Microbe Interactions[M]. Cham: Springer, 2015. 215-224. |
| [8] | Kuypers M M M, Marchant H K, Kartal B. The microbial nitrogen-cycling network[J]. Nature Reviews Microbiology, 2018, 16(5): 263-276. DOI:10.1038/nrmicro.2018.9 |
| [9] | Liu L, Gao Z Y, Yang Y, et al. Long-term high-P fertilizer input shifts soil P cycle genes and microorganism communities in dryland wheat production systems[J]. Agriculture, Ecosystems & Environment, 2023, 342. DOI:10.1016/j.agee.2022.108226 |
| [10] | Liang J L, Liu J, Jia P, et al. Novel phosphate-solubilizing bacteria enhance soil phosphorus cycling following ecological restoration of land degraded by mining[J]. The ISME Journal, 2020, 14(6): 1600-1613. DOI:10.1038/s41396-020-0632-4 |
| [11] | Zhang L, Fan J Q, Ding X D, et al. Hyphosphere interactions between an arbuscular mycorrhizal fungus and a phosphate solubilizing bacterium promote phytate mineralization in soil[J]. Soil Biology and Biochemistry, 2014, 74: 177-183. DOI:10.1016/j.soilbio.2014.03.004 |
| [12] | Xun W B, Huang T, Zhao J, et al. Environmental conditions rather than microbial inoculum composition determine the bacterial composition, microbial biomass and enzymatic activity of reconstructed soil microbial communities[J]. Soil Biology and Biochemistry, 2015, 90: 10-18. DOI:10.1016/j.soilbio.2015.07.018 |
| [13] | Ji B Y, Hu H, Zhao Y L, et al. Effects of deep tillage and straw returning on soil microorganism and enzyme activities[J]. The Scientific World Journal, 2014, 2014. DOI:10.1155/2014/451493 |
| [14] | Štursová M, Baldrian P. Effects of soil properties and management on the activity of soil organic matter transforming enzymes and the quantification of soil-bound and free activity[J]. Plant and Soil, 2011, 338(1-2): 99-110. DOI:10.1007/s11104-010-0296-3 |
| [15] | Zhou Z H, Wang C K, Zheng M H, et al. Patterns and mechanisms of responses by soil microbial communities to nitrogen addition[J]. Soil Biology and Biochemistry, 2017, 115: 433-441. DOI:10.1016/j.soilbio.2017.09.015 |
| [16] | Wu X J, Rensing C, Han D F, et al. Genome-resolved metagenomics reveals distinct phosphorus acquisition strategies between soil microbiomes[J]. mSystems, 2022, 7(1). DOI:10.1128/msystems.01107-21 |
| [17] | 鲁如坤. 土壤农业化学分析方法[M]. 北京: 中国农业科技出版社, 2000. |
| [18] | Zhou J Z, Bruns M A, Tiedje J M. DNA recovery from soils of diverse composition[J]. Applied and Environmental Microbiology, 1996, 62(2): 316-322. DOI:10.1128/aem.62.2.316-322.1996 |
| [19] |
唐浩琪, 张娜, 孙波, 等. 典型农田土壤中丛枝菌根真菌-根际细菌互作及与氮磷利用的关系[J]. 微生物学报, 2020, 60(6): 1117-1129. Tang H Q, Zhang N, Sun B, et al. Effect of interaction between arbuscular mycorrhizal fungi and rhizosphere bacteria in farmland soils on nutrients utilization[J]. Acta Microbiologica Sinica, 2020, 60(6): 1117-1129. DOI:10.13343/j.cnki.wsxb.20200204 |
| [20] |
张淼, 刘俊杰, 刘株秀, 等. 黑土区农田土壤氮循环关键过程微生物基因丰度的分布特征[J]. 土壤学报, 2022, 59(5): 1258-1269. Zhang M, Liu J J, Liu Z X, et al. Distribution characteristics of microbial gene abundance in key processes of soil nitrogen cycling in black soil zone[J]. Acta Pedologica Sinica, 2022, 59(5): 1258-1269. |
| [21] | Mao Y J, Yannarell A C, Mackie R I. Changes in N-transforming archaea and bacteria in soil during the establishment of bioenergy crops[J]. PLoS One, 2011, 6(9). DOI:10.1371/journal.pone.0024750 |
| [22] | Wang Q, Fish J A, Gilman M, et al. Xander: employing a novel method for efficient gene-targeted metagenomic assembly[J]. Microbiome, 2015, 3. DOI:10.1186/s40168-015-0093-6 |
| [23] | Tu Q C, He Z L, Wu L Y, et al. Metagenomic reconstruction of nitrogen cycling pathways in a CO2-enriched grassland ecosystem[J]. Soil Biology and Biochemistry, 2017, 106: 99-108. DOI:10.1016/j.soilbio.2016.12.017 |
| [24] | Zhou J Z, He Z L, Yang Y F, et al. High-throughput metagenomic technologies for complex microbial community analysis: open and closed formats[J]. mBio, 2015, 6(1). DOI:10.1128/mBio.02288-14 |
| [25] |
董志新, 孙波, 殷士学, 等. 气候条件和作物对黑土和潮土固氮微生物群落多样性的影响[J]. 土壤学报, 2012, 49(1): 130-138. Dong Z X, Sun B, Yin S X, et al. Impacts of climate and cropping on community diversity of diazotrophs in pachic udic argiboroll and fluventic ustochrept[J]. Acta Pedologica Sinica, 2012, 49(1): 130-138. |
| [26] |
钟泽坤, 杨改河, 任成杰, 等. 黄土丘陵区撂荒农田土壤酶活性及酶化学计量变化特征[J]. 环境科学, 2021, 42(1): 411-421. Zhong Z K, Yang G H, Ren C J, et al. Effects of farmland abandonment on soil enzymatic activity and enzymatic stoichiometry in the loess hilly region, China[J]. Environmental Science, 2021, 42(1): 411-421. |
| [27] | Dungait J A J, Cardenas L M, Blackwell M S A, et al. Advances in the understanding of nutrient dynamics and management in UK agriculture[J]. Science of the Total Environment, 2012, 434: 39-50. DOI:10.1016/j.scitotenv.2012.04.029 |
| [28] | Bowles T M, Raab P A, Jackson L E. Root expression of nitrogen metabolism genes reflects soil nitrogen cycling in an organic agroecosystem[J]. Plant and Soil, 2015, 392(1-2): 175-189. DOI:10.1007/s11104-015-2412-x |
| [29] | Xu C M, Xiao D S, Chen S, et al. Changes in the activities of key enzymes and the abundance of functional genes involved in nitrogen transformation in rice rhizosphere soil under different aerated conditions[J]. Journal of Integrative Agriculture, 2022, 22(3): 923-934. |
| [30] | Enwall K, Philippot L, Hallin S. Activity and composition of the denitrifying bacterial community respond differently to long-term fertilization[J]. Applied and Environmental Microbiology, 2005, 71(12): 8335-8343. DOI:10.1128/AEM.71.12.8335-8343.2005 |
| [31] | Hallin S, Jones C M, Schloter M, et al. Relationship between N-cycling communities and ecosystem functioning in a 50-year-old fertilization experiment[J]. The ISME Journal, 2009, 3(5): 597-605. DOI:10.1038/ismej.2008.128 |
| [32] | Song H, Che Z, Cao W C, et al. Changing roles of ammonia-oxidizing bacteria and archaea in a continuously acidifying soil caused by over-fertilization with nitrogen[J]. Environmental Science and Pollution Research, 2016, 23(12): 11964-11974. |
| [33] | Krause H M, Thonar C, Eschenbach W, et al. Long term farming systems affect soils potential for N2 O production and reduction processes under denitrifying conditions[J]. Soil Biology and Biochemistry, 2017, 114: 31-41. |
| [34] | Coyotzi S, Doxey A C, Clark I D, et al. Agricultural soil denitrifiers possess extensive nitrite reductase gene diversity[J]. Environmental Microbiology, 2017, 19(3): 1189-1208. |
| [35] | Dandie C E, Wertz S, Leclair C L, et al. Abundance, diversity and functional gene expression of denitrifier communities in adjacent riparian and agricultural zones[J]. FEMS Microbiology Ecology, 2011, 77(1): 69-82. |
| [36] | Li F G, Li M C, Shi W C, et al. Distinct distribution patterns of proteobacterial nirK- and nirS-type denitrifiers in the Yellow River estuary, China[J]. Canadian Journal of Microbiology, 2017, 63(8): 708-718. |
| [37] | Richardson A E, Simpson R J. Soil microorganisms mediating phosphorus availability update on microbial phosphorus[J]. Plant Physiology, 2011, 156(3): 989-996. |
| [38] | Fraser T D, Lynch D H, Gaiero J, et al. Quantification of bacterial non-specific acid(phoC) and alkaline(phoD) phosphatase genes in bulk and rhizosphere soil from organically managed soybean fields[J]. Applied Soil Ecology, 2017, 111: 48-56. |
| [39] | Yadav B K, Verma A. Phosphate solubilization and mobilization in soil through microorganisms under arid ecosystems[A]. Ali M(Ed.). The Functioning of Ecosystems[M]. Rijeka: InTech, 2012. |
| [40] | Spohn M, Kuzyakov Y. Distribution of microbial- and root-derived phosphatase activities in the rhizosphere depending on P availability and C allocation-coupling soil zymography with 14 C imaging[J]. Soil Biology and Biochemistry, 2013, 67: 106-113. |
| [41] | Sun Q, Qiu H S, Hu Y J, et al. Cellulose and lignin regulate partitioning of soil phosphorus fractions and alkaline phosphomonoesterase encoding bacterial community in phosphorus-deficient soils[J]. Biology and Fertility of Soils, 2019, 55(1): 31-42. |
| [42] | Fraser T, Lynch D H, Entz M H, et al. Linking alkaline phosphatase activity with bacterial phoD gene abundance in soil from a long-term management trial[J]. Geoderma, 2015, 257-258: 115-122. |
| [43] | Tan H, Barret M, Mooij M J, et al. Long-term phosphorus fertilisation increased the diversity of the total bacterial community and the phoD phosphorus mineraliser group in pasture soils[J]. Biology and Fertility of Soils, 2013, 49(6): 661-672. |
| [44] | Luo G W, Ling N, Nannipieri P, et al. Long-term fertilisation regimes affect the composition of the alkaline phosphomonoesterase encoding microbial community of a vertisol and its derivative soil fractions[J]. Biology and Fertility of Soils, 2017, 53(4): 375-388. |
| [45] | Xu L, He N P, Li X Z, et al. Local community assembly processes shape β-diversity of soil phoD-harbouring communities in the northern Hemisphere steppes[J]. Global Ecology and Biogeography, 2021, 30(11): 2273-2285. |
 2023, Vol. 44
2023, Vol. 44


