2. 黑龙江大学现代农业与生态环境学院, 哈尔滨 150080
2. College of Advanced Agriculture and Ecological Environment, Heilongjiang University, Harbin 150080, China
随着人们对绿色食品需求的不断上升, 有机肥在农业生产中被广泛施用.施用畜禽粪便有机肥可使蔬菜的产量增加[1], 但畜禽养殖业中抗生素的广泛使用使畜禽粪便中残留有大量的抗生素、抗生素抗性基因(antibiotic resistance genes, ARGs)和各种可移动遗传元件(mobile genetic elements, MGEs), 导致AGRs在土壤中不断积累和传播[2, 3].此外, 土壤环境中的细菌菌群在选择压力下会产生抗生素抗性和ARGs, 这些AGRs和抗性菌能够通过食物链进入到人体当中, 严重威胁人类的身体健康[4].因此, 研究农用土壤中ARGs的丰度水平和转移能力具有十分重要的意义.
农用土壤中ARGs在土壤环境中的分布和丰度受农业生产活动的影响.有研究表明, 在畜禽养殖厂废水灌溉、畜禽粪便施用后, 土壤中检测到了磺胺类(sul1)、大环内酯类(ermB)等ARGs和MGEs[5~7].气候环境、土壤的利用方式以及种植年限等因素也会对土壤中ARGs的积累产生影响.有研究表明, 不同地区的农田土壤中sul的分布存在显著差异, 在北部地区土壤中相对丰度(以16S rRNA计)最高可达1.85×10-3[8].此外, 有研究还发现ARGs的相对丰度随着种植年限的延长而逐年增加, 且施加粪肥的设施土壤中ARGs的丰度明显高于露地土壤[9, 10].整合子是一种十分常见的MGEs, 参与ARGs在微生物之间的传播[11, 12], 有研究表明整合子可通过水平基因转移促进ARGs在土壤中的积累[13], 其中整合子intl1、intl2和intl3与ARGs的转移密切相关[2].
设施菜田、露地菜田和粮田等不同利用方式土壤中施肥量不同[14, 15]、气候环境不同导致ARGs的丰度也存在差异, 我国不同地区不同利用方式土壤中ARGs的积累特征目前鲜见报道[16~19].磺胺类和大环内酯类ARGs是农用土壤中常见的ARGs, 在我国多个地区均有检出, 污染隐患较大[20~22], 且其传播与整合子intl1有密切的联系[2].因此, 本研究选取了这两类ARGs和一种MEGs基因intl1进行定性和定量分析, 从而明确不同地区、不同利用方式和不同种植年限的土壤中ARGs的积累特征.这不仅能为农业安全生产提供参考, 也进一步为食品安全提供有力支撑.
1 材料与方法土壤样品ARGs的检测在东北农业大学园艺园林学院农业农村部东北地区园艺作物生物学与种质创新重点实验室完成.
1.1 试验材料 1.1.1 采样点本试验土壤取自我国黑龙江(HLJ)、吉林(JL)、辽宁(LN)、内蒙古(NMG)、新疆(XJ)、甘肃(GS)、云南(YN)、江苏(JS)、湖北(HB)、广东(GD)、海南(HN)、山西(SX)、河南(HEN)和山东(SD)共计14个省份, 通过运用生态学的原理和方法, 将各省份进行了整合与分区, 将14个省份划分为5个生态区, 分别是北部地区、西部地区、长江地区、中部地区与南部地区, 具体地点详见表 1.
|
|
表 1 试验取样地点 Table 1 Sampling location |
1.1.2 样品采集与处理
在2017年7~8月, 分别选取各省份具有代表性的不同利用方式(设施菜田、露地菜田和露地粮田)以及不同种植年限(1~3、7~10和15 a以上)土壤, 取土深度为5~20 cm.即每个省份选取设施菜田(SS)以及与设施菜田相邻近的露地菜田(CT)和露地粮田(LT)这3种不同利用方式和3种种植年限的9种土壤(处理), 共126个土壤样品, 每种土壤选取3个地点作为3次重复, 共378个样品.所有样品与付彦祥[23]所用的土壤一致, 采集后迅速放入保温箱, 带至实验室-80℃冰箱中保存, 用于DNA的提取.
1.2 试验方法 1.2.1 DNA的提取采用Power Soil® DNA Isolation Kit(MO BIO Laboratories, CA, USA)按说明提取DNA.用1%琼脂糖凝胶电泳检测提取的土壤DNA的完整性.使用NanoDropND-1000 UV-Vis分光光度计(Thermo Scientific, Rockwood, TN, USA)检测提取物的DNA浓度和质量(A260/A280).
1.2.2 土壤中抗生素抗性基因的定性和定量分析本研究选择了2种磺胺类(sul1、sul2)、2种大环内酯类(ermB、mefA)的ARGs和1种整合子基因(intl1), 共5种基因进行检测.通过普通PCR仪(PTC-200, 美国Bio-Rad)确定土壤中存在的目标基因, 检出基因通过实时荧光定量PCR仪(qTOWER, Analytik Jena, Germany)进行定量检测, 所用引物序列、扩增目标基因片段大小和退火温度(T)见表 2. qPCR反应体系(10 μL): 5 μL PCR-Mix、0.5 μL引物(10 μmol ·L-1)、1 μL DNA模板、3 μL无菌去离子水.qPCR反应条件: 95℃预变性3 min; 94℃变性20 s、T(℃)退火30 s; 72℃延伸30 s, 共40个循环, 72℃延伸7 min; 溶解曲线为65~95℃之间, 每5 s增加0.5℃, 期间停留30 s.
|
|
表 2 引物序列1) Table 2 Primer sequences |
1.3 数据分析
采用Microsoft Excel (Office 2010)对原始数据进行整理, 采用IBM SPSS Statistics 19.0软件中Tukey's HSD检验进行数据分析, 检验水平P < 0.05.柱形图和箱线图采用Origin 8.5软件进行图片绘制.线性回归分析采用R软件ggplot2包进行分析.
2 结果与分析 2.1 不同土壤中抗生素抗性基因的定性检测在14个省份的3、7和15 a设施菜田、露地菜田和露地粮田土壤中sul1、sul2和intl1在不同利用方式土壤中的检出率均为100%, 在设施菜田土壤中ermB和mefA的检出率也为100%, 而在露地菜田和粮田土壤中ermB的检出率为94%, mefA的检出率分别为92%和90%(表 3).
|
|
表 3 不同地区土壤中ARGs和intl1的检出情况1) Table 3 Detection of ARGs and intl1 in different soils |
2.2 同一利用方式不同种植年限土壤磺胺类抗生素抗性基因差异分析 2.2.1 同一利用方式不同种植年限土壤中sul1绝对丰度差异分析
粮田土壤中sul1的绝对丰度范围是1.28×106~4.53×107 copies ·g-1, 3、7和15 a粮田土壤中sul1的绝对丰度在江苏地区最高, 3 a粮田土壤中sul1的绝对丰度在广东地区最低, 7 a和15 a粮田土壤中sul1的绝对丰度在云南地区最低[图 1(a)].露地菜田土壤中sul1的绝对丰度范围是1.04×107~6.56×107 copies ·g-1, 3、7和15 a露地土壤中sul1的绝对丰度在黑龙江地区最高, 在湖北地区最低[图 1(b)].设施土壤中sul1的绝对丰度范围是5.08×107~3.11×108 copies ·g-1, 3 a和15 a设施土壤中sul1的绝对丰度在黑龙江地区最高, 7 a在山东地区最高. 3 a设施菜田土壤中sul1的绝对丰度在海南地区最低, 7 a在湖北地区最低, 15 a在云南地区最低[图 1(c)]. sul1的绝对丰度在黑龙江地区的15 a的设施菜田土壤中最高, 在广东3 a的粮田土壤中最低.

|
LT表示设施菜田邻近的露地粮田土壤, CT表示设施菜田邻近的露地菜田土壤, SS表示设施菜田土壤; 不同小写字母表示在0.05水平上有显著差异, 下同 图 1 不同地区同一利用方式和种植年限土壤中sul1绝对丰度的差异 Fig. 1 Difference in absolute abundance of sul1 in soils with the same utilization pattern and planting years in different areas |
粮田土壤中sul2的绝对丰度范围是1.78×106~2.83×107copies ·g-1, 3 a和7 a粮田土壤中sul2的绝对丰度在江苏地区最高, 15 a在黑龙江地区最高, 3 a粮田土壤中sul2的绝对丰度在广东地区最低, 7 a粮田土壤中sul2的绝对丰度在云南地区最低, 15 a粮田土壤中sul2的绝对丰度在河南地区最低[图 2(a)].露地菜田土壤中sul2的绝对丰度范围是1.13×107~6.44×107 copies ·g-1, 3、7和15 a露地菜田土壤中sul2的绝对丰度在吉林地区最高, 最低均在湖北地区[图 2(b)].设施菜田土壤中sul2的绝对丰度范围是2.67×107~2.85×108 copies ·g-1, 3 a和15 a设施菜田土壤sul2的绝对丰度在黑龙江地区最高, 7 a为山东地区, 3 a设施菜田土壤中sul2的绝对丰度在河南地区最低, 7 a在海南地区, 15 a在云南地区[图 2(c)]. sul2的绝对丰度在黑龙江地区的15 a棚室土壤中最高, 在广东3 a粮田土壤中最低(图 2).
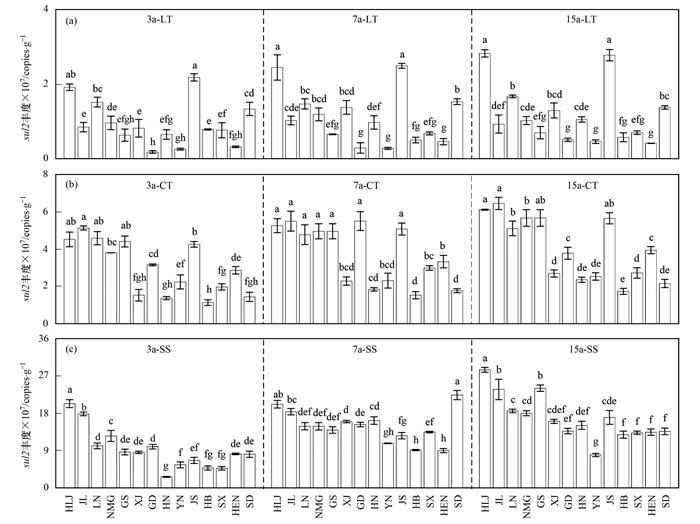
|
图 2 不同地区同一利用方式和种植年限土壤中sul2绝对丰度的差异 Fig. 2 Difference in absolute abundance of sul2 in soils with the same utilization pattern and planting years in different areas |
粮田土壤中ermB的绝对丰度范围是0~6.74×105copies ·g-1, 3 a和7 a粮田土壤中丰度在内蒙古地区最高, 15 a为黑龙江地区最高.3 a粮田土壤中丰度在海南, 湖北和山东地区最低, 7 a和15 a在黑龙江地区最低[图 3(a)].露地菜田土壤中ermB的绝对丰度范围是5.90×104~2.62×106copies ·g-1, 3、7和15 a露地菜田土壤中丰度均在内蒙古地区最高, 在云南地区最低[图 3(b)].设施菜田土壤中ermB的绝对丰度范围是1.52×105~8.78×106 copies ·g-1, 3 a和7 a设施土壤中丰度在内蒙古地区最高, 15 a在黑龙江地区最高, 最低均在云南地区[图 3(c)]. ermB的绝对丰度在黑龙江地区的15 a设施菜田土壤中最高, 在海南、湖北和山东3 a粮田土壤中最低(图 3).

|
图 3 不同地区同一利用方式和种植年限土壤中ermB绝对丰度的差异 Fig. 3 Difference in absolute abundance of ermB in soils with the same utilization pattern and planting years in different areas |
粮田土壤中mefA的绝对丰度范围是0~4.97×105 copies ·g-1, 3、7和15 a粮田土壤中绝对丰度在黑龙江地区最高, 3 a粮田土壤中绝对丰度在内蒙古、海南、云南、湖北和山东地区为最低, 7 a和15 a粮田土壤中绝对丰度最低为云南地区[图 4(a)].露地菜田土壤中mefA的绝对丰度范围是1.28×104~1.12×106 copies ·g-1, 3、7和15 a露地菜田土壤中丰度均在吉林地区最高, 最低均在海南地区[图 4(b)].设施菜田土壤中mefA的绝对丰度范围是1.16×105~5.44×106 copies ·g-1, 3、7和15 a设施菜田土壤中丰度均在黑龙江地区最高, 最低均在云南地区[图 4(c)]. mefA的绝对丰度在黑龙江地区的7 a设施菜田土壤中最高, 在内蒙古、海南、云南、湖北和山东的3 a粮田土壤中最低(图 4).
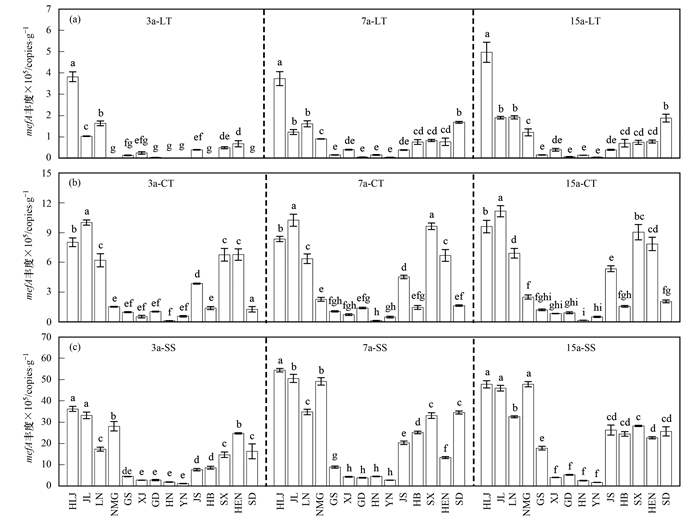
|
图 4 不同地区同一利用方式和种植年限土壤中mefA绝对丰度的差异 Fig. 4 Difference in absolute abundance of mefA in soils with the same utilization pattern and planting years in different areas |
在粮田土壤中 intl1的绝对丰度范围是2.66×106~5.28×107 copies ·g-1, 3、7和15 a粮田土壤中绝对丰度在新疆地区最高, 最低均在云南地区[图 5(a)].露地菜田土壤中 intl1的绝对丰度范围是9.97×106~2.34×108 copies ·g-1, 3、7和15 a露地菜田土壤中丰度均在黑龙江地区最高, 最低均在湖北地区[图 5(b)].设施菜田土壤中 intl1的绝对丰度范围是7.67×107~8.18×108copies ·g-1, 7 a和15 a设施菜田土壤中丰度在内蒙古地区最高, 3 a在黑龙江地区最高, 3 a设施菜田土壤中 intl1的丰度在海南地区最低, 7 a设施菜田土壤中 intl1的丰度在河南地区最低, 15 a设施菜田土壤中 intl1的丰度在湖北地区最低[图 5(c)]. intl1的绝对丰度在内蒙古地区的7 a棚室土壤中最高, 在云南15 a粮田土壤中最低(图 5).

|
图 5 不同地区同一利用方式和种植年限土壤中 intl1绝对丰度的差异 Fig. 5 Difference in absolute abundance of intl1 in soils with the same utilization pattern and planting years in different areas |
各生态区设施菜田土壤中 sul1、sul2、ermB、mefA和 intl1的绝对丰度显著高于露地菜田和粮田土壤(图 6).北部地区露地菜田土壤中 sul1和 sul2的绝对丰度显著高于露地粮田土壤[图 6(a)和6(b)].北部与西部地区露地菜田土壤中 intl1的绝对丰度显著高于露地粮田土壤[图 6(e)].
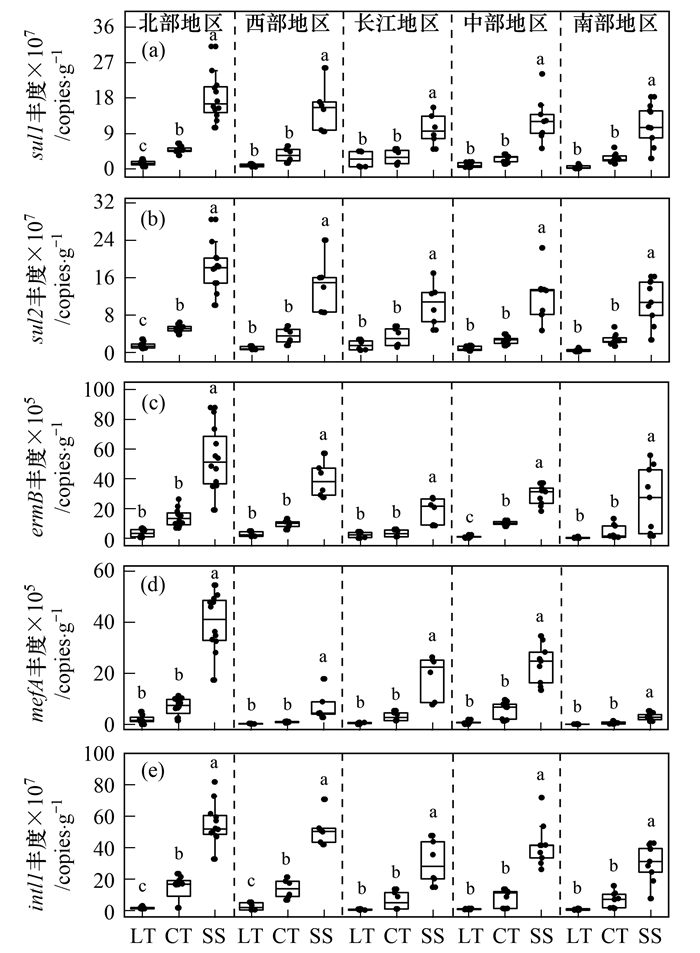
|
图 6 不同生态区不同利用方式土壤中ARGs与MGEs的绝对丰度差异 Fig. 6 Differences in ARGs and MGEs in soils of different utilization patterns and ecoregions |
设施菜田土壤的 sul1、sul2、ermB、mefA和 intl1的绝对丰度与土壤种植年限呈显著线性正相关关系[图 7(c)].除露地菜田土壤中的 sul1和 sul2外, 露地菜田土壤和粮田土壤中的ARGs与土壤种植年限没有显著线性相关[图 7(a)和7(b)].

|
(a) 露地粮田土壤, (b) 露地菜田土壤, (c) 设施菜田土壤 图 7 ARGs和MGEs与种植年限的线性回归 Fig. 7 Linear regression between ARGs, MGEs, and planting years |
抗生素抗性基因 sul1、sul2、ermB和mefA的绝对丰度呈显著正相关, 且各抗生素抗性基因的绝对丰度与整合子基因 intl1的绝对丰度均呈显著正相关(表 4).
|
|
表 4 ARGs和MGEs间Spearman相关性分析1) Table 4 Relationship analysis between ARGs and MGEs |
3 讨论
畜禽粪便是环境中ARGs的主要来源, 农用土壤中畜禽粪便的广泛施用加剧了其在土壤环境中的传播和扩散, 严重威胁着人类健康.本研究对来自14个省份不同利用方式的土壤进行了磺胺类(sul1、sul2)、大环内酯类(mefA、mefB)的ARGs以及整合子(intl1)的定性和定量分析, 发现在不同的土壤中均检测到了这两类ARGs和 intl1, 表明农用土壤中存在磺胺类和大环内酯类ARGs.Chen等[27]研究发现在所有类型的检测土壤中大环内酯类抗性基因ermB的检出频率较高, 这与本研究的结果基本一致.张兰河等[28]研究也发现长期施用有机肥的温室和大田土壤中 sul1和 sul2的检出率为100%.产生这种现象的原因可能是sul基因在土壤中产生了广泛的传播. sul基因一般可以通过整合子、移动质粒和转座子这3种方式进行转移及传播[29, 30], 且sul基因和整合子 intl1总是存在正相关关系[31].本试验研究结果中 intl1的检出率也是100%, 并且和sul基因的绝对丰度也存在着显著的正相关, 表明sul基因可以通过整合子来进行水平转移, 从而使其在环境中大量传播.
我国不同省份的气候条件、土壤类型以及抗生素的使用方式均会影响ARGs的绝对丰度[32].在本试验中3种土壤利用方式ARGs的绝对丰度存在显著差异, 各地区3、7和15 a设施土壤中 sul1、sul2、ermB、mefA和 intl1的绝对丰度显著高于相同种植年限下的露地菜田与粮田土壤.有机肥的施用可以增加ARGs的丰度, 与露地菜田和粮田土壤相比, 设施蔬菜生产过程中施用了大量的有机肥, 且设施环境密闭, 温度较高, 不易受到外界环境因素的影响, 有助于ARGs的大量积累, 这可能是导致设施土壤中ARGs的绝对丰度高于露地菜田和粮田的原因[33, 34].此外, 在本研究中, 各地区ARGs的丰度在露地菜田和粮田土壤中也存在着差异, 露地菜田土壤除施用化肥之外也会伴随着施用有机肥, 这可能导致了露地菜田土壤中ARGs的丰度高于粮田土壤.但是湖北、海南和山东等几个地区中露地粮田和菜田土壤中ARGs丰度之间无显著性差异, 这一结果与前人的研究相反[35].通过对土壤背景的调查发现湖北、海南和山东这几个地区露地粮田和菜田土壤所使用的肥料基本一致, 而其他地区露地菜田土壤中施肥量高于粮田土壤.这可能是导致3个地区ARGs的绝对定量值无差异的原因.此外, 有研究表明微生物之间ARGs的传播主要依靠横向基因的转移, ARGs被整合到一些可移动元件上在微生物之间进行传播, 从而影响了ARGs的丰度[36].在本试验中尽管 intl1在各地区中均有检出, 但是这3个地区的整合子 intl1的绝对丰度在露地粮田和菜田土壤之间无显著差异, 这可能是导致3个地区露地粮田和菜田土壤中ARGs的绝对丰度无显著差异的另一个原因.
将14个省份分为5个生态区, 本研究发现不同生态区不同利用方式土壤中, 均是设施土壤中基因的绝对丰度最高, 各生态区相比发现达到最高丰度的地区大部分位于北部地区.Zhou等[37]研究结果也表明北部省份ARGs的污染高于南部.有研究表明, 温度是影响ARGs污染程度的一个重要的因素, 温度过高有利于生化反应的进行, 使得环境中菌群的DNA组成结构发生改变, 从而去除ARGs[38~40].因此, 造成本试验结果的原因可能是由于温度的不同, 从而使得北部地区中各类ARGs的丰度偏高.同时, 栽培方式也是影响ARGs污染程度的因素之一[37].ARGs独特的空间分布格局可能受土壤共存污染物、土壤特性和气候条件的区域差异的影响[41].因此我国北部地区ARGs的污染水平绝对丰度较高, 可能与人类活动、土壤类型和气候条件有关[37].
长期施用粪肥会增加土壤中ARGs和MGEs的含量[42].有研究表明, 施用有机肥的温室土壤中ARGs的丰度与种植年限呈显著正相关[43].本研究通过对不同利用方式土壤和种植年限之间进行相关性分析也发现, 设施菜田土壤中 sul1、sul2、ermB、mefA和intl1的绝对丰度与土壤的种植年限呈显著正相关, 除露地菜田土壤 sul1和 sul2外, 露地菜田和粮田土壤中ARGs与土壤种植年限没有显著线性相关.为了提高经济效益, 改善土壤质量以及提高农作物对土壤养分的需求, 设施菜田中畜禽粪便的添加量和添加次数明显高于露地菜田, 畜禽粪便中含有大量的抗生素及ARGs, 设施菜田中畜禽粪便的大量施用导致设施土壤中ARGs的丰度逐年升高.
ARGs在土壤中的扩散与MGEs密切相关.有研究表明, 磺胺类抗性基因 sul1与整合子intl, 大环内酯类抗性基因ermB和mefA与转座子 tnpA03均呈显著正相关[44, 45].王百羽等[32]研究表明菜田土壤中磺胺类和氯霉素类ARGs的绝对丰度与MGEs呈显著正相关, 表明较高的MGEs丰度有助于ARGs发生水平转移.何燕等[46]研究表明, 稻田土壤中氯霉素类、四环素类抗性基因和MGEs的丰度呈极显著正相关, 说明ARGs的水平转移可能促进其在土壤中的迁移和富集.ARGs的水平迁移是导致抗生素耐药性在环境中快速传播和扩散的主要原因[47].本研究中, Spearman相关分析表明, 磺胺类和大环内酯类ARGs的丰度与MGEs的丰度呈显著正相关, 表明MGEs对促进ARGs在农用土壤中的传播和转移具有重要作用.
4 结论(1) 磺胺类抗性基因和整合子 intl1在不同利用方式土壤中的检出率均为100%, 大环内酯类抗性基因在设施菜田土壤中的检出率为100%, 而在露地粮田和菜田土壤中的检出率为90% ~94%.
(2) 磺胺类和大环内酯类ARGs的绝对丰度在黑龙江地区种植15 a的设施土壤中最高, 整合子 intl1的绝对丰度在内蒙古地区种植15 a的设施土壤中最高.
(3) 各地区3、7和15 a设施土壤中磺胺类、大环内酯类ARGs和整合子 intl1的绝对丰度显著高于相同年限下的露地菜田与粮田土壤, 设施菜田7 a和15 a土壤中ARGs与intl1的绝对丰度显著高于3 a土壤.
(4) 设施菜田土壤中ARGs与intl1的绝对丰度与种植年限呈显著正相关.ARGs的绝对丰度与intl1的绝对丰度呈显著正相关.
| [1] |
杨文叶, 季淑枫, 李丹, 等. 连续施用商品有机肥对耕地质量及蔬菜产量的影响[J]. 农业资源与环境学报, 2014, 31(4): 319-322. Yang W Y, Ji S F, Li D, et al. Effect of continuous application of commercial organic manure on farmland quality and vegetable yield[J]. Journal of Agricultural Resources and Environment, 2014, 31(4): 319-322. DOI:10.13254/j.jare.2014.0123 |
| [2] |
李金阳. 施用牛粪/堆肥对农田土壤和生菜中抗生素抗性基因传播的影响[D]. 济南: 济南大学, 2021. Li J Y. Effects of application of cow manure/compost on the transmission of antibiotic resistance genes in farmland soil and lettuce[D]. Jinan: University of Jinan, 2021. |
| [3] |
彭晶, 高英志, 谷月, 等. 鸡粪有机肥对土壤中抗生素抗性基因和整合酶基因的影响[J]. 环境工程学报, 2019, 13(4): 984-991. Peng J, Gao Y Z, Gu Y, et al. Effects of chicken manure organic fertilizer on antibiotic resistance genes and integrase genes in soil[J]. Chinese Journal of Environmental Engineering, 2019, 13(4): 984-991. |
| [4] |
李晓天, 黄焯燊, 汤有千, 等. 畜禽养殖废物中抗生素和重金属抗性基因的产生机制和控制方法研究进展[J]. 应用生态学报, 2022, 33(6): 1719-1728. Li X T, Huang Z S, Tang Y Q, et al. Generation mechanism and control methods of antibiotic and heavy metal resistance genes in poultry waste: a review[J]. Chinese Journal of Applied Ecology, 2022, 33(6): 1719-1728. DOI:10.13287/j.1001-9332.202210.029 |
| [5] | Chen Q L, An X L, Li H, et al. Long-term field application of sewage sludge increases the abundance of antibiotic resistance genes in soil[J]. Environment International, 2016, 92-93: 1-10. DOI:10.1016/j.envint.2016.03.026 |
| [6] | Dungan R S, McKinney C W, Leytem A B. Tracking antibiotic resistance genes in soil irrigated with dairy wastewater[J]. Science of the Total Environment, 2018, 635: 1477-1483. DOI:10.1016/j.scitotenv.2018.04.020 |
| [7] | Pu C J, Liu H, Ding G C, et al. Impact of direct application of biogas slurry and residue in fields: in situ analysis of antibiotic resistance genes from pig manure to fields[J]. Journal of Hazardous Materials, 2018, 344: 441-449. DOI:10.1016/j.jhazmat.2017.10.031 |
| [8] |
周雨婷. 中国农田土壤中抗生素抗性基因分布及典型污染物浓度特征、对其影响与风险评估[D]. 杭州: 浙江大学, 2020. Zhou Y T. Distribution of antibiotic resistance genes and concentration characteristics, influence on antibiotic resistance genes, and risk assessments of typical pollutants in agricultural soils in China[D]. Hangzhou: Zhejiang University, 2020. |
| [9] |
龚小雅, 宋建宇, 吴凤芝. 我国不同利用方式土壤四环素类抗性基因积累的研究[J]. 农业环境科学学报, 2023, 42(1): 87-100. Gong X, Song J, Wu F. Study on the accumulation of tetracycline resistance genes in soil with different utilization patterns in China[J]. Journal of Agro-Environment Science, 2023, 42(1): 87-100. |
| [10] | Fang H, Wang H F, Cai L, et al. Prevalence of antibiotic resistance genes and bacterial pathogens in long-term manured greenhouse soils as revealed by metagenomic survey[J]. Environmental Science & Technology, 2015, 49(2): 1095-1104. |
| [11] | Li J J, Xin Z H, Zhang Y Z, et al. Long-term manure application increased the levels of antibiotics and antibiotic resistance genes in a greenhouse soil[J]. Applied Soil Ecology, 2017, 121: 193-200. DOI:10.1016/j.apsoil.2017.10.007 |
| [12] | Zhu Y G, Zhao Y, Li B, et al. Continental-scale pollution of estuaries with antibiotic resistance genes[J]. Nature Microbiology, 2017, 2(4). DOI:10.1038/nmicrobiol.2016.270 |
| [13] | Elizabeth R, Chanda D D, Chakravarty A, et al. Association of glycerol kinase gene with class 3 integrons: a novel cassette array within Escherichia coli[J]. Indian Journal of Medical Microbiology, 2018, 36(1): 104-107. DOI:10.4103/ijmm.IJMM_17_188 |
| [14] | 王娟娟. 我国蔬菜施肥现状调查研究[J]. 中国农技推广, 2016, 32(6): 11-13. DOI:10.3969/j.issn.1002-381X.2016.06.003 |
| [15] |
由海霞. 近十一年我国蔬菜播种面积的变化规律分析[J]. 北方园艺, 2016(6): 168-170. You H X. Variation of vegetable sown areas in China during the past eleven years[J]. Northern Horticulture, 2016(6): 168-170. |
| [16] | Gao P, Munir M, Xagoraraki I. Correlation of tetracycline and sulfonamide antibiotics with corresponding resistance genes and resistant bacteria in a conventional municipal wastewater treatment plant[J]. Science of the Total Environment, 2012, 421-422: 173-183. |
| [17] | Huang X, Liu C X, Li K, et al. Occurrence and distribution of veterinary antibiotics and tetracycline resistance genes in farmland soils around swine feedlots in Fujian Province, China[J]. Environmental Science and Pollution Research, 2013, 20(12): 9066-9074. DOI:10.1007/s11356-013-1905-5 |
| [18] | Luo Y, Xu L, Rysz M, et al. Occurrence and transport of tetracycline, sulfonamide, quinolone, and macrolide antibiotics in the Haihe River Basin, China[J]. Environmental Science & Technology, 2011, 45(5): 1827-1833. |
| [19] | Ji X L, Liu F, Shen Q H, et al. Quantitative detection of sulfonamides and tetracycline antibiotics and resistance genes in sewage farms[J]. Ecology and Environmental Sciences, 2011, 20(5): 927-933. |
| [20] |
陈运杰, 郭欣妍, 杨艺, 等. 玉米根际土壤中大环内酯类抗性基因的分布特征[J]. 生态与农村环境学报, 2019, 35(9): 1182-1189. Chen Y J, Guo X Y, Yang Y, et al. Distribution pattern of genes conferring resistance to macrolides in rhizosphere soil of maize[J]. Journal of Ecology and Rural Environment, 2019, 35(9): 1182-1189. |
| [21] |
何轮凯. 粪肥施用农田土壤中抗生素与耐药基因污染特征与归趋研究[D]. 广州: 中国科学院大学(中国科学院广州地球化学研究所), 2018. He L K. Characteristics and fate of antibiotics and antibiotic resistance genes in manure application soil[D]. Guangzhou: Guangzhou Institute of Geochemistry, Chinese Academy of Sciences, 2018. |
| [22] |
鲁艺杰. 农田土壤磺胺类抗生素及抗性基因污染调查研究[D]. 新乡: 河南师范大学, 2019. Lu Y J. Investigation on sulfonamide antibiotics and resistance gene pollution in farmland and soil[D]. Xinxiang: Henan Normal University, 2019. |
| [23] |
付彦祥. 我国不同生态区及不同土地利用方式下土壤细菌群落结构的变化[D]. 哈尔滨: 东北农业大学, 2020. Fu Y X. Changes of soil bacterial communities by land-use intensity in different ecological regions in China[D]. Harbin: Northeast Agricultural University, 2020. |
| [24] | Pei R T, Kim S C, Carlson K H, et al. Effect of river landscape on the sediment concentrations of antibiotics and corresponding antibiotic resistance genes (ARG)[J]. Water Research, 2006, 40(12): 2427-2435. DOI:10.1016/j.watres.2006.04.017 |
| [25] | Eitel Z, Sóki J, Urbán E, et al. The prevalence of antibiotic resistance genes in Bacteroides fragilis group strains isolated in different European countries[J]. Anaerobe, 2013, 21: 43-49. DOI:10.1016/j.anaerobe.2013.03.001 |
| [26] |
赵祥. 施用粪肥的设施菜地土壤中抗生素及抗性基因多样性及丰度的研究[D]. 泰安: 山东农业大学, 2017. Zhao X. Diversity and abundance of antibiotics and ARGs in vegetable soil with manure application[D]. Tai'an: Shandong Agricultural University, 2017. |
| [27] | Chen J, Michel F C Jr, Sreevatsan S, et al. Occurrence and persistence of erythromycin resistance genes (erm) and tetracycline resistance genes (tet) in waste treatment systems on swine farms[J]. Microbial Ecology, 2010, 60(3): 479-486. DOI:10.1007/s00248-010-9634-5 |
| [28] |
张兰河, 王佳佳, 高敏, 等. 施用畜禽粪便有机肥土壤抗生素抗性基因污染状况[J]. 生态与农村环境学报, 2016, 32(4): 664-669. Zhang L H, Wang J J, Gao M, et al. Pollution of antibiotics resistant genes in farmland amended with livestock organic manure[J]. Journal of Ecology and Rural Environment, 2016, 32(4): 664-669. |
| [29] | Smalla K, Heuer H, Gotz A, et al. Exogenous isolation of antibiotic resistance plasmids from piggery manure slurries reveals a high prevalence and diversity of IncQ-like plasmids[J]. Applied and Environmental Microbiology, 2000, 66(11): 4854-4862. DOI:10.1128/AEM.66.11.4854-4862.2000 |
| [30] | Antunes P, Machado J, Sousa J C, et al. Dissemination of sulfonamide resistance genes (sul1, sul2, and sul3) in portuguese Salmonella enterica strains and relation with integrons[J]. Antimicrobial Agents and Chemotherapy, 2005, 49(2): 836-839. |
| [31] | Heuer H, Smalla K. Manure and sulfadiazine synergistically increased bacterial antibiotic resistance in soil over at least two months[J]. Environmental Microbiology, 2007, 9(3): 657-666. |
| [32] |
王百羽, 张珣, 王宝玉, 等. 沈阳蔬菜地土壤中典型抗生素抗性基因与可移动元件分布特征[J]. 生态学杂志, 2021, 40(7): 2113-2119. Wang B Y, Zhang X, Wang B Y, et al. Distribution of typical antibiotic resistance genes and mobile genetic elements in vegetable soils of Shenyang[J]. Chinese Journal of Ecology, 2021, 40(7): 2113-2119. |
| [33] | Li C, Chen J Y, Wang J H, et al. Occurrence of antibiotics in soils and manures from greenhouse vegetable production bases of Beijing, China and an associated risk assessment[J]. Science of the Total Environment, 2015, 521-522: 101-107. |
| [34] | Zheng X R, Pfordt A, Khatri L, et al. Contrasting patterns of colonization with Verticillium longisporum in winter-and spring-type oilseed rape (Brassica napus) in the field and greenhouse and the role of soil temperature[J]. Plant Disease, 2019, 103(8): 2090-2099. |
| [35] | Marti R, Scott A, Tien Y C, et al. Impact of manure fertilization on the abundance of antibiotic-resistant bacteria and frequency of detection of antibiotic resistance genes in soil and on vegetables at harvest[J]. Applied and Environmental Microbiology, 2013, 79(18): 5701-5709. |
| [36] | Zhu Y G, Johnson T A, Su J Q, et al. Diverse and abundant antibiotic resistance genes in Chinese swine farms[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(9): 3435-3440. |
| [37] | Zhou Y T, Niu L L, Zhu S Y, et al. Occurrence, abundance, and distribution of sulfonamide and tetracycline resistance genes in agricultural soils across China[J]. Science of the Total Environment, 2017, 599-600: 1977-1983. |
| [38] | Ahmed I, Zhang Y P, Sun P Y, et al. Sensitive response mechanism of ARGs and MGEs to initial designed temperature during swine manure and food waste co-composting[J]. Environmental Research, 2023, 216. DOI:10.1016/j.envres.2022.114513 |
| [39] | Yu Q L, Han Q, Shi S Q, et al. Metagenomics reveals the response of antibiotic resistance genes to elevated temperature in the Yellow River[J]. Science of the Total Environment, 2023, 859. DOI:10.1016/j.scitotenv.2022.160324 |
| [40] |
许继飞, 张秋萍, 朱天骄, 等. 温度和搅拌对牛粪厌氧消化系统抗生素抗性基因变化和微生物群落的影响[J]. 环境科学, 2021, 42(6): 2992-2999. Xu J F, Zhang Q P, Zhu T J, et al. Effects of temperature and stirring on the changes of antibiotic resistance genes and microbial communities in anaerobic digestion of dairy manure[J]. Environmental Science, 2021, 42(6): 2992-2999. |
| [41] | Wu N, Qiao M, Zhang B, et al. Abundance and diversity of tetracycline resistance genes in soils adjacent to representative swine feedlots in China[J]. Environmental Science & Technology, 2010, 44(18): 6933-6939. |
| [42] |
何轮凯, 张敏, 刘有胜, 等. 长期施用粪肥的土壤中抗生素耐药基因的消减规律[J]. 华南师范大学学报(自然科学版), 2018, 50(1): 1-10. He L K, Zhang M, Liu Y S, et al. Dynamics of antibiotic resistance genes in soil chronically fertilized by swine manure[J]. Journal of South China Normal University (Natural Science Edition), 2018, 50(1): 1-10. |
| [43] | Zhang H P, Zhang Q K, Song J J, et al. Tracking resistomes, virulence genes, and bacterial pathogens in long-term manure-amended greenhouse soils[J]. Journal of Hazardous Materials, 2020, 396. DOI:10.1016/j.jhazmat.2020.122618 |
| [44] | Li W, Li Y Y, Zheng N G, et al. Occurrence and distribution of antibiotics and antibiotic resistance genes in the guts of shrimp from different coastal areas of China[J]. Science of the Total Environment, 2022, 815. DOI:10.1016/j.scitotenv.2021.152756 |
| [45] | Wang R, Chen D W, Wang F, et al. An insight into the exploration of proliferation of antibiotic resistance genes in high-fat diet induced obesity mice[J]. Genomics, 2021, 113(4): 2503-2512. |
| [46] |
何燕, 朱冬, 王东, 等. 四川省稻田土壤的抗生素抗性基因多样性研究[J]. 农业环境科学学报, 2020, 39(6): 1249-1258. He Y, Zhu D, Wang D, et al. Diversity of antibiotic resistance genes in paddy soils in Sichuan Province, China[J]. Journal of Agro-Environment Science, 2020, 39(6): 1249-1258. |
| [47] | Dantas G, Sommer M O A. How to fight back against antibiotic resistance[J]. American Scientist, 2014, 102(1): 42-51. |
 2023, Vol. 44
2023, Vol. 44


