2. 山西大学黄土高原研究所, 黄土高原生态恢复山西省重点实验室, 太原 030006;
3. 山西省林业和草原工程总站, 太原 030006;
4. 忻州师范学院生物系, 忻州 034003
2. Shanxi Key Laboratory of Ecological Restoration for Loess Plateau, Institute of Loess Plateau, Shanxi University, Taiyuan 030006, China;
3. Shanxi Forestry and Grassland General Engineering Station, Taiyuan 030006, China;
4. Department of Biology, Xinzhou Normal University, Xinzhou 034003, China
草地提供重要的生态服务, 在生物地球化学循环和温室气体调节中发挥重要作用.草地生态系统的功能在很大程度上取决于地下微生物群落的功能多样性和活性[1, 2].土壤微生物调节着许多对草地生态系统功能至关重要的生物地球化学过程, 包括养分循环, 其组成和多样性对干扰很敏感[3, 4].土壤微生物群落的结构和功能被认为是草地退化的关键指标[5].因此, 研究草地退化对土壤微生物群落的影响, 对于提高草地退化机制的认识, 预测和控制退化草地的发展具有十分重要的意义.
草地退化对土壤微生物群落影响的研究主要集中在内蒙古草原的温带草原和青藏高原的高寒草甸[4, 6, 7], 而草地退化对黄土高原亚高山草甸微生物群落的影响知之甚少.Li等[6]发现青藏高原高寒草甸退化显著增加了土壤微生物多样性, 细菌和真菌的多样性与植物多样性都不显著相关[8].Che等[7]的研究结果表明草地退化斑块的形成显著降低了微生物呼吸速率, 改变了微生物类群间的互作模式, 增加了真菌多样性, 但对微生物丰度没有显著影响.以上这些研究主要关注微生物群落的分类多样性, 由于其具有高度的功能冗余, 无法准确预测微生物的功能特征[9].相比于物种分类特征, 功能基因能更好地预测微生物群落对生境变化的响应[10].特别是近年来, 鸟枪法宏基因组测序的进展极大地提高了描述环境宏基因组的能力, 为了解微生物群落的复杂性及其功能特征提供了许多新的见解[11].因此, 利用宏基因组测序评估微生物功能基因多样性的变化可以更好地阐明亚高山草甸生态系统退化的过程与机制.
亚高山草甸是高寒草甸的一种类型, 以耐寒冷、密丛短根茎嵩草以及苔草、禾草、杂类草为建群植物的草甸群落.在山西省较大的山系中, 几乎都有亚高山草甸类草地的分布(约35.3万hm2), 占全省草地总面积的10%左右, 其分布位置主要在五台山、太岳山、吕梁山、云中山、中条山、恒山等山系林线以上的高海拔地带(一般在海拔1 700 m以上).多年来, 在自然因素与人为因素的双重叠加干扰下, 山西亚高山草甸面临严重的退化问题, 导致生态系统服务和生态功能降低, 严重威胁着区域的生态安全[12].本研究采用空间-时间替代法, 以五台山不同退化演替阶段亚高山草甸土壤微生物群落为研究对象, 借助宏基因组学的优势, 探讨:①草甸退化对土壤微生物群落分类和功能多样性的影响; ②草甸退化过程中微生物群落潜在功能途径的变化; ③土壤微生物群落分类和功能结构的变化及主要驱动因子.本研究通过理解土壤微生物群落结构和功能对亚高山草甸退化的响应机制, 旨在为亚高山草甸生态监测和保护修复提供理论依据和数据支撑.
1 材料与方法 1.1 研究区概况研究区五台山位于山西省忻州市东北部, 主峰叶斗峰海拔3 061 m, 是华北最高峰.沿海拔梯度有明显的植被垂直带谱, 海拔由低到高依次包括落叶阔叶林、落叶针叶林、常绿针叶林、亚高山灌丛和草甸等.五台山亚高山草甸是华北地区特有的亚高山草甸自然景观代表, 也是华北面积最大的高山亚高山草甸生态系统之一, 主要分布在海拔2 000 m林线以上的地带, 面积106 993 hm2[3].亚高山地区属于高山气候区, 年平均气温为-4.2℃, 极端最低气温为-44.8℃, 年降水量为966.3 mm.五台山亚高山草甸是优良的高山夏季牧场, 全年放牧期集中在6~8月, 为了便于畜养管理, 大量牲畜高密度地集中在一起, 导致超载, 植被破坏严重, 部分地区形成退化斑块甚至裸露地[13]. 据估计, 不同退化程度的草地约占五台山草地总面积的3/5.退化草甸植被覆盖度和产草量明显下降, 植物群落结构趋于单一, 优质牧草减少, 杂草、毒草比例增加, 土壤侵蚀严重等一系列次生问题相继出现[14].
1.2 样地设置与样品采集在五台山草甸自然保护区建立4个不同退化水平亚高山草甸样地, 包括未退化(ND)、轻度退化(LD)、中度退化(MD)和重度退化(HD)草甸.在植被调查的基础上, 采用Wang等[15]描述的指标评价草甸退化程度.每个草甸在地形条件相似的条件下随机设置100 m×100 m的样地, 样地间距不超过500 m. 2018年8月, 用完全随机设计, 在每个样地中设置3个样方(1 m×1 m)进行植被调查和土壤采样, 样方之间距离大于40 m, 共12个样方.采用直径为10 cm的无菌土钻, 按对角线多点(每样方5芯)混合取样模式, 对每个样方10 cm深度土壤进行采样.将样方中的5子样均匀混合形成复合样品.所有土壤样本就地保存在干冰箱中, 并在一天内带回实验室.每个土壤样本去除根系、凋落物、石块和碎屑后, 过2 mm的筛子, 分成两部分.一部分土壤样品保存在-80℃进行DNA提取, 另一部分土壤风干进行理化分析.土壤理化性质测定和植被调查方法和结果参见课题组前期研究[3].
1.3 DNA提取、宏基因组测序及生物信息学分析使用E.Z.N.A.®土壤DNA试剂盒(Omega Bio-tek, USA)按照说明书从0.5 g土壤样品中提取土壤DNA.使用NanoDrop 2000光谱仪(Thermo Scientific, Wilmington, DE, USA)评估DNA的质量和浓度. 取1 μg检测合格的DNA样品送往上海美吉生物医药技术有限公司Illumina HiSeq4000平台(Illumina Inc., San Diego, CA, USA)上进行末端配对测序.使用软件fastp(https://github.com/OpenGene/fastp)剪切序列3′端和5′端的adapter序列, 去除质量剪切后长度小于50 bp、平均质量值低于20以及含N碱基的reads, 保留高质量的pair-end reads和single-end reads用于宏基因组分析.基于succinct de Bruijn graphs方法, 使用MEGAHIT(https://github.com/voutcn/megahit)组装具有不同测序深度的序列.选择长度大于等于300 bp的Contigs作为最终组装结果, 并用于下一步的基因预测和注释.使用MetaGene(http://metagene.cb.k.u-tokyo.ac.jp/)对contigs中的开放阅读框(ORFs)进行预测.选取核酸长度大于等于100bp的基因, 翻译成氨基酸序列, 得到每个样本的基因预测结果统计表.使用CD-HIT对所有样本的预测基因序列进行聚类分析(identity≥95%, coverage ≥90%), 每个类中最长序列为代表序列, 构建非冗余基因集.使用SOAPaligner (http://soap.genomics.org.cn/)将每个经过质检reads映射到非冗余基因集(identity ≥95%), 并计算相应样本中基因的丰度信息.使用BLASTP(http://blast.ncbi.nlm.nih.gov/Blast.cgi)将非冗余基因集与NCBI-NR、KEGG(Kyoto Encyclopedia of Genes and Genomes, http://kobas.cbi.pku.edu.cn/home.do)数据库比对, 获得物种、功能注释信息, 然后使用物种或功能类别对应的基因丰度总和计算该物种分类和功能的丰度(BLAST比对参数设置期望值E-value为1e-5).
1.4 数据分析使用R studio vegan包中“diversity”函数计算微生物分类(基于宏基因组物种水平)和功能(基于KEGG level 3)α多样性指数(Shannon指数).利用非度量多维尺度(non-metric multidimensional scaling, NMDS)和相似性分析(analysis of similarity, ANOSIM)对不同退化阶段草甸土壤微生物群落的分类和功能之间的Bray-Curtis距离进行分析.采用冗余分析(redundancy analysis, RDA) 评价微生物群落结构与土壤物理化学变量之间的相关性.使用R中“forward. sel”函数对土壤理化变量进行999次蒙特卡洛排列测试的正向选择, 选择具有统计学意义(P<0.05)的变量进行下一步的冗余分析.用STAMP(statistical analysis of metagenomic profiles)软件比较未退化和退化草甸土壤微生物功能基因相对丰度的差异.利用Pearson相关系数计算微生物与植物群落α和β多样性之间的相关性. 通过加权的UniFrac距离矩阵, 使用Mantel和偏Mantel分析来确定土壤性质和植被特征与微生物群落之间的相关性.利用IBM SPSS statistics 20, 采用单因素方差分析和Duncan多重比较法对微生物门和属相对丰度、多样性进行比较, 显著性水平设为P<0.05.
2 结果与分析 2.1 不同退化阶段亚高山草甸土壤微生物群落的组成和丰度通过宏基因组测序, 12个土壤样品共产生了548 335 800个原始reads(平均每个样品45 694 650个), 98.9%的reads通过下游分析的质量控制.通过序列组装和基因预测, 分别产生了1 624 586条contigs序列和1 851 886条ORFs序列.去冗余后, 构建非冗余基因集, 基因数为20 270个, 总序列长度为8 930 268 bp, 平均序列长度440.57 bp.将样本序列与NCBI-NR数据库中的序列进行比较, 获得了物种分类学丰度数据, 共筛选出43 037 988个基因, 其中细菌基因42 662 408个, 占所有基因的99.1%, 真菌基因9 086个, 古生菌基因124 962个, 病毒基因6 940个.共鉴定出49个门、101个纲、202个目、346个科、910个属和2 543个种.8个优势门(相对丰度>1%)均为细菌门, 相对丰度从高到低依次为: 变形菌门(Proteobacteria, 43.79%)、放线菌门(Actinobacteria, 28.28%)、酸杆菌门(Acidobacteria, 7.76%)、绿弯菌门(Chloroflexi, 3.14%)、芽单胞菌门(Gemmatimonadetes, 2.32%)、硝化螺旋菌门(Nitrospirae, 2.08%)、厚壁菌门(Firmicutes, 1.68%)和疣微菌门(Verrucomicrobia, 1.33%)(图 1).4种不同退化阶段亚高山草甸之间放线菌门、拟杆菌门、硝化螺旋菌门和Parcubacteria的相对丰度存在显著差异(P<0.05, 图 1).在属水平, 慢生根瘤菌属(Bradyrhizobium)、土壤红杆菌属(Solirubrobacter)、鞘脂单胞菌属(Sphingomonas)、类诺卡氏菌属(Nocardioides)、硝化螺菌属(Nitrospira)、链霉菌属(Streptomyces)、分枝杆菌属(Mycobacterium)和Pyrinomonas在不同退化阶段亚高山草甸间存在显著差异(P<0.05, 图 1).
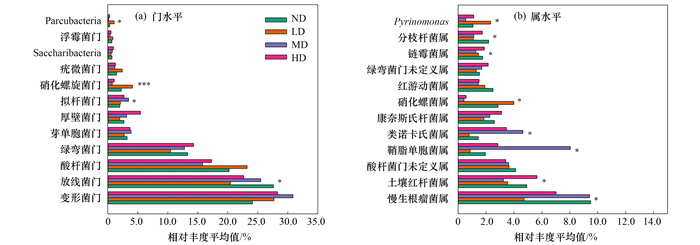
|
选取相对丰度平均值最高的12个门和属分别定义为优势门和优势属; ***表示P<0.001; **表示P<0.01; *表示P<0.05 图 1 宏基因组测序分析土壤微生物优势门和属水平相对丰度平均值差异 Fig. 1 Differences in the relative abundance of the dominant microbialphyla and genera by metagenomic sequencing |
基于KEGG数据库, 对不同退化程度亚高山草甸土壤中获得的非冗余基因集进行了基因功能预测, 共有21 158 140条reads可以对应到相应的功能注释结果中.在level 1水平上, 属于代谢(metabolism)类别的reads有16 728 792条, 属于遗传信息处理(genetic information processing)类别的有1 903 764条, 属于环境信息处理(environmental information processing)类别的有1 514 323条, 属于细胞过程(cellular processes)类别的有1 240 690条, 属于人类疾病(human diseases)类别的有922 396条, 属于生物系统(organismal systems)类别的有642 890条.
在KEGG level 3水平上, 退化草甸中参与碳代谢(carbon metabolism)、氨基酸生物合成(biosynthesis of amino acids)、丙酮酸代谢(pyruvate metabolism)、柠檬酸循环(即TCA循环, TCA cycle)、丙酸代谢(propanoate metabolism)、丁酸代谢(butanoate metabolism)和脂肪酸代谢(fatty acid metabolism)功能基因相对丰度显著高于未退化草甸(P<0.05), 而退化草甸中磷酸戊糖途径(pentose phosphate pathway)、精氨酸和脯氨酸代谢(arginine and proline metabolism)、有机含硒化合物代谢(selenocompound metabolism)、长寿调节途径-蠕虫(longevity regulating pathway-worm)、酪氨酸代谢(tyrosine metabolism)、肺结核(tuberculosis)、硝基甲苯降解(nitrotoluene degradation)和长寿调节途径-多物种(longevity regulating pathway-multiple species)相关功能基因相对丰度显著低于未退化草甸(P<0.05; 图 2).
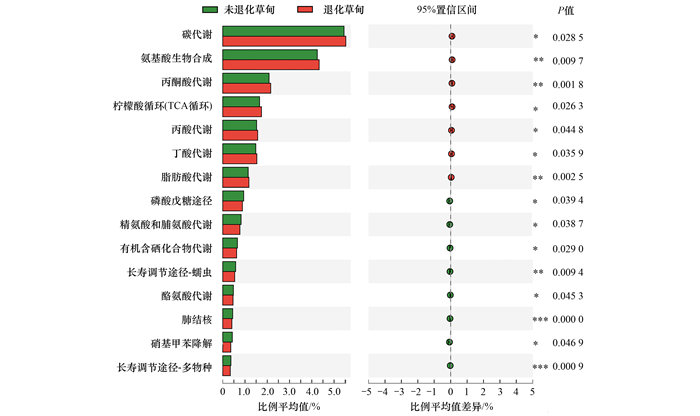
|
***表示P<0.001; **表示P<0.01; *表示P<0.05 图 2 未退化和退化草甸(包括轻度、中度、重度退化)土壤功能基因(KEGG level 3)相对丰度的STAMP分析 Fig. 2 STAMP analysis of the relative abundance of functional gene categories at KEGG level 3 obtained from undegraded meadow soils versus degraded (LD, MD, and HD) meadow soils |
不同退化程度亚高山草甸的土壤微生物宏基因组分类(基于宏基因组物种水平计算)和功能α多样性(基于KEGG level 3计算)如图 3所示.与ND草甸土壤微生物的分类α多样性相比, LD草甸显著升高(P<0.05), MD草甸显著降低(P<0.05), 而HD草甸没有显著变化(P>0.05)[图 3(a)].然而, 与ND草甸土壤微生物的功能α多样性相比, LD草甸没有显著的变化(P>0.05), 而MD和HD草甸均显著降低(P<0.05)[图 3(b)].这些结果表明, 草地退化总体上改变了土壤微生物分类和功能α多样性, 特别是退化程度较大的MD和HD草甸, 微生物功能多样性显著降低(P<0.05).
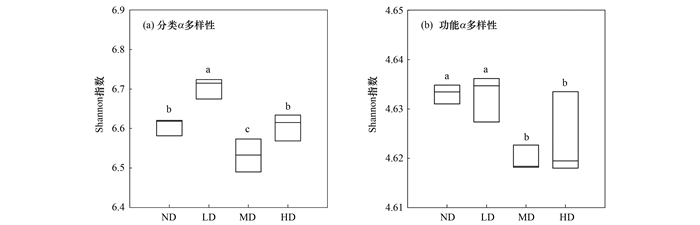
|
不同的字母表示组间具有P<0.05水平上的差异 图 3 不同退化阶段草甸土壤微生物群落的分类和功能α多样性 Fig. 3 Taxonomic and functional alpha diversity of soil microbial communities in different degradation stages of meadow |
NMDS分析结果表明, 不同退化程度草甸间土壤微生物群落的分类和功能存在明显差异(图 4).利用相似性分析(ANOSIM)进一步对基于土壤微生物物种分类和功能组成差异性进行了分析.结果显示4种不同退化程度草甸间土壤微生物物种分类(r=0.892, P<0.001)和功能组成(r=0.972, P<0.001)均有显著性差异(图 4).
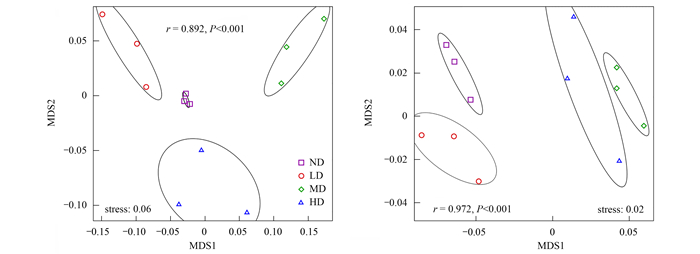
|
(a)微生物群落分类(基于NR属水平)的NMDS, (b)微生物群落功能(基于KEGG level 3)的NMDS; r和P为ANOSIM检验结果; stress值表示整体降维效果, 一般要求该值<0.1 图 4 不同退化阶段草甸土壤微生物群落分类和功能属性的NMDS分析 Fig. 4 NMDS plots of the microbial community compositions in different degradation stages of meadow according to the taxonomic and functional profiles |
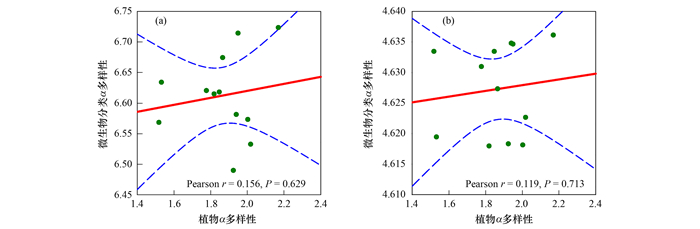
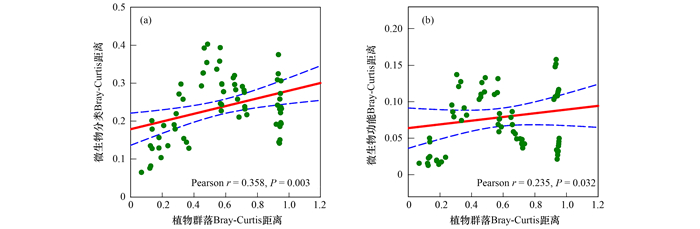
植物与微生物分类学和功能α多样性的Pearson相关关系发现, 微生物群落分类学和功能Shannon指数均与植物的Shannon指数没有显著相关关系(分别为r=0.156, P=0.629和r=0.119, P=0.713; 图 5).然而, 微生物分类学和功能β多样性均与植物β多样性显著正相关(分别为r=0.358, P=0.003和r=0.235, P=0.032; 图 6), 说明在草地退化过程中, 植物群落结构的变化对土壤微生物群落结构和功能具有重要影响.
|
红色实线表示植物和微生物之间α多样性的线性回归; 蓝色虚线间的区域表示拟合的95%置信区间 图 5 植物α多样性与微生物分类(基于NR属水平)和功能(基于KEGG level 3)α多样性之间的相关关系 Fig. 5 Relationships between plant α diversity and microbial taxonomic (based on NR genus level) and functional (based on KEGG level 3) α diversity |
|
红色实线表示植物和微生物群落成对Bray-Curtis距离间的线性回归; 蓝色虚线间的区域表示拟合的95%置信区间 图 6 植物群落与微生物群落分类和功能β多样性之间的关系 Fig. 6 Relationships between plant and microbial taxonomicand functional β diversity |
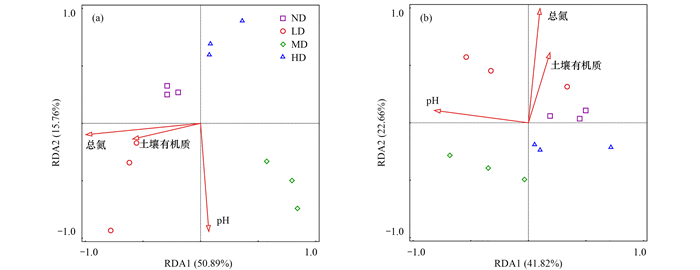
RDA结果表明总氮、pH值和土壤有机质含量对微生物群落分类和功能组成具有显著影响(P<0.05; 图 7), 分别解释了群落变量的66.65%和64.48%.其中, 总氮对微生物群落分类和功能组成均有最大的解释, 分别解释了50.5%和51.2%.
|
(a)微生物群落物种分类组成的RDA分析; (b)微生物群落功能组成的RDA分析 图 7 不同退化程度草甸土壤微生物群落分类和功能组成的冗余分析 Fig. 7 RDA of soil microbial taxonomic and functional composition in meadows with different degradation degrees |
土壤环境因子在联系地上植物群落和地下微生物群落方面具有极为重要的作用.Mantel分析发现土壤理化因子对微生物分类学组成(r=0.370 2, P=0.011)和功能组成(r=0.601, P =0.015)均具有显著的影响.植物群落直接对土壤微生物群落分类(r=0.352 4, P=0.014)和功能(r=0.884 7, P<0.001)组成也产生显著的影响, 且对微生物功能组成的影响均大于微生物分类组成的影响(表 1).用偏Mantel分析发现土壤理化因子对土壤微生物群落分类组成(r=0.333 9, P=0.005)的影响大于功能组成(r=0.190 5, P=0.045), 而植物群落对土壤微生物群落分类学组成的影响小于功能组成(分别为r=0.233 9, P=0.014和r=0.424 5, P=0.014)(表 1).总之, 土壤理化因子和植物群落共同影响着微生物群落的种类和功能组成, 其中土壤因子对微生物群落的分类组成影响较大, 而植物群落对微生物群落功能影响较大(表 1).
|
|
表 1 植物群落和土壤理化对微生物群落的偏Mantel和Mantel分析1) Table 1 Influence of plant communities and soil chemical properties on microbial communities determined by partial Mantel and Mantel test |
3 讨论 3.1 土壤微生物群落的分类和功能多样性对亚高山草甸退化的响应
不同退化阶段亚高山草甸土壤微生物群落的分类和功能α多样性均存在明显差异.土壤微生物分类和功能α多样性对草地退化的响应模式可能不一样, ND和HD草甸中分类α多样性没有显著差异, 但在HD草甸的功能α多样性显著低于ND草甸.这与Zhang等[16]在内蒙古草原开展的为期5 a的植物功能群移除实验结果一致, 植物多样性与生产力降低导致土壤微生物功能多样性降低, 但是对微生物群落分类多样性影响较小.LD草甸土壤微生物的分类α多样性显著高于ND草甸, 这可能是由于LD草甸中土壤微环境的异质性更高, 使土壤微生物多样性也更高.随着草甸退化程度加剧后, 许多原生植物逐渐消失, 植物群落结构趋于单一, 植物多样性下降, 输入到土壤的枯落物和根系分泌物的多样性也会相应降低[17], 从而导致在MD和HD草甸的土壤微生物功能多样性显著低于ND草甸.除了植物多样性, 植物生产力下降也可能是退化草地土壤微生物功能多样性降低的主要原因之一[18].生产力下降限制了提供微生物的新鲜植物资源, 强化了生态过滤, 偏好负责能源生产/转化的代谢基因, 从而导致许多具有其他功能的基因丧失[16].虽然LD草甸具有较高的分类多样性, 但功能多样性与ND草甸差异不大, 说明LD草甸具有较高的功能冗余性.微生物多样性的增加可以为特定生态功能提供充分的特性冗余保障, 以应对草地退化过程中的显著变化(包括植物多样性和覆盖度)[19].
3.2 土壤微生物功能类群和潜在功能途径对亚高山草甸退化的响应本研究结果表明退化草甸中参与碳代谢、氨基酸生物合成、丙酮酸代谢、柠檬酸循环、丙酸代谢、丁酸代谢和脂肪酸代谢功能基因相对丰度显著高于未退化草甸, 而退化草甸中磷酸戊糖途径、精氨酸和脯氨酸代谢、有机含硒化合物代谢和酪氨酸代谢相关功能基因相对丰度显著低于未退化草甸.其中碳代谢、丙酮酸代谢、柠檬酸循环、丙酸代谢、丁酸代谢和磷酸戊糖途径属于KEGG level 2中碳水化合物代谢; 氨基酸生物合成、精氨酸和脯氨酸代谢和酪氨酸代谢均属于KEGG level 2中的氨基酸代谢.说明在退化草甸中, 碳水化合物代谢、脂质代谢和氨基酸代谢发生了明显的变化[20].这可能是由于过度放牧导致的草地退化, 地上植被大部分被家畜采食, 只有少量的粪便和凋落物归还土壤[21].土壤得不到有机质的输入, 便会加速分解其腐殖质层中的有机质, 碳水化合物、氨基酸代谢和核苷酸代谢, 导致退化草地中相应的微生物代谢功能基因丰度增加, 以维持植物生长所需的养分有效性[5].这也体现了土壤微生物对逆境胁迫和干扰环境的适应性[20, 22]. 有研究证实随着草地退化程度的增加, 土壤微生物对活性碳库进行同化后, 可能会加速代谢顽固性碳库来适应土壤有机质输入不足[23].Che等[7]发现青藏高原高寒草甸退化斑块形成过程中, 氨氧化微生物丰度明显增加, 是退化过程中土壤氮流失的重要驱动因素.草地退化过程中碳氮代谢功能的改变可能会导致土壤中分解有机碳、氮的微生物及酶活性增加, 从而促进土壤温室气体(CO2和N2O)排放通量增加[23].
3.3 植物群落β多样性与微生物群落分类和功能β多样性的显著相关尽管植物群落α多样性与土壤微生物群落分类和功能α多样性没有显著的相关关系, 但是植物群落β多样性可以显著预测土壤微生物群落分类和功能的β多样性(图 6).说明在草地退化过程中, 植物群落结构变化对土壤微生物群落结构和功能产生直接或间接的影响.目前植物和土壤微生物之间β多样性的关系并不明确.有研究表明, 植被类型和土壤微生物模式在全球[24]、区域[25]和局域[26]尺度上存在显著相关性, 而其他研究则发现植物群落和土壤微生物群落受到不同环境驱动因素的影响[27].本研究中植物群落可能通过多种途径直接或间接影响微生物的群落结构和功能[26, 28].首先, 由于草地退化而导致植物地上和地下生物量的减少, 降低了输入到土壤中的凋落物和根的碳含量, 从而影响了微生物利用底物的有效性.不同的植被产生不同数量和质量的植物凋落物, 主要导致土壤碳氮的变化, 最终导致微生物群落组成和功能的变化[29].其次, 植物组成的显著改变, 如退化过程中莎草科植物的减少和非禾本植物的增加, 也可能对微生物群落产生显著影响.植物物种对其相关微生物的选择性效应已被广泛报道[30].
3.4 土壤特性决定了亚高山草甸退化过程中土壤微生物群落结构的变化不同退化程度草甸土壤微生物群落和组成差异显著, 说明微生物群落分类和功能的结构均对亚高山草甸退化敏感.土壤特性是塑造土壤微生物群落的最重要因素之一.有研究表明[4~6, 31], 土壤物理化学变量在草地退化过程中对微生物群落的形成起着至关重要的作用.李海云等[32]的研究表明, 土壤速效钾、全氮、速效氮和有机碳是祁连山不同退化高寒草地土壤真菌群落结构的主要驱动因子.Li等[6]对青藏高原退化高寒草甸的研究发现, 土壤总碳、总有机碳、总氮、总磷和总钾等土壤营养条件显著影响细菌和真菌群落结构.在本研究中, 总氮、pH值和土壤有机质含量对微生物群落分类和功能组成均具有显著影响(P<0.05), 这表明草地退化过程中微生物多样性组成和结构的差异很可能是土壤特性造成的.与本研究结果相似, 先前的研究也证明[14, 19], 上述因素影响了土壤微生物群落.目前, 许多研究集中在氮梯度对土壤微生物群落的影响[33, 34], 因为全球的生态系统都受到高水平氮的影响.例如, Ramirez等[35]发现短期施用氮肥后, 草地土壤微生物群落规模将会扩大.相反, 长期施用氮肥会导致草地土壤微生物群落组成发生改变, 增加氮肥用量可能对土壤中碳循环产生潜在的负面影响, 同时促进具有已知致病性状的真菌属的形成[36].在草地生态系统中, 土壤pH的变化会直接影响草地土壤微生物酶的活性, 从而导致草地土壤微生物代谢功能的改变[37].土壤有机质的含量和质量对微生物的功能过程具有重要的调节作用[38].许多土地利用方式改变的研究均发现有机质在形成微生物群落中起关键作用, 包括森林砍伐[39]、放牧管理[40]和植被恢复[19].综上所述, 亚高山草甸退化显著改变了土壤微生物分类和功能多样性, 土壤特性在塑造微生物群落多样性方面发挥着重要作用.
4 结论(1) 亚高山草甸过程中土壤微生物群落物种组成和丰度存在明显的变化.退化草甸中参与碳代谢、氨基酸生物合成、丙酮酸代谢、柠檬酸循环、丙酸代谢、丁酸代谢和脂肪酸代谢功能基因相对丰度显著高于未退化草地.草地退化改变了亚高山草甸土壤微生物群落能量代谢和营养循环的代谢潜力.
(2) 草地退化总体上改变了土壤微生物分类和功能α多样性, 特别是退化程度较大的MD和HD草甸的微生物功能多样性显著降低.地上植物群落与地下土壤真菌群落之间的α多样性没有显著相关关系, 而微生物分类和功能β多样性均与植物β多样性显著正相关.
(3) 不同退化阶段亚高山草甸土壤微生物群落的分类和功能结构存在显著差异.总氮、pH值和土壤有机质含量对微生物群落分类和功能组成具有显著影响.土壤理化因子和植物群落共同影响着微生物群落的种类和功能组成, 其中土壤因子对微生物群落的分类组成影响较大, 而植物群落对微生物群落功能影响较大.
致谢: 感谢山西省五台山国有林管理局在采样过程中的协助与大力支持.
| [1] | Fierer N. Embracing the unknown: disentangling the complexities of the soil microbiome[J]. Nature Reviews Microbiology, 2017, 15(10): 579-590. DOI:10.1038/nrmicro.2017.87 |
| [2] | Coban O, De Deyn G B, van der Ploeg M. Soil microbiota as game-changers in restoration of degraded lands[J]. Science, 2022, 375(6584). DOI:10.1126/science.abe0725 |
| [3] |
罗正明, 赫磊, 刘晋仙, 等. 土壤真菌群落对五台山亚高山草甸退化的响应[J]. 环境科学, 2022, 43(6): 3328-3337. Luo Z M, He L, Liu J X, et al. Responses of soil fungal communities to subalpine meadow degradation in Mount Wutai[J]. Environmental Science, 2022, 43(6): 3328-3337. DOI:10.13227/j.hjkx.202109174 |
| [4] | Che R X, Wang Y F, Li K X, et al. Degraded patch formation significantly changed microbial community composition in alpine meadow soils[J]. Soil and Tillage Research, 2019, 195. DOI:10.1016/j.still.2019.104426 |
| [5] | Yu Y, Zheng L, Zhou Y J, et al. Changes in soil microbial community structure and function following degradation in a temperate grassland[J]. Journal of Plant Ecology, 2021, 14(3): 384-397. DOI:10.1093/jpe/rtaa102 |
| [6] | Li Y M, Wang S P, Jiang L L, et al. Changes of soil microbial community under different degraded gradients of alpine meadow[J]. Agriculture, Ecosystems & Environment, 2016, 222: 213-222. |
| [7] | Che R X, Wang F, Wang W J, et al. Increase in ammonia-oxidizing microbe abundance during degradation of alpine meadows may lead to greater soil nitrogen loss[J]. Biogeochemistry, 2017, 136(3): 341-352. DOI:10.1007/s10533-017-0399-5 |
| [8] | Cai X B, Peng Y L, Yang M N, et al. Grassland degradation decrease the diversity of arbuscular mycorrhizal fungi species in Tibet Plateau[J]. Notulae Botanicae, Horti Agrobotanici, Cluj-Napoca, 2014, 42(2): 333-339. |
| [9] | Fierer N, Leff J W, Adams B J, et al. Cross-biome metagenomic analyses of soil microbial communities and their functional attributes[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(52): 21390-21395. DOI:10.1073/pnas.1215210110 |
| [10] | Burke C, Steinberg P, Rusch D, et al. Bacterial community assembly based on functional genes rather than species[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(34): 14288-14293. DOI:10.1073/pnas.1101591108 |
| [11] | Knight R, Vrbanac A, Taylor B C, et al. Best practices for analysing microbiomes[J]. Nature Reviews Microbiology, 2018, 16(7): 410-422. DOI:10.1038/s41579-018-0029-9 |
| [12] |
章异平, 江源, 刘全儒, 等. 放牧压力下五台山高山、亚高山草甸的退化特征[J]. 资源科学, 2008, 30(10): 1555-1563. Zhang Y P, Jiang Y, Liu Q R, et al. Degradation features of alpine and subaipine meadows under grazing pressure in Mt. Wutai[J]. Resources Science, 2008, 30(10): 1555-1563. DOI:10.3321/j.issn:1007-7588.2008.10.017 |
| [13] |
江源, 章异平, 杨艳刚, 等. 放牧对五台山高山、亚高山草甸植被-土壤系统耦合的影响[J]. 生态学报, 2010, 30(4): 837-846. Jiang Y, Zhang Y P, Yang Y G, et al. Impacts of grazing on the system coupling between vegetation and soil in the alpine and subalpine meadows of Wutai Mountain[J]. Acta Ecologica Sinica, 2010, 30(4): 837-846. |
| [14] | Zhang L, Wang X T, Wang J, et al. Alpine meadow degradation depresses soil nitrogen fixation by regulating plant functional groups and diazotrophic community composition[J]. Plant and Soil, 2022, 473(1-2): 319-335. DOI:10.1007/s11104-021-05287-z |
| [15] | Wang C T, Long R J, Wang Q L, et al. Changes in plant diversity, biomass and soil C, in alpine meadows at different degradation stages in the headwater region of three rivers, China[J]. Land Degradation & Development, 2009, 20(2): 187-198. |
| [16] | Zhang X M, Johnston E R, Barberán A, et al. Decreased plant productivity resulting from plant group removal experiment constrains soil microbial functional diversity[J]. Global Change Biology, 2017, 23(10): 4318-4332. DOI:10.1111/gcb.13783 |
| [17] | Paredes S H, Lebeis S L. Giving back to the community: microbial mechanisms of plant-soil interactions[J]. Functional Ecology, 2016, 30(7): 1043-1052. DOI:10.1111/1365-2435.12684 |
| [18] | Millard P, Singh B K. Does grassland vegetation drive soil microbial diversity?[J]. Nutrient Cycling in Agroecosystems, 2010, 88(2): 147-158. DOI:10.1007/s10705-009-9314-3 |
| [19] | Guo Y Q, Chen X T, Wu Y Y, et al. Natural revegetation of a semiarid habitat alters taxonomic and functional diversity of soil microbial communities[J]. Science of the Total Environment, 2018, 635: 598-606. DOI:10.1016/j.scitotenv.2018.04.171 |
| [20] | Lai C M, Li C Y, Peng F, et al. Plant community change mediated heterotrophic respiration increase explains soil organic carbon loss before moderate degradation of alpine meadow[J]. Land Degradation & Development, 2021, 32(18): 5322-5333. |
| [21] | Zhou G Y, Zhou X H, He Y H, et al. Grazing intensity significantly affects belowground carbon and nitrogen cycling in grassland ecosystems: a meta-analysis[J]. Global Change Biology, 2017, 23(3): 1167-1179. DOI:10.1111/gcb.13431 |
| [22] | Chen J, Elsgaard L, van Groenigen K J, et al. Soil carbon loss with warming: new evidence from carbon-degrading enzymes[J]. Global Change Biology, 2020, 26(4): 1944-1952. DOI:10.1111/gcb.14986 |
| [23] | Breidenbach A, Schleuss P M, Liu S B, et al. Microbial functional changes mark irreversible course of Tibetan grassland degradation[J]. Nature Communications, 2022, 13(1). DOI:10.1038/s41467-022-30047-7 |
| [24] | Prober S M, Leff J W, Bates S T, et al. Plant diversity predicts beta but not alpha diversity of soil microbes across grasslands worldwide[J]. Ecology Letters, 2015, 18(1): 85-95. DOI:10.1111/ele.12381 |
| [25] | Griffiths R I, Thomson B C, James P, et al. The bacterial biogeography of British soils[J]. Environmental Microbiology, 2011, 13(6): 1642-1654. DOI:10.1111/j.1462-2920.2011.02480.x |
| [26] | Li H, Xu Z W, Yan Q Y, et al. Soil microbial beta-diversity is linked with compositional variation in aboveground plant biomass in a semi-arid grassland[J]. Plant and Soil, 2018, 423(1-2): 465-480. DOI:10.1007/s11104-017-3524-2 |
| [27] | Soininen J. Macroecology of unicellular organisms-patterns and processes[J]. Environmental Microbiology Reports, 2012, 4(1): 10-22. DOI:10.1111/j.1758-2229.2011.00308.x |
| [28] | Luo Z M, Liu J X, Jia T, et al. Soil bacterial community response and nitrogen cycling variations associated with subalpine meadow degradation on the Loess Plateau, China[J]. Applied and Environmental Microbiology, 2020, 86(9). DOI:10.1128/AEM.00180-20 |
| [29] | Sariyildiz T, Anderson J M. Interactions between litter quality, decomposition and soil fertility: a laboratory study[J]. Soil Biology and Biochemistry, 2003, 35(3): 391-399. DOI:10.1016/S0038-0717(02)00290-0 |
| [30] | Micallef S A, Shiaris M P, Colón-Carmona A. Influence of Arabidopsis thaliana accessions on rhizobacterial communities and natural variation in root exudates[J]. Journal of Experimental Botany, 2009, 60(6): 1729-1742. DOI:10.1093/jxb/erp053 |
| [31] | Yang Y F, Wu L W, Lin Q Y, et al. Responses of the functional structure of soil microbial community to livestock grazing in the Tibetan alpine grassland[J]. Global Change Biology, 2013, 19(2): 637-648. DOI:10.1111/gcb.12065 |
| [32] |
李海云, 姚拓, 高亚敏, 等. 退化高寒草地土壤真菌群落与土壤环境因子间相互关系[J]. 微生物学报, 2019, 59(4): 678-688. Li H Y, Yao T, Gao Y M, et al. Relationship between soil fungal community and soil environmental factors in degraded alpine grassland[J]. Acta Microbiologica Sinica, 2019, 59(4): 678-688. DOI:10.13343/j.cnki.wsxb.20180257 |
| [33] | De Deyn G B, Van Der Putten W H. Linking aboveground and belowground diversity[J]. Trends in Ecology & Evolution, 2005, 20(11): 625-633. |
| [34] | Leff J W, Jones S E, Prober S M, et al. Consistent responses of soil microbial communities to elevated nutrient inputs in grasslands across the globe[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(35): 10967-10972. DOI:10.1073/pnas.1508382112 |
| [35] | Ramirez K S, Craine J M, Fierer N. Consistent effects of nitrogen amendments on soil microbial communities and processes across biomes[J]. Global Change Biology, 2012, 18(6): 1918-1927. DOI:10.1111/j.1365-2486.2012.02639.x |
| [36] | Paungfoo-Lonhienne C, Yeoh Y K, Kasinadhuni N R P, et al. Nitrogen fertilizer dose alters fungal communities in sugarcane soil and rhizosphere[J]. Scientific Reports, 2015, 5. DOI:10.1038/srep08678 |
| [37] | Zhang Y, Cao C, Guo L, et al. Soil properties, bacterial community composition, and metabolic diversity responses to soil salinization of a semiarid grassland in northeast China[J]. Journal of Soil and Water Conservation, 2015, 70(2): 110-120. DOI:10.2489/jswc.70.2.110 |
| [38] | Ding J J, Zhang Y G, Wang M M, et al. Soil organic matter quantity and quality shape microbial community compositions of subtropical broadleaved forests[J]. Molecular Ecology, 2015, 24(20): 5175-5185. DOI:10.1111/mec.13384 |
| [39] | Navarrete A A, Tsai S M, Mendes L W, et al. Soil microbiome responses to the short-term effects of Amazonian deforestation[J]. Molecular Ecology, 2015, 24(10): 2433-2448. DOI:10.1111/mec.13172 |
| [40] | Xun W B, Yan R R, Ren Y, et al. Grazing-induced microbiome alterations drive soil organic carbon turnover and productivity in meadow steppe[J]. Microbiome, 2018, 6(1). DOI:10.1186/s40168-018-0544-y |
 2023, Vol. 44
2023, Vol. 44


