2. 北京师范大学自然科学高等研究院, 珠海 519087;
3. 华东师范大学地理信息科学教育部重点实验室, 上海 200241
2. Advanced Institute of Natural Sciences, Beijing Normal University, Zhuhai 519087, China;
3. Key Laboratory of Geographic Information Science, Ministry of Education, East China Normal University, Shanghai 200241, China
青藏高原是我国重要的生态安全屏障, 拥有大量重要的受人类活动干扰较小的自然生态系统和荒野, 孕育着丰富的生物多样性[1, 2].但近年来, 受气候变化和人类活动等影响, 高原生态系统面临退化风险[3~5].微生物作为河流生态系统的重要组成部分[6], 在河流生态系统的物质循环及营养分配等过程中承担了主要作用[7], 是反映流域健康的重要指标.同时, 微生物对河流生态环境的变化非常敏感[8], 而河流微生物更能反映流域的总体变化, 其多样性和群落特征对环境变化有较好的指示意义[9].因此, 全面系统地了解青藏高原河流生态系统的微生物群落组成及多样性具有较高的科学研究价值[10].
目前, 对青藏高原河流微生物群落的研究大多针对单一流域尺度, 如Zhang等[8]采用高通量测序技术对青藏高原黑河流域上游水体浮游细菌群落进行了调查; 程豹等[11]探究了澜沧江流域的浮游细菌群落结构特征.然而仍缺乏从青藏高原多个流域层面开展大尺度的微生物群落结构差异研究.
16S rRNA基因广泛存在于细菌中, 其基因序列由9个可变区和10个保守区交替组成.保守区有利于扩增引物的设计, 可变区由于体现了物种间的进化差异可以用于物种的分类鉴定[12, 13].相比传统的微生物多样性研究方法, 基于16S rRNA基因的高通量测序无需培养微生物, 已经成为当前微生物多样性分析最常用方法[14].
本研究基于16S rRNA测序技术, 对青藏高原河流的细菌群落组成进行分析, 范围上跨越了黄河、长江、澜沧江、怒江、雅鲁藏布江和柴达木盆地这6大区域.目的在于探究:①青藏高原河流细菌群落多样性的组成和分布特点; ②不同流域的细菌群落多样性特征; ③流域间细菌群落差异; ④细菌群落与环境因子的关系, 以期为推动青藏高原环境变化预测和提高生态环境治理提供参考.
1 材料与方法 1.1 采样点位及样本采集采样范围跨越了黄河、柴达木盆地、长江、澜沧江、怒江和雅鲁藏布江这6大区域, 包括各流域干流和支流在内共65个采样点(图 1). 于2021年7月, 在每个采样点位采集3 L表层水样, 放入保温箱内冷藏保存, 24 h内使用0.22 μm混合纤维滤膜对水样进行抽滤(每个点位3个平行样, 每个平行样1 L水), 得到的滤膜放置在1.5 mL离心管内, 将离心管放置在干冰中低温保存, 等待后续DNA的提取.
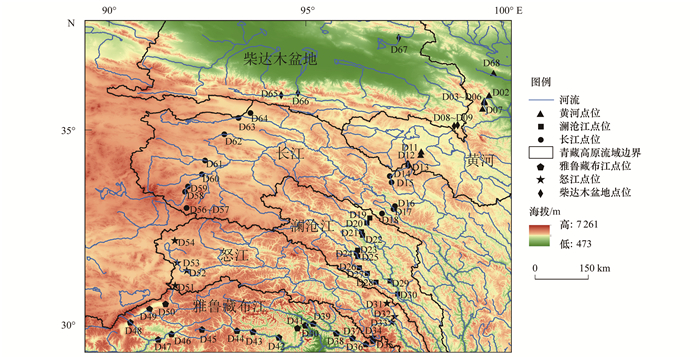
|
图 1 青藏高原河流采样点示意 Fig. 1 River sampling sites on the Qinghai-Tibet Plateau |
(1) 流域边界数据 流域边界数据集来源于国家青藏高原科学数据中心[15, 16], 65个采样点分布于黄河、柴达木盆地、长江、澜沧江、怒江和雅鲁藏布江这6个流域.
(2) DEM数据 数字高程模型(digital elevation model, DEM)数据来源于中国科学院计算机网络信息中心地理空间数据云平台(http://www.gscloud.cn), 空间分辨率为90 m的SRTMDEM原始高程数据.本文利用DEM数据绘制采样点, 并提取各采样点以上流域的平均高程、平均坡度和平均海拔等信息.
(3) NDVI数据 归一化植被指数(normalized differential vegetation index, NDVI)数据来源于NASA官网(https://ladsweb.modaps.eosdis.nasa.gov/)的MODIS NDVI的16 d合成产品, 产品获取时间为2021年7月28日, 与样品采集时间相近.
(4) 地表覆盖类型数据 地表覆盖类型数据来源于30 m全球地表覆盖数据GlobeLand 30[17]的2020版(http://www.globallandcover.com/), 共包括10个一级类型, 分别是:耕地、林地、草地、灌木地、湿地、水体、苔原、人造地表、裸地、冰川和永久积雪.经过数据处理, 发现本文所有采样点以上流域均没有苔原分布.
1.3 水环境指标的测定将采集了水样的水桶保存在放置有冰袋的保温箱内并运送至实验室, 各指标的分析测试方法参考文献[18].电导率(electrical conductivity, Cond)和pH用水质检测仪YSI-Pro进行测定, 总磷(total phosphorus, TP)使用钼酸铵分光光度法, 溶解态活性磷(soluble reactive phosphorus, SRP)用锑钼酸抗坏血酸盐法, 总氮(total nitrogen, TN)使用碱性过硫酸钾消解紫外分光光度法, NO3--N使用酚二磺酸分光光度法, NH4+-N使用纳氏试剂分光光度法, 溶解性硅(dissolved silicon, DSi)使用硅钼蓝自动比色法, 溶解性有机碳(dissolved organic carbon, DOC)用总有机碳分析仪进行分析.SRP/TP的比值可用来表征泥沙或水体悬浮颗粒物的含量[6], 也将用于之后的环境因子分析.
1.4 DNA提取及PCR扩增参照E.Z.N.A.® Soil DNA Kit (Omega Bio-tek, Norcross, GA, U.S.) 试剂盒说明书, 提取样本中的DNA.细菌群落结构研究采用16S rRNA V4-V5区引物515F (上游引物)5′- GTGCCAGCMGCC GCGG-3′和907R(下游引物)5′-CCGTCAATTCM TTTRAGTTT-3′进行PCR扩增, 每个样本的扩增引物含有8碱基标签序列用以区分样本.准备20 μL的PCR反应体系, 如下:4 μL的5xFastPfu Buffer, 2μL的2.5 mm dNTPs, 上下游引物各0.8 μL (5 μm), 0.4 μL的FastPfu Polymerase和10 ng模板DNA.PCR扩增实验程序如下:首先将获得的DNA放在95℃下进行5 min预变性, 为DNA充分解旋做准备; 之后再在95℃下进行30 s, 在Tm℃下进行30 s, 使DNA充分解旋; 最后在72℃下进行45 s, 使DNA聚合酶催化子链延伸.上述3个步骤进行25个循环, 最后72℃保持10 min, 使DNA聚合酶充分发挥作用, 完成DNA的充分复制.扩增产物经2%琼脂糖凝胶电泳后采用AxyPrep DNA Gel Extraction Kit (Axygen Biosciences, Union City, CA, U.S.) 试剂盒参照说明书操作流程进行纯化.
1.5 文库构建及测序利用Qubit® 3.0 (Life Invitrogen)对纯化后的PCR产物进行精准定量, 然后将带有不同标签序列的24个扩增子样本进行等量混合.混池后的DNA产物参照Illumina基因组测序文库构建流程, 构建Illumina双端测序的PE文库.该扩增子文库参照标准流程采用Illumina平台进行PE250模式测序, 建库测序工作由上海凌恩生物科技有限公司完成.
1.6 测序数据前处理测序得到原始序列.根据原始序列首尾两端的条形码和引物区分样品, 并调整序列方向.数据拆分后进行数据除杂, 参数要求如下:①过滤read尾部质量值20以下的碱基, 设置10bp的窗口, 如果窗口内的平均质量值低于20, 从窗口开始截去后端碱基, 过滤质控后50bp以下的read; ②拼接序列的重叠区允许的最大错配比率为0.2, 筛除不符合序列; ③根据PE reads之间的重叠关系, 将成对reads拼接成一条序列, 最小重叠长度为10bp.
使用UCHIME软件鉴定并移除嵌合序列, 采用UPARSE(version 7.1 http://drive5.com/uparse/) 软件将相似性达97%的序列聚类为OTU.采用RDP classifier (http://rdp.cme.msu.edu/) 贝叶斯算法对OTU代表序列进行分类学分析, 比对Silva Release138.1(http://www.arb-silva.de)数据库, 置信度阈值为0.7, 最终获得每个OTU在各个分类学水平上的物种信息, 并由此统计各个分类水平上各样本的群落构成.
1.7 统计分析在R语言中进行统计分析和绘图.使用ggplot2包进行可视化绘图[19].使用vegan包的rarecurve函数绘制稀释性曲线.使用stats包的pairwise.wilcox.test函数分别判定流域间环境因子、多样性指数和微生物组成差异(P<0.05表明具有显著性差异).使用psych包分别进行多样性指数和环境因子, 细菌类群和环境因子的Spearman相关性分析, P值运用FDR方法进行矫正.使用vegan包做主坐标分析(principal coordinates analysis, PCoA).使用vegan包进行典范对应分析(canonical correspondence analysis, CCA), 确定与群落结构差异呈显著性相关(P<0.05)的环境因子.
2 结果与分析 2.1 青藏高原河流水环境指标、流域特征与地表覆盖特征本文选取了pH值、电导率、TP、TN、SRP、NO3--N、NH4+-N、SRP/TP值、DOC和DSi作为水环境指标变量; 平均海拔、流域面积、平均NDVI、平均坡度和河网长度作为流域特征变量; 耕地、林地、草地、灌木地、湿地、人造地表、裸地、冰川和永久积雪各地表覆盖类型占总流域面积的百分比作为地表覆盖特征变量, 并对其进行流域间的差异性分析(表 1).柴达木盆地为干旱区, 其pH值最高, 为7.70.雅鲁藏布江的电导率显著低于其他流域, 为157 μS·cm-1.总体而言, 青藏高原河流的TP质量浓度处于Ⅱ类水下; TN质量浓度处于Ⅲ类水下.长江流域的ρ(NH4+-N)最高, 为0.01 mg·L-1.澜沧江和雅鲁藏布江的DOC显著低于其他流域.黄河和雅鲁藏布江的SRP/TP值显著高于澜沧江.所有采样点流域的平均海拔范围为3 596~5 708 m, 怒江的平均海拔最高, 为5 000 m.澜沧江的平均NDVI显著高于其他流域, 为0.61.雅鲁藏布江的平均坡度显著高于其他流域, 为23.43°.在所有地表覆盖类型中, 草地面积占比最高, 平均占比63%; 其次为裸地, 平均占比15%.
|
|
表 1 青藏高原不同流域环境因子1) Table 1 Environmental variables in watersheds on the Qinghai-Tibet Plateau |
2.2 物种注释及群落多样性指数
经过16s rRNA测序及97%相似性聚类后, 共得到24 705个OTU.各样点稀释性曲线趋于平坦(图 2), 说明测序数据量合理.测序深度指数(Coverage指数)均值为95.65%, 能够反映样本微生物群落的真实情况.对OTU进行物种注释后, 共得到分类地位及物种信息确定的有65门、149纲、383目、540科、1 311属和383种.
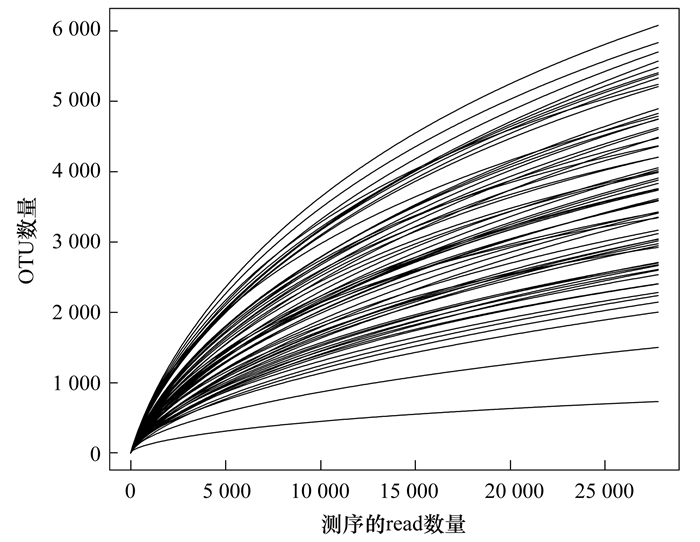
|
图 2 青藏高原河流细菌稀释性曲线 Fig. 2 Rarefaction curve of bacteria in river ecosystems on the QTP |
对每个样点进行α多样性分析, 得到一系列估计群落物种丰富度和多样性的指数, 并按流域进行分组展示(图 3).Chao指数和OTU richness指数用于反映物种丰富度, Chao指数或OTU richness指数值越大, 说明样品中群落丰富度越大.Shannon指数和Simpson指数用于反映物种的多样性, Shannon指数越高, 说明物种多样性越高; Simpson指数越高, 说明物种多样性越低.
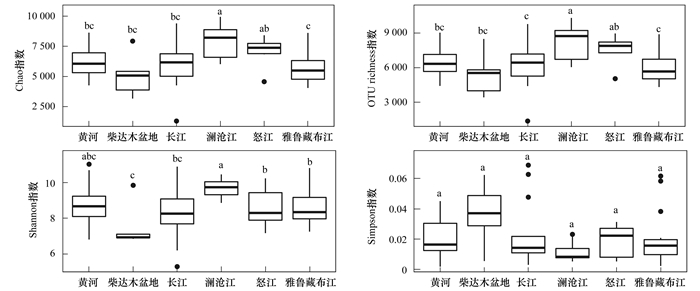
|
相同小写字母表示组间差异不显著,不同小写字母表示组间差异显著;箱体内横线代表各流域多样性指数的中位数 图 3 不同流域细菌群落的多样性指数及差异 Fig. 3 The α diversity indexes of bacterial communities in different watersheds |
结果显示, 青藏高原所有采样点的Chao、OTU richness、Shannon和Simpson指数(平均值±标准差)分别为6 402±1 714、6 722±1 795、8.7±1.3和0.02±0.018, 表明青藏高原河流细菌的丰富度和多样性均处于较高水平.其中, 澜沧江的细菌丰富度和多样性最高, 柴达木盆地的细菌丰富度和多样性最低.差异性分析结果显示:在丰富度上, 澜沧江与黄河、柴达木盆地、长江和雅鲁藏布江存在显著性差异; 在多样性上, Shannon指数显示澜沧江与柴达木盆地、长江、怒江和雅鲁藏布江之间有显著性差异.
将多样性指数与环境因子进行Spearman相关性分析(图 4).Chao指数和OTU richness指数均与SRP/TP的值有极显著负相关关系.
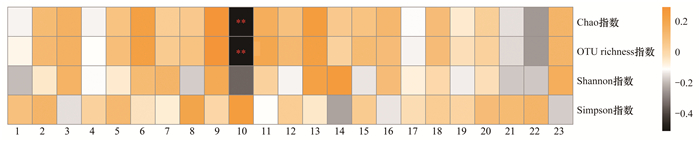
|
1.pH, 2.电导率, 3.TP, 4.SRP, 5.TN, 6.NO3--N, 7.NH4+-N, 8.DOC, 9.DSi, 10.SRP/TP, 11.平均海拔, 12.流域面积, 13.平均NDVI, 14.平均坡度, 15.河网长度, 16.耕地, 17.林地, 18.草地, 19.灌木地, 20.湿地, 21.人造地表, 22.裸地, 23.冰川和永久积雪; **表示显著性水平P < 0.01 图 4 多样性指数与环境因子的Spearman相关性分析 Fig. 4 Spearman correlations between α diversity indexes and environmental variables |
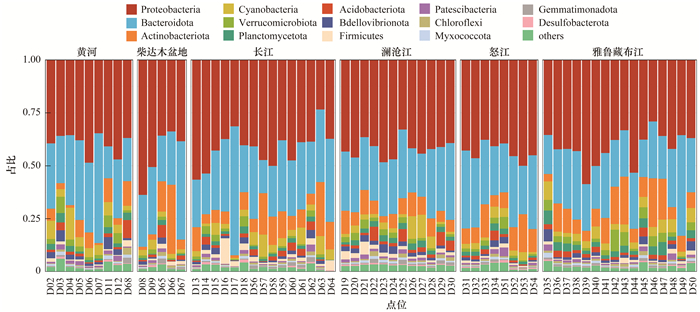
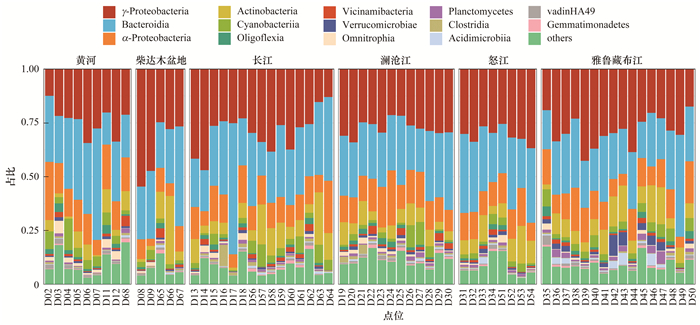
总体而言, 青藏高原河流微生物群落在门水平上以变形菌门(Proteobacteria)和拟杆菌门(Bacteroidota)为主(图 5), 相对丰度分别为41.72%和28.46%.另外两个相对丰度较高的门为放线菌门(Actinobacteriota, 9.38%)和蓝细菌门(Cyanobacteria, 4.12%).在纲水平上(图 6), 总体而言, 最常见的类群为γ-变形菌纲(28.70%)、拟杆菌纲(28.15%)和α-变形菌纲(13.02%).其在不同流域所占相对丰度范围分别为24.14%~36.24%、25.23%~30%和8.24%~14.47%.在属水平上, 总体而言, Flavobacterium为各流域中占比最高的优势类群, 相对丰度范围为6.12%~15.78%. Limnohabitans为柴达木盆地、长江、澜沧江和怒江流域的第二优势类群, 相对丰度达到2%~10.16%. hgcI clade是雅鲁藏布江流域的第二优势类群. Vibrionimonas是黄河流域的第二优势类群.对属水平总体相对丰度前5的类群进行不同流域之间的差异性分析(表 2), 发现Flavobacterium在雅鲁藏布江和黄河之间、雅鲁藏布江和澜沧江之间、雅鲁藏布江和怒江之间呈现出显著性差异; Limnohabitans在较多流域之间均呈现显著性差异; 黄河流域hgcI clade的相对丰度与其他流域均呈显著性差异.
|
图 5 青藏高原河流微生物在门水平上的组成 Fig. 5 Bacterial community composition at phylum level in river ecosystems on the Qinghai-Tibet Plateau |
|
图 6 青藏高原河流微生物在纲水平上的组成 Fig. 6 Bacterial community composition at class level in river ecosystems on the Qinghai-Tibet Plateau |
|
|
表 2 青藏高原不同流域主要属水平上微生物相对丰度平均值差异1)/% Table 2 Differences in average relative abundance of major genera between watersheds on the Qinghai-Tibet Plateau/% |
2.4 青藏高原各流域间微生物群落结构的差异
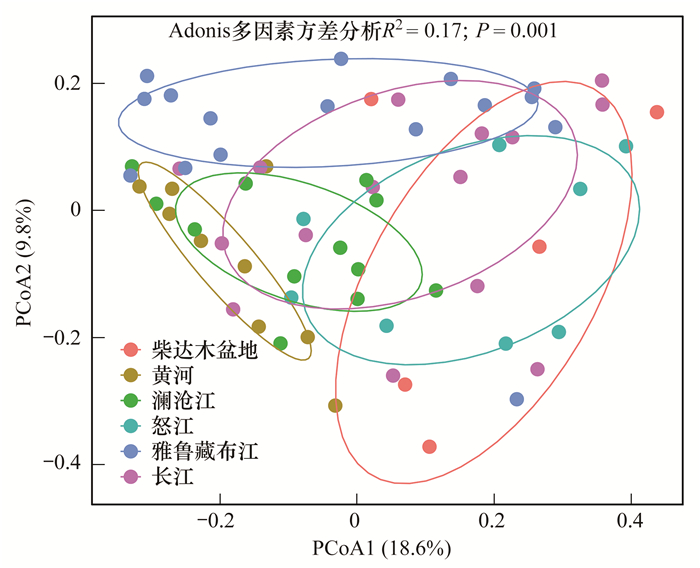
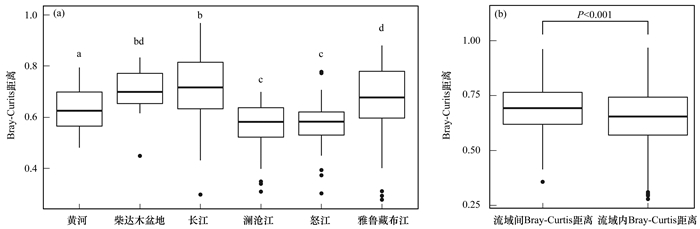
为了比较青藏高原各流域间微生物群落结构的相似性或差异性, 本文利用PCoA分析对数据进行降维并可视化(图 7).两个主坐标共解释28.4%的差异.各组互相交叉重合部分较多, 在图上并没有显著分开.利用Adonis多因素方差分析对所有流域在统计学意义上进行显著性分析(R2=0.17, P=0.001), 可知微生物群落结构在各流域间整体上有极显著差异.用Bray-Curtis距离表征各流域内细菌群落的差异[图 8(a)], 结果显示各流域内群落的异质性较强.这可能是因为部分流域在相隔较远的上下游位置采样, 流域异质性高, 导致同流域内的点位微生物组成有较大差异.但总体上各流域细菌群落的组间差异仍然大于组内差异, 且差异极显著[图 8(b)].
|
图 7 各流域微生物群落的主坐标分析 Fig. 7 Principal coordinate analysis of microbial communities in different watersheds |
|
相同小写字母表示组间差异不显著,不同小写字母表示组间差异显著;箱体内横线代表Bray-Curtis距离的中位数 图 8 流域微生物群落结构差异比较 Fig. 8 Differences in composition characteristics of microbial communities |
为探究微生物群落结构与环境因子的关系, 本文将水环境指标变量、流域特征变量和地表覆盖特征变量作为环境变量, 把OTU水平上的群落组成作为物种变量, 进行CCA分析(图 9).采用envfit函数确定与微生物群落结构呈显著性相关的环境变量(表 3).在所有环境因子中, 电导率、pH、平均NDVI、DOC、DSi和平均坡度对微生物群落有极显著影响(P < 0.01), 流域面积、河网长度、灌木所占流域面积的百分比和平均海拔对微生物群落有显著影响(P < 0.05).
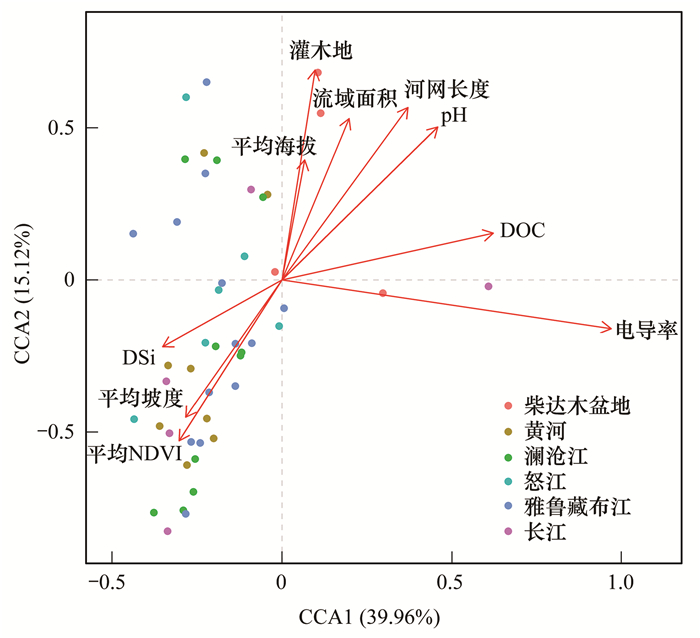
|
仅显示显著性相关的环境变量 图 9 OTU水平上群落组成与环境因子的CCA分析 Fig. 9 Canonical correspondence analysis of bacterial community (at OTU level) and environmental variables |
|
|
表 3 与微生物群落结构呈显著性相关的环境变量 Table 3 Significant environmental variables associated with the variation in the bacterial communities |
将细菌门、纲和属分类单元的优势类群与环境因子进行Spearman相关性分析(图 10).在门水平上, 放线菌门(Actinobacteriota)和DSi显著负相关; 蓝细菌门(Cyanobacteria)和TP显著负相关, 和SRP/TP显著正相关.在纲水平上, α-变形菌纲和SRP显著负相关.在属水平上, hgcI clade与TN、NO3--N、DSi和平均NDVI显著负相关; 与平均海拔、流域面积、河网长度和灌木面积占比显著正相关.
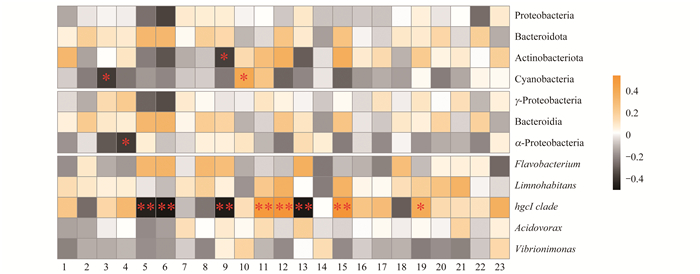
|
1.pH, 2.电导率, 3.TP, 4.SRP, 5.TN, 6.NO3--N, 7.NH4+-N, 8.DOC, 9.DSi, 10.SRP/TP, 11.平均海拔, 12.流域面积, 13.平均NDVI, 14.平均坡度, 15.河网长度, 16.耕地, 17.林地, 18.草地, 19.灌木地, 20.湿地, 21.人造地表, 22.裸地, 23.冰川和永久积雪; *和**分别表示显著性水平P < 0.05和P < 0.01 图 10 门、纲和属分类单元优势类群与环境因子的Spearman相关性分析 Fig. 10 Spearman correlations between enriched bacteria and environmental variables |
青藏高原所有采样点的多样性指数均表明青藏高原河流细菌的丰富度和多样性处于较高水平.其中, 表征物种丰富度的Chao指数和OTU richness指数与SRP/TP的值有极显著负相关关系, 这与刘洋等[20]研究的结果一致.当水体流速达到一定值时, 水流不断冲刷岸边土壤, 将大量泥沙带入水体, 导致水体浊度增加.从岸边土壤带入的较多碳源和氮磷等营养物质, 为颗粒附着型细菌提供了良好的生存条件[11].
经过物种注释, 共在青藏高原各河流样点中注释到微生物65门, 优势类群包括变形菌门、拟杆菌门、放线菌门和蓝细菌门.这一结果符合典型淡水细菌群落在门水平上的构成规律[21].在纲水平上, γ-变形菌纲和拟杆菌纲分别是第一和第二优势类群, 这可能是因为青藏高原海拔高、气温低, 而这两种类群能够适应寒冷, 在低温环境中具有较高的竞争力[8, 22].
属水平上各流域的第一优势类群均为Flavobacterium, 柴达木盆地、长江、澜沧江和怒江的第二优势类群是Limnohabitans, 雅鲁藏布江的第二优势类群是hgcI clade.这些细菌类群都与氮循环过程紧密相关:Flavobacterium属于反硝化细菌[23], Limnohabitans在硝酸盐还原过程中起到关键作用[24], hgcI clade具有较强的吸收氮化合物的能力[22].这些参与氮循环的细菌相对丰度较高可能是因为青藏高原近年来氮沉降显著增加[25].而细菌作为氮循环过程的关键组成部分, 其群落结构因此受到影响[26].差异性分析显示, Limnohabitans的相对丰度在多个流域之间呈现出显著性差异.这可能是因为本文采样点的pH值范围较大(5.58~8.74), 而Limnohabitans偏好非酸性的环境[27].如Limnohabitans相对丰度最低的黄河流域, 其平均pH值也是最低的.黄河流域的hgcI clade相对丰度与其他流域呈显著性差异, 这可能是因为hgcI clade对低氧环境具有耐受性[22], 在高海拔地区具有较强的竞争力.黄河流域采样点的平均海拔最低, 因而hgcI clade的相对丰度较低.
此外, 在OTU注释结果中包含模糊条目的物种共有2门43纲792属, 说明青藏高原河流中仍存在大量不能确定分类层次或物种信息的微生物资源, 具有深入研究的价值.
3.2 微生物与环境因子的关系本文在分析环境因子和微生物群落之间的相关性时, 在水环境指标之上加入了流域特征因子和地表覆盖类型因子.不同地表覆盖类型向河流输入不同颗粒物和营养物[28], 影响细菌群落分布[29].河流是开放流动的系统, 能够从周围土壤输入外源细菌[28], 因而河流的细菌群落结构在某种程度上也是流域特征的体现.CCA结果显示, 较多环境因子对微生物群落有显著影响.其中电导率的R2为所有因子中最大的(表 3), 说明电导率是青藏高原河流细菌群落分布的最大影响因素.电导率受到溶质盐含量的影响.刘睿等[30]研究发现, 溶解性营养盐对低温水体中的部分优势菌群具有积极影响.青藏高原海拔高、气温低, 一般营养元素较为匮乏, 溶质盐含量可能成为影响细菌群落分布的主要限制性因子.
由于不同细菌对生长环境的酸碱性要求不同, pH被认为是显著影响微生物群落组成的因素[31].流域的平均海拔同样对微生物群落组成有显著性影响, 这可能是因为不同微生物对含氧量的要求不同所致.如对hgcI clade与平均海拔的Spearman相关性分析显示, 两者具有显著正相关, 可能是因为hgcI clade对低氧具有耐受性[22], 随着海拔升高水中含氧量下降, 其相对丰度增高.河网长度与流域面积对微生物群落组成的显著影响与Read等[32]研究结果一致, 不同空间距离引起的河水滞留时间差异是影响群落结构的原因.平均坡度在一定程度上影响降雨对地表物质的冲刷能力[8], 两岸颗粒物通过地表径流的方式进入河流, 将陆地上的生物信息带入水体[30], 从而造成河流微生物群落结构的差异.平均NDVI和灌木面积占比对微生物群落构成有显著影响.Ren等[33]研究发现, 青藏高原植被退化会导致下游河流细菌群落发生变化, 这与本文研究结果一致.Wang等[34]研究发现, 受气候变暖影响, 青藏高原高山灌木线显著爬升.流域来源于岸边土壤的生物与非生物信息都会通过径流的方式进入到水体中, 从而对下游河流的细菌群落结构产生影响[35].青藏高原河流的细菌群落组成, 或许能够反映流域整体特征的变化.
Jackson等[36]在比较密西西比河浮游和颗粒附着细菌群落时, 发现蓝细菌门在浮游群落中的比例明显较低.SRP/TP值可用来表征水体悬浮颗粒物的含量[6].在本研究中, 用SRP/TP与蓝细菌门的相对丰度做Spearman相关性分析, 结果呈显著正相关关系, 这与Jackson等[36]得到的结论相反.将TP的值与蓝细菌门相对丰度做Spearman相关性分析, 发现TP与蓝细菌门相对丰度有显著负相关关系, 这与Bridgeman等[37]所发现的结论相反.说明蓝细菌门在青藏高原水体中所呈现出的性质具有特殊性, 仍需深入研究.
4 结论(1) 所调查研究的青藏高原河流中共检测出24 705个细菌OTUs, 涵盖65门1 311属.门水平上优势类群主要有变形菌门、拟杆菌门和放线菌门; 纲水平优势类群主要有γ-变形菌纲、拟杆菌纲和α-变形菌纲; 属水平优势类群主要有Flavobacterium和Limnohabitans.青藏高原河流中存在大量不能确定分类层次或物种信息的微生物资源, 具有深入研究的价值.
(2) 青藏高原河流细菌的丰富度和多样性均处于较高水平.其中, 表征物种丰富度的Chao指数和OTU richness指数均与SRP/TP的值有极显著负相关关系.PCoA分析和Adonis多因素方差分析表明, 微生物群落组成在青藏高原各流域间有极显著差异.
(3) 电导率、pH、平均NDVI、DSi、DOC、平均坡度、河网长度、流域面积、灌木面积占比和平均海拔对微生物群落产生显著影响.其中, 电导率是青藏高原河流细菌群落分布的最大影响因素.
(4) 河流细菌群落可用来反映流域环境特征.
致谢: 感谢国家青藏高原科学数据中心(http://data.tpdc.ac.cn)提供流域边界数据.
| [1] |
邓冉, 邵怀勇, 黄宝荣, 等. 青藏高原生态系统完整性远程评价与国家公园群建设时序研究[J]. 生态学报, 2021, 41(3): 847-860. Deng R, Shao H Y, Huang B R, et al. Remote assessment on ecosystem integrity of the Qinghai-Tibet Plateau and research on National Parks Group construction sequence[J]. Acta Ecologica Sinica, 2021, 41(3): 847-860. |
| [2] | Mi X C, Feng G, Hu Y B, et al. The global significance of biodiversity science in China: an overview[J]. National Science Review, 2021, 8(7). DOI:10.1093/nsr/nwab032 |
| [3] | Yao T D, Thompson L G, Mosbrugger V, et al. Third pole environment (TPE)[J]. Environmental Development, 2012, 3: 52-64. DOI:10.1016/j.envdev.2012.04.002 |
| [4] | Li M, Zhang X Z, Wu J S, et al. Declining human activity intensity on alpine grasslands of the Tibetan Plateau[J]. Journal of Environmental Management, 2021, 296. DOI:10.1016/j.jenvman.2021.113198 |
| [5] | Liang J, Lupien R L, Xie H C, et al. Lake ecosystem on the Qinghai-Tibetan Plateau severely altered by climatic warming and human activity[J]. Palaeogeography, Palaeoclimatology, Palaeoecology, 2021, 576. DOI:10.1016/j.palaeo.2021.110509 |
| [6] |
胡安谊, 李姜维, 杨晓永, 等. 宁波三江口水域原核生物群落结构分析[J]. 环境科学, 2015, 36(7): 2487-2495. Hu A Y, Li J W, Yang X Y, et al. Analysis of prokaryotic community structure in river waters of the Ningbo Sanjiang Mouth[J]. Environmental Science, 2015, 36(7): 2487-2495. DOI:10.13227/j.hjkx.2015.07.022 |
| [7] |
李轶, 雷梦婷, 杨楠, 等. 河流微生物生态学的研究进展[J]. 水资源保护, 2022, 38(1): 190-197. Li Y, Lei M T, Yang N, et al. Research and prospect on river microbial ecology[J]. Water Resources Protection, 2022, 38(1): 190-197. |
| [8] | Zhang Q Q, Jian S L, Li K M, et al. Community structure of bacterioplankton and its relationship with environmental factors in the upper reaches of the Heihe River in Qinghai Plateau[J]. Environmental Microbiology, 2021, 23(2): 1210-1221. DOI:10.1111/1462-2920.15358 |
| [9] |
但言, 沈子伟, 余凤琴, 等. 嘉陵江合川段秋季微生物多样性研究[J]. 淡水渔业, 2019, 49(1): 3-8. Dan Y, Shen Z W, Yu F Q, et al. The research on microbial diversity of Hechuan reach for Jialing River in autumn[J]. Freshwater Fisheries, 2019, 49(1): 3-8. DOI:10.3969/j.issn.1000-6907.2019.01.001 |
| [10] |
周宁一. 青藏高原微生物多样性研究[J]. 微生物学通报, 2014, 41(11): 2378. Zhou N Y. Microbial diversity on the Tibetan Plateau[J]. Microbiology China, 2014, 41(11): 2378. DOI:10.13344/j.microbiol.china.148011 |
| [11] |
程豹, 望雪, 徐雅倩, 等. 澜沧江流域浮游细菌群落结构特征及驱动因子分析[J]. 环境科学, 2018, 39(8): 3649-3659. Cheng B, Wang X, Xu Y Q, et al. Bacterioplankton community structure in the Lancang River basin and the analysis of its driving environmental factors[J]. Environmental Science, 2018, 39(8): 3649-3659. |
| [12] | Johnson J S, Spakowicz D J, Hong B Y, et al. Evaluation of 16S rRNA gene sequencing for species and strain-level microbiome analysis[J]. Nature Communications, 2019, 10(1). DOI:10.1038/s41467-019-13036-1 |
| [13] |
黄志强, 邱景璇, 李杰, 等. 基于16S rRNA基因测序分析微生物群落多样性[J]. 微生物学报, 2021, 61(5): 1044-1063. Huang Z Q, Qiu J X, Li J, et al. Exploration of microbial diversity based on 16S rRNA gene sequence analysis[J]. Acta Microbiologica Sinica, 2021, 61(5): 1044-1063. |
| [14] | Tringe S G, Hugenholtz P. A renaissance for the pioneering 16S rRNA gene[J]. Current Opinion in Microbiology, 2008, 11(5): 442-446. DOI:10.1016/j.mib.2008.09.011 |
| [15] | Zhang G Q, Yao T D, Xie H J, et al. Increased mass over the Tibetan Plateau: from lakes or glaciers?[J]. Geophysical Research Letters, 2013, 40(10): 2125-2130. DOI:10.1002/grl.50462 |
| [16] | 张国庆. 青藏高原流域边界数据集(2016)[R]. 北京: 国家青藏高原科学数据中心, 2019. |
| [17] | Jun C, Ban Y F, Li S N. Open access to earth land-cover map[J]. Nature, 2014, 514(7523). DOI:10.1038/514434c |
| [18] | 中国生态系统研究网络科学委员会. 水域生态系统观测规范[M]. 北京: 中国环境科学出版社, 2007. |
| [19] | Wickham H. ggplot2: elegant graphics for data analysis[M]. Switzerland: Springer, 2016. |
| [20] |
刘洋, 刘琦, 田雨露, 等. 滦河干流中上游浮游细菌群落多样性及其影响因素研究[J]. 生态学报, 2022, 42(12): 5103-5114. Liu Y, Liu Q, Tian Y L, et al. Characteristics of bacterioplankton community with relations to environmental parameters in upstream and midstream of the Luanhe River, China[J]. Acta Ecologica Sinica, 2022, 42(12): 5103-5114. |
| [21] | Zwart G, Crump B C, Kamst-Van Agterveld M P, et al. Typical freshwater bacteria: an analysis of available 16S rRNA gene sequences from plankton of lakes and rivers[J]. Aquatic Microbial Ecology, 2002, 28(2): 141-155. |
| [22] |
王松鸽, 赖子尼, 麦永湛, 等. 珠江河网冬季浮游细菌群落结构及其影响因素[J]. 中国水产科学, 2019, 26(3): 522-533. Wang S G, Lai Z N, Mai Y Z, et al. Bacterioplanktonic community's composition and their environmental impact factors of the Pearl River Delta in winter[J]. Journal of Fishery Sciences of China, 2019, 26(3): 522-533. |
| [23] |
张紫薇, 陈召莹, 张甜娜, 等. 基于高通量绝对定量测序解析岗南水库微生物群落的时空分布特征及关键驱动因素[J]. 环境科学学报, 2022, 42(2): 224-239. Zhang Z W, Chen Z Y, Zhang T N, et al. Spatiotemporal characteristics and key driving factors of microbial community evolution based on high-throughput absolute quantification sequencing in the Gangnan Reservoir[J]. Acta Scientiae Circumstantiae, 2022, 42(2): 224-239. |
| [24] | Liu S M, Zhao F B, Fang X. The relationship between the community structure and function of bacterioplankton and the environmental response in Qingcaosha Reservoir[J]. Water, 2021, 13(22). DOI:10.3390/w13223155 |
| [25] | Li J J, Yang C, Zhou H K, et al. Responses of plant diversity and soil microorganism diversity to water and nitrogen additions in the Qinghai-Tibetan Plateau[J]. Global Ecology and Conservation, 2020, 22. DOI:10.1016/j.gecco.2020.e01003 |
| [26] |
刘乐冕, 杨军, 余小青, 等. 厦门后溪流域沿城乡梯度浮游细菌多样性及其与环境因子的关系[J]. 应用与环境生物学报, 2012, 18(4): 591-598. Liu L M, Yang J, Yu X Q, et al. Bacterioplankton diversity in Xiamen Houxi River watershed along an urban-rural gradient and its relation to environmental factors[J]. Chinese Journal of Applied and Environmental Biology, 2012, 18(4): 591-598. |
| [27] |
李亚莉, 杨正健, 许尤, 等. 清江上游利川段浮游细菌群落结构特征及其影响因素[J]. 生态学杂志, 2020, 39(11): 3756-3765. Li Y L, Yang Z J, Xu Y, et al. Community structure of planktonic bacteria and its driving factors along the upstream area of Qingjiang River in Lichuan City, Hubei Province[J]. Chinese Journal of Ecology, 2020, 39(11): 3756-3765. |
| [28] |
赵君, 王鹏, 徐启渝, 等. 袁河流域不同土地利用方式下河流浮游细菌群落结构特征[J]. 环境科学学报, 2020, 40(3): 890-900. Zhao J, Wang P, Xu Q Y, et al. Effects of land use in drainage basin on bacterioplankton communities in the Yuan River[J]. Acta Scientiae Circumstantiae, 2020, 40(3): 890-900. |
| [29] | Lear G, Washington V, Neale M, et al. The biogeography of stream bacteria[J]. Global Ecology and Biogeography, 2013, 22(5): 544-554. |
| [30] |
刘睿, 吴巍, 周孝德, 等. 渭河浮游细菌群落结构特征及其关键驱动因子[J]. 环境科学学报, 2017, 37(3): 934-944. Liu R, Wu W, Zhou X D, et al. Bacterioplankton community structure in Weihe River and its relationship with environmental factors[J]. Acta Scientiae Circumstantiae, 2017, 37(3): 934-944. |
| [31] | Xia N, Xia X H, Liu T, et al. Characteristics of bacterial community in the water and surface sediment of the Yellow River, China, the largest turbid river in the world[J]. Journal of Soils and Sediments, 2014, 14(11): 1894-1904. |
| [32] | Read D S, Gweon H S, Bowes M J, et al. Catchment-scale biogeography of riverine bacterioplankton[J]. The ISME Journal, 2015, 9(2): 516-526. |
| [33] | Ren Z, Niu D C, Ma P P, et al. Bacterial communities in stream biofilms in a degrading grassland watershed on the Qinghai-Tibet Plateau[J]. Frontiers in Microbiology, 2020, 11. DOI:10.3389/fmicb.2020.01021 |
| [34] | Wang Y F, Liang E Y, Lu X M, et al. Warming-induced shrubline advance stalled by moisture limitation on the Tibetan Plateau[J]. Ecography, 2021, 44(11): 1631-1641. |
| [35] | Ren Z, Gao H K, Luo W, et al. Bacterial communities in surface and basal ice of a glacier terminus in the headwaters of Yangtze River on the Qinghai-Tibet Plateau[J]. Environmental Microbiome, 2022, 17(1). DOI:10.1186/s40793-022-00408-2 |
| [36] | Jackson C R, Millar J J, Payne J T, et al. Free-living and particle-associated bacterioplankton in large rivers of the Mississippi River basin demonstrate biogeographic patterns[J]. Applied and Environmental Microbiology, 2014, 80(23): 7186-7195. |
| [37] | Bridgeman T B, Chaffin J D, Kane D D, et al. From river to lake: phosphorus partitioning and algal community compositional changes in Western Lake Erie[J]. Journal of Great Lakes Research, 2012, 38(1): 90-97. |
 2023, Vol. 44
2023, Vol. 44


