2. 华东理工大学资源与环境工程学院, 国家环境保护化工过程环境风险评价与控制重点实验室, 上海 200237;
3. 上海化学工业区中法水务发展有限公司, 上海 201400;
4. 上海市污染控制与生态安全研究院, 上海 200240
2. State Environmental Protection Key Laboratory of Environmental Risk Assessment and Control on Chemical Process, School of Resources and Environmental Engineering, East China University of Science and Technology, Shanghai 200237, China;
3. Chemical Industrial Zone of Shanghai Zhongfa Water Affair Devlopment Limited Company, Shanghai 201400, China;
4. Shanghai Institute of Pollution Control and Ecological Security, Shanghai 200240, China
石油化工产业是国家的支柱产业, 在国民经济中扮演着极其重要的角色[1].石油化工废水来源多样, 且排放量巨大[2, 3].据2019年中国石油化工行业报告显示, 我国石化废水年排放量已达到40亿t[3], 主要来自炼油和石油产品加工过程[4].石油化工废水的处理已成为石油化工行业绿色发展主要挑战之一[5~7].以石油产品加工过程中产生的废水为例, 该种废水通常含有不同浓度的氨氮与氮氧化物[2], 直接排放会加剧水体的富营养化污染.在石化行业逐步削减工业取新水总量, 并极力推行污水资源化利用的大背景下, 石化废水的脱氮处理更是关键环节[8].
活性污泥法已被广泛应用于石化废水脱氮处理[9~11], 然而活性污泥法中的功能微生物群落高度复杂[12], 相关的脱氮过程交错多样[13].据报道在活性污泥体系中, 最核心的脱氮途径是硝化-反硝化结合的脱氮途径[12, 14, 15], 但同时也可能伴生异化硝酸盐还原(DNRA)[16], 厌氧氨氧化(ANAMMOX)等过程[17, 18].脱氮微生物的群落组成及状态直接影响废水中氮素生物转化的途径和效率[19].全面地了解脱氮微生物群落结构和功能特征可为石化废水脱氮处理的过程诊断与定向调控提供微生物学依据.然而, 以往对石化废水处理工艺中微生物的研究都基于16S rRNA基因高通量测序分析[20, 21], 但由于扩增过程带来的偏向性以及很难直接从功能基因角度获取结果[22], 将导致无法精确地反映复杂系统中实际的菌群结构与功能特征, 使人们对石化废水处理中脱氮微生物结构与功能的理解受到限制.
近年来, 基于Illumina测序的宏基因组测序分析技术发展迅速, 测序分析成本逐渐降低, 在微生物群落深度分析中显示出巨大的优势, 已被广泛应用于饮用水、土壤、实验室模拟反应器和城市污水处理厂等自然或工程环境中微生物群落的分析[13, 14, 23, 24].宏基因组测序分析已经被证明是一个强有力的工具, 可以助力解开具有较高多样性的工程生态系统中的“暗物质”[25, 26].然而, 宏基因组测序只是潜能而非实际功能或活性的测量, 联合采用宏基因组和宏转录组的多组学方法能更好地揭示活性污泥微生物内部的物种相互作用和基因表达活性对环境的响应[27, 28].基于多组学分析方法的石化废水处理厂中脱氮微生物的分析研究未见报道[29, 30].本研究通过宏基因组和宏转录组技术对一实际石化废水生物处理系统中脱氮微生物进行探究, 将有助于明晰石化废水生物脱氮处理的生物过程.
1 材料与方法 1.1 污水处理厂运行数据比较及污泥样品采集在2021年1~8月期间, 对位于上海某化工区污水处理厂的某条生物脱氮A/O工艺线, 监测了运行数据并收集了污泥样本.该条工艺线主要处理化工区内多家石化企业排出的数十种废水源的混合废水, 工艺日处理水量平均值为(6 788±99) m3·d-1.该A/O工艺缺氧池和好氧池水力停留时间分别为9 h和27 h, 污泥停留时间约为20 d.
工艺线的运行数据由在线水质监测探头定时监测, 并收集了这期间工艺运行的实际监测数据, 通过对进出水氨氮(NH4+-N)、硝态氮(NO3--N)和亚硝态氮(NO2--N)浓度的统计比较, 厘清工艺运行效能.污泥样本的采集, 选择了工艺运行的两个典型时期作为采样的时间点, 即工艺出水水质稳定时期与工艺出水水质波动时期, 以反映工艺由“稳定”状态到“失稳”状态的过程, 聚焦此过程中的微生物特征.第一次样本采集在3月底, 此期间出水水质稳定; 第二次样本采集在6月初, 某家石化企业在5月31日至6月5日间进行的检修工作造成了工艺线进水中总油浓度的增加, 此期间进水ρ(总油)为35.4 mg·L-1, 增加了近3倍, 是工艺“失稳”期间主要的进水水质变化, 并在6月1~10日间出水NH4+-N浓度出现波动.每次污泥采样在好氧池首尾两端各设置一个采样位点, 共计2个采样点, 记为Of和Ob; 缺氧池设置1个采样位点, 记为A.为降低系统误差, 每个采样点均进行两次重复采样.污泥样品采集后用液氮快速冷冻, 立即放入-80℃冰箱保存.
1.2 DNA及RNA的提取及测序利用FastDNATM Spin及RNA PowerSoil® (MoBio Laboratories Inc, USA) 试剂盒对所采集的污泥样品进行总DNA和RNA提取.总DNA的提取步骤为:适量污泥混合液经低温离心后取约0.5 g加入到裂解介质管, 并向其中依次加入磷酸钠缓冲液和MT Buffer, 振荡离心后保留上清液至洁净离心管.加入250 μL PPS试剂混匀, 离心后保留上清液至洁净离心管并加入1.0 mL Binding Matrix, 振荡静置后小心去除500 μL上清液, 剩余混合物转移到SPINTM Filter.多次洗涤离心后, 加入100 μL ddH2O洗脱离心, 所得液体即为提取的总DNA, 测序前置于-20℃冰箱保存.总RNA的提取步骤为:适量污泥混合液经低温离心后去除上清液, 加入100 μL TE缓冲液和350 μL RL裂解液, 振荡混匀后加入250 μL的无水乙醇, 混合液转移至CR3吸附柱.低温离心后去除收集管中废液, 多次洗涤离心后, 加入30 μL ddH2O洗脱离心, 所得液体即为提取的总RNA, 测序前置于-80℃冰箱保存.污泥样品的DNA及RNA序列分别由Hiseq-PE150和HiSeq xten sequencer (Illumina)测序平台进行测序(派森诺, 上海).具体测序流程为:将DNA样品加入Fragmentation Buffer, 采用超声破碎仪进行随机打断, 将打断后得到的短片段DNA用于文库构建, 对于质检合格的文库将采用Illumina HiSeq 2500高通量测序平台进行PE150测序.RNA的测序流程类似, 合格的RNA样品用以文库的构建, 然后采用Illumina HiSeq Xten测序平台进行测序.
1.3 宏基因组与宏转录组分析基于DIAMOND (版本0.8.34)[31]分析手段, 将污泥样本的DNA和RNA序列与氮循环关键酶的基因数据库进行比较, 该数据库包含13个氮循环关键酶基因[32].基因组的组装使用Megahit, 超过80% 的测序序列组装成重叠区, 大于500 bp的重叠区将用于进一步的分析.CheckM (版本1.0.11)[33]用于评估拼接出的基因组草图的完整性和污染情况.基于Kaiju分析手段, 对微生物物种的组成与比例特征进行分析, 评价不同样品中微生物的多样性差异[34].
2 结果与讨论 2.1 工艺脱氮效能系统分析了两次污泥采样前后A/O工艺共7个月的脱氮效能相关数据, 对进出水NH4+-N、NO3--N和NO2--N浓度以及脱氮效率进行整理, 如图 1所示.工艺进水ρ(总氮)平均值为(52.5±1.7) mg·L-1 (图 1).监测期内, 总氮去除率平均值为(79.9±0.6)%, 去除率随温度变化无明显波动.出水ρ(NH4+-N)平均值为(1.3±0.1) mg·L-1, 在6月初的出水NH4+-N浓度波动期间, 出水ρ(NH4+-N)平均值达到(9.8±0.8) mg·L-1.出水ρ(NO2--N)和ρ(NO3--N)平均值分别为 < 0.2 mg·L-1和(2.8±0.2) mg·L-1.结果表明在监测期内, 体系中NO2--N和NO3--N的转化效果良好, 但NH4+-N转化效果的波动幅度大, 初步判断NH4+-N的转化可能是A/O体系脱氮过程中的薄弱环节.

|
(a) 温度变化和总氮去除效率, (b) 进、出水NH4+-N浓度, (c) 进、出水NO3--N浓度, (d) 进、出水NO2--N浓度; 虚线标记为污泥采样时间点 图 1 A/O工艺脱氮效能 Fig. 1 Nitrogen removal efficiency of the A/O process |
为了深入分析体系中氮代谢过程, 以测序获得的DNA及RNA序列, 比对硝化过程关键基因(氨氧化:amoA和hao; 亚硝酸盐氧化:nxrA)、厌氧氨氧化过程关键基因(hzsA和hdh)、反硝化过程关键基因(napA、narG、nirK、nirS、cnorB、qnorZ、nosZⅠ和nosZⅡ), 用标准化序列丰度及表达活性代表样品中关键功能基因的丰度与表达活性, 用每百万序列数(reads per million, RPM), 即每百万条测得的序列中比对到的目标物种基因组的序列条数作为评估标准, 衡量氮代谢中关键功能基因丰度的大小与表达活性的强弱[24, 27].
如图 2所示, 从基因丰度来看, 3月底和6月初不同采样时期的基因丰度变化差异不明显, 说明系统中脱氮菌群结构具有一定的稳定性.其中, 厌氧氨氧化过程功能基因丰度平均值低于(0.05±0.01) RPM [图 2(a)和2(c)].反硝化过程功能基因丰度平均值为(91.65±7.43) RPM, 约占工艺内氮代谢关键基因丰度的90%.相比之下, 氨氧化过程功能基因丰度相对较低, amoA基因约占工艺内氮代谢关键基因丰度的0.17%, 比以往的研究报道都要小[35~37].
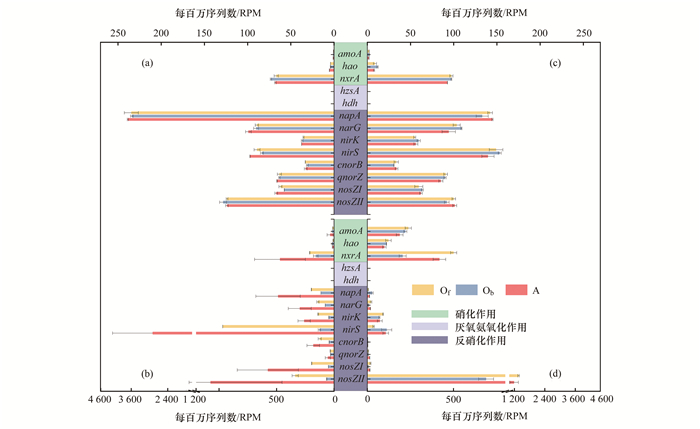
|
(a) 3月底样品硝化、厌氧氨氧化和反硝化的关键基因丰度, (b) 3月底样品硝化、厌氧氨氧化和反硝化的关键基因表达活性, (c) 6月初样品硝化、厌氧氨氧化和反硝化的关键基因丰度, (d) 6月初样品硝化、厌氧氨氧化和反硝化的关键基因表达活性; Of、Ob和A分别表示好氧池前端、好氧池后端和缺氧池采样点 图 2 A/O系统中氮代谢功能基因分析 Fig. 2 Analysis of nitrogen metabolism genes in the A/O system |
此外, 本研究还测定了硝化、厌氧氨氧化和反硝化关键基因的表达活性[图 2(b)和2(d)].但几乎监测不到厌氧氨氧化过程基因的表达.对于硝化作用来说, 6月初采样点氨氧化功能基因(amoA和hao)的表达活性是3月底采样点的近10倍, 6月初采样点亚硝酸盐氧化功能基因(nxrA)的表达活性是3月底采样点的近1.5倍, 硝化作用的功能基因表达活性都有增长, 可能是由于反应池内NH4+-N浓度的增加, 促进了相应氨氧化功能基因的表达.虽然6月初amoA基因的表达活性更高, 但体系中amoA基因的低丰度情况并未改变, 因此出水NH4+-N浓度依旧升高.
2.3 功能微生物组成分析对原始样品的宏基因组序列数据通过Kaiju分析手段, 计算体系中微生物物种组成与比例特征, 如图 3所示.原始样品中有近50%的序列不能被注释, 活性污泥内仍有大部分生物不能被识别.在门水平中, 变形菌门(Proteobacteria)占可注释细菌总数的近60%, 这与以往的报道相似[38, 39], 在污水处理厂中变形菌门是活性污泥中最主要的门类.在属水平中, 陶厄氏菌属(Thauera)和固氮弧菌属(Azoarcus)是主要的优势菌属, 占注释菌属比例为62%, 是常见的反硝化菌属[40~42], 而且都属于变形菌门(Proteobacteria)中的β-变形菌(β-Proteobacteria). β-变形菌通常被认为是活性污泥法群落中的主要成员, 在其他的活性污泥微生物研究中也报告了这一发现[42].同时, Kaiju分析结果发现氨氧化功能菌的丰度小于0.5%.
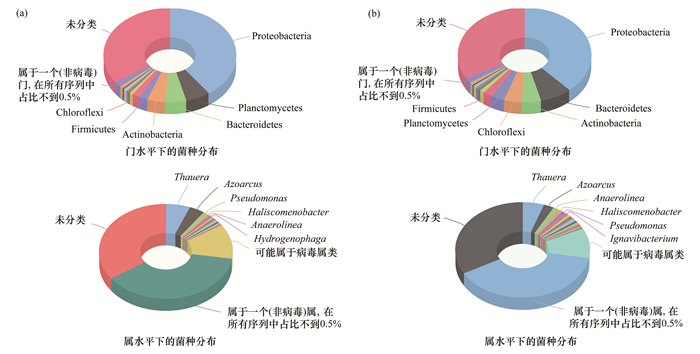
|
(a) 3月底采样门和属水平下的物种组成; (b) 6月初采样门和属水平下的物种组成 图 3 宏基因组数据分类评价不同层次微生物群落组成 Fig. 3 Metagenomic data were used to assess the composition of microbial communities at different levels |
通过原始DNA序列数据, 利用Megahit组装共获得约1 255 238条500 bp以上的重叠区(contigs), 通过质量筛选(完整度>70%, 污染度 < 10%), 获得126个高质量的基因组草图(draft metagenome-assembled genomes).对这些基因组草图进行分类注释, 发现共涉及近20个门类, 其中, 主要门类为变形菌门(Proteobacteria)、浮霉菌门(Planctomycetes)和拟杆菌门(Bacteroidetes).同时, 利用脱氮功能基因与筛选出的基因组草图做比对, 进行功能注释进而筛选出脱氮功能微生物.发现126个高质量基因组草图中携带反硝化过程基因的草图个数为81个, 且未发现有携带amoA基因的基因组草图.为了更深入地了解系统中的优势功能菌, 本研究将基因组草图与原始序列数据进行比对, 计算得到相对丰度与表达活性, 以此判断体系中的优势菌, 筛选相对丰度与表达活性最高的前5株菌, 如表 1所示.结果发现, 这5株优势菌都携带有反硝化过程的功能基因, 尤其是PRO1这株菌携带有硝酸盐还原酶(napA/narG)、亚硝酸盐还原酶(nirK/nirS)及一氧化氮还原酶(cnorB/qnorZ)的编码基因.
|
|
表 1 A/O工艺体系中的5株优势菌 Table 1 Five dominant bacteria in the system |
为了进一步探究体系中的氨氧化功能微生物, 利用宏转录组的序列数据进行深度挖掘, 筛选出携带有amoA基因的序列, 通过Kaiju工具分析体系中氨氧化功能菌的组成情况, 如图 4所示.由分析结果可知, 两个采样时期下, 体系中主要的氨氧化功能菌属是亚硝化单胞菌(Nitrosomonas), 约占注释序列的近80%.而在第一次采样点中, 主要的氨氧化菌种为Nitrosomonas communis, 而在第二次采样点中, 主要的氨氧化菌种为Nitrosomonas ureae, 据以往的研究报导, N. communis和N. ureae都是Nitrosomonas属中常见的物种, 在代谢过程中未发现有特殊差异, 在实际污水厂中常被检测到[43].体系中氨氧化菌种的变化可能是由于季节变化发生的演替, 据报导, 季节温度的变化是硝化菌群落演替的关键因素[26, 4].工艺线中氨氧化菌群丰度低和结构单一, 这可能是体系中氨氧化作用处于劣势的主要原因[44, 45].

|
(a) 3月底属和种水平下的携带amoA基因的物种组成, (b) 6月初属和种水平下的携带amoA基因的物种组成; Of、Ob和A分别表示好氧池前端、好氧池后端和缺氧池采样点 图 4 宏转录组数据分析携带amoA基因的微生物群落组成 Fig. 4 Composition of the microbial community carrying the amoA gene according to metatranscriptomic analysis |
本研究通过宏基因组和宏转录组, 对上海某化工区的某条石化废水A/O脱氮工艺线的活性污泥样品进行了深入分析, 聚焦工艺运行由“稳定”状态到“失稳”状态的过程, 发现工艺主要的脱氮途径为硝化-反硝化脱氮, 且未发现工艺内有厌氧氨氧化发生.反硝化功能基因占关键脱氮功能基因比例约90%, 其次是亚硝酸盐氧化功能基因约9%, 氨氧化功能基因amoA基因最少, 仅占约0.17%.氨氧化功能菌在工艺中丰度极低且结构单一, 主要的氨氧化功能菌属是Nitrosomonas, 这可能是工艺的NH4+-N转化“失稳”的主要原因.提高工艺中的氨氧化菌丰度以及丰富氨氧化功能菌的种类可能对石化废水A/O工艺出水的稳定达标具有一定的积极作用.
| [1] | Jafarinejad S. Petroleum waste treatment and pollution control[M]. Oxford: Butterworth-Heinemann, 2016. |
| [2] | Ghimire N, Wang S. Biological treatment of petrochemical wastewater[A]. In: Zoveidavianpoor M (Ed. ). Petroleum Chemicals-Recent Insight[M]. London: IntechOpen, 2018. |
| [3] | Tian X M, Song Y D, Shen Z Q, et al. A comprehensive review on toxic petrochemical wastewater pretreatment and advanced treatment[J]. Journal of Cleaner Production, 2020, 245. DOI:10.1016/j.jclepro.2019.118692 |
| [4] | Jiang Y M, Huang H Y, Tian Y R, et al. Stochasticity versus determinism: microbial community assembly patterns under specific conditions in petrochemical activated sludge[J]. Journal of Hazardous Materials, 2021, 407. DOI:10.1016/j.jhazmat.2020.124372 |
| [5] | Priyadarshini M, Ahmad A, Das S, et al. Application of microbial electrochemical technologies for the treatment of petrochemical wastewater with concomitant valuable recovery: a review[J]. Environmental Science and Pollution Research, 2021. DOI:10.1007/s11356-021-14944-w |
| [6] | Jothinathan L, Cai Q Q, Ong S L, et al. Organics removal in high strength petrochemical wastewater with combined microbubble-catalytic ozonation process[J]. Chemosphere, 2021, 263. DOI:10.1016/j.chemosphere.2020.127980 |
| [7] | Wang L X, Liu T, Chen S, et al. Enhancing the treatment of petrochemical wastewater using redox mediator suspended biofilm carriers[J]. Biochemical Engineering Journal, 2021, 173. DOI:10.1016/j.bej.2021.108087 |
| [8] | Andreides D, Varga Z, Pokorna D, et al. Performance evaluation of sulfide-based autotrophic denitrification for petrochemical industry wastewater[J]. Journal of Water Process Engineering, 2021, 40. DOI:10.1016/j.jwpe.2020.101834 |
| [9] | Wang D, Sun D X, Tian X, et al. Role of microbial communities on organic removal during petrochemical wastewater biological treatment with pure oxygen aeration[J]. Journal of Water Process Engineering, 2021, 42. DOI:10.1016/j.jwpe.2021.102151 |
| [10] | Wang S, Ghimire N, Xin G, et al. Efficient high strength petrochemical wastewater treatment in a hybrid vertical anaerobic biofilm (HyVAB) reactor: a pilot study[J]. Water Practice and Technology, 2017, 12(3): 501-513. DOI:10.2166/wpt.2017.051 |
| [11] | Chu L B, Ding P Y, Ding M C. Pilot-scale microaerobic hydrolysis-acidification and anoxic-oxic processes for the treatment of petrochemical wastewater[J]. Environmental Science and Pollution Research, 2021, 28(41): 58677-58687. DOI:10.1007/s11356-021-14810-9 |
| [12] |
张博雅, 余珂. 微生物基因数据库在氮循环功能基因注释中的应用[J]. 微生物学通报, 2020, 47(9): 3021-3038. Zhang B Y, Yu K. Application of microbial gene databases in the annotation of nitrogen cycle functional genes[J]. Microbiology China, 2020, 47(9): 3021-3038. |
| [13] |
鞠峰, 张彤. 活性污泥微生物群落宏组学研究进展[J]. 微生物学通报, 2019, 46(8): 2038-2052. Ju F, Zhang T. Advances in meta-omics research on activated sludge microbial community[J]. Microbiology China, 2019, 46(8): 2038-2052. |
| [14] | Yu K, Zhang T. Metagenomic and metatranscriptomic analysis of microbial community structure and gene expression of activated sludge[J]. PLoS One, 2012, 7(5). DOI:10.1371/journal.pone.0038183 |
| [15] | Winkler M K H, Straka L. New directions in biological nitrogen removal and recovery from wastewater[J]. Current Opinion in Biotechnology, 2019, 57: 50-55. DOI:10.1016/j.copbio.2018.12.007 |
| [16] | Pandey C B, Kumar U, Kaviraj M, et al. DNRA: a short-circuit in biological N-cycling to conserve nitrogen in terrestrial ecosystems[J]. Science of the Total Environment, 2020, 738. DOI:10.1016/j.scitotenv.2020.139710 |
| [17] | Xie G J, Liu T, Cai C, et al. Achieving high-level nitrogen removal in mainstream by coupling anammox with denitrifying anaerobic methane oxidation in a membrane biofilm reactor[J]. Water Research, 2018, 131: 196-204. DOI:10.1016/j.watres.2017.12.037 |
| [18] | Xu S Y, Wu X L, Lu H J. Overlooked nitrogen-cycling microorganisms in biological wastewater treatment[J]. Frontiers of Environmental Science & Engineering, 2021, 15(6). DOI:10.1007/s11783-021-1426-2 |
| [19] |
房平, 李雨娥, 魏东洋, 等. 污水处理过程中微生物群落多样性及其对环境因子响应的研究进展[J]. 微生物学通报, 2020, 47(9): 3004-3020. Fang P, Li Y E, Wei D Y, et al. Microbial community diversity and its response to environmental factors during sewage treatment[J]. Microbiology China, 2020, 47(9): 3004-3020. |
| [20] | Behnami A, Farajzadeh D, Isazadeh S, et al. Diversity of bacteria in a full-scale petrochemical wastewater treatment plant experiencing stable hydrocarbon removal[J]. Journal of Water Process Engineering, 2018, 23: 285-291. DOI:10.1016/j.jwpe.2018.04.015 |
| [21] | Liang J W, Mai W N, Tang J F, et al. Highly effective treatment of petrochemical wastewater by a super-sized industrial scale plant with expanded granular sludge bed bioreactor and aerobic activated sludge[J]. Chemical Engineering Journal, 2019, 360: 15-23. DOI:10.1016/j.cej.2018.11.167 |
| [22] | Eisenstein M. Microbiology: making the best of PCR bias[J]. Nature Methods, 2018, 15(5): 317-320. DOI:10.1038/nmeth.4683 |
| [23] | Zhuang J L, Zhou Y Y, Liu Y D, et al. Flocs are the main source of nitrous oxide in a high-rate anammox granular sludge reactor: insights from metagenomics and fed-batch experiments[J]. Water Research, 2020, 186. DOI:10.1016/j.watres.2020.116321 |
| [24] | Li W, Zhuang J L, Zhou Y Y, et al. Metagenomics reveals microbial community differences lead to differential nitrate production in anammox reactors with differing nitrogen loading rates[J]. Water Research, 2020, 169. DOI:10.1016/j.watres.2019.115279 |
| [25] | Gernaey K V, van Loosdrecht M C M, Henze M, et al. Activated sludge wastewater treatment plant modelling and simulation: state of the art[J]. Environmental Modelling & Software, 2004, 19(9): 763-783. |
| [26] | Datta T, Racz L A, Kotay S M, et al. Seasonal variations of nitrifying community in trickling filter-solids contact (TF/SC) activated sludge systems[J]. Bioresource Technology, 2011, 102(3): 2272-2279. DOI:10.1016/j.biortech.2010.10.043 |
| [27] | Li W, Gao J, Zhuang J L, et al. Metagenomics and metatranscriptomics uncover the microbial community associated with high S0 production in a denitrifying desulfurization granular sludge reactor[J]. Water Research, 2021, 203. DOI:10.1016/j.watres.2021.117505 |
| [28] | Ji X M, Wu Z Y, Sung S, et al. Metagenomics and metatranscriptomics analyses reveal oxygen detoxification and mixotrophic potentials of an enriched anammox culture in a continuous stirred-tank reactor[J]. Water Research, 2019, 166. DOI:10.1016/j.watres.2019.115039 |
| [29] | Rodríguez E, García-Encina P A, Stams A J M, et al. Meta-omics approaches to understand and improve wastewater treatment systems[J]. Reviews in Environmental Science and Bio/Technology, 2015, 14(3): 385-406. DOI:10.1007/s11157-015-9370-x |
| [30] | Dang H Y, Cupples A M. Identification of the phylotypes involved in cis-dichloroethene and 1, 4-dioxane biodegradation in soil microcosms[J]. Science of the Total Environment, 2021, 794. DOI:10.1016/j.scitotenv.2021.148690 |
| [31] | Buchfink B, Xie C, Huson D H. Fast and sensitive protein alignment using DIAMOND[J]. Nature Methods, 2015, 12(1): 59-60. DOI:10.1038/nmeth.3176 |
| [32] | Nadeau S A, Roco C A, Debenport S J, et al. Metagenomic analysis reveals distinct patterns of denitrification gene abundance across soil moisture, nitrate gradients[J]. Environmental Microbiology, 2019, 21(4): 1255-1266. DOI:10.1111/1462-2920.14587 |
| [33] | Parks D H, Imelfort M, Skennerton C T, et al. CheckM: assessing the quality of microbial genomes recovered from isolates, single cells, and metagenomes[J]. Genome Research, 2015, 25(7): 1043-1055. DOI:10.1101/gr.186072.114 |
| [34] | Menzel P, Ng K L, Krogh A. Fast and sensitive taxonomic classification for metagenomics with Kaiju[J]. Nature Communications, 2016, 7. DOI:10.1038/ncomms11257 |
| [35] | Wang Z, Zhang X X, Lu X, et al. Abundance and diversity of bacterial nitrifiers and denitrifiers and their functional genes in tannery wastewater treatment plants revealed by high-throughput sequencing[J]. PLoS One, 2014, 9(11). DOI:10.1371/journal.pone.0113603 |
| [36] | Guo J H, Ni B J, Han X Y, et al. Unraveling microbial structure and diversity of activated sludge in a full-scale simultaneous nitrogen and phosphorus removal plant using metagenomic sequencing[J]. Enzyme and Microbial Technology, 2017, 102: 16-25. DOI:10.1016/j.enzmictec.2017.03.009 |
| [37] |
马切切, 袁林江, 牛泽栋, 等. 活性污泥微生物群落结构及与环境因素响应关系分析[J]. 环境科学, 2021, 42(8): 3886-3893. Ma Q Q, Yuan L J, Niu Z D, et al. Microbial community structure of activated sludge and its response to environmental factors[J]. Environmental Science, 2021, 42(8): 3886-3893. |
| [38] | Jiang X, Ma M C, Li J, et al. Bacterial diversity of active sludge in wastewater treatment plant[J]. Earth Science Frontiers, 2008, 15(6): 163-168. |
| [39] | Anand V, Pandey A. Molecular biological techniques used in environmental engineering: current prospects and challenges[A]. In: Shah M P, Rodriguez-Couto S (Eds. ). Wastewater Treatment Reactors: Microbial Community Structure[M]. Amsterdam: Elsevier, 2021. 509-536. |
| [40] | Heider J, Fuchs G. Thauera[A]. In: Whitman W B (Ed. ). Bergey's Manual of Systematics of Archaea and Bacteria[M]. Hoboken: Wiley, 2015. |
| [41] | Ma Q, Qu Y Y, Zhang X W, et al. Identification of the microbial community composition and structure of coal-mine wastewater treatment plants[J]. Microbiological Research, 2015, 175: 1-5. |
| [42] | Ma Q, Qu Y Y, Shen W L, et al. Bacterial community compositions of coking wastewater treatment plants in steel industry revealed by Illumina high-throughput sequencing[J]. Bioresource Technology, 2015, 179: 436-443. |
| [43] | Fan X Y, Gao J F, Pan K L, et al. Temporal heterogeneity and temperature response of active ammonia-oxidizing microorganisms in winter in full-scale wastewater treatment plants[J]. Chemical Engineering Journal, 2019, 360: 1542-1552. |
| [44] | Siripong S, Rittmann B E. Diversity study of nitrifying bacteria in full-scale municipal wastewater treatment plants[J]. Water Research, 2007, 41(5): 1110-1120. |
| [45] | Limpiyakorn T, Shinohara Y, Kurisu F, et al. Communities of ammonia-oxidizing bacteria in activated sludge of various sewage treatment plants in Tokyo[J]. FEMS Microbiology Ecology, 2005, 54(2): 205-217. |
 2022, Vol. 43
2022, Vol. 43


