在过去的几十年里, 化肥使用量急剧增加, 旨在提高农业产量以满足日益增长的粮食需求[1].尽管带来了巨大的农业效益, 然而越来越多的研究发现长期过量投入造成的不利影响和环境风险直接导致现代农业不可持续, 全球农业正面临着前所未有的威胁[2, 3].作为世界最大的农业生产国之一, 中国同样面临着严峻的农业环境问题.为此, 中国政府于2015年决定实施化肥减施行动, 以缓解紧迫的农业环境问题并保障粮食安全和农业可持续发展[4].
为实现农业可持续发展, 必须降低化肥用量[5], 并通过添加有机物料补充养分[6].秸秆和有机肥作为主推的有机物料能够维持和提升作物产量, 有利于健康土壤的培育[7], 归因于其改善了土壤养分循环, 提升了土壤肥力[8~10], 同时提高了土壤物理结构稳定性, 形成了健康的土壤微环境[11].目前, 已有研究主要关注有机添加对土壤理化性质、酶活性、微生物群落和作物产量的影响[12~15], 而对土壤N循环变化却缺乏了解.
N是作物必需的常量元素, 决定着农业生产[16].土壤N循环是由固氮、氨氧化和反硝化等多个相互关联过程构成的闭合网络, 由包括固氮细菌(nitrogen-fixing bacteria)、氨氧化微生物(ammonia-oxidizing microorganisms)和反硝化微生物(denitrifying microorganisms)在内的多种微生物所驱动[17].施肥措施变化所致的N投入改变将直接影响N循环微生物, 功能基因标志物的应用为研究这些N循环微生物如何响应施肥措施变化提供了技术方法. nifH基因用于表征固定大气N2的固氮细菌, chiA基因用于表征分解有机N的细菌[18], 这两种微生物能够提升土壤中生物有效N含量.氨氧化是硝化作用的第一步也是限速步骤, 主要由氨氧化细菌和古菌(ammonia-oxidizing bacteria and archaea, AOB和AOA)驱动, 其生物标志物为amoA基因[19].亚硝酸盐还原为NO的过程主要由nirK和nirS型反硝化细菌介导[20], 随后NO被还原成N2O, 成为气态N损失和温室气体增加的主要根源[21].近期研究发现含nirK基因的真菌在土壤反硝化过程中同样发挥着关键作用[22~24].含nosZ基因的细菌能够将N2O还原为N2, 其系统发育包括nosZⅠ和nosZⅡ共2个进化枝, 对于减少农田土壤N2O排放至关重要[25].
鉴于N循环功能微生物的重要性, 本研究通过田间定位试验探究小麦-玉米轮作制度下化肥减量结合有机物料添加对土壤微生物N循环功能基因丰度和N转化遗传潜力的影响, 并明确其与土壤性质变化的关系.笔者假设:①由于化肥施用量减少和有机物料投入, 与N获取相关的chiA和nifH基因丰度增加, 进而提升N存储潜力; ②amoA基因丰度因有效N减少和能量获取需求减弱而降低, 导致NO3-淋溶潜力下降; ③由于营异养生活, 细菌和真菌nirK基因以及细菌nirS基因丰度均因有机物料的添加而增加, 将增强气态N排放潜力, 同样营异养生活的N2O还原细菌的nosZ基因丰度也将有所提升.本研究以期为化肥减量结合有机物料添加对土壤微生物N循环策略的潜在生态学影响提供新的见解, 并且为集约化种植制度下施肥措施合理调整和农业可持续发展提供技术支撑.
1 材料与方法 1.1 研究区概况与试验设计本研究布设于农业农村部环境保护科研监测所试验站, 该试验站坐落于天津市宁河区(117°43′E, 39°28′N), 属于典型的温带大陆性季风气候, 土壤类型为潮土, 耕作方式为小麦-玉米轮作.本试验始于2015年10月, 共包括5种处理:①常规施肥(NPK), 同时作为对照; ②化肥减量(NPKR); ③化肥减量配施秸秆(NPKRS); ④化肥减量配施有机肥(NPKRO); ⑤化肥减量配施秸秆和有机肥(NPKROS).各处理(22 m×22 m)按照随机区组设计布设, 由于小区面积较大能够反映耕作措施的影响, 因此未设置重复.在小麦季和玉米季, 化肥施用总量均为200 kg·hm-2(以N计)、187.5 kg·hm-2(以P2O5计)和63 kg·hm-2(以K2O计), 而减量后施用总量均为140 kg·hm-2(以N计)、131.25 kg·hm-2(以P2O5计)和44.1 kg·hm-2(以K2O计).所施用的化肥为无机复合肥、尿素、过磷酸钙和氯化钾. 60%的氮肥(无机复合肥)以及全量磷肥和钾肥作为基肥于小麦和玉米播种时施入, 而40%的氮肥(尿素)作为追肥于小麦的苗期和玉米的拔节期施入.按照当地常规管理方式, 所有处理中小麦秸秆全部还田, 那么NPKRS和NPKROS处理中的秸秆专指玉米秸秆, 于玉米收获后粉碎还田.有机肥仅在小麦季施入相应处理中, 施用量为15 t·hm-2.
2019年9月, 在玉米收获前采集土壤样品.在每个小区中, 随机选择3个假重复样带[26].在每个假重复样带中, 使用直径5 cm的土壤采样器按照5点采样法采集5个土芯(0~20 cm), 随后混合成一个复合样品.所有土壤样品置于低温保藏箱中, 迅速运送至实验室.将所有土壤样品均匀混合并过2 mm筛以去除可见的石块、作物残体和动物残骸等杂质.用于分子生物学分析的子样品于-80℃冰箱中保存.本研究所采用的化学性质分析方法见文献[14], 其中用土壤有机碳(soil organic carbon, SOC)代替土壤有机质(soil organic matter, SOM), 相关试验数据见文献[14]和表 1.
|
|
表 1 不同施肥措施下土壤化学性质1) Table 1 Soil chemical properties under different fertilization practices |
1.2 土壤基因组DNA提取和实时定量PCR分析
使用DNeasy PowerSoil Isolation Kit(Qiagen, Hilden, USA)从0.25 g冻存土壤样品中提取土壤基因组DNA, 按照操作说明进行.通过1.0%(10 mg·mL-1)琼脂糖凝胶电泳验证是否成功提取DNA, 并使用NanoDrop ND-2000 spectrophotometer(NanoDrop Technologies, Thermo Scientific, MA, USA)检测DNA的纯度和浓度.在分子生物学分析前, 所有DNA样品均保存于-80℃冰箱中.
使用CFX 96 touch实时定量PCR系统(Bio-Rad, Hercules, CA, USA)测定N功能基因丰度. 25 μL的PCR反应体系包括12.5 μL的TB GreenTM Premix Ex TaqTM(TaKaRa, Japan), 0.5 μL的每种引物(10 μmol·L-1)(除真菌nirK基因测定中加入1.25 μL外), 0.05 μL的T4 gene 32 protein(10 ng·μL-1)(Roche, Laval, Quebec, Canada)和1.0 μL的10倍稀释的DNA模板, 最后加入相应体积的灭菌去离子水.用于实时定量PCR分析的引物集和热反应程序见表 2.试验过程中设置阴性对照以判定是否存在环境污染.使用10倍连续稀释的含有靶标基因片段的质粒绘制标准曲线.样品、标准质粒和阴性对照均设置3次重复.在延伸步骤收集荧光数据.为证实扩增的特异性, 在qPCR程序的最后进行熔解曲线分析.靶标基因的扩增效率范围为82.5% ~92.5%, R2值范围为0.998~1.000.
|
|
表 2 实时定量PCR的引物集和热循环程序 Table 2 Primer sets and thermal procedures for real-time quantitative PCR |
1.3 统计分析
为满足方差齐性假设, 将功能基因拷贝数进行对数转换.采用N循环功能基因的相对丰度评价微生物N转化中的能量投入, 用每种基因的丰度对基因总丰度的百分比表示[35].考虑到真菌在土壤反硝化作用中的重要地位, 本研究中加入了真菌nirK基因丰度分析.(chiA+nifH)/(AOA amoA+AOB amoA)、(AOA amoA+AOB amoA)/(细菌nirK+真菌nirK+nirS)和(nosZⅠ+nosZⅡ)/(细菌nirK+真菌nirK+nirS)的丰度比分别用于评价土壤微生物的N存储潜力[36, 37]、NO3-淋溶潜力[36]和N2O还原潜力(本研究).此外, 采用(细菌nirK+真菌nirK+nirS) 的总丰度评价土壤微生物的气态N排放潜力(本研究).采用单因素方差分析(one-way analysis of variance, ANOVA)并结合Duncan多重比较分析5种施肥处理间基因丰度的差异显著性, P<0.05时差异显著.采用Pearson相关分析评价N循环功能基因丰度和N转化遗传潜力与化学性质间的相关性.根据N循环功能基因丰度, 采用基于距离的冗余分析(distance-based redundancy analysis, db-RDA)评价施肥处理间的关系, 并挖掘关键驱动因子, 采用Canoco 5.0 for Windows软件完成(Microcomputer Power, Inc., Ithaca. NY).使用Rstudio中“vegan”包的adonis功能进行置换多元方差分析.
2 结果与分析 2.1 N循环功能基因丰度除AOA和AOB amoA基因外, 施肥措施变化对N循环功能基因丰度影响显著(图 1, P<0.05).对于chiA基因丰度, NPKROS处理最高, NPK处理最低, 并且NPKROS处理显著高于其他处理(P<0.05).相似地, 施肥措施变化提高了nifH基因丰度, NPKROS处理最高, 并且NPKROS处理显著高于NPK和NPKRO处理(P<0.05).细菌nirK基因丰度因施肥措施改变而增加, 相比于NPK处理, NPKROS和NPKRO处理增幅显著(P<0.05).真菌nirK基因丰度整体上升, 最低值出现在NPKRS处理, 而非NPK处理, 最高值亦然出现在NPKROS处理. nirS基因丰度明显高于两种nirK基因, NPKROS和NPKR处理的nirS基因丰度较NPK处理显著增加(P<0.05). nosZⅠ和nosZⅡ基因丰度也因施肥措施变化而增加, 与NPK处理相比, NPKROS、NPKR和NPKRO处理的nosZⅠ基因丰度显著升高(P<0.05), NPKROS和NPKRO处理的nosZⅡ基因丰度增幅显著(P<0.05).
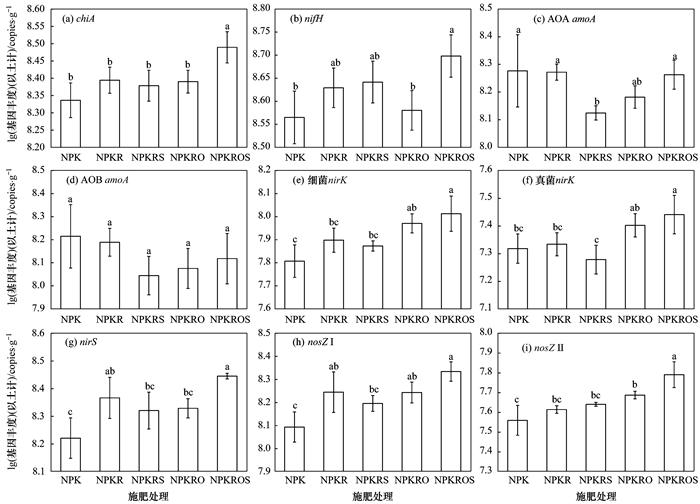
|
不同小写字母表示差异达到显著水平(P<0.05); NPK代表常规施肥, NPKR代表化肥减量, NPKRS代表化肥减量配施秸秆, NPKRO代表化肥减量配施有机肥, NPKROS代表化肥减量配施秸秆和有机肥, 下同 图 1 不同施肥措施下土壤微生物N循环功能基因丰度 Fig. 1 Abundances of soil microbial N-cycling functional genes under different fertilization practices |
由图 2可见, chiA基因的相对丰度(约占基因总丰度的16%)未发生显著变化. nifH基因丰度在基因总丰度中的占比超过25%, 其中NPKRS处理的nifH基因比例显著高于其他处理(P<0.05).从整体上看, 施肥措施变化后AOA和AOB amoA基因的相对丰度均显著降低.与NPK处理相比, 4种施肥处理中细菌nirK基因的相对丰度均增加, 其中NPKRO和NPKROS处理增幅显著(P<0.05).所有处理的真菌nirK基因占比均低于2%. nirS和nosZⅠ基因的相对丰度均因施肥措施改变而显著增加(P<0.05).对于nosZⅡ基因, NPKRO和NPKROS处理的相对丰度较高, 分别为NPK处理的1.24倍和1.26倍.
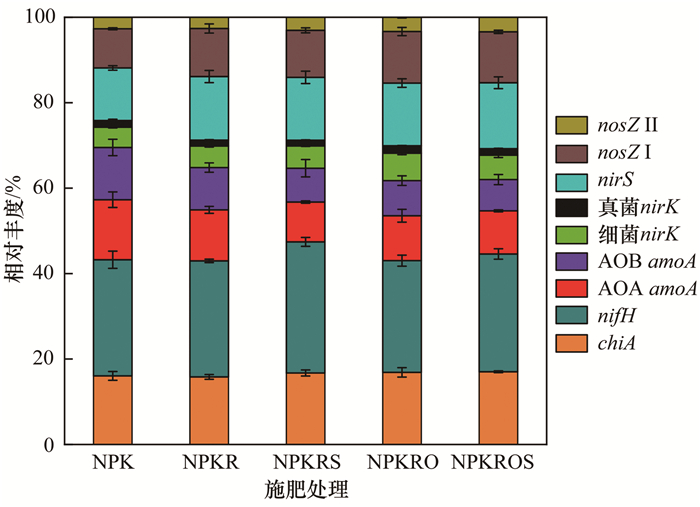
|
图 2 不同施肥措施下土壤微生物N循环功能基因的相对丰度 Fig. 2 Relative abundances of soil microbial N-cycling functional genes under different fertilization practices |
微生物N存储潜力因有机物料的添加而显著增加[图 3(a)], 其中NPKRO、NPKROS和NPKRS处理分别为NPK处理的1.38、1.54和1.67倍. NO3-淋溶潜力因施肥措施变化而显著降低, 添加有机物料处理的数值几乎为NPK处理的一半[图 3(b)].与N存储潜力相似, 气态N排放潜力因施肥措施变化而增加, 其中NPKROS、NPKR和NPKRO处理与NPK处理间差异显著[P<0.05, 图 3(c)].此外, 施肥措施变化也提高了N2O还原潜力, 但差异并不显著[P>0.05, 图 3(d)].
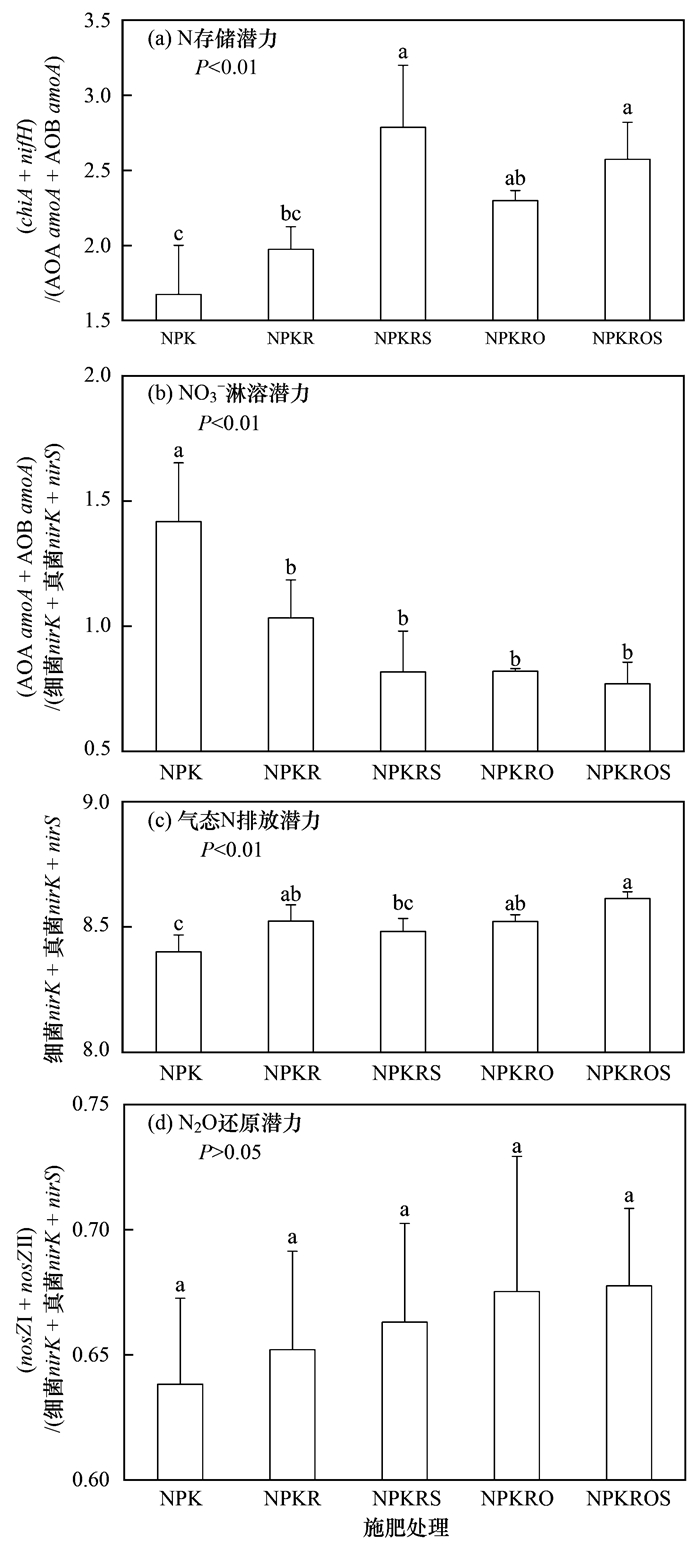
|
不同小写字母表示差异达到显著水平(P<0.05) 图 3 不同施肥措施下土壤微生物N转化遗传潜力 Fig. 3 Soil microbial N-converting genetic potentials under different fertilization practices |
如db-RDA排序所示(图 4), 前两轴共计解释约83.59%的N循环功能基因丰度总变异.PERMANOVA(permutational multivariate analysis of variance, R2=0.645, P=0.002)表明5种施肥处理间差异显著.在所选择的7种土壤化学性质中, NH4+是影响N循环功能基因丰度的决定因素[图 4(a)].对于N循环功能基因丰度, 添加有机肥的处理(NPKRO和NPKROS处理)的分布表现出明显的相似性.同时, 基于样品分布与N循环功能基因丰度的关系[图 4(b)], 有机肥投入有利于除AOA和AOB amoA基因外的N循环功能基因丰度, 而秸秆的加入增强了这种影响.
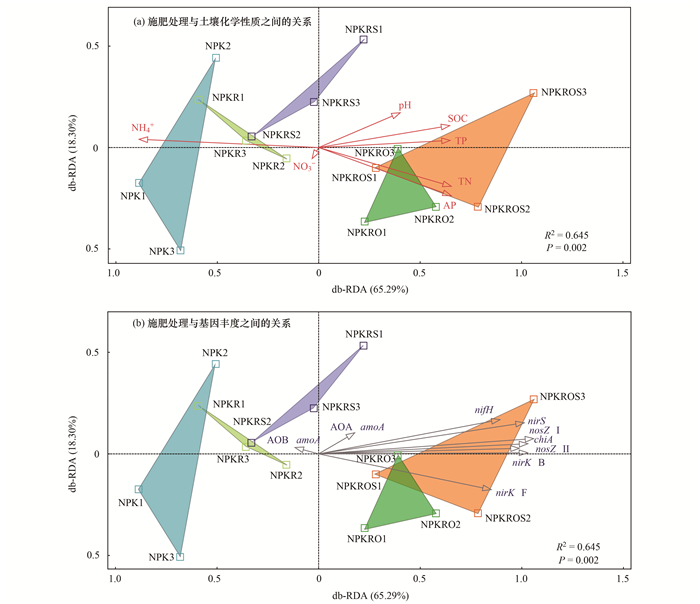
|
图 4 基于N循环功能基因丰度的db-RDA排序 Fig. 4 Distance-based redundancy analysis (db-RDA) according to the abundances of N-cycling genes |
Pearson相关性分析表明(表 3), chiA基因丰度与NH4+显著负相关(P<0.05). AOB amoA基因丰度与TN显著负相关(P<0.05).此外, 细菌nirK基因拷贝数与SOC、AP和TN间均表现出显著正相关关系(P<0.05).同样, 真菌nirK基因丰度与AP显著正相关(P<0.05). nosZⅡ基因丰度与SOC、AP和TN显著正相关(P<0.05), 而与NH4+显著负相关(P<0.01).然而, 未见nifH、AOA amoA、nirS和nosZⅠ基因拷贝数与任何化学指标呈现显著相关关系.N存储潜力与NO3-淋溶潜力(P<0.01)和NH4+(P<0.05)间均呈现显著负相关关系. NO3-淋溶潜力与气态N排放潜力(P<0.05)、SOC(P<0.05)和TN(P<0.01)间均显著负相关, 而与NH4+(P<0.05)显著正相关.气态N排放潜力与NH4+显著负相关(P<0.05).然而, N2O还原潜力与所有化学性质间均无显著相关关系.
|
|
表 3 N循环功能基因丰度和N转化遗传潜力与化学性质间的Pearson相关性分析1) Table 3 Relationships of N-cycling functional gene abundances and N-converting genetic potentials with soil chemical properties as revealed by Pearson correlation analysis |
3 讨论 3.1 有机N分解和N固定表征的土壤微生物N存储
与第一个假设一致, 相比于NPK处理, 施肥措施发生变化的处理中chiA基因丰度增加.含chiA基因的细菌能够以几丁质作为C和N源[38].有机物料带来的大量C和N源刺激了分解几丁质细菌的生长和繁殖.出乎意料的是仅化肥减量配施秸秆和有机肥的处理(NPKROS处理)中, chiA基因丰度显著高于对照(NPK处理).细菌分解几丁质在有氧和缺氧条件下均可以发生, 而有氧条件更为有利[38].在农田中, 几丁质分解主要发生在充气的表土中[39].秸秆还田形成的富氧微环境能够促进几丁质分解细菌, 然而秸秆中相对较少的底物类型可能会限制几丁质分解者.有机肥因其含有丰富且易分解的底物同样会激发几丁质分解细菌, 但仅添加有机肥无法形成与秸秆还田相似的通风条件.同时添加秸秆和有机肥能够形成底物类型多样和富氧条件的微区, 几丁质分解细菌快速繁殖.仅降低化肥施用量同样增加了几丁质分解细菌的丰度, 可能与化肥对微生物的抑制作用减弱有关.
固氮细菌通过生物N固定将大气N转变为生物可利用N, 此过程是农田土壤有效N的第二大来源[40].与几丁质分解细菌相似, N固定细菌数量也因施肥措施变化而增加, 并且在NPKROS处理中增幅显著.化肥施用量降低直接导致生物有效N减少, 一方面, 可能会减弱化学施肥的不利影响, 另一方面, 可能会引发作物与微生物间的N竞争, 最终表现在N固定细菌的N捕获过程得到增强[41].有研究报道有机物料单独或与化肥联合施用均有利于N固定细菌[42], 归因于其对有机物质和能量的依赖[29], 因此土壤中较高的有机质含量有利于固氮细菌, 这与本研究的结果相一致.与同时添加有机肥和秸秆的处理(NPKROS处理)相比, 单独添加有机肥(NPKRO处理)却显著降低了固氮菌细丰度, 可能是由于有机肥富含的有机N矿化后补充了无机N, 而富C的秸秆的加入改变了C和N平衡, 增强了N素利用, 激发固氮群落增殖以执行N固定.此外, 土壤通透性的改善也会提供充足的N2, 但固氮菌通过何种机制保证固氮酶免受同时引入的O2的抑制作用有待深入研究.
N循环微生物倾向于降低N存储并增加N损失以应对N富集, 这是通过调控富N环境中N功能群丰度实现的[36], 反之亦然.综合以上研究结果, 由于有效N投入降低和有机C投入增加, 参与有机N分解和N固定的微生物数量增加, 土壤N存储潜力显著增强, 土壤向N自给的方向发展.
3.2 氨氧化主导的土壤NO3-淋溶损失化能自养型氨氧化微生物群落(如AOA和AOB)的丰度、组成和结构受土壤N有效性的强烈影响[43, 44].本研究中降低化肥施用量直接导致用作电子供体和能量来源的无机N底物减少[45].与常规施肥措施相比, 仅降低化肥用量表现出对AOA和AOB的轻微抑制作用, 说明在这两种施肥措施下N底物供应是相当的. 然而, 有机物料的添加表现出明显的抑制效果, 这不同于Xue等[46]的研究结果.对于AOA, 化肥减量配施秸秆处理(NPKRS处理)中丰度最低, 可能归因于富C贫N秸秆所引发的无机N源被大量利用.有机肥本身含有一定数量的无机N, 而且其富含的有机N在微生物N矿化下生成无机N, 同时秸秆添加提升了有氧条件和土壤C含量, 进一步刺激有机N分解, 进而产生更多的可用于硝化作用的N底物.AOB表现出相似的丰度变化, 尽管处理间差异并不显著, 产生这种结果的原因可能与AOA相似.尽管有机N分解和N固定增强, 但在作物和其他微生物利用后, 剩余的可为氨氧化微生物直接利用的N可能是不足的.此外, 氨氧化微生物通过氧化氨获得电子和能量以固定大气CO2用于合成有机化合物[19], 有机物料的添加可能会改变微生物对有机C的需求, 异养型微生物因有机物料的投入而快速增殖, 压缩了AOA和AOB的生存空间.
总之, 在化肥减量的基础上添加有机物料降低了氨氧化微生物的丰度, 暗示由氨向下转化直接供应NO3-能力的减弱, 同时结合产N2O微生物丰度的有机C升高, 表明可能在遗传水平上直接缓解NO3-淋溶损失, 说明该措施的实施可能会减轻硝态氮淋溶进入地下水的环境风险.
3.3 反硝化介导的土壤气态N损失和N2O还原与硝化作用紧密关联的反硝化作用是陆地生态系统中主要的N2O排放途径之一[47], 其2个关键步骤(NO2-和N2O还原)通常被用于研究气态N损失和N2O汇[48, 49].正如所预期的, 参与NO2-还原为NO的2种细菌亚硝酸还原酶基因的丰度因施肥措施变化而增加, 并且NPKROS处理下丰度最高. nirK和nirS型反硝化细菌均为严格的异养菌, 其通过分解有机化合物获取能量用于生长和繁殖[50], 因此, 与常规施肥措施相比, 有机物料投入增加了C底物并对这2种细菌产生积极影响.值得注意的是, 仅实施化肥减量的处理(NPKR处理)同样增加了这2种细菌数量, 可能是因为NPKR处理中AOA和AOB丰度较高, 其为细菌N2O生产者提供可直接利用的无机N源, 并且与减弱的化学施肥不利影响有关.此外, 在所有处理中nirS基因拷贝数均高于nirK基因, 这与Zhao等[49]的研究结果一致, 其研究表明nirS型N2O生产者在农田土壤中占据数量优势.与细菌N2O生产者相似, 施肥措施变化同样提高了真菌nirK基因数量, 除NPKRS处理的丰度略低于NPK处理.化肥减量配施秸秆下真菌nirK基因丰度的降低可能与相对较少的有机底物类型和有限的无机N源有关.此外, 与细菌反硝化微生物相似, 化肥减量配施有机肥和秸秆对真菌反硝化菌群最为有利.
本研究中进化枝Ⅰ和Ⅱ的nosZ基因丰度均因有机物料添加而增加, 并与产N2O微生物表现出相似的生态偏好.作为异养型微生物, N2O还原细菌同样需要有机物质作为能量源和电子供体[51], 因此有机物料投入有利于2个进化枝的生长和繁殖.与NPK处理相比, 仅实施化肥减量的处理同样表现出较高的丰度, 可能是由于在连续还原过程中产N2O微生物产生了更多可以直接利用的电子受体, 另一个原因可能是化肥减量缓解了对土壤微生物的不利影响. 2种进化枝丰度间的比较表明进化枝Ⅰ在数量上高于进化枝Ⅱ, 与此前在农田中进行的研究一致[49].然而, 据报道nosZⅡ型N2O还原微生物决定了N2O排放潜力[49], 说明数量优势并不一定代表高生物化学活性[52], 并且本研究中是否也存在这种现象需要进一步探索.
N2O生产者和消费者间的平衡一定程度上决定了土壤的净N2O排放[53].尽管气态N排放潜力的增强意味着会产生更多的N2O, 但N2O还原微生物数量同样增加(N2O还原潜力增加), 则N2O被还原的量增加.尽管如此, 必须认识到虽然反硝化作用是兼性厌氧过程[54], 但由于其偏爱厌氧环境[55], 本研究所采用的参数可能无法全面反映与N2O产生和消耗有关的真实环境, 但当遇到灌溉、降雨事件, 甚至是土壤含水量较高的微环境时, 所获得的结果为预测这种潜力提供了有价值的信息.值得注意的是硝化微生物的反硝化作用被证实是另一个重要的N2O排放途径[45], 仅采用细菌nirK+真菌nirK+细菌nirS丰度总和可能低估了真实的气态N排放潜力, 因此必须进行长期监测以证实气态N排放和N2O还原潜力的可应用性.
3.4 N循环功能微生物的能量投入策略和潜在驱动因素本研究中, 除营自养生活的氨氧化微生物外, 其他微生物均为异养型.相比于常规施肥, 施肥措施的变化, 尤其是有机物料添加, 有利于异养菌群, 反而抑制了自养菌群, 说明微生物能量投入与平衡策略因能量、资源和养分组成变化而发生改变[36].由于C源增加, C利用和有效N供应中的能量投入相应增加, 而能量获取途径中的投入则会降低, 以调节C/N化学计量平衡.由于固氮细菌和亚硝酸盐还原微生物偏爱富含有机C的土壤环境[21, 56], 因此参与耗能的N供应和还原过程的微生物比例整体上升. nifH基因在数量上占主导地位, 说明本研究的土壤具有极大的生物N固定遗传潜力.
db-RDA分析表明5种施肥处理间差异显著, 说明施肥措施强烈影响N循环功能基因丰度, NH4+在此过程中发挥关键作用.NPKRO和NPKROS处理间关系紧密, 均对异养型N循环微生物数量产生积极影响, 说明有机肥可能是潜在的积极因素, 并且秸秆的加入起到促进作用.N存储和NO3-淋溶潜力与NH4+分别呈显著负相关和正相关关系, 表明化肥施用不利于土壤N存储并且极易引发N淋溶, 较低的气态N排放能力同样会增加NO3-淋溶, 可能与反硝化作用受到抑制有关.
4 结论化肥减量结合有机物料投入对异养N循环微生物产生了积极的影响, 而对自养N循环微生物造成了不利影响.N转化过程的能量投入和多数N循环微生物的生态位因施肥措施变化而发生改变.施肥措施变化提高了微生物N存储、气态N排放和N2O还原潜力, 而降低NO3-淋溶潜力.与化肥投入密切相关的NH4+在驱动N功能微生物丰度变化中发挥关键作用.总之, 化肥减量耦合有机物料投入具有促进土壤N存储, 抑制淋溶所致的N损失, 并缓解因N2O排放所致的环境风险的潜力, 暗示可以从N循环功能基因丰度及N转化遗传潜力角度揭示健康和可持续农业土壤培育的生物学机制.
| [1] | Galloway J N, Townsend A R, Erisman J W, et al. Transformation of the nitrogen cycle: recent trends, questions, and potential solutions[J]. Science, 2008, 320(5878): 889-892. DOI:10.1126/science.1136674 |
| [2] | Guo J H, Liu X J, Zhang Y, et al. Significant acidification in major Chinese croplands[J]. Science, 2010, 327(5968): 1008-1010. DOI:10.1126/science.1182570 |
| [3] | Raza S, Miao N, Wang P Z, et al. Dramatic loss of inorganic carbon by nitrogen-induced soil acidification in Chinese croplands[J]. Global Change Biology, 2020, 26(6): 3738-3751. DOI:10.1111/gcb.15101 |
| [4] | 农业部种植业管理司. 农业部020年化肥使用量零增长行动方案》和《到2020年农药使用量零增长行动方案》的通知[EB/OL]. http://www.zzys.moa.gov.cn/gzdt/201503/t20150318_6309945.htm, 2015-03-18. |
| [5] | Ju X T, Xing G X, Chen X P, et al. Reducing environmental risk by improving N management in intensive Chinese agricultural systems[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(9): 3041-3046. DOI:10.1073/pnas.0813417106 |
| [6] | Bai H Z, Tao F L. Sustainable intensification options to improve yield potential and eco-efficiency for rice-wheat rotation system in China[J]. Field Crops Research, 2017, 211: 89-105. DOI:10.1016/j.fcr.2017.06.010 |
| [7] | Sun R B, Zhang X X, Guo X S, et al. Bacterial diversity in soils subjected to long-term chemical fertilization can be more stably maintained with the addition of livestock manure than wheat straw[J]. Soil Biology and Biochemistry, 2015, 88: 9-18. DOI:10.1016/j.soilbio.2015.05.007 |
| [8] | Steiner C, Teixeira W G, Lehmann J, et al. Long term effects of manure, charcoal and mineral fertilization on crop production and fertility on a highly weathered Central Amazonian upland soil[J]. Plant and Soil, 2007, 291(1-2): 275-290. DOI:10.1007/s11104-007-9193-9 |
| [9] | Zhu H H, Wu J S, Huang D Y, et al. Improving fertility and productivity of a highly-weathered upland soil in subtropical China by incorporating rice straw[J]. Plant and Soil, 2010, 331(1-2): 427-437. DOI:10.1007/s11104-009-0263-z |
| [10] | Zhao H L, Ning P, Chen Y L, et al. Effect of straw amendment modes on soil organic carbon, nitrogen sequestration and crop yield on the North-Central Plain of China[J]. Soil Use and Management, 2019, 35(3): 511-525. DOI:10.1111/sum.12482 |
| [11] | Lin Y X, Ye G P, Kuzyakov Y, et al. Long-term manure application increases soil organic matter and aggregation, and alters microbial community structure and keystone taxa[J]. Soil Biology and Biochemistry, 2019, 134: 187-196. DOI:10.1016/j.soilbio.2019.03.030 |
| [12] |
靳玉婷, 李先藩, 蔡影, 等. 秸秆还田配施化肥对稻-油轮作土壤酶活性及微生物群落结构的影响[J]. 环境科学, 2021, 42(8): 3985-3996. Jin Y T, Li X F, Cai Y, et al. Effects of straw returning with chemical fertilizer on soil enzyme activities and microbial community structure in rice-rape rotation[J]. Environmental Science, 2021, 42(8): 3985-3996. |
| [13] | Zhu J, Peng H, Ji X H, et al. Effects of reduced inorganic fertilization and rice straw recovery on soil enzyme activities and bacterial community in double-rice paddy soils[J]. European Journal of Soil Biology, 2019, 94. DOI:10.1016/j.ejsobi.2019.103116 |
| [14] | Wu X, Zhang T, Zhao J N, et al. Variation of soil bacterial and fungal communities from fluvo-aquic soil under chemical fertilizer reduction combined with organic materials in North China Plain[J]. Journal of Soil Science and Plant Nutrition, 2021, 21(1): 349-363. DOI:10.1007/s42729-020-00365-0 |
| [15] | Chen A L, Zhang W Z, Sheng R, et al. Long-term partial replacement of mineral fertilizer with in situ crop residues ensures continued rice yields and soil fertility: a case study of a 27-year field experiment in subtropical China[J]. Science of the Total Environment, 2021, 787. DOI:10.1016/j.scitotenv.2021.147523 |
| [16] | Thomas R Q, Brookshire E N J, Gerber S. Nitrogen limitation on land: how can it occur in Earth system models?[J]. Global Change Biology, 2015, 21(5): 1777-1793. DOI:10.1111/gcb.12813 |
| [17] | Kuypers M M M, Marchant H K, Kartal B. The microbial nitrogen-cycling network[J]. Nature Reviews Microbiology, 2018, 16(5): 263-276. DOI:10.1038/nrmicro.2018.9 |
| [18] | Geisseler D, Horwath W R, Joergensen R G, et al. Pathways of nitrogen utilization by soil microorganisms? A review[J]. Soil Biology and Biochemistry, 2010, 42(12): 2058-2067. DOI:10.1016/j.soilbio.2010.08.021 |
| [19] | Lehtovirta-Morley L E. Ammonia oxidation: ecology, physiology, biochemistry and why they must all come together[J]. FEMS Microbiology Letters, 2018, 365(9). DOI:10.1093/femsle/fny058 |
| [20] | Zumft W G. Cell biology and molecular basis of denitrification[J]. Microbiology and Molecular Biology Reviews, 1997, 61(4): 533-616. |
| [21] | Levy-Booth D J, Prescott C E, Grayston S J. Microbial functional genes involved in nitrogen fixation, nitrification and denitrification in forest ecosystems[J]. Soil Biology and Biochemistry, 2014, 75: 11-25. DOI:10.1016/j.soilbio.2014.03.021 |
| [22] | Ma S T, Shan J, Yan X Y. N2O emissions dominated by fungi in an intensively managed vegetable field converted from wheat-rice rotation[J]. Applied Soil Ecology, 2017, 116: 23-29. DOI:10.1016/j.apsoil.2017.03.021 |
| [23] | Xu H F, Sheng R, Xing X Y, et al. Characterization of fungal nirK-containing communities and N2O emission from fungal denitrification in arable soils[J]. Frontiers in Microbiology, 2019, 10. DOI:10.3389/fmicb.2019.00117 |
| [24] | Aldossari N, Ishii S. Fungal denitrification revisited - Recent advancements and future opportunities[J]. Soil Biology and Biochemistry, 2021, 157. DOI:10.1016/j.soilbio.2021.108250 |
| [25] | Jones C M, Graf D R, Bru D, et al. The unaccounted yet abundant nitrous oxide-reducing microbial community: a potential nitrous oxide sink[J]. The ISME Journal, 2013, 7(2): 417-426. DOI:10.1038/ismej.2012.125 |
| [26] | Merloti L F, Mendes L W, Pedrinho A, et al. Forest-to-agriculture conversion in Amazon drives soil microbial communities and N-cycle[J]. Soil Biology and Biochemistry, 2019, 137. DOI:10.1016/j.soilbio.2019.107567 |
| [27] | Xiao X, Yin X B, Lin J, et al. Chitinase genes in lake sediments of Ardley Island, Antarctica[J]. Applied and Environmental Microbiology, 2005, 71(12): 7904-7909. DOI:10.1128/AEM.71.12.7904-7909.2005 |
| [28] | Huang L N, Tang F Z, Song Y S, et al. Biodiversity, abundance, and activity of nitrogen-fixing bacteria during primary succession on a copper mine tailings[J]. FEMS Microbiology Ecology, 2011, 78(3): 439-450. DOI:10.1111/j.1574-6941.2011.01178.x |
| [29] | Francis C A, Roberts K J, Beman J M, et al. Ubiquity and diversity of ammonia-oxidizing archaea in water columns and sediments of the ocean[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(41): 14683-14688. DOI:10.1073/pnas.0506625102 |
| [30] | Rotthauwe J H, Witzel K P, Liesack W. The ammonia monooxygenase structural gene amoA as a functional marker: molecular fine-scale analysis of natural ammonia-oxidizing populations[J]. Applied and Environmental Microbiology, 1997, 63(12): 4704-4712. DOI:10.1128/aem.63.12.4704-4712.1997 |
| [31] | Hallin S, Lindgren P E. PCR detection of genes encoding nitrite reductase in denitrifying bacteria[J]. Applied and Environmental Microbiology, 1999, 65(4): 1652-1657. DOI:10.1128/AEM.65.4.1652-1657.1999 |
| [32] | Wei W, Isobe K, Shiratori Y, et al. Development of PCR primers targeting fungal nirK to study fungal denitrification in the environment[J]. Soil Biology and Biochemistry, 2015, 81: 282-286. DOI:10.1016/j.soilbio.2014.11.026 |
| [33] | Su M X, Kleineidam K, Schloter M. Influence of different litter quality on the abundance of genes involved in nitrification and denitrification after freezing and thawing of an arable soil[J]. Biology and Fertility of Soils, 2010, 46(5): 537-541. DOI:10.1007/s00374-010-0449-9 |
| [34] | Melero S, Pérez-de-Mora A, Murillo J M, et al. Denitrification in a vertisol under long-term tillage and no-tillage management in dryland agricultural systems: key genes and potential rates[J]. Applied Soil Ecology, 2011, 47(3): 221-225. DOI:10.1016/j.apsoil.2010.12.003 |
| [35] | Stone M M, Kan J J, Plante A F. Parent material and vegetation influence bacterial community structure and nitrogen functional genes along deep tropical soil profiles at the Luquillo Critical Zone Observatory[J]. Soil Biology and Biochemistry, 2015, 80: 273-282. DOI:10.1016/j.soilbio.2014.10.019 |
| [36] | Tang Y Q, Yu G R, Zhang X Y, et al. Changes in nitrogen-cycling microbial communities with depth in temperate and subtropical forest soils[J]. Applied Soil Ecology, 2018, 124: 218-228. DOI:10.1016/j.apsoil.2017.10.029 |
| [37] | Song Z L, Wang J, Liu G B, et al. Changes in nitrogen functional genes in soil profiles of grassland under long-term grazing prohibition in a semiarid area[J]. Science of the Total Environment, 2019, 673: 92-101. DOI:10.1016/j.scitotenv.2019.04.026 |
| [38] | Wieczorek A S, Schmidt O, Chatzinotas A, et al. Ecological functions of agricultural soil bacteria and microeukaryotes in chitin degradation: a case study[J]. Frontiers in Microbiology, 2019, 10. DOI:10.3389/fmicb.2019.01293 |
| [39] | Fernandez C W, Koide R T. The role of chitin in the decomposition of ectomycorrhizal fungal litter[J]. Ecology, 2012, 93(1): 24-28. DOI:10.1890/11-1346.1 |
| [40] | Ollivier J, Töwe S, Bannert A, et al. Nitrogen turnover in soil and global change[J]. FEMS Microbiology Ecology, 2011, 78(1): 3-16. DOI:10.1111/j.1574-6941.2011.01165.x |
| [41] | Vitousek P M, Menge D N L, Reed S C, et al. Biological nitrogen fixation: rates, patterns and ecological controls in terrestrial ecosystems[J]. Philosophical Transactions of the Royal Society B: Biological Sciences, 2013, 368(1621). DOI:10.1098/rstb.2013.0119 |
| [42] | Liao H K, Li Y Y, Yao H Y. Fertilization with inorganic and organic nutrients changes diazotroph community composition and N-fixation rates[J]. Journal of Soils and Sediments, 2018, 18(3): 1076-1086. DOI:10.1007/s11368-017-1836-8 |
| [43] | Zhang Y, Chen L J, Dai T J, et al. Ammonia manipulates the ammonia-oxidizing archaea and bacteria in the coastal sediment-water microcosms[J]. Applied Microbiology and Biotechnology, 2015, 99(15): 6481-6491. DOI:10.1007/s00253-015-6524-2 |
| [44] | Norman J S, Barrett J E. Substrate availability drives spatial patterns in richness of ammonia-oxidizing bacteria and archaea in temperate forest soils[J]. Soil Biology and Biochemistry, 2016, 94: 169-172. DOI:10.1016/j.soilbio.2015.11.015 |
| [45] | Wrage-Mönnig N, Horn M A, Well R, et al. The role of nitrifier denitrification in the production of nitrous oxide revisited[J]. Soil Biology and Biochemistry, 2018, 123: A3-A16. DOI:10.1016/j.soilbio.2018.03.020 |
| [46] | Xue C, Zhang X, Zhu C, et al. Quantitative and compositional responses of ammonia-oxidizing archaea and bacteria to long-term field fertilization[J]. Scientific Reports, 2016, 6. DOI:10.1038/srep28981 |
| [47] | Butterbach-Bahl K, Baggs E M, Dannenmann M, et al. Nitrous oxide emissions from soils: how well do we understand the processes and their controls?[J]. Philosophical Transactions of the Royal Society B: Biological Sciences, 2013, 368(1621). DOI:10.1098/rstb.2013.0122 |
| [48] | Li D D, Zhang X Y, Green S M, et al. Nitrogen functional gene activity in soil profiles under progressive vegetative recovery after abandonment of agriculture at the Puding Karst Critical Zone Observatory, SW China[J]. Soil Biology and Biochemistry, 2018, 125: 93-102. DOI:10.1016/j.soilbio.2018.07.004 |
| [49] | Zhao S Y, Zhou J M, Yuan D D, et al. NirS-type N2O-producers and nosZ Ⅱ-type N2O-reducers determine the N2O emission potential in farmland rhizosphere soils[J]. Journal of Soils and Sediments, 2020, 20(1): 461-471. DOI:10.1007/s11368-019-02395-3 |
| [50] | Zhang Y, Cai Z C, Zhang J B, et al. C: N ratio is not a reliable predictor of N2O production in acidic soils after a 30-day artificial manipulation[J]. Science of the Total Environment, 2020, 725. DOI:10.1016/j.scitotenv.2020.138427 |
| [51] | Groffman P M. Terrestrial denitrification: challenges and opportunities[J]. Ecological Processes, 2012, 1(1). DOI:10.1186/2192-1709-1-11 |
| [52] | Liu Z B, Zhang C J, Wei Q Y, et al. Temperature and salinity drive comammox community composition in mangrove ecosystems across southeastern China[J]. Science of the Total Environment, 2020, 742. DOI:10.1016/j.scitotenv.2020.140456 |
| [53] | Domeignoz-Horta L A, Spor A, Bru D, et al. The diversity of the N2O reducers matters for the N2O∶N2 denitrification end-product ratio across an annual and a perennial cropping system[J]. Frontiers in Microbiology, 2015, 6. DOI:10.3389/fmicb.2015.00971 |
| [54] | Liu B B, Zhang X J, Bakken L R, et al. Rapid succession of actively transcribing denitrifier populations in agricultural soil during an anoxic spell[J]. Frontiers in Microbiology, 2019, 9. DOI:10.3389/fmicb.2018.03208 |
| [55] | Philippot L, Hallin S, Schloter M. Ecology of denitrifying prokaryotes in agricultural soil[J]. Advances in Agronomy, 2007, 96: 249-305. |
| [56] | Chen Z, Luo X Q, Hu R G, et al. Impact of long-term fertilization on the composition of denitrifier communities based on nitrite reductase analyses in a paddy soil[J]. Microbial Ecology, 2010, 60(4): 850-861. DOI:10.1007/s00248-010-9700-z |
 2022, Vol. 43
2022, Vol. 43


