总氮提标改造工程的微生物群落结构分析
李海松,
王柯丹,
陈晓蕾,
阎登科,
许子聪,
胡培基
环境科学  2022, Vol. 43 2022, Vol. 43 Issue (9): 4727-4735 Issue (9): 4727-4735 |
|
市政WWTPs普遍使用活性污泥法进行污染物的去除, 活性污泥作为一种独特的人工微生物生态系统, 具有较高微生物多样性.微生物与碳(C)、氮(N)、硫(S)和磷(P)的转化有着密不可分的关系, 是污水生物处理系统实现污染物去除和稳定运行的基础[1].随着高通量技术的发展, WWTP活性污泥中微生物的群落结构研究已经成为热点[2~4].有研究报道:市政WWTPs普遍存在的微生物有Proteobacteria、Bacteroidetes、Actinobacteria、Firmicutes、Planctomycetes和Chloroflexi等[5~7].然而对市政WWTPs活性污泥的研究更关注多个污水厂甚至是全球的好氧池微生物群落结构及影响因素[8, 9], 对同一污水处理厂的不同工段活性污泥微生物群落结构间的差异关注较少, 尤其是特殊环境下的微生物群落结构.随着我国对生态环保问题的日益重视, 污水排放标准日趋严格, 多数污水处理厂面临提标改造.在整个提标改造过程中, 总氮(TN)是短板.为了满足高要求的排放标准, 研究各种方式降低TN的出水浓度[10~12], 河南省某一市政WWTP在二沉池后端加设深度脱氮装置进行反硝化, 实现ρ(TN)<1.5 mg ·L-1.此工况下的微生物群落结构尚未研究, 且同一污水厂不同工段的微生物群落结构是否存在差异尚不清楚, 尤其是在将TN从15 mg ·L-1降低至1.5 mg ·L-1的环境下, 微生物的组成是否存在演替, 多样性分布及功能是否存在差异也需研究.
因此, 本文基于河南省郑州市五龙口市政污水处理厂的TN提标改造工程, 研究原有氧化沟工艺及新增设的深度脱氮装置中活性污泥的微生物群落结构.分析常规生化工段及深度脱氮特殊环境下微生物多样性的分布、影响因素、物种的组成和优势菌群等; 进行组间比较, 研究不同工段之间微生物群落结构的差异, 寻找各工段中的生物标识; 通过共线性网络模型研究促使微生物群落结构差异性的原因, 以及环境参数与微生物间相互作用; 通过PICRUSt(phylogenetic investigation of communities by reconstruction of unobserved states)对功能基因进行预测, 分析两工段微生物在功能上存在的差异.将微生物组学与工艺选择、参数优化和生物强化结合, 以期为污水处理厂提供更完善的活性污泥微生物信息.
1 材料与方法 1.1 样本采集及指标检测对河南省郑州市五龙口WWTP的WLK_OD(氧化沟)及WLK_AD(深度脱氮装置)工段中的活性污泥进行采集.郑州市五龙口WWTP的日处理量10万m3·d-1, 采用氧化沟工艺进行生化处理.其出水经过二沉池后满足一级A排放标准. 2017~2019年, 在氧化沟出水处采集的样本分别为WLK_OD1、WLK_OD2、WLK_OD3和WLK_OD4.在二沉池后新增设深度脱氮装置, 该装置主要是进行硝态氮的去除, 其日处理量3万m3 ·d-1. 2018年3月16日, 对WLK_AD内不同高度、不同时间(白天和晚上)的活性污泥进行采集, 分别命名为WLK_AD1、WLK_AD2、WLK_AD3、WLK_AD4、WLK_AD5、WLK_AD6、WLK_AD7和WLK_AD8.样本采集后进行-20℃保存, 并在一周内进行送样测序.取样点如图 1所示.pH值用pH计(PHS-3E)检测, COD采用HJ828-2017中重铬酸钾法测定, 氨氮(NH4+-N)、硝态氮(NO3--N)和TN采用分光光度法测定.
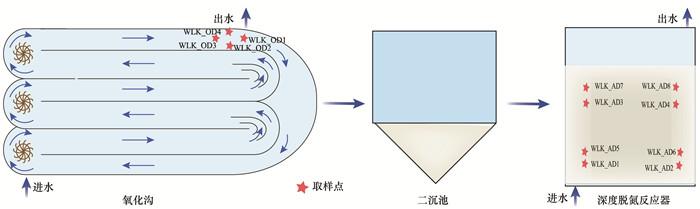
|
图 1 WLK_OD和WLK_AD两工段活性污泥取样点示意 Fig. 1 Schematic diagram of activated sludge collected from WLK_OD and WLK_AD |
将采集的样本直接送往生工生物工程(上海)股份有限公司进行DNA的提取, PCR扩增以及高通量测序.提取DNA采用E.Z.N.ATM Mag-Bind Soil DNA Kit的试剂盒, 并按说明书提供的步骤对WWTP采集的所有样本进行细菌DNA提取.之后利用Qubit3.0 DNA检测试剂盒对基因组DNA精确定量, 对细菌16S rDNA中的V3-V4高变区进行PCR扩增, 引物序列为341F和805R, 利用Illumina MiSeq PE300平台进行测序分析.
1.3 微生物数据分析生物信息分析依照扩增子数据分析指南[13, 14].分别对序列进行双端合并、质控和去重复.然后用USEARCH v10.0的-unoise命令将非冗余序列去噪为扩增序列变体(OTUs), 并生成特征表.基于RDP训练集v16对特征序列(OTUs)进行分类学并注释.在R语言v4.0.2环境下, 多样性分析使用vegan v2.5-6包, 数据可视化采用ggplot2 v3.3.2包.主坐标分析(PCoA)用Bray-Curtis距离评估, 微生物共现网络图通过Gephi 0.9.2将微生物之间的相关性结果可视化.使用线性判别分析影响(LEfSe)、曼哈顿图和火山图进行组间比较.环境参数与微生物之间的冗余分析结果通过Canoco5进行可视化.此外, 通过PICRUSt对活性污泥中的微生物基于KEGG (Kyoto Encyclopedia of Genes and Genomes)数据库进行功能预测, 其结果通过Stamp软件进行可视化.
2 结果与讨论 2.1 污水处理厂深度脱氮反应器脱氮性能对采集样本的水质指标进行检测, 结果如表 1所示.在生化段, TN的去除率为63%左右, NH4+-N降低到0.5 mg ·L-1以下, 其去除率高达98%, 而TN基本上以NO3--N的形式存在.经过深度脱氮处理后, NO3--N的去除率高达98%, 总氮降低到1.5 mg ·L-1以下, 其去除率达到90%以上, 主要进行NO3--N的去除.两工段活性污泥中微生物的生态位不同, 脱氮性能也存在差异, 其原因可归于微生物群落结构的不同.
|
|
表 1 所有样本的物理化学指标 Table 1 Physical chemistry parameters of samples |
为了进一步分析两工段脱氮性能的差异, 进行WLK_OD与WLK_AD的微生物多样性分析, 如图 2所示.分别用Richness指数和Shannon指数解释微生物群落的丰度和多样性[图 2(a)和2(b)], 结果表明WLK_AD的Richness指数均值为993, 比WLK_OD(940)高; 但其Shannon指数均值为5.18低于WLK_AD(5.78).推断WLK_AD微生物群落具有较低的均匀性, 归因于特定的脱氮环境对微生物进行自然选择, 形成了独特的菌群.图 2(c)为两工段微生物的稀释曲线, 测序深度均达到85%时, 微生物的丰度趋于平衡, 表明测序结果具有可靠性.图 2(d)展示了PCoA的结果, WLK_AD的样本聚集在一起; 尽管WLK_OD样本相对分散, 但是在PCoA1轴比较接近.此外, 两工段的样本在主坐标轴1分离(P<0.01).在PCoA中两个样本越接近, 两个样本的微生物组成越相似[15].结果表明各工段中微生物群落结构相似, 两工段间有显著差异, 进一步推测不同的水质环境对微生物群落结构具有影响.
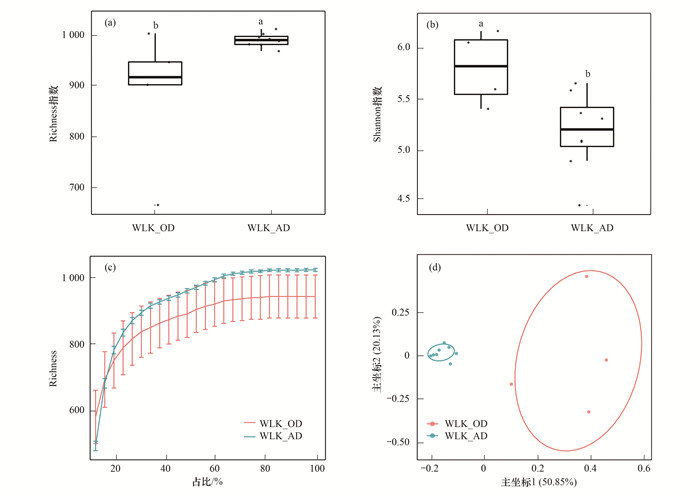
|
不同小写字母表示两组间有显著差异 图 2 不同工段样本的多样性分析、稀释曲线分析和主坐标分析 Fig. 2 Diversity, rarefaction curve and PCoA of samples in WLK_OD and WLK_AD |
该WWTP的微生物共检测到1 244个OTUs, 被注释分配到27个门, 245个属.在门水平: Proteobacteria(变形菌门)、Bacteroidetes(拟杆菌门)、Actinobacteria(放线菌门)、Firmicutes(厚壁菌门)、Planctomycetes(浮霉菌门)、Chloroflexi(绿弯菌门)、Parcubacteria(帕库菌门)和Verrucomicrobia(疣微菌门)在两个工段中均作为优势菌(相对丰度>1%), 如图 3所示.此结果与已有的研究结果一致[1, 2, 6~9].Proteobacteria在WLK_AD中富集, 相对丰度为70.11%, 比WLK_OD增加了近25%.Proteobacteria包含有多种好氧-厌氧-兼性细菌, 可降解有机污染物, 去除氮磷, 易适应微生态环境的变化.为了进一步分析该微生物在两工段间的差异, 图 3中显示了其纲水平的5种微生物, 发现β-Proteobacteria从12.06%增加到40.13%.有研究报道β-Proteobacteria的大部分细菌都可以参与氮的代谢[12].此外, 从WLK_OD到WLK_AD, Candidatus_Saccharibacteria和Acidobacteria(酸杆菌门)的丰度分别由3.58%和2.29%降到0.50%和0.69%.有研究报道Candidatus_Saccharibacteria中的大部分细菌可以代谢多种碳源, 如蛋白胨、葡萄糖和乙酸钠等[16].然而WLK_AD的进水为二沉池出水, 其中有机物大部分为难降解物, pH值相对较高, 因此不适合深度脱氮环境的Candidatus_Saccharibacteria及酸杆菌被淘汰.
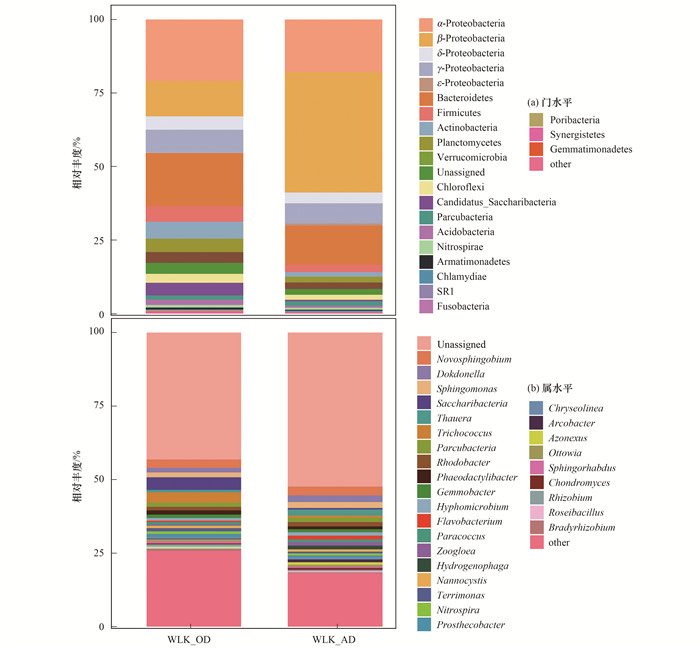
|
图 3 不同工段活性污泥微生物在门水平和属水平组成及分布 Fig. 3 Bacterial composition of activated sludge from different reactors at phylum and genus levels |
在属水平, 不同工段中检测到的共有优势菌(相对丰度>1%)分别为Novosphingobium、Dokdonella、Sphingomonas(鞘氨醇单胞菌)、Parcubacteria、Rhodobacter(红杆菌)和Gemmobacter (巨大芽殖杆菌).然而WLK_OD中的功能反硝化菌Trichococcus(3.50%)和Terrimonas(1.10%)在WLK_AD中的相对丰度分别降到0.70%和0.59%, Thauera(2.04%)、Flavobacterium(1.27%)、Hydrogenophaga(1.26%)和Zoogloea(1.18%)分别是WLK_AD中的优势反硝化菌群[17~22], 它们在WLK_OD中的相对丰度分别是0.81%, 0.50%, 0.050%和0.28%.有文献报道Thauera、Hydrogenophaga和Zoogloea可以利用难降解物质为碳源进行反硝化[15].结果表明在WLK_AD中反硝化菌群发生演替, 富集了独特的反硝化功能菌群.原因可归于不同工段微生物的生态位和生长动力学不同.WLK_OD的微生物经历了厌氧, 缺氧和好氧环境, 脱氮过程涉及到氨化、硝化和反硝化; 而WLK_AD的微生物处于缺氧环境, 主要进行硝态氮的去除.此外, 相较于WLK_OD, WLK_AD处理的污水生化性较低, 且含氮污染物浓度较低, 微生物处在寡营养环境, 独特的优势反硝化菌可利用难降解物质为碳源实现富集; WLK_OD中富集的反硝化菌可利用易生物降解有机物实现快速生长从而成为优势菌.
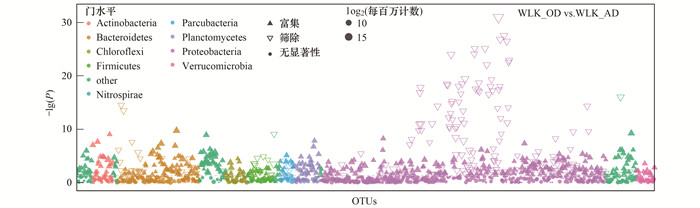
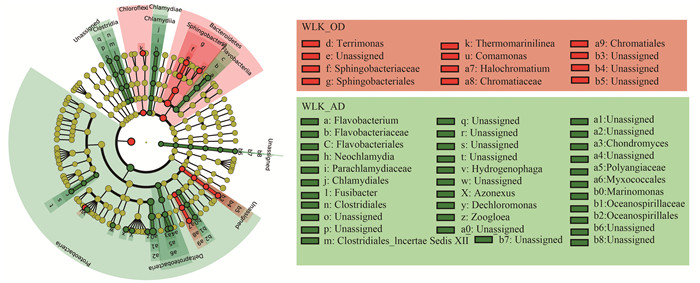
2.4 组间比较火山图展示了所有OTUs在两工段的差异性(图 4), 其中红色/绿色分别表示显著富集/筛除的差异OTUs.通过组间比较, 具有显著差异OTUs总数量为622, 在WLK_AD中富集的OTUs数量201(P<0.05).曼哈顿图的结果(图 5)表明, 在WLK_AD中富集的具有差异的OUTs是Proteobacteria, 且这些OTUs的差异性更大[-lg(P)>20)].统计学检验结果进一步证明两个工段间的微生物组成具有显著差异.此外, LEfSe的结果进一步展示了两个工段中具有显著差异的微生物(生物标识).生物标识是指在区别不同组具有显著性差异的微生物中发挥很重要作用[9].在WLK_AD中演替的优势反硝化菌Flavobacterium、Hydrogenophaga和Zoogloea具有显著性差异(图 6), 对体系内高脱氮效率发挥很重要的作用.此外一些非优势菌(如:Thermomarinilinea、Comamonas和Dechloromonas等)作为生物标识被检测到, 且与工段内的大部分优势菌呈正相关关系(图 7), 表明这些微生物在各工段内可以协同优势菌共同去除污染物.
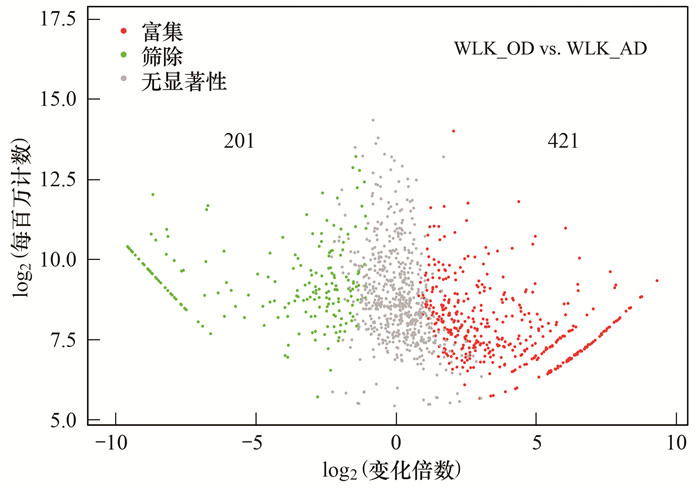
|
图 4 不同工段的火山图分析 Fig. 4 Volcano analysis between WLK_OD and WLK_AD |
|
图 5 不同工段的曼哈顿分析 Fig. 5 Manhattan analysis between WLK_OD and WLK_AD |
|
图 6 不同工段微生物的LEfSe分析 Fig. 6 LEfSe analysis between WLK_OD and WLK_AD |
|
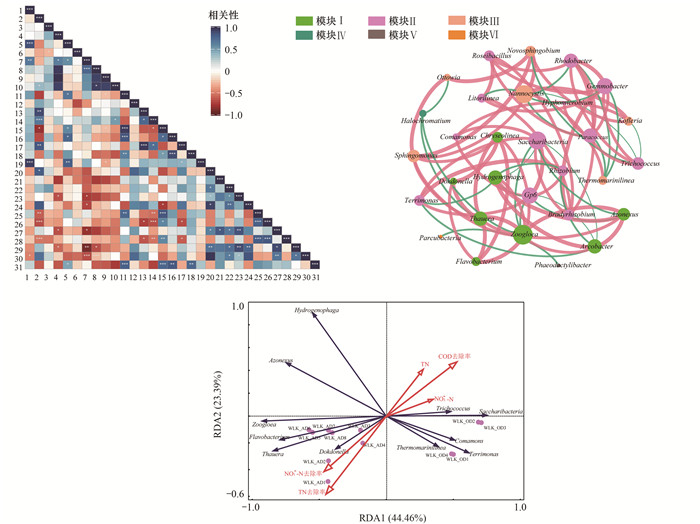
1.Novosphingobium, 2. Saccharibacteria, 3. Phaeodactylibacter, 4. Thermomarinilinea, 5. Sphingomonas, 6. Parcubacteria, 7. Nannocystis, 8. Kofleria, 9. Halochromatium, 10. Comamonas, 11. Thauera, 12. Nitrospira, 13. Terrimonas, 14. Gp6, 15. Zoogloea, 16. Chryseolinea, 17. Rhizobium, 16. Dokdonella, 19. Ottowia, 20. Bradyrhizobium, 21. Hyphomicrobium, 22. Roseibacillus, 23. Rhodobacter, 24. Litorilinea, 25. Azonexus, 26. Arcobacter, 27. Paracoccus, 28. Hydrogenophaga, 29. Gemmobacter, 30. Trichococcus, 31. Flavobacterium; ***表示P<0.001, **表示P<0.01, *表示P<0.05; 蓝色箭头表示微生物, 红色箭头表示环境因子, 圆圈表示样本 图 7 微生物的相互关系、共现网络分析和冗余分析 Fig. 7 Interrelationships, co-occurrence network, and RDA of microorganisms |
为了研究促使五龙口WWTP不同工段的微生物群落结构差异性的原因, 进一步分析微生物间的相关性、共现性以及与环境因子之间的相互作用.图 7展示了31种菌属的共线性模式(Spearman's系数>0.6且P<0.05), 共有80条边(红色是正相关, 绿色是负相关); 边的粗细与相关性强度成正比.经过模块化分析, 微生物主要被分成3个模块.模块Ⅰ中的微生物主要是WLK_AD中的优势菌, 可以利用难降解有机物为碳源进行反硝化[17, 23, 24]; 模块Ⅱ中的微生物主要是WLK_OD中优势菌, 多为生活WWTPs常见的异养菌, 可以代谢多种易生物降解有机物及营养元素[19, 20, 25~28]; 模块Ⅲ中的微生物在两个工段中的相对丰度波动较小, 普遍存在于各种环境中.结果表明不同的水质环境影响微生物群落结构.
对群落结构影响最大的环境因子是COD和TN的去除率.COD的去除率、TN和NO3--N浓度之间呈正相关, 并与Saccharibacteria、Thermomarinilinea、Terrimonas和Comamonas密切相关[17].TN的去除率与NO3--N的去除率呈正相关, 它们与Dokdonella、Thauera、Flavobacterium和Zoogloea密切相关[29].此外, WLK_OD和WLK_AD的样本分别坐落在第二、三象限.结合表 1的结果, 由于两工段的微生物的生态位不同, WLK_OD的微生物快速利用污水中可降解有机物及较高浓度的营养物质进行硝化反硝化, 实现脱氮除碳; 而WLK_AD的微生物在寡营养环境下主要进行NO3--N的去除, 最终实现出水ρ(TN)<1.5 mg ·L-1.
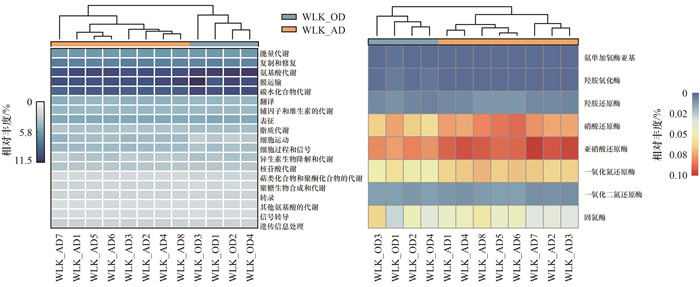
2.6 微生物功能预测图 8展示了所有样本基于KEGG数据库的功能信息, 热图上部分展示的聚类结果表明两个工段微生物在功能作用上存在差异.在WWTP中均富集的功能是氨基酸代谢、碳水化合物代谢和膜运输.相比较于WLK_OD, 细胞运动性能在WLK_AD中丰度更高.此外, 编码氨单加氧酶的基因在WLK_OD中具有更高的丰度, 主要进行硝化反应; 而编码硝酸还原酶、亚硝酸还原酶和一氧化氮还原酶的基因在WLK_AD中具有更高的相对丰度, 进行反硝化实现较高的总氮去除率.这与WLK_AD中存在的独特优势反硝化菌携带的基因有关.
|
图 8 所有样本中的微生物功能代谢途径以及与氮代谢相关的功能热图分析 Fig. 8 Heatmap analysis of metabolic function pathway and nitrogen metabolism function from all samples |
(1) 相较于WLK_OD, WLK_AD中的微生物具有较低的均匀性, 展现其微生物多样性分布的特异性.
(2) 两个工段优势的反硝化菌群发生演替, WLK_OD中是Trichococcus和Terrimonas; 在WLK_AD中为Thauera、Flavobacterium、Hydrogenophaga和Zoogloea.
(3) 导致两工段微生物群落结构差异的原因是不同的水质环境, 且COD和TN的去除率对该WWTP微生物群落影响最大.
(4) 两个工段的微生物在功能上也展现差异.WLK_AD中编码硝酸还原酶、亚硝酸还原酶和一氧化氮还原酶的基因丰度更高, 使ρ(TN)<1.5 mg ·L-1, 并实现较高的总氮去除率.
| [1] |
贺赟, 李魁晓, 王佳伟, 等. 不同季节城市污水处理厂微生物群落特性[J]. 环境科学, 2021, 42(3): 1488-1495. He Y, Li K X, Wang J W, et al. Microbial community structure of waste water treatment plants in different seasons[J]. Environmental Science, 2021, 42(3): 1488-1495. |
| [2] | Luo Y S, Yao J Q, Wang X Y, et al. Efficient municipal wastewater treatment by oxidation ditch process at low temperature: bacterial community structure in activated sludge[J]. Science of the Total Environment, 2020, 703. DOI:10.1016/j.scitotenv.2019.135031 |
| [3] | Xue J, Schmitz B W, Caton K, et al. Assessing the spatial and temporal variability of bacterial communities in two Bardenpho wastewater treatment systems via Illumina MiSeq sequencing[J]. Science of the Total Environment, 2019, 657: 1543-1552. DOI:10.1016/j.scitotenv.2018.12.141 |
| [4] | Wu L W, Ning D L, Zhang B, et al. Global diversity and biogeography of bacterial communities in wastewater treatment plants[J]. Nature Microbiology, 2019, 4(7): 1183-1195. DOI:10.1038/s41564-019-0426-5 |
| [5] | Nelson M B, Martiny A C, Martiny J B H. Global biogeography of microbial nitrogen-cycling traits in soil[J]. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(29): 8033-8040. DOI:10.1073/pnas.1601070113 |
| [6] |
鞠峰, 张彤. 活性污泥微生物群落宏组学研究进展[J]. 微生物学通报, 2019, 46(8): 2038-2052. Ju F, Zhang T. Advances in meta-omics research on activated sludge microbial community[J]. Microbiology China, 2019, 46(8): 2038-2052. |
| [7] | Zhang L, Shen Z, Fang W K, et al. Composition of bacterial communities in municipal wastewater treatment plant[J]. Science of the Total Environment, 2019, 689: 1181-1191. DOI:10.1016/j.scitotenv.2019.06.432 |
| [8] |
马切切, 袁林江, 牛泽栋, 等. 活性污泥微生物群落结构及与环境因素响应关系分析[J]. 环境科学, 2021, 42(8): 3886-3893. Ma Q Q, Yuan L J, Niu Z D, et al. Microbial community structure of activated sludge and its response to environmental factors[J]. Environmental Science, 2021, 42(8): 3886-3893. |
| [9] |
张冰, 吴林蔚, 文湘华. 全国城市污水处理厂中微生物群落的溯源分析[J]. 环境科学, 2019, 40(8): 3769-3705. Zhang B, Wu L W, Wen X H. Potential source environments for microbial communities in wastewater treatment plants (WWTPs) in China[J]. Environmental Science, 2019, 40(8): 3769-3705. |
| [10] | Chen Y F, Shao Z Y, Kong Z, et al. Study of pyrite based autotrophic denitrification system for low-carbon source stormwater treatment[J]. Journal of Water Process Engineering, 2020, 37. DOI:10.1016/j.jwpe.2020.101414 |
| [11] | Zhu Z H, Zhao Y C, Guo Y Y, et al. A novel additional carbon source derived from rotten fruits: application for the denitrification from mature landfill leachate and evaluation the economic benefits[J]. Bioresource Technology, 2021, 334. DOI:10.1016/j.biortech.2021.125244 |
| [12] | Xu Z C, Chen X L, Li H S, et al. Combined heterotrophic and autotrophic system for advanced denitrification of municipal secondary effluent in full-scale plant and bacterial community analysis[J]. Science of the Total Environment, 2020, 717. DOI:10.1016/j.scitotenv.2020.136981 |
| [13] | Liu Y X, Qin Y, Chen T, et al. A practical guide to amplicon and metagenomic analysis of microbiome data[J]. Protein & Cell, 2021, 12(5): 315-330. |
| [14] |
刘永鑫, 秦媛, 郭晓璇, 等. 微生物组数据分析方法与应用[J]. 遗传, 2019, 41(9): 845-862. Liu Y X, Qin Y, Guo X X, et al. Methods and applications for microbiome data analysis[J]. Hereditas, 2019, 41(9): 845-862. |
| [15] | Zheng M S, Zhou N, Liu S F, et al. N2 O and NO emission from a biological aerated filter treating coking wastewater: main source and microbial community[J]. Journal of Cleaner Production, 2019, 213: 365-374. DOI:10.1016/j.jclepro.2018.12.182 |
| [16] |
马思佳, 顾卓江, 丁丽丽, 等. 碳源对活性污泥微生物细胞膜特性和群落结构影响[J]. 微生物学通报, 2017, 44(3): 561-573. Ma S J, Gu Z J, Ding L L, et al. Effects of carbon sources on cell membrane properties and microbial community of activated sludge[J]. Microbiology China, 2017, 44(3): 561-573. |
| [17] |
唐义, 马邕文, 万金泉, 等. 外加固体缓释碳源的两段反硝化工艺脱氮性能[J]. 环境科学, 2021, 42(7): 3392-3399. Tang Y, Ma Y W, Wan J Q, et al. Two-stage denitrification process performance with solid slow-release carbon source[J]. Environmental Science, 2021, 42(7): 3392-3399. |
| [18] | Yang Y K, Wang L F, Xiang F, et al. Activated sludge microbial community and treatment performance of wastewater treatment plants in industrial and municipal zones[J]. International Journal of Environmental Research and Public Health, 2020, 17(2). DOI:10.3390/ijerph17020436 |
| [19] |
杨文焕, 石大钧, 张元, 等. 高原湖泊沉积物中反硝化微生物的群落特征——以包头南海湖为例[J]. 中国环境科学, 2020, 40(1): 431-438. Yang W H, Shi D J, Zhang Y, et al. Community characteristics of denitrifying microorganisms in plateau lake sediments-taking Nanhaihu lake as example[J]. China Environmental Science, 2020, 40(1): 431-438. DOI:10.3969/j.issn.1000-6923.2020.01.048 |
| [20] | Pishgar R, Dominic J A, Sheng Z Y, et al. Denitrification performance and microbial versatility in response to different selection pressures[J]. Bioresource Technology, 2019, 281: 72-83. DOI:10.1016/j.biortech.2019.02.061 |
| [21] | Zhao F Z, Ju F, Huang K L, et al. Comprehensive insights into the key components of bacterial assemblages in pharmaceutical wastewater treatment plants[J]. Science of the Total Environment, 2019, 651: 2148-2157. DOI:10.1016/j.scitotenv.2018.10.101 |
| [22] | Hamza R A, Zaghloul M S, Iorhemen O T, et al. Optimization of organics to nutrients (COD: N: P) ratio for aerobic granular sludge treating high-strength organic wastewater[J]. Science of the Total Environment, 2019, 650: 3168-3179. DOI:10.1016/j.scitotenv.2018.10.026 |
| [23] | Liu W L, Wu Y, Zhang S J, et al. Successful granulation and microbial differentiation of activated sludge in anaerobic/anoxic/aerobic (A2O) reactor with two-zone sedimentation tank treating municipal sewage[J]. Water Research, 2020, 178. DOI:10.1016/j.watres.2020.115825 |
| [24] | Banerjee S, Táncsics A, Tóth E, et al. Hydrogenophaga aromaticivorans sp. nov., isolated from a para-xylene-degrading enrichment culture, capable of degrading benzene, meta- and para-xylene[J]. International Journal of Systematic and Evolutionary Microbiology, 2021, 71(3). DOI:10.1099/ijsem.0.004743 |
| [25] | Kindaichi T, Yamaoka S, Uehara R, et al. Phylogenetic diversity and ecophysiology of Candidate phylum Saccharibacteria in activated sludge[J]. FEMS Microbiology Ecology, 2016, 92(6). DOI:10.1093/femsec/fiw078 |
| [26] | Wang X J, Wang W Q, Zhang J, et al. Dominance of Candidatus saccharibacteria in SBRs achieving partial denitrification: effects of sludge acclimating methods on microbial communities and nitrite accumulation[J]. RSC Advances, 2019, 9(20): 11263-11271. DOI:10.1039/C8RA09518C |
| [27] |
王薇, 蔡祖聪, 钟文辉, 等. 好氧反硝化菌的研究进展[J]. 应用生态学报, 2007, 18(11): 2618-2625. Wang W, Cai Z C, Zhong W H, et al. Research advances in aerobic denitrifiers[J]. Chinese Journal of Applied Ecology, 2007, 18(11): 2618-2625. |
| [28] | Parshina S N, Strepis N, Aalvink S, et al. Trichococcus shcherbakoviae sp. nov., isolated from a laboratory-scale anaerobic EGSB bioreactor operated at low temperature[J]. International Journal of Systematic and Evolutionary Microbiology, 2019, 69(2): 529-534. DOI:10.1099/ijsem.0.003193 |
| [29] |
池玉蕾, 石烜, 任童, 等. 溶解氧对低碳源城市污水处理系统脱氮性能与微生物群落的影响[J]. 环境科学, 2021, 42(9): 4374-4382. Chi Y L, Shi X, Ren T, et al. Effects of dissolved oxygen on nutrient removal performance and microbial community in low carbon/nitrogen municipal wastewater treatment process[J]. Environmental Science, 2021, 42(9): 4374-4382. |