抗生素抗性基因(antibiotic resistance genes, ARGs)是可使微生物在暴露于抗生素的条件下能够生长和繁殖的基因片段.由于其复杂的抗性产生机制, ARGs的传播扩散是一个多维度和多机制联合作用的过程[1].这一过程受多种因素的影响, 除抗生素残留外, 还包括重金属[2]、消毒剂及其副产物[3]、抗生素抗性菌[4]、可移动遗传元件[5, 6]以及环境参数(TN、TP、NH4+、有机物和pH等)[7, 8]等.其中, 抗生素和重金属是影响环境中ARGs传播重要的选择性压力.抗生素可以杀死或者抑制敏感细菌的生长, 使得少数耐药菌获得生存空间而大量繁殖, 是ARGs传播的直接压力源[9].重金属具有和抗生素相似的抗性产生机制, 可以通过共抗性、交叉抗性和共调控这3种方式对ARGs产生选择压力[10].目前抗生素和重金属对ARGs的影响机制及其环境赋存特征已有大量研究, 然而这些参数常常是在特定的时空背景下被单独评估, 目前只有少数研究同时量化了特定位点的环境介质中ARGs、抗生素和重金属的丰度, 且不同研究的相关结果并不一致, 缺乏汇总分析, 妨碍了对三者因果关系的识别、判断、量化和表征[11].
基于此, 本文系统收集和汇总了现有环境介质中抗生素、重金属含量和ARGs丰度的数据, 利用统计回归方法, 调查地表水、沉积物、废水和土壤等环境介质中ARGs的丰度同抗生素和重金属的含量是否存在相关关系, 以期为评估抗生素和重金属污染对抗生素耐药性传播的影响以及抗生素耐药性控制提供支撑.
1 材料与方法 1.1 文献检索策略在常用期刊数据库ISI Web of Science中, 以"resistance gene"和"distribution/prevalence/occurrence/hotspot/reservoir/contamination/pollution/character*/correlation/dissemination/accum-ulation/source/abundance/presence/metagenom*/qPCR/monitor/evaluation/quantification"为关键词对1990年1月1日到2020年1月1日公开发表的文献进行检索, 共检索出抗性基因相关文献5 213篇.
1.2 文献和数据纳入标准对检索文献的题目和摘要逐一进行阅读, 剔除实验设计、培养菌株ARGs检测和耐药机制研究等文章, 只纳入有关自然环境介质中ARGs检出情况的文献, 保留原始研究, 剔除综述或二次研究, 剔除重复报告, 共筛查出310篇相关文献.
经阅读全文后, 筛查出同时检测了ARGs丰度以及抗生素或重金属含量, 且在文献或补充材料中提供有一次或二次图表数据的文章共47篇, 其中8篇使用宏基因组检测ARGs丰度, 39篇使用实时定量基因扩增荧光检测方法(real-time fluorescent quantitative polymerase chain reaction, qPCR)检测ARGs丰度.qPCR是此前研究中ARGs定量检测的最常用方法, 只纳入使用qPCR方法的文章, 最终39篇文献的数据被收集和进一步处理.其中, 27篇文献的采样点位于中国, 另外12篇文献的采样点分布在美国、越南、瑞士和罗马尼亚等8个国家.
1.3 数据提取从所纳入文献中依次提取和整理以下信息: 采样年份、采样地区、环境基质类型、抗生素含量、重金属含量和ARGs丰度, 制成信息提取表.其中图表形式表示的数据使用WebPlotDigitizer 4.2软件提取.低于检测限或定量限的数据没有收集进行分析, 同一样本的重复数据收集平均值(优先选择)或中值.最后, 共收集和分析了465份环境样本的数据.对所纳入样本根据环境基质类型分类为地表水、废水、沉积物和土壤这4类.
1.4 数据转化地表水和废水中抗生素的浓度均转化到以ng ·L-1为单位, 沉积物和土壤中抗生素的含量(以dw计, 下同)均转化到以ng·g-1为单位.抗生素含量的计算见式(1):
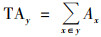
|
(1) |
式中, Ax表示样本中抗生素x的含量, TAy表示属于y类抗生素的抗生素x含量之和.抗生素按照解剖学、治疗学及化学分类(ATC)系统进行分类.
地表水和废水中重金属的浓度均转化到以μg·L-1为单位, 沉积物和土壤中重金属的含量均转化到以μg·g-1为单位.重金属含量的计算见式(2):
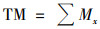
|
(2) |
式中, Mx表示样本中重金属x的含量, TM表示样本中重金属的总含量.
为消除生物量的影响, ARGs的绝对丰度使用16S rRNA基因的绝对丰度进行归一化, ARGs相对丰度的计算见式(3)和式(4):
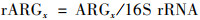
|
(3) |
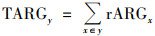
|
(4) |
式中, ARGx表示样本中抗生素抗性基因x的拷贝数, 16S rRNA是该样本中基因16S rRNA的拷贝数, rARGx表示该样本中抗性基因x的相对丰度.TARGy是对y类抗生素产生耐药性的所有抗性基因x相对丰度之和.根据McArthur等[12]的整理结果对应抗生素及相应的抗性基因.除抗性基因外, 整合子intl1、intl2和intl3作为ARGs动员和传播的重要媒介, 其丰度数据也被整理并对应所有种类的抗生素.
1.5 统计分析使用SPSS 18.0软件对环境介质中ARGs丰度、抗生素含量和重金属含量数据进行单变量和多变量线性回归分析.
2 结果与讨论 2.1 ARGs丰度与抗生素含量的相关性 2.1.1 总ARGs丰度与总抗生素含量的相关性本文共收集到同时检测抗生素含量和ARGs丰度的水相环境样本数据203个, 其中127个为地表水样品, 76个为废水样品, 固相环境样本数据有223个, 其中168个为沉积物样品, 55个为土壤样品(图 1).对总ARGs丰度和总抗生素含量数据的线性回归结果表明(表 1), 地表水、沉积物和土壤样本中总ARGs丰度与总抗生素含量之间均存在显著正相关关系(P<0.05), 随抗生素含量增加, ARGs丰度提高.这与大多数研究的结果一致, 一方面, 抗生素能够选择性地抑制敏感菌的生长, 使得少数耐药菌获得生存空间而大量繁殖, 加速ARGs的扩增[13].另一方面, 一些杀菌类的抗生素, 如β-内酰胺类(β-lactams, β-Ls)和氟喹诺酮类抗生素, 也可刺激细胞产生活性氧(reactive oxygen species, ROS), 激活SOS反应进而诱导耐药突变和ARGs重组, 促进ARGs的传播[14, 15].因此, 抗生素的使用和污染是影响ARGs传播的主要原因[16, 17].同时, 本研究中总ARGs丰度和总抗生素含量之间为对数相关关系, 说明ARGs增速随抗生素含量增加而不断降低.与此相似, Kohanski等[15]的研究发现与抑菌浓度的抗生素相比, 亚抑制浓度抗生素暴露条件下更有利于抗生素耐药菌的生长和富集, 因为亚抑制浓度环境中大多数种群都可存活, 可连续积累多个小的表型效应突变, 渐进性地选择抗生素耐药菌[14, 18].然而废水中没有发现总ARGs丰度与总抗生素残留浓度之间的显著相关关系(P<0.05), 可能是因为废水中抗生素和其他污染物的浓度和毒性水平较高, 主要表现为毒性选择作用, 抗生素的选择压力不再凸显, 因此高浓度抗生素污染废水中ARB和ARGs丰度与抗生素浓度的相关性较弱.
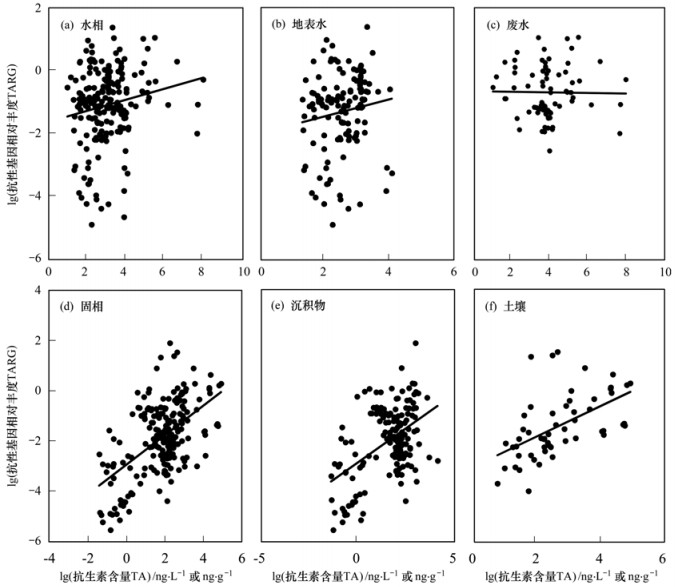
|
图 1 环境样本中总ARGs丰度和总抗生素含量 Fig. 1 Total ARGs and total antibiotics in environmental samples |
|
|
表 1 环境样本中总ARGs丰度与总抗生素含量线性回归结果1) Table 1 Linear regression results of total ARGs and total antibiotics in environmental samples |
虽然地表水、沉积物和土壤样本中总ARGs丰度与总抗生素含量均存在显著正相关关系, 但R2只有0.035~0.295, 抗生素含量只能解释一小部分ARGs丰度的方差, 大部分方差不能解释.抗生素和ARGs之间较差的相关性可以归因为以下几个因素: 首先, 抗生素和ARGs的环境命运不同.抗生素的半衰期较短, 在环境中容易发生降解, 而环境选择压力产生的ARGs却会在环境中持久存在, 难以消除[19].抗生素发生降解或者ARGs是由之前的环境压力选择产生并在种群中保存下来的, 都会影响相关分析结果.此外, 环境中还存在其他可能影响ARGs丰度的因素.例如, 有研究发现重金属[20]、消毒剂及其副产物[21]、杀菌剂季铵盐[22]、防腐剂三氯生[23]、纳米材料[24]和离子液体[25]等都可能会影响水平基因转移, 进而影响ARGs的传播.由于不同研究样本中物理化学因素的复杂性和差异性, 较低的R2不难被解释.同时, 固相样本的回归R2远大于水相环境样本的回归R2.一种可能的解释是因为水文条件的影响, 水相环境中生物地球化学因素随时间和采样地点变化存在较大的不稳定性, 而固相环境样本中的生物地球化学因素较为稳定, 因此水相环境样本测量结果存在更大比例不能解释的误差[26].
2.1.2 不同种类抗生素含量与配对ARGs丰度的相关性本文共收集到10大类抗生素及配对ARGs的数据, 其中四环素类(tetracyclines, TCs)、磺胺类(sulfonamides, SAs)、氟喹诺酮类(fluoroquinolones, FQs)和大环内酯类(macrolides, MLs)抗生素及配对ARGs(TRGs、SRGs、FRGs和MRGs)的数据占总样本数据量的95%.这4大类抗生素是常见的抗细菌感染药物, 被广泛用于人类和牲畜疾病的治疗和预防[27], 因此常通过动物排泄进入环境, 在环境中的检出率较高[28, 29].不同环境介质中4大类抗生素及配对ARGs的丰度如图 2所示.TCs和TRGs在地表水、沉积物和土壤中均有大量检出, 这与TCs在医疗、兽药和水产养殖中的广泛使用有关[30].SAs的使用量虽然相对较低, 但在环境中不易降解, 在各种环境中均有较高检出率.地表水中SAs和SRGs的平均水平和检出频率要高于沉积物, 因为SAs的水溶性较高, 对土壤和沉积物的吸附亲和力较低[28].MLs在地表水中也表现出比沉积物中更高的检出水平.然而, FQs和FRGs表现出相反趋势, 沉积物的检出水平更高, 这与FQs倾向于吸附沉积物和悬浮颗粒的趋势一致[31].
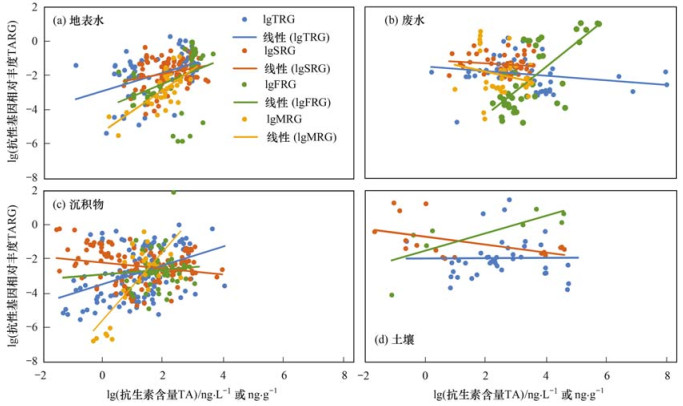
|
图 2 环境样本中不同种类抗生素的含量和配对ARGs的丰度 Fig. 2 Different classes of antibiotics and corresponding ARGs in environmental samples |
对不同种类抗生素及配对ARGs的丰度数据进行线性回归(表 2), 地表水中4类抗生素及配对ARGs丰度之间均存在显著正相关关系(P<0.05), 废水中只有FQs和FRGs丰度呈正相关, 沉积物中MLs和TCs与配对ARGs的丰度呈正相关, 土壤中FQs和FRGs的丰度呈正相关, 而沉积物和土壤中SAs与SRGs的丰度是负相关关系.其余样本抗生素与ARGs丰度的回归中没有发现显著相关关系.同一环境介质中, 不同种类的抗生素及配对ARGs丰度的相关关系和回归斜率不同, 虽然有其他环境因素的干扰, 但结果在一定程度上表明不同种类的抗生素对ARB和ARGs产生的选择作用和富集能力不同, 可能是因为它们不同的作用机制和抗性产生机制[13].与此相似, He等[32]在TCs、SAs和β-Ls的污染模拟实验中发现, ARGs的发生顺序为TRGs、SRGs和β-RGs, 说明TRGs相比于SRGs、β-RGs可能对抗生素更加敏感, 更容易被诱导产生和富集.不同环境介质中, 同类抗生素与配对ARGs丰度的相关关系也不相同, 这主要是因为抗生素在不同环境中的吸附、传输和转化等环境行为存在差异, 可能导致环境中直接与微生物接触的抗生素实际浓度(生物有效浓度)不同[33], 进而对微生物产生不同程度的实际影响[34, 35].固相样本中SAs与SRGs的负相关性可能与部分文献中其他因素而非抗生素对ARGs丰度的影响更为显著、以及样本性质与其他文献的样本性质差异较大有关, 其他文献样本点回归以及每单篇文献的样本点回归中两者均表现为正相关关系或不显著相关关系.
|
|
表 2 不同种类抗生素含量与配对ARGs丰度线性回归结果 Table 2 Linear regression results of different classes of antibiotics and corresponding ARGs in environmental samples |
2.2 ARGs丰度与重金属含量的相关性
本文共收集到112个同时监测了ARGs丰度和重金属含量的环境样本数据, 其中52个为水相环境样本, 60个为固相环境样本(图 3).水相和固相环境样本中的重金属含量范围分别为1.75~22 577.6 μg ·L-1和20.15~804.1 μg ·g-1.近年来, 工业三废排放、机动车尾气排放、化肥农药使用和矿山开发, 大大加速了重金属在环境中的释放[36], 重金属已经成为环境中一种重要的污染物和选择压力源.
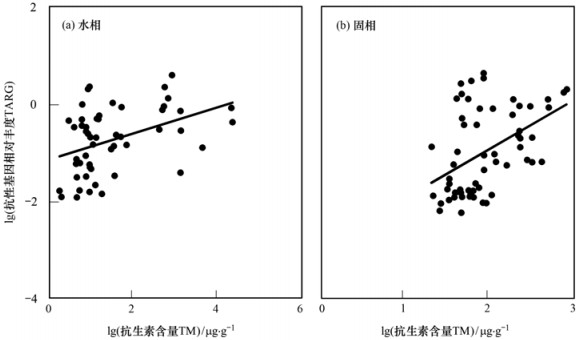
|
图 3 环境样本中总ARGs丰度和重金属含量 Fig. 3 Total ARGs and total heavy metals in environmental samples |
对环境样本中ARGs丰度和重金属含量的线性回归结果表明(表 3), 水相环境和固相环境中, ARGs丰度和重金属含量之间均存在显著正相关关系(P<0.01), R2分别为0.171和0.213, 说明重金属对细菌抗生素耐药性传播具有显著影响[37, 38].大多数研究的结果与本文相似, Knapp等[39]对90份苏格兰土壤样品的研究发现, 重金属含量与耐药基因水平之间存在显著的正相关关系(R>0.70), 除Se和Co之外的大多数金属都与部分耐药基因相关.Xu等[40]对接近金属矿区、重金属污染严重的湘江下游研究发现, 部分重金属与某些ARG亚型显著正相关(P<0.05), 重金属的选择性压力而非抗生素源排放对湘江中ARGs的分布格局有显著影响.耐药机制研究也表明, 一方面, 重金属具有和抗生素相似的抗性产生机制, 如外排泵过表达、细胞膜通透性改变和作用靶标改变等, 这些相似的抗性机制可能导致细菌对抗生素和重金属的交叉抗性或共抗性耐药[41].另一方面, 有研究报道, 重金属暴露条件下, 细胞内会产生过量的ROS, 促进基因的抗性突变和抗性基因的水平转移[42], ROS的生成也会刺激细胞产生应激反应, 改变细胞膜的通透性, 从而促进抗性基因的水平转移[43].因此, 重金属已被认为是影响ARGs传播的重要因素.
|
|
表 3 环境样本中总ARGs丰度与重金属含量线性回归结果 Table 3 Linear regression results of total ARGs and total heavy metals in environmental samples |
2.3 ARGs丰度对抗生素和重金属含量的线性回归
抗生素和重金属均为影响环境中ARGs分布的重要选择压力, 并且两者之间可能存在协同作用.因此, 对同时监测了3个参数的44个水相环境样本和41个固相环境样本数据, 以重金属含量、抗生素含量以及抗生素含量和重金属含量的交互项为自变量对ARGs丰度进行多元线性回归, 剔除不显著项, 回归结果如表 4.
|
|
表 4 总ARGs丰度与抗生素和重金属含量线性回归结果 Table 4 Linear regression results of total ARGs with antibiotics and heavy metals |
对抗生素和重金属两个变量进行多变量回归, 水相环境和固相环境样本最终回归模型的R2分别为0.482和0.707, 比只用单变量回归的R2更高.Mao等[44]对东海海湾沉积物中ARGs丰度的逐步回归模型中也发现, 最终回归模型中抗生素和重金属均在统计学意义上显著, 两个因素能够更好地解释环境样本中ARGs丰度的差异, 两者均为影响ARGs分布的重要因素[45, 46].但最终模型只解释了方差的一部分, 说明仍有影响ARGs传播的环境因素未包含在模型内.水相样本的单变量和最终回归模型中, 变量抗生素含量的斜率为负值, 这与水相环境样本的主要来源是废水有关(n=30).而其它单变量回归的斜率均为正值, 与前面的研究结果一致.在水相和固相环境样本的最终回归模型中, 抗生素含量和重金属含量的交互项对ARGs的传播均存在显著的正效应(P<0.05), 说明抗生素和重金属的交互作用对ARGs的传播也具有显著影响[44].与此相似, He等[32]通过鱼缸内SAs、TCs和Cu、Zn污染的模拟实验发现, 抗生素和重金属污染的综合效应大于个体选择效应的总和, 且协同效应随共选择因子(污染物种类)的增加而增加[47].抗生素和重金属协同效应的存在一方面是因为细菌可能通过同一机制对抗生素和重金属产生抗性, 两者施加的环境选择压力, 均可能会诱导细胞通过共调控作用产生多重抗性[41].另一方面, 通常发现ARGs和重金属抗性基因经常同时出现在可移动遗传元件上[48], 抗生素和重金属施加的选择压力会共同促进可移动遗传元件及其上ARGs在微生物间的传播[49].由于动物养殖和水产养殖中多种抗生素和重金属的长期交叉使用, 畜禽粪便和周边土壤、粪肥施用农田以及水产养殖水体和沉积物中重金属和抗生素的复合污染现象普遍存在[2, 50], 两者的协同作用可能导致更为严重的耐药性传播风险.
3 结论本文整理了现有的同时检测了环境介质中抗生素含量、重金属含量和ARGs丰度的文献数据, 通过单变量和多变量线性回归模型分析发现: 抗生素和重金属含量均为影响环境中ARGs丰度的重要因素, 两者的交互作用与ARGs的传播也有显著相关关系(P<0.05).引入两个变量的统计回归模型的最终R2分别为0.482和0.707, 比单变量模型的R2更高, 能够更好地解释环境样本中ARGs丰度的差异, 但也只能解释方差的一部分.抗生素诱导产生和富集ARGs的能力与抗生素种类和环境基质类型有很大关系.
| [1] | Bengtsson-Palme J, Kristiansson E, Larsson D G J. Environmental factors influencing the development and spread of antibiotic resistance[J]. FEMS Microbiology Reviews, 2018, 42(1). DOI:10.1093/femsre/fux053 |
| [2] | Gorovtsov A V, Sazykin I S, Sazykina M A. The influence of heavy metals, polyaromatic hydrocarbons, and polychlorinated biphenyls pollution on the development of antibiotic resistance in soils[J]. Environmental Science and Pollution Research, 2018, 25(10): 9283-9292. DOI:10.1007/s11356-018-1465-9 |
| [3] | Zhang Y, Gu A Z, He M, et al. Subinhibitory concentrations of disinfectants promote the horizontal transfer of multidrug resistance genes within and across genera[J]. Environmental Science & Technology, 2017, 51(1): 570-580. |
| [4] | Shao S C, Hu Y Y, Cheng J H, et al. Research progress on distribution, migration, transformation of antibiotics and antibiotic resistance genes(ARGs)in aquatic environment[J]. Critical Reviews in Biotechnology, 2018, 38(8): 1195-1208. DOI:10.1080/07388551.2018.1471038 |
| [5] | Guo J H, Li J, Chen H, et al. Metagenomic analysis reveals wastewater treatment plants as hotspots of antibiotic resistance genes and mobile genetic elements[J]. Water Research, 2017, 123: 468-478. DOI:10.1016/j.watres.2017.07.002 |
| [6] | Yuan W, Tian T T, Yang Q X, et al. Transfer potentials of antibiotic resistance genes in Escherichia spp. strains from different sources[J]. Chemosphere, 2020, 246. DOI:10.1016/j.chemosphere.2019.125736 |
| [7] | Sun W, Qian X, Gu J, et al. Mechanism and effect of temperature on variations in antibiotic resistance genes during anaerobic digestion of dairy manure[J]. Scientific Reports, 2016, 6. DOI:10.1038/srep30237 |
| [8] | Zhao W X, Wang B, Yu G. Antibiotic resistance genes in China: occurrence, risk, and correlation among different parameters[J]. Environmental Science and Pollution Research, 2018, 25(22): 21467-21482. DOI:10.1007/s11356-018-2507-z |
| [9] | Andersson D I, Hughes D. Microbiological effects of sublethal levels of antibiotics[J]. Nature Reviews Microbiology, 2014, 12(7): 465-478. DOI:10.1038/nrmicro3270 |
| [10] | Wright M S, Peltier G L, Stepanauskas R, et al. Bacterial tolerances to metals and antibiotics in metal-contaminated and reference streams[J]. FEMS Microbiology Ecology, 2006, 58(2): 293-302. DOI:10.1111/j.1574-6941.2006.00154.x |
| [11] | Larsson D G J, Andremont A, Bengtsson-Palme J, et al. Critical knowledge gaps and research needs related to the environmental dimensions of antibiotic resistance[J]. Environment International, 2018, 117: 132-138. DOI:10.1016/j.envint.2018.04.041 |
| [12] | McArthur A G, Waglechner N, Nizam F, et al. The comprehensive antibiotic resistance database[J]. Antimicrobial Agents and Chemotherapy, 2013, 57(7): 3348-3357. DOI:10.1128/AAC.00419-13 |
| [13] | Gullberg E, Cao S, Berg O G, et al. Selection of resistant bacteria at very low antibiotic concentrations[J]. PLoS Pathogens, 2011, 7(7). DOI:10.1371/journal.ppat.1002158 |
| [14] | Hughes D, Andersson D I. Selection of resistance at lethal and non-lethal antibiotic concentrations[J]. Current Opinion in Microbiology, 2012, 15(5): 555-560. DOI:10.1016/j.mib.2012.07.005 |
| [15] | Kohanski M A, DePristo M A, Collins J J. Sublethal antibiotic treatment leads to multidrug resistance via radical-induced mutagenesis[J]. Molecular Cell, 2010, 37(3): 311-320. DOI:10.1016/j.molcel.2010.01.003 |
| [16] | Ben W W, Wang J, Cao R K, et al. Distribution of antibiotic resistance in the effluents of ten municipal wastewater treatment plants in China and the effect of treatment processes[J]. Chemosphere, 2017, 172: 392-398. DOI:10.1016/j.chemosphere.2017.01.041 |
| [17] | Ben W W, Wang J, Pan X, et al. Dissemination of antibiotic resistance genes and their potential removal by on-farm treatment processes in nine swine feedlots in Shandong Province, China[J]. Chemosphere, 2017, 167: 262-268. DOI:10.1016/j.chemosphere.2016.10.013 |
| [18] | Sandegren L, Andersson D I. Bacterial gene amplification: implications for the evolution of antibiotic resistance[J]. Nature Reviews Microbiology, 2009, 7(8): 578-588. DOI:10.1038/nrmicro2174 |
| [19] | Bean D C, Livermore D M, Papa I, et al. Resistance among Escherichia coli to sulphonamides and other antimicrobials now little used in man[J]. Journal of Antimicrobial Chemotherapy, 2005, 56(5): 962-964. DOI:10.1093/jac/dki332 |
| [20] | Hu H W, Wang J T, Li J, et al. Field-based evidence for copper contamination induced changes of antibiotic resistance in agricultural soils[J]. Environmental Microbiology, 2016, 18(11): 3896-3909. DOI:10.1111/1462-2920.13370 |
| [21] | Li D, Zeng S Y, He M, et al. Water disinfection byproducts induce antibiotic resistance-role of environmental pollutants in resistance phenomena[J]. Environmental Science & Technology, 2016, 50(6): 3193-3201. |
| [22] |
魏媛媛. 季铵化合物对抗生素抗性基因传播扩散的影响和机理探究[D]. 杭州: 浙江大学, 2018. Wei Y Y. The effect and mechanism of quaternary ammonium compound on the distribution of antibiotic resistant genes[D]. Hangzhou: Zhejiang University, 2018. |
| [23] | Jutkina J, Marathe N P, Flach C F, et al. Antibiotics and common antibacterial biocides stimulate horizontal transfer of resistance at low concentrations[J]. Science of the Total Environment, 2018, 616-617: 172-178. DOI:10.1016/j.scitotenv.2017.10.312 |
| [24] | Wang X L, Yang F X, Zhao J, et al. Bacterial exposure to ZnO nanoparticles facilitates horizontal transfer of antibiotic resistance genes[J]. NanoImpact, 2018, 10: 61-67. DOI:10.1016/j.impact.2017.11.006 |
| [25] | Luo Y, Wang Q, Lu Q, et al. An Ionic liquid facilitates the proliferation of antibiotic resistance genes mediated by class I integrons[J]. Environmental Science & Technology Letters, 2014, 1(5): 266-270. |
| [26] | Duarte D J, Oldenkamp R, Ragas A M J. Modelling environmental antibiotic-resistance gene abundance: a meta-analysis[J]. Science of the Total Environment, 2019, 659: 335-341. DOI:10.1016/j.scitotenv.2018.12.233 |
| [27] | Zhang Q Q, Ying G G, Pan C G, et al. Comprehensive evaluation of antibiotics emission and fate in the river basins of China: source analysis, multimedia modeling, and linkage to bacterial resistance[J]. Environmental Science & Technology, 2015, 49(11): 6772-6782. |
| [28] | Li S, Shi W Z, Liu W, et al. A duodecennial national synthesis of antibiotics in China's major rivers and seas(2005-2016)[J]. Science of the Total Environment, 2018, 615: 906-917. DOI:10.1016/j.scitotenv.2017.09.328 |
| [29] | Qin Y W, Wen Q, Ma Y Q, et al. Antibiotics pollution in Gonghu Bay in the period of water diversion from Yangtze River to Taihu Lake[J]. Environmental Earth Sciences, 2018, 77(11). DOI:10.1007/s12665-018-7558-4 |
| [30] | Sarmah A K, Meyer M T, Boxall A B A. A global perspective on the use, sales, exposure pathways, occurrence, fate and effects of veterinary antibiotics(VAs)in the environment[J]. Chemosphere, 2006, 65(5): 725-759. DOI:10.1016/j.chemosphere.2006.03.026 |
| [31] | Liu X H, Lu S Y, Guo W, et al. Antibiotics in the aquatic environments: a review of lakes, China[J]. Science of the Total Environment, 2018, 627: 1195-1208. DOI:10.1016/j.scitotenv.2018.01.271 |
| [32] | He X L, Xu Y B, Chen J L, et al. Evolution of corresponding resistance genes in the water of fish tanks with multiple stresses of antibiotics and heavy metals[J]. Water Research, 2017, 124: 39-48. DOI:10.1016/j.watres.2017.07.048 |
| [33] | Ding C, He J Z. Effect of antibiotics in the environment on microbial populations[J]. Applied Microbiology and Biotechnology, 2010, 87(3): 925-941. DOI:10.1007/s00253-010-2649-5 |
| [34] | Jia J, Guan Y J, Cheng M Q, et al. Occurrence and distribution of antibiotics and antibiotic resistance genes in Ba River, China[J]. Science of the Total Environment, 2018, 642: 1136-1144. DOI:10.1016/j.scitotenv.2018.06.149 |
| [35] | Wang J, Ben W W, Yang M, et al. Dissemination of veterinary antibiotics and corresponding resistance genes from a concentrated swine feedlot along the waste treatment paths[J]. Environment International, 2016, 92-93: 317-323. DOI:10.1016/j.envint.2016.04.020 |
| [36] |
陈欣瑶. 重金属—抗生素单一及复合污染胁迫下土壤生态功能稳定性及其微生物调控机制研究[D]. 苏州: 苏州科技大学, 2019. Chen X Y. Study on stability of soil ecological function and microbial regulation mechanisms under the single and combined pollution of heavy metal and antibiotic[D]. Suzhou: Suzhou University of Science and Technology, 2019. |
| [37] | Devarajan N, Laffite A, Graham N D, et al. Accumulation of clinically relevant antibiotic-resistance genes, bacterial load, and metals in freshwater lake sediments in Central Europe[J]. Environmental Science & Technology, 2015, 49(11): 6528-6537. |
| [38] | McKinney C W, Loftin K A, Meyer M T, et al. Tet and sul antibiotic resistance genes in livestock lagoons of various operation type, configuration, and antibiotic occurrence[J]. Environmental Science & Technology, 2010, 44(16): 6102-6109. |
| [39] | Knapp C W, McCluskey S M, Singh B K, et al. Antibiotic resistance gene abundances correlate with metal and geochemical conditions in archived Scottish soils[J]. PLoS One, 2011, 6(11). DOI:10.1371/journal.pone.0027300 |
| [40] | Xu Y, Xu J, Mao D Q, et al. Effect of the selective pressure of sub-lethal level of heavy metals on the fate and distribution of ARGs in the catchment scale[J]. Environmental Pollution, 2017, 220: 900-908. DOI:10.1016/j.envpol.2016.10.074 |
| [41] |
陈帅, 邹海燕, 高方舟, 等. 抗生素、重金属和杀生剂抗性共选择机制[J]. 生态毒理学报, 2020, 15(2): 1-10. Chen S, Zou H Y, Gao F Z, et al. Co-selection mechanism of antibiotic, metal and biocide resistance[J]. Asian Journal of Ecotoxicology, 2020, 15(2): 1-10. |
| [42] | Zhang S, Wang Y, Song H L, et al. Copper nanoparticles and copper ions promote horizontal transfer of plasmid-mediated multi-antibiotic resistance genes across bacterial genera[J]. Environment International, 2019, 129: 478-487. DOI:10.1016/j.envint.2019.05.054 |
| [43] | Zhang Y, Gu A Z, Cen T Y, et al. Sub-inhibitory concentrations of heavy metals facilitate the horizontal transfer of plasmid-mediated antibiotic resistance genes in water environment[J]. Environmental Pollution, 2018, 237: 74-82. DOI:10.1016/j.envpol.2018.01.032 |
| [44] | Mao D Q, Yu S, Rysz M, et al. Prevalence and proliferation of antibiotic resistance genes in two municipal wastewater treatment plants[J]. Water Research, 2015, 85: 458-466. DOI:10.1016/j.watres.2015.09.010 |
| [45] | Gao P, He S, Huang S L, et al. Impacts of coexisting antibiotics, antibacterial residues, and heavy metals on the occurrence of erythromycin resistance genes in urban wastewater[J]. Applied Microbiology and Biotechnology, 2015, 99(9): 3971-3980. DOI:10.1007/s00253-015-6404-9 |
| [46] | Guo X P, Liu X R, Niu Z S, et al. Seasonal and spatial distribution of antibiotic resistance genes in the sediments along the Yangtze Estuary, China[J]. Environmental Pollution, 2018, 242: 576-584. DOI:10.1016/j.envpol.2018.06.099 |
| [47] | Chen J Y, Su Z G, Dai T J, et al. Occurrence and distribution of antibiotic resistance genes in the sediments of the East China Sea bays[J]. Journal of Environmental Sciences, 2019, 81: 156-167. DOI:10.1016/j.jes.2019.01.016 |
| [48] | Wright G D. The antibiotic resistome: the nexus of chemical and genetic diversity[J]. Nature Reviews Microbiology, 2007, 5(3): 175-186. DOI:10.1038/nrmicro1614 |
| [49] | Wang N, Guo X Y, Yan Z, et al. A comprehensive analysis on spread and distribution characteristic of antibiotic resistance genes in livestock farms of southeastern China[J]. PLoS One, 2016, 11(7). DOI:10.1371/journal.pone.0156889 |
| [50] | Seiler C, Berendonk T U. Heavy metal driven co-selection of antibiotic resistance in soil and water bodies impacted by agriculture and aquaculture[J]. Frontiers in Microbiology, 2012, 3. DOI:10.3389/fmicb.2012.00399 |
 2021, Vol. 42
2021, Vol. 42


