2. 中国科学院生态环境研究中心, 北京 100085
2. Research Center for Eco-Environmental Sciences, Chinese Academy of Sciences, Beijing 100085, China
生物因素带来的安全问题自古以来是人类面临的巨大挑战.人类历史上多次由细菌和病毒引起的传染性疾病大暴发, 造成全球大量人口死亡.生物武器的使用以及生物恐怖对军事安全和社会稳定造成巨大的影响.食品安全和食源性疾病的危害是人们关注的重大问题.此外, 现代新兴生物技术和生物体改造手段的快速发展, 为科学伦理和生物安全带来了新的挑战.政治经济的全球化、国际贸易和旅游业的发展加快了微生物病原体的流动和促进了传染性疾病的传播.2009~2019年, 世界卫生组织(WHO)已宣布过5次"国际关注的突发公共卫生事件", 包括2009年H1N1猪流感病毒疫情、2014年野生型脊髓灰质炎病毒疫情、2014年西非埃博拉病毒疫情、2015~2016年寨卡病毒疫情和2018年刚果埃博拉病毒疫情等. 2020年新冠疫情的全球大流行对人民健康、社会经济和政治均产生了深远的影响.
2020年3月2日, 习近平总书记指出: 生命安全和生物安全领域的重大科技成果也是国之重器, 疫病防控和公共卫生应急体系是国家战略体系的重要组成部分.要完善关键核心技术攻关的新型举国体制, 加快推进人口健康和生物安全等领域科研力量布局, 整合生命科学生物技术、医药卫生和医疗设备等领域的国家重点科研体系, 布局一批国家临床医学研究中心, 加大卫生健康领域科技投入, 加强生命科学领域的基础研究和医疗健康关键核心技术突破, 加快提高疫病防控和公共卫生领域战略科技力量和战略储备能力.
生物安全是国家安全的重要组成部分, 对人民健康、社会经济发展及生态环境具有不可估量的影响.其重要性不亚于金融安全和能源安全, 并与粮食安全和生态安全紧密相关.生物安全关系到国家公共卫生、社会稳定、经济发展和国防建设, 各国如美国和英国等主要发达国家均已或拟将生物安全战略纳入国家安全战略, 制定相关的国家生物安全战略规划[1].生物安全相较于政治、经济和军事等传统安全问题, 其本身具有鲜明的特点[2].大多数新发和烈性传染病具有传染性强和传播速度快等特点, 而城市作为现代主要人群的生活和工作场所, 人口密集, 城市居民聚集性社交频繁; 同时城市工商业发达, 是区域政治、经济和文化中心.城市一旦暴发生物安全问题, 将给居民健康、社会经济发展和生态环境带来重大影响, 因此亟需从国家战略的高度采取积极有效的应对策略, 保障城市环境生物安全, 维护人群健康、国家经济和生态系统安全.
1 生物安全的内涵关于生物安全的定义国际上尚无广泛认可的统一概念, 其研究内涵有狭义和广义之分[2].狭义的生物安全包括两个方面: 一是指病原微生物的安全防护与管理措施, 保护实验室人员免受感染, 防止病原微生物等实验材料的意外泄漏, 保证病原微生物不被有意窃取或有意地滥用等所带来的危害[3, 4]; 二是指对现代生物技术的研究、开发和应用的管理措施, 以及转基因生物体的利用与转移, 使其符合科学伦理规范, 防止转基因生物体的滥用带来的食品安全和生态安全等问题.
广义的生物安全则指与生物有关的人为或非人为因素对国家和区域社会、经济、人民健康及生态环境所产生的危害或潜在风险, 以及对这些危害或风险进行防范、管理的战略性和综合性措施.这里的生物因素包括了自然界中天然的生物因子、转基因生物和现代生物技术[2].广义的生物安全涵盖了传染性疾病、生物武器和生物恐怖、食品安全、微生物实验室安全、现代生物技术安全以及生态安全的部分内容, 符合当前国家安全、人群健康、现代生物技术管理和生态环境压力等多个方面对生物安全的需求.
2 城市环境生物安全的范畴健康是促进人的全面发展的必然要求, 是经济社会发展的基础条件.健康中国建设已上升为国家战略, 2016年国务院印发"健康中国2030"规划纲要, 旨在推进健康中国建设, 提高人民健康水平, 实现国民健康长寿, 是国家富强、民族振兴的重要标志, 也是全国各族人民的共同愿望[5].我国城市化水平不断提高, 2012年我国城市人口首次超过农村人口, 当前农村人口迁移到城市的趋势还在持续.然而随着城市化的发展, 城市中人口高度密集, 城乡居民交流广泛, 为城市发展提供了主要推动力, 同时也给城市发展带来愈发严峻的新挑战.
居民健康离不开健康的城市环境.近年来的新发传染病多为人畜共患病, 许多病原微生物可通过多种环境介质在人和动物间传播, 人类、动物和生态系统的健康紧密相关(图 1)[6], 人们逐渐认识到需要采用健康一体化(One Health)的理念, 通过跨学科、跨部门和跨国家(地区)多方面的合作来防控传染性疾病的发生和扩散[7~9], 因此保持城市环境健康安全是保障生物安全和保护人群健康的重要组成部分.
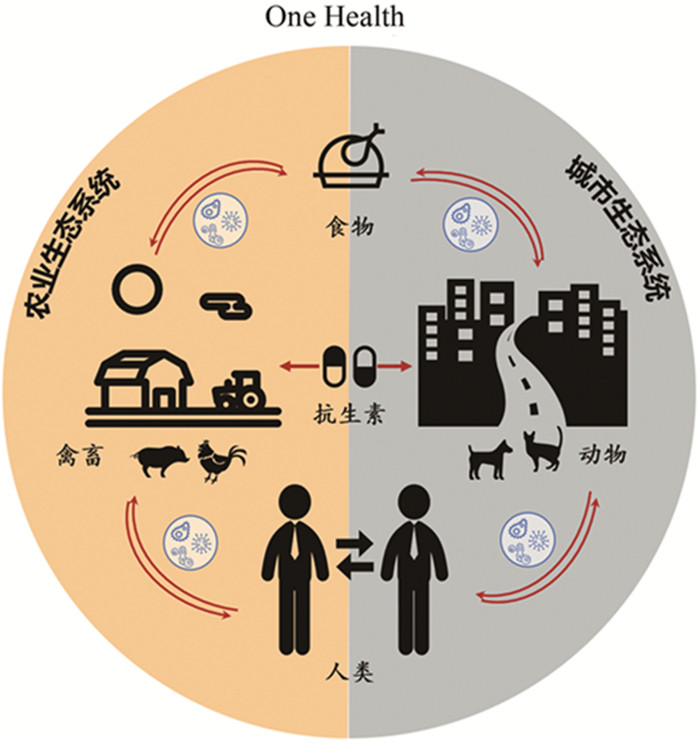
|
修改自文献[6] 图 1 人、动物和环境共享复杂微生物世界 Fig. 1 Humans, animals, and the environment share a complex microbial world |
城市环境生物安全即指城市生态系统中由生物带来的对人群健康、经济和生态环境的潜在风险和危害, 以及相应的防控措施.这里的生物因素同样包括了自然界中天然的生物污染物、转基因生物和现代生物技术, 但更侧重于环境方面的内容, 包括人为和非人为因素导致的生物污染物向城市环境的释放、人群通过多种环境介质(包括空气、土壤、水体、食品及城市建成环境介质等)暴露于生物污染物的健康风险、以及对生物污染物的监测、减排和灭活等综合性管理措施.
病原微生物、耐药微生物及其耐药基因是环境中主要的微生物污染物.与病原微生物相关的传染性疾病是当前生物安全领域面临的最重要的问题, 主要是病原细菌、真菌、病毒和原生动物等, 如美国CDC所列具有潜在严重健康威胁的病原微生物清单(https://www.selectagents.gov/SelectAgentsandToxinsList.html).此外常见病原菌抗生素耐药性的增强和全球范围内的扩散也严重威胁了人类健康和经济发展[10~12], 相关的耐药微生物和耐药基因也引起了广泛关注[13~15]. 城市环境中存在大量微生物, 特定条件下少量微生物物种可以迁移到人类和动物体内并引发疾病, 由微生物引发的疾病往往具有隐蔽性、潜伏期长、突发性和致病性强烈等特点, 尤其是对于新型致病微生物引发的传染性疾病的防控难度巨大.国际上对于生物实验室和转基因生物等现代生物技术目前已有相对较为完善的分级和管理方案[3, 16, 17], 然而目前对城市环境微生物组成、功能、传播途径和扩散机制尚未有清晰的认知.城市环境是人类对自然环境干预最强烈和自然环境变化最剧烈的场所, 具有人口密度高、人口流动性强、人群来源复杂、物质和能源流动性大和环境变化剧烈等特征.城市化发展进程带来的生态退化、环境污染、气候变化和野生动物接触等剧烈变化, 使得城市环境中动物、微生物组成和多样性也会发生剧烈变化, 因此, 城市环境也是微生物-人群-环境相互作用最为强烈和最为复杂的场所.从城市生态系统健康角度出发, 对城市环境微生物开展系统研究将有利于城市微生物安全风险防控和治理体系建设, 从而将传染性疾病预防的关口进一步提前.
3 城市环境微生物安全研究进展城市微生物组是城市不同环境介质中多种土著和瞬态微生物的集合, 这些微生物栖息在城市生态系统中的自然环境和建成环境中, 包括大气, 绿化植物, 土壤, 水体, 建筑物表面, 室内环境, 人体以及城市基建环境(如医院、污水处理厂、下水管道、道路和地铁系统等)等.城市微生物组在城市生态系统中具有最高的物种和遗传多样性, 为维持人类活动提供主要的生态系统服务[18, 19].有研究表明, 美国城市大气中具有高度多样化的瞬态微生物群落, 存在着至少1 800种不同的细菌[20]; 非洲和加勒比海沙尘暴每克土中约含有10 000个细菌, 每年约有100万t的沙尘土壤在大气中移动, 因此估计在大气中至少约有1016个灰尘细菌[21, 22]; 城市公园土壤具有其他生物群区(如北极区、热带区和沙漠地区)同样多的微生物物种分类和群落种类[23].城市微生物丰度、多样性和功能会随着不同土地利用类型发生变化, 土壤深度增加微生物多样性增加[23, 24].湿度、温度、植物、宠物和人类等因素也会影响微生物从室外环境向建成环境内部的迁移[25~27].最近的研究也表明中国香港和纽约城市地铁系统具有高度多样的微生物, 温度、相对湿度和人流高峰时段都会影响地铁内微生物的多样性[28, 29].
城市环境微生物群落中包含多种病原微生物, 可以通过人类活动在城市环境中传播扩散, 直接影响人群健康, 而且近年来的研究表明环境微生物组成和多样性也跟人体健康紧密相关[30~32].目前, 传染病仍然是全球卫生和安全的重大威胁, 下呼吸道感染、腹泻、艾滋病和结核病等是国际关注的传染性疾病[33], 感染性细菌耐药性的增强和扩散极大地降低了抗生素的疗效.围绕着城市环境生物安全和人群健康, 各国科学家们主要开展了微生物污染物(包括病原微生物、耐药微生物和耐药基因)的来源和组成、迁移与传播和健康效应与风险评估、防控策略等方面的研究.
3.1 微生物污染物的来源城市环境生物安全研究中首要需鉴别各类别微生物污染物的主要来源, 从而有针对性地对不同污染源进行防控.自然环境中病原微生物较少[34], 但在人和动物聚集环境中其丰度显著增加, 人群主导的生态系统中人畜共患病原微生物宿主多样性也显著升高[35].医院是城市环境中微生物污染物的重要来源, 医院环境病人聚集, 常见病原微生物和耐药基因均可在医院环境中检出, 并且多重耐药菌比例较高[36].医院环境微生物污染物多为人类病原菌和抗生素治疗高度相关的耐药基因, 具有较高的人群健康威胁[11], 因此受到广泛关注.
城市生活污水和污水处理系统是城市环境微生物污染物的另一重要来源[37, 38].城市生活污水汇集了大量人体肠道微生物、耐药细菌和耐药基因等, 未经处理的生活污水排放可造成城市土壤和水体污染, 即使是处理后出水中仍可含有一定量的微生物污染物[39].
动物养殖业也是城市环境微生物污染物来源之一.动物养殖场通常不位于城市中, 其直接影响较小.然而动物粪便及其制备的有机肥常常含有微生物污染物, 有机肥农用及城市绿地用肥可导致城市土壤污染[40], 并可使植物如蔬菜中微生物污染物丰度增加[41], 从而对食品安全造成潜在威胁.
动物是人畜共患病原微生物的主要携带者, 驯养动物、灵长类和蝙蝠比其它哺乳动物携带有更多种的传染性动物病毒[42].动物源食品安全和家养宠物传染性微生物也一直是生物安全关注的部分.近年来, 城市野生动物逐渐引起关注, 如有研究表明美国纽约城区的家鼠是致病细菌和病毒的潜在储库, 家鼠粪便中检测到36种病毒, 其中6种是新病毒, 同时也携带4种可引起人类肠道感染疾病的细菌[43]; 澳大利亚墨尔本附近的水鸟携带27种病毒, 包括2种多宿主病毒(禽类冠状病毒和甲型流感病毒)及1种新型轮状病毒[44].相应地城市农贸市场、批发市场等可能成为微生物污染物的重要节点, 有研究表明城市农贸市场活禽交易区耐药基因丰度显著高于周边样品[45].
此外, 城市环境中其它一些微生物富集区域也影响城市生物安全.比如城市生活垃圾堆放点容易滋生多种潜在病原菌, 威胁人类和动物健康[46, 47].室内空调系统可富集病原菌和病毒, 如嗜肺军团菌跟空调系统紧密相关, 可引起感染性肺炎, 影响室内环境微生物安全[48].
3.2 微生物污染物在城市环境中的迁移与传播人、动物活动以及自然因素驱动了微生物污染物在城市环境中的迁移和传播(图 2)[35].一项基于数学模型的研究显示热带森林地区土地利用方式改变及哺乳动物多样性与动物传染性疾病的暴发显著相关[49].人和动物是微生物污染物的主要携带者, 人和动物在城市环境中的活动必然伴随着微生物污染物在城市环境中的扩散、迁移和循环.目前已有多项针对城市交通工具表面和空气中的微生物组、潜在病原微生物以及耐药基因的组成、分布特征的研究[50~54].
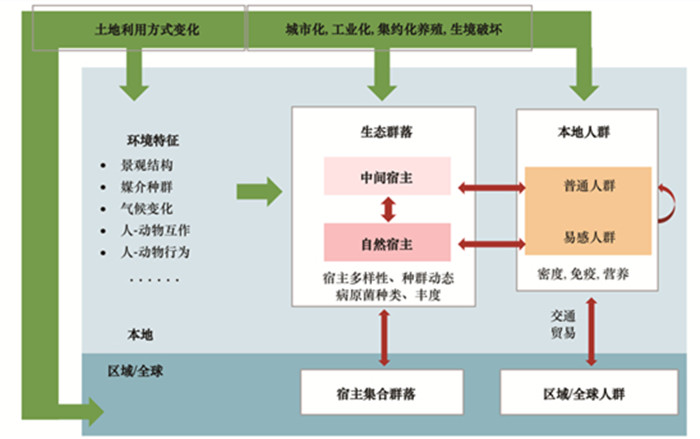
|
修改自文献[35] 图 2 土地利用方式变化对人畜共患疾病传播的影响 Fig. 2 Conceptual framework for the effects of land use change on zoonotic disease transmission |
联合国环境规划署鉴别了驱动动物传染性疾病暴发的7个主要人为因子, 包括人类对动物蛋白需求的增加、不可持续的集约农业、城市化等带来的对自然资源不可持续性的利用、对野生动物的开发利用、食物供应链的变化、旅行和交通以及气候变化[55].多种人为活动均可促进微生物污染物在城市环境不同介质中扩散和迁移, 如污水的排放、动物有机肥的施用、医疗和生活垃圾的填埋、动物屠宰和交易等.
微生物污染物在城市环境中的迁移、传播和循环还受到微生物自身的生物学特性和自然环境驱动力的影响.不同类型的病原微生物在不同类型环境介质中其赋存特征具有极大差异.城市环境中空气和水体流动等是重要的自然驱动力.有研究表明城市灰尘中含有多种耐药基因, 并且具有明显的季节差异, 这些灰尘中耐药基因可通过空气流动进入大气环境中, 影响空气中的耐药基因组成[56, 57].空气质量对空气耐药基因的影响受到了广泛的关注, 空气污染严重时, 尤其是雾-霾天气中空气耐药基因的多样性和丰度通常高于正常水平[58~62].
大量对人类健康构成威胁的病原微生物, 包括细菌、病毒、真菌和原生动物等也广泛分布于城市水体中.已被识别出的水传播病原微生物超过140种[63], 它们可通过饮水传播、水依赖传播以及与水相关的生物携带传播等方式导致传染病的发生和流行.近年来全球频发的极端降雨事件, 给城市水体的生物安全保障带来了极大威胁: 2005年Katrina飓风、2015年Soudelor超强台风和2018年Harvey飓风分别导致美国新奥尔良、中国厦门以及美国休斯敦地区城市排水系统超载, 造成未经处理的污水溢流, 极大地增加了城市水体、饮水管网、地表路面以及低层建筑中病原微生物的暴露风险[64~66].最近的一项研究表明空气污染可通过降雪导致耐药基因的全球扩散[67].
3.3 微生物污染物的人群暴露健康风险在医学领域病原微生物的健康风险已有较为深入研究, 针对细菌耐药性问题, WHO也于2017年发布了"指导新抗生素研发的抗生素耐药细菌优先性列表"[14].在城市环境生物安全研究中主要侧重人群通过饮食、呼吸和接触等途径微生物污染物的暴露健康风险.定量微生物风险评价(quantitative microbial risk assessment, QMRA)是一种以定量的方式评价病原微生物暴露对人体健康的风险, 包括危害鉴定、暴露评价、剂量-反应关系评价和风险表征, 已被WHO、美国、加拿大、欧盟和澳大利亚等国家和组织应用于水环境风险管理[68, 69].国外学者应用QMRA对城市水体病原微生物暴露风险开展了广泛研究[70], 该研究发现多种人类粪便指示菌和指示病毒可替代水传播病原微生物来评估污水原水和二级出水污染休闲水体造成的人群肠道健康风险.我国学者基于隐孢子虫、蓝氏贾第鞭毛虫、粪大肠菌群和总大肠菌群等常规指标, 对饮用水、景观水体及再生水应用场所开展了QMRA分析[72~74].
相对于病原微生物, 目前关于环境中耐药基因的健康效应和风险评估目前研究较少, 是该领域当前研究的难点和重点[75].虽然目前已有人提出了基于抗性基因的抗性类型、可移动性以及是否与病原菌相关等标准的抗性基因风险分级框架[76], 鉴别抗性基因通过呼吸、摄食等人群暴露途径, 进而研究环境耐药暴露对肠道微生物和耐药组的影响[77], 量化抗性基因的暴露量, 从而结合微生物风险评估的方法初步对抗性基因风险进行评价, 然而针对耐药基因的健康风险的评价仍处于起步阶段[75, 78].主要因为受限于健康风险评价所用的各种定量数据及模型方法较为缺乏, 比如大尺度的抗性基因种类和丰度的定量数据, 抗性基因水平转移频率的估算, 抗生素和重金属等选择制剂的选择压力与抗性基因突变和转移的定量关系, 与抗性基因相关的流行病学数据的收集及其量化模型的构建等因素[79, 80].
3.4 防控策略目前城市环境生物安全的防控策略首要以源头控制为主, 包括完善城市公共卫生基础设施, 严格消毒医疗废弃物, 改进工艺(如高级氧化法)提高污水处理对微生物污染物的去除率, 发展超高温堆肥、动物堆肥和生物炭制备等技术进行动物粪便和剩余污泥的处理等措施, 从而减少中水排放和有机肥施用对受纳环境的污染, 并基于流行病学数据鉴别和预测传染病高发风险地区, 从而有针对性地开展防控[49, 81].其次, 建立健全监测报告预警系统, 如由疾病防御控制中心负责和管理的传染病疫情监测和报告系统[82], 国家卫生计生委合理用药专家委员会负责的全国细菌耐药监测网(CARSS)以及由农业农村部负责的动物源细菌耐药性监测计划等.再次, 针对微生物污染物主要传播媒介和途径的阻断措施, 如此次新冠疫情中采取的感染者隔离、勤洗手和戴口罩等措施.最后, 采用物理和化学等方法对环境介质中微生物污染物的消杀措施.
4 城市环境生物安全的挑战和展望尽管国内外科学家在该领域已开展了较多的研究, 但仍然存在许多科学问题亟待解决.首先, 现有研究手段难以满足城市环境生物安全对微生物污染物监测和预警的需求.微生物污染物包括多种病原微生物和耐药基因等, 然而我国现行3个水质标准《生活饮用水卫生标准》(GB 5749-2006)、《地表水环境质量标准》(GB 3838-2002)和《地下水环境质量标准》(GB 14848-2017), 仅收录了总大肠菌群、粪大肠菌群、大肠埃希菌、沙门菌、大肠埃希菌O157 ∶H7、蓝氏贾第鞭毛虫和隐孢子虫等病原微生物指标, 远不足以反映城市水体的水质状况, 更谈不上维护生物安全.近年来已发展出高通量病原菌检测技术[83, 84], 但尚未推广应用于环境生物安全领域, 微生物污染物的实时在线监测更是该领域急需突破的技术.除了病原菌以外, 病毒是人群健康的主要威胁, 尽管当前高通量测序技术极大地支持了该领域的研究, 但仍有许多问题尚未解决[85], 特别是对未知病毒的威胁缺乏有效的识别手段, 并且目前尚未有针对病毒的高通量定量监测技术.未来需要特别加强环境病毒组的研究及其相关数据库的构建.
正是由于研究手段的缺乏, 目前相关研究多聚焦于少数几种微生物污染物, 尚未能全面描绘微生物污染物在城市环境中的分布特征.同时, 由于城市生态系统的复杂性, 微生物污染物的生物学特征、人为活动和自然因素共同影响了微生物污染物在城市环境中的生消动态和扩散过程, 尚未能全面解析其迁移传播途径和规律, 甑别主控影响因素.
对微生物污染物环境暴露风险研究不足.目前暴露风险研究同样集中于少数几种微生物污染物, 而对于多数病原微生物缺乏不同病原微生物的生理毒性及人群暴露参数等基础数据的积累, 同时缺乏对QMRA各个环节的不确定性分析[69].
针对这些挑战, 聚焦城市多生境多物种的微生物组学和生物安全, 综合考虑病因、宿主和环境的相互关系, 将来在城市环境生物安全急需开展以下研究: ①研发城市环境微生物组监测新方法, 建立城市环境微生物监测系统, 探明城市环境微生物组和典型人类病原菌的时空变化规律, 构建城市环境生物安全大数据平台; ②明确城市环境微生物组分布规律和扩散传播途径, 解析环境微生物组与人体微生物组的相互作用机制; ③识别城市环境病原微生物的人群暴露途径, 构建病原菌传播预警模型, 评估微生物污染健康风险, 并提出防控对策.从而形成系统完备的"实验-监测-大数据"的综合平台, 为国家生物安全防控提供科技支撑.此外, 未来还应加强与疾控和临床领域的合作, 在早期监测、诊断、预防、控制和治疗全链条联合攻关, 构建城市微生物安全风险防控和治理体系.
| [1] |
陈方, 张志强, 丁陈君, 等. 国际生物安全战略态势分析及对我国的建议[J]. 中国科学院院刊, 2020, 35(2): 204-211. Chen F, Zhang Z Q, Ding C J, et al. Analysis of global biosafety strategy and recommendations to China[J]. Bulletin of the Chinese Academy of Sciences, 2020, 35(2): 204-211. |
| [2] | 中国科学院武汉文献情报中心, 生物安全战略情报研究中心. 生物安全发展报告: 科技保障安全[R]. 北京: 科学出版社, 2014. |
| [3] | WHO. Laboratory biosafety manual, 3rd edition[R]. Geneva: WHO, 2004. |
| [4] | WHO. FACT SHEET biosafety and biosecurity[R]. 2018. |
| [5] | 中共中央政治局. 中共中央政治局召开会议审议"健康中国2030"规划纲要[R]. 北京: 中共中央政治局, 2016. |
| [6] | Zhu Y G, Gillings M, Penuelas J. Integrating biomedical, ecological, and sustainability sciences to manage emerging infectious diseases[J]. One Earth, 2020, 3(1): 23-26. DOI:10.1016/j.oneear.2020.06.004 |
| [7] | Rabinowitz P M, Kock R, Kachani M, et al. Toward proof of concept of a one health approach to disease prediction and control[J]. Emerging Infectious Disease Journal, 2013, 19(12). DOI:10.3201/eid1912.130265 |
| [8] | Trinh P, Zaneveld J R, Safranek S, et al. One health relationships between human, animal, and environmental microbiomes: a mini-review[J]. Frontiers in Public Health, 2018, 6. DOI:10.3389/fpubh.2018.00235 |
| [9] | van Bruggen A H C, Goss E M, Havelaar A, et al. One health-cycling of diverse microbial communities as a connecting force for soil, plant, animal, human and ecosystem health[J]. Science of the Total Environment, 2019, 664: 927-937. DOI:10.1016/j.scitotenv.2019.02.091 |
| [10] | Cassini A, Högberg L D, Plachouras D, et al. Attributable deaths and disability-adjusted life-years caused by infections with antibiotic-resistant bacteria in the EU and the European Economic Area in 2015:a population-level modelling analysis[J]. The Lancet Infectious Diseases, 2019, 19(1): 56-66. DOI:10.1016/S1473-3099(18)30605-4 |
| [11] | Laxminarayan R, Van Boeckel T, Frost I, et al. The Lancet Infectious Diseases Commission on antimicrobial resistance: 6 years later[J]. The Lancet Infectious Diseases, 2020, 20(4): e51-e60. DOI:10.1016/S1473-3099(20)30003-7 |
| [12] | Van Boeckel T P, Pires J, Silvester R, et al. Global trends in antimicrobial resistance in animals in low-and middle-income countries[J]. Science, 2019, 365(6459). DOI:10.1126/science.aaw1944 |
| [13] | IACG. No time to wait: securing the future from drug-resistant infections[R]. Geneva: WHO, 2019. |
| [14] | WHO. WHO priority pathogens list for R&D of new antibiotics[R]. Geneva: WHO, 2017. |
| [15] |
朱永官, 欧阳纬莹, 吴楠, 等. 抗生素耐药性的来源与控制对策[J]. 中国科学院院刊, 2015, 30(4): 509-516. Zhu Y G, Ouyang W Y, Wu N, et al. Antibiotic resistance: sources and mitigation[J]. Bulletin of the Chinese Academy of Sciences, 2015, 30(4): 509-516. |
| [16] | Akbari O S, Bellen H J, Bier E, et al. Safeguarding gene drive experiments in the laboratory[J]. Science, 2015, 349(6251): 927-929. DOI:10.1126/science.aac7932 |
| [17] | CDC. Biosafety in Microbiological and Biomedical Laboratories (BMBL): 5th edition[R]. U.S. Department of Health and Human Services, 2010. |
| [18] | Charlop-Powers Z, Pregitzer C C, Lemetre C, et al. Urban park soil microbiomes are a rich reservoir of natural product biosynthetic diversity[J]. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(51): 14811-14816. DOI:10.1073/pnas.1615581113 |
| [19] | King G M. Urban microbiomes and urban ecology: how do microbes in the built environment affect human sustainability in cities?[J]. Journal of Microbiology, 2014, 52(9): 721-728. DOI:10.1007/s12275-014-4364-x |
| [20] | Brodie E L, DeSantis T Z, Parker J P M, et al. Urban aerosols harbor diverse and dynamic bacterial populations[J]. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(1): 299-304. DOI:10.1073/pnas.0608255104 |
| [21] | Griffin D W, Kellogg C A. Dust storms and their impact on ocean and human health: dust in earth's atmosphere[J]. EcoHealth, 2004, 1(3): 284-295. DOI:10.1007/s10393-004-0120-8 |
| [22] | Kellogg C A, Griffin D W. Aerobiology and the global transport of desert dust[J]. Trends in Ecology & Evolution, 2006, 21(11): 638-644. |
| [23] | Ramirez K S, Leff J W, Barberán A, et al. Biogeographic patterns in below-ground diversity in New York City's Central Park are similar to those observed globally[J]. Proceeding of the Royal Society B: Biological Sciences, 2014, 281(1795). DOI:10.1098/rspb.2014.1988 |
| [24] | Bowers R M, McLetchie S, Knight R, et al. Spatial variability in airborne bacterial communities across land-use types and their relationship to the bacterial communities of potential source environments[J]. The ISME Journal, 2011, 5(4): 601-612. DOI:10.1038/ismej.2010.167 |
| [25] | Fujimura K E, Johnson C C, Ownby D R, et al. Man's best friend? The effect of pet ownership on house dust microbial communities[J]. Journal of Allergy and Clinical Immunology, 2010, 126(2): 410-412. DOI:10.1016/j.jaci.2010.05.042 |
| [26] | Frankel M, Bekö G, Timm M, et al. Seasonal variations of indoor microbial exposures and their relation to temperature, relative humidity, and air exchange rate[J]. Applied and Environmental Microbiology, 2012, 78(23): 8289-8297. DOI:10.1128/AEM.02069-12 |
| [27] | Berg G, Mahnert A, Moissl-Eichinger C, et al. Beneficial effects of plant-associated microbes on indoor microbiomes and human health?[J]. Frontiers in Microbiology, 2014, 5. DOI:10.3389/fmicb.2014.00015 |
| [28] | Leung M H Y, Wilkins D, Li E K T, et al. Indoor-air microbiome in an urban subway network: diversity and dynamics[J]. Applied and Environmental Microbiology, 2014, 80(21): 6760-6770. DOI:10.1128/AEM.02244-14 |
| [29] | Kang K, Ni Y, Li J, et al. The environmental exposures and inner-and intercity traffic flows of the metro system may contribute to the skin microbiome and resistome[J]. Cell Reports, 2018, 24(5): 1190-1202. DOI:10.1016/j.celrep.2018.06.109 |
| [30] | Ege M J, Mayer M, Normand A C, et al. Exposure to environmental microorganisms and childhood asthma[J]. The New England Journal of Medicine, 2011, 364(8): 701-709. DOI:10.1056/NEJMoa1007302 |
| [31] | Gilbert J A, Stephens B. Microbiology of the built environment[J]. Nature Reviews Microbiology, 2018, 16(11): 661-670. DOI:10.1038/s41579-018-0065-5 |
| [32] | Rook G A. Regulation of the immune system by biodiversity from the natural environment: an ecosystem service essential to health[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(46): 18360-18367. DOI:10.1073/pnas.1313731110 |
| [33] |
张玲霞, 王永怡, 王姝, 等. 聚焦当今传染病[J]. 传染病信息, 2012, 25(1): 1-6. Zhang L X, Wang Y Y, Wang S, et al. Focus on current infectious diseases[J]. Infectious Disease Information, 2012, 25(1): 1-6. DOI:10.3969/j.issn.1007-8134.2012.01.001 |
| [34] | Microbiology by numbers[J]. Nature Reviews Microbiology, 2011, 9(9): 628. |
| [35] | Gibb R, Redding D W, Chin K Q, et al. Zoonotic host diversity increases in human-dominated ecosystems[J]. Nature, 2020, 584(7821): 398-402. DOI:10.1038/s41586-020-2562-8 |
| [36] | Mody L, Washer L L, Kaye K S, et al. Multidrug-resistant organisms in hospitals: what is on patient hands and in their rooms?[J]. Clinical Infectious Diseases, 2019, 69(11): 1837-1844. DOI:10.1093/cid/ciz092 |
| [37] | Rizzo L, Manaia C, Merlin C, et al. Urban wastewater treatment plants as hotspots for antibiotic resistant bacteria and genes spread into the environment: a review[J]. Science of the Total Environment, 2013, 447: 345-360. DOI:10.1016/j.scitotenv.2013.01.032 |
| [38] | Su J Q, An X L, Li B, et al. Metagenomics of urban sewage identifies an extensively shared antibiotic resistome in China[J]. Microbiome, 2017, 5. DOI:10.1186/s40168-017-0298-y |
| [39] | An X L, Su J Q, Li B, et al. Tracking antibiotic resistome during wastewater treatment using high throughput quantitative PCR[J]. Environment International, 2018, 117: 146-153. DOI:10.1016/j.envint.2018.05.011 |
| [40] | Pu Q, Zhao L X, Li Y T, et al. Manure fertilization increase antibiotic resistance in soils from typical greenhouse vegetable production bases, China[J]. Journal of Hazardous Materials, 2020, 391. DOI:10.1016/j.jhazmat.2020.122267 |
| [41] | Zhu B K, Chen Q L, Chen S C, et al. Does organically produced lettuce harbor higher abundance of antibiotic resistance genes than conventionally produced?[J]. Environment International, 2017, 98: 152-159. DOI:10.1016/j.envint.2016.11.001 |
| [42] | Johnson C K, Hitchens P L, Pandit P S, et al. Global shifts in mammalian population trends reveal key predictors of virus spillover risk[J]. Proceeding of the Royal Society B: Biological Sciences, 2020, 287(1924). DOI:10.1098/rspb.2019.2736 |
| [43] | Williams S H, Che X Y, Paulick A, et al. New York city house mice (Mus musculus) as potential reservoirs for pathogenic bacteria and antimicrobial resistance determinants[J]. mBio, 2018, 9(2). DOI:10.1128/mBio.00624-18 |
| [44] | Cao J, Hu Y F, Liu F, et al. Metagenomic analysis reveals the microbiome and resistome in migratory birds[J]. Microbiome, 2020, 8(1). DOI:10.1186/s40168-019-0781-8 |
| [45] |
房文艳, 高新磊, 李继, 等. 城市社区农贸市场空气微生物及抗生素抗性基因研究[J]. 生态毒理学报, 2015, 10(5): 95-99. Fang W Y, Gao X L, Li J, et al. Investigation on airborne bacteria and antibiotic resistance genes in urban community farmers market[J]. Asian Journal of Ecotoxicology, 2015, 10(5): 95-99. |
| [46] | Becker D J, Streicker D G, Altizer S. Linking anthropogenic resources to wildlife-pathogen dynamics: a review and meta-analysis[J]. Ecology Letters, 2015, 18(5): 483-495. DOI:10.1111/ele.12428 |
| [47] | Murray M H, Hill J, Whyte P, et al. Urban compost attracts coyotes, contains toxins, and may promote disease in urban-adapted wildlife[J]. Ecohealth, 2016, 13(2): 285-292. DOI:10.1007/s10393-016-1105-0 |
| [48] | Cunha B A, Burillo A, Bouza E. Legionnaires' disease[J]. The Lancet, 2016, 387(10016): 376-385. DOI:10.1016/S0140-6736(15)60078-2 |
| [49] | Allen T, Murray K A, Zambrana-Torrelio C, et al. Global hotspots and correlates of emerging zoonotic diseases[J]. Nature Communications, 2017, 8(1). DOI:10.1038/s41467-017-00923-8 |
| [50] | Afshinnekoo E, Meydan C, Chowdhury S, et al. Geospatial resolution of human and bacterial diversity with city-scale metagenomics[J]. Cell Systems, 2015, 1(1): 72-87. DOI:10.1016/j.cels.2015.01.001 |
| [51] | Hsu T, Joice R, Vallarino J, et al. Urban transit system microbial communities differ by surface type and interaction with humans and the environment[J]. mSystems, 2016, 1(3). DOI:10.1128/mSystems.00018-16 |
| [52] | Hsu T, Tiffany R, Vallarino J, et al. Urban transit system microbial communities differ by surface type and interaction with humans and the environment[J]. MSystems, 2016, 1(3). DOI:10.1128/mSystems.00018-16 |
| [53] | Ling F Q, Hwang C, LeChevallier M W, et al. Core-satellite populations and seasonality of water meter biofilms in a metropolitan drinking water distribution system[J]. The ISME Journal, 2016, 10(3): 582-595. DOI:10.1038/ismej.2015.136 |
| [54] | The MetaSUB International Consortium. The metagenomics and metadesign of the subways and urban biomes (METASUB) international consortium inaugural meeting report[J]. Microbiome, 2016, 4(1). DOI:10.1186/s40168-016-0168-z |
| [55] | UN EP, International Livestock Research Institute. Preventing the next pandemic: zoonotic diseases and how to break the chain of transmission[J]. Nairobi, Kenya: UNEP, International Livestock Research Institute, 2020. |
| [56] | Mazar Y, Cytryn E, Erel Y, et al. Effect of Dust storms on the atmospheric microbiome in the eastern mediterranean[J]. Environmental Science & Technology, 2016, 50(8): 4194-4202. |
| [57] | Zhou H, Wang X L, Li Z H, et al. Occurrence and distribution of urban dust-associated bacterial antibiotic resistance in northern China[J]. Environmental Science & Technology Letters, 2018, 5(2): 50-55. |
| [58] | Hu J L, Zhao F Z, Zhang X X, et al. Metagenomic profiling of ARGs in airborne particulate matters during a severe smog event[J]. Science of the Total Environment, 2018, 615: 1332-1340. DOI:10.1016/j.scitotenv.2017.09.222 |
| [59] | Li J, Cao J J, Zhu Y G, et al. Global survey of antibiotic resistance genes in air[J]. Environmental Science & Technology, 2018, 52(19): 10975-10984. |
| [60] | Pal C, Bengtsson-Palme J, Kristiansson E, et al. The structure and diversity of human, animal and environmental resistomes[J]. Microbiome, 2016, 4(1). DOI:10.1186/s40168-016-0199-5 |
| [61] | Xie J W, Jin L, He T T, et al. Bacteria and antibiotic resistance genes (ARGs) in PM2.5 from China: implications for human exposure[J]. Environmental Science & Technology, 2019, 53(2): 963-972. |
| [62] | Xie J W, Jin L, Luo X S, et al. Seasonal disparities in airborne bacteria and associated antibiotic resistance genes in PM2.5 between urban and rural sites[J]. Environmental Science & Technology Letters, 2018, 5(2): 74-79. |
| [63] | Reynolds K A, Mena K D, Gerba C P. Risk of waterborne illness via drinking water in the United States[A]. In: Whitacre D M (Ed. ). Reviews of Environmental Contamination and Toxicology[M]. New York: Springer, 2008. 117-158. |
| [64] | Sinigalliano C D, Gidley M L, Shibata T, et al. Impacts of Hurricanes Katrina and Rita on the microbial landscape of the New Orleans area[J]. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(21): 9029-9034. DOI:10.1073/pnas.0610552104 |
| [65] | Wang H J, Yang X Y, Chen Q F, et al. Response of prokaryotic communities to extreme precipitation events in an urban coastal lagoon: a case study of Yundang lagoon, China[J]. Science of the Total Environment, 2020, 706. DOI:10.1016/j.scitotenv.2019.135937 |
| [66] | Yu P F, Zaleski A, Li Q L, et al. Elevated levels of pathogenic indicator bacteria and antibiotic resistance genes after Hurricane Harvey's flooding in Houston[J]. Environmental Science & Technology Letters, 2018, 5(8): 481-486. |
| [67] | Zhu G B, Wang X M, Yang T, et al. Air pollution could drive global dissemination of antibiotic resistance genes[J]. The ISME Journal, 2020. DOI:10.1038/s41396-020-00780-2 |
| [68] | WHO. Quantitative microbial risk assessment: application for water safety management[R]. Geneva: WHO, 2016. |
| [69] |
张振兴, 王江权, 郑祥. 水体病原微生物定量风险评价: 历史、现状与发展趋势[J]. 环境科学学报, 2016, 36(1): 2-15. Zhang Z X, Wang J Q, Zheng X. Quantitative microbial risk assessment of waterborne pathogens: History and progress[J]. Acta Scientiae Circumstantiae, 2016, 36(1): 2-15. |
| [70] | Zhang Q, Gallard J, Wu B L, et al. Synergy between quantitative microbial source tracking (qMST) and quantitative microbial risk assessment (QMRA): a review and prospectus[J]. Environment International, 2019, 130. DOI:10.1016/j.envint.2019.03.051 |
| [71] | Ahmed W, Hamilton K A, Lobos A, et al. Quantitative microbial risk assessment of microbial source tracking markers in recreational water contaminated with fresh untreated and secondary treated sewage[J]. Environment International, 2018, 117: 243-249. DOI:10.1016/j.envint.2018.05.012 |
| [72] | Xiao S M, An W, Chen Z M, et al. The burden of drinking water-associated cryptosporidiosis in China: the large contribution of the immunodeficient population identified by quantitative microbial risk assessment[J]. Water Research, 2012, 46(13): 4272-4280. DOI:10.1016/j.watres.2012.05.012 |
| [73] |
陈磊, 李蕾芳, 郅晓沙, 等. 北运河粪源微生物分布特征及健康风险评价[J]. 环境科学, 2019, 40(2): 633-639. Chen L, Li L F, Zhi X S, et al. Pollution characteristics and health risk assessment of microorganism pollutions in the Beiyun River[J]. Environmental Science, 2019, 40(2): 633-639. |
| [74] |
王江权, 康敉, 郑祥, 等. 海河流域典型河流粪源性指示微生物的污染特征及其时空分布[J]. 环境科学学报, 2017, 37(1): 138-145. Wang J Q, Kang M, Zheng X, et al. Occurrence and temporal-spatial distribution of fecal indicator microorganisms in three rivers of the Haihe River Basin[J]. Acta Scientiae Circumstantiae, 2017, 37(1): 138-145. |
| [75] | Ashbolt N J, Amézquita A, Backhaus T, et al. Human Health Risk Assessment (HHRA) for environmental development and transfer of antibiotic resistance[J]. Environmental Health Perspectives, 2013, 121(9): 993-1001. DOI:10.1289/ehp.1206316 |
| [76] | Martínez J L, Coque T M, Baquero F. What is a resistance gene? Ranking risk in resistomes[J]. Nature Reviews Microbiology, 2015, 13(2): 116-123. DOI:10.1038/nrmicro3399 |
| [77] | Sun J, Liao X P, D'Souza A W, et al. Environmental remodeling of human gut microbiota and antibiotic resistome in livestock farms[J]. Nature Communications, 2020, 11(1). DOI:10.1038/s41467-020-15222-y |
| [78] | Leonard A F C, Zhang L H, Balfour A J, et al. Exposure to and colonisation by antibiotic-resistant E. coli in UK coastal water users: environmental surveillance, exposure assessment, and epidemiological study (Beach Bum Survey)[J]. Environment International, 2018, 114: 326-333. DOI:10.1016/j.envint.2017.11.003 |
| [79] | 崔亮亮, 杜艳君, 李湉湉. 环境健康风险评估方法第二讲危害识别(续一)[J]. 环境与健康杂志, 2015, 32(4): 362-365. |
| [80] |
安新丽, 苏建强. 活性污泥抗生素抗性基因研究进展[J]. 微生物学通报, 2019, 46(8): 2069-2079. An X L, Su J Q. Resistome in activated sludge: current knowledge and future directions[J]. Microbiology China, 2019, 46(8): 2069-2079. |
| [81] | Carlson C J, Kracalik I T, Ross N, et al. The global distribution of Bacillus anthracis and associated anthrax risk to humans, livestock and wildlife[J]. Nature Microbiology, 2019, 4(8): 1337-1343. DOI:10.1038/s41564-019-0435-4 |
| [82] |
潘恩春. 从SARS流行剖析我国现行疫情报告系统[J]. 疾病控制杂志, 2004, 8(5): 435-437. Pan E C. Analysis on present reporting system from the prevalence of SARS[J]. Chinese Journal of Disease Control & Prevention, 2004, 8(5): 435-437. DOI:10.3969/j.issn.1674-3679.2004.05.017 |
| [83] | An X L, Wang J Y, Pu Q, et al. High-throughput diagnosis of human pathogens and fecal contamination in marine recreational water[J]. Environmental Research, 2020, 190. DOI:10.1016/j.envres.2020.109982 |
| [84] | Lee Y J, van Nostrand J D, Tu Q C, et al. The PathoChip, a functional gene array for assessing pathogenic properties of diverse microbial communities[J]. The ISME Journal, 2013, 7(10): 1974-1984. DOI:10.1038/ismej.2013.88 |
| [85] | Khan Mirzaei M, Xue J L, Costa R, et al. Challenges of studying the human virome-relevant emerging technologies[J]. Trends in Microbiology, 2020. DOI:10.1016/j.tim.2020.05.021 |
 2021, Vol. 42
2021, Vol. 42


