2. 河海大学环境学院, 南京 210098
2. College of Environment, Hohai University, Nanjing 210098, China
硅藻是一种真核单细胞藻类, 在湖泊、河流、海洋、河口和极地冰原等生境广泛分布[1].硅藻种类繁多、生命周期短、繁殖能力强且对环境变化敏感, 可作为主要的初级生产者直接影响食物链中较高营养等级生物的生长代谢, 在水域生态系统的物质循环和能量流动中发挥极其重要的作用[1~4].硅藻的多样性与生态分布一直以来都是环境微生物领域研究的热点问题.已有大量研究关注海洋中的硅藻资源及其在碳循环中的作用, 以及硅藻对于环境变化的响应[2, 5~7], 然而, 目前河流中硅藻的地理分布模式和群落建成机制仍不清楚.此外, 由于微生物群落在水体和沉积物两种环境介质中差异显著[8, 9], 以往研究大多仅针对单一介质(水体或沉积物)的微生物分布特征[3, 4, 10], 而河流中浮游硅藻和底栖硅藻的比较研究尚有不足.
环境筛选作用(与环境异质性相关)和扩散限制作用(与空间距离相关)被认为是影响微生物地理分布格局的重要驱动因素[11].目前关于硅藻生态分布的主导控制因素仍存在争议, 对于环境选择和扩散限制对硅藻分布的相对贡献, 不同研究得出的结论也不尽相同[12~15].如Jamoneau等[4]的研究表明, 法国全境的河流中底栖硅藻群落主要受环境筛选作用的影响, 而Virtanen等[16]研究了芬兰4个盆地中河流底栖硅藻的群落分布, 其发现扩散限制作用对硅藻群落的影响大于环境筛选作用.已有的这些对硅藻分布格局驱动过程的研究大多集中在支流、小型河流、河段和河口区域[17~19], 而大型河流中浮游硅藻和底栖硅藻分布的主导控制过程仍不清楚.除环境选择和扩散限制这两种非生物过程以外, 生物过程(物种间相互作用)也是影响微生物生态分布的重要因素[20, 21].随着高通量测序技术的发展和生物信息学分析方法的不断深入, 基于18S rRNA基因扩增子测序的真核生物解析正逐渐成为硅藻多样性研究的重要技术手段[22, 23].以微生物测序数据集为基础, 共现网络分析已成为解析微生物种间相互作用关系的重要手段[21, 24].然而, 目前对大型河流中浮游硅藻和底栖硅藻种间互作关系的研究仍有待加强.
澜沧江-湄公河是亚洲第一的国际河流, 位于中国境内的澜沧江流经青海、西藏和云南三省, 是西南地区的典型大河之一.澜沧江流域水能资源丰富, 在云南境内干流沿线规划了16座梯级水电站, 其中有7座电站目前正在运行, 形成了首尾相连的深水、梯级水库群.梯级水电开发改变了河流原有的流动性[25], 影响了库区水环境特征, 同时对河流微生物的地理分布格局也产生影响[26].有研究发现, 澜沧江水体浮游和底栖细菌群落组成受梯级大坝影响显著, 环境筛选作用是浮游和底栖细菌分布的主要驱动因素[26, 27], 而关于澜沧江硅藻的相关研究仍十分缺乏.本文从18S rRNA基因扩增子测序数据中获得硅藻门物种信息, 研究澜沧江干流浮游硅藻和底栖硅藻的群落结构和多样性, 探明影响硅藻群落结构的关键环境因子, 分析环境筛选作用和扩散限制作用对两类硅藻生态分布的贡献, 基于共现网络分析探究澜沧江硅藻的种间互作关系.本研究结果补充了大型河流浮游硅藻和底栖硅藻多样性和生态分布的认知不足, 同时有助于理解梯级水电开发下河流硅藻的生态响应.
1 材料与方法 1.1 样品采集及处理于2017年2月沿澜沧江干流进行水体和沉积物样品采集, 研究河段范围从上游盐井(YJ)至出境断面勐腊(ML), 覆盖河流长度为1 200 km, 海拔梯度为420~2 600 m.共布设采样点位35个, 包括上游自然河段(目前未建大坝)10个点位(从YJ至DHQ)和下游梯级库区影响段25个点位(从Mwei至ML), 其中在已建大坝的点位分别设置坝前(BQ即大坝上游5 km范围内)和坝下(BX即大坝下游5 km范围内)采样点, 各样点示意如图 1所示, 具体的样点信息参照文献[26].
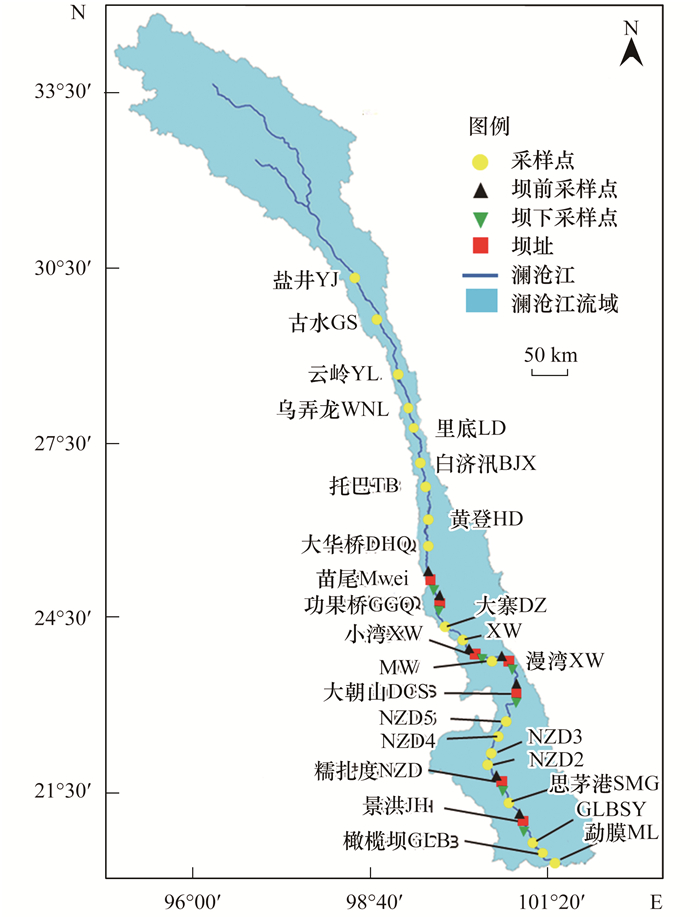
|
图 1 澜沧江采样点分布示意 Fig. 1 Distribution of Lancang River sampling sites |
使用GPSMAP 62s(Garmin, KS, USA)现场测定每个点位的经度、纬度和海拔, 使用Hydrobios Ruttner采水器(Altenholz, Germany)和UWITEC采泥器分别采集各个样点的表层水样(水深0.5 m)和表层沉积物样品(0~20 cm).每个样点的样品随机取3份, 混合后作为该样点的样品.由于野外采样条件限制, 其中9个点位无法获得沉积物样品, 最终得到35份水样和26份沉积物样.水样采集后立即用0.22 μm聚碳酸酯滤膜过滤收集浮游微生物; 滤膜和沉积物样品在-80℃冰箱保存备用.
使用HQ40d多参数水质分析仪(Hach, CO, USA)现场测量水温(t)、pH和溶解氧(DO), 使用2100P浊度仪(Hach, CO, USA)现场测定水体浊度.水样的总氮(TN)、硝氮(NO3-)、氨氮(NH4+)、总磷(TP)、可溶性活性磷(SRP)、可溶性硅酸盐(DSi)和可溶性有机碳(DOC)参照文献[27, 28]的方法进行测定.沉积物的pH、电导率(Ec)、总氮(TN)、硝氮(NO3-)、氨氮(NH4+)、总磷(TP)、总碳(TC)和总有机碳(TOC)参照文献[29]的方法进行测定.采用钼酸盐蓝分光光度法测定沉积物样品中的活性硅酸盐(RSi)和生物硅酸盐(BSi)含量[30, 31].各样品的理化指标信息已在文献[26, 27]中详细列出.
1.2 DNA提取和高通量测序收集水体微生物的滤膜用无菌剪刀剪碎后, 用PowerWater DNA提取试剂盒(MoBio, CA, USA)提取浮游微生物总基因组DNA; 沉积物DNA提取使用FastDNA SPIN试剂盒(MP Biomedicals, CA, USA), 整个提取操作步骤参照试剂盒说明.参照Xue等[21]和Malviya等[23]的研究, 使用带barcode的特异性引物528F(5′-GCGGTAATTCCAGCTCCAA-3′)和706R(5′-AATCCRAGAATTTCACCTCT-3′)对18S rRNA基因的V4高变区进行扩增.参照文献[28, 32]所述进行基因文库的构建, 将每个样品的PCR产物以等摩尔量混合到一起, 文库检测合格后, 使用Ion S5TM XL平台(Thermo Fisher Scientific)上机测序.
根据样品特异性片段和扩增引物序列从下机数据中拆分出各个样品的数据, 截去特异性片段和引物序列, 对每个样品的数据使用FLASH 1.2.7进行拼接, 得到原始序列, 使用QIIME平台(v.1.8.0)对原始测序序列进行过滤, 以获得高质量序列[33].使用UCHIME算法将读取的序列与Silva数据库进行比较, 以检测去除嵌合体序列[34].使用UPARSE(v.7.1)将过滤后的高质量序列按照97%的相似度聚类成可操作分类单元(operational taxonomic units, OTUs), 选择每个OTU中出现频数最高的序列作为代表序列.利用RDP分类器的Silva数据库对OTUs代表序列进行物种注释, 获得真核生物的分类学信息[34].使用MUSCLE软件(v.3.8.31)进行了快速多序列比对, 得到所有OTU代表序列的系统发生关系, 最后以序列最少的样本为标准将OTU进行均一化处理, 最大程度地减少样本间的不同测序深度的影响.参照Malviya等[23]的研究, 从测序得到的真核生物数据集中选择物种注释属于硅藻门的OTUs, 用于后续分析.
1.3 统计分析使用R3.2.1软件的“vegan”程序包, 用物种注释属于硅藻门的OTUs数值计算各样品的α多样性指数(物种丰富度); Beta多样性根据样品之间的Bray-Curtis距离计算, 并且使用了非度量多维尺度分析(NMDS)可视化不同样品之间硅藻群落结构的差异, 使用ANOSIM分析检测各群落之间组成差异的显着性.使用SPSS 22.0软件, 进行α多样性与环境因子的Pearson相关分析.
使用R中的“vegan”包进行Mantel检验, 分析单个地化指标与硅藻群落结构的相关性.参照Chen等[26]的方法进行硅藻群落相似度和地理距离、硅藻群落相似度和环境差异性的距离衰减分析, 其中硅藻群落相似性用Bray-Curtis距离计算, 环境差异性使用欧式距离计算.使用R中的“vegan”软件包进行方差分解分析(variance partitioning analysis, VPA), 计算地理距离和环境筛选对硅藻群落结构的解释率, 其中地理距离基于PCNM变量, 它是由R中“vegan”包的PCNM函数对每个采样点的经纬度计算得到.
为探究硅藻群落的种间相互作用关系, 使用Cytoscape软件(v.3.7.1), 根据测序数据中硅藻门OTUs的相对丰度分别构建了浮游硅藻和底栖硅藻在全河段、上游自然河段和梯级库区段的共现网络(共6个网络).选择了4种方法(Pearson、Spearman、Bray-Curtis和Kullback-Leibler)用于确定OTUs之间的相关关系[35].每个节点代表一个OTU, 每条边代表两个节点之间的显著相关性.使用R中的“igraph”程序包计算网络拓扑指标, 包括节点和边的数量, 平均路径长度, 平均聚类系数, 平均连接性, 图密度, 平均度和模块化[36], 同时使用了交互式平台Gephi(v.0.9.2)实现共现网络的可视化[37].
2 结果与分析 2.1 澜沧江浮游硅藻和底栖硅藻的α多样性本研究从35个水体样品中共得到了218 349个浮游硅藻的高质量序列, 从29个沉积物样品中共得到307 930个底栖硅藻的高质量序列, 这些高质量序列以97%的相似度分别归类成274个和376个OTUs. α多样性(物种丰富度)分析结果显示, 底栖硅藻多样性在澜沧江的上游自然河段显著高于梯级库区影响河段[图 2(e)]; 而浮游硅藻多样性在上游自然河段(YJ至DHQ)逐渐增加, 进入梯级库区后自Mwei至NZD2逐渐下降, 但在下游河段NZDBQ至ML逐渐上升[图 2(a)].可见, 澜沧江两类硅藻α多样性的沿程变化存在较大差异.
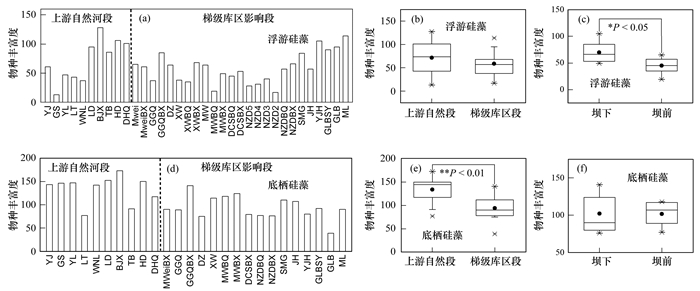
|
图 2 澜沧江浮游硅藻和底栖硅藻物种丰富度指数的沿程变化 Fig. 2 Species richness index of planktonic and sedimentary diatoms in Lancang River |
为进一步探明建坝对河流硅藻多样性的影响, 本研究对比分析了单个大坝坝前(BQ)和坝下(BX)样点的物种丰富度指标差异.结果表明, 底栖硅藻多样性在BQ和BX样点间并无明显差异[图 2(f)], 对于浮游硅藻而言, 除Mwei(苗尾), 其余6个大坝BQ样点的物种丰富度均低于BX样点, 说明建坝降低了库区水体中浮游硅藻的多样性. Pearson相关性分析结果显示(表 1), 浮游硅藻的物种丰富度与水体中的可溶性硅酸盐含量显著相关(r=0.34, P<0.05), 而与其他地理化学指标的相关性并不显著; 底栖硅藻的物种丰富度与采样点海拔(r=0.62, P<0.01)、沿程采样距离(r=-0.63, P<0.01)、pH(r=0.42, P<0.05)、总氮(r=-0.06, P<0.01)、总有机碳(r=-0.58, P<0.01)和硝态氮(r=-0.48, P<0.05)呈显著相关.
|
|
表 1 澜沧江浮游硅藻和底栖硅藻的物种丰富度与地化指标的Pearson相关性1) Table 1 Pearson's correlation of species richness and geochemical variables of planktonic and sedimentary diatoms in Lancang River |
2.2 澜沧江浮游硅藻和底栖硅藻的β多样性
基于Bray-Curtis距离, 本研究用NMDS显示所有水样和沉积物样品中硅藻群落结构差异.如图 3(a)所示, 所有样品在NMDS上总体分为4组, 分别为上游自然段水体、梯级库区影响段水体、上游自然段沉积物和梯级库区影响段沉积物.根据NMDS分组结果, 对浮游硅藻和底栖硅藻进行ANOSIM、MRPP和ADONIS差异性分析结果显示(表 2), 两类硅藻的群落结构差异显著(P<0.01), 并且对于同一类硅藻而言, 其群落组成在上游自然河段和梯级库区影响段间存在显著差异(P<0.01).
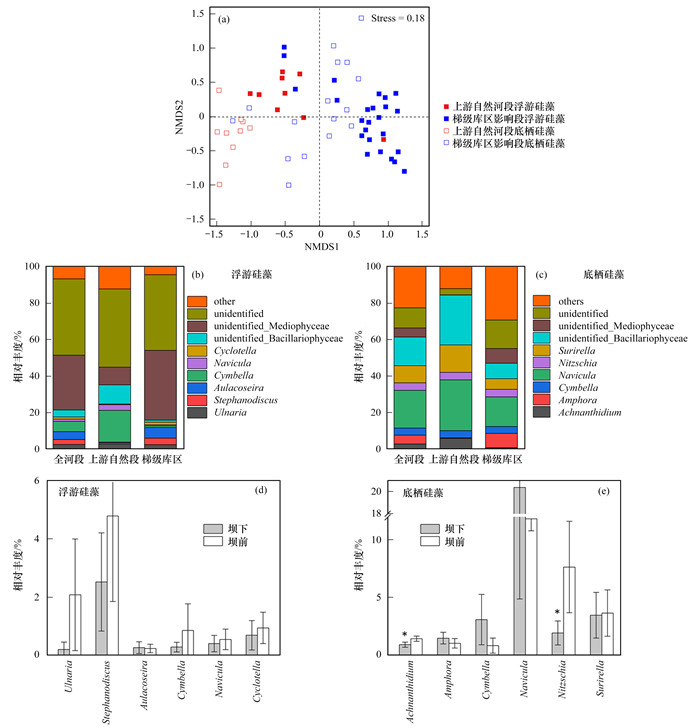
|
(a)NMDS分析; (b)~(e)优势属的相对丰度 图 3 澜沧江浮游硅藻和底栖硅藻群落结构的NMDS分析和优势属的相对丰度 Fig. 3 NMDS analysis of community compositions of planktonic and sedimentary diatoms in Lancang River and relative abundances of the dominant diatom genera |
|
|
表 2 澜沧江浮游和底栖硅藻的ANOSIM、MRPP和ADONIS检验1) Table 2 ANOSIM, MRPP, and ADONIS inspection of planktonic and sedimentary diatoms in Lancang River |
根据硅藻的物种注释信息和相对丰度数据, 本研究在属水平上分析了澜沧江优势硅藻类群的分布特征.浮游硅藻注释到属水平的OTUs相对占比较低, 仅占20%左右, 而底栖硅藻注释到属水平的占比可达50%左右.如图 3(b)所示, 优势浮游硅藻主要包括肘形藻属(Ulnaria)、冠盘藻属(Stephanodiscus)、浮生直链藻属(Aulacoseira)、桥弯藻属(Cymbella)、舟形藻属(Navicula)和小环藻属(Cyclotella); 其中上游自然河段舟形藻属和桥弯藻属的相对丰度分别为3.06%和17.4%, 明显高于梯级库区影响段; 而小环藻属、浮生直链藻属和冠盘藻属的相对丰度在梯级库区影响段较高.如图 3(c)所示, 优势底栖硅藻主要包括链状弯壳藻属(Achnanthidium)、双眉藻属(Amphora)、桥弯藻属(Cymbella)、舟形藻属(Navicula)、菱形藻属(Nitzschia)和双菱藻属(Surirella); 其中上游自然河段舟形藻属和双菱藻属的相对丰度分别为27.8%和15.1%, 明显高于梯级库区影响段; 而双眉藻属的相对丰度在梯级库区影响段较高.可见, 浮游硅藻和底栖硅藻的优势种属组成在不同河段间存在明显差异.
单个大坝坝前和坝下样点的优势浮游硅藻属对比结果显示, 浮游硅藻的优势类群相对丰度没有显著变化[图 3(d)], 而优势底栖硅藻的相对丰度在坝前坝下表现出显著差异(P<0.05), 链状弯壳藻属由坝下的0.89%提高至坝前样点的1.42%, 菱形藻属由坝下的1.92%提高至坝前样点的7.64%[图 3(e)].以上结果表明, 建坝对河流中底栖硅藻的优势种属组成产生显著影响.
2.3 澜沧江浮游硅藻和底栖硅藻地理分布的关键驱动因素为探明地理和环境因素对澜沧江硅藻群落结构的影响, 本研究用Mantel检验法分析了不同地化因子与硅藻群落差异性的关系.如表 3所示, 浮游硅藻群落差异性与海拔、经纬度、水温、浊度、pH、DO和营养盐类等地化因子均显著相关(P<0.01), 其中与水温的相关性最高(r=0.53).底栖硅藻群落差异性与距离、经度、纬度和TN的相关系数较高, 分别为0.49、0.47、0.46和0.43, 但与总磷、Ec和生物硅含量无显著相关性(P>0.05). Mantel检验结果说明, 澜沧江浮游硅藻和底栖硅藻的群落结构受地理因素和环境因素共同影响, 而主导的地化因子在两类硅藻间存在差异.
|
|
表 3 Mantel检验显示澜沧江浮游硅藻和底栖硅藻群落差异性与地化因子的关系1) Table 3 Mantel test showing the relationship between the geochemical variables and the planktonic and sedimentary diatomic communities in Lancang River |
为进一步探明地理距离和环境异质性对澜沧江硅藻群落结构的影响, 基于两类硅藻全河段的数据, 通过距离衰减分析建立了硅藻群落相似性与地理距离和环境差异性之间的线性关系(图 4).结果显示, 澜沧江浮游硅藻和底栖硅藻的群落相似性与地理距离和环境异质性均存在显著的负相关关系(P<0.01), 说明浮游硅藻和底栖硅藻的地理分布符合距离-衰减模式, 即随着地理距离和环境异质性的增大, 硅藻群落相似性逐渐降低.本结果也说明澜沧江硅藻群落分布受扩散限制和环境选择共同影响.
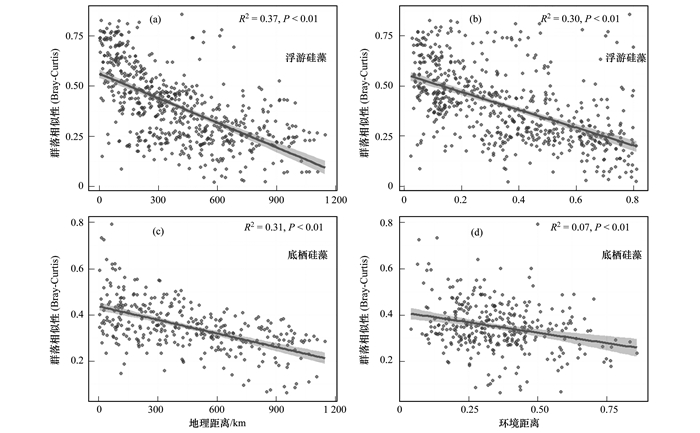
|
图 4 澜沧江浮游硅藻和底栖硅藻与地理距离和环境差异性的距离衰减关系 Fig. 4 Distance-decay relationships of planktonic and sedimentary diatoms in Lancang River with geographical distance and environmental distance |
为明确地理距离和环境异质性对浮游硅藻和底栖硅藻群落的相对影响, 本研究采用方差分解分析(VPA)计算两种作用对硅藻群落差异性的解释率.如图 5所示, 两种作用(地理距离和环境异质性)对浮游硅藻和底栖硅藻群落差异性的全部解释率分别为38.6%和36.5%, 未解释率分别为61.4%和63.5%.地理距离分别解释了浮游硅藻和底栖硅藻群落差异性的16.7%和29.8%, 显著高于环境异质性的影响(解释了浮游硅藻群落的10.3%, 底栖硅藻群落的2.17%), 说明相比环境筛选, 扩散限制作为主导驱动因素对两类硅藻群落差异性的贡献较大.相比浮游硅藻, 底栖硅藻的地理距离解释率更高, 说明底栖硅藻比浮游硅藻更易受扩散限制的影响.
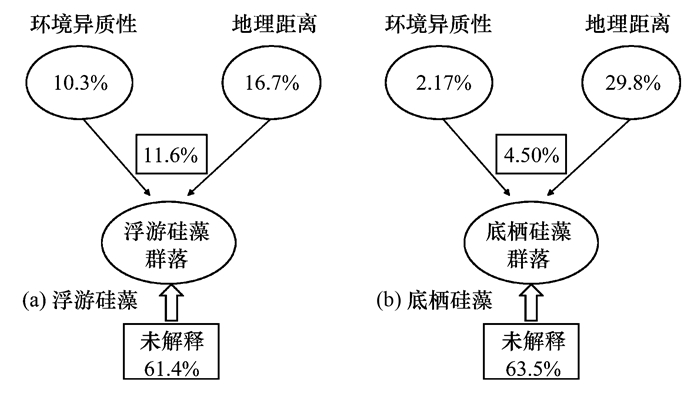
|
图 5 方差分解分析显示地理距离和环境异质性对浮游硅藻和底栖硅藻群落差异性的解释率 Fig. 5 Variance partitioning analysis of the proportion of variation in planktonic and sedimentary diatomic communities that can be explained by environment dissimilarity and geographic distance |
为探明澜沧江浮游硅藻和底栖硅藻的种间互作关系, 本研究分别对全河段、上游自然河段和梯级库区段的水体和沉积物样品中硅藻门OTUs进行共现网络分析, 共现网络(图 6)和网络拓扑结构指标(表 4)可以直接反映微生物的种间交互关系[38].对于全河段, 浮游硅藻的网络图中有25个节点(OTUs)和57条边(相连的OTUs显著相关), 正相关和负相关占比分别为26.3%和73.4%;底栖硅藻的共现网络显示39个节点和31条边, 其中正相关占比较高(77.4%).在不同河段间的网络分析结果显示, 浮游硅藻上游自然河段的网络中边的数量为39条, 明显高于梯级库区段的13条, 而底栖硅藻的边条数在不同河段差别不大.浮游硅藻上游自然河段网络的正相关占比(92.3%)明显高于梯级库区段(59%), 而底栖硅藻节点间的正相关关系在自然河段和梯级库区段分别占95.7%和100%, 说明浮游硅藻的物种间竞争作用在梯级库区段增强, 而底栖硅藻的种间协作在不同河段中均占主导.此外, 6个网络的模块化为0.47~0.76, 表明所构建的网络具有模块化结构[38]; 平均路径长度为1.65~2.47, 接近网络节点总数的对数, 并且与已报道的显示“小世界”特性的网络平均路径长度在类似范围[38], 说明这6个网络都具有“小世界”属性.
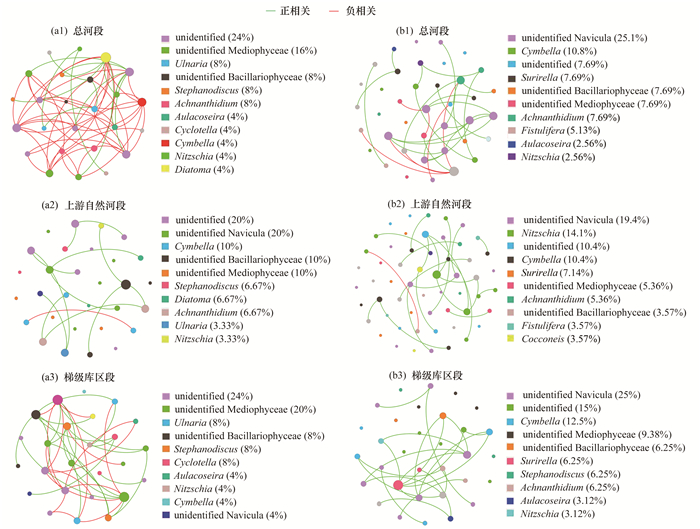
|
(a)浮游硅藻; (b)底栖硅藻 图 6 澜沧江浮游硅藻和底栖硅藻的共现网络 Fig. 6 Co-occurrence networks of planktonic and sedimentary diatomic communities in Lancang River |
|
|
表 4 澜沧江浮游硅藻和底栖硅藻共现网络的关键拓扑结构特征 Table 4 Key topological features of the co-occurrence network of planktonic and sedimentary diatoms in Lancang River |
网络拓扑指标中的密度、聚类系数和平均连通性可以反映物种间相互作用的复杂程度和联通程度[39, 40].相比底栖硅藻, 浮游硅藻的网络密度和平均连通性在全河段、上游自然河段和梯级库区段均较高, 说明浮游硅藻物种间互作关系较为复杂、物种间联通性更强.相比上游自然河段, 两类硅藻的平均度, 聚类系数和图密度在梯级库区段均较高, 表明硅藻物种交互关系在梯级库区段更紧密.浮游硅藻的网络在物种组成方面也是不同于底栖硅藻的, 在浮游硅藻的3个共现网络中, 超过40%的节点属于等片藻属(Diatoma)、冠盘藻属(Stephanodiscus)、浮生直链藻属(Aulacoseira)、桥弯藻属(Cymbella)和小环藻属(Cyclotella), 其中上游自然河段网络的桥弯藻属所占比例(10%)要高于梯级库区影响段(4%), 且上游自然河段网络出现的等片藻属(Diatoma)在梯级库区段消失了.在底栖硅藻的3个共现网络中, 50%以上的节点属于链状弯壳藻属(Achnanthidium)、桥弯藻属(Cymbella)、菱形藻属(Nitzschia)、双菱藻属(Surirella)和浮生直链藻属(Aulacoseira), 其中, 上游的自然河段菱形藻属所占比例(14.1%)高于梯级库区段(3.12%), 而桥弯藻属在梯级库区段的网络中更为丰富(12.5%).
3 讨论文献[41, 42]报道了东江流域底栖硅藻的多样性沿程表现出先上升后下降的趋势, 该研究指出可能是上游自然河段营养盐浓度较低, 沿河流向下人类活动逐渐增加, 营养盐浓度增加, 使得底栖硅藻的多样性增加, 而下游河段的城市化带来了众多环境污染问题, 例如重金属污染, 使得底栖硅藻的多样性被抑制.与上述研究的结果相似, 本研究中浮游硅藻与底栖硅藻的α多样性总体表现为上游自然河段较高, 进入梯级库区后降低(图 2).这种α多样性的沿程变化可能受梯级库区段的人类活动影响, 同样, Chen等[26]和Wang等[27]的研究也指出澜沧江梯级库区段的人类活动可能会导致微生物的多样性降低.河流水体和沉积物中的营养盐是微生物生长代谢的物质基础, 是影响微生物多样性的重要因素[16, 22].基于Pearson分析, 本研究发现浮游硅藻α多样性与水体中溶解性硅酸盐显著正相关, 推测澜沧江上游自然河段风化作用产生的硅酸盐可能是浮游硅藻多样性较高的原因之一.而底栖硅藻多样性与TN、NO3-和TOC浓度呈负相关, 可能是梯级库区影响段中沉积物中营养盐浓度过高, 抑制了底栖硅藻的多样性[22, 26], 而上游自然河段沉积物中营养盐浓度变化不大, 故总体呈现出负相关.
本研究中浮游硅藻和底栖硅藻的群落结构存在显著差异, 这可能是河流中水体和沉积物两种环境介质的不同而导致的[8, 9]. Chen等[26]对澜沧江河岸带细菌地理分布格局的研究结果表明, 沉积物中细菌的群落结构在上游和下游河段之间存在显著差异.根据NMDS和ANOSIM分析结果, 本研究发现浮游硅藻和底栖硅藻群落在上游自然河段和梯级库区段之间也存在显著差异[图 3(a)].物种注释和相对丰度结果表明, 桥弯藻属、舟形藻属和链状弯壳藻属等典型淡水硅藻类群作为优势种属在澜沧江水体和沉积物中广泛存在.有研究发现菱形藻属在高氯离子和硝酸盐浓度的水体中丰度较高[43], 小环藻属和浮生直链藻属多存在于较高水温和SRP浓度的水体中.由于澜沧江梯级库区段水体氮磷营养盐浓度较高[27], 这或许可以解释本研究中浮游小环藻属和浮生直链藻属相对丰度在梯级库区段较高的原因.有研究还发现桥弯藻属和舟形藻属在低浓度氮磷环境中相对丰度最高[44]; 另有研究发现链状弯壳藻属多分布在流速快、营养盐浓度低、污染较小的河源地区[45, 46].本研究中上游自然河段浮游硅藻的桥弯藻属和舟形藻属、以及底栖硅藻的链状弯壳藻相对丰度较高, 可能与澜沧江上游自然河段水体和沉积物中较低的氮磷营养盐含量有关[26, 27].
已有研究通过对比坝前和坝下样点的微生物指标差异来表征单个大坝的影响[27, 47].本研究发现坝前样点的浮游硅藻多样性要低于坝下, 多数坝前样点的底栖硅藻多样性高于坝下, 说明建坝降低了浮游硅藻多样性, 而增加了底栖硅藻多样性.这种建坝引起的浮游硅藻和底栖硅藻多样性的变化在其他微生物类群(如浮游细菌、真菌和固氮菌)中也发现类似的响应规律[27, 29, 47].这可能是大坝阻隔了水体流动, 也阻隔了营养盐的运输[25], 水库内流速缓慢, 生态系统变得相对独立, 环境条件逐渐均一化[48], 并且水库内沉降作用增大, 使得悬浮颗粒大量沉降, 造成了水体的EC和DOC下降, 沉积物中氮磷营养盐含量上升, 导致了水体中硅藻多样性下降, 沉积物内硅藻多样性上升[27, 49], 此外, 定植于悬浮颗粒的浮游硅藻也可能因颗粒沉降作用沉淀到水库底部, 致使浮游硅藻的多样性下降[50].有研究表明, 建坝对微生物优势类群的相对丰度也产生显著影响.例如, Chen等[26]对澜沧江沉积物中细菌的研究, 报道了梯级大坝显著提高了酸杆菌门、疣微菌门和绿弯菌门的相对丰度, 降低了变形菌门和厚壁菌门的相对丰度.本研究中, 建坝对底栖硅藻的优势属造成了显著影响, 显著提高了链状弯壳藻属和菱形藻属的相对丰度.这种影响可能与建坝后库区水动力条件、营养盐浓度和水温等环境特征的变化相关[48, 49].
微生物地理分布格局的关键驱动机制是目前环境微生物学和微生物生态学的研究热点问题[51, 52].本研究的Mantel检验结果表明, 浮游硅藻和底栖硅藻的群落差异性与多种地理因素(如经度、海拔)和环境因素(如pH、TOC、TN等)显著相关(表 3).已有研究报道水温、电导率、pH和营养盐等是硅藻群落组成的关键因素[53~55], 因为水温和营养盐对硅藻的生长和繁殖至关重要, 并且pH和电导率也会影响硅藻的生长和分布.距离衰减模型已被广泛应用于评估环境筛选和扩散限制作用, 二者会极大地影响距离衰减的模式[35]. Wetzel等[56]的研究发现, 巴西内格罗河中的浮游和底栖硅藻群落均有显著的距离衰减关系, 与其研究结果相似, 本研究发现澜沧江的浮游硅藻和底栖硅藻群落均符合距离衰减模型, 与地理距离和环境异质性呈显著负相关(图 4), 说明扩散限制和环境筛选两种过程可能共同影响了澜沧江硅藻的分布.
尽管扩散限制作用和环境筛选作用已被认为是控制微生物地理分布的重要驱动过程[11, 57], 但这两种作用过程对微生物群落建成的相对影响或相对贡献仍不清楚, 关于硅藻群落分布的主导驱动过程(扩散限制或环境筛选作用)也存在争议[12, 13, 58].例如, Urrea等[59]的研究认为, 环境筛选作用对西班牙瓜迪亚纳河中底栖硅藻群落的影响大于扩散限制作用, 而Liu等[10]的研究认为, 中国钱塘江中的底栖硅藻主要受扩散限制作用的影响.根据VPA结果(图 5), 本研究发现扩散限制对澜沧江两类硅藻群落的影响均大于环境筛选.对于这种扩散限制对硅藻的主导驱动作用可以从以下两方面进行解释.一方面, 由于本研究对澜沧江的采样区域空间跨度较大(覆盖河流长度达1200 km), 硅藻在长距离空间尺度上可能难以传播扩散; 另一方面, 梯级大坝的建设影响了河流的连通性, 造成了不同库区间硅藻群落的空间隔离, 进而增加了扩散传播的障碍[29, 60].此外, 相对浮游硅藻, 扩散限制对底栖硅藻地理分布的影响更大, 这与Wetzel等[56]和Wang等[22]对水体和沉积物中硅藻分布的研究结果一致. Wetzel等[56]认为浮游硅藻能够随水流快速迁移, 而底栖硅藻在沉积物中难以扩散传播, 因此扩散限制对于底栖硅藻的群落分布影响更大. Wang等[22]认为建坝造成了河流流速缓慢、河道加深等变化, 这些因素会导致更少的底栖硅藻迁移到水体中, 进一步限制了底栖硅藻的传播扩散.
近年来, 基于数学神经网络模型的微生物共现网络分析, 为理解微生物种群间的相互作用关系提供了新的研究方法和表征手段[21, 24].本研究通过对6个共现网络分析, 探明了澜沧江浮游硅藻和底栖硅藻在全河段、上游自然河段和梯级库区影响段的相互作用关系.在共现网络中, 两个节点间边的正、负相关性分别代表相连OTUs之间的互惠和竞争关系[39].本研究发现浮游硅藻在梯级库区段网络中的负相关占比要高于上游自然河段网络, 说明梯级库区段浮游硅藻种间竞争关系增强, 而协作关系减弱.这个结果与Chen等[47]的研究结果一致, 其研究发现梯级建坝对澜沧江固氮菌的种间共生协作关系产生不利影响.相比浮游硅藻, 底栖硅藻网络的正相关性占比较高, 说明底栖硅藻共生或互惠作用要强于浮游硅藻.网络的拓扑结构特征可以反映微生物种间的连通性[38], 有研究认为具有较高连接性的网络对于环境干扰反应更为迅速[39, 61].本研究发现浮游硅藻的网络连通性比底栖硅藻更高且更为复杂, 由此可推测浮游硅藻可能对澜沧江的环境变化反应更加敏感.从VPA结果也可以发现, 环境异质性对浮游硅藻的影响大于底栖硅藻.此外, 微生物网络的联通性可能代表微生物之间的相互作用或生态位共享[62].本研究发现, 浮游硅藻和底栖硅藻梯级库区段网络的密度、连通性和复杂性均高于上游自然河段网络, 这可能由于建坝等人类活动促进了硅藻群落之间的相互作用或生态位共享.这与Chen等[26]对于细菌的研究相反, 其认为梯级大坝阻碍了细菌的相互作用和耦合.可见, 梯级水电开发的河流中不同微生物类群的共现网络模式可能存在明显区别, 但具体的影响因素和驱动机制仍需进一步研究.
4 结论(1) 澜沧江浮游硅藻和底栖硅藻的α多样性在上游自然河段较高, 两类硅藻表现出不同的地理分布格局, 群落结构和优势属相对丰度在不同河段间差异显著; 建坝降低了浮游硅藻α多样性, 影响了两类硅藻的优势物种组成.
(2) 扩散限制作用和环境筛选作用是澜沧江浮游硅藻和底栖硅藻地理分布格局的重要驱动因子, 而扩散限制作用对两类硅藻分布的影响均较大; 与浮游硅藻相比, 底栖硅藻受到扩散限制的影响更大.
(3) 共现网络分析表明, 相比浮游硅藻, 底栖硅藻物种间协同作用较强, 但网络连通性较弱; 相比上游自然河段, 两类硅藻的网络密度、复杂度和连通性在梯级库区段较高.
| [1] | Raphael S, Matschullat J, Oeste F D. The diatoms:applications for the environmental and earth sciences[J]. Journal of Soils and Sediments, 2002, 2(2): 103-104. DOI:10.1007/BF02987879 |
| [2] | Rabosky D L, Sorhannus U. Diversity dynamics of marine planktonic diatoms across the Cenozoic[J]. Nature, 2009, 457(7226): 183-186. DOI:10.1038/nature07435 |
| [3] | Liess A, Lange K, Schulz F, et al. Light, nutrients and grazing interact to determine diatom species richness via changes to productivity, nutrient state and grazer activity[J]. Journal of Ecology, 2008, 97(2): 326-336. |
| [4] | Jamoneau A, Passy S I, Soininen J, et al. Beta diversity of diatom species and ecological guilds:response to environmental and spatial mechanisms along the stream watercourse[J]. Freshwater Biology, 2018, 63(1): 62-73. DOI:10.1111/fwb.12980 |
| [5] | Armbrust E V. The life of diatoms in the world's oceans[J]. Nature, 2009, 459(7244): 185-192. DOI:10.1038/nature08057 |
| [6] | Kafouris S, Smeti E, Spatharis S, et al. Nitrogen as the main driver of benthic diatom composition and diversity in oligotrophic coastal systems[J]. Science of the Total Environment, 2019, 694: 133773. DOI:10.1016/j.scitotenv.2019.133773 |
| [7] | Cibic T, Comici C, Bussani A. Benthic diatom response to changing environmental conditions[J]. Estuarine, Coastal and Shelf Science, 2012, 115: 158-169. DOI:10.1016/j.ecss.2012.03.033 |
| [8] | Feng B W, Li X R, Wang J H, et al. Bacterial diversity of water and sediment in the Changjiang estuary and coastal area of the East China Sea[J]. FEMS Microbiology Ecology, 2009, 70(2): 236-248. DOI:10.1111/j.1574-6941.2009.00772.x |
| [9] | Xia N, Xia X H, Liu T, et al. Characteristics of bacterial community in the water and surface sediment of the Yellow River, China, the largest turbid river in the world[J]. Journal of Soils and Sediments, 2014, 14(11): 1894-1904. DOI:10.1007/s11368-014-0974-5 |
| [10] | Liu S R, Xie G X, Wang L Z, et al. Different roles of environmental variables and spatial factors in structuring stream benthic diatom and macroinvertebrate in Yangtze River Delta, China[J]. Ecological Indicators, 2016, 61: 602-611. DOI:10.1016/j.ecolind.2015.10.011 |
| [11] | Hanson C A, Fuhrman J A, Horner-Devine M C, et al. Beyond biogeographic patterns:processes shaping the microbial landscape[J]. Nature Reviews Microbiology, 2012, 10(7): 497-506. DOI:10.1038/nrmicro2795 |
| [12] | Zorzal-Almeida S, Soininen J, Bini L M, et al. Local environment and connectivity are the main drivers of diatom species composition and trait variation in a set of tropical reservoirs[J]. Freshwater Biology, 2017, 62(9): 1551-1563. DOI:10.1111/fwb.12966 |
| [13] | Soininen J, Weckström J. Diatom community structure along environmental and spatial gradients in lakes and streams[J]. Fundamental and Applied Limnology/Archiv für Hydrobiologie, 2009, 174(3): 205-213. DOI:10.1127/1863-9135/2009/0174-0205 |
| [14] | Dong X Y, Li B, He F Z, et al. Flow directionality, mountain barriers and functional traits determine diatom metacommunity structuring of high mountain streams[J]. Scientific Reports, 2016, 6(1): 24711. DOI:10.1038/srep24711 |
| [15] | Plante C J, Hill-Spanik K, Lowry J. Environmental and spatial influences on biogeography and community structure of benthic diatoms[J]. Phycologia, 2017, 56(4): 149-150. |
| [16] | Virtanen L, Soininen J. The roles of environment and space in shaping stream diatom communities[J]. European Journal of Phycology, 2012, 47(2): 160-168. DOI:10.1080/09670262.2012.682610 |
| [17] | Liu J, Soininen J, Han B P, et al. Effects of connectivity, dispersal directionality and functional traits on the metacommunity structure of river benthic diatoms[J]. Journal of Biogeography, 2013, 40(12): 2238-2248. DOI:10.1111/jbi.12160 |
| [18] | Kheiri S, Solak C N, Edlund M B, et al. Biodiversity of diatoms in the Karaj River in the Central Alborz, Iran[J]. Diatom Research, 2018, 33(3): 355-380. DOI:10.1080/0269249X.2018.1557747 |
| [19] | Centis B, Tolotti M, Salmaso N. Structure of the diatom community of the River Adige (North-Eastern Italy) along a hydrological gradient[J]. Hydrobiologia, 2010, 639(1): 37-42. DOI:10.1007/s10750-009-0019-z |
| [20] | Fan K K, Cardona C, Li Y T, et al. Rhizosphere-associated bacterial network structure and spatial distribution differ significantly from bulk soil in wheat crop fields[J]. Soil Biology and Biochemistry, 2017, 113: 275-284. DOI:10.1016/j.soilbio.2017.06.020 |
| [21] | Xue Y Y, Chen H H, Yang J R, et al. Distinct patterns and processes of abundant and rare eukaryotic plankton communities following a reservoir cyanobacterial bloom[J]. The ISME Journal, 2018, 12(9): 2263-2277. DOI:10.1038/s41396-018-0159-0 |
| [22] | Wang J W, Liu Q X, Zhao X F, et al. Molecular biogeography of planktonic and benthic diatoms in the Yangtze River[J]. Microbiome, 2019, 7(1): 153. DOI:10.1186/s40168-019-0771-x |
| [23] | Malviya S, Scalco E, Audic S, et al. Insights into global diatom distribution and diversity in the world's ocean[J]. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(11): E1516-E1525. DOI:10.1073/pnas.1509523113 |
| [24] | Chow C E T, Kim D Y, Sachdeva R, et al. Top-down controls on bacterial community structure:Microbial network analysis of bacteria, T4-like viruses and protists[J]. The ISME Journal, 2013, 8(4): 816-829. |
| [25] | Kunz M J, Wüest A, Wehrli B, et al. Impact of a large tropical reservoir on riverine transport of sediment, carbon, and nutrients to downstream wetlands[J]. Water Resources Research, 2011, 47(12): W12531. |
| [26] | Chen J, Wang P F, Wang C, et al. Bacterial communities in riparian sediments:A large-scale longitudinal distribution pattern and response to dam construction[J]. Frontiers in Microbiology, 2018, 9: 999. DOI:10.3389/fmicb.2018.00999 |
| [27] | Wang X, Wang C, Wang P F, et al. How bacterioplankton community can go with cascade damming in the highly regulated Lancang-Mekong River Basin[J]. Molecular Ecology, 2018, 27(22): 4444-4458. DOI:10.1111/mec.14870 |
| [28] | Liu L M, Chen H H, Liu M, et al. Response of the eukaryotic plankton community to the cyanobacterial biomass cycle over 6 years in two subtropical reservoirs[J]. The ISME Journal, 2019, 13(9): 2196-2208. DOI:10.1038/s41396-019-0417-9 |
| [29] | Chen J, Wang P F, Wang C, et al. Fungal community demonstrates stronger dispersal limitation and less network connectivity than bacterial community in sediments along a large river[J]. Environmental Microbiology, 2019, 22(3): 832-849. |
| [30] | DeMaster J D. The supply and accumulation of silica in the marine environment[J]. Geochimica et Cosmochimica Acta, 1981, 45(10): 1715-1732. DOI:10.1016/0016-7037(81)90006-5 |
| [31] | Mortlock R A, Froelich P N. A simple method for the rapid determination of biogenic opal in pelagic marine sediments[J]. Deep Sea Research Part A. Oceanographic Research Papers, 1989, 36(9): 1415-1426. DOI:10.1016/0198-0149(89)90092-7 |
| [32] | Dissanayake A J, Purahong W, Wubet T, et al. Direct comparison of culture-dependent and culture-independent molecular approaches reveal the diversity of fungal endophytic communities in stems of grapevine (Vitis vinifera)[J]. Fungal Diversity, 2018, 90(1): 85-107. DOI:10.1007/s13225-018-0399-3 |
| [33] | Caporaso J G, Kuczynski J, Stombaugh J, et al. QIIME allows analysis of high-throughput community sequencing data[J]. Nature Methods, 2010, 7(5): 335-336. DOI:10.1038/nmeth.f.303 |
| [34] | Edgar R C, Haas B J, Clemente J C, et al. UCHIME improves sensitivity and speed of chimera detection[J]. Bioinformatics, 2011, 27(16): 2194-2200. DOI:10.1093/bioinformatics/btr381 |
| [35] | Xiao X, Liang Y T, Zhou S, et al. Fungal community reveals less dispersal limitation and potentially more connected network than that of bacteria in bamboo forest soils[J]. Molecular Ecology, 2018, 27(2): 550-563. DOI:10.1111/mec.14428 |
| [36] | Csárdi G, Nepusz T. The igraph software package for complex network research[J]. InterJournal Complex Systems, 2006, 1695. |
| [37] | Bastian M, Heymann S, Jacomy M. Gephi: an open source software for exploring and manipulating networks[A]. In: Proceedings of the 3rd International AAAI Conference on Weblogs and Social Media[C]. San Jose: AAAI, 2009.361-362. |
| [38] | Deng Y, Jiang Y H, Yang Y F, et al. Molecular ecological network analyses[J]. BMC Bioinformatics, 2012, 13(1): 113. |
| [39] | Zhang B G, Zhang J, Liu Y, et al. Co-occurrence patterns of soybean rhizosphere microbiome at a continental scale[J]. Soil Biology and Biochemistry, 2018, 118: 178-186. DOI:10.1016/j.soilbio.2017.12.011 |
| [40] | Jiao S, Chen W M, Wei G H. Biogeography and ecological diversity patterns of rare and abundant bacteria in oil-contaminated soils[J]. Molecular Ecology, 2017, 26(19): 5305-5317. DOI:10.1111/mec.14218 |
| [41] | 刘静.东江流域底栖硅藻多样性及集合群落的研究[D].广州: 暨南大学, 2013. |
| [42] | 林罗敏.东江干流底栖硅藻分布特征及生态系统健康评价研究[D].广州: 暨南大学, 2017. |
| [43] | Stenger-Kovács C, Lengyel E, Crossetti L O, et al. Diatom ecological guilds as indicators of temporally changing stressors and disturbances in the small Torna-stream, Hungary[J]. Ecological Indicators, 2013, 24: 138-147. DOI:10.1016/j.ecolind.2012.06.003 |
| [44] | Stenger-Kovács C, Buczkó K, Hajnal é, et al. Epiphytic, littoral diatoms as bioindicators of shallow lake trophic status:Trophic Diatom Index for Lakes (TDIL) developed in Hungary[J]. Hydrobiologia, 2007, 589(1): 141-154. DOI:10.1007/s10750-007-0729-z |
| [45] | Van Dam H, Mertens A, Sinkeldam J. A coded checklist and ecological indicator values of freshwater diatoms from the Netherlands[J]. Netherlands Journal of Aquatic Ecology, 1994, 28(1): 117-133. DOI:10.1007/BF02334251 |
| [46] |
邱礼城, 韦桂峰, 李湘姣, 等. 广东鉴江水系底栖硅藻多样性与时空分布特征[J]. 热带亚热带植物学报, 2016, 24(2): 197-207. Qiu L C, Wei G F, Li X J, et al. Species diversity and temporal-spatial distribution of benthic diatoms in Jianjiang River, Guangdong Province[J]. Journal of Tropical and Subtropical Botany, 2016, 24(2): 197-207. |
| [47] | Chen J, Wang P F, Wang C, et al. Dam construction alters function and community composition of diazotrophs in riparian soils across an environmental gradient[J]. Soil Biology and Biochemistry, 2019, 132: 14-23. DOI:10.1016/j.soilbio.2019.01.020 |
| [48] | Poff N L, Olden J D, Merritt D M, et al. Homogenization of regional river dynamics by dams and global biodiversity implications[J]. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(14): 5732-5737. DOI:10.1073/pnas.0609812104 |
| [49] | Zhang W, Yuan J, Han J Q, et al. Impact of the Three Gorges Dam on sediment deposition and erosion in the middle Yangtze River:a case study of the Shashi Reach[J]. Hydrology Research, 2016, 47(S1): 175-186. DOI:10.2166/nh.2016.092 |
| [50] | Chen J, Wang P F, Wang C, et al. Distinct assembly mechanisms underlie similar biogeographic patterns of rare and abundant bacterioplankton in cascade reservoirs of a large river[J]. Frontiers in Microbiology, 2020, 11: 158. DOI:10.3389/fmicb.2020.00158 |
| [51] | Nemergut D R, Schmidt S K, Fukami T, et al. Patterns and processes of microbial community assembly[J]. Microbiology and Molecular Biology Reviews, 2013, 77(3): 342-356. DOI:10.1128/MMBR.00051-12 |
| [52] | Morrison-Whittle P, Goddard M R. Quantifying the relative roles of selective and neutral processes in defining eukaryotic microbial communities[J]. The ISME Journal, 2015, 9(9): 2003-2011. DOI:10.1038/ismej.2015.18 |
| [53] | Chen X, Zhou W Q, Pickett S T A, et al. Diatoms are better indicators of urban stream conditions:a case study in Beijing, China[J]. Ecological Indicators, 2016, 60: 265-274. DOI:10.1016/j.ecolind.2015.06.039 |
| [54] | Yao M, Li Y L, Yang X D, et al. Three-year changes in planktonic diatom communities in a eutrophic lake in Nanjing, Jiangsu Province, China[J]. Journal of Freshwater Ecology, 2011, 26(1): 133-141. DOI:10.1080/02705060.2011.553932 |
| [55] | Tan X, Xia X L, Zhao Q L, et al. Temporal variations of benthic diatom community and its main influencing factors in a subtropical river, China[J]. Environmental Science and Pollution Research, 2013, 21(1): 434-444. |
| [56] | Wetzel C E, de C., Bicudo D, Ector L, et al. Distance decay of similarity in neotropical diatom communities[J]. PLoS One, 2012, 7(9): e45071. DOI:10.1371/journal.pone.0045071 |
| [57] | Zhang W J, Pan Y B, Yang J, et al. The diversity and biogeography of abundant and rare intertidal marine microeukaryotes explained by environment and dispersal limitation[J]. Environmental Microbiology, 2018, 20(2): 462-476. DOI:10.1111/1462-2920.13916 |
| [58] | Burlig A L, Torgan L C, de Andrade E A N, et al. Changes in diatom associations with altitudinal gradient and land use in Itajaí-Mirim River, Southern Brazil[J]. Iheringia Serie Botanica, 2014, 69(2): 451-464. |
| [59] | Urrea G, Sabater S. Epilithic diatom assemblages and their relationship to environmental characteristics in an agricultural watershed (Guadiana River, SW Spain)[J]. Ecological Indicators, 2009, 9(4): 693-703. DOI:10.1016/j.ecolind.2008.09.002 |
| [60] | Ruiz-González C, Proia L, Ferrera I, et al. Effects of large river dam regulation on bacterioplankton community structure[J]. FEMS Microbiology Ecology, 2013, 84(2): 316-331. DOI:10.1111/1574-6941.12063 |
| [61] | Zhou J Z, Deng Y, Luo F, et al. Functional molecular ecological networks[J]. mBio, 2010, 1(14): e00169-10. |
| [62] | Berry D, Widder S. Deciphering microbial interactions and detecting keystone species with co-occurrence networks[J]. Frontiers in Microbiology, 2014, 5: 219. |
 2020, Vol. 41
2020, Vol. 41


