2. 华南师范大学环境学院, 广州 510006
2. School of Environment, South China Normal University, Guangzhou 510006, China
抗生素的大量使用以及不合理应用促进了耐药基因(antibiotic resistance genes, ARGs)的发展及其在临床、环境中的传播[1, 2].近年来, 抗生素耐药性已得到各国家及地区的广泛关注[3~5].耐药基因和耐药菌(antibiotic resistance bacteria, ARB)随生活废水、医疗废水和畜禽养殖等废水排放迁移至土壤、地表水、沉积物, 通过淋溶、渗透或地表水-地下水相互交换等途径渗透到地下水中[5~8], 从而对地下水形成污染.地下水环境中的耐药基因可通过遗传或者水平转移(horizontal gene transfer, HGT)在细菌间进行扩散, 或通过饮用水和食物链进行转移, 对环境卫生与人体健康形成潜在威胁[9].
地下水在许多国家和地区被用作饮用水或农业用水, 是重要的公共给水来源.城市或农业等人为活动增强了耐药基因在地下水环境中的污染与扩散[10, 11].地下水微生物虽然处于低丰度水平, 却天然携带多种类型质粒, 具有快速适应环境变化的潜在能力[12].在污水处理厂、垃圾填埋场、畜禽养殖场等相关地下水中, 不仅发现了多种抗生素和耐药基因, 也发现粪大肠菌群、假单胞菌属、肠球菌、大肠杆菌等细菌已对多种抗生素形成耐药[13~18].
我国对环境中的抗生素与耐药基因研究集中在土壤、地表水和沉积物[19~23].由于地下水监测的难度与复杂度, 目前对地下水中耐药基因的研究报道还很少[11, 24].笔者前期调查了南方地区东江流域地表水和地下水中药物和个人护理用品(PPCPs)和人工甜味剂(ASs)的分布情况, 研究表明城市活动对地下水造成了严重污染[25], 由此可见, 地下水中耐药基因的分布特征及其潜在风险亦不容忽视.本文选择深圳市民用井为研究对象, 分析我国南方地区浅层地下水中耐药基因的分布特征及生态风险, 以期为地下水的污染防治措施提供基础数据.
1 材料与方法 1.1 研究区域及样品采集深圳市位于北回归线以南, 本地水资源匮乏, 人均水资源拥有量低于世界水危机标准, 是全国严重缺水城市之一, 流域城区的河流普遍受到污染[26].随着人口密度的增长, 地下水开采量不断加大, 成为重要的水资源.据文献[27]的数据, 深圳市地下水资源量为6.38亿m3, 年供水量为318.31万m3, 且全为浅层地下水, 要用于居民生活、农业、矿泉水和工业, 其中农业灌溉用水井和居民生活用水井是主要的取水用途.
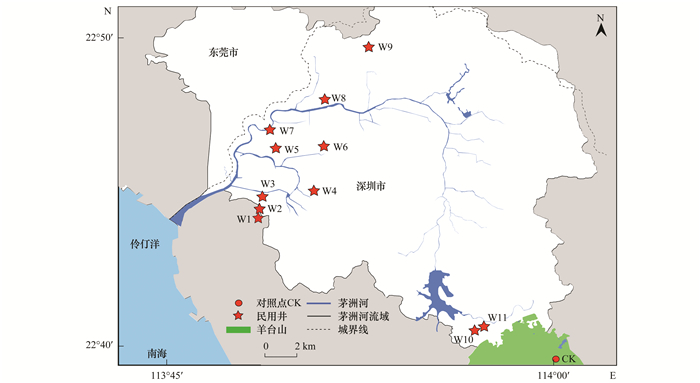
本研究采集深圳市茅洲河流域内11口居民生活区地下水进行分析(图 1).采样涉及宝安区的沙井街道、松岗街道、燕罗街道、江碧工业区以及光明新区的石岩街道.W1~W8主要位于人口较为密集的河流中下游区域, 其中, W1、W2、W3、W5、W6和W8位于居民区院落内, 附近生活人口密集; W4与W7位于居民区菜地旁.W9位于城郊山区某林场, 为数百居民日常生活用水.W10与W11位于河流上游、羊台山脚下.以羊台山泉水为井水环境本底对照样品(CK).采集的井水保存在灭菌处理过的10 L白色不透明聚乙烯塑料瓶中, 并在24 h内前处理完毕.
|
图 1 民用井采样地示意 Fig. 1 Map of sampling wells |
取1.5~2 L水样经0.22 μm孔径混合醋酸酯纤维素膜过滤, 每个样品收集3个平行滤膜.取滤膜用灭菌后的手术剪在无菌操作下剪碎, 按照PowerSoil DNA Isolation Kit (MoBio Laboratories, USA)试剂盒内的操作步骤提取滤膜上的DNA.随后用1.0%琼脂凝胶电泳检测DNA样品的质量和完整性.采用分光光度计(NanoDrop ND-1000, Thermo Scientific, Waltham, MA)对提取的DNA进行浓度与纯度的检测.DNA样品保存在-80℃备用.
1.3 抗生素耐药基因检测与定量采用高通量荧光定量PCR (high-throughput qPCR, HT-qPCR)技术对井水中的抗生素耐药基因(ARGs)进行分析.目标基因为248个环境常见以及临床相关的耐药基因和可移动元件基因, 包括:① 9大类耐药基因:氨基糖苷类(aminoglycosides)、β-内酰胺类(β-lactamases)、氯霉素类(chloramphenicols)、大环内酯类-林可酰胺类-链阳性菌素B类(macrolide- lincosamide-streptogramin B, MLSB)、多重耐药类(multidrug)、磺胺类(sulfonamides)、四环素类(tetracyclines)、万古霉素类(vancomycins)及其他(other); ②10种可移动遗传元件(mobile genetic elements, MGEs): 8种转座子、一类整合子整合酶基因(intl1)、临床一类整合子整合酶基因(cintl1); ③16S rRNA基因.引物设计与验证参考文献[28]进行.
反应体系的总体积为100 nL, 包括1×LightCycler 480 SYBR Gree I Master, 500 nmol·L-1引物, 2 ng·μL-1 DNA模板.反应程序为:预变性95℃, 10 min; 95℃, 30 s; 退火温度60℃, 30 s; 循环40次.每个芯片设置一组阴性对照.目标基因的检测与定量在安徽微分基因有限公司Wafergen Smartchip反应平台进行(Microanaly Genetech Co. Limited, Anhui, China).根据SmartChip Real-Time PCR Systems的仪器性能, 选取CT值< 31, 且在3个平行中有两个及以上检出、偏差 < 20%, 且满足曲线拟合分析的样本判为阳性检出.
1.4 16S rRNA基因丰度测定采用荧光定量PCR仪QuantStudioTM 7 Flex System (ABI, 美国)对样品中16S rRNA基因丰度进行绝对定量.qPCR反应体系为20 μL: 10 μL的2×SYBRⓇ Premix Ex TaqTM (Tli RNaseH Plus), 0.05 mmol·L-1的上下游引物[18]各0.4 μL, 50×ROX参比染料0.4 μL, 2 μL的DNA模板(DNA < 80 ng), 以及6.8 μL灭菌超纯水.qPCR的反应条件为:95℃预变性5 min; 95℃变性15 s, 60℃退火30 s, 72℃延伸30 s, 总共40个循环.阴性对照为灭菌的超纯水代替样品, 每个样品均做3次技术重复实验. 16S rRNA基因的标准曲线是通过将携带该基因的质粒进行10倍稀释得到的, 从107~101个拷贝, 每个浓度设置3次重复. 16S rRNA基因的拷贝数通过qPCR结果中每个基因的CT值所对应的标准曲线进行计算.标准曲线相关系数的平方(r2)范围为0.99~0.998, 扩增效率为95%~110%.
1.5 数据分析样品中ARGs的相对拷贝数计算参照已有研究方法[8, 28], 采用公式(1)进行计算.采用公式(2)[29]计算样品中ARGs的绝对丰度.
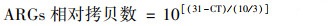
|
(1) |

|
(2) |
根据核糖体RNA操纵子拷贝数数据库(Ribosomal RNA Operon Copy Number Database)所述, 每个细菌平均含有4.1个16S rRNA基因[30], 因此取每个细菌平均含有4个16S rRNA基因.因此, 每个细菌携带的耐药基因或可移动遗传元件的相对丰度计算公式如下:
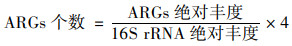
|
(3) |
采用Excel 2010进行数据处理, 采用SPSS 24.0进行Pearson相关性分析, 使用Origin 9.0进行相关图形绘制.对目标基因的绝对量取log10值, 采用Rstudio绘制热图.
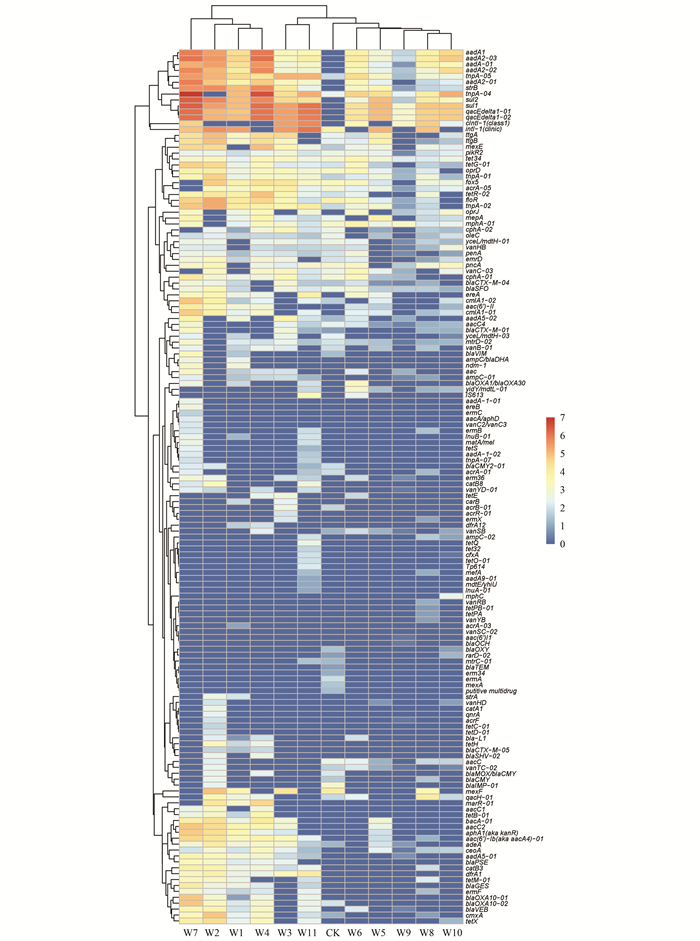
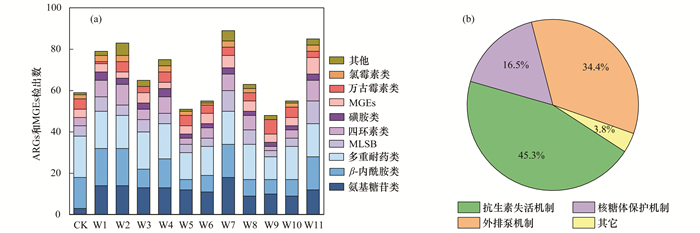
2 结果与讨论 2.1 民用井中耐药基因检出情况根据HT-qPCR筛查结果, 11口民用井中共检出141种耐药基因和8种可移动遗传元件(6种转座子基因以及2种整合酶基因)(图 2).在11口民用井分别检出48种至89种基因, 平均每口井检出68种[图 3(a)].在环境本底对照样品(羊台山泉水CK)检出59种目标基因.检出的耐药基因主要分属于以下9大类抗生素:氨基糖苷类、β-内酰胺类、氯霉素类、大环内酯类-林可酰胺类-链阳性菌素B类(MLSB)、多重耐药类、磺胺类、四环素类、万古霉素类及其他[31].其中, 氨基糖苷类、β-内酰胺类与多重耐药类为检出频次最高的类型.Szekeres等[11]使用qPCR技术对11种耐药基因在邻近城市的地下水中进行了检测, 该研究中耐药基因的检出频率为β-内酰胺类>磺胺类>四环素类>其他>MLSB>氯霉素类>氨基糖苷类.在城市水环境中, 如自来水厂[32]、医院废水[33]、生活污水[34]及养殖废水中[18], β-内酰胺类、氯霉素类、MLSB、磺胺类和四环素类耐药基因检出频繁, 在天然水环境中, 如河流、湖泊、水库以及地下水中, β-内酰胺类、氨基糖苷类和磺胺类耐药基因相对含量最高, 为最常见的耐药基因类型[35].
|
图 2 民用井中耐药基因(ARGs)和可移动元件(MGEs)分布特征 Fig. 2 Distribution characteristics of ARGs and MGEs in domestic wells |
|
(a)民用井中耐药基因与可移动元件检出数; (b)不同耐药机制的基因在总耐药基因中所占比例 图 3 民用井中耐药基因与可移动元件的检出情况 Fig. 3 Detected ARGs and MGEs in domestic wells |
按照耐药机制的不同[图 3(b)], 检出的耐药基因主要属于抗生素失活机制(45.3%)、外排泵机制(34.3%)、核糖体保护机制(16.5%)以及其他机制(3.8%).本研究结果也与Chen等[30]在生活垃圾填埋场附近的地下水中耐药基因的检测结果十分相似, 抗生素失活机制和外排泵机制成为微生物适应抗生素选择压力的最主要机制.抗生素失活酶可将细胞内抗生素转换为低活性产物, 从而有效降低微生物胞内抗生素的浓度, 如氨基糖苷类耐药基因(aac6i、aadA2和aadD等).外排泵机制可介导抗生素和重金属等多种污染物排至细胞外, 如emrY和macA等耐药基因, 也是微生物适应环境胁迫条件的主要途径[36].
2.2 耐药基因在民用井分布特征及其健康风险民用井中耐药基因绝对总量为8.0×103~4.4×107 copies·mL-1 [图 4(a)], 其中位于松岗街道的民用井W4中耐药基因的丰度最高, 其次为茅洲河沿岸的民用井W7.来自林场的民用井W9与松岗街道的W6中耐药基因的丰度最低, 甚至低于环境本底对照点CK.在所有井水中, 磺胺类耐药基因及多重耐药类基因的丰度占据最高比例, 此外, 在W2、W4及W7等井中, 氨基糖苷类耐药基因也呈现出较高丰度[图 3(a)].在对照点CK中, 耐药基因的总丰度为3.9×104 copies·mL-1.Chen等[30]通过高通量qPCR技术在城市垃圾填埋场的地下水中检测得出耐药基因的丰度为2.5×106~1.27×107 copies·mL-1, 其中主要的耐药基因类型为多重耐药类、β-内酰胺类和四环素类.

|
图 4 民用井中耐药基因检出水平 Fig. 4 Abundance of ARGs detected from domestic wells |
为了降低细菌生物量所引起的差异, 将耐药基因的绝对丰度与细菌细胞数进行了均一化处理.目前, 根据核糖体RNA操纵子拷贝数数据库(Ribosomal RNA Operon Copy Number Database)的描述, 每个细菌平均含有4.1个16S rRNA基因[34].因而, 根据每口民用井中测得的16S rRNA的拷贝数, 将耐药基因的个数分别估算至每个细菌细胞体内, 其结果如图 4(b)所示.结果显示, 在民用井W7、W8、W10及W11的井水中, 平均每个细菌携带有至少一个耐药基因, 经均一化处理后的W11井水中甚至平均每个细菌携带有4个耐药基因.
在本研究所检测到的141种耐药基因中, aadA-01、aadA1、aadA2、pikR2、qacEdelta1-01、qacEdelta1-02、strB、sul1、sul2和tet34在所有样本中均可检出, acrA、floR、cmlA1-01、blaCTX-M-04、blaSFO、ceoA、cphA-01、fox5、mphA-01、mtrD-02、oleC、oprD、penA、pncA、tetG、ttgB、vanHB和ycel/mdtH-01等基因在样本中的检出率超过82%.由于磺胺类、四环素类以及季铵盐类化合物的普遍应用[9], 磺胺类(sul1、sul2)、四环素类(tet34、tetG)和季铵盐类耐药基因(qacEdelta1、qacEdealta2)在环境中普遍检出, 是环境常见的耐药基因类型[11, 37, 38].同时, 氨基糖苷类抗生素是临床上使用量最多的五大类抗生素之一[39], 有研究认为氨基糖苷类耐药基因是在污水、废水和动物粪便中最频繁检出的一类耐药基因, 在天然水体中也有检出[40].
然而, 临床相关耐药基因(如超广谱β-内酰胺酶基因blaSHV、blaTEM、blaCTX和blaOXA-1等, 万古霉素类基因vanB和vanC-03等, 氟苯尼考耐药基因floR等)在民用井水中的高频检出, 指示一定程度的健康风险.国内外多名学者[41~43]的研究发现大肠杆菌、沙门氏菌等多种致病菌携带floR基因, 甚至同时携带其他耐药基因如tetA基因, 形成多重耐药菌[44, 45].肠球菌是院内感染的重要致病菌, 对万古霉素耐药主要由vanA、vanB、vanC和vanD等基因介导[46].万古霉素耐药基因的肠球菌阳性率呈逐年上升的趋势[46, 47], 对临床治疗形成一定影响.此外, 汪亚斯等[48]从医院分离获得的耐碳青霉烯酶大肠埃希菌中检出有50%的菌株同时携带有超广谱β-内酰胺酶基因blaTEM、blaCTX和blaCTX.然而, 碳青霉烯类抗生素是治疗严重细菌感染最主要的药物之一, 被认为是抗产超广谱β-内酰胺酶菌株的最后一道防线[49].
民用井作为当地居民的饮用水源之一, 耐药基因(尤其是临床相关耐药基因)带来的潜在健康风险不容忽视.若将本研究所调查的11口民用井看作河流域内饮用水的代表, 则平均每毫升井水中含有1.6×107个细菌.将检测并计算得到的民用井井水中平均每个细菌细胞所携带的耐药基因量取平均值, 可知井水中平均每个细菌细胞携带有1.03个耐药基因.文献[50]中指出, 我国健康成年人每天需饮水1 500~1 700 mL, 假设该流域内人均喝水量为1 700 mL, 并以井水为唯一水源, 则当地居民平均每人每天摄入的耐药基因量将达到2.8×1010个. Xu等[51]对杭州两个自来水厂的源水中ARGs的丰度检测结果显示, 钱塘江江水作为自来水源水中ARGs的丰度为108~109 copies·L-1, 而出水中ARGs的丰度在105~108 copies·L-1.可见, 地下水中的耐药基因对人体健康形成潜在威胁.
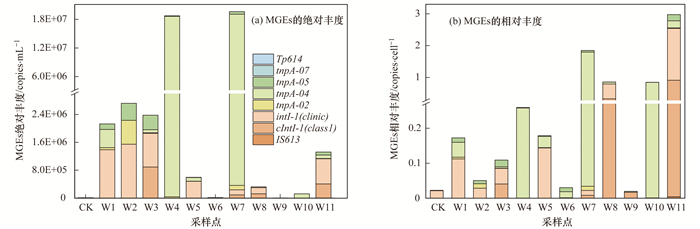
2.3 可移动元件在民用井中的分布特征及耐药基因水平转移风险可移动元件基因MGEs在11口民用井中的绝对丰度为2.3×103~1.96×107 copies·mL-1, 其中民用井W7与W4中MGEs的丰度最高[图 5(a)].在对照点CK中, MGEs的总丰度为9.4×103copies·mL-1.经均一化处理后每个细菌细胞体内的可移动遗传元件数量估算结果如图 5 (b)所示.W7、W8和W10中每个细菌所携带的MGEs在1~2个, 井W11中平均每个细菌携带有3个MGEs, 显示出较高的基因水平转移风险.W4、W7及W10井水中检出的MGEs主要为tnpA-04, W8和W11井水中主要为cIntI-1 (class1)与intI-1 (clinic) [图 5(b)].值得注意的是, 可移动元件基因与耐药基因的分布情况相吻合[图 2、图 4(a)及图 5(a)], 民用井W4、W1、W2和W7不仅含有高丰度的耐药基因, 也含有多种MGEs.可见, 相比环境本底对照点(CK), 民用井水含有丰富的ARGs和MGEs.
|
图 5 民用井中移动元件检出水平 Fig. 5 Abundance of MGEs detected from domestic wells |
进一步对耐药基因与可移动元件进行相关性分析(表 1), 结果显示磺胺类、多重耐药类、氨基糖苷类、β-内酰胺类以及氯霉素类耐药基因与可移动元件呈显著正相关(相关系数为0.777~0.985, P < 0.01).Chen等[30]在城市垃圾填埋场附近的地下水中亦发现磺胺类(P < 0.05)、多重耐药(P < 0.05)和氯霉素类(P < 0.01)耐药基因与可移动元件呈正相关, 除此之外, 四环素类(P < 0.01)和万古霉素类(P < 0.05)耐药基因也具有相关规律.
|
|
表 1 ARGs和MGEs之间的相关性1) Table 1 Correlations between various ARGs and MGEs |
水平基因转移通常被认为是细菌获得抗性的重要分子机制[10, 52, 53].Zhu等[28]的研究指出, 整合酶基因(如intI-1、cintI-1)和转座酶基因(tnpA)常被用作可移动遗传元件的标志基因.一类整合子基因int1被认为是耐药细菌和人为污染的指示基因[10].有研究表明, 利用intI-1或cintI-1编码的整合酶, Ⅰ类整合子以环形基因盒的形式可以捕获不同的ARGs[10, 54, 55].水平转移也是临床相关致病菌从环境细菌中获取抗性基因的重要途径.
耐药基因的风险评估主要围绕基因的水平转移能力及其与致病菌的关联来进行.Martínez等[56]对ARGs的人体健康风险提供了一种评估模型, 根据该基因是否为耐药基因、ARGs是否在MGEs上以及该基因是否在人类致病菌中存在而将ARGs的风险分为7个等级.Oh等[57]基于宏基因组测序的数据, 通过计算对ARGs的相对丰度、可移动性以及在病原体内的存在情况进行评估, 使用三维风险空间模型对ARGs的耐药风险进行评分.有研究指出环境中ARGs的分布特征和多样性变化不仅与水平转移相关[58~60], 也与细菌群落结构密切关联[61].由于微生物群落的多样性和实际环境的复杂性, 迄今尚未形成完整且可靠的耐药基因风险评估框架.
3 结论(1) 城市密集活动对地下水耐药基因具有较大影响, 民用井水中耐药基因主要类型为磺胺类、多重耐药类以及氨基糖苷类, 其检出水平和丰度远高于环境背景值.
(2) 民用井水不仅含有多种环境常见基因, 也含有多种临床重要的耐药基因; 同时可移动元件基因与耐药基因密切相关, 井水中的耐药基因具有较高的转移风险, 对环境卫生和人体健康具有潜在威胁.
| [1] | Zaffiri L, Gardner J, Toledo-Pereyra L H. History of antibiotics. From salvarsan to cephalosporins[J]. Journal of Investigative Surgery, 2012, 25(2): 67-77. DOI:10.3109/08941939.2012.664099 |
| [2] | Pruden A, Pei R T, Storteboom H, et al. Antibiotic resistance genes as emerging contaminants:studies in northern Colorado[J]. Environmental Science & Technology, 2006, 40(23): 7445-7450. |
| [3] | 国家卫生计生委, 国家发展改革委, 教育部, 等.关于印发遏制细菌耐药国家行动计划(2016-2020年)的通知.国卫医发[2016]43号[EB/OL]. http://www.gov.cn/xinwen/2016-08/25/content_5102348.htm, 2016-08-05. |
| [4] | World Health Organization. Antimicrobial resistance. Global report on surveillance[EB/OL]. http://apps.who.int/bookorders/anglais/detart1.jsp?codlan=1&codcol=15&codcch=870, 2014. |
| [5] | Sui Q, Cao X Q, Lu S G, et al. Occurrence, sources and fate of pharmaceuticals and personal care products in the groundwater:a review[J]. Emerging Contaminants, 2015, 1(1): 14-24. |
| [6] | Balzer F, Zühlke S, Hannappel S. Antibiotics in groundwater under locations with high livestock density in Germany[J]. Water Supply, 2016, 16(5): 1361-1369. DOI:10.2166/ws.2016.050 |
| [7] | Hou J, Wan W N, Mao D Q, et al. Occurrence and distribution of sulfonamides, tetracyclines, quinolones, macrolides, and nitrofurans in livestock manure and amended soils of Northern China[J]. Environmental Science and Pollution Research, 2015, 22(6): 4545-4554. DOI:10.1007/s11356-014-3632-y |
| [8] | Chen Q L, An X L, Li H, et al. Do manure-borne or indigenous soil microorganisms influence the spread of antibiotic resistance genes in manured soil?[J]. Soil Biology and Biochemistry, 2017, 114: 229-237. DOI:10.1016/j.soilbio.2017.07.022 |
| [9] | Qiao M, Ying G G, Singer A C, et al. Review of antibiotic resistance in China and its environment[J]. Environment International, 2018, 110: 160-172. DOI:10.1016/j.envint.2017.10.016 |
| [10] | Gillings M R. Lateral gene transfer, bacterial genome evolution, and the Anthropocene[J]. Annals of the New York Academy of Sciences, 2017, 1389(1): 20-36. DOI:10.1111/nyas.13213 |
| [11] | Szekeres E, Chiriac C M, Baricz A, et al. Investigating antibiotics, antibiotic resistance genes, and microbial contaminants in groundwater in relation to the proximity of urban areas[J]. Environmental Pollution, 2018, 236: 734-744. DOI:10.1016/j.envpol.2018.01.107 |
| [12] | Kothari A, Wu Y W, Chandonia J M, et al. Large circular plasmids from groundwater plasmidomes span multiple incompatibility groups and are enriched in multimetal resistance genes[J]. Mbio, 2019, 10(1): e02899-18. DOI:10.1128/mbio.02899-18 |
| [13] | Li X D, Watanabe N, Xiao C L, et al. Antibiotic-resistant E.coli in surface water and groundwater in dairy operations in Northern California[J]. Environmental Monitoring and Assessment, 2014, 186(2): 1253-1260. DOI:10.1007/s10661-013-3454-2 |
| [14] | Yu Z, He P, Shao L, et al. Co-occurrence of mobile genetic elements and antibiotic resistance genes in municipal solid waste landfill leachates:A preliminary insight into the role of landfill age[J]. Water Research, 2016, 106: 583-592. DOI:10.1016/j.watres.2016.10.042 |
| [15] | Boy-Roura M, Mas-Pla J, Petrovic M, et al. Towards the understanding of antibiotic occurrence and transport in groundwater:Findings from the Baix Fluvia alluvial aquifer (NE Catalonia, Spain)[J]. Science of the Total Environment, 2018, 612: 1387-1406. DOI:10.1016/j.scitotenv.2017.09.012 |
| [16] | Tolentino F M, De Almeida I A Z C, Dos Santos C C M, et al. Phenotypic and genotypic profile of the antimicrobial resistance of bacterial isolates and evaluation of physical and chemical potability indicators in groundwater in Brazil[J]. International Journal of Environmental Health Research. DOI:10.1080/09603123.2019.1640354 |
| [17] | He L Y, Liu Y S, Su H C, et al. Dissemination of antibiotic resistance genes in representative broiler feedlots environments:identification of indicator ARGs and correlations with environmental variables[J]. Environmental Science & Technology, 2014, 48(22): 13120-13129. |
| [18] | He L Y, Ying G G, Liu Y S, et al. Discharge of swine wastes risks water quality and food safety:antibiotics and antibiotic resistance genes from swine sources to the receiving environments[J]. Environment International, 2016, 92-93: 210-219. DOI:10.1016/j.envint.2016.03.023 |
| [19] | Tao R, Ying G G, Su H C, et al. Detection of antibiotic resistance and tetracycline resistance genes in Enterobacteriaceae isolated from the Pearl rivers in South China[J]. Environmental Pollution, 2010, 158(6): 2101-2109. DOI:10.1016/j.envpol.2010.03.004 |
| [20] | Yang J F, Ying G G, Zhao J L, et al. Spatial and seasonal distribution of selected antibiotics in surface waters of the Pearl Rivers, China[J]. Journal of Environmental Science and Health, Part B, 2011, 46(3): 272-280. DOI:10.1080/03601234.2011.540540 |
| [21] | Zhou L J, Ying G G, Zhao J L, et al. Trends in the occurrence of human and veterinary antibiotics in the sediments of the Yellow River, Hai River and Liao River in northern China[J]. Environmental Pollution, 2011, 159(7): 1877-1885. DOI:10.1016/j.envpol.2011.03.034 |
| [22] | Su H C, Pan C G, Ying G G, et al. Contamination profiles of antibiotic resistance genes in the sediments at a catchment scale[J]. Science of the Total Environment, 2014, 490: 708-714. DOI:10.1016/j.scitotenv.2014.05.060 |
| [23] | Ying G G, He L Y, Ying A J, et al. China must reduce its antibiotic use[J]. Environmental Science & Technology, 2017, 51(3): 1072-1073. |
| [24] | Zhang Y, Zhang Y Z, Kuang Z M, et al. Comparison of microbiomes and resistomes in two karst groundwater sites in Chongqing, China[J]. Groundwater, 2019, 57(5): 807-818. DOI:10.1111/gwat.12924 |
| [25] | Yang Y Y, Zhao J L, Liu Y S, et al. Pharmaceuticals and personal care products (PPCPs) and artificial sweeteners (ASs) in surface and ground waters and their application as indication of wastewater contamination[J]. Science of the Total Environment, 2018, 616-617: 816-823. DOI:10.1016/j.scitotenv.2017.10.241 |
| [26] | 深圳市水务局.深圳基本水情[EB/OL]. http://swj.sz.gov.cn/xxgk/zfxxgkml/szswgk/szjbsq/201911/t20191105_18496533.htm, 2019. |
| [27] | 深圳市水务局.深圳市水资源公报[EB/OL]. http://swj.sz.gov.cn/xxgk/zfxxgkml/szswgk/tjsj/szygb/201910/t20191023_18451949.htm, 2018. |
| [28] | Zhu Y G, Johnson T A, Su J Q, et al. Diverse and abundant antibiotic resistance genes in Chinese swine farms[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(9): 3435-3440. DOI:10.1073/pnas.1222743110 |
| [29] |
程建华, 唐翔宇, 刘琛. 紫色土丘陵区畜禽养殖场土壤中抗生素抗性基因分布特征[J]. 环境科学, 2019, 40(7): 3257-3262. Cheng J H, Tang X Y, Liu C. Characteristics of antibiotic resistance genes in various livestock feedlot soils of the hilly purple soil region[J]. Environmental Science, 2019, 40(7): 3257-3262. |
| [30] | Chen Q L, Li H, Zhou X Y, et al. An underappreciated hotspot of antibiotic resistance:the groundwater near the municipal solid waste landfill[J]. Science of the Total Environment, 2017, 609: 966-973. DOI:10.1016/j.scitotenv.2017.07.164 |
| [31] |
黄福义, 安新丽, 陈青林, 等. 梅花鹿养殖场抗生素抗性基因分布特征[J]. 环境科学, 2016, 37(11): 4402-4409. Huang F Y, An X L, Chen Q L, et al. Distribution characteristics of antibiotic resistance genes in sika deer farm[J]. Environmental Science, 2016, 37(11): 4402-4409. |
| [32] | Bergeron S, Boopathy R, Nathaniel R, et al. Presence of antibiotic resistant bacteria and antibiotic resistance genes in raw source water and treated drinking water[J]. International Biodeterioration & Biodegradation, 2015, 102: 370-374. |
| [33] | Rodriguez-Mozaz S, Chamorro S, Marti E, et al. Occurrence of antibiotics and antibiotic resistance genes in hospital and urban wastewaters and their impact on the receiving river[J]. Water Research, 2015, 69: 234-242. DOI:10.1016/j.watres.2014.11.021 |
| [34] | Lefkowitz J R, Duran M. Changes in antibiotic resistance patterns of Escherichia coli during domestic wastewater treatment[J]. Water Environment Research, 2009, 81(9): 878-885. DOI:10.2175/106143009X426068 |
| [35] |
李林云, 谭璐, 崔玉晓, 等. 饮用水中细菌耐药及其健康风险研究进展[J]. 生态毒理学报, 2018, 13(2): 1-12. Li L Y, Tan L, Cui Y X, et al. Bacterial resistance and human health risk in drinking water[J]. Asian Journal of Ecotoxicology, 2018, 13(2): 1-12. |
| [36] | Chen B W, Lin L, Fang L, et al. Complex pollution of antibiotic resistance genes due to beta-lactam and aminoglycoside use in aquaculture farming[J]. Water Research, 2018, 134: 200-208. DOI:10.1016/j.watres.2018.02.003 |
| [37] | Xu Y, Guo C S, Luo Y, et al. Occurrence and distribution of antibiotics, antibiotic resistance genes in the urban rivers in Beijing, China[J]. Environmental Pollution, 2016, 213: 833-840. DOI:10.1016/j.envpol.2016.03.054 |
| [38] | Ling Z H, Yang Y, Huang Y L, et al. A preliminary investigation on the occurrence and distribution of antibiotic resistance genes in the Beijiang River, South China[J]. Journal of Environmental Sciences, 2013, 25(8): 1656-1661. DOI:10.1016/S1001-0742(12)60223-X |
| [39] |
张红, 程寒飞. 水环境中氨基糖苷类抗性基因污染及研究进展[J]. 环境科学与技术, 2018, 41(10): 121-130. Zhang H, Cheng H F. Aquatic environmental pollution of aminoglycoside resistance genes:a review[J]. Environmental Science & Technology, 2018, 41(10): 121-130. DOI:10.19672/j.cnki.1003-6504.2018.10.018 |
| [40] | Hsu C Y, Hsu B M, Ji W T, et al. A potential association between antibiotic abuse and existence of related resistance genes in different aquatic environments[J]. Water, Air, & Soil Pollution, 2015, 226. DOI:10.1007/s11270-014-2235-z |
| [41] | Bischoff K M, White D G, McDermott P F, et al. Characterization of chloramphenicol resistance in beta- hemolytic Escherichia coli associated with diarrhea in Neonatal swine[J]. Journal of Clinical Microbiology, 2002, 40(2): 389-394. DOI:10.1128/JCM.40.2.389-394.2002 |
| [42] |
杜向党, 吴蓓蓓, 沈建忠. 牛源致病性大肠杆菌耐氟苯尼考floR基因的克隆与序列分析[J]. 中国兽医学报, 2005, 25(3): 247-250. Du X D, Wu B B, Shen J Z. Cloning and sequence analysis of floR genes isolated from bovine pathogenic Escherichia coli[J]. Chinese Journal of Veterinary Science, 2005, 25(3): 247-250. DOI:10.3969/j.issn.1005-4545.2005.03.007 |
| [43] |
杜向党, 李新生, 张素梅, 等. 鸡源大肠杆菌氟苯尼考耐药基因floR的分子检测[J]. 河南农业大学学报, 2007, 41(2): 188-191. Du X D, Li X S, Zhang S M, et al. Detection of the floR genes in florfenicol-resistant Escherichia coli isolated from diseased chickens[J]. Journal of Henan Agricultural University, 2007, 41(2): 188-191. DOI:10.3969/j.issn.1000-2340.2007.02.014 |
| [44] | Mayers D L. Antimicrobial drug resistance:mechanisms of drug resistance[M]. New York: Humana Press, 2009. |
| [45] | Hradecka H, Karasova D, Rychlik I. Characterization of Salmonella enterica serovar typhimurium conjugative plasmids transferring resistance to antibiotics and their interaction with the virulence plasmid[J]. Journal of Antimicrobial Chemotherapy, 2008, 62(5): 938-941. DOI:10.1093/jac/dkn286 |
| [46] | Kak V, Chow J W. Acquired antibiotic resistances in enterococci[A]. In: Gilmore M S (Ed.). The Enterococci: Pathogenesis, Molecular biology, and Antibiotic Resistance[M]. Washington, DC: ASM Press, 2002. 355-384. |
| [47] |
郑波, 王珊, 李耘, 等. 万古霉素耐药肠球菌基因及水平转移研究[J]. 中国临床药理学杂志, 2007, 23(2): 133-136. Zheng B, Wang S, Li Y, et al. Study of gene and horizontal gene transfer of vancomycin resistant Enterococci[J]. The Chinese Journal of Clinical Pharmacology, 2007, 23(2): 133-136. DOI:10.3969/j.issn.1001-6821.2007.02.013 |
| [48] |
汪亚斯, 董爱英, 周海健, 等. 不同感染途径CREC分子分型特点及耐药分析[J]. 中国抗生素杂志. Wang Y S, Dong A Y, Zhou H J, et al. Molecular typing and drug resistance analysis of carbapenem-resistant Escherichia coli in different infection[J]. Chinese Journal of Antibiotics. DOI:10.13461/j.cnki.cja.006801 |
| [49] | Abbas H A, Kadry A A, Shaker G H, et al. Impact of specific inhibitors on metallo-β-carbapenemases detected in Escherichia coli and Klebsiella pneumoniae isolates[J]. Microbial Pathogenesis, 2019, 132: 266-274. DOI:10.1016/j.micpath.2019.05.022 |
| [50] | 中国营养学会. 中国居民膳食指南(2016)[M]. 北京: 人民卫生出版社, 2016. |
| [51] | Xu L K, Ouyang W Y, Qian Y Y, et al. High-throughput profiling of antibiotic resistance genes in drinking water treatment plants and distribution systems[J]. Environmental Pollution, 2016, 213: 119-126. DOI:10.1016/j.envpol.2016.02.013 |
| [52] | Gaze W H, Zhang L H, Abdouslam N A, et al. Impacts of anthropogenic activity on the ecology of class 1 integrons and integron-associated genes in the environment[J]. The ISME Journal, 2011, 5(8): 1253-1261. DOI:10.1038/ismej.2011.15 |
| [53] | Marshall B M, Levy S B. Food animals and antimicrobials:impacts on human health[J]. Clinical Microbiology Reviews, 2011, 24(4): 718-733. DOI:10.1128/CMR.00002-11 |
| [54] | Heuer H, Binh C T, Jechalke S, et al. IncP-1ε plasmids are important vectors of antibiotic resistance genes in agricultural systems:diversification driven by class 1 Integron gene cassettes[J]. Frontiers in Microbiology, 2012, 3: 2. DOI:10.3389/fmicb.2012.00002 |
| [55] | Partridge S R, Tsafnat G, Coiera E, et al. Gene cassettes and cassette arrays in mobile resistance integrons[J]. FEMS Microbiology Reviews, 2009, 33(4): 757-784. DOI:10.1111/j.1574-6976.2009.00175.x |
| [56] | Martínez J L, Coque T M, Baquero F. What is a resistance gene? Ranking risk in resistomes[J]. Nature Reviews Microbiology, 2015, 13(2): 116-123. DOI:10.1038/nrmicro3399 |
| [57] | Oh M, Pruden A, Chen C Q, et al. MetaCompare:a computational pipeline for prioritizing environmental resistome risk[J]. FEMS Microbiology Ecology, 2018, 94(7): fiy079. DOI:10.1093/femsec/fiy079 |
| [58] | Jia S Y, Shi P, Hu Q, et al. Bacterial community shift drives antibiotic resistance promotion during drinking water chlorination[J]. Environmental Science & Technology, 2015, 49(20): 12271-12279. |
| [59] | Sun M M, Ye M, Wu J, et al. Impact of bioaccessible pyrene on the abundance of antibiotic resistance genes during Sphingobium sp.-and sophorolipid-enhanced bioremediation in soil[J]. Journal of Hazardous Materials, 2015, 300: 121-128. DOI:10.1016/j.jhazmat.2015.06.065 |
| [60] | Chen Q L, An X L, Li H, et al. Long-term field application of sewage sludge increases the abundance of antibiotic resistance genes in soil[J]. Environment International, 2016, 92-93: 1-10. DOI:10.1016/j.envint.2016.03.026 |
| [61] | Forsberg K J, Patel S, Gibson M K, et al. Bacterial phylogeny structures soil resistomes across habitats[J]. Nature, 2014, 509(7502): 612-616. DOI:10.1038/nature13377 |
 2020, Vol. 41
2020, Vol. 41


