2. 江西省红壤研究所, 南昌 331717;
3. 江苏省地理信息资源开发与利用协同创新中心, 南京 210023
2. Jiangxi Institute of Red Soil, Nanchang 331717, China;
3. Jiangsu Center for Collaborative Innovation in Geographical Information Resource Development and Application, Nanjing 210023, China
氮循环是土壤生态系统中重要的元素循环之一, 主要包括氮素固定、有机氮矿化以及各种形态无机氮的相互转化[1].微生物是介导土壤氮循环过程的载体和引擎, 刻画和关联功能微生物类群(或功能基因簇)与氮循环生态过程是环境组学和微生物生态学研究的重要内容之一[2].为此, 行业先驱们做出了很多努力, 通过分析nifH、amoA、narG和nir/nos等功能基因来研究单个氮循环生态过程[3~6].然而, 氮循环各过程承先启后、相互关联, 且部分功能基因簇缺少合适的扩增引物, 因此全面、综合地研究复杂土壤生态系统中的氮循环过程十分受限[7, 8]. 2010年后基因芯片技术的发展, 使得环境样品的功能基因研究快速发展[9, 10];但受现有数据库及芯片制作技术所限, 该技术对新发现基因及多样性较高的基因簇研究存在缺陷[11].近年来, 随着宏基因组(metagenome)测序技术的发展, 使得在一个生态系统中同步对多个氮循环功能基因进行研究成为可能;即在宏基因组深度测序的基础上, 通过与COG、KEGG、eggNOG和NCycDB等综合数据库比对, 筛选出氮循环过程中的功能基因类群及相对丰度[12, 13].因此, 基于宏基因组测序技术, 可全面、综合地评估气候变化[12, 14]和人为活动[13]引起的土壤生态系统演变下的微生物功能基因簇变化及其生态过程潜力.
酸度值低于5.5的酸性红壤主要分布在我国南方地区, 约占全国土地总面积的22.7%;酸性土壤有机质含量低、生物可利用养分含量低、土壤淋溶强、氧化势高, 使得该土壤肥力普遍较低[15].长期定位施肥试验发现, 相比于不施肥土壤, 30 a的长期有机粪肥施加显著提高了我国江西地区酸性玉米地土壤有机质含量近40%[3], 而该地区另一长期定位施肥试验研究发现长期施用化肥也能提升土壤肥力, 但对土壤有机质的提升较小(3.8% ~6.1%)[16].因此, 施有机肥是提升酸性土壤有机质含量、提高土壤质量的最有效措施之一, 也是提高土壤通气性、缓解土壤酸化的重要措施之一[3, 17, 18].相关研究发现, 土壤有机质提升会显著增加土壤硝化活性[3], 改变土壤氨氧化微生物群落丰度和组成[3, 16, 19], 可见酸性土壤有机质提升确实显著影响了土壤微生物群落及其生态功能.但是, 有机质提升后对土壤多个氮循环过程、氮循环功能基因及相关功能微生物的研究, 目前还未见报道.
本文以位于江西进贤的长期施有机粪肥处理的酸性红壤及不施肥土壤为研究材料, 结果发现, 相比于不施肥处理, 32a的有机粪肥施加显著提升了土壤有机质含量, 增幅达38.2%.采用基于Illumina HiSeq高通量测序平台的宏基因组测序技术定量土壤氮循环功能基因及氮循环相关功能微生物类群的相对丰度, 结合基于室内培养试验的土壤硝化强度及氨氧化微生物绝对丰度数据评估土壤硝化过程功能基因丰度与功能微生物丰度及硝化作用强度的匹配程度, 综合研究酸性土壤氮循环功能基因及关键功能微生物类群对有机质提升的响应.本研究有助于全面把握土壤中的无机氮和有机氮循环功能基因及无机氮循环功能微生物类群, 以期为把握酸性红壤氮循环特征提供试验数据, 也可为酸性土壤改良提供思路.
1 材料与方法 1.1 土壤样品采集和预处理本研究依托位于江西进贤的江西省红壤研究所红壤生态实验站, 以其长期施肥旱地玉米地试验田为研究平台.该地区属亚热带季风气候(28°21′04″ N, 116°10′16″E), 年均温17.7℃, 种植期日平均气温23.1℃, 年降水量1 727 mm;该试验田建于1986年, 设双季玉米轮作种植方式, 种植期为每年的4~11月[3, 17].试验田设有不施肥(CK)和长期施有机肥(OM)两个处理, 有机肥为猪粪, 施用量为2 000 kg ·hm-2, 施肥时间为早季玉米种植前;每个处理设有3个随机分布的重复小区, 各小区面积约为20 m2.于2018年4月早季玉米种植前, 使用土钻采集耕作层土壤样本(0~15 cm), 每个小区采用S型采集10个点混合后作为1份土壤样品.土壤样品立即于4℃下保存, 并于2 d内去除杂物和植物残留, 过2 mm筛后, 分成3份.一份储存于-80℃用于土壤总DNA提取;一份储存于4℃用于无机氮含量测定及土壤硝化强度测定;一份风干后用于基础理化性质测定.
1.2 土壤理化性质及硝化强度测定土壤理化性质测定方法参照文献[3, 18].采用玻璃电极法测定土壤pH值(Mettler-Toledo, 瑞士), 浸提液分别为去离子水和2 mol ·L-1 KCl溶液, 水土比为2.5 :1.采用重铬酸钾氧化法测定土壤总有机碳(Total organic carbon, TOC)含量, 采用微波消解-凯氏定氮法测定土壤总氮(Total nitrogen, TN)含量.采用KCl浸提-流动分析仪法(SKALA, 荷兰)测定土壤铵态氮和硝态氮含量.采用硝态氮累计法测定土壤净硝化活性[3], 采用氯酸钾抑制-亚硝态氮累计法测定土壤氨氧化潜势[19].测定结果均以土壤干重计.
1.3 土壤总DNA提取及宏基因组测序称取0.5 g储存于-80℃的土壤样品, 采用FastDNA(Spin Kit for Soil MP Biomedicals, 美国)试剂盒提取土壤总DNA, 提取步骤及所用设备参照试剂盒说明书.采用NanoDrop 2000(Thermo Fisher, 美国)测定提取液中DNA浓度及纯度.检测合格的DNA溶液加入fragmentation缓冲液(Thermo Fisher, 美国), 超声破碎法随机打断后得到短片段DNA用于测序文库构建, 采用HiSeq2500高通量测序平台(Illumina, 美国)进行PE150测序.本研究宏基因组测序工作委托广东美格基因科技有限公司完成.获得每个样本的宏基因组测序数据后, 首先采用Trimmomatic(http://www.usadellab.org/cms/index.php?page=trimmomatic)进行质量控制来去除低质量数据, 获得Clean Reads, 以保证结果可信度;采用MEGAHIT(http://www.l3-bioinfo.com/products/megahit.html)对Clean Reads进行de novo拼接获得500 bp以上的Scaftig, k-min=35, k-max=95, k-step=20;采用MetaGeneMark(http://exon.gatech.edu/meta_gmhmmp.cgi)进行开放阅读框预测[20];采用Linclust进行基因聚类及去冗余, 得到Unigenes序列[21];将非冗余的Unigenes序列与NCBI-NR数据库(https://www.ncbi.nlm.nih.gov/)比对, 结合MetaPhlAn2(http://www.huttenhower.org/metaphlan2)对Clean Reads快速分类, 共同对物种进行注释.同时, 利用预测出来的蛋白序列与NCycDB数据库进行比对, 以获取相关氮循环功能基因注释信息[11];该数据库综合了KEGG、COG、eggNOG、Uniprot等数据库中的氮循环功能基因信息, 共收录68个氮循环功能基因家族的基因, 包含219 146条参考序列, 数据库信息及比对方法参见https://github.com/qichao1984/NCyc.
1.4 古菌和细菌amoA基因的定量PCR分析采用基于SYBR染料法的定量PCR分析土壤中氨氧化古菌(ammonia-oxidizing archaea, AOA)amoA基因(amoA-A)和氨氧化细菌(ammonia-oxidizing bacteria, AOB)amoA基因(amoA-B)的绝对丰度.amoA-A和amoA-B基因扩增引物分别为Arch-amoAF(5′-STA ATG GTC TGG CTT AGA CG-3′)/Arch-amoAR(5′-GCG GCC ATC CAT CTG TAT GT-3′)[22]和amoA-1F(5′-GGG GTT TCT ACT GGT GGT-3′)/amoA-2R(5′-CCC CTC KGS AAA GCC TTC TTC-3′)[23].定量PCR扩增设备为CFX96(Bio-Rad Laboratories, 美国), 扩增试剂为HiScript II Q RT SuperMix for qPCR(Vazyme Biotech, 中国), 反应体系、扩增程序及熔解曲线程序见文献[3, 18].采用10倍稀释含有标靶基因的重组质粒制作标准曲线, AOA和AOB的amoA基因标准曲线模板浓度分别为1.39×102~1.39×108 copies ·μL-1和2.30×102~2.30×108 copies ·μL-1, 扩增效率分别为94.0%(R2>0.991)和102.8%(R2>0.985).
1.5 数据分析采用SPSS 16.0(IBM, 美国)统计软件对试验数据进行分析, 采用独立样本T检验比较CK和OM土壤中各指标的显著性差异(n=3, P < 0.05).采用CIRCOS平台(http://circos.ca/)绘制微生物群落组成图, 以表征不同功能微生物属在氮循环类群和土壤样品中的分布.
2 结果与分析 2.1 土壤理化性质和硝化强度长期有机物质输入后, 土壤理化性质显著变化(表 1).与CK土壤相比, OM土壤pH值显著提升(P < 0.05), 有效缓解了土壤酸化;土壤总有机碳和总氮含量显著增加, 增幅分别为38.2%和44.1%(P < 0.05).OM土壤硝态氮含量显著高于CK土壤(P < 0.05), 而两种土壤铵态氮含量无差异(P>0.05).与CK土壤相比, OM土壤净硝化速率和氨氧化潜势显著升高, 增幅分别为70.6%和78.6%(P < 0.05).
|
|
表 1 土壤基本理化性质、硝化强度及amoA基因丰度1) Table 1 Basic physicochemical properties, nitrification activity, and amoA gene abundance of soils |
2.2 土壤无机氮循环功能基因丰度
有机质提升后, 酸性红壤中与无机氮循环过程相关的功能基因丰度发生明显变化(图 1).好氧硝化过程中, 有机质提升显著增加了amoA-A基因丰度(P < 0.05), 增幅为64.1%, 但对amoA-B基因丰度没有影响(P>0.05);定量PCR结果发现有机质提升后土壤古菌和细菌amoA基因丰度均显著增长(表 1), 增幅分别为134%和147%.有机质提升显著降低了编码羟胺氧化酶的hao基因丰度(P < 0.05), 降幅为32.7%;有机质提升对编码亚硝酸盐氧化酶的nxrAB基因丰度无显著影响(P>0.05).有机质提升显著增加了反硝化过程中各功能基因的丰度, 包括编码硝酸盐还原酶的narGHIJVWYZ基因和napABC基因丰度、编码亚硝酸盐还原酶的nirKS基因、编码NO还原酶的norBC基因及编码N2O还原酶的nosZ基因(P < 0.05), 增幅分别达到56.3%、63.7%、94.3%、100.6%和71.2%.硝酸盐异化还原成铵(DNRA)过程, 有机质提升显著增加了nrfABCD基因丰度(P < 0.05).有机质提升对厌氧氨氧化过程(ANAMMOX)和生物固氮(Nitrogen fixation)过程中的功能基因丰度无显著影响(P>0.05).有机质提升后, 土壤氨氧化过程功能基因amoA丰度与硝化强度(表 1)变化趋势一致.
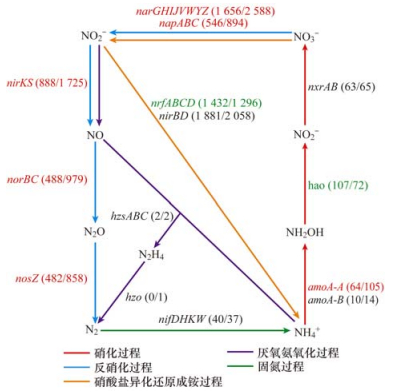
|
基因名称旁括号内的左边和右边数字分别代表CK和OM土壤中基于宏基因组测序得出的标准化功能基因丰度数据;有机质提升后丰度显著增加的基因用红色标注(T-test, n=3, P < 0.05);有机质提升后丰度显著降低的基因用绿色标注(T-test, n=3, P < 0.05);CK和OM土壤中丰度无显著差异的基因用黑色标注(T-test, n=3, P>0.05);不同颜色的带箭头线代表不同的氮循环过程 图 1 土壤无机氮循环功能基因丰度 Fig. 1 Abundance of soil inorganic nitrogen cycling genes |
有机质提升也改变了酸性土壤中无机氮循环过程主要微生物属(>0.5%)的相对丰度(图 2).CK土壤中氨氧化古菌属Nitrososphaera和Nitrosopumilus相对丰度分别为40.8%和3.4%, 有机质提升后分别显著下降至33.1%和2.1%(P < 0.05).CK土壤中氨氧化细菌属Nitrosospira和Nitrosomonas相对丰度分别为4.0%和1.1%, 有机质提升后分别上升至6.8%和1.6%(P < 0.05);而Nitrosococcus相对丰度则由3.2%显著下降至2.6%(P < 0.05).有机质提升后, 土壤中亚硝酸盐氧化菌属Nitrolance和Nitrospina相对丰度分别从23.4%和2.5%下降至6.0%和1.9%(P < 0.05), 而Nitrospira相对丰度从14.9%上升至40.1%(P < 0.05);有机质提升对Nitrobacter和Nitrospirillum相对丰度无显著影响(P>0.05).本研究还检测到厌氧氨氧化菌属Candidatus Brocadia和Candidatus Kuenenia, 其相对丰度均未受有机质提升影响(P>0.05).
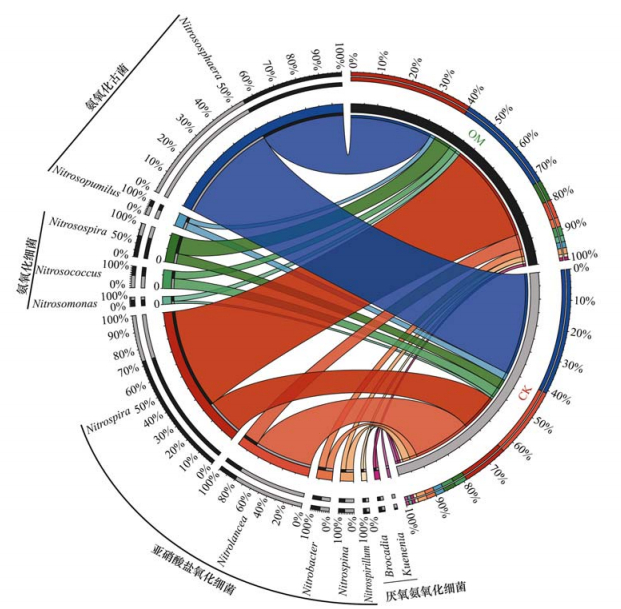
|
不同颜色的模块代表执行不同无机氮循环过程的功能微生物;同一颜色的不同拆分模块所占比例数值代表的是不同微生物属占各自功能微生物的相对丰度以及在CK和OM土壤中的相对丰度 图 2 土壤氮循环功能微生物类群及其相对丰度 Fig. 2 Abundance of soil inorganic nitrogen cycling genes |
酸性土壤有机氮循环过程主要包括硝酸盐同化过程和有机氮代谢过程, 也显著受有机质提升影响(图 3);而铵盐的同化过程功能基因在本研究土壤中并未检测到.硝酸盐同化还原过程中, 有机质提升显著增加了编码硝酸盐还原酶的nasAB基因丰度(增幅为16.5%, P < 0.05), 显著降低了编码亚硝酸盐还原酶的nirA和NR基因丰度(降幅分别为22.5%和20.2%, P < 0.05), 对编码另一类硝酸盐还原酶的narBC基因丰度无显著影响(P>0.05).有机氮代谢过程中, 有机质提升显著增加了编码谷氨酰胺合成酶的glnA、编码谷氨酸脱氢酶的gdh、编码谷氨酰胺酶的glsA/ansB和编码硝基烷氧化酶的nao基因丰度(P < 0.05), 增幅分别为10.1%、11.4%、141.5%、85.8%和72.5%, 而对其它有机氮代谢功能基因丰度无显著影响(P>0.05).
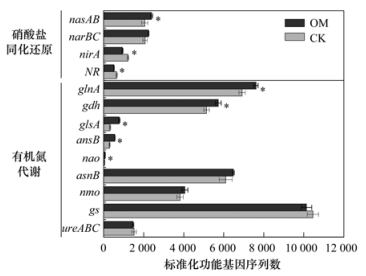
|
横坐标代表基于宏基因组测序得出的标准化功能基因序列数;柱子右侧的星号表示该功能基因丰度在CK和OM土壤中有显著差异(T-test, n=3, P < 0.05) 图 3 土壤有机氮循环功能基因丰度 Fig. 3 Abundance of soil organic nitrogen cycling genes |
过去20 a, 许多报道调查了酸性土壤中的氮循环过程、功能基因及功能微生物[3, 5, 24, 25], 但同时关联多个氮循环功能基因及微生物的研究鲜有报道.本研究基于宏基因组测序及NCycDB氮循环功能基因数据库, 研究了有机质提升对酸性红壤氮循环功能基因丰富及相关功能微生物组成的影响.虽然本研究仅测定了一个时间点的数据, 但是研究对象是经过32 a严格、持续的长期定位施肥管理的具有稳定生态系统的土壤, 且选取冬休结束时刻进行采样以避免当年的人为干扰, 其结果应该能够代表该地区酸性红壤中氮循环功能基因丰度及微生物组成的变化对有机质提升的响应.
硝化过程被认为是氮循环过程的中心环节, 氨氧化是硝化过程的第一及限速步骤[15].本研究发现有机质提升显著增加了土壤净硝化活性和氨氧化潜势(P < 0.05), 增幅分别为70.6%和78.6%(表 1), 和已报道研究结果一致[3, 16, 19].Wang等[3]在2014年调查该地区土壤, 也发现有机质提升增加了土壤净硝化活性45.5%;He等[19]调查了湖南祁阳红壤实验站小麦-玉米轮作的酸性红壤, 相比于不施肥土壤, 16 a的长期有机肥-无机肥配施提升了56.6%的土壤有机质含量, 土壤氨氧化潜势也增加了196%;Liu等[16]调查江西千烟洲生态试验站桔园施肥试验也发现类似结果.氨氧化古菌(AOA)和氨氧化细菌(AOB)是执行氨氧化过程的功能微生物[22].AOA普遍存在于各种土壤环境中[26], 且通常丰度较高[24];大部分研究结果均认为pH[3, 25, 27]、底物浓度[28]、有机物含量[29, 30]和氧气含量[31]等是影响氨氧化微生物在土壤中生长并发挥氨氧化功能的主要因素.例如, 研究者运用稳定同位素探针技术(SIP)发现在中性或者偏碱性(pH 6.9~8.0)土壤中AOB具有较高氨氧化活性[27, 32];但在强酸性土壤(pH <5.5)中仅检测到AOA具有自养氨氧化活性[3, 25, 33].本研究土壤经过32 a的有机质输入, 土壤有机质提升的同时, 土壤pH值(水溶液提取)从4.97上升至6.35(表 1);前期SIP研究也发现CK土壤中活性氨氧化微生物是AOA, 但OM土壤中活性氨氧化微生物转变为AOB[3], 可见土壤酸化缓解应是有机质提升增加酸化红壤硝化强度的最主要因素.本研究还发现有机质提升后, 古菌和细菌amoA基因绝对丰度均显著增加(表 1), 但是宏基因组测定结果发现仅古菌amoA基因丰度显著增加, 细菌amoA基因丰度略有增加, 但统计上不显著(图 1).绝对定量和相对丰度的结果不一致的原因可能是:土壤中古菌/细菌amoA基因绝对丰度比是151~160, 即酸性红壤中AOA占据明显数量优势, AOB群落丰度较低;而宏基因组测序结果仅在CK和OM土壤中找到10~14条细菌amoA基因序列, 即测序深度不足可能是CK和OM土壤中细菌amoA基因相对丰度无差异的主要原因.另外, 有研究还发现AOA具有独特的代谢途径和生理代谢特点, 也可能在有机质含量较高的土壤具有生长优势.Alonso-Sáez等[29]在海洋中发现AOA具有尿素水解为基础的氨氧化代谢路径[30], Lu等[33, 34]在酸性土壤(pH 3.75~5.40)硝化作用中占据主导地位的AOA中也发现了脲酶基因, 证实复杂的土壤环境中也存在以尿素水解为基础的古菌氨氧化代谢路径.许多研究还表明AOA具有利用有机底物的潜力[35, 36]或能够受有机底物刺激生长[37, 38].本研究有机质提升降低了土壤AOA属Nitrososphaera和Nitrosopumilus相对丰度, 而增加了AOB属Nitrosospira和Nitrosomonas相对丰度(图 2).相比于AOB和亚硝酸盐氧化细菌, AOA代时较长、生长较慢[22, 28], 导致有机质提升后古菌amoA基因丰度显著上升(表 1), 而Nitrososphaera和Nitrosopumilu相对丰度分别降低了7.7%和1.3%(图 2).大数据分析发现Nitrososphaera分布与土壤pH值呈正相关[39], 但是SIP试验证明Nitrososphaera在酸性红壤中有重要生态活性[40];本研究有机质提升后土壤pH值上升了近1.5个单位(表 1), 导致Nitrososphaera相对丰度有所下降.而Nitrosopumilus是海洋中普遍存在的AOA类群[26], 在土壤中丰度较低, 在低营养的酸性土壤中有分布[3], 本研究中Nitrosopumilus相对丰度也较低(2.1% ~3.4%), pH上升后其生长也不占优势.本研究还发现有机质提升显著降低了编码羟胺氧化酶的hao基因丰度(图 1), 但是目前对于hao基因主要还集中在纯菌株的基因结果和功能蛋白研究上[41], 所以尚无法解释在复杂的土壤生态系统中基因丰度的变化.土壤中亚硝酸氧化过程是非限速步骤[15], 因此本研究发现有机质提升对编码亚硝酸盐氧化酶的nxrAB基因丰度无显著影响, 该结果也得到其他研究证实[32, 33].亚硝酸盐氧化菌优势类群从CK土壤中的Nitrolancea变为OM土壤中的Nitrospira, 因为Nitrospira具有更强的适应性和更高的亚硝酸盐氧化能力[32, 42].
酸性红壤有机质提升显著刺激了反硝化过程各还原酶的功能基因丰度(图 1).Dambreville等[43]的研究发现长期粪肥输入提升了土壤有机质含量6.5%, 显著刺激反硝化过程, 刺激了N2O还原酶活性, 从而降低了土壤N2O排放量;Yu等[5]在调查酸性红壤反硝化活性时, 发现高有机质含量的针叶林土壤NO排放量显著高于低有机质含量的农田土壤.由于缺乏合适的保守序列和分类标准, 目前对于反硝化微生物的研究还停留在功能基因上.反硝化微生物是异养微生物[44], 有机质提升后带来的TOC和TN含量的增加(表 1), 以及土壤pH的提升[5], 应是反硝化过程各还原酶的功能基因丰度上升的主要原因.以往一直认为细菌是主要的反硝化微生物, 近年来也发现真菌在酸性土壤N2O产生过程中具有重要作用[45, 46], 值得进一步研究.本研究材料是旱地玉米土壤, 不适合需要严格厌氧环境的厌氧氨氧化过程发生[47], 好氧氨氧化过程是铵盐转化的最主要过程, 因此有机质提升并未显著影响厌氧氨氧化过程相关功能基因丰度及功能微生物类群(图 1和图 2).本研究检测到的nif基因丰度较低, 且CK和OM土壤间无显著差异(图 1), 表明该土壤的固氮能力较弱.另外, 厌氧或缺氧条件下, 硝酸盐还能被还原为铵盐, 即DNRA过程, 由于反应能耗高于反硝化过程[48], DNRA过程通常被忽视.虽然本研究是好氧的旱地土壤, 但是也检测出大量的DNRA过程功能基因nrf和nir, 且有机质提升后降低了DNRA过程功能基因nrfABCD的丰度(图 1);相关研究发现土壤总有机碳含量及厌氧环境通常是影响DNRA活性和功能基因nrfA丰度的主要因子[2, 49], 与本研究结论一致.该结果可为旱地土壤无机氮蓄持研究提供启示.
氮源是微生物生长的基本要素之一, 无机氮是土壤微生物生长的主要氮源类型, 尤其是在土壤含氮量较低的酸性土壤中尤为明显;酸性土壤中, 大量无机氮通过硝酸盐淋溶、反硝化和N2O排放等途径流失, 因此增加酸性土壤无机氮同化量可能是提高土壤氮素固持水平的重要途径[15].有研究表明, 微生物会优先同化铵态氮作为氮源[50, 51], 但在高有机碳含量土壤中, 硝态氮也可被微生物迅速同化[52, 53];大数据分析也发现土壤有机碳含量是土壤有机-无机态氮转化的重要影响因子[54].nasAB基因是本研究土壤中执行硝酸盐同化功能丰度最高的功能基因, 有机质提升显著增加了nasAB基因丰度(图 3), 表明有机碳增加会促进硝酸盐同化;有机质提升降低nirA和NR基因丰度的原因, 目前尚不清楚.本研究共检测到24种与有机氮循环相关的功能基因, 涵盖了尿素分解、氨基酸合成和分解代谢[11], 数据具有一定的代表性.长期的有机质输入提高了编码氨基酸、谷氨酸、谷氨酰胺等含氮有机物合成或者分解代谢的功能基因丰度(图 3);Tu等[12]的研究发现大气CO2浓度升高后, 植株分泌的光合作用碳增加, 土壤中编码硝基烷氧化酶的nao基因丰度也相应增加, 与本研究的结果一致.
4 结论(1) 有机质提升显著增加了酸性红壤总有机碳和总氮含量, 缓解了土壤酸化, 增加了土壤净硝化活性和氨氧化潜势.
(2) 有机质提升显著增长了酸性红壤无机氮代谢过程中编码古菌氨单加氧酶及反硝化过程各还原酶的功能基因丰度, 降低了编码羟胺氧化酶及执行DNRA过程的功能基因丰度, 改变了硝化过程功能微生物群落组成.有机质提升显著增加了酸性红壤有机氮代谢过程中谷氨酰胺转化功能基因丰度, 改变了硝酸盐同化过程功能基因丰度.
(3) 有机质提升后土壤酸化的缓解(上升约1.5个单位)及TOC含量的提升(增幅为38.2%)是影响氮循环各过程功能基因丰度及功能微生物组成的最主要因素.研究结果全面关联了无机氮和有机氮循环过程中的功能基因及相关功能微生物类群, 为把握酸性红壤氮循环特征提供数据依据, 并为酸性土壤改良提供思路.
| [1] | Gruber N, Galloway J N. An Earth-system perspective of the global nitrogen cycle[J]. Nature, 2008, 451(7176): 293-296. |
| [2] |
贺纪正, 张丽梅. 土壤氮素转化的关键微生物过程及机制[J]. 微生物学通报, 2013, 40(1): 98-108. He J Z, Zhang L M. Key processes and microbial mechanisms of soil nitrogen transformation[J]. Microbiology China, 2013, 40(1): 98-108. |
| [3] | Wang X L, Han C, Zhang J B, et al. Long-term fertilization effects on active ammonia oxidizers in an acidic upland soil in China[J]. Soil Biology and Biochemistry, 2015, 84: 28-37. |
| [4] |
郭安宁, 段桂兰, 赵中秋, 等. 施加碳酸钙对酸性土壤微生物氮循环的影响[J]. 环境科学, 2017, 38(8): 3483-3488. Guo A N, Duan G L, Zhao Z Q, et al. Effects of CaCO3 application on soil microbial nitrogen cycle in an acid soil[J]. Environmental Science, 2017, 38(8): 3483-3488. |
| [5] | Yu Y J, Zhang J B, Chen W W, et al. Effect of land use on the denitrification, abundance of denitrifiers, and total nitrogen gas production in the subtropical region of China[J]. Biology and Fertility of Soils, 2014, 50(1): 105-113. |
| [6] | Harter J, Krause H M, Schuettler S, et al. Linking N2O emissions from biochar-amended soil to the structure and function of the N-cycling microbial community[J]. The ISME Journal, 2014, 8(3): 660-674. |
| [7] | Danczak R E, Johnston M D, Kenah C, et al. Members of the Candidate Phyla Radiation are functionally differentiated by carbon-and nitrogen-cycling capabilities[J]. Microbiome, 2017, 5: 112. |
| [8] | Souza R C, Cantão M E, Vasconcelos A T R, et al. Soil metagenomics reveals differences under conventional and no-tillage with crop rotation or succession[J]. Applied Soil Ecology, 2013, 72: 49-61. |
| [9] | Ding JJ, Zhang YG, Deng Y, et al. Integrated metagenomics and network analysis of soil microbial community of the forest timberline[J]. Scientific Reports, 2015, 5: 7994. |
| [10] |
孙欣, 汪诗平, 林巧燕, 等. 基于分子生态学网络探究西藏草地放牧对土壤微生物群落的影响[J]. 微生物学通报, 2015, 42(9): 1818-1831. Sun X, Wang S P, Lin Q Y, et al. Molecular ecological network analyses revealing the effects of livestock grazing on soil microbial community in the Tibetan grassland[J]. Microbiology China, 2015, 42(9): 1818-1831. |
| [11] | Tu Q C, Lin L, Cheng L, et al. NCycDB:a curated integrative database for fast and accurate metagenomic profiling of nitrogen cycling genes[J]. Bioinformatics, 2019, 35(6): 1040-1048. |
| [12] | Tu Q C, He Z L, Wu L Y, et al. Metagenomic reconstruction of nitrogen cycling pathways in a CO2-enriched grassland ecosystem[J]. Soil Biology and Biochemistry, 2017, 106: 99-108. |
| [13] | Nelson M B, Martiny A C, Martiny J B H. Global biogeography of microbial nitrogen-cycling traits in soil[J]. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(29): 8033-8040. |
| [14] | Xue K, Yuan M M, Shi Z J, et al. Tundra soil carbon is vulnerable to rapid microbial decomposition under climate warming[J]. Nature Climate Change, 2016, 6(6): 595-600. |
| [15] | De Boer W, Kowalchuk G A. Nitrification in acid soils:micro-organisms and mechanisms[J]. Soil Biology and Biochemistry, 2001, 33(7-8): 853-866. |
| [16] | Liu H F, Wu X, Wang Q, et al. Responses of soil ammonia oxidation and ammonia-oxidizing communities to land-use conversion and fertilization in an acidic red soil of southern China[J]. European Journal of Soil Biology, 2017, 80: 110-120. |
| [17] | Zhong W H, Gu T, Wang W, et al. The effects of mineral fertilizer and organic manure on soil microbial community and diversity[J]. Plant and Soil, 2010, 326(1-2): 511-522. |
| [18] |
徐白璐, 钟文辉, 黄欠如, 等. 长期施肥酸性旱地土壤硝化活性及自养硝化微生物特征[J]. 环境科学, 2017, 38(8): 3473-3482. Xu B L, Zhong W H, Huang Q R, et al. Nitrification activity and autotrophic nitrifiers in long-term fertilized acidic upland soils[J]. Environmental Science, 2017, 38(8): 3473-3482. |
| [19] | He J Z, Shen J P, Zhang L M, et al. Quantitative analyses of the abundance and composition of ammonia-oxidizing bacteria and ammonia-oxidizing archaea of a Chinese upland red soil under long-term fertilization practices[J]. Environmental Microbiology, 2007, 9(9): 2364-2374. |
| [20] | Zhu W C, Lomsadze A, Borodovsky M. Ab initio gene identification in metagenomic sequences[J]. Nucleic Acids Research, 2010, 38(12): e132. |
| [21] | Steinegger M, Söding J. Clustering huge protein sequence sets in linear time[J]. Nature Communications, 2018, 9: 2542. |
| [22] | Francis C A, Roberts K J, Beman J M, et al. Ubiquity and diversity of ammonia-oxidizing archaea in water columns and sediments of the ocean[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(41): 14683-14688. |
| [23] | Rotthauwe J H, Witzel K P, Liesack W. The ammonia monooxygenase structural gene amoA as a functional marker:molecular fine-scale analysis of natural ammonia-oxidizing populations[J]. Applied and Environmental Microbiology, 1997, 63(12): 4704-4712. |
| [24] | He J Z, Hu H W, Zhang L M. Current insights into the autotrophic thaumarchaeal ammonia oxidation in acidic soils[J]. Soil Biology and Biochemistry, 2012, 55: 146-154. |
| [25] | Zhang L M, Hu H W, Shen J P, et al. Ammonia-oxidizing archaea have more important role than ammonia-oxidizing bacteria in ammonia oxidation of strongly acidic soils[J]. The ISME Journal, 2012, 6(5): 1032-1045. |
| [26] | Hatzenpichler R. Diversity, physiology, and niche differentiation of ammonia-oxidizing archaea[J]. Applied and Environmental Microbiology, 2012, 78(21): 7501-7510. |
| [27] | Jia Z J, Conrad R. Bacteria rather than Archaea dominate microbial ammonia oxidation in an agricultural soil[J]. Environmental Microbiology, 2009, 11(7): 1658-1671. |
| [28] | Martens-Habbena W, Berube PM, Urakawa H, et al. Ammonia oxidation kinetics determine niche separation of nitrifying Archaea and Bacteria[J]. Nature, 2009, 461(7266): 976-979. |
| [29] | Alonso-Sáez L, Waller A S, Mende D R, et al. Role for urea in nitrification by polar marine Archaea[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(44): 17989-17994. |
| [30] | Kirchman D L. Marine archaea take a short cut in the nitrogen cycle[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(44): 17732-17733. |
| [31] | Wang B Z, Zhao J, Guo Z Y, et al. Differential contributions of ammonia oxidizers and nitrite oxidizers to nitrification in four paddy soils[J]. The ISME Journal, 2015, 9(5): 1062-1075. |
| [32] | Xia W W, Zhang C X, Zeng X W, et al. Autotrophic growth of nitrifying community in an agricultural soil[J]. The ISME Journal, 2011, 5(7): 1226-1236. |
| [33] | Lu L, Han W Y, Zhang J B, et al. Nitrification of archaeal ammonia oxidizers in acid soils is supported by hydrolysis of urea[J]. The ISME Journal, 2012, 6(10): 1978-1984. |
| [34] | Lu L, Jia Z J. Urease gene-containing Archaea dominate autotrophic ammonia oxidation in two acid soils[J]. Environmental Microbiology, 2013, 15(6): 1795-1809. |
| [35] | Hallam S J, Mincer T J, Schleper C, et al. Pathways of carbon assimilation and ammonia oxidation suggested by environmental genomic analyses of marine Crenarchaeota[J]. PLoS Biology, 2006, 4(4): e95. |
| [36] | Mußmann M, Brito I, Pitcher A, et al. Thaumarchaeotes abundant in refinery nitrifying sludges express amoA but are not obligate autotrophic ammonia oxidizers[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(40): 16771-16776. |
| [37] | Levi Dč nik-Höfferle Š, Nicol G W, Ausec L, et al. Stimulation of thaumarchaeal ammonia oxidation by ammonia derived from organic nitrogen but not added inorganic nitrogen[J]. FEMS Microbiology Ecology, 2012, 80(1): 114-123. |
| [38] |
徐杰, 韩成, 张金波, 等. 有机碳氮添加对酸性森林土壤氨氧化过程的影响[J]. 土壤学报, 2017, 54(4): 1029-1037. Xu J, Han C, Zhang J B, et al. Effects of amendment of organic carbon or nitrogen on ammonia oxidation in the subtropical acidic forest soil[J]. Acta Pedologica Sinica, 2017, 54(4): 1029-1037. |
| [39] | Gubry-Rangin C, Hai B, Quince C, et al. Niche specialization of terrestrial archaeal ammonia oxidizers[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(52): 21206-21211. |
| [40] | Wang B Z, Zheng Y, Huang R, et al. Active ammonia oxidizers in an acidic soil are phylogenetically closely related to neutrophilic archaeon[J]. Applied and Environmental Microbiology, 2014, 80(5): 1684-1691. |
| [41] | Moran M A, Buchan A, González J M, et al. Genome sequence of Silicibacter pomeroyi reveals adaptations to the marine environment[J]. Nature, 2004, 432(7019): 910-913. |
| [42] | Daebeler A, Bodelier P L E, Yan Z, et al. Interactions between Thaumarchaea, Nitrospira and methanotrophs modulate autotrophic nitrification in volcanic grassland soil[J]. The ISME Journal, 2014, 8(12): 2397-2410. |
| [43] | Dambreville C, Hénault C, Bizouard F, et al. Compared effects of long-term pig slurry applications and mineral fertilization on soil denitrification and its end products (N2O, N2)[J]. Biology and Fertility of Soils, 2006, 42(6): 490-500. |
| [44] | Zhang J B, Cai Z C, Zhu T B. N2O production pathways in the subtropical acid forest soils in China[J]. Environmental Research, 2011, 111(5): 643-649. |
| [45] | Zhang J B, Müller C, Cai Z C. Heterotrophic nitrification of organic N and its contribution to nitrous oxide emissions in soils[J]. Soil Biology and Biochemistry, 2015, 84: 199-209. |
| [46] | Shoun H, Fushinobu S, Jiang L, et al. Fungal denitrification and nitric oxide reductase cytochrome P450nor[J]. Philosophical Transactions of the Royal Society B:Biological Sciences, 2012, 367(1593): 1186-1194. |
| [47] | Qin H, Han C, Jin Z, et al. Vertical distribution and community composition of anammox bacteria in sediments of a eutrophic shallow lake[J]. Journal of Applied Microbiology, 2018, 125(1): 121-132. |
| [48] | Fdz-Polanco F, Fdz-Polanco M, Fernández N, et al. Combining the biological nitrogen and sulfur cycles in anaerobic conditions[J]. Water Science and Technology, 2001, 44(8): 77-84. |
| [49] | Rütting T, Boeckx P, Müller C, et al. Assessment of the importance of dissimilatory nitrate reduction to ammonium for the terrestrial nitrogen cycle[J]. Biogeosciences, 2011, 8(7): 1779-1791. |
| [50] | Recous S, Machet J M, Mary B. The partitioning of fertilizer-N between soil and crop:comparison of ammonium and nitrate applications[J]. Plant and Soil, 1992, 144(1): 101-111. |
| [51] |
程谊, 黄蓉, 余云飞, 等. 应重视硝态氮同化过程在降低土壤硝酸盐浓度中的作用[J]. 土壤学报, 2017, 54(6): 1326-1331. Cheng Y, Huang R, Yu Y F, et al. Role of microbial assimilation of soil in reducing soil concentration[J]. Acta Pedologica Sinica, 2017, 54(6): 1326-1331. |
| [52] | Cheng Y, Wang J, Wang J Y, et al. The quality and quantity of exogenous organic carbon input control microbial NO3- immobilization:a meta-analysis[J]. Soil Biology and Biochemistry, 2017, 115: 357-363. |
| [53] | Romero C M, Engel R, Chen C C, et al. Microbial immobilization of nitrogen-15 labelled ammonium and nitrate in an agricultural soil[J]. Soil Science Society of America Journal, 2015, 79(2): 595-602. |
| [54] | Colman B P, Schimel J P. Drivers of microbial respiration and net N mineralization at the continental scale[J]. Soil Biology and Biochemistry, 2013, 60: 65-76. |
 2020, Vol. 41
2020, Vol. 41


