2. 北京市农林科学院北京农业生物技术研究中心, 北京 100097
2. Beijing Agricultural Biotechnology Research Center, Beijing Academy of Agriculture and Forestry Sciences, Beijing 100097, China
抗生素作为20世纪最重要的发现之一, 在保障人类及动物健康以及促进畜禽养殖业发展方面起着不可替代的作用[1]. 1950年美国食品与药品监管局(FDA)首次批准抗生素可作为饲料添加剂, 抗生素因此被全面推广应用于动物养殖业[2].然而近年研究发现, 摄入动物体内的抗生素大约有30%~90%以原药和代谢产物的形式通过粪尿排出体外进入环境中[3], 导致土壤和水环境中大量抗生素的残留, 从而引起耐药菌的富集.抗生素抗性基因(antibiotic resistance genes, ARGs)通常位于质粒、转座子、整合子等可移动遗传元件(mobile genetic elements, MGEs)上, 可通过基因水平转移在菌群间迁移, 引起抗生素耐药菌的扩散;甚至可能转移到人类共生微生物和病原体中[4]. 2006年, Pruden等[5]首次提出ARGs是一种新型的环境污染物, 有关ARGs在环境中传播和污染等的报道日益增多[6].
土壤被认为是抗生素残留的最重要的受纳体之一, 抗生素和ARGs主要是通过粪肥与污泥的施用[7]以及废水或再生水灌溉[8]等方式释放到土壤中.中国是畜禽养殖大国, 全国各种规模畜禽粪便鲜重产量为22.28亿t[9].畜禽粪便作为有机肥料广泛应用于农业生产中, 尤其是蔬菜种植业, 然而畜禽粪便中普遍富含抗生素, 不可避免地会造成土壤中抗生素的残留污染[10~16].土壤中残留的抗生素可能被蔬菜等作物吸收积累, 抗性菌与抗性基因亦可能通过食用生食蔬菜过程进入人体, 引起人类的健康风险.因此, 研究蔬菜基地土壤中抗生素含量、抗性基因的丰度及其水平转移能力是十分有必要的.
目前关于蔬菜基地土壤中的抗生素的研究较多, 主要集中在蔬菜基地土壤中抗生素的检出率、种类、丰度以及不同蔬菜与土壤类型中抗生素的组成差异.综合分析蔬菜基地土壤中检测率较高的抗生素为四环素、土霉素、金霉素与强力霉素[17].我国北方地区蔬菜基地土壤中残留的抗生素浓度在0.1~2 683 μg·kg-1, 其中土霉素含量较高[18].不同蔬菜土壤中抗生素的种类和含量有较大差异, 蔬菜基地土壤抗生素残留水平普遍高于陆地土壤, 部分蔬菜基地土壤中的抗生素残留如四环素、诺氟沙星和氧氟沙星的含量超过了欧盟规定的生态安全触发线(100 μg·kg-1)[12].
目前对于土壤中抗生素抗性基因的分布特征亦有较多报道, 主要针对的是畜禽养殖场及周边土壤中抗生素抗性基因的研究, Ji等[19]在上海地区养猪场、养鸡场和养牛场畜禽粪便及周围土壤中均检测到八类抗性基因, 丰度较高的抗生素抗性基因是sulA和sulⅡ.Wu等[20]分析北京、天津及浙江嘉兴三地的猪饲养场周边土壤发现了15种四环素类抗性基因, 其中丰度较高的抗生素抗性基因型为tetM、tetO、tetQ、tetW和tetT;在我国南方地区, 邹世春等[21]在珠海某养殖场周边土壤检测到多种四环素类抗性基因, 其中含量较高的抗性基因亚型是tetG、tetC和 tetO.研究者应用定量PCR对哈尔滨粪肥施用土壤中的四环素类和大环内酯类抗生素抗性基因进行检测, 发现tetM、tetW、emrB和emrF的丰度较高[22].新近研究发现, 与不施肥相比, 长期施用粪肥的蔬菜基地土壤中有十五类抗生素抗性基因亚型得到富集, 如sul1、sul2、tetX、tetL、vanR、tcmA、rosB和mexF等[23].综上研究表明养殖场周边或施用粪肥土壤中抗生素抗性基因的多样性增加、丰度升高, 但是关于不同的蔬菜基地土壤中抗生素抗性基因与可移动元件的分布特征缺乏系统地横向比较研究, 哪些抗生素、抗生素抗性基因及可移动元件在蔬菜基地土壤中的丰度较高?不同蔬菜基地土壤中是否存在一些共性特征?仍然未知.
因此, 本文应用高通量荧光定量PCR方法(同时检测285种ARGs、10种MGEs及16S rRNA), 对北京顺义、昌平和通州这3个区, 5个蔬菜基地土壤中抗生素抗性基因与可移动元件进行全面系统研究.以掌握北京主要蔬菜基地土壤中ARGs与MGEs的分布特征, 以期为控制ARGs的传播提供基础数据.
1 材料与方法 1.1 样品采集选取北京顺义、昌平和通州区的5个典型蔬菜种植基地作为采样点, 每个采样点采集3份重复样品, 共15份土壤样品.土壤过2 mm筛, 保存于-80℃冰箱中, 用于后续抗生素含量测定和DNA提取.
样本的基本信息如表 1所示.
|
|
表 1 样本的基本信息 Table 1 Basic information of the samples |
1.2 抗生素的检测方法
依据本实验室已建立的固相萃取-超高效液相色谱-串联质谱(UPLC-MS/MS)的方法[24], 同时对蔬菜基地土壤中的四环素类[金霉素(CTC)、四环素(TC)、土霉素(OTC)、强力霉素(DOX)]、磺胺类[磺胺二甲氧嘧啶(SDM)、磺胺喹啉(SQ)、磺胺氯哒嗪(SCP)、磺胺甲唑(SMX)、磺胺二甲嘧啶(SM2)]、氟喹诺酮类[诺氟沙星(NOR)、恩诺沙星(ENR)、环丙沙星(CIP)]和大环内酯类[红霉素(ERY)、罗红霉素(ROX)、泰乐菌素(TS)]15种抗生素进行检测.以50%乙腈(体积分数)的磷酸盐缓冲溶液(pH=4)提取3次, 经过超声、离心、旋蒸和稀释后, SAX-HLB串联小柱净化富集, 用10 mL甲醇洗脱, 35℃氮吹近干后, 用含0.1%甲酸-甲醇(1:1, 体积比)定容, 在UPLC-MS/MS反应检测模式下进行定量分析, 其中四环素类、磺胺类、氟喹诺酮类和大环内酯类抗生素的平均加标回收率为56.4%~94.6%, 相对标准偏差(RSD)在2.6%~19.8%之间, 方法检出限(LOD, S/N=3)和定量限(LOQ, S/N=10)分别为0.01~2.50μg·kg-1和0.05~7.90μg·kg-1[24].
1.3 DNA提取样品的基因组提取使用MoBio PowerSoil DNA Isolation Kit (MoBio Laboratories, Carlsbad, CA, USA), 按照说明书要求进行提取.DNA提取完毕后, 用微量核酸蛋白质分析仪(Nanodrop)检测含量以及纯度, A260/A280在1.8~2.0之间, 表明DNA纯度可进行后续分析.
1.4 抗生素抗性基因与可移动元件的HT-qPCR定量检测为了测量样品中抗生素抗性基因的丰富度和多样性, 采用Wafergen智能芯片实时PCR系统进行抗生素抗性基因的高通量定量PCR分析.PCR设置过程中设置了296对引物, 包括284种靶向抗生素抗性基因、8个转座酶、1个16S rRNA基因、1个blaNDM-1基因, 2个1类整合子基因(intI1和cinT).这284个抗生素抗性基因的引物几乎覆盖了所有已知的抗生素抗性基因亚型.HT-qPCR的384板每个孔样品预混液体系包括:12.8 μL样品预混液[SYBR Green I Mix (2X) 8 μL, 1 mg·mL-1牛血清蛋白0.8 μL, 无菌水4 μL]与3.2 μL稀释好的DNA(20~40 ng·μL-1);384板每个孔引物预混液体系包括:9.6 μL引物预混液[SYBR Green I Mix (2X) 6 μL, 无菌水3.6 μL]与2.4 μL引物.将引物预混液与样品预混液喷于同一芯片上, 进行后续扩增分析.热循环过程是:95℃10 min, 然后在95℃下30 s, 60℃下30 s, 进行40次循环.ARGs和MGE的相对拷贝数可通过公式计算得出(1), 然后将16S rRNA进行绝对定量, 计算其绝对拷贝数, 依据16S rRNA的绝对拷贝数计算抗生素抗性基因和MGE的绝对拷贝数[25].
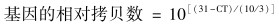
|
式中, CT值为检测基因的荧光信号达到设定的并阈值时所需的循环数.
1.5 数据分析采用R语言PCoA软件包绘制主坐标分析(PCoA)图, 分析不同处理中总的抗生素抗性基因的组成特征.利用R语言pheatmap软件包绘制热图, 对比分析不同处理中抗生素抗性基因丰度差异.采用R语言TukeyHSD方法对不同处理中抗生素抗性基因丰度是否存在差异进行统计分析.应用Origin绘图软件进行柱形图与气泡图的绘制.
2 结果与讨论 2.1 抗生素抗性基因组成与多样性分析蔬菜基地土壤中检测到92~121种抗生素抗性基因亚型, 分别隶属于氨基糖苷类抗生素抗性基因(Aminoglycoside)、β-内酰胺酶抗生素抗性基因(β-Lactamase)、氯霉素抗生素抗性基因(Chloramphenicol)、大环内酯类-林肯酰胺类-链阳性菌素B类抗生素抗性基因(MLSB)、多重耐药抗生素抗性基因(Multidrug)、磺胺类抗生素抗性基因(Sulfonamide)、四环素抗生素抗性基因(Tetracycline)和万古霉素抗生素抗性基因(Vancomycin)等;检测到4~6种可移动元件.顺义S2基地土壤的ARGs和MGEs的数目最高, 通州T蔬菜基地土壤的数目最低(图 1).
|
C1、C2分别表示昌平的2个蔬菜基地;SI、S2表示顺义的 2个蔬菜基地;T表示通州蔬菜基地,下同 图 1 不同蔬菜基地土壤中抗生素抗性基因与可移动元件的数目分析 Fig. 1 Number analysis of antibiotic resistance genes and mobile genetic elements in soil of different vegetable base soils |
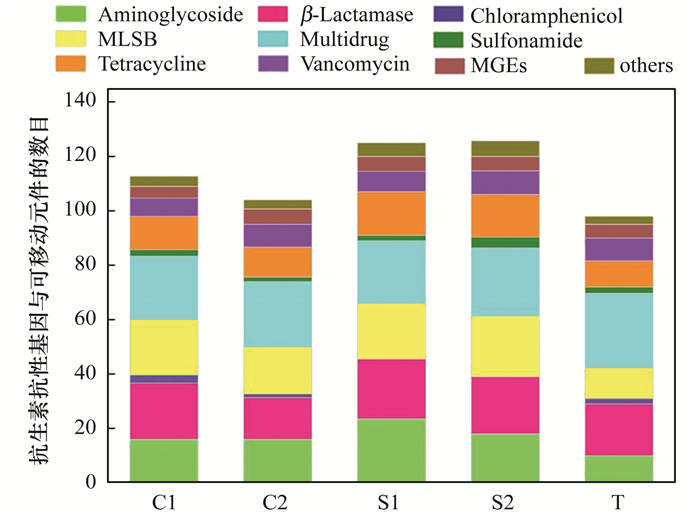
为研究不同蔬菜基地中抗生素抗性基因组成的特点, 通过PCoA分析对抗性基因数据进行分析(图 2), 不同地区的抗生素抗性基因存在差异, 昌平两个蔬菜基地(C1和C2)土壤中的抗生素抗性基因聚集在一起、顺义区两个蔬菜基地(S1和S2)土壤中的抗生素抗性基因聚集在一起, 与通州区基地(T)土壤中的抗生素抗性基因分开.表明同一区域内的抗生素抗性基因组成相似, 不同区内的抗生素抗性基因差异较大.前人研究发现大田土壤和蔬菜基地土壤中的抗生素抗性基因种类存在一定差异, 但均检测到较高含量的磺胺类、四环素类、喹诺酮类抗生素抗性基因以及可移动元件intI1[26], 然而在本研究中没有检测到高丰度的喹诺酮类抗生素抗性基因.而Chen等[27]也用HT-qPCR的方法对土壤进行了抗生素抗性基因的检测, 检测结果和本研究的结果类似.多重耐药类抗生素抗性基因都是检测含量最高的, 大约占到20%, 其次是大环内酯类-林肯酰胺类-链阳性菌素B类抗生素抗性基因、万古霉素、氨基糖苷类和四环素类抗性基因.由此说明, 不同地区土壤中抗生素抗性基因、整合子的种类与丰度存在差异, 推测不同地区抗生素抗性基因存在差异的原因可能与土壤类型、施肥方式、重金属含量、抗生素含量等相关.
|
图 2 不同蔬菜基地土壤中抗生素抗性基因与可移动元件组成的差异 Fig. 2 Differences of antibiotic resistance genes and mobile genetic elements in soil of different vegetable base soils |
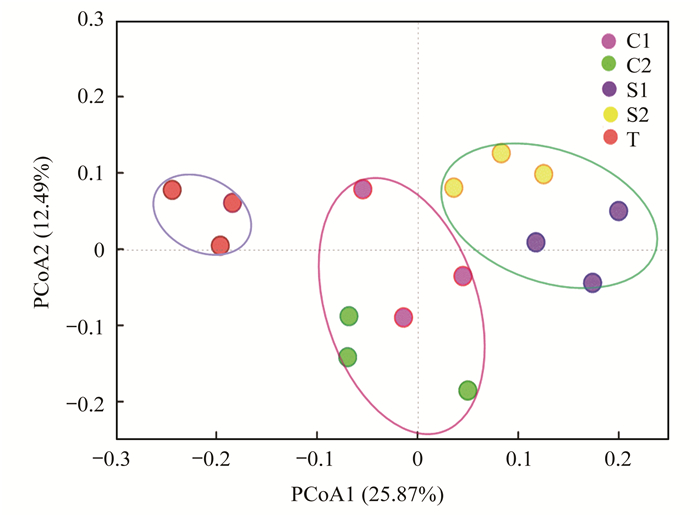
不同地区蔬菜基地土壤的抗生素抗性基因组成存在明显差异, 不同地区的主要抗生素抗性基因型、抗性机制存在哪些共性或差异, 其是否与可移动元件存在相关性?为阐明此问题, 应用Heatmap及Mantel test对抗性基因及其与可移动元件的相关性进行比较分析.结果显示5个蔬菜基地中均丰度较高的抗生素抗性基因型为多重耐药、大环内酯类-林可霉素类-链阳霉素和β-内酰胺类抗性基因型[图 3(a)].不同区丰度较高的抗性基因型亦存在一定差异, 顺义区(S1)的磺胺类、氨基糖苷类、四环素类抗性基因的相对丰度显著高于昌平区(C1和C2)与通州区(T)(P < 0.05).通州区(T)土壤万古霉素抗性基因的相对丰度较高, C1、S1与T区MGEs的相对丰度较高, 但与其它区相比差异未达到显著水平(P>0.05).抗生素抗性基因与可移动元件丰度分析结果也表明丰度较高的抗性基因与热图结果一致.总抗生素抗性基因丰度比较为S1>S2>C1>T>C2, 其中顺义S1基地土壤中总抗生素抗性基因丰度显著高于其它基地(P < 0.05);总的可移动元件丰度为S1>C1>T>S2>C2 [图 3(b)].可见, 顺义S1基地土壤中抗性基因与可移动元件的丰度均较高.
|
(a)抗生素抗性基因热图;(b)相对丰度 图 3 不同蔬菜基地土壤中抗生素抗性基因型比较分析 Fig. 3 Comparative analysis of antibiotic resistance genotypes in different vegetable base soils |
之前对河南菜田土壤中的ARGs及MGEs进行检测研究, 结果表明丰度较高的抗生素抗性基因和可移动元件分别为四环素类抗生素抗性基因, 磺胺类抗生素抗性基因, β-内酰胺酶抗生素抗性基因和intI1、intI2和ISCR2这3种可移动元件[28].四环素是当前世界上使用最广泛的抗生素之一[20], 因此在环境中特别是农田土壤环境中, 四环素类抗生素抗性基因丰度较高.与四环素类抗生素一样, 磺胺类抗生素抗性基因的检出率也非常高, 二者均被证实广泛存在于土壤和废水处理厂等环境中[29, 30].本研究中北京地区5个蔬菜基地中四环素类和磺胺类抗生素抗性基因并不是丰度较高抗生素抗性基因;而β-内酰胺类抗性基因和可移动元件intI1的丰度较高, 与前人的研究结果一致.由此表明ARGs和可移动元件的分布可能是多种环境因素共同选择的结果.
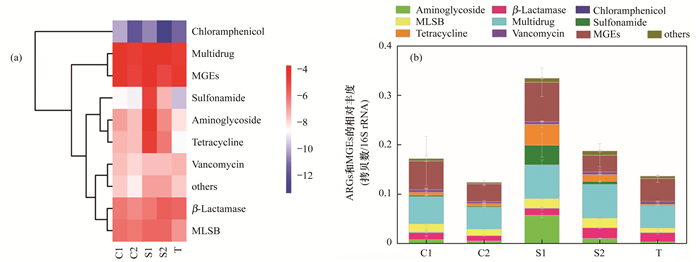
深入比较分析不同蔬菜基地土壤中抗生素抗性基因的机制, 结果显示细胞外排泵与抗生素失活均为5个基地土壤中主要的抗性机制(图 4).但不同区抗性机制的相对丰度存在差异, 与其它地区相比, 顺义S1蔬菜基地土壤抗生素失活和细胞保护的相对丰度较高, 而细胞外排泵抗性机制的相对丰度较低.通州区T蔬菜基地则细胞外排泵抗性机制占比较大, 抗生素失活和细胞保护占比较小.不同区抗生素抗性机制的差异亦反映了不同蔬菜基地土壤中抗生素抗性微生物与抗性基因的差异.
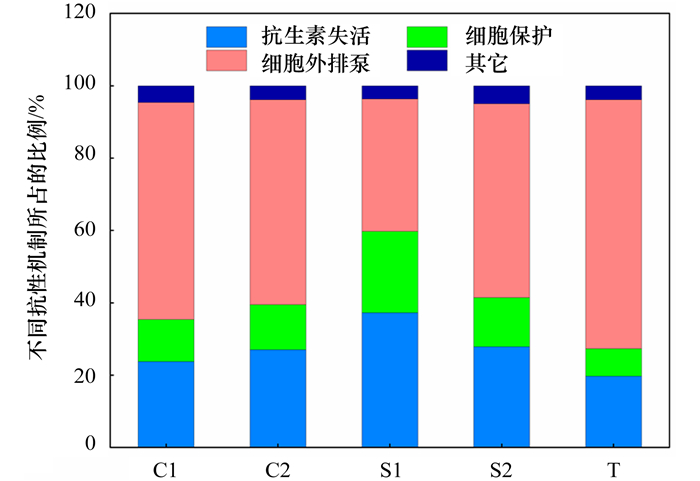
|
图 4 抗生素抗性机制分析 Fig. 4 Mechanism analysis of antibiotic resistance |
为评估蔬菜基地土壤中抗生素抗性基因水平转移风险, 研究不同类型抗生素抗性基因与可移动元件的相关性(表 2).发现氨基糖苷类与磺胺类抗生素抗性基因与MGEs呈显著正相关(P < 0.05).表明这两类抗性基因存在水平转移的风险.
|
|
表 2 抗生素抗性基因与MGEs的相关性分析 Table 2 Correlation analysis between antibiotic resistance genes and MGEs |
关于可移动元件与抗生素抗性基因之间的关系, 之前Ma等[31]应用高通量测序技术和宏基因组学分析证明了不同生态系统中可移动元件与ARGs丰度之间都表现了较高的联系, 但是在不同的生态系统中也是存在较大差异的, 其中在污水处理厂之间的相关系数较低, 而在河水, 养殖场附近土壤中的相关系数较高.其它研究结果也表明, 河口环境可移动元件与ARGs丰度之间也表现了很高的相关性, 其中多重耐药类、氨基糖苷类、β-内酰胺酶类抗性基因丰度与可移动元件丰度相关系数较高[32].以上研究均说明, 可移动元件与抗生素抗性基因之间普遍存在着较高的相关性, 表明环境中抗生素抗性基因存在水平转移风险.
2.3 共有抗性基因与可移动元件组成分析蔬菜基地土壤中均存在丰富的抗生素抗性基因与可移动元件, 其数目及组成在不同蔬菜基地土壤中存在一定差异, 那么不同蔬菜基地中是否存在一些共有的抗生素抗性基因亚型及可移动元件?为此本研究对不同蔬菜基地土壤中的共有的抗性基因亚型的数目、组成及相对丰度进行分析.各蔬菜基地中共有抗性基因数目为39, 共有可移动元件为intI1, 占各基地中总的抗性基因与可移动元件数目的31.4%~41.2%.共有的抗生素抗性基因在通州区T蔬菜基地土壤中的比例最高, 占T检测到总抗性基因数目的41.2%.共有抗生素抗性基因在顺义区S1与S2基地土壤中的比例较低, 约占检测到总抗性基因数目30% [图 5(a)].进一步对共有的抗生素抗性基因进行PCoA分析, 结果发现, 共有抗性基因与总的抗性基因的分布一致, 共有抗性基因与可移动元件的组成亦是按区分开[图 5(b)].不同区共有抗生素抗性基因亚型的数目一致, 但组成分布不同, 表明共有抗生素抗性基因亚型的丰度存在差异.
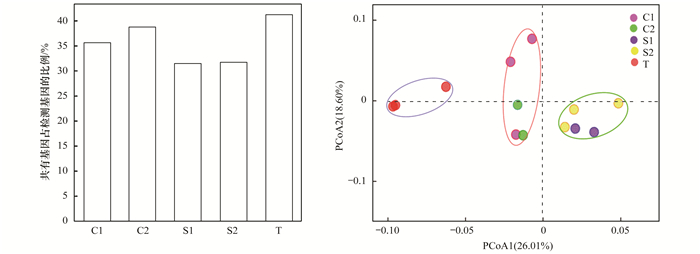
|
(a)共有抗生素抗性基因数目占各区中总抗性基因数目的比例;(b)蔬菜基地中共有抗生素抗性基因的组成差异分析 图 5 共有抗生素抗性基因比例及其组成差异 Fig. 5 Proportional and compositional differences of antibiotic resistance genes |
进一步对蔬菜基地中共有抗性基因亚型的丰度进行分析(图 6), 各基地土壤中主要的抗生素抗性基因亚型为多重耐药类抗生素抗性基因oprD、acrA-04和acrA-05, MLSB类抗生素抗性基因mphA-01, β-内酰胺酶类抗生素抗性基因fox5, 万古霉素类抗性基因vanC-03.S1中多重耐药抗生素抗性基因qacEdelta1-01, β-内酰胺酶类的ampC-04, 氨基糖苷类aadE、aadA-1-02和aacC抗性基因型的丰度高于其它蔬菜基地土壤.已有研究对上海养猪场附近的农田土壤进行抗生素抗性基因的分析, 得到丰度较高的抗生素抗性基因为磺胺类sulA和sulI及四环素tetM和tetW[33], 对磺胺甲恶唑污染过的土壤进行抗生素抗性基因分析, 丰度较高的抗生素抗性基因为磺胺抗性基因sulI和sulⅡ;喹诺酮抗性基因qnrS1、qnrS2、gyrA1、cmlA1和floR;红霉素抗性基因ermB;四环素抗性基因tetA/P、tetM1、tetG1、tetG2、tet(34)和tetM等[34].可见蔬菜基地土壤中抗生素抗性基因与大田土壤中主要的抗生素抗性基因不同, 施用不同类型的粪肥或土壤受到抗生素污染的种类不同均会引起土壤中抗生素抗性基因组成的差异, 亦说明土壤中的抗生素抗性菌易受到人为活动影响.
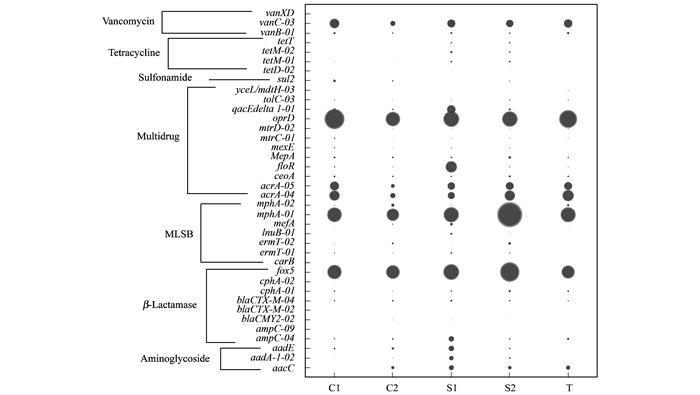
|
图 6 蔬菜基地土壤中共有抗性基因亚型分析 Fig. 6 Analysis of resistance gene subtypes in vegetable base soils |
不同蔬菜基地中抗生素抗性基因亚型及丰度存在差异, 蔬菜基地土壤中的抗生素种类是否存在差异, 其是否与抗性基因的分布存在相关性?为此, 本研究对蔬菜基地中的土壤进行了15种常用的抗生素检测, 共检测到7种抗生素, 其中恩诺沙星(ENR)抗生素丰度最高, 顺义S2蔬菜基地恩诺沙星含量大约为154.41 μg·kg-1, 其次是诺氟沙星(NOR)为38.68 μg·kg-1.顺义S2蔬菜基地中磺胺二甲嘧啶(SM2)抗生素要高于其它4个区, 抗生素的浓度随地区的变化而有所差异.各基地中共有的抗生素为恩诺沙星(ENR)与磺胺二甲嘧啶(SM2);顺义区S1与S2基地检测到的抗生素种类较多, 如金霉素(CTC)、环丙沙星(CIP)和土霉素(OTC)等也有检出. 5个蔬菜基地中抗生素的丰度依次是S2>S1>T>C2>C1.相关性分析结果表明蔬菜基地土壤中抗生素的丰度与抗性基因的丰度存在显著的相关性[P < 0.01, 图 7(b)].
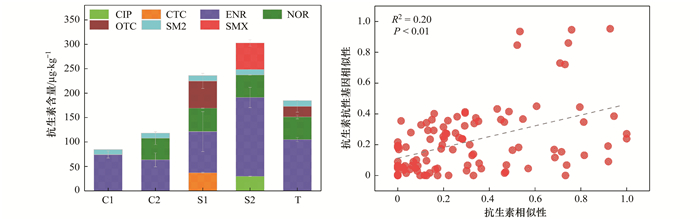
|
(a)土壤中抗生素分析;(b)抗性基因与抗生素的相关性分析; 相似性是指不同处理中抗生素或抗生素抗性基因组成的相似性, 包括抗生素和抗生素抗性基因的种类和丰度的比较 图 7 蔬菜基地土壤抗性基因与抗生素的相关性分析 Fig. 7 Correlation analysis of soil resistance genes and antibiotics in vegetable base soils |
Li等[35]对北京温室蔬菜土壤中抗生素进行检测, 发现温室土壤中的抗生素浓度高于大田土壤, 且四环素(102 μg·kg-1)>喹诺酮类(86 μg·kg-1)>磺胺类(1.1 μg·kg-1)>大环内酯类(0.62 μg·kg-1), 土霉素、氯霉素、诺氟沙星、环丙沙星和恩诺沙星对土壤微生物具有较高的风险.环境中的抗生素残留物会影响陆生生物, 改变土壤中的微生物活动和群落组成, 并促进抗生素抗性基因的发展, 对人类和动物健康均构成威胁.土壤中抗生素主要来源于粪肥、污泥或养殖废水的灌溉, 土壤中抗生素累积及削减受多种因素影响, 例如施肥量和施用方法, 栽培密度, 土壤性质或温室栽培年龄[18, 36, 37].而对于抗生素与抗性基因之间关系的研究, 不同的研究者得到的结论也不尽相同, Smith等[38]发现养殖场污水中四环素ARGs与抗生素之间的相关性不明显;Ji等[19]对猪场粪污及周边土壤的研究也得到了相似的结果.而Tang等[39]和McKinney等[40]发现农田土壤和养殖场粪污中四环素和磺胺ARGs与抗生素含量显著正相关.以上不一致的研究结果暗示ARGs不仅仅与抗生素相关, 还可能受其他因素影响, 比如重金属、pH值及有机质含量等[40].
3 结论(1) 蔬菜基地土壤中检测到92~121种抗生素抗性基因亚型, 4~6种可移动元件.各蔬菜基地中共有抗性基因数目为39, 共有可移动元件为intI1.总的抗生素抗性基因与可移动元件组成按地区分开, 共有抗性基因与可移动元件的组成亦是按地区分开.
(2) 蔬菜基地中丰度较高的抗生素抗性基因型为多重耐药类oprD、acrA-04和acrA-05, MLSB类mphA-01, β-内酰胺酶类fox5, 万古霉素类vanC-03.
(3) 检测到蔬菜基地土壤中抗生素抗性基因的抗性机制主要为抗生素失活与细胞外排泵.
(4) 可移动元件与氨基糖苷类、万古霉素、多重耐药及MLSB类抗性基因均呈显著正相关性.土壤中抗生素的丰度与抗性基因的丰度存在显著的正相关性.
| [1] | Moore P R, Evenson A, Luckey T D, et al. Use of sulfasuxidine, streptothricin, and streptomycin in nutritional studies with the chick[J]. Journal of Biological Chemistry, 1946, 165(2): 437-441. |
| [2] | 马越, 李景云, 金少鸿. 美国食品药品监督管理局修订有关人用抗生素类药品说明书的新规定[J]. 中国药事, 2003, 17(10): 643-645. DOI:10.3969/j.issn.1002-7777.2003.10.027 |
| [3] | Fang H, Han Y L, Yin Y M, et al. Variations in dissipation rate, microbial function and antibiotic resistance due to repeated introductions of manure containing sulfadiazine and chlortetracycline to soil[J]. Chemosphere, 2014, 96: 51-56. DOI:10.1016/j.chemosphere.2013.07.016 |
| [4] | Forsberg K J, Reyes A, Wang B, et al. The shared antibiotic resistome of soil bacteria and human pathogens[J]. Science, 2012, 337(6098): 1107-1111. DOI:10.1126/science.1220761 |
| [5] | Pruden A, Pei R T, Storteboom H, et al. Antibiotic resistance genes as emerging contaminants:studies in northern Colorado[J]. Environmental Science & Technology, 2006, 40(23): 7445-7450. |
| [6] |
苏建强, 黄福义, 朱永官, 等. 环境抗生素抗性基因研究进展[J]. 生物多样性, 2013, 21(4): 481-487. Su J Q, Huang F Y, Zhu Y G, et al. Antibiotic resistance genes in the environment[J]. Biodiversity Science, 2013, 21(4): 481-487. |
| [7] | Chee-Sanford J C, Mackie R I, Koike S, et al. Fate and transport of antibiotic residues and antibiotic resistance genes following land application of manure waste[J]. Journal of Environmental Quality, 2009, 38(3): 1086-1108. DOI:10.2134/jeq2008.0128 |
| [8] | Chen C Q, Li J, Chen P P, et al. Occurrence of antibiotics and antibiotic resistances in soils from wastewater irrigation areas in Beijing and Tianjin, China[J]. Environmental Pollution, 2014, 193: 94-101. DOI:10.1016/j.envpol.2014.06.005 |
| [9] |
谢光辉, 包维卿, 刘继军, 等. 中国畜禽粪便资源研究现状述评[J]. 中国农业大学学报, 2018, 23(4): 75-87. Xie G H, Bao W Q, Liu J J, et al. An overview of researches on livestock and poultry excreta resource in China[J]. Journal of China Agricultural University, 2018, 23(4): 75-87. |
| [10] |
史奕, 赵牧秋, 王俊, 等. 设施菜地土壤-植物系统中有机肥源抗生素的影响研究进展[J]. 农业环境科学学报, 2010, 29(S1): 240-244. Shi Y, Zhao M Q, Wang J, et al. Effect of antibiotics from organic manure on protected vegetable soil-plant system:a review[J]. Journal of Agro-Environment Science, 2010, 29(S1): 240-244. |
| [11] |
张慧敏, 章明奎, 顾国平. 浙北地区畜禽粪便和农田土壤中四环素类抗生素残留[J]. 生态与农村环境学报, 2008, 24(3): 69-73. Zhang H M, Zhang M K, Gu G P. Residues of tetracyclines in livestock and poultry manures and agricultural soils from North Zhejiang Province[J]. Journal of Ecology and Rural Environment, 2008, 24(3): 69-73. DOI:10.3969/j.issn.1673-4831.2008.03.014 |
| [12] |
张志强, 李春花, 黄绍文, 等. 土壤及畜禽粪肥中四环素类抗生素固相萃取-高效液相色谱法的优化与初步应用[J]. 植物营养与肥料学报, 2013, 19(3): 713-726. Zhang Z Q, Li C H, Huang S W, et al. Optimization of residual tetracycline analysis in soils and manure using SPE-HPLC and pilot survey in Tianjin[J]. Journal of Plant Nutrition and Fertilizer, 2013, 19(3): 713-726. DOI:10.11674/zwyf.2013.0323 |
| [13] |
李云辉, 吴小莲, 莫测辉, 等. 畜禽粪便中喹诺酮类抗生素的高效液相色谱-荧光分析方法[J]. 江西农业学报, 2011, 23(8): 147-150. Li Y H, Wu X L, Mo C H, et al. High performance liquid chromatogram-fluorimetric analytical method for detection of quinolone antibiotics in livestock's manure[J]. Acta Agriculturae Jiangxi, 2011, 23(8): 147-150. DOI:10.3969/j.issn.1001-8581.2011.08.049 |
| [14] |
王德全, 何思远, 宋伟明, 等. 加速溶剂萃取-超高效液相色谱-串联质谱法测定畜禽粪便堆肥过程中磺胺及其代谢产物的残留量[J]. 理化检验-化学分册, 2017, 53(5): 532-537. Wang D Q, He S Y, Song W M, et al. ASE-UHPLC-MS/MS determination of residual amounts of sulfonamides and their metabolitesin livestock manure during its composting course[J]. Physical Testing and Chemical Analysis (Part B:Chemical Analysis), 2017, 53(5): 532-537. |
| [15] |
贾伟, 李宇虹, 陈清, 等. 京郊畜禽粪肥资源现状及其替代化肥潜力分析[J]. 农业工程学报, 2014, 30(8): 156-167. Jia W, Li Y H, Chen Q, et al. Analysis of nutrient resources in livestock manure excretion and its potential of fertilizers substitution in Beijing suburbs[J]. Transactions of the Chinese Society of Agricultural Engineering, 2014, 30(8): 156-167. DOI:10.3969/j.issn.1002-6819.2014.08.019 |
| [16] |
王虹, 蒋卫杰, 余宏军, 等. 禽畜废弃物中的抗生素及其在蔬菜等农作物的富集[J]. 中国蔬菜, 2011, 1(12): 10-15. Wang H, Jiang W J, Yu H J, et al. Antibiotics in livestock wastes and its enrichment in vegetable crops[J]. China Vegetables, 2011, 1(12): 10-15. |
| [17] |
邰义萍, 莫测辉, 李彦文, 等. 长期施用粪肥菜地土壤中四环素类抗生素的含量与分布特征[J]. 环境科学, 2011, 32(4): 1182-1187. Tai Y P, Mo C H, Li Y W, et al. Concentrations and distribution of tetracycline antibiotics in vegetable field soil chronically fertilized with manures[J]. Environmental Science, 2011, 32(4): 1182-1187. |
| [18] | Hu X G, Zhou Q X, Luo Y. Occurrence and source analysis of typical veterinary antibiotics in manure, soil, vegetables and groundwater from organic vegetable bases, northern China[J]. Environmental Pollution, 2010, 158(9): 2992-2998. DOI:10.1016/j.envpol.2010.05.023 |
| [19] | Ji X L, Shen Q H, Liu F, et al. Antibiotic resistance gene abundances associated with antibiotics and heavy metals in animal manures and agricultural soils adjacent to feedlots in Shanghai; China[J]. Journal of Hazardous Materials, 2012, 235-236: 178-185. DOI:10.1016/j.jhazmat.2012.07.040 |
| [20] | Wu N, Qiao M, Zhang B, et al. Abundance and diversity of tetracycline resistance genes in soils adjacent to representative swine feedlots in China[J]. Environmental Science & Technology, 2010, 44(18): 6933-6939. |
| [21] |
邹世春, 李青, 贺竹梅, 等. 禽畜养殖场土壤抗生素抗性基因污染的初步研究[J]. 中山大学学报(自然科学版), 2012, 51(6): 87-91. Zou S C, Li Q, He Z M, et al. A Preliminary study on the tetracycline resistance genes in the livestock soil, South China[J]. Acta Scientiarum Naturalium Universitatis Sunyatseni, 2012, 51(6): 87-91. |
| [22] |
彭晶, 高英志, 谷月, 等. 鸡粪有机肥对土壤中抗生素抗性基因和整合酶基因的影响[J]. 环境工程学报, 2019, 13(4): 984-991. Peng J, Gao Y Z, Gu Y, et al. Effects of chicken manure organic fertilizer on antibiotic resistance genes and integrase genes in soil[J]. Chinese Journal of Environmental Engineering, 2019, 13(4): 984-991. |
| [23] | Sun Y M, Qiu T L, Gao M, et al. Inorganic and organic fertilizers application enhanced antibiotic resistome in greenhouse soils growing vegetables[J]. Ecotoxicology and Environmental Safety, 2019, 179: 24-30. DOI:10.1016/j.ecoenv.2019.04.039 |
| [24] |
吴丹, 韩梅琳, 邹德勋, 等. 超高效液相色谱-串联质谱法检测鸡粪中16种残留抗生素[J]. 分析化学, 2017, 45(9): 1389-1396. Wu D, Han M L, Zou D X, et al. Ultra performance liquid chromatography-tandem mass spectrometry analysis of 16 kinds of residual antibiotics in chicken manure[J]. Chinese Journal of Analytical Chemistry, 2017, 45(9): 1389-1396. |
| [25] | Su J Q, Wei B, Ouyang W Y, et al. Antibiotic resistome and its association with bacterial communities during sewage sludge composting[J]. Environmental Science & Technology, 2015, 49(12): 7356-7363. |
| [26] |
张兰河, 王佳佳, 哈雪姣, 等. 北京地区菜田土壤抗生素抗性基因的分布特征[J]. 环境科学, 2016, 37(11): 4395-4401. Zhang L H, Wang J J, Ha X J, et al. Distribution characteristics of antibiotic resistance genes in vegetable soils in Beijing[J]. Environmental Science, 2016, 37(11): 4395-4401. |
| [27] | Chen Q L, An X L, Li H, et al. Long-term field application of sewage sludge increases the abundance of antibiotic resistance genes in soil[J]. Environment International, 2016, 92-93: 1-10. DOI:10.1016/j.envint.2016.03.026 |
| [28] |
刘佳, 赵小学, 李红萍, 等. 河南某工厂附近农田土壤中抗生素抗性基因与可移动遗传元件的分布[J]. 郑州大学学报(医学版), 2016, 51(3): 301-305. Liu J, Zhao X X, Li H P, et al. Prevalence of antibiotic resistance genes and mobile genetic elements in farmland soil near a factory of a certain city in Henan province[J]. Journal of Zhengzhou University (Medical Sciences), 2016, 51(3): 301-305. |
| [29] |
冀秀玲, 刘芳, 沈群辉, 等. 养殖场废水中磺胺类和四环素抗生素及其抗性基因的定量检测[J]. 生态环境学报, 2011, 20(5): 927-933. Ji X L, Liu F, Shen Q H, et al. Quantitative determination of sulfonamides and tetracycline antibiotics and their resistance genes in farm wastewater[J]. Ecology and Environmental Sciences, 2011, 20(5): 927-933. DOI:10.3969/j.issn.1674-5906.2011.05.025 |
| [30] | Gao P, Munir M, Xagoraraki I, et al. Correlation of tetracycline and sulfonamide antibiotics with corresponding resistance genes and resistant bacteria in a conventional municipal wastewater treatment plant[J]. Science of the Total Environment, 2012, 421-422: 173-183. DOI:10.1016/j.scitotenv.2012.01.061 |
| [31] | Ma L P, Li A D, Yin X L, et al. The prevalence of integrons as the carrier of antibiotic resistance genes in natural and man-made environments[J]. Environmental Science & Technology, 2017, 51(10): 5721-5728. |
| [32] | Zhu Y G, Zhao Y, Li B, et al. Continental-scale pollution of estuaries with antibiotic resistance genes[J]. Nature Microbiology, 2017, 2(4): 16270. DOI:10.1038/nmicrobiol.2016.270 |
| [33] |
沈群辉.养殖场及周边农田土壤抗生素抗性基因和重金属污染初步研究[D].上海: 东华大学, 2013. Shen Q H. Preliminary studies on the pollution levels of antibiotic resistance genes and heavy metals in feedlots and agricultural soils adjacent to feedlots in Shanghai, China[D]. Shanghai: Donghua University, 2013. |
| [34] |
张海丰, 史明明, 孙艳梅, 等. 磺胺甲唑污染土壤中微生物群落结构与抗生素抗性基因的分布特征[J]. 环境科学, 2019, 40(10): 4678-4684. Zhang H F, Shi M M, Sun Y M, et al. Microbial community structures and the distribution of antibiotic resistance genes in soil contaminated by sulfamethoxazole[J]. Environmental Science, 2019, 40(10): 4678-4684. |
| [35] | Li C, Chen J Y, Wang J H, et al. Occurrence of antibiotics in soils and manures from greenhouse vegetable production bases of Beijing, China and an associated risk assessment[J]. Science of the Total Environment, 2015, 521-522: 101-107. DOI:10.1016/j.scitotenv.2015.03.070 |
| [36] | Li Y X, Zhang X L, Li W, et al. The residues and environmental risks of multiple veterinary antibiotics in animal faeces[J]. Environmental Monitoring and Assessment, 2013, 185(3): 2211-2220. DOI:10.1007/s10661-012-2702-1 |
| [37] | Wu X L, Xiang L, Yan Q Y, et al. Distribution and risk assessment of quinolone antibiotics in the soils from organic vegetable farms of a subtropical city, Southern China[J]. Science of the Total Environment, 2014, 487: 399-406. DOI:10.1016/j.scitotenv.2014.04.015 |
| [38] | Smith M S, Yang R K, Knapp C W, et al. Quantification of tetracycline resistance genes in feedlot lagoons by real-time PCR[J]. Applied and Environmental Microbiology, 2004, 70(12): 7372-7377. DOI:10.1128/AEM.70.12.7372-7377.2004 |
| [39] | Tang X J, Lou C L, Wang S X, et al. Effects of long-term manure applications on the occurrence of antibiotics and antibiotic resistance genes (ARGs) in paddy soils:evidence from four field experiments in south of China[J]. Soil Biology and Biochemistry, 2015, 90: 179-187. DOI:10.1016/j.soilbio.2015.07.027 |
| [40] | McKinney C W, Loftin K A, Meyer M T, et al. tet and sul antibiotic resistance genes in livestock lagoons of various operation type, configuration, and antibiotic occurrence[J]. Environmental Science & Technology, 2010, 44(16): 6102-6109. |
 2020, Vol. 41
2020, Vol. 41


