抗生素的滥用诱导和加速了抗生素抗性基因(antibiotic resistance genes, ARGs)的产生、传播以及抗生素耐药菌(antibiotic resistant bacteria, ARB)形成的风险[1, 2], 并且ARGs的存在是细菌表现耐药性的根本原因[3].ARGs作为一类新型的环境污染物[4], 可通过质粒、整合子以及转座子等可在细菌同种属菌株间和不同种属的菌株之间发生水平基因转移, 即使ARB死亡后, 携带ARGs的裸露DNA会在脱氧核苷酸酶的保护作用下长期存在, 故ARGs在环境中的持久性残留、传播和扩散比抗生素本身的危害还要大.目前, ARGs广泛存在于不同的生态环境中, 如水环境[5]、土壤[6]和大气[7], 其中作为水环境之一的污水厂被认为是ARB和ARGs巨大的储存地[8~10].以前针对ARGs的大多数研究主要集中在处理医院和生活污水的城市污水厂上[11~13].然而, 迄今为止, 很少有报道从医药化工废水中检测出ARB和ARGs.
ARGs的检测方法有可培方法和非培方法这两大类.可培方法是通过培养基进行微生物分离, 而后根据所分离微生物的耐药性, 结合各种分子生物学方法或者对ARB进行全基因组.但是由于自然环境中超过99%的微生物不能用传统培养基方法进行分离培养[14], 故可培方法会遗漏大量ARB和ARGs.非培方法有PCR技术、qPCR技术、基因芯片技术及宏基因组学技术.无论是普通PCR技术还是qPCR技术只能局限于已知序列的单一耐药基因检测, 并且只能对少数的ARGs进行同时检测, 最后还需要测序分析来进一步确认.基因芯片技术虽然可以同时检测出大量的ARGs, 但仍然要依赖于根据已知的耐药基因序列设计出相应的特异性强的探针, 并且其制作复杂, 费用昂贵.而随着生物技术的不断发展, 宏基因组测序也越来越多地应用于医学研究及临床诊断领域, 如感染类型诊断、抗性基因的鉴定和传染病的防控等[15].宏基因组技术通过高通量测序, 打破了传统以培养为主的微生物研究方式, 直接提取遗传物质进行遗传操作, 不仅可以准确获得样品微生物物种组成及丰度等信息, 进入数据库预测微生物功能, 还能分析微生物代谢网络等多方面的信息, 并且可同时检测大量的ARGs, 为寻找新基因、研究微生物多样性提供了便利的工具[16].
本研究借助宏基因组技术调查医药化工废水厂中微生物群落组成, 并分析ARGs的多样性及两者之间的关系, 对掌握医药化工废水中ARGs的污染现状及控制其传播, 保护微生态环境具有重要指导意义.
1 材料与方法 1.1 样品采集本实验样品取自江苏某医药化工园区废水处理厂, 该废水厂主要功能是处理园区各医药化工企业排放的工业废水, 纳入废水厂管网的废水包括抗生素中间体废水及农药中间体废水等多种医药化工废水.该废水厂日处理量10万t, 收集的废水中含β-内酰胺类、氨基糖苷类、大环内酯类、喹诺酮类和四环素类等几大类抗生素生产过程中产生的高浓发酵废水、化学合成废水、其他制剂废水及混合废水(清洗水、冷却循环水和生活污水等).废水中含大量残留的抗生素原材料、副反应产物, 还含有机溶剂二甲基甲酰胺、丙酮、乙腈、四氢呋喃和二氯甲烷等.废水厂主要采用厌氧-缺氧-好氧(A2O)工艺.本实验样品为活性污泥, 具体取自完全混合式好氧生物处理池.取样时先在池内前中后三段各取约500 mL泥水混合样品, 以保证样品具有代表性, 现场混合均匀后, 用无菌50 mL离心管分装后冰袋低温冷藏, 当日即进行基因组DNA提取.
1.2 基因组DNA提取使用FastDNA® Spin Kit for Soil (MP Biomedicals, CA, USA)试剂盒根据产品操作手册提取样品基因组DNA.采用1%琼脂凝胶电泳检测DNA样品的质量和完整性.用NanoDropTM 2 000分光光度计(Thermo Fisher Scientific, MA, USA)和Qubit® 2.0荧光光谱仪(Thermo Fisher Scientific, MA, USA)检测DNA的产量和纯度.基因组DNA样品用干冰保藏并立即送往上海美吉生物医药科技有限公司进行测序.
1.3 ARGs生物信息学分析采用Illumina HiSeq Xten平台进行PE150双端测序.首先对原始测序数据进行质量控制, 去除碱基数大于40 bp但是其质量分数低于38的序列, 去掉歧义核苷酸超过10个的序列, 去掉与adapter序列有15 bp重叠的序列.采用SOAPdenovo软件组装clean reads[17].当选择最高的scaffold N50 value来进行进一步分析时, 选择不同的K-mer值(49、55和59)用于使用于不同的-d1、-M3、-u和-F参数.将得到的scaffold片段在N段打断, 进而得到scaftig片段.仅仅大于500 bp的scaftig片段被保留以作进一步分析.采用默认参数的MetaGeneMark软件从组装好的scaftig片段来预测开放阅读框(ORFs)[18], 采用CD-HIT来获得非冗余基因.采用SOAPaligner软件将保留的clean read与基因库进行比对, 以计算匹配的数目[8].通过去除含有少于3个mapped reads映射片段的基因类别来获得unigene.用DIAMOND软件将unigene与NCBI-NR数据库进行比对, 设置blastp参数e-value≤1e-5, 并且采用MEGAN软件LCA算法进行注释[19].采用DIAMOND软件(blastp, evalue ≤ 1e-5)将unigene对应的蛋白序列在KEGG数据库、eggNOG数据库和CAZy数据库中进行比对.利用BLAST软件(blastp, evalue≤1e-5)将unigene在CARD数据库进行比对, 进行ARGs注释.采用Cytoscape软件进行网络分析, 以研究ARGs与微生物分类的关系.
2 结果与讨论 2.1 医药化工污水厂中微生物群落结构在该好氧活性污泥样品中, 经过质量控制后, 共得到reads数目109 523 076条, 并由此组装得到55 255个scaftig, 并预测了105 892个unigene.利用DIAMOND软件将unigene在NCBI-NR数据库中进行比对, 后续用MEGAN软件进行注释, 统计样本在门水平(表 1)及属水平(图 1)上的前10个丰度最大的群落组成.
|
|
表 1 废水厂中10个最丰富门的相对丰度 Table 1 Relative abundance of the ten most abundant phyla in the studied wastewater treatment plant |
|
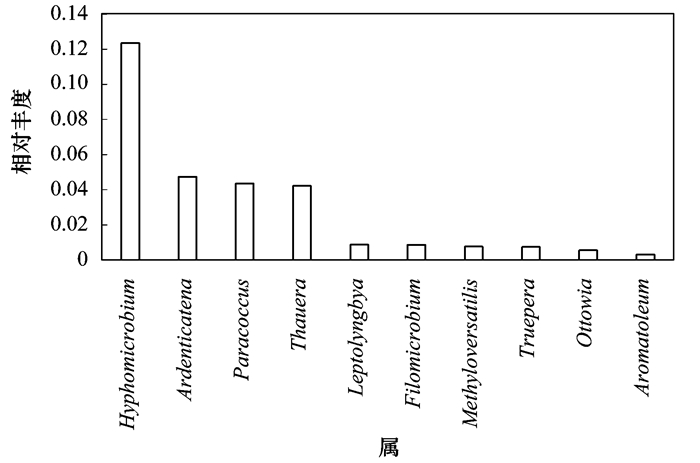
图 1 废水厂中10个最丰富属的相对丰度 Fig. 1 Relative abundance of the ten top identified genera in the studied wastewater treatment plant |
根据分类结果, 细菌域(Bacteria)是该医药化工园区废水厂中主要的微生物类群.并由表 1可知, 该废水厂丰度在前10的优势菌门是Proteobacteria、Chloroflexi、Bacteroidetes、Acidobacteria、Actinobacteria、Cyanobacteria、Deinococcus-Thermus、Firmicutes、Planctomycetes和Candidatus Saacharibacteria.其中最优势的菌门是Proteobacteria, 这与其他关于污水厂报道相符合[8, 20, 21], 丰度接近50%;其次是Chloroflexi, 丰度约占10%.Chloroflexi菌株可以在极端环境中生存, 比如热泉[22]和高盐环境[23], 并且也经常从污水厂环境[24]中分离得到, Chloroflexi类群中的丝状菌是污水处理厂活性污泥中的常见组成, 其大量出现会产生污泥膨胀现象[25].细菌菌群丰度居第三的是Bacteroidetes, 约占5%;其他7个优势菌群的丰度在0.5%~3%之间.
在该混合污泥样品中, 共检测到265个属, 丰度在前10的优势属为Hyphomicrobium、Ardenticatena、Paracoccus、Thauera、Leptolyngbya、Filomicrobium、Methyloversatilis、Truepera、Ottowia和Aromatoleum, 其中Hyphomicrobium是最主要的类群, 属于革兰氏阴性菌, 占据约12%;Ardenticatena、Paracoccus和Thauera这3个优势属的丰度相似, 均在4.5%左右, 其中Ardenticatena丰度稍高, 为4.7%;其他6个优势属的丰度在0.2%~1.0%之间(图 1).在属水平上, Hyphomicrobium是最优势属, 而在种水平上, Hyphomicrobium zavarzinii是最优势种.根据报道, Hyphomicrobium属类菌株也经常出现在污水环境中[26~28], 属于限制性的兼性甲基氧化菌, 能够利用甲醇、甲基化胺或甲酸盐等一碳化合物作为唯一碳源生长.而Hyphomicrobium zavarzinii能通过甲醇脱氢酶的过量表达, 在污水甲基营养型代谢中起着关键作用[29].
相比之下, 城市污水处理厂(wastewater treatment plants, WWTPs)中处理的废水大部分为生活污水, 可生化性好, 废水中的抗生素一般来源于医院排放的医疗废水, 含量较少, 尤其经大量其他废水稀释之后浓度一般为痕量.从WWTPs中分析到的主要细菌种类属于粪便污染的常见指标:大肠杆菌、总大肠菌群和肠球菌.监测到的主要耐药菌包括耐甲氧的Staphylococcus aureus、耐万古霉素的Enterococcus spp.及革兰氏阴性菌(如Enterobacteria、Pseudomonads和Acinetobacter), 这些菌对氟喹诺酮类、碳青霉烯类和β-内酰胺类等抗生素具有抗性.医药化工废水处理厂的抗生素浓度高于WWTPs的10~100倍, 因此其抗性菌种的种类和耐药性也一般强于WWTPs[30].
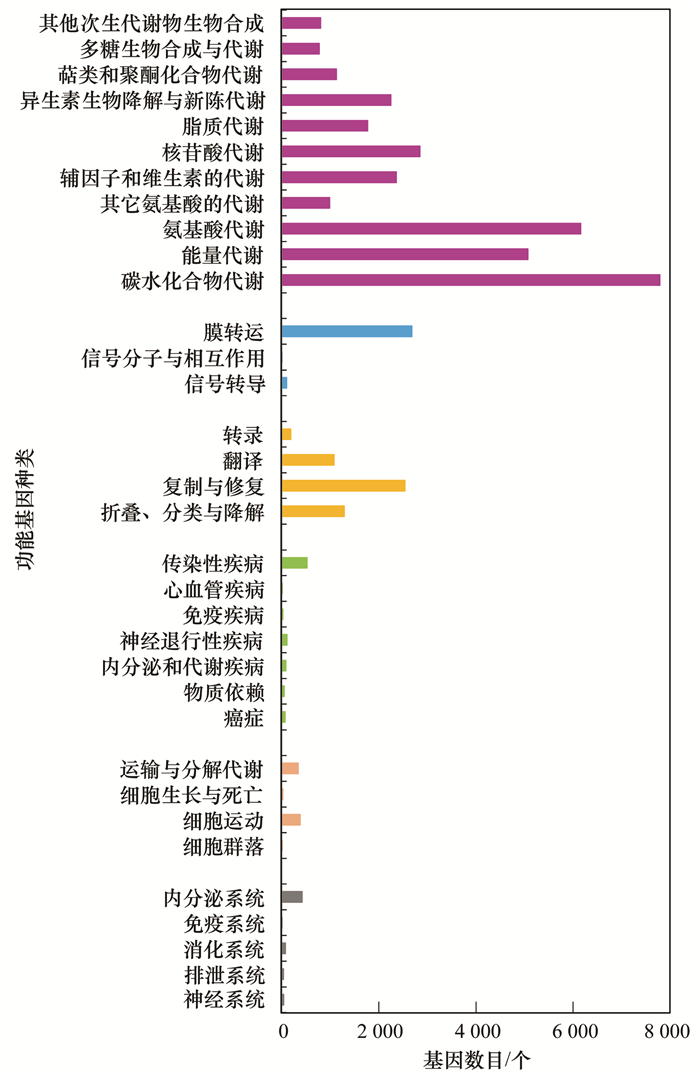
2.2 医药化工废水厂中微生物群落功能注释用DIAMOND软件将unigene在KEGG数据库进行比对, 共注释了54564个unigene, 结果如图 2所示.从中可知, 该微生物菌群的功能通路相关的基因数量依次为:代谢>遗传信息处理>环境信息处理>人类疾病>细胞过程>生物系统, 其中最主要的功能通路是代谢相关, 其次是遗传信息处理, 第三是环境信息处理.属于代谢类别的功能分类中丰度依次为:碳代谢>氨基酸代谢>能量代谢>核苷酸代谢>辅因子和维生素的代谢>异生素生物降解与新陈代谢>脂质代谢>萜类和聚酮化合物代谢>其他氨基酸代谢>其他次生代谢物生物合成>多糖生物合成与代谢, 其中碳代谢、氨基酸代谢和能量代谢的基因数量尤为丰富, 分别为7 788、6 155和5 067个.属于遗传信息处理类别的功能分类丰度依次为:复制与修复>折叠、分类与降解>翻译>转录, 其中复制与修复是最主要部分, 基因数量为2 535个.该微生物菌群功能通路中还有一部分的环境信息处理类别的功能, 其分类类别丰度依次为:膜转运>信号转导>信号分子与相互作用, 其中最主要的是膜转运, 基因数据为2 678个, 其他两个类别的基因合计100个, 数目甚少.属于人类疾病类别丰度依次为:传染性疾病>神经退行性疾病>内分泌和代谢疾病>癌症>物质依赖>免疫疾病>心血管疾病, 其中细菌传染性疾病相关的基因数目尤其突出, 为517个, 是其他人类疾病基因总数的约1.6倍.而在传染病疾病中, 细菌感染性疾病相关的基因占据了大部分, 约80%.
|
图 2 基于KEGG数据库注释的废水厂微生物群落功能基因分布 Fig. 2 Distribution of genes according to functional classes annotated with KEGG database for the studied in wastewater treatment plant |
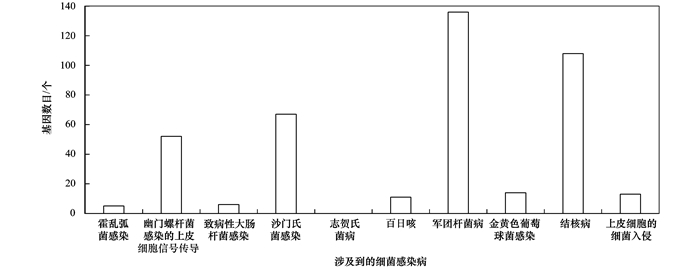
在该废水厂中细菌传染疾病涉及到9类(图 3), 其丰度依次为军团杆菌病>结核病>沙门氏菌感染>幽门螺杆菌感染的上皮细胞信号传导>金黄色葡萄球菌感染>上皮细胞的细菌入侵>百日咳>致病性大肠杆菌感染>霍乱弧菌感染, 其中最主要的传染病为军团杆菌病, 其基因数目为136个;其次是结核病, 基因数目为108个;沙门氏菌感染和幽门螺杆菌感染的上皮细胞信号传导, 基因数目均在60个左右;其他疾病的基因数目在15个以下.
|
图 3 废水厂中细菌感染疾病相关的基因数量 Fig. 3 Numbers of genes related to bacterial infectious diseases in the studied wastewater treatment plant |
根据功能注释结果, 该废水厂菌群中最主要的功能通路是代谢相关, 并且还存在许多与人类疾病相关的基因, 其中主要是细菌感染性疾病.到目前为止, 全球新出现的传染病事件的发生率有所增加, 废水厂等特殊环境可以作为许多病原体的天然宿主对人类健康存在潜在威胁[31].
2.3 医药化工废水厂中ARGs及耐药机制用BLAST软件将unigene在抗性基因数据库CARD中进行比对, 结果如图 4所示.经过氨基酸大于50%标准控制后, 共注释到74类抗生素功能基因, 涉及包括β-内酰胺类、头孢菌素类、氨基糖苷类、大环内酯类、多肽类、喹诺酮类及磺胺类等多种抗生素抗性基因.
|
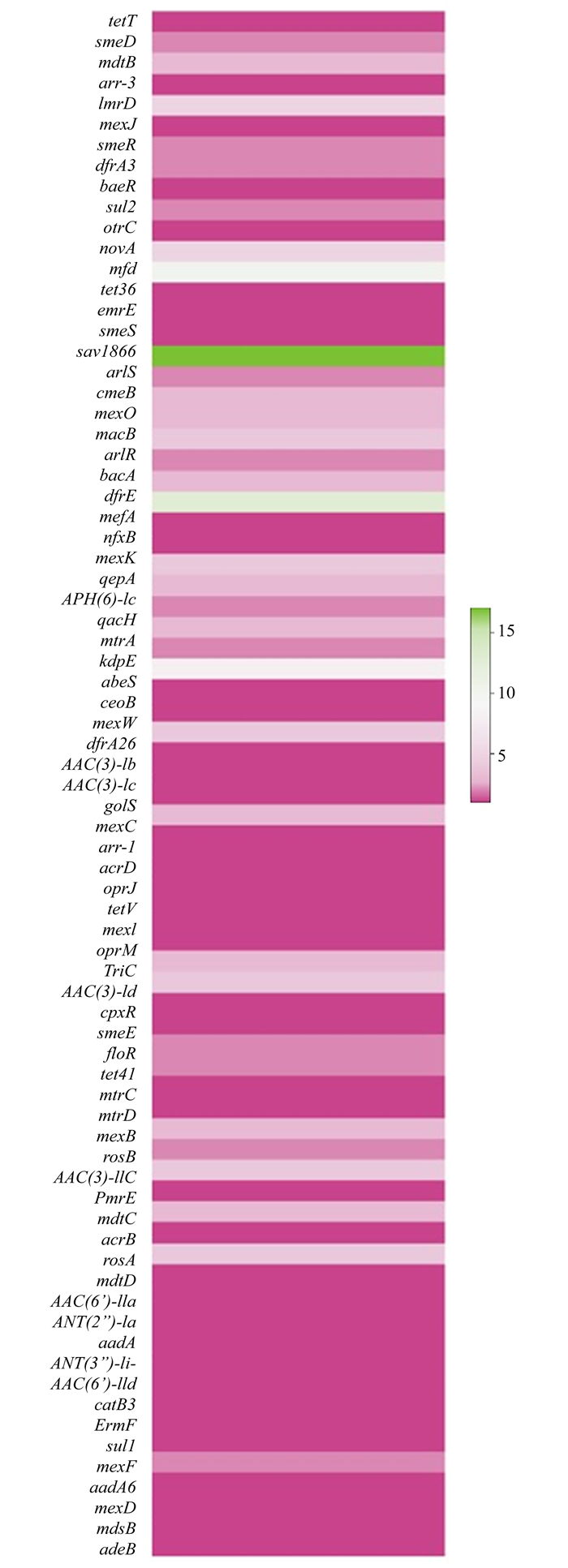
图 4 污水厂中不同ARGs的相对丰度 Fig. 4 Relative abundance of different ARGs in the studied wastewater treatment plant |
在这74类ARGs中sav1866、dfrE和mfd是最主要ARGs.sav1866基因属于多重耐药基因, 其编码的蛋白是ABC膜转运蛋白, 其耐药机制属于ABC外排泵.在细菌中, 许多ABC家族转运蛋白都与抗生素耐受性有关[32].sav1866被检测为丰度最大的ARGs, 推测这与该废水厂处理多种抗生素中间体废水及多种农药中间体废水密切相关.dfrE基因首次被发现在粪肠球菌中, 其编码的蛋白是一种耐甲氧苄啶二氢叶酸还原酶(dihydrofolate reductases, DHFRs), 该种DHFRs具有高的甲氧苄啶抗性[33], 其耐药机制是抗生素靶位点改变.mfd基因的产物Mfd是转录-修复偶联因子蛋白, 属于DNA移位酶, 也是DNA修复的必须因子, 其耐药机制是抗生素靶位点保护.Mfd主要功能是促进DNA修复[34], 但在某些情况下, 如在复制-转录冲突区域和在稳定阶段的突变中, Mfd反而增加了突变, 如Mfd促使空肠弯曲杆菌产生了对喹诺酮药物的抗性.在新的研究结果中, Mfd被认为是一种促进突变的进化因子, 加速了抗生素耐药性的发展, 是不同类群的细菌快速获得各种抗生素耐药性所必须的[35].由此推测, mfd基因的大量存在及积累可能会促进废水厂中微生物产生更多抗生素抗性.
统计这些ARGs的耐药机制, 以调查该废水厂的耐药机制情况, 如图 5中(a)所示.在该医药化工废水好氧活性污泥处理系统中, 微生物存在耐药的机制有5种, 分别是抗生素失活(8%)、抗生素靶位点改变(4%)、抗生素靶位点保护(7%)、抗生素靶位点替换(11%)和抗生素特异的外排泵(70%), 其中抗生素特异的外排泵是主要的耐药机制, 占比50%以上.在所研究的菌群中的外排泵组成如图 5(b)所以, 主要由ABC外排泵(18%)、MFS外排泵(10%)、RND外排泵(34%)和SMR外排泵(3%)组成, 其中RND外排泵是该微生物菌群抗生素外排泵耐药机制的最主要类型, 其次是ABC外排泵.据报道, 细菌细胞膜上的主动外排泵可将抗菌药物排至细菌外, 从而阻碍抗菌药物与细菌内的靶点结合, 引起耐药.并且, 外排泵在细菌中广泛存在, 在介导细菌多重耐药中起着重要作用[36].本研究表明, 氯霉素、四环素、磺胺类、氟喹诺酮类和大环内酯类抗生素均能通过细菌的主动外排机制而被排出细胞外.而该研究的医药化工污水系统所具有的外排泵中, RND外排泵是主要机制.RND外排泵的外排底物比较广泛, 包括多种抗菌药物、消毒剂和染料等[37], 由此可推测该菌群对多种抗生素具有抗性.结合图 4, 属于RND外排泵的ARGs有smeD、mdtB、mexJ、smeR、cmeB和mexQ等, 涉及氨基香豆素抗生素、四环素抗生素、大环内酯类抗菌素、氨基糖苷抗生素、三氯沙抗生素和氟喹诺酮类抗生素、头孢菌素等ARGs;属于ABC外排泵的ARGs有lmrD、otrC、novA、macB和sav1866, 涉及林可酰胺类抗生素、四环素类抗生素和大环内酯类抗生素的耐药.
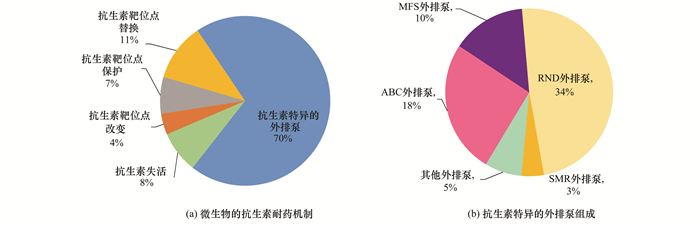
|
(a)微生物的抗生素耐药机制; (b)抗生素特异的外排泵组成 图 5 污水厂中抗生素耐药机制类型占比 Fig. 5 Percentage of the antibiotic resistance mechanism types in the studied wastewater treatment plant |
对废水厂中ARGs与微生物类群的共存模式进行网络分析(图 6), 结果显示有44类ARGs与污水厂中属级的微生物分类群高度相关, 并且主要类群的属都是多重耐药菌株, 同时携带多种ARGs, 涉及到4大类ARGs耐药机制.Hyphomicrobium属和Thauera属均可能携带有近20个ARGs, Paracoccus属和Methyloversatilis属可能携带有近10个ARGs.如Hyphomicrobium属中可能携带的抗性基因有dfrA26、mdsB、qepA、tetT、mexQ、mexF、mexL、rosB、sul2、dfrE、mfd和bacA等;Thauera属则可能携带有mdtD、bacA、mexJ、tet36、oprM、acrB和novA等.
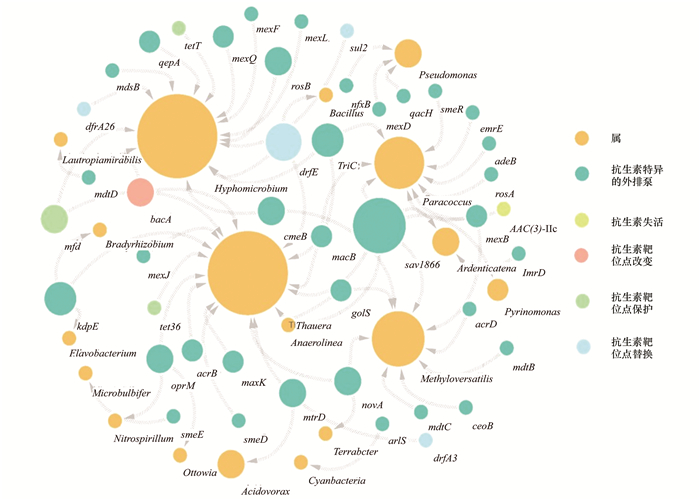
|
图 6 污水厂中ARGs与微生物类群的相关性网络分析 Fig. 6 Network analysis of the correlation between ARGs and microbial taxa in the studied wastewater treatment plant |
(1) 该医药化工废水处理厂的菌群组成结构中, 细菌域是主要微生物类类群, 并且最优势的菌门是Proteobacteria, 其次是Chloroflexi.最优势属是Hyphomicrobium, 最优势种为Hyphomicrobium zavarzinii.
(2) 该医药化工废水厂中微生物群落最主要的功能通路是代谢相关, 并还存在许多与人类疾病相关的基因, 其中主要是细菌感染性疾病.
(3) 该医药化工废水厂中ARGs共检测到74类ARGs, 涉及包括β-内酰胺类、头孢菌素类、氨基糖苷类、大环内酯类、多肽类、喹诺酮类、磺胺类等多种抗生素抗性基因, 其中sav1866、dfrE和mfd是最主要ARGs, 而最主要的Hyphomicrobium属也同时携带这3种主要的ARGs.该废水厂菌群的耐药机制主要是抗生素特异的外排泵, 其中RND外排泵是主要机制.
(4) 该废水厂活性污泥中微生物属级分类与ARGs高度相关, 并且主要类群的属都是多重耐药菌株.
| [1] |
沈怡雯, 黄智婷, 谢冰. 抗生素及其抗性基因在环境中的污染、降解和去除研究进展[J]. 应用与环境生物学报, 2015, 21(2): 181-187. Shen Y W, Huang Z T, Xie B. Advances in research of pollution, degradation and removal of antibiotics and antibiotic resistance genes in the environment[J]. Chinese Journal of Applied and Environmental Biology, 2015, 21(2): 181-187. |
| [2] |
张衍, 陈吕军, 谢辉, 等. 两座污水处理系统中细胞态和游离态抗生素抗性基因的丰度特征[J]. 环境科学, 2017, 38(9): 3823-3830. Zhang Y, Chen L J, Xie H, et al. Abundance of cell-associated and cell-free antibiotic resistance genes in two wastewater treatment systems[J]. Environmental Science, 2017, 38(9): 3823-3830. |
| [3] |
张昊, 王盼亮, 杨清香, 等. 畜禽粪便中多重耐药细菌及耐药基因的分布特征[J]. 环境科学, 2018, 39(1): 460-466. Zhang H, Wang P L, Yang Q X, et al. Distribution of multidrug-resistant bacteria and antibiotic-resistant genes in livestock manures[J]. Environmental Science, 2018, 39(1): 460-466. |
| [4] |
孙薇.畜禽粪便厌氧发酵过程中抗生素抗性基因变化机理研究[D].杨凌: 西北农林科技大学, 2017. Sun W. Varition mechanism of antibiotic resistance genes during anaerobic digestion with liverstock manure[D]. Yangling: Northwest A&F University, 2017. |
| [5] |
庄榆佳, 赵忆, 苏建强, 等. 抗生素抗性基因在养殖废水中的分布与去除[J]. 环境化学, 2017, 36(11): 2311-2318. Zhuang Y J, Zhao Y, Su J Q, et al. Distribution and removal of antibiotic resistance genes in swine wastewater[J]. Environmental Chemistry, 2017, 36(11): 2311-2318. DOI:10.7524/j.issn.0254-6108.2017041801 |
| [6] | Xiao K Q, Li B, Ma L P, et al. Metagenomic profiles of antibiotic resistance genes in paddy soils from South China[J]. FEMS Microbiology Ecology, 2016, 92(3): fiw023. DOI:10.1093/femsec/fiw023 |
| [7] |
高新磊, 邵明非, 贺小萌, 等. 污水处理厂空气介质抗生素抗性基因的分布[J]. 生态毒理学报, 2015, 10(5): 89-94. Gao X L, Shao M F, He X M, et al. Distribution of airborne antibiotic resistance gene in wastewater treatment plant[J]. Asian Journal of Ecotoxicology, 2015, 10(5): 89-94. |
| [8] | Guo J H, Li J, Chen H, et al. Metagenomic analysis reveals wastewater treatment plants as hotspots of antibiotic resistance genes and mobile genetic elements[J]. Water Research, 2017, 123: 468-478. DOI:10.1016/j.watres.2017.07.002 |
| [9] | Jałowiecki Ł, Żur J, Chojniak J, et al. Properties of antibiotic-resistant bacteria isolated from onsite wastewater treatment plant in relation to biofilm formation[J]. Current Microbiology, 2018, 75(5): 639-649. DOI:10.1007/s00284-017-1428-2 |
| [10] |
李奥林, 陈吕军, 张衍, 等. 抗生素抗性基因在两级废水处理系统中的分布和去除[J]. 环境科学, 2018, 39(10): 4593-4600. Li A L, Chen L J, Zhang Y, et al. Distribution and removal of antibiotic resistance genes in two sequential wastewater treatment plants[J]. Environmental Science, 2018, 39(10): 4593-4600. |
| [11] | Szekeres E, Baricz A, Chiriac C M, et al. Abundance of antibiotics, antibiotic resistance genes and bacterial community composition in wastewater effluents from different Romanian hospitals[J]. Environmental Pollution, 2017, 225: 304-315. DOI:10.1016/j.envpol.2017.01.054 |
| [12] | Yu H, Qu F, Shan B, et al. Detection of the mcr-1 colistin resistance gene in carbapenem-resistant Enterobacteriaceae from different hospitals in China[J]. Antimicrobial Agents and Chemotherapy, 2016, 60(8): 5033-5035. DOI:10.1128/AAC.00440-16 |
| [13] | Galler H, Feierl G, Petternel C, et al. Multiresistant bacteria isolated from activated sludge in austria[J]. International Journal of Environmental Research and Public Health, 2018, 15(3): 479. DOI:10.3390/ijerph15030479 |
| [14] |
邢磊, 赵圣国, 郑楠, 等. 未培养微生物分离培养技术研究进展[J]. 微生物学通报, 2017, 44(12): 3053-3066. Xing L, Zhao S G, Zheng N, et al. Advance in isolation and culture techniques of uncultured microbes: a review[J]. Microbiology China, 2017, 44(12): 3053-3066. |
| [15] |
李林海, 陈丽丹, 肖斌, 等. 宏基因组测序在感染性疾病病原体检测中的应用[J]. 传染病信息, 2018, 31(1): 15-18. Li L H, Chen L D, Xiao B, et al. Application of metagenomic sequencing in detecting the pathogens of infectious diseases[J]. Infectious Disease Information, 2018, 31(1): 15-18. DOI:10.3969/j.issn.1007-8134.2018.01.004 |
| [16] |
陈泓宇, 闫海亚, 赵胤丞, 等. 宏基因组学及其在鱼类肠道微生物中的研究进展[J]. 水产科学, 2018, 37(5): 699-706. Chen H Y, Yan H Y, Zhao Y C, et al. Metagenomics and its advances in research of fish gut microbiome[J]. Fisheries Science, 2018, 37(5): 699-706. |
| [17] | Luo R B, Liu B H, Xie Y L, et al. SOAPdenovo2: an empirically improved memory-efficient short-read de novo assembler[J]. GigaScience, 2012, 1(1): 18. DOI:10.1186/2047-217X-1-18 |
| [18] | Zhu W H, Lomsadze A, Borodovsky M. Ab initio gene identification in metagenomic sequences[J]. Nucleic Acids Research, 2010, 38(12): e132. DOI:10.1093/nar/gkq275 |
| [19] | Huson D H, Beier S, Flade I, et al. MEGAN community edition-interactive exploration and analysis of large-scale microbiome sequencing data[J]. PLoS Computational Biology, 2016, 12(6): e1004957. DOI:10.1371/journal.pcbi.1004957 |
| [20] | Yang Y, Li B, Zou S C, et al. Fate of antibiotic resistance genes in sewage treatment plant revealed by metagenomic approach[J]. Water Research, 2014, 62: 97-106. DOI:10.1016/j.watres.2014.05.019 |
| [21] | Behnami A, Farajzadeh D, Isazadeh S, et al. Diversity of bacteria in a full-scale petrochemical wastewater treatment plant experiencing stable hydrocarbon removal[J]. Journal of Water Process Engineering, 2018, 23: 285-291. DOI:10.1016/j.jwpe.2018.04.015 |
| [22] | Lavrentyeva E V, Radnagurueva A A, Barkhutova D D, et al. Bacterial diversity and functional activity of microbial communities in hot springs of the baikal rift zone[J]. Microbiology, 2018, 87(2): 272-281. DOI:10.1134/S0026261718020078 |
| [23] | Nunoura T, Nishizawa M, Hirai M, et al. Microbial diversity in sediments from the bottom of the Challenger Deep, the Mariana Trench[J]. Microbes and Environments, 2018, 33(2): 186-194. DOI:10.1264/jsme2.ME17194 |
| [24] | Buelow E, Bayjanov J R, Majoor E, et al. Limited influence of hospital wastewater on the microbiome and resistome of wastewater in a community sewerage system[J]. FEMS Microbiology Ecology, 2018, 94(7): fiy087. DOI:10.1093/femsec/fiy087 |
| [25] | Nierychlo M, Miłobędzka A, Petriglieri F, et al. The morphology and metabolic potential of the Chloroflexi in full-scale activated sludge wastewater treatment plants[J]. FEMS Microbiology Ecology, 2018, 95(2): fiy228. |
| [26] | Wang X H, Zheng Q, Yuan Y, et al. Bacterial community and molecular ecological network in response to Cr2O3 nanoparticles in activated sludge system[J]. Chemosphere, 2017, 188: 10-17. DOI:10.1016/j.chemosphere.2017.08.072 |
| [27] | Gao F, Li Z W, Chang Q B, et al. Effect of florfenicol on performance and microbial community of a sequencing batch biofilm reactor treating mariculture wastewater[J]. Environmental Technology, 2017, 39(3): 363-372. |
| [28] | Li E C, Lu S G. Denitrification processes and microbial communities in a sequencing batch reactor treating nanofiltration (NF) concentrate from coking wastewater[J]. Water Science and Technology, 2017, 76(11-12): 3289-3298. |
| [29] | Salerno C, Benndorf D, Kluge S, et al. Metaproteomics applied to activated sludge for industrial wastewater treatment revealed a dominant methylotrophic metabolism of Hyphomicrobium zavarzinii[J]. Microbial Ecology, 2016, 72(1): 9-13. |
| [30] | Karkman A, Do T T, Walsh F, et al. Antibiotic-resistance genes in waste water[J]. Trends in Microbiology, 2018, 26(3): 220-228. DOI:10.1016/j.tim.2017.09.005 |
| [31] | Sandifer P A, Sutton-Grier A E, Ward B P. Exploring connections among nature, biodiversity, ecosystem services, and human health and well-being: opportunities to enhance health and biodiversity conservation[J]. Ecosystem Services, 2015, 12: 1-15. DOI:10.1016/j.ecoser.2014.12.007 |
| [32] | 任远, 司维, 任恒, 等. ABC家族转运蛋白及应对抗生素耐药性的概述[J]. 河南医学研究, 2016, 25(1): 82-84. DOI:10.3969/j.issn.1004-437X.2016.01.036 |
| [33] | Coque T M, Singh K V, Weinstock G M, et al. Characterization of dihydrofolate reductase genes from trimethoprim-susceptible and trimethoprim-resistant strains of Enterococcus faecalis[J]. Antimicrobial Agents and Chemotherapy, 1999, 43(1): 141-147. DOI:10.1128/AAC.43.1.141 |
| [34] | Million-Weaver S, Samadpour A N, Moreno-Habel D A, et al. An underlying mechanism for the increased mutagenesis of lagging-strand genes in Bacillus subtilis[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(10): E1096-E1105. DOI:10.1073/pnas.1416651112 |
| [35] | Ragheb M N, Thomason M K, Hsu C, et al. Inhibiting the evolution of antibiotic resistance[J]. Molecular Cell, 2019, 73(1): 157-165. DOI:10.1016/j.molcel.2018.10.015 |
| [36] | 欧阳净, 陈耀凯. 细菌多重耐药外排泵的研究进展[J]. 临床内科杂志, 2018, 35(3): 149-151. DOI:10.3969/j.issn.1001-9057.2018.03.001 |
| [37] |
陈小燕, 潘珏. 细菌耐多药外排泵的研究进展[J]. 微生物与感染, 2016, 11(3): 183-187. Chen X Y, Pan J. Research progress on bacterial multidrug efflux pumps and biofilm formation[J]. Journal of Microbes and Infections, 2016, 11(3): 183-187. |
 2020, Vol. 41
2020, Vol. 41


