2. 中国科学院遗传与发育生物学研究所, 石家庄 100101;
3. 中国农业科学院农业资源与农业区划研究所, 北京 100081
2. Institute of Genetics and Developmental Biology, Chinese Academy of Sciences, Shijiazhuang 100101, China;
3. Institute of Agricultural Resource and Regional Planning, Chinese Academy of Agricultural Sciences, Beijing 100081, China
作物秸秆是农业生产中很普遍的有机物料, 秸秆还田是土壤养分管理中的常用措施, 能够提供植物生长所需的C、N以及其他营养元素;提高土壤的供肥保肥能力;还能改善土壤的物理性状.水稻秸秆及根是稻田的主要碳源, 据估计每年输入稻田的有机质约有1 700~3 470 kg·hm-2[1, 2], 其中约有65%的有机碳来自水稻残体.然而稻秸还田显著促进稻田甲烷的产生和排放[3].稻田土壤系统是研究厌氧系统微生物学的模式体系.稻田土壤的微生物菌群降解水稻残体.稻秸主要由纤维素、半纤维素和木质素组成.稻秸厌氧降解是稻田土壤有机碳的主要来源[4].有研究证明添加稻秸的水稻土泥浆是研究植物残体厌氧降解代谢过程的模式体系[5~7].
土壤微生物在维持土壤生态功能及元素循环中起着非常重要的作用.土壤微生物对环境变化很敏感, 其群落结构的改变在一定程度上能够反映土壤生态功能的变化[8].有研究发现稻秸还田会显著改变土壤微生物学特性, 如微生物数量、群落结构、活性以及微生物功能多样性等[9~11].土壤微生物在保持生态系统稳定性方面也具有重要作用, 不同微生物具有不同的生态位, 如细菌多样性降低会导致其生态功能的缺失, 对生态系统产生不利的影响. Weber等[7]的研究发现, 在水稻土厌氧培养的早期和晚期, 土壤细菌群落结构不同.在稻秸降解过程中, 稻秸的不同部位由不同的细菌菌群所降解.稻秸还田后, 稻田土壤养分发生短期空间梯度变化, 参与稻秸降解的细菌菌群在稻秸整个降解过程中存在演替规律. Rui等[12]的研究发现Clostridium(clusters Ⅰ and Ⅲ)在稻秸早期降解过程中成为优势菌群, 它们以易降解有机质为底物. Bacteroidetes和Chlorobi在稻秸降解的后期阶段占主导地位, 以难降解的有机质为底物.有研究称长期稻秸还田对细菌群落产生影响是因为增加了土壤的C营养[13].有报道也称土壤中如果投入大量的营养元素, 则会抑制一些寡营养型细菌的生长, 进而影响细菌群落[14].另有报道称稻秸还田配合化肥施用对细菌群落结构和多样性没有显著影响, 但能够提高一些特定细菌的数量, 如氨化细菌、硝化细菌、反硝化细菌和纤维分解菌等[9].因此, 研究稻秸对土壤细菌菌群的影响, 在评价稻秸还田对土壤生态的影响方面具有重要意义.
产甲烷古菌是一类以甲烷为代谢终产物的微生物, 稻田甲烷是产甲烷古菌在严格厌氧条件下有机质降解的最终产物.产甲烷古菌是目前已知的地球上对氧极其敏感的生物类群, 在系统发育学上隶属于古菌域广古菌门(Euryarchaeota), 是已知的唯一能够代谢产甲烷的一种微生物, 只能利用CO2/H2、乙酸和甲基类化合物等简单物质产生甲烷并合成自身生长所需的物质[15].大量培养实验研究发现秸秆降解过程中产甲烷古菌群落结构表现出较高的动态变化, 不同产甲烷古菌对秸秆类型、培养温度及培养时间的响应是不同的[16~22].有研究认为在稻秸降解过程中产生的乙酸和氢气浓度是调控产甲烷古菌群落变化的关键因素[23]在稻秸添加处理中一些特定产甲烷古菌如Methanosarcinaceae和Methanobacteriales的相对丰度增加, 但抑制了RC-I及Methanomicrobiales的生长[18, 19]水稻土厌氧培养中添加稻秸使产甲烷古菌菌群的数量增加[21].稻秸降解过程中发现有大量Methanosarcina、Methanobacterium和RC-I附着在稻秸上[6]
稻秸添加对水稻土细菌和产甲烷古菌菌群的影响已有较多研究[10, 19, 21], 然而这些研究均基于水稻土的短期厌氧培养, 稻秸添加后水稻土经过相对较长时间的厌氧培养后细菌和产甲烷古菌的响应研究仍较缺乏.本研究基于细菌16S rDNA及产甲烷古菌功能基因mcrA, 运用T-RFLP及q-PCR微生物分子技术, 对两种典型水稻土在添加不同量稻秸并经相对较长时间厌氧培养后的细菌及产甲烷古菌的响应进行分析, 以期为评价稻秸还田对土壤微生物生态的影响及寻找环境友好型农业措施提供依据.
1 材料与方法 1.1 水稻土预处理及厌氧培养过程实验土壤分别采自江西鹰潭(JX)和广东雷州半岛(GD)水稻田0~20 cm的耕层土, JX和GD土壤类型分别为粉砂壤土和粉质黏壤土、pH分别为6.42和5.24、总C分别为10.27 g·kg-1和11.68 g·kg-1、总N分别为0.54 g·kg-1和0.38 g·kg-1.土样风干后储存在室温, 培养开始前, 将土样过2 mm筛且搅拌均匀, 土壤样品进行预培养两周以活化土壤微生物.稻秸施用前将其粉碎至粉末状.灭菌的100 mL模制瓶中装入30 g干土, 稻秸以0.5%、1%和2%干土重的量加入模制瓶并与土壤充分混匀.灭菌去氧的去离子水以水土比1.5:1的量加入模制瓶.盖上丁基胶塞, 用压盖钳压上铝盖密封, 充入5 min的高纯氮气后抽真空, 反复3次, 最后充入氮气后剧烈振荡培养瓶30 s, 之后放在30℃培养箱中黑暗静置培养9个月.每处理设置3个重复.所有处理分别命名为JX/GD-Ctrl、JX/GD-0.5%RS、JX/GD-1%RS和JX/GD-2%RS.培养的第0 d、第180 d和第270 d以破坏性取样的方式进行取样, 分析产甲烷古菌及细菌群落结构和数量的变化.
1.2 土壤微生物DNA的提取采用FastDNA® SPIN Kit for Soil (Q Bio gene)试剂盒方法, 称取-20℃保存的土样0.5 g, 按试剂盒实验操作说明进行土壤微生物总DNA提取.提取的DNA用1%(质量分数)琼脂糖电泳EB(Ethidium Bromide, 溴化乙锭)染色检查DNA提取效果, 90 V, 40 min电泳检测, 在Alpbalmager 2200成像系统拍照.
1.3 产甲烷古菌功能基因mcrA的PCR扩增产甲烷古菌功能基因mcrA的PCR扩增采用引物对ME1/ME2, T-RFLP分析的PCR前引物ME1的5′端有6-carboxyfluorescein标记. 50 μL PCR反应体系为:10×PCR反应缓冲液;15 mmol·L-1MgCl2;10 mmol·L-1 dNTP;各引物10 μmol·L-1;5U Taq DNA聚合酶和1 μL的DNA模板, 用灭菌高纯水补足到50 μL. PCR反应程序为:94℃预变性3 min;94℃变性45 s;49℃退火30 s;72℃延伸1 min;运行30个循环;最后72℃延伸10 min. PCR产物取3 μL用1%(质量分数)琼脂糖凝胶电泳检测并拍照, 样品在-20℃保存.
1.4 PCR扩增产物DNA凝胶回收及纯化采用蓝罡普通PCR产物凝胶回收试剂盒方法对PCR产物进行凝胶回收, 并用1%(质量分数)琼脂糖电泳EB(Ethidium Bromide, 溴化乙锭)染色检查纯化效果.
1.5 产甲烷古菌T-RFLP(末端限制性片段多态性)分析(1) PCR产物的酶切纯化后的PCR产物使用限制性内切酶Sau96I(Fermentas, Canada)对mcrA基因进行酶切.酶切反应条件:37℃条件下酶切3 h.酶切反应体系(20 μL):灭菌高纯水0~5 μL;10×缓冲液2 μL;0.1% BSA 2 μL;Sau96I 0.5 μL;纯化后的PCR产物10~15 μL.酶切完后用2% (质量分数)琼脂糖电泳EB(Ethidium Bromide, 溴化乙锭)染色检查酶切效果.
(2) 乙醇沉淀法纯化酶切产物在酶切产物中加入1/10体积的3 mol·L-1NaAC溶液和2倍体积的无水乙醇, 混匀离心后-20℃过夜.然后将冷冻过夜的酶切产物在14 000 r·min-1、4℃条件下离心15 min, 去上清;加入200 μL 70%乙醇, 然后14 000 r·min-1、4℃条件下离心5 min, 去上清, 通风橱中风干10 min或在室温放置20 min左右;最后加入10 μL灭菌高纯水溶解DNA, -20℃保存.
(3) T-RFLP上机酶切产物在AppliedBiosystems 3730测序仪上进行聚丙烯酰胺凝胶电泳自动测序仪进行检测, 末端带荧光标记的片段能被检测到, 而其它没有带荧光标记的片段则检测不到, T-RFLP图谱用Genemaper 4.0(ABI)软件进行分析.
1.6 产甲烷古菌mcrA基因的克隆文库构建及序列分析利用不加荧光标记的引物进行mcrA基因的PCR扩增;PCR扩增产物凝胶回收及纯化(同上);PCR产物通过pMD 19-T vector进行连接, 连接产物进行质粒(JM109感受态细胞)载体的转化.转化产物在SOC培养基中培养1 h(37℃).然后取一定量的细胞液涂在LB平板培养基上培养12 h(37℃), 形成单菌落.利用引物M13-47/RV-M对单菌落进行PCR检验阳性克隆, 将筛选好的阳性克隆送至测序公司进行测序, 采用MEGA(4.0)软件的N-J法(Neighbor-Joining)建立系统发育树. JX水稻土产甲烷古菌克隆序列在NCBI数据库的序列号为KX233727~KX233778;GD水稻土产甲烷古菌克隆序列在NCBI数据库的序列号为KX233779~KX233832.
1.7 实时荧光定量PCR(1) 质粒DNA的提取根据定量的目标基因在克隆文库中选取适合的单克隆, 使用UNIQ-10柱式质粒小量抽提试剂盒提取质粒DNA, 具体步骤参照试剂盒使用说明.使用Quant-iT PicoGreen DsDNA Reagent and Kits(Invitrogen)试剂盒, 用酶标仪测定质粒DNA浓度.具体操作参照试剂盒.
(2) 标准曲线的制备将已知浓度的质粒DNA进行10倍的系列梯度稀释, 质粒DNA标准在109~101拷贝数之间选择6个不同数量级的拷贝数作为定量PCR的标准.
(3) 反应体系及反应程序样品实时荧光定量PCR产甲烷古菌功能基因mcrA的定量引物为ME1f/ME3r. 25 μL反应体系为:12.5 μL的2 × Power SYBR@Green PCR Master Mix, 10 μmol·L-1引物, 10 μg·μL-1 BSA, DNA模板5 μL, 加灭菌高纯水补足到25 μL.反应程序为:94℃预变性3 min;94℃变性45 s;49℃退火30 s;72℃延伸1 min;83℃收集荧光信号10 s;运行40个循环;溶解曲线, 从55℃开始, 每个循环的温度以0.5℃递增, 每个循环进行10 s, 95℃结束.单个DNA样品和标准在进行定量PCR时做3个重复, 定量的结果用7500 System SDS Software(ABI)分析.
1.8 统计分析Excel 2007计算数据的平均值和标准误.末端标记限制性片段T-RFs由PeakScan 1.0软件分析计算.选择片段大小>50 bp且相对丰度>1%的片段进行Cluster分析.细菌序列相似度>97%的被认为是同一个OTU, R语言绘制细菌OTU热图及维恩图. CANOCO 4.5软件对T-RFLP数据进行主成分分析(PCA analysis).
2 结果与分析 2.1 两种水稻土在添加稻秸后产甲烷古菌群落结构的变化由图 1所示, JX水稻土中共检测到8个不同片段长度的T-RF(88、120、136、494、639、652、667和726 bp), 其中片段长度为136 bp的产甲烷古菌为优势菌, 其次是片段长度为639 bp和726 bp的产甲烷古菌.该水稻土在厌氧培养的第180 d和第270 d, 726 bp的产甲烷古菌相对丰度随着稻秸添加量的增加而有所增加, 而88 bp和136 bp的产甲烷古菌相对丰度随着稻秸添加量的增加略有降低.对于GD水稻土, 产甲烷古菌群落结构与JX水稻土相比有明显的不同, 仅检测到5个不同片段长度的T-RF(120、136、639、667和726 bp), 其中136 bp的产甲烷古菌仍为优势菌群, 以726 bp的产甲烷古菌为第二大优势菌群, 120 bp和639 bp的产甲烷古菌的相对丰度最低.在培养的第270 d, 片段长度为667 bp和726 bp的产甲烷古菌相对丰度在对照及各个稻秸处理中均明显增加, 而136 bp的产甲烷古菌相对丰度明显降低. 120 bp和639 bp的产甲烷古菌的相对丰度均无变化.
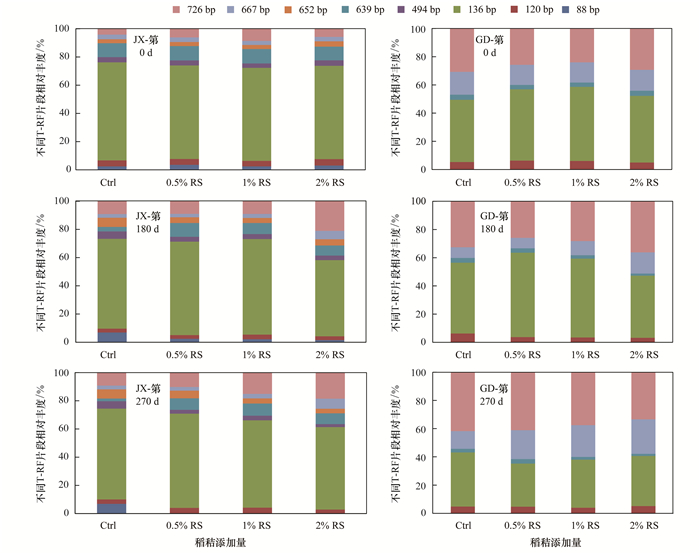
|
JX表示江西鹰潭水稻土, GD表示广东雷州半岛水稻土;Ctrl、0.5% RS、1% RS及2% RS分别表示对照、添加0.5%(质量分数)稻秸、添加1%稻秸和添加2%稻秸, 下同 图 1 两种水稻土在不同培养时间下稻秸添加量对产甲烷古菌群落结构的影响(基于mcrA基因的T-RFLP分析) Fig. 1 Community structures of methanogenic archaea based on T-RFLP analysis targeting mcrA genes in different added RS amounts and sampling time in the two soil types |
对两种水稻土不同处理mcrA基因的T-RFLP图谱进行了主成分分析(PCA), 结果表明两种水稻土的产甲烷古菌群落结构明显不同.不同稻秸添加量对JX水稻土产甲烷古菌群落结构有一定的影响, 而对GD水稻土的产甲烷古菌群落结构的影响不大(图 2).
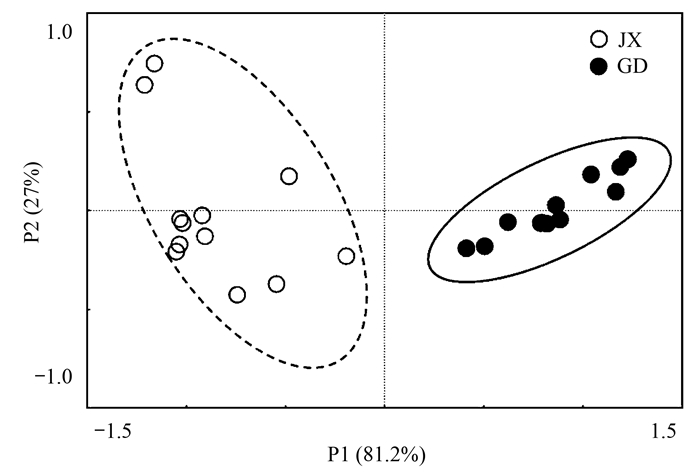
|
双轴上的数值表示每轴能够解释总变异的百分比 图 2 不同处理mcrA基因的T-RFLP图谱主成分分析(PCA) Fig. 2 Principal component analysis of T-RFLP profiles for mcrA genes retrieved from different treatments |
对2% RS处理的两种水稻土培养第270 d的产甲烷古菌mcrA基因进行了克隆测序, 并与NCBI数据库中的相似序列进行了比对, 系统发育分析结果见图 3.从中可知, GD和JX水稻土中均检测到3种不同科的产甲烷古菌, 为Methanosarcinaceae、Methanobacteriaceae及Methanocellaceae.不同科的产甲烷古菌在两种水稻土中所占的比例不同.对系统发育分析结果进行的分类统计表明(表 1), JX水稻土的优势菌为Methanobacteriaceae, 占总测序列的44.59%;Methanosarcinaceae为第二优势菌, 占总测序列的29.73%;Methanocellaceae占总测序列的25.68%.而GD水稻土中以Methanosarcinaceae为优势菌, 占总测序列的59.65%;Methanobacteriaceae为次之, 占总测序列的28.07%;Methanocellaceae为第三, 占总测序列的10.52%;检测到的未知古菌仅有一例.
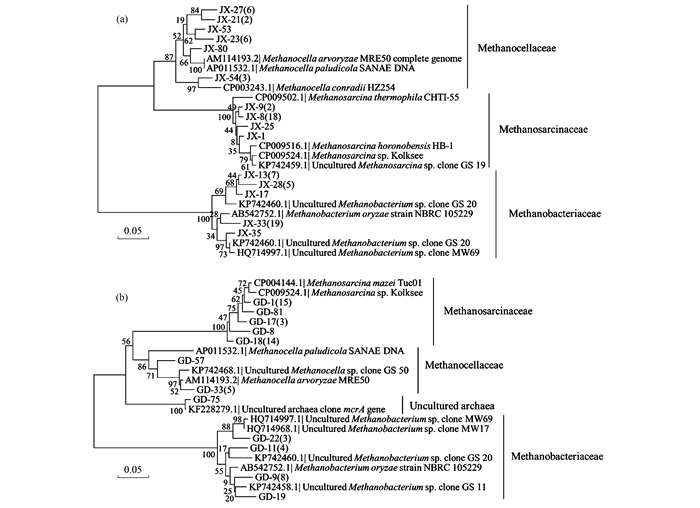
|
(a)和(b)分别是JX和GD水稻土基于2% RS处理的第270 d土壤样品构建;以“JX-27(6)”为例:JX表示江西鹰潭水稻土, 27表示该条序列在克隆文库中的序号, (6)表示在所有序列中与JX-27这条序列相似度>95%的一共有6条;克隆序列相似度>95%的所有序列被认为是同一操作分类单元(OTU) 图 3 N-J法构建的产甲烷古菌系统发育分析 Fig. 3 Phylogenetic analysis of methanogenic archaea constructed through the neighbor-joining method |
|
|
表 1 RS处理的两种水稻土中产甲烷古菌mcrA基因序列在系统发育归类中的数量及所占百分含量1) Table 1 Number and proportion of mcrA gene sequences of methanogenic archaea in phylogenetic classification with 2% treatment in the two soil types |
对序列进行In silico分析得到的片段与T-RFLP数据中片段结合比较, 以及根据前人已发表的数据信息, 得出T-RFs的系统分支归类.本研究中产甲烷古菌mcrA基因的T-RFs多数都能归类于特定的产甲烷古菌类群.一些T-RFs能够归类于多个不同的产甲烷古菌类群.如末端限制片段为120、136及652 bp的T-RFs可以归类为Methanosarcinales;667及726 bp的T-RFs归类于Methanobacteriaceae;88 bp的T-RF归类于Methanomicrobiales;639bp的T-RFs归类于Methanocellaceae.在JX水稻土中的494 bp的T-RFs未归类于特定的产甲烷古菌类群的系统分支.另外, 本研究中由测序得到的各产甲烷古菌类群在总测序列中所占的比例与T-RFLP分析得到的各T-RFs的相对丰度结果基本一致.
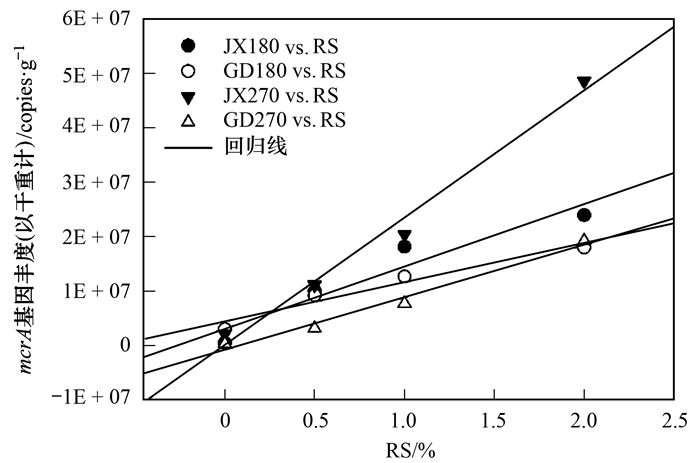
2.3 不同稻桔添加量对两种水稻土产甲烷古菌mcrA基因拷贝数的影响对于产甲烷古菌mcrA基因拷贝数, 本研究对培养第180 d和270 d的土样进行了测定(图 4).分析结果表明, 在培养第180 d, 两种水稻土的产甲烷古菌mcrA基因拷贝数随稻秸添加量的增加而增加.相同稻秸量添加条件下, JX水稻土产甲烷古菌mcrA基因拷贝数大于GD水稻土. JX-2% RS处理中产甲烷古菌mcrA基因拷贝数显著大于JX-Ctrl.在培养第270 d, 两种水稻土的产甲烷古菌mcrA基因拷贝数同样随稻秸添加量的增加而增加. 2% RS处理的两种水稻土产甲烷古菌mcrA基因拷贝数显著大于其他RS处理.相同稻秸量添加条件下, JX水稻土产甲烷古菌mcrA基因拷贝数明显大于GD水稻土.对稻秸添加量与产甲烷古菌mcrA基因拷贝数之间进行的相关性分析表明, 稻秸添加量与mcrA基因拷贝数之间呈显著正相关关系(图 5).其中JX水稻土的产甲烷古菌mcrA基因拷贝数变化与稻秸量添加之间线性关系的斜率更高, 表明JX水稻土的产甲烷古菌mcrA基因丰度对不同稻秸量添加的响应更敏感.
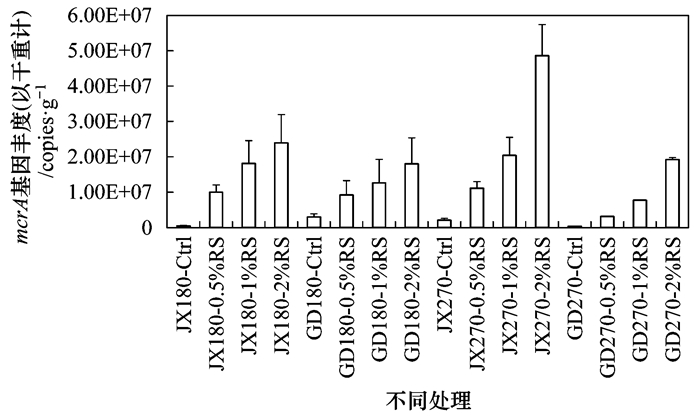
|
以JX180-2%RS为例:JX180表示JX水稻土培养第180 d, 2%RS表示添加2%稻秸处理;图内数值为平均值±标准误, n=3;方差分析仅在同一土壤在相同培养时间下的不同RS处理之间进行(P < 0.05) 图 4 两种水稻土在培养第180 d和第270 d时不同RS处理的mcrA基因丰度 Fig. 4 Abundance of mcrA genes in all RS treatments over sampling times of 180 days and 270 days in the two paddy soils |
|
以JX180 vs. RS为例:表示JX水稻土培养第180 d的mcrA基因丰度与稻秸量的线性回归 图 5 mcrA基因丰度与稻秸添加量之间的相关性分析 Fig. 5 Analysis of correlation between mcrA gene abundance and RS addition |
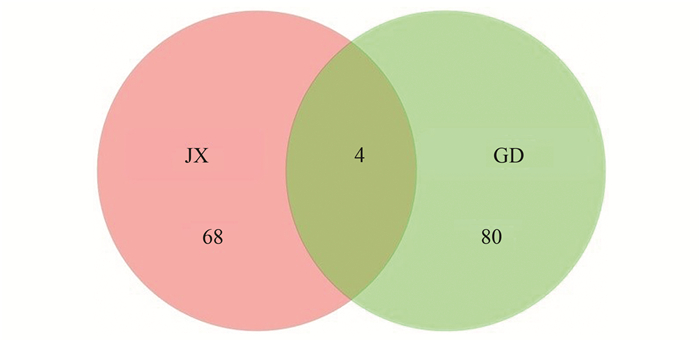
对两种水稻土培养第270 d时2% RS处理的细菌群落结构差异进行了分析(如图 6和7).由图 6可知两种水稻土的细菌OTU数明显不同. JX和GD水稻土细菌OTU数分别为72和84个, 其中特有的OUT数分别为68和80个, 两者共有的OUT数为4(图 7).表明两种水稻土细菌群落结构有很大差异, 且GD水稻土细菌多样性高于JX水稻土.

|
人为定义97%水平为属的序列差异阈值, 每条横线代表一个OTU, 将相似度≥97%的序列被认为是同一个OTU 图 6 2% RS处理的两种水稻土培养第270 d的细菌多样性在0.03水平的热图 Fig. 6 Heat map showing the bacteria diversity with 2% RS treatment at the 270th day of incubation in the two soils |
|
人为定义97%水平为属的序列差异阈值;图内数值OUT数量 图 7 2% RS处理的两种水稻土培养第270 d时细菌OTU在0.03水平下的维恩图 Fig. 7 Venn diagram showing the bacteria OTU at a 0.03 level with 2% RS treatment at the 270th day of incubation in the two soils |
对两种水稻土培养第270 d的细菌16S rRNA基因进行了克隆测序, 所测的JX和GD水稻土细菌16S rRNA基因序列分别有100条和96条.对所测序列进行了系统归类统计(表 2).由表 2结果显示, 两种水稻土细菌在属水平上有很大差异. JX水稻土的优势细菌(同一属的序列数≥5被认为是优势菌)有Bacillus、Desulfovirgula、Thermosporothrix、Acidobacteria/Gp1、Acidobacteria/Gp3及Ktedonobacter;而GD水稻土的优势细菌为Longilinea、Acidobacteria/Gp6、Bellilinea及Thermosporothrix.
|
|
表 2 2% RS处理培养第270 d水稻土细菌16S rRNA基因序列的系统归类 Table 2 Systematic classification of bacteria 16S rRNA gene sequences with 2% RS treatment at the 270th day of incubation in the two soils |
3 讨论 3.1 稻秸添加对两种水稻土产甲烷古菌群落结构及丰度的影响
本研究发现添加不同稻秸量对JX水稻土的产甲烷古菌群落结构有一定的影响.比如在培养第180 d和第270 d, 添加2%稻秸量使Methanobacteriaceae和Methanocellaceae的相对丰度增加, 但降低了Methanosarcinaceae的相对丰度.而对于GD水稻土, 不同稻秸量添加对产甲烷古菌群落结构的影响不明显.由此可见, 不同水稻土类型中添加不同稻秸量对产甲烷古菌群落结构的影响不尽相同, 这可能与土壤特性有关, 本研究中的JX水稻土属于粉砂壤土, GD水稻土属于粉质黏壤土, 其土壤黏粒含量高于JX水稻土.有研究表明土壤微生物活性与土壤黏粒含量之间存在负相关关系[24], 这些微生物包括有机质降解菌, GD水稻土黏粒含量高, 对该类菌群的活性不利, 因此可能影响了稻秸的降解程度或降解速度, 使得稻秸降解的终产物如H2和乙酸等(也是产甲烷古菌能量底物)也较低, 底物不充足可能影响产甲烷古菌的生长, 最终表现为GD水稻土中产甲烷古菌群落结构对稻秸添加的响应不明显.另外, JX和GD水稻土中的K含量、pH及C/N等都存在差异, 这可能也是造成两种水稻土的产甲烷古菌群落结构对稻秸添加有不同响应的原因之一. Peng等[19]及Cornad等[21]研究了不同类型秸秆对产甲烷古菌群落结构的影响, 结果表明秸秆类型对产甲烷古菌群落结构产生不同的影响, 而这些研究中并未设置不同稻秸量的处理.
本研究中两种水稻土的产甲烷古菌mcrA基因拷贝数均随稻秸添加量的增加而增加, 2%RS处理的产甲烷古菌mcrA基因拷贝数显著高于其他RS处理(图 4).相同稻秸量添加条件下, JX水稻土产甲烷古菌mcrA基因拷贝数大于GD水稻土.研究发现秸秆降解产生的乙酸、H2等为产甲烷古菌提供底物, 从而促进其生长[17, 20].表明底物作为能量物质是微生物生长的限制因子.稻秸添加量与产甲烷古菌丰度(mcrA基因拷贝数)之间存在显著正相关关系, JX水稻土的产甲烷古菌丰度变化对不同稻秸量的响应更敏感(图 5), 这可能与土壤特性及土著菌群降解稻秸的能力等因素有关.
另外, 本研究发现产甲烷古菌群落结构在两种水稻土之间也存在明显差异(图 1和图 2). JX水稻土产甲烷古菌多样性高于GD水稻土. JX水稻土共检测到的产甲烷古菌有Methanosarcinaceae(120 bp和136 bp)、Methanocellaceae(639 bp)、Methanomicrobiaceae(88 bp)、Methanobacteriaceae(667 bp及726 bp)以及未知菌群(494 bp). GD水稻土中仅有3大类产甲烷古菌, 分别为Methanobacteriaceae、Methanosarcinaceae和Methanocellaceae. Peng等[19]的研究中也检测到以上的产甲烷古菌, 且为优势菌群. JX水稻土中Methanosarcinaceae始终为绝对优势菌群, Methanobacteriaceae为第二大优势菌群.而在GD水稻土中, 产甲烷古菌群落存在演替现象, 随着培养时间的延长, 优势菌群由Methanosarcinaceae转变为Methanobacteriaceae.另外, Methanosarcinaceae和Methanocellaceae的相对丰度在JX水稻土中较高, 而Methanobacteriaceae的相对丰度在GD水稻土中较高.这种群落结构的差异反映了水稻土土壤类型对产甲烷古菌具有重要影响.研究发现Methanosarcinaceae是一类生长快且底物多样化的一类产甲烷古菌, 不仅能利用高浓度的乙酸(0.2~1.2 mmol·L-1), 也能利用H2-CO2或甲酸[23, 25].在前期的类似研究中发现, 稻秸添加后的培养体系中产生大量乙酸等小分子有机酸和H2[26], 这为Methanosarcinaceae的生长提供充足底物. Conrad等[18]的研究也发现Methanosarcinaceae在秸秆降解过程中利用H2-CO2进行繁殖. Methanobacteriaceae是一类使用H2-CO2的产甲烷古菌, 在水稻土中可以较容易地富集得到[27].有研究发现稻秸添加的厌氧体系中Methanobacteriaceae的响应归因于H2浓度的变化[19, 28]. Methanocellaceae是一类新发现的氢营养型的产甲烷古菌, 广泛分布在甲烷生成的环境中, 属于RC-I, 该类菌在水稻根际区域CH4产生过程中起非常关键的作用[28].低H2浓度的环境有利于Methanocellaceae的生长, 另外Methanocellaceae也是具有潜在的抗O2毒的一类产甲烷古菌, 它们在厌氧和好氧条件下均能生存.有研究发现在CH4大量产生阶段之后, 体系中的优势菌群为Methanosarcinaceae和Methanocellacellales[29].
3.2 两种水稻土的细菌菌群对稻秸添加的响应本研究发现两种水稻土的细菌群落结构明显不同, 二者之间相同的OTU数仅有4个(图 7). GD水稻土的多样性(OTU数为80)高于JX水稻土(OTU数为68).细菌多样性影响其生态功能, 在细菌多样性低的生态体系中, 细菌的生态功能也相对缺乏, 不利于生态系统的稳定.以往的研究发现, 土壤pH是影响土壤细菌群落地理分布的主要因素, 在农田生态系统中, 也有学者发现pH对细菌群落的改变起主要作用.孙瑞波等[13]的研究发现, 长期秸秆还田导致土壤pH降低, 使细菌多样性降低.本研究的JX水稻土本底pH值(pH=6.42)高于GD水稻土(pH=5.24), 而在培养过程中其细菌多样性低于GD水稻土, 这可能是由于在JX水稻土中稻秸添加后相对较长时间的厌氧培养造成大量有机酸的生成, 使JX水稻土培养体系中的pH降低, 因此降低了JX水稻土细菌多样性.同时, 有研究表明秸秆还田增加土壤碳含量, 进而影响细菌群落[13].本研究GD水稻土的总C(11.68 g·kg-1)高于JX水稻土的总C含量(10.27 g·kg-1), 这为土壤细菌提供了更多的本底生长底物碳源, 这也可能是两种水稻土细菌群落结构存在差异的原因之一.有研究发现营养物质丰富会促进富营养型的细菌群落生长, 而寡营养型细菌生长受到抑制, 细菌群落受到影响[11].
本研究对两种水稻土培养第180 d的细菌菌群进行了测序分析, 发现两种水稻土在属水平上有很大差异. JX水稻土的优势细菌共有6种:Bacillus、Desulfovirgula、Thermosporothrix、Acidobacteria/Gp1、Acidobacteria/Gp3和Ktedonobacter;而GD水稻土的优势细菌共有4种:Longilinea、Acidobacteria/Gp6、Bellilinea和Thermosporothrix. Rui等[12]的研究发现水稻土厌氧培养体系中细菌以Clostridium为优势菌, 尤其在培养早期.培养后期则以Bacteroidetes和Chlorobi为优势菌.本研究仅在JX水稻土中发现Clostridium, 且只占所测序列的2%. Bacteroidetes在两种水稻土中也仅发现2%的比例. Chlorobi在两种水稻土中均未发现. Acidobacteria在JX水稻土中占11%, 在GD水稻土中占15%.另外, 本研究的两种水稻土的优势细菌Acidobacteria, 在Rui等[12]的研究中仅在高温(45℃)培养体系中作为优势菌存在, 这种差异可能与水稻土的土壤类型及土壤特性有关.
Clostridia属革兰氏阳性菌, 是孢子形成的细菌.研究表明它是水稻土细菌菌群普遍存在的优势菌[6, 30~32].通过细菌16S rRNA基因检测到Bacteroidetes和Acidobacteria普遍存在于各种环境中. Bacteroidetes能够在各种复杂底物上消化和生长, 如纤维素、木质素和糖等.另有研究发现Bacteroidetes能够在底物有效性很低时也能够生长得很好[33, 34]. Acidobacteria由于很难进行培养, 目前其生态功能未知.有研究表明这类微生物生长缓慢, 且一般适宜在寡营养的环境中生存[35, 36].本研究的两种水稻土中均仅有两条序列属于Bacteroidetes, 而Acidobacteria在JX水稻土中有11条, GD水稻土中有10条.这可能与本研究测定的序列相对较少, 未能覆盖体系中所有的细菌也有关系. Wegner等[37]对秸秆降解早期的水稻土细菌菌群进行了分析, 发现Clostridiaceae、Lachnospiraceae、Ruminococcaceae、Veillonellaceae和Pseudomonadaceae是丰度最高且最活跃的菌群, 这些细菌对秸秆早期降解贡献很大.而在本研究中, 这些细菌群的丰度非常低, 仅占总序列的0~2%, 这可能与本研究培养时间相对较长有关. Wegner等[37]的研究也发现Firmicutes参与纤维素和几丁质的降解, 而Acidobacteria, Bacteriodetes及Chloroflexi参与半纤维素的降解.本研究中Firmicutes在GD水稻土中占所测序列的26%, 而在JX水稻土中占7%, 表明在GD水稻土中更多的Firmicutes与稻秸的后期降解有关. Cahyani等[38]的研究结果表明, α-Proteobacteria参与稻秸初始的降解, Thermophilic Bacillus spp.和Actinomycetes参与稻秸降解的嗜热阶段, Cytophaga和Clostridial members参与稻秸降解的固化阶段, 而Mesophilic Bacillus spp.一直参与稻秸降解的整个阶段. Weber等[6, 7]的研究发现稻秸厌氧降解的优势细菌属于Clostridium spp..日本水稻土秸秆降解中分离培养出了Actinobacteria, 尤其有Cellulomonas spp.[39].另有学者发现日本水稻土中参与稻秸降解的主要细菌有Proteobacteria、Cytophaga-Flavobacterium-Bacteroidetes[CFB]群及Spirochaetes[40].可以看出, 不同的研究得到了不同的结果, 表明秸秆添加在不同类型的水稻土时, 参与秸秆降解的细菌及土壤中的细菌菌群不尽相同.
4 结论不同稻秸添加量对JX水稻土产甲烷古菌群落结构有一定的影响, 而对GD水稻土的产甲烷古菌群落结构的影响不大.两种水稻土的产甲烷古菌mcrA基因拷贝数均随稻秸添加量的增加而增加.在相同稻秸量添加条件下两种水稻土产甲烷古菌群落不同. JX水稻土中的产甲烷古菌有Methanosarcinaceae、Methanocellaceae、Methanomicrobiaceae、Methanobacteriaceae以及未知菌群(494 bp). GD水稻土中的产甲烷古菌为Methanobacteriaceae、Methanosarcinaceae和Methanocellaceae.细菌群落结构在两种水稻土之间也有明显差异, GD水稻土的细菌多样性高于JX水稻土.综上表明, 稻秸量添加对不同水稻土产甲烷古菌群落结构的影响有差异, 但均能促进产甲烷古菌的生长.不同类型的水稻土经相对较长时间的厌氧培养后其产甲烷古菌及细菌菌群结构不同.
| [1] | Kimura M, Murase J, Lu Y H. Carbon cycling in rice field ecosystems in the context of input, decomposition and translocation of organic materials and the fates of their end products (CO2 and CH4)[J]. Soil Biology and Biochemistry, 2004, 36(9): 1399-1416. DOI:10.1016/j.soilbio.2004.03.006 |
| [2] | Lu Y H, Watanabe A, Kimura M. Input and distribution of photosynthesized carbon in a flooded rice soil[J]. Global Biogeochemical Cycles, 2002, 16(4): 1085. |
| [3] | Watanabe A, Yoshida M, Kimura M. Contribution of rice straw carbon to CH4 emission from rice paddies using 13C-enriched rice straw[J]. Journal of Geophysical Research, 1998, 103(D7): 8237-8242. DOI:10.1029/97JD03460 |
| [4] | Liesack W, Schnell S, Revsbech N P. Microbiology of flooded rice paddies[J]. FEMS Microbiology Reviews, 2000, 24(5): 625-645. DOI:10.1111/j.1574-6976.2000.tb00563.x |
| [5] | Glissmann K, Weber S, Conrad R. Localization of processes involved in methanogenic degradation of rice straw in anoxic paddy soil[J]. Environmental Microbiology, 2001, 3(8): 502-511. DOI:10.1046/j.1462-2920.2001.00212.x |
| [6] | Weber S, Lueders T, Friedrich M W, et al. Methanogenic populations involved in the degradation of rice straw in anoxic paddy soil[J]. FEMS Microbiology Ecology, 2001, 38(1): 11-20. |
| [7] | Weber S, Stubner S, Conrad R. Bacterial populations colonizing and degrading rice straw in anoxic paddy soil[J]. Applied and Environmental Microbiology, 2001, 67(3): 1318-1327. DOI:10.1128/AEM.67.3.1318-1327.2001 |
| [8] | Kennedy A C, Smith K L. Soil microbial diversity and the sustainability of agricultural soils[J]. Plant and Soil, 1995, 170(1): 75-86. DOI:10.1007/BF02183056 |
| [9] |
李秀英, 赵秉强, 李絮花, 等. 不同施肥制度对土壤微生物的影响及其与土壤肥力的关系[J]. 中国农业科学, 2005, 38(8): 1591-1599. Li X Y, Zhao B Q, Li X H, et al. Effects of different fertilization systems on soil microbe and its relation to soil fertility[J]. Scientia Agriculutra Sinica, 2005, 38(8): 1591-1599. DOI:10.3321/j.issn:0578-1752.2005.08.014 |
| [10] |
杨滨娟, 黄国勤, 钱海燕. 秸秆还田配施化肥对土壤温度、根际微生物及酶活性的影响[J]. 土壤学报, 2014, 51(1): 150-157. Yang B J, Huang G Q, Qian H Y. Effects of straw incorporation plus chemical fertilizer on soil temperature, root micro-organisms and enzyme activities[J]. Acta Pedologica Sinica, 2014, 51(1): 150-157. |
| [11] |
伍玉鹏, 彭其安, Shaaban M, 等. 秸秆还田对土壤微生物影响的研究进展[J]. 中国农学通报, 2014, 30(29): 175-183. Wu Y P, Peng Q A, Shaaban M, et al. Research progress of effect of straw returning on soil microorganism[J]. Chinese Agricultural Science Bulletin, 2014, 30(29): 175-183. DOI:10.11924/j.issn.1000-6850.2014-0064 |
| [12] | Rui J P, Peng J J, Lu Y H. Succession of bacterial populations during plant residue decomposition in rice field soil[J]. Applied and Environmental Microbiology, 2009, 75(14): 4879-4886. DOI:10.1128/AEM.00702-09 |
| [13] |
孙瑞波, 郭熙盛, 王道中, 等. 长期施用化肥及秸秆还田对砂姜黑土细菌群落的影响[J]. 微生物学通报, 2015, 42(10): 2049-2057. Sun R B, Guo X S, Wang D Z, et al. The impact of long-term application of chemical fertilizers and straw returning on soil bacterial community[J]. Microbiology China, 2015, 42(10): 2049-2057. |
| [14] | Shen C C, Xiong J B, Zhang H Y, et al. Soil pH drives the spatial distribution of bacterial communities along elevation on Changbai Mountain[J]. Soil Biology and Biochemistry, 2013, 57: 204-211. DOI:10.1016/j.soilbio.2012.07.013 |
| [15] |
李煜珊, 李耀明, 欧阳志云. 产甲烷微生物研究概况[J]. 环境科学, 2014, 35(5): 2025-2030. Li Y S, Li Y M, Ouyang Z Y. A research overview of methanogens[J]. Environmental Science, 2014, 35(5): 2025-2030. |
| [16] | Tanahashi T, Murase J, Matsuya K, et al. Bacterial communities responsible for the decomposition of rice straw compost in a Japanese rice paddy field estimated by DGGE analysis of amplified 16s rDNA and 16s rRNA fragments[J]. Soil Science and Plant Nutrition, 2005, 51(3): 351-360. DOI:10.1111/j.1747-0765.2005.tb00040.x |
| [17] |
卜元卿, 黄为一. 稻秸对土壤细菌群落分子多态性的影响[J]. 土壤学报, 2005, 42(2): 270-277. Bu Y Q, Huang W Y. Effect of rice straw on molecular phylogenetic diversify of soil bacterial communities[J]. Acta Pedologica Sinica, 2005, 42(2): 270-277. DOI:10.3321/j.issn:0564-3929.2005.02.015 |
| [18] | Conrad R, Klose M. Dynamics of the methanogenic archaeal community in anoxic rice soil upon addition of straw[J]. European Journal of Soil Science, 2006, 57(4): 476-484. DOI:10.1111/j.1365-2389.2006.00791.x |
| [19] | Peng J J, Lü Z, Rui J P, et al. Dynamics of the methanogenic archaeal community during plant residue decomposition in an anoxic rice field soil[J]. Applied and Environmental Microbiology, 2008, 74(9): 2894-2901. DOI:10.1128/AEM.00070-08 |
| [20] | Shrestha M, Shrestha P M, Conrad R. Bacterial and archaeal communities involved in the in situ degradation of 13C-labelled straw in the rice rhizosphere[J]. Environmental Microbiology Reports, 2011, 3(5): 587-596. DOI:10.1111/j.1758-2229.2011.00267.x |
| [21] | Conrad R, Klose M, Lu Y H, et al. Methanogenic pathway and archaeal communities in three different anoxic soils amended with rice straw and maize straw[J]. Frontiers in Microbiology, 2012, 3: 4. |
| [22] | Lu Y, Fu L, Lu Y H, et al. Effect of temperature on the structure and activity of a methanogenic archaeal community during rice straw decomposition[J]. Soil Biology and Biochemistry, 2015, 81: 17-27. DOI:10.1016/j.soilbio.2014.10.031 |
| [23] | Conrad R. Microbial ecology of methanogens and methanotrophs[J]. Advances in Agronomy, 2007, 96: 1-63. DOI:10.1016/S0065-2113(07)96005-8 |
| [24] | Müller T, Höper H. Soil organic matter turnover as a function of the soil clay content:consequences for model applications[J]. Soil Biology and Biochemistry, 2004, 36(6): 877-888. DOI:10.1016/j.soilbio.2003.12.015 |
| [25] | Jetten M S M, Stams A J M, Zehnder A J B. Acetate threshold values and acetate activating enzymes in methanogenic bacteria[J]. FEMS Microbiology Letters, 1990, 73(4): 339-344. DOI:10.1111/j.1574-6968.1990.tb03958.x |
| [26] | Bao Q L, Xiao K Q, Chen Z, et al. Methane production and methanogenic archaeal communities in two types of paddy soil amended with different amounts of rice straw[J]. FEMS Microbiology Ecology, 2014, 88(2): 372-385. DOI:10.1111/1574-6941.12305 |
| [27] | Asakawa S, Morii H, Akagawa-Matsushita M, et al. Characterization of Methanobrevibacter arboriphilicus SA isolated from a paddy field soil and DNA-DNA hybridization among M. arboriphilicus strains[J]. International Journal of Systematic and Evolutionary Microbiology, 1993, 43(4): 683-686. |
| [28] | Lu Y H, Conrad R. In situ stable isotope probing of methanogenic archaea in the rice rhizosphere[J]. Science, 2005, 309(5737): 1088-1090. DOI:10.1126/science.1113435 |
| [29] | Angel R, Claus P, Conrad R. Methanogenic archaea are globally ubiquitous in aerated soils and become active under wet anoxic conditions[J]. The ISME Journal, 2012, 6(4): 847-862. DOI:10.1038/ismej.2011.141 |
| [30] | Hengstmann U, Chin K J, Janssen P H, et al. Comparative phylogenetic assignment of environmental sequences of genes encoding 16S rRNA and numerically abundant culturable bacteria from an anoxic rice paddy soil[J]. Applied and Environmental Microbiology, 1999, 65(11): 5050-5058. |
| [31] | Lüdemann H, Arth I, Liesack W. Spatial changes in the bacterial community structure along a vertical oxygen gradient in flooded paddy soil cores[J]. Applied and Environmental Microbiology, 2000, 66(2): 754-762. DOI:10.1128/AEM.66.2.754-762.2000 |
| [32] | Stackebrandt E, Kramer I, Swiderski J, et al. Phylogenetic basis for a taxonomic dissection of the genus Clostridium[J]. FEMS Immunology & Medical Microbiology, 1999, 24(3): 253-258. |
| [33] | Kirchman D L. The ecology of Cytophaga-flavobacteria in aquatic environments[J]. FEMS Microbiology Ecology, 2002, 39(2): 91-100. |
| [34] | Sharp R, Macfarlane G T. Chemostat enrichments of human feces with resistant starch are selective for adherent butyrate-producing clostridia at high dilution rates[J]. Applied and Environmental Microbiology, 2000, 66(10): 4212-4221. DOI:10.1128/AEM.66.10.4212-4221.2000 |
| [35] | Eichorst S A, Breznak J A, Schmidt T M. Isolation and characterization of soil bacteria that define Terriglobus gen. nov., in the phylum Acidobacteria[J]. Applied and Environmental Microbiology, 2007, 73(8): 2708-2717. DOI:10.1128/AEM.02140-06 |
| [36] | Fierer N, Bradford M A, Jackson R B. Toward an ecological classification of soil bacteria[J]. Ecology, 2007, 88(6): 1354-1364. DOI:10.1890/05-1839 |
| [37] | Wegner C E, Liesack W. Microbial community dynamics during the early stages of plant polymer breakdown in paddy soil[J]. Environmental Microbiology, 2016, 18(9): 2825-2842. DOI:10.1111/1462-2920.12815 |
| [38] | Cahyani V R, Matsuya K, Asakawa S, et al. Succession and phylogenetic composition of bacterial communities responsible for the composting process of rice straw estimated by PCR-DGGE analysis[J]. Soil Science and Plant Nutrition, 2003, 49(4): 619-630. DOI:10.1080/00380768.2003.10410052 |
| [39] | Hiroshi A, Izawa T, Ueki K, et al. Phylogeny of numerically abundant culturable anaerobic bacteria associated with degradation of rice plant residue in Japanese paddy field soil[J]. FEMS Microbiology Ecology, 2003, 43(2): 149-161. DOI:10.1111/j.1574-6941.2003.tb01054.x |
| [40] | Sugano A, Tsuchimoto H, Tun C C, et al. Succession and phylogenetic profile of eubacterial communities in rice straw incorporated into a rice field:estimation by PCR-DGGE analysis[J]. Soil Science and Plant Nutrition, 2005, 51(1): 51-60. DOI:10.1111/j.1747-0765.2005.tb00006.x |
 2019, Vol. 40
2019, Vol. 40


