2. 山西水利职业技术学院水利工程系, 运城 044000
2. Department of Hydraulic Engineering, Shanxi Water Conservancy Technical College, Yuncheng 044000, China
河流在生态生源要素循环中起着至关重要作用[1], 也是地表水的重要水源之一.近年来, 西北地区城市的工农业不断发展, 在城镇居民比较集中的地区有大量无机氮通过生活污水和人畜粪便排放进入水体, 在农业比较发达的地方, 无机氮通过化肥的使用排放到河流中, 致使河流中无机氮浓度逐年上升.目前, 自然水环境污染越来越严峻, 使得淡水资源日益短缺, 对人类的生活与生产发展构成了严重阻碍[2, 3], 恶化的水体不仅使河流生态系统失去了自净功能, 而且导致了水环境中生物多样性的锐减[4].
微生物对自然河流生态系统的生化反应过程有较大影响, 包括氨氧化[5]、硝化[6]、硫酸盐还原以及甲烷的产生等.一般来说, 细菌种类繁多且很难培养, 传统技术较难分析细菌群结构与功能.目前, 已有克隆文库法[7]、变性梯度凝胶电泳技术[8]、荧光定量PCR技术和高通量DNA测序[9]等技术用来分析细菌群落, 且大多用于湿地[10]、湖泊[11]、水库[12]、河流[13, 14]和海洋[15]等水环境.姚炎红等[16]通过定量PCR及克隆文库结合T-RFLP研究了土壤中反硝化细菌的群落结构, 结果表明, 土壤中影响nirK型反硝化细菌群落的重要因子是PAHs.赵昕燕等[17]利用聚合酶链式反应-变性梯度凝胶电泳技术(PCR-DGGE)对比分析了MBBR反应器的短程硝化生物膜中的微生物群落结构.程建华等[18]采用荧光定量PCR技术分析季节和污染类型对沉积物样品中硝化(amoA)和反硝化(nirS、nirK)功能基因丰度的影响.由此可见, 学者们关于污染土壤、水源水库、河流沉积物等生态系统反硝化菌群结构的研究较多, 对于自然河流研究却鲜见报道.因此研究自然河流的微生物(反硝化细菌)群结构以及其与无机氮的相互影响关系, 对于改变水体中无机氮的含量并进一步缓解河流无机氮污染问题, 有着重要的意义.
汾河是山西第一大河, 位于山西省的中南部, 下游流域位于东经110°30′~112°34′, 北纬35°20′~36°57′, 河段全长210.5 km, 流域面积11 276 km2.随着汾河流域城市化进程的影响, 人类活动对于水质的影响逐渐加剧, 越来越多的学者对汾河下游无机氮进行了研究.通常通过水质与土地类型相关性来分析水中无机氮污染源[19], 近几年, 学者利用同位素来解析水体中无机氮的来源, 如孟志龙等[20]基于氮、氧同位素对汾河下游硝酸盐污染源进行了解析.对汾河下游水体中反硝化细菌群落结构与无机氮关系的研究鲜见报道.本研究选取汾河下游9个采样点的水样作为对象, 在综合水质理化因子分析的基础上, 运用特定基因DNA测序技术分析汾河下游nirS型反硝化细菌群落结构, 探知汾河下游无机氮与反硝化细菌群落结构的相互联系, 以期为后期汾河下游环境检测以及无机氮治理提供科学的基础数据依据.
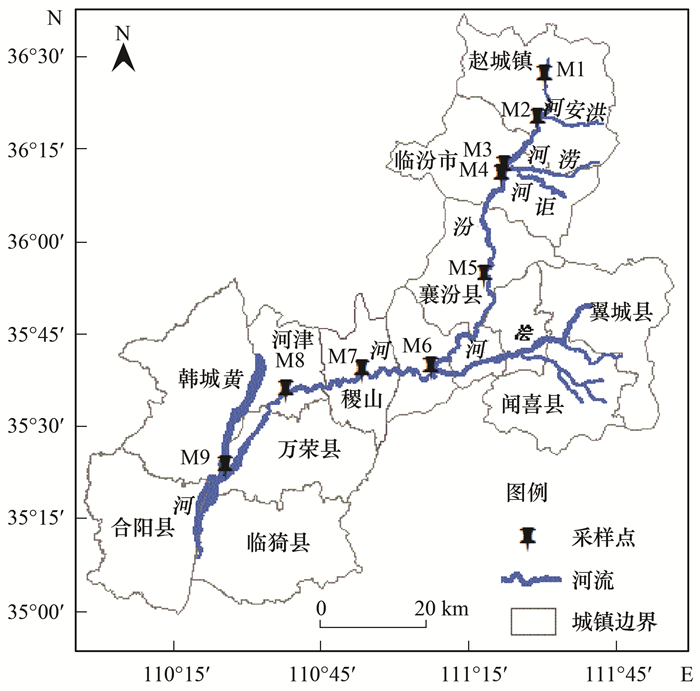
1 材料与方法 1.1 研究地点和样本采集汾河是黄河的一级支流, 下游无机氮污染严重.本研究沿汾河下游赵城镇到入黄口的9个点进行水样采集.如图 1所示, 分别为赵城镇(M1, 36°23′1.1″N, 111°39′43″E)、洪洞(M2, 36°15′13.9″N, 111°38′17.7″E)、临汾(M3, 36°4′49.7″N, 111°29′11.4″E)、涝河口(M4, 36°7′39.2″N, 111°30′4.2″E)、襄汾(M5, 35°52′42.3″N, 111°25′38.9″E)、浍河口(M6, 35°35′27.6″N, 111°11′58.6″E)、稷山(M7, 35°35′25.5″N, 110°58′22.4″E)、河津(M8, 35°33′23.5″N, 110°42′20.1″E)和入黄口(M9, 35°21′9.3″N, 110°28′41.1″E).用采样器在汾河下游河流中距离表面0.2 m处取水样, 并立即转移到无菌瓶中.水样被带回实验室后, 首先通过0.45 μm孔径滤膜过滤, 去除较大的悬浮颗粒, 然后于-4℃下冷藏保存.
|
图 1 采样点示意 Fig. 1 Sampling point schematic |
使用便携式多参数分析仪(HACH-HQ30d)现场测定水样中的溶解氧(dissolved oxygen, DO)、温度T和pH值. TN、NO3--N采用紫外分光光度法(HJ 636-2012)测定; NH4+-N采用纳氏试剂分光光度法(HJ 535-2009)测定; NO2--N采用N-(1-萘基)-乙二胺光度法测定.
1.3 DNA提取与高通量测序用OMEGA Soil DNA Kit提取截留在滤膜上的微生物DNA, 并用NanoDrop2000分光光度计(美国Hudson, Thermo Fisher)和琼脂糖凝胶电泳(Resp)检测DNA的浓度和完整性. DNA电泳结果符合质量要求, 可以进行后续实验.以提取的DNA为模板, 用引物(Cd3Af/R3cd)进行PCR扩增微生物16S rRNA基因V3-V4高变区. PCR反应体系(30 μL):2×HiFi Hot Start Ready Mix 12.5 μL, 引物cd3aF (10 μmol ·L-1) 0.5 μL, 引物R3cd (10 μmol ·L-1) 0.5 μL, DNA模板12.5ng. PCR反应条件:95℃ 3 min, 95℃ 30 s, 60℃ 30 s, 72℃ 30 s, 25个循环; 72℃ 10 min.然后检测PCR扩增产物的纯度, 检测后的产物使用磁珠纯化.经过第1阶段PCR产物纯化后, 进行第二阶段PCR扩增, PCR反应体系(30 μL):2×HiFi Hot Start Ready Mix 12.5 μL, 引物cd3aF (10 μmol ·L-1) 0.5 μL, 引物R3cd (10 μmol ·L-1) 0.5 μL, 第一轮PCR纯化产物10 ng. PCR反应条件:95℃ 3 min, 95℃ 30 s, 55℃ 30 s, 72℃ 30 s, 8个循环, 72℃ 10 min.并再次检测产物纯度, 检测后使用磁珠纯化, 对纯化后的PCR产物进行Qubit定量.按照PCR产物的浓度进行等量混样, 在Illumina MiSeq测序平台上测序.
1.4 测序分析使用Trimmomatic软件对原始双端序列进行去杂, 为保证结果的准确性, 可进行精准去杂, 精准去杂的参数为:去掉含有N碱基的序列, 保留碱基质量分数Q20达到75%以上的序列.去杂后的双端序列利用FLASH软件进行.同时, 利用UCHIME检测序列.在测序数据经预处理生成优质序列之后, 采用Vsearch软件, 根据序列的相似性, 将序列归为多个OTUs(序列相似度≥97%).使用QIIME软件包挑选出各个OTUs的代表序列, 将所有主要序列与数据库Silva(version123)[21]比对后最终得到系统发育树和OTUs代表的分类信息, 并在属的水平上分析每个样品的群落结构组成.
1.5 统计分析使用SPSS 21.0软件(SPSS, USA)对9个水样的环境参数的差异进行单因素方差分析(ANOVA).基于细菌群落组成及水体物理环境因子数据, 利用CANOCO(Version 5.0)进行主成分分析(PCA)及相关性分析, 探明汾河下游反硝化细菌群落与无机氮的相互联系. PCA分析图中, 主要解释变量在轴PC1与轴PC2上, 实心箭头表示不同水质因子, 向量长短表示相应水质指标对主轴的解释量.采用R软件(Version 2.1.1)制作热图(heat map), 通过颜色梯度反映不同采样点水体反硝化菌群在属水平上群落组成差异[22, 23].
2 结果与分析 2.1 环境表征汾河下游9个点采集水样的理化参数如表 1所示. T和pH值差异性不高(P>0.05), 属于正常的水环境指标范围.溶解氧(DO)沿着河流明显降低, 在稷山、河津及入黄口这3个采样点的含量小于3mg ·L-1(F=98.8, P < 0.01), 这可能由于下游水流缓慢, 有利于好氧微生物的生存, 消耗了大量水中溶解氧; 下游9个取样点的NH4+-N浓度范围为(6.49±0.05)~(10.28±0.05)mg ·L-1(F=59.62, P < 0.01), 均高于Ⅴ类水标准(≤2.0mg ·L-1); NO3--N浓度范围为(9.24±0.08)~(14.27±0.05)·L-1(F=63.58, P < 0.01), 除了洪洞(M2)和临汾(M3)这2个点的NO3--N浓度未超过10mg ·L-1, 其余7个点均超出了国家水质标准(10mg ·L-1), 主要因为河流两岸分布大量城镇和农业用地, 导致含有NO3--N、NH4+-N的污水、粪肥进入到河流.整体来看, 汾河下游无机氮污染较为严重, 水体水质属于Ⅴ类水标准.
|
|
表 1 不同采样点水体水质指标分析1) Table 1 Analysis of water quality indicators at different sampling points |
2.2 反硝化细菌群落结构 2.2.1 细菌群落多样性
通过MiSeq高通量测序, 汾河下游9个样点水样共得到高质量16S rRNA基因序列290 364条, 以97%的序列相似度对得到的基因序列进行OTUs划分, 共获得4 440条OTUs, 单个样点水样的OTUs数量为233~794之间.各样点的OTUs数量和多样性指数如表 2所示, M8点的OTUs数目最多, 为794; M2点的OTUs数目最少, 为233; 9个点共有76个相同的OTUs, 占OTUs总数的6.29%, 表明汾河下游不同空间的水环境对反硝化细菌群落组成影响显著.各样本文库的覆盖度(coverage)范围为88.82%~97.2%, 平均为94.26%, 表明测序深度可以反映汾河下游水体中微生物群落的真实情况.由Chao1丰度指数可知, M1样点微生物丰度最高; M2、M3样点Chao1指数较低, 微生物丰度相对也较低. 9个样点的Simpson指数在0.81~0.98之间, 其中M1、M4和M5样点Simpson指数值较高, 分别为0.98、0.96和0.97, 与其它样点Simpson指数(0.80~0.90)有差异, 说明此3个样点水体较其他水体具有丰富的群落结构.
|
|
表 2 各取样点水体反硝化细菌群落多样性指数 Table 2 Denitrifying bacteria community diversity index for water samples at each sampling point |
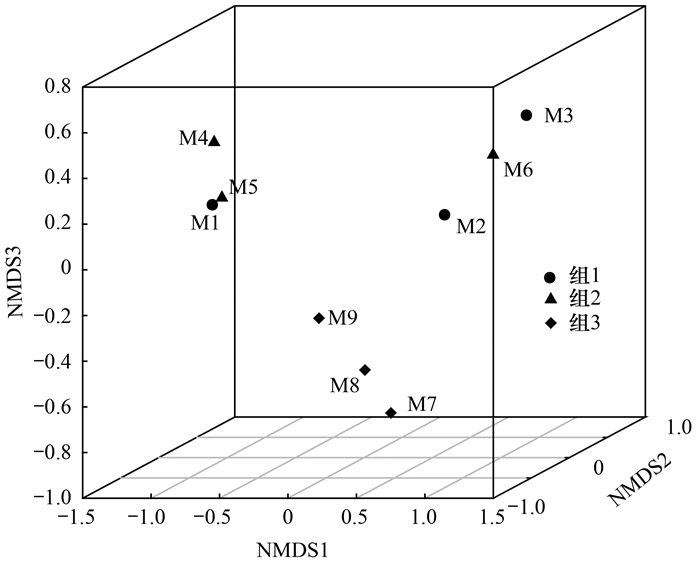
Shannon指数反映了反硝化细菌的群落多样性.如表 2中, 汾河下游9个采样点的Shannon指数变化范围为3.36~7.54, 平均值为5.32, M1样品Shannon指数最高, M2样品Shannon指数最低, 说明M1处的反硝化细菌多样性最高, M2处多样性最低.在接近入黄口的3个点水样Shannon指数较均匀, 变化范围为4.66~5.52, 说明入黄口处的反硝化细菌的多样性差异较小.根据空间位置将采样点分为3个组, 非度量多维尺度分析(nonmetric multidimensional scaling, NMDS)表明(图 2), 汾河下游组2内的M4和M5样点反硝化细菌群落结构具有极高的相似性, 组3内M7、M8和M9的反硝化细菌的群落结构组也具有一定的相似性, 而组1内的M1、M2和M3这3个样点的反硝化细菌的群落结构差异性较高.说明靠近入黄口的3个采样点水流缓慢, 地势平坦, 周边土地类型主要为农业用地, 水中的无机氮主要来源于农业氮肥的使用, 且水中溶解氧均低于3mg ·L-1, 相似的水环境条件为反硝化细菌提供了一样的生存基础; 汾河下游前3个采样点(M1、M2和M3)周围分布有大量焦化厂以及矿场, 各种污水的排放使得水体中水环境有很大的不同, 致使不同空间的反硝化细菌群落结构有明显的差异性.
|
图 2 NMDS分析 Fig. 2 NMDS analysis plot |
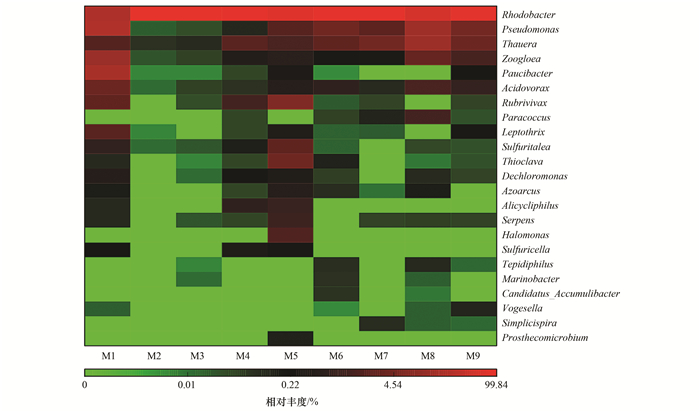
对97%的相似水平下的OUTs进行生物信息统计分析, 结果表明4 440个OUTs主要23分为个属.热图(heat map)可以直观地将数据的大小以定义的颜色深浅表示出来, 在属水平上对各样品中不同种属丰度做热图. 图 3表明, 汾河下游水体具有丰富的反硝化细菌群落结构, 但主要菌群存在差异. M1与M5样点水体具有较丰富的群落结构, M2和M3样点水体的群落结构较单一, 这主要是因为M1和M5采样点分别接近赵城镇、襄汾县(未流经), 河流流速缓慢, 周边生活、工厂废弃物排放严重, 使得水体水环境更为复杂, 水体中悬浮固体物较高, 为微生物生存提供了良好的环境.相反, M2和M3采样点位于洪洞县以及临汾市城中, 河流流经城市公园以及湿地保护区(区域内有大量的植物)时水体经过了简单的净化, 且周边严禁污水排放, 水体污染较小, 水环境适合少量的微生物生存.从优势属来看, M1采样点优势菌属为假单胞菌属Pseudomonas, 占比为23.3%, 主要菌属为红细菌属Rhodobacter及Paucibacter, 分别占比21.4%和19.9%.假单胞菌属中多个种属于反硝化过程的典型菌株, 其中施氏假单胞菌种Pseudomonas stutzeri可以去除97%的硝酸盐氮[24], 因此假单胞菌属作为优势属可能参与水体脱氮作用.其余8个样点的优势菌属均为红细菌属Rhodobacter, 在M2和M3样点中分别占比高达99.6%和98.7%.除了优势菌属外, 各样点的其他菌属差异性很大.副球菌属Paracoccus更适合生存在总氮含量较高水环境中, 是一种好氧反硝化细菌, 对于促进反硝化作用具有较大影响.在M1和M8样点总氮含量较高的水体中检测到副球菌属Paracoccus占了总菌属的2.9%和2.6%, 而总氮浓度较低的其他样点的副球菌属Paracoccus占比很小, 甚至没有检测出; 假单胞菌属Pseudomonas不仅是M1样点的优势菌属, 而且在M7、M8和M9样点也占有较高的比例, 这为入黄口处硝态氮转化为其他形态氮做出很大贡献; 在M5样点中检测到了大量变形菌门Proteobacteria中的红长命菌属Rubrivivax[25], 虽然不是优势菌属, 但是相对其他菌属也占了一定的比例, 对反硝化作用有很大的影响; 另外, 9个样点中, 陶厄氏菌属Thauera占比也比较高, 对反硝化作用有着促进作用[26], 其余菌属虽然占比不高, 但是对水体中反硝化作用也发挥了一定的作用.
|
图 3 各样点优势菌属热图 Fig. 3 Heat map of the dominant bacteria |
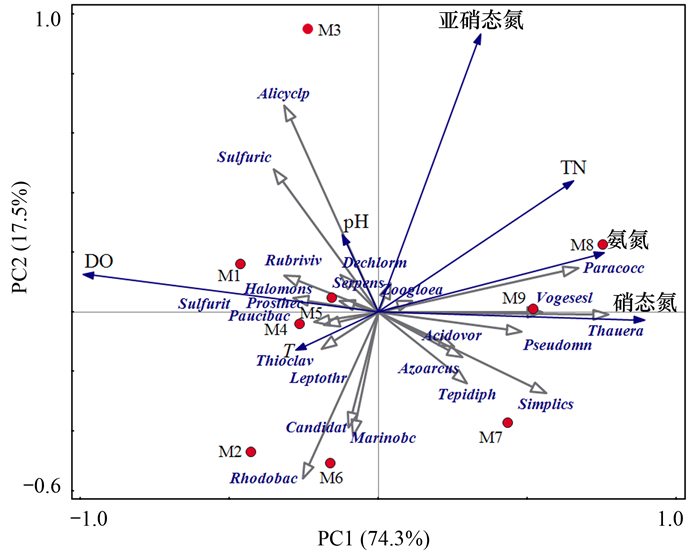
基于反硝化细菌菌属类别及水体水质数据基础, 对细菌菌属群落与水体无机氮进行主成分分析(principal component analysis, PCA)及Spearman相关性分析.主成分(PC1和PC2)可以解释菌属结构的91.8%, 绝大多数nirS型反硝化细菌属显示在PC1轴上, PC1解释总变异的74.3%, PC2解释总变异的17.5%.由图 4可知, 9个样点中M3与其他样点的反硝化细菌群落互异, 其他8个样点可分为相似度较高的3组群落结构, M1、M4和M5样点水体反硝化细菌群落相似度极高, M2和M6样点水体反硝化细菌群落相似度高, 同样, M7、M8和M9样点水体也具有相似度较高的反硝化细菌菌属群落.无机氮与不同反硝化细菌菌属群落有着较大的关联, 在反硝化细菌菌属群落具有相似度较高的M7、M8和M9样点水体中, TN、NH4+-N和NO3--N是反硝化细菌菌属群落的主要影响因子, 红细菌属Rhodobacter、假单胞菌属Pseudomonas以及陶厄氏菌属Thauera这3种菌属与无机氮的浓度显著关系; 温度(T)、DO是M1、M4和M5样点水体中的多种反硝化细菌菌属的主要影响因子, 而TN、NH4+-N、NO3--N、NO2--N与浮球菌属Paracocccu、红细菌属Rhodobacter以及假单胞菌属Pseudomonas、陶厄氏菌属Thauera呈负相关, 对降低无机氮浓度有一定促进作用; pH值与NO2--N是M3样点水体中反硝化细菌菌属的主要环境影响因子.
|
图 4 反硝化细菌群落的主成分分析 Fig. 4 Principal component analysis for the denitrifying bacterial populations |
由主要菌属与水质因子的Spearman相关性分析可知(表 3), 汾河下游水体中反硝化细菌的优势菌属红细菌属Rhodobacter与T、DO和pH值呈正相关, 但相关性不高(|F| < 0.3), 与NO3--N、NO2--N和TN等无机氮呈负相关, 且相关性较高(|F|>0.5). M1、M4和M5样点水体反硝化细菌群落结构主要受DO、T和pH值这3个水质因子影响(P < 0.05), 其中DO的影响最为显著(P < 0.01), 而无机氮NO3--N、NO2--N和TN等对群落结构几乎没有影响(P=NS), 说明在M1、M4和M5样点水体中反硝化细菌对于无机氮的浓度没有明显影响; T是M2和M6样点水体反硝化细菌群落结构的影响因子(P < 0.05), 但影响显著性不高; 接近入黄口M7、M8和M9样点处, NO3--N、NO2--N和TN这3个水质因子对反硝化细菌群落结构的影响作用较高, 3个水样中检测到了大量红细菌属Rhodobacter以及假单胞菌属Pseudomonas、陶厄氏菌属Thauera, 这3种nirS型反硝化菌属群落结构与盐度、氨氮和硝态氮浓度显著相关[27].
|
|
表 3 主要菌属与环境因子的Spearman相关性分析 Table 3 Spearman correlation analysis between the dominant bacteria and environmental factors |
3 讨论
汾河下游无机氮污染较为严重, 在9个采样点的水样中, 7个点的NO3--N含量均超出了国家水质标准(10mg ·L-1), 占比为78%, NH4+-N浓度均高于Ⅴ类水标准(≤2.0mg ·L-1).这主要因为所选取的9个取样点都是在城镇区以及河流交汇处, 在城镇区有工业以及生活污水排放, 如襄汾(M5)的NH4+-N浓度较高, 主要和城内的生活污水大量排放有关.而河流交汇处, 很多NH4+-N都是由支流汇流带进来的.浍河口处(M6)和稷山(M7)这2个点的NH4+-N浓度分别为8.52mg ·L-1和7.63mg ·L-1.氨氮含量最高处河津市为9.64mg ·L-1, 其次为入黄口9.54mg ·L-1, 主要因为入黄口往上是万荣县, 该县城拥有大面积的农业耕地, 大量的农业化肥通过灌溉用水和雨水汇入河道, 造成NH4+-N的增加, 同时上游河津市的3个污水处理厂的排水也会影响该地区NH4+-N的浓度.另外, 7月正处于农耕繁忙时期, 由于雨水的冲刷与汇入, 农田的化肥氮素(NO3--N为主)进入河流, 导致汾河河流水体中的NO3--N浓度快速增加, 同时在入黄口断面处, 黄河水流量过大就会产生河水倒灌, 稀释了河流交汇处NO3--N的浓度, 交汇处河道宽阔平坦, 水流速缓慢, NO3--N浓度由于微生物反硝化的降解作用有所降低[28].整体来看, 汾河下游无机氮污染较为严重, 水体水质属于Ⅴ类水标准.
细菌多样性指数即Shannon多样性是反映多样性综合性指数, 既包含物种丰富度又包括均匀度[29].本研究中, 在9个水样中Shannon指数有很大的差异, 这主要是由于汾河下游周围环境不同, 各个样点处的水环境较为复杂, 给不同的反硝化细菌提供了良好的生存环境. M1与M2样品Shannon指数相差最大, 说明M1处的反硝化细菌多样性较M2处多样性丰富.虽然M1与M2样点相距较近, 但是M1样点周围分布有大量的焦化厂以及煤场、矿场, 不同的废水排放致使河流水环境更为复杂, 且大量的煤粉、矿粉进入河流为微生物提供了良好的生存环境, 这与一些河流细菌多样性研究基本一致[30]. 9个采样点中赵城镇(M1)水体中优势菌属为假单胞菌属Pseudomonas, 其余8个点水体中优势菌属均为红细菌属Rhodobacter, 在M2和M3样品中分别占比高达99.6%和98.7%, 是绝对的优势菌属.另外, 9个样点中, 陶厄氏菌属Thauera占比也比较高, 在M7、M8和M9样品中占比分别为5.7%、13.5%和5.4%, 陶厄氏菌属Thauera能够很好地促进反硝化[31], 对水体脱氮起很大的作用.
由优势菌属与水质指标间的主成分分析及Spearman相关性分析知, 汾河下游水体中优势菌属红细菌属Rhodobacter与T、DO和pH值呈正相关, 与NO3--N、NO2--N和TN等无机氮呈负相关, 相关系数均小于-0.5, 与NH4+-N呈正相关.另外, 稷山、河津和入黄口这3处的主要菌属假单胞菌属Pseudomonas与NO3--N呈负相关关系(R=-0.551, P < 0.05), 与NO2--N呈负相关关系(R=-0.220, P < 0.05), 与NH4+-N呈正相关(R=0.637, P < 0.05), 陶厄氏菌属Thauera与NO3--N和NO2--N呈负相关关系.这说明水体中发生了复杂的反硝化作用, 且红细菌属Rhodobacter和假单胞菌属Pseudomonas对脱氮有一定的促进作用, 陶厄氏菌属Thauera是一类革兰氏阴性菌, 可以参与反硝化作用.在M7、M8和M9采样点处, 汾河地势平坦, 水流速度缓慢, DO均低于3 mg ·L-1, 有利用反硝化反应进行, 这对水体发生反硝化作用提供了一定的环境基础, 对河流中无机氮含量变化具有一定的影响.
4 结论(1) 汾河下游无机氮污染较为严重, 河流整体水质为Ⅴ类水标准, 其中NH4+-N浓度均高于2.0mg ·L-1, 78%的水体中NO3--N含量均超出了国家水质标准(10mg ·L-1).
(2) 汾河下游不同空间水体中的nirS反硝化细菌群落结构差异显著.赵城镇、涝河口、襄汾这3处水体群落结构相似, 稷山、河津、入黄口这3处也具有相似的群落结构, 但洪洞县、临汾、浍河口却与其他样点的群落结构相异. 9个采样点中除赵城镇水体中优势菌属为假单胞菌属Pseudomonas外, 其余8个点水体中优势菌属均为红细菌属Rhodobacter.
(3) 汾河下游赵城镇、临汾、襄汾水体中反硝化细菌群落结构主要受DO、T、pH值这3个水质因子影响(P < 0.05), T是洪洞、浍河口两处水体中反硝化细菌群落结构的影响因子, 但影响显著性不高; 下游的稷山、河津、入黄口水体中, NO3--N、NO2--N和TN是影响反硝化细菌群落结构的重要环境因子(P < 0.05).各采样点处群落结构中的优势菌属红细菌属Rhodobacter、假单胞菌属Pseudomonas、陶厄氏菌属Thauera与NO3--N和NO2--N等无机氮呈负相关, 与NH4+-N呈正相关, 对水体中无机氮的含量有一定的影响.
| [1] | Williamson C E, Dodds W, Kratz T K, et al. Lakes and streams as sentinels of environmental change in terrestrial and atmospheric processes[J]. Frontiers in Ecology and the Environment, 2008, 6(5): 247-254. DOI:10.1890/070140 |
| [2] |
秦伯强, 高光, 朱广伟, 等. 湖泊富营养化及其生态系统响应[J]. 科学通报, 2013, 58(10): 855-864. Qin B Q, Gao G, Zhu G W, et al. Lake eutrophication and its ecosystem response[J]. Chinese Science Bulletin, 2013, 58(9): 961-970. |
| [3] |
郝利霞, 孙然好, 陈利顶. 海河流域河流生态系统健康评价[J]. 环境科学, 2014, 35(10): 3692-3701. Hao L X, Sun R H, Chen L D. Health assessment of river ecosystem in haihe river basin, China[J]. Environmental Science, 2014, 35(10): 3692-3701. |
| [4] | Weijters M J, Janse J H, Alkemade R, et al. Quantifying the effect of catchment land use and water nutrient concentrations on freshwater river and stream biodiversity[J]. Aquatic Conservation, 2009, 19(1): 104-112. DOI:10.1002/aqc.v19:1 |
| [5] | Nam Y D, Sung Y, Chang H W, et al. Characterization of the depth-related changes in the microbial communities in Lake Hovsgol sediment by 16S rRNA gene-based approaches[J]. The Journal of Microbiology, 2008, 46(2): 125-136. DOI:10.1007/s12275-007-0189-1 |
| [6] | Xia X H, Zhou J S, Yang Z F. Nitrogen contamination in the Yellow River basin of China[J]. Journal of Environmental Quality, 2002, 31(3): 917-925. DOI:10.2134/jeq2002.9170 |
| [7] | Gao J, Hou L J, Zheng Y L, et al. nirS-Encoding denitrifier community composition, distribution, and abundance along the coastal wetlands of China[J]. Applied Microbiology and Biotechnology, 2016, 100(19): 8573-8582. DOI:10.1007/s00253-016-7659-5 |
| [8] | Yasumoto J, Yasumoto-Hirose M, Kudeken T, et al. Microbial community analysis of groundwater related to the denitrification in Ryukyu limestone aquifer[J]. Journal of Groundwater Hydrology, 2015, 57(2): 153-169. DOI:10.5917/jagh.57.153 |
| [9] | Wang Z, Zhang X X, Lu X, et al. Abundance and diversity of bacterial nitrifiers and denitrifiers and their functional genes in tannery wastewater treatment plants revealed by high-throughput sequencing[J]. PLoS One, 2014, 9(11): e113603. DOI:10.1371/journal.pone.0113603 |
| [10] |
魏佳明, 崔丽娟, 李伟, 等. 表流湿地细菌群落结构特征[J]. 环境科学, 2016, 37(11): 4357-4365. Wei J M, Cui L J, Li W, et al. Characteristics of bacterial communities in surface-flow constructed wetlands[J]. Environmental Science, 2016, 37(11): 4357-4365. |
| [11] |
张雅洁, 李珂, 朱浩然, 等. 北海湖微生物群落结构随季节变化特征[J]. 环境科学, 2017, 38(8): 3319-3329. Zhang Y J, Li K, Zhu H R, et al. Community structure of microorganisms and Its seasonal variation in Beihai Lake[J]. Environmental Science, 2017, 38(8): 3319-3329. |
| [12] |
陈兆进, 丁传雨, 朱静亚, 等. 丹江口水库枯水期浮游细菌群落组成及影响因素研究[J]. 中国环境科学, 2017, 37(1): 336-344. Chen Z J, Ding C Y, Zhu J Y, et al. Community structure and influencing factors of bacterioplankton during low water periods in Danjiangkou Reservoir[J]. China Environmental Science, 2017, 37(1): 336-344. |
| [13] | Staley C, Johnson D, Gould T J, et al. Frequencies of heavy metal resistance are associated with land cover type in the Upper Mississippi River[J]. Science of the Total Environment, 2015, 511: 461-468. DOI:10.1016/j.scitotenv.2014.12.069 |
| [14] |
刘睿, 吴巍, 周孝德, 等. 渭河浮游细菌群落结构特征及其关键驱动因子[J]. 环境科学学报, 2017, 37(3): 934-944. Liu R, Wu W, Zhou X D, et al. Bacterioplankton community structure in Weihe River and its relationship with environmental factors[J]. Acta Scientiae Circumstantiae, 2017, 37(3): 934-944. |
| [15] |
白洁, 刘小沙, 侯瑞, 等. 南海南部海域浮游细菌群落特征及影响因素研究[J]. 中国环境科学, 2014, 34(11): 2950-2957. Bai J, Liu X S, Hou R, et al. Community structure and influencing factors of bacterioplankton in the southern South China Sea[J]. China Environmental Science, 2014, 34(11): 2950-2957. |
| [16] |
姚炎红, 王明霞, 左小虎, 等. 典型油田多环芳烃污染对土壤反硝化微生物群落结构的影响[J]. 环境科学, 2016, 37(12): 4750-4759. Yao Y H, Wang M X, Zuo X H, et al. Effects of PAHs pollution on the community structure of denitrifiers in a typical oilfield[J]. Environmental Science, 2016, 37(12): 4750-4759. |
| [17] |
赵昕燕, 卞伟, 侯爱月, 等. 季节性温度对短程硝化系统微生物群落的影响[J]. 中国环境科学, 2017, 37(4): 1366-1374. Zhao X Y, Bian W, Hou A Y, et al. Characteristics of microbial community structure in the stable operation of the partial cut nitrification system with seasonal temperature[J]. China Environmental Science, 2017, 37(4): 1366-1374. DOI:10.3969/j.issn.1000-6923.2017.04.021 |
| [18] |
程建华, 窦智勇, 孙庆业. 铜陵市河流沉积物中硝化和反硝化微生物分布特征[J]. 环境科学, 2016, 37(4): 1362-1370. Cheng J H, Dou Z Y, Sun Q Y. Distribution characteristics of nitrifiers and denitrifiers in the river sediments of Tongling City[J]. Environmental Science, 2016, 37(4): 1362-1370. |
| [19] | Chang C C Y, Kendall C, Silva S R, et al. Nitrate stable isotopes:tools for determining nitrate sources among different land uses in the Mississippi River Basin[J]. Canadian Journal of Fisheries and Aquatic Sciences, 2002, 59(12): 1874-1885. DOI:10.1139/f02-153 |
| [20] |
孟志龙, 杨永刚, 秦作栋, 等. 汾河下游流域水体硝酸盐污染过程同位素示踪[J]. 中国环境科学, 2017, 37(3): 1066-1072. Meng Z L, Yang Y G, Qin Z D, et al. Isotopic tracing for nitrate pollution process of water body in the lower reaches of Fenhe River[J]. China Environmental Science, 2017, 37(3): 1066-1072. |
| [21] | Quast C, Pruesse E, Yilmaz P, et al. The SILVA ribosomal RNA gene database project:improved data processing and web-based tools[J]. Nucleic Acids Research, 2013, 41(D1): D590-D590. |
| [22] | Oberauner L, Zachow C, Lackner S, et al. The ignored diversity:complex bacterial communities in intensive care units revealed by 16S pyrosequencing[J]. Scientific Reports, 2013, 3: 1413. DOI:10.1038/srep01413 |
| [23] | Jami E, Israel A, Kotser A, et al. Exploring the bovine rumen bacterial community from birth to adulthood[J]. The ISME Journal, 2013, 7(6): 1069-1079. DOI:10.1038/ismej.2013.2 |
| [24] | Huang T L, Guo L, Zhang H H, et al. Nitrogen-removal efficiency of a novel aerobic denitrifying bacterium, Pseudomonas stutzeri strain ZF31, isolated from a drinking-water reservoir[J]. Bioresource Technology, 2015, 196: 209-216. DOI:10.1016/j.biortech.2015.07.059 |
| [25] |
王春香, 刘常敬, 郑林雪, 等. 厌氧氨氧化耦合脱氮系统中反硝化细菌研究[J]. 中国环境科学, 2014, 34(7): 1878-1883. Wang C X, Liu C J, Zheng L X, et al. Denitrifying bacteria of anaerobic ammonium-oxidizing denitrifying system[J]. China Environmental Science, 2014, 34(7): 1878-1883. |
| [26] | Ma W J, Liu C S, Zhao D F, et al. Microbial characterization of denitrifying sulifde removal sludge using high-throughput amplicon sequencing method[J]. China Petroleum Processing and Petrochemical Technology, 2015, 17(4): 89-95. |
| [27] | Yang J K, Cheng Z B, Li J, et al. Community composition of nirS-type denitrifier in a shallow Eutrophic Lake[J]. Microbial Ecology, 2013, 66(4): 796-805. DOI:10.1007/s00248-013-0265-5 |
| [28] | Pardo L H, Kendall C, Pett-Ridge J, et al. Evaluating the source of streamwater nitrate using δ15N and δ18O in nitrate in two watersheds in New Hampshire, USA[J]. Hydrological Processes, 2004, 18(14): 2699-2712. DOI:10.1002/(ISSN)1099-1085 |
| [29] |
张华, 胡鸿钧, 晁爱敏, 等. 浙江紧水滩水库浮游植物群落结构季节变化特征[J]. 生态学报, 2013, 33(3): 944-956. Zhang H, Hu H J, Chao A M, et al. Seasonal changes of phytoplankton community structure in Jinshuitan Reservoir, Zhejiang, China[J]. Acta Ecologica Sinica, 2013, 33(3): 944-956. |
| [30] | Xia N, Xia X H, Liu T, et al. Characteristics of bacterial community in the water and surface sediment of the Yellow River, China, the largest turbid river in the world[J]. Journal of Soils and Sediments, 2014, 14(11): 1894-1904. DOI:10.1007/s11368-014-0974-5 |
| [31] |
洪璇, 洪有为, 陈仲巍, 等. 九龙江河口区nirS型反硝化细菌多样性及系统发育学分析[J]. 微生物学通报, 2015, 42(9): 1639-1650. Hong X, Hong Y W, Chen Z W, et al. Phylogenetic diversity of nirS-type denitrifying bacteria in Jiulong River estuary[J]. Microbiology China, 2015, 42(9): 1639-1650. |
 2019, Vol. 40
2019, Vol. 40


