2. 中国科学院生态环境研究中心, 北京 100085;
3. 西安工程大学化学与环境工程学院, 西安 710048
2. Research Center for Eco-Environmental Sciences, Chinese Academy of Sciences, Beijing 100085, China;
3. School of Environmental and Chemical Engineering, Xi'an Polytechnic University, Xi'an 710048, China
在我国革兰阳性菌引起的医院内感染率中, 肠球菌是排名第二的重要院内感染条件致病菌, 可引起尿路感染、皮肤软组织感染以及危及生命的腹腔感染、败血症、心内膜炎和脑内膜炎等[1~3].此外, 人们在养殖业中动物来源分离出的肠球菌对多种抗生素具有高水平耐药性[4, 5].肠球菌的耐药性和致病性污染对人类健康和养殖业经济收入都有重要影响作用, 因此控制肠球菌耐药性和致病性在环境中传播有重要意义.
城市污水和污泥中含有大量的肠球菌, 人们已经在城市污水和污泥中分离的肠球菌检测出其携带了多种抗生素抗性基因(antibiotic resistance genes, ARGs)及毒力基因(virulence genes, VGs)[6~8].常见的肠球菌毒力因子主要有表面蛋白(surface adhesions, esp)、溶血素(cytolysin, cylA)、蛋白明胶酶(gelatinase, gelE)、透明质酸酶(hyalronidase, hyl)等.已有研究表明污泥厌氧消化过程可能会导致肠球菌携带的部分ARGs阳性率增加, 同时耐药性增强[9].虽然病原菌携带的VGs常与耐药基因之间存在某些联系, 且VGs可影响病原菌的耐药性[10, 11], 然而在污泥处理过程中肠球菌的ARGs及VGs之间的具体关系仍未明确.
热水解-厌氧消化是近年来新兴的城市污泥强化厌氧消化处理工艺, 但已有研究缺乏针对城市污泥强化厌氧消化处理过程中肠球菌的ARGs与VGs的研究, 尤其对携带ARGs、VGs的肠球菌对其耐药性的影响尚未明确.因此, 本研究考察热水解污泥在中温/高温厌氧消化过程中肠球菌的耐药表型、基因型及携带的VGs变化规律, 探讨ARGs和VGs在肠球菌中, 及其肠球菌耐药性之间的共现关系(co-occurrence), 以期为今后控制城市污泥处理过程中肠球菌的耐药性和致病性传播提供科学依据.
1 材料与方法 1.1 实验设备及样品采集以某城市再生水厂热水解-厌氧消化(40℃)污泥处理工艺的热水解污泥作为本研究厌氧消化进泥, 以两个有效容积分别为200 L和800 L的完全混合式厌氧消化罐, 进行40℃中温和55℃高温厌氧消化.反应罐每天进泥1次, 中温厌氧罐进泥10 kg·d-1, 高温厌氧罐进泥27 kg·d-1, 进泥的同时排出等体积消化污泥, 维持两个厌氧罐HRT均为20 d.在厌氧罐稳定运行40 d后, 分别取中温/高温厌氧进泥及对应的厌氧出泥用于检测分析, 每隔5 d取样一次, 各取样3次.高温厌氧启动晚于中温厌氧:中温厌氧稳定期取样时间为2018年1月(冬季), 高温厌氧稳定期取样时间为2018年6月(夏季).
1.2 肠球菌的筛选与计数肠球菌采用滤膜法进行筛选计数[12], 将污泥样品振荡均匀后, 用PBS梯度稀释菌液, 每个泥样选择3梯度3平行稀释样, 经过0.45 μm醋酸纤维滤膜过滤, 将滤膜以截留面朝上贴在肠球菌滤膜琼脂平板培养基上, 37℃下恒温培养48~60 h后计数.根据标准方法[13]检测污泥总固体(TS), 折算出单位重量污泥中的肠球菌.采用胆汁七叶苷方法进行肠球菌鉴定:用灭菌牙签将肠球菌滤膜琼脂培养基上光滑饱满的粉红至紫红色的单菌落挑至Muller-Hinton (Oxoid, UK)肉汤液体培养基中, 37℃下150 r·min-1振荡培养24 h; 将富集的菌液滴在胆汁七叶苷(Oxoid, UK)琼脂平板培养基上, 37℃下培养24 h, 滴菌液处显深棕色者为肠球菌.相应肠球菌菌液4℃保存, 用于耐药表型与基因型的分析.
1.3 肠球菌耐药表型分析采用美国临床和实验室标准协会(CLSI)推荐的Kirby-Bauer纸片法[14], 通过药敏实验进行肠球菌的耐药表型分析.用无菌生理盐水调节待检菌株菌液的浊度至0.5麦氏浊度后, 用棉签蘸取菌液均匀涂布于Muller-Hinton琼脂平板上, 贴上螺旋霉素(SP, 30 μg)、阿奇霉素(AZM, 15 μg)和四环素(TE, 30 μg)这3种抗生素的药敏纸片(Oxoid, UK), 将平板倒置于生化培养箱中37℃下培养24 h, 以ATCC29212菌株为质量控制标准菌株, 根据抑菌圈直径及CLSI标准判断菌株的耐药情况.
1.4 抗生素抗性基因和毒力基因的检测采用菌液PCR的方法检测肠球菌携带的四环素类抗性基因tetA、tetM和tetX, 大环内酯类抗性基因ermB、ermF和mefA/E, 以及转移元件转座子Tn916/1545, 4种肠球菌毒力基因gelE(编码明胶酶)、esp(编码表面蛋白)、hyl(编码透明质酸酶)、clyA(编码溶血素).各基因引物信息见表 1所示.菌液PCR采用25 μL反应体系:12.5 μL Taq PCR Master Mix, 0.5 μL引物(F), 0.5 μL引物(R), 菌液1μL, 加ddH2O至25μL.反应程序为:预变性95℃, 10 min; 变性95℃, 30 s; 退火30 s; 72℃延伸, 1 min; 进行34个循环; 72℃后延伸, 5 min; 使用无菌水做阴性对照.PCR产物以1.5%的琼脂凝胶电泳分析.
|
|
表 1 ARGs、转移元件和VGs所用引物序列 Table 1 Primer information for ARGs, mobile elements, and VGs |
1.5 数据分析
采用IBM SPSS Statistics 25.0软件对中温、高温厌氧消化前后肠球菌的耐药率、ARGs及VGs检出率进行卡方检验, 以确定各检出率变化的显著性(P < 0.05代表有显著性差异, P < 0.01代表非常显著差异).
2 结果与讨论 2.1 肠球菌的削减本研究的厌氧进泥为某再生水厂现场的热水解污泥, 虽然城市污水厂的污泥经过165℃, 6×105 Pa的热水解处理, 但热水解污泥中(以TS计)仍含有1.97×107~2.17×107CFU·g-1的肠球菌(图 1).中温和高温厌氧消化均可以有效削减肠球菌:在中温厌氧消化后降低为6.98×106 CFU·g-1, 削减了35.42%, 降低1个数量级; 在高温厌氧消化后降低至3.44×105CFU·g-1, 削减了98.41%, 降低了2个数量级, 肠球菌削减效果更佳.刘和等[20]的研究结果也表明高温厌氧消化比中温厌氧消化有更好的肠球菌削减效果.
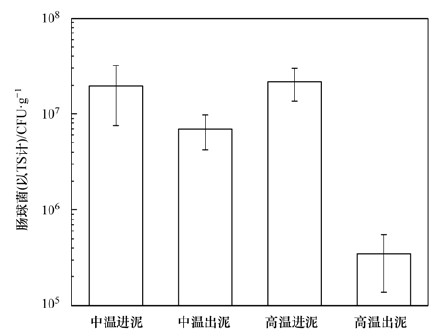
|
图 1 厌氧消化过程肠球菌的变化 Fig. 1 Variation in the abundance of enterococci during anaerobic digestion |
本研究在中温/高温厌氧进泥和出泥中共筛选出217株肠球菌.肠球菌在中温、高温厌氧消化过程对四环素(TE), 以及大环内酯类的螺旋霉素(SP)和阿奇霉素(AZM)的耐药表型变化如表 2所示.由于中温和高温厌氧启动时间不同, 稳定运行后的取样季节不同, 即中温厌氧取样时间为冬季(1月), 高温厌氧为夏季(6月), 虽然进泥中的肠球菌计数相似, 且耐药表型有所差异:厌氧进泥即热水解污泥中, 肠球菌对四环素和螺旋霉素的耐药率冬季高于夏季(P < 0.01和P>0.05), 但中温和高温厌氧进泥中的肠球菌对阿奇霉素的耐药率均显著高于其他两种(P < 0.01), 分别为67.14%和97.67%.
|
|
表 2 厌氧消化过程中肠球菌耐药率的变化 Table 2 Variations in the antibiotic resistance rates of enterococci during anaerobic digestion |
中温、高温厌氧消化对肠球菌的耐药率表现出完全不同的作用趋势:高温厌氧消化后, 四环素耐药率升高了2.43%(P>0.05), 对螺旋霉素的耐药率降低了2.33%(P>0.05), 而对阿奇霉素的耐药率显著降低了16.72%(P < 0.05);而中温厌氧消化后, 对四环素的耐药性显著下降了24.94%(P < 0.05), 对螺旋霉素耐药率增加1.47%(P>0.05), 对阿奇霉素的耐药率则显著升高23.18%(P < 0.01).整体来看, 高温厌氧消化后肠球菌的耐药性增加并不显著, 而显著降低了对阿奇霉素的耐药率, 说明高温厌氧相比中温厌氧对肠球菌的耐药性具有更好的控制及削减效果.中温、高温厌氧消化后, 肠球菌对阿奇霉素的耐药率依然显著高于其他两种抗生素(P < 0.01).
此外, 中温厌氧进泥中肠球菌同时耐药四环素与螺旋霉素、四环素与阿奇霉素、螺旋霉素与阿奇霉素、及同时耐药3种抗生素的占比分别为4.29%、20%、7.14%和4.29%.经过中温厌氧消化, 肠球菌对以上抗生素组合的耐药率分别增加0.55%(P>0.05), 降低3.87%(P>0.05), 增加4.15%(P>0.05), 降低4.29%(P>0.05).高温厌氧消化进泥中仅有2.33%的肠球菌对四环素与阿奇霉素同时耐药, 高温厌氧消化后, 肠球菌同时对四环素和阿奇霉素耐药升高了2.34%(P>0.05).由此可见, 中温、高温厌氧消化可促使肠球菌对部分种类抗生素双/多重耐药性增加, 但其影响并不显著.
2.3 肠球菌耐药基因型的变化本研究筛选出的217株肠球菌耐药基因型变化如图 2所示.所有污泥样品中的肠球菌均未检出tetX.中温厌氧进泥与出泥肠球菌中均未检出tetA; 进泥中tetM的检出率高达78.57%, 显著高于其他ARGs(P < 0.01).中温厌氧消化可显著降低tetM的检出率至30.65%(P < 0.01), 这一现象与中温厌氧后肠球菌对四环素的耐药率降低结果一致.对于高温厌氧消化, tetA检出率由18.61%降低至11.91%(P>0.05);而tetM检出率由11.63%升高至26.19%(P>0.05), 与高温厌氧后肠球菌的四环素耐药率增加结果一致.以上结果表明, tetM可能是引起中温、高温厌氧消化后肠球菌对四环素的耐药率变化的重要原因.此外, 中温厌氧消化后, 携带tetA或tetM且对四环素耐药的肠球菌菌株比例从56.36%增加到57.89%(P>0.05);高温厌氧进泥中携带tetA或tetM的肠球菌并未表达对四环素的耐药性, 即耐药率为0, 但在高温厌氧消化后, 携带tetA或tetM的肠球菌对四环素的耐药率增加到14.29%(P>0.05).这一结果表明, 高温厌氧消化可能会导致肠球菌中四环素类抗性基因的表达增强.
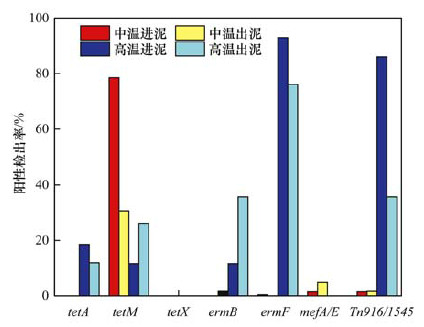
|
图 2 肠球菌耐药基因型的变化 Fig. 2 Variations in the antibiotic resistance genotypes of enterococci |
对于大环内酯类抗性基因, 中温厌氧进泥中的肠球菌ermB、ermF和mefA/E的检出率分别为0、1.43%和1.43%, 出泥肠球菌的ermB检出率升高到1.61%(P>0.05), mefA/E显著升高到4.84%(P < 0.05), 而ermF检出率降低到0(P>0.05).高温厌氧消化进泥、出泥中均未检测出mefA/E, 但进泥中肠球菌ermB、ermF的检出率分别为11.63%、93.02%.高温厌氧消化后ermB检出率显著增长至35.71%(P < 0.01), ermF检出率则显著降低至76.19%(P < 0.05).以上结果表明, 中温、高温厌氧消化均可导致ermB检出率增加, ermF检出率降低.此外, 中温厌氧消化前后, 携带ermB、ermF和mefA/E的肠球菌对螺旋霉素及阿奇霉素的耐药率并无变化; 高温厌氧对螺旋霉素耐药率虽无影响, 但可导致携带ermB、ermF和mefA/E的肠球菌对阿奇霉素的耐药率降低13.51%.
厌氧消化前后肠球菌同时携带ARGs与转座子的检出情况如图 3所示.无论中温、高温厌氧消化, 均可导致同时携带tetM+ermB和tetM+Tn916/1545的肠球菌检出率增加:在中温厌氧后, 分别增加了1.61%(P>0.05)和0.18%(P>0.05);在高温厌氧后分别增加了2.58%(P>0.05)和9.8%(P>0.05).中温厌氧消化进泥和出泥均未检测出同时携带ermB+Tn916/1545和tetM+ermB+Tn916/1545的肠球菌, 而高温厌氧消化则可促使以上基因检出率增加2.54%(P>0.05)和0.11%(P>0.05).转座子Tn916/1545是引起抗性基因水平转移的重要因素, 也是导致细菌多重耐药的重要机制之一[21], 可同时携带tetM和ermB等多种ARGs[9, 22], 从而导致肠球菌对四环素类和大环内酯类抗生素同时耐药.以上结果表明, 虽然厌氧之后ARGs检出率的增加不显著, 但中温、高温厌氧消化都有促使ARGs在肠球菌中发生水平转移的潜在趋势, 从而导致其潜在双重甚至多重耐药性的可能性增加.
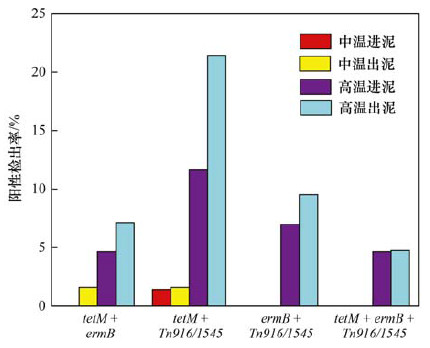
|
图 3 肠球菌中ARGs与Tn916/1545的共现关系 Fig. 3 Co-occurrence of ARGs and Tn916/1545 in enterococci |
在中温/高温厌氧消化进/出污泥中筛选出的217株肠球菌均未检出esp、clyA和hyl.肠球菌中毒力基因gelE检出率的变化如图 4所示.中温厌氧可显著降低肠球菌的gelE检出率, 即从80%降低到54.84%(P < 0.01).高温厌氧则促使gelE检出率由11.63%升高到21.43%(P>0.05).其他研究者也发现从地表水[23, 24]、海鲜[25]和奶酪[26]中分离出的肠球菌具有较高的gelE检出率.
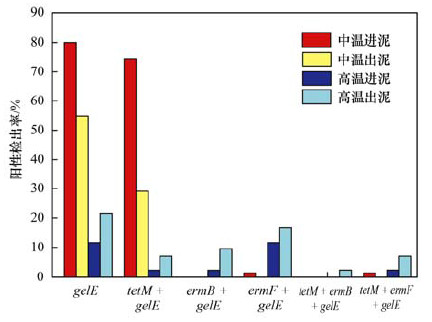
|
图 4 肠球菌中VGs与ARGs的共现关系 Fig. 4 Co-occurrence of VGs and ARGs in enterococci |
肠球菌同时检出ARGs和VGs的比例在污泥厌氧消化过程中的变化趋势如图 4所示.中温厌氧消化进泥的肠球菌同时携带ARGs与VGs的情况有tetM+gelE和ermF+gelE, 且同时携带ermF+gelE组合的菌株也同时为tetM阳性.在中温进泥中74.29%的肠球菌检出tetM+gelE, 即74.29%的肠球菌既有潜在致病性, 又对四环素类抗生素有潜在耐药性.中温厌氧消化可显著削减tetM+gelE检出率到29.03%(P < 0.01);此外, ermF+gelE的检出率也由1.43%降为0.而经过高温厌氧消化肠球菌tetM+gelE的检出率由2.33%增长到7.14%(P < 0.05), ermB+gelE和ermF+gelE的比例也分别升高了7.19%(P>0.05)和5.04%(P>0.05).这个结果表明, 中温厌氧消化有降低肠球菌同时携带ARGs与VGs几率的潜在趋势, 而高温厌氧消化则可能会增加肠球菌同时携带ARGs与VGs的几率, 从而增加其同时具有致病性和耐药性的潜在风险.
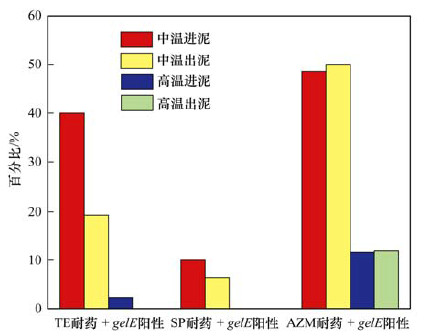
已有研究发现VGs可能促进细菌对抗生素耐药性的增加[11].例如, Duprè等[27]的研究表明esp基因对糖肽类抗生素耐药性有密切关系, 携带esp基因的肠球菌在抗生素选择压力下可导致其对不同抗生素耐药性的增加.因此本研究考察了携带VGs的肠球菌对3种抗生素的耐药性变化(图 5).中温进泥中gelE阳性且对四环素、螺旋霉素、阿奇霉素耐药的肠球菌比例分别为40.0%、10.0%和48.57%;除gelE阳性且对阿奇霉素耐药的肠球菌比例在出泥中升高了1.43%(P>0.05)之外, 既携带gelE且对四环素和阿奇霉素耐药的肠球菌在中温厌氧消化后占比均有所下降, 分别为20.65%(P < 0.01)和3.5%(P>0.05).而高温厌氧消化进泥中gelE阳性且对四环素、螺旋霉素、阿奇霉素耐药的肠球菌比例分别为2.33%、0和11.63%, 出泥中gelE阳性且对阿奇霉素耐药的肠球菌比例升高了0.27%(P>0.05), 携带gelE且对四环素、螺旋霉素耐药的比例均为0.综上所述, gelE阳性且对四环素、螺旋霉素耐药的菌株比例在中温和高温厌氧消化后都实现了削减, 而gelE阳性且对阿奇霉素耐药的菌株比例均有小幅上升但并不显著(P>0.05).
|
图 5 肠球菌中VGs与耐药性的共现关系 Fig. 5 Co-occurrence of VGs in enterococci and the antibiotic resistance of enterococci |
(1) 热水解污泥中仍含有107数量级的肠球菌, 高温厌氧比中温厌氧对肠球菌削减效果更佳.
(2) 热水解污泥中肠球菌对阿奇霉素的耐药率显著高于其螺旋霉素和四环素的耐药率; 高温厌氧比中温厌氧对肠球菌的耐药性具有更好的控制及削减效果.同时, 中温、高温厌氧消化后肠球菌双重耐药几率均有所增加.
(3) 中温、高温厌氧消化均可导致ermB检出率增加, ermF检出率降低, 并可促进ARGs在肠球菌中发生水平转移, 导致其潜在双重甚至多重耐药性增加.高温厌氧可能会促进四环素类抗性基因在肠球菌的表达.
(4) 中温厌氧消化具有降低肠球菌同时携带ARGs与VGs的几率的趋势, 而高温厌氧消化则可能会增加肠球菌同时携带抗性基因与毒力基因的几率.中温和高温厌氧消化都可削减gelE阳性且对四环素、螺旋霉素耐药的肠球菌.
| [1] | Anderson A C, Jonas D, Huber I, et al. Enterococcus faecalis from food, clinical specimens, and oral sites:prevalence of virulence factors in association with biofilm formation[J]. Frontiers in Microbiology, 2016, 6: 1534. |
| [2] | Talebi M, Asghari Moghadam N, Mamooii Z, et al. Antibiotic resistance and biofilm formation of enterococcus faecalis in patient and environmental samples[J]. Jundishapur Journal of Microbiology, 2015, 8(10): e23348. |
| [3] |
张肖肖, 马传玲, 刘海洋, 等. 临床分离肠球菌生物膜的形成能力及毒力基因的相关性研究[J]. 中国抗生素杂志, 2016, 41(11): 869-873. Zhang X X, Ma C L, Liu H Y, et al. The ability of biofilm formation in clinical Enterococcus and their correlation with virulence factors[J]. Chinese Journal of Antibiotics, 2016, 41(11): 869-873. DOI:10.3969/j.issn.1001-8689.2016.11.011 |
| [4] |
张慧, 叶丹娟, 何晴, 等. 人和鸡来源粪肠球菌耐药性以及毒力基因分布研究[J]. 现代预防医学, 2014, 41(14): 2611-2613, 2616. Zhang H, Ye D J, He Q, et al. Study on drug resistance and gene distribution of virulent factors in Enterococcus faecalis from human and chicken[J]. Modern Preventive Medicine, 2014, 41(14): 2611-2613, 2616. |
| [5] |
陈丽颖, 张留君, 秦明明, 等. 不同动物源肠球菌耐药性及毒力基因测定与分析[J]. 中国人兽共患病学报, 2017, 33(11): 984-990. Chen L Y, Zhang L J, Qin M M, et al. Drug resistance spectrum and virulence gene distribution in Enterococcus isolates of animal origin[J]. Chinese Journal of Zoonoses, 2017, 33(11): 984-990. DOI:10.3969/j.issn.1002-2694.2017.11.007 |
| [6] | Lanthier M, Scott A, Lapen D R, et al. Frequency of virulence genes and antibiotic resistances in Enterococcus spp. isolates from wastewater and feces of domesticated mammals and birds, and wildlife[J]. Canadian Journal of Microbiology, 2010, 56(9): 715-729. DOI:10.1139/W10-046 |
| [7] | Rad P K, Eslami G. Distribution of gelE, esp and hyl virulence genes among enteroccus facium isolated from urban sewage samples intehran[J]. Iranian Journal of Public Health, 2014(43): 122. |
| [8] | Da Costa P M, Vaz-Pires P, Bernardo F. Antimicrobial resistance in Enterococcus spp. isolated in inflow, effluent and sludge from municipal sewage water treatment plants[J]. Water Research, 2006, 40(8): 1735-1740. DOI:10.1016/j.watres.2006.02.025 |
| [9] | Tong J, Lu X T, Zhang J Y, et al. Occurrence of antibiotic resistance genes and mobile genetic elements in enterococci and genomic DNA during anaerobic digestion of pharmaceutical waste sludge with different pretreatments[J]. Bioresource Technology, 2017, 235: 316-324. DOI:10.1016/j.biortech.2017.03.104 |
| [10] | Da Silva G J, Mendonça N. Association between antimicrobial resistance and virulence in Escherichia coli[J]. Virulence, 2012, 3(1): 18-28. DOI:10.4161/viru.3.1.18382 |
| [11] | Soto S M. Relationship between virulence and antimicrobial resistance in bacteria[J]. Reviews in Medical Microbiology, 2009, 20(4): 84-90. DOI:10.1097/MRM.0b013e3283342903 |
| [12] | Da Silva M F, Tiago I, Veríssimo A, et al. Antibiotic resistance of enterococci and related bacteria in an urban wastewater treatment plant[J]. FEMS Microbiology Ecology, 2006, 55(2): 322-329. |
| [13] | 国家环境保护总局. 水和废水监测分析方法[M]. (第四版).北京: 中国环境科学出版社, 2002. |
| [14] | M100-S24, Performance standards for antimicrobial susceptibility testing; Twenty-fourth informational supplement[S]. |
| [15] | Zhang J Y, Chen M X, Sui Q W, et al. Fate of antibiotic resistance genes and its drivers during anaerobic co-digestion of food waste and sewage sludge based on microwave pretreatment[J]. Bioresource Technology, 2016, 217: 28-36. DOI:10.1016/j.biortech.2016.02.140 |
| [16] | Tong J, Liu J B, Zheng X, et al. Fate of antibiotic resistance bacteria and genes during enhanced anaerobic digestion of sewage sludge by microwave pretreatment[J]. Bioresource Technology, 2016, 217: 37-43. DOI:10.1016/j.biortech.2016.02.130 |
| [17] | Kwon K H, Hwang S Y, Moon B Y, et al. Occurrence of antimicrobial resistance and virulence genes, and distribution of enterococcal clonal complex 17 from animals and human beings in Korea[J]. Journal of Veterinary Diagnostic Investigation, 2012, 24(5): 924-931. DOI:10.1177/1040638712455634 |
| [18] | Meena B, Anburajan L, Sathish T, et al. Enterococcus species diversity and molecular characterization of biomarker genes in Enterococcus faecalis in Port Blair Bay, Andaman and Nicobar Islands, India[J]. Marine Pollution Bulletin, 2015, 94(1-2): 217-227. DOI:10.1016/j.marpolbul.2015.02.027 |
| [19] | Vankerckhoven V, Van Autgaerden T, Vael C, et al. Development of a multiplex PCR for the detection of asa1, gelE, cylA, esp, and hyl genes in enterococci and survey for virulence determinants among european hospital isolates of Enterococcus faecium[J]. Journal of Clinical Microbiology, 2004, 42(10): 4473-4479. DOI:10.1128/JCM.42.10.4473-4479.2004 |
| [20] | 刘和, 符波, 姜谦, 等.城市剩余污泥厌氧消化对病原微生物的杀灭机理研究[A].见: 第十五次全国环境微生物学学术研讨会论文集[C].大连: 中国微生物学会, 2012. http://www.wanfangdata.com.cn/details/detail.do?_type=conference&id=7935558 |
| [21] | Roberts A P, Mullany P. Tn916-like genetic elements:a diverse group of modular mobile elements conferring antibiotic resistance[J]. FEMS Microbiology Reviews, 2011, 35(5): 856-871. DOI:10.1111/j.1574-6976.2011.00283.x |
| [22] | Hegstad K, Mikalsen T, Coque T M, et al. Mobile genetic elements and their contribution to the emergence of antimicrobial resistant Enterococcus faecalis and Enterococcus faecium[J]. Clinical Microbiology and Infection, 2010, 16(6): 541-554. DOI:10.1111/j.1469-0691.2010.03226.x |
| [23] | Molale L G, Bezuidenhout C C. Antibiotic resistance, efflux pump genes and virulence determinants in Enterococcus spp. from surface water systems[J]. Environmental Science and Pollution Research, 2016, 23(21): 21501-21510. |
| [24] | Sidhu J P S, Skelly E, Hodgers L, et al. Prevalence of Enterococcus species and their virulence genes in fresh water prior to and after storm events[J]. Environmental Science & Technology, 2014, 48(5): 2979-2988. |
| [25] | De Souza Carneiro C, Evangelista-Barreto N S, Da Silveira-Oliveira C S, et al. Antagonistic activity, antimicrobial susceptibility and potential virulence factors of Enterococcus faecalis[J]. Journal of Life Sciences, 2015, 9: 318-326. |
| [26] | Hammad A M, Hassan H A, Shimamoto T. Prevalence, antibiotic resistance and virulence of Enterococcus spp. in Egyptian fresh raw milk cheese[J]. Food Control, 2015, 20: 815-820. |
| [27] | Duprè I, Zanetti S, Schito A M, et al. Incidence of virulence determinants in clinical Enterococcus faecium and Enterococcus faecalis isolates collected in Sardinia (Italy)[J]. Journal of Medical Microbiology, 2003, 52: 491-498. DOI:10.1099/jmm.0.05038-0 |
 2019, Vol. 40
2019, Vol. 40


