2. 日本东北大学工学部, 仙台市 980-8579
2. Department of Civil and Environmental Engineering, Graduate School of Engineering, Tohoku University, Sendai 980-8579, Japan
我国已成为世界最大的制药生产与出口国, 制药发酵生产过程中产生大量的抗生素菌渣, 据不完全统计, 我国每年制药菌渣的产生量约为130万t[1, 2].现有的菌渣减量化处置技术如焚烧、填埋以及制造肥料和饲料等普遍存在成本高、二次污染、残留药物积累等负面效应和隐患.如何实现制药菌渣合理有效利用与安全处置是我国制药工业亟待解决的难题[3~5].厌氧消化适合处理有机质含量高的底物, 而制药菌渣含有高蛋白、高能量的特点, 具有较高的沼气生产潜能, 是解决菌渣资源化合理有效利用的途径之一[6~8]. Zhong等[9]和Zhang等[10]利用头孢菌渣厌氧消化的研究表明, 经过预处理的抗生素菌渣厌氧消化取得了较好的消化效果.孙效新等[11]对多种抗生素菌渣厌氧生物法处理的研究证明:青霉素、链霉素、麦迪霉素菌渣液无论是单独处理还是混合处理都具有较高的产气潜能.何品晶等的研究表明[12]:含固率为3%、接种比为3的工况中, 林可霉素菌渣的挥发性固体(volatile solid, VS)累积产甲烷达到106 mL·g-1.
青霉素、头孢类抗生素菌渣含有机硫, 在厌氧发酵过程中有机硫很容易分解为无机SO42-和S2-[13, 14].高负荷的SO42-会引起厌氧发酵过程硫酸盐还原菌(SRB)与甲烷生成菌(MPA)围绕基质的竞争, 导致甲烷发酵过程受阻, 产气量下降[15, 16].掌握SO42-还原竞争产生的关键条件对于避免其对产甲烷的抑制作用十分必要.
发酵基质中COD/SO42-是影响甲烷生成与硫酸盐还原过程竞争的主要因素[17~21].影响过程竞争的COD/SO42-范围变化很大. Li等[22]针对磺胺类制药废水厌氧发酵的研究发现, COD/SO42-减小为1.5时, 对产甲烷产生轻微抑制. Kiyuna等的研究表明[21], COD/SO42-比小于7.5时甲烷产生量相比较于COD/SO42-比为12的处理下降了35%, 而Rinzema等[23]发现COD/SO42-比小于10就会导致产甲烷过程的失败. Ren等的研究发现[24], COD/SO42-比小于3时, 反应器内的电子主要为硫酸盐还原过程利用.当SO42-浓度过高时, 不仅会导致产甲烷效果下降, 而且也会导致SO42-去除效果下降, 这是因为高浓度S2-不仅对甲烷菌具有抑制作用[25~28], 而且对硫酸盐还原菌也具有抑制作用[29].
COD/SO42-对产甲烷过程的抑制阈值大小不仅与厌氧消化工艺类型, 操作条件等有关系, 还与厌氧消化基质类型有关, 已有研究针对的都是合成废水, 其共同特点为基质成分较为简单, 并且大都是以SO42-为唯一S源.青霉素、头孢类抗生素菌渣是实际废弃物, 其基质成分十分复杂, 所含S源主要为有机硫, 对于有机硫存在条件下SO42-对厌氧消化过程的影响规律还不清楚.另外, 厌氧发酵是由物质的水解、酸化、甲烷化、三阶段、多菌群参与的微生物共代谢过程.了解厌氧消化过程物质分解特性是提高厌氧消化效率的重要手段[30, 31].青霉素类菌渣富含发酵菌丝体, 被微生物的细胞壁包裹, 难以被微生物直接利用, 水解酸化过程成为其厌氧发酵的限制性步骤, 为了提高厌氧发酵的效率, 对产酸相物质分解特性的讨论是菌渣厌氧消化需要解决的另一个问题.
本研究以青霉素菌渣为基质, 采用批试实验研究不同COD/SO42-对青霉素菌渣厌氧发酵产甲烷的影响, 以把握SO42-对青霉素菌渣厌氧消化的影响规律, 并在此基础上探讨其厌氧消化酸生成相物质利用特性, 以期为具有复杂组成的含有机S源抗生素菌渣厌氧发酵工程实践提供理论指导.
1 材料与方法 1.1 基质实验所用基质来自某制药厂青霉素生产车间储渣池, 利用高速离心粉碎机将青霉素菌渣粉碎, 加水调制成总固体(total solid, TS)为10%的均浆状基质.
1.2 接种污泥实验所用接种污泥取自实验室连续稳定运行150 d的中温食品废物厌氧发酵反应器.采取接种污泥时连续实验连续3 d停止进基质, 以便消耗发酵液中的剩余COD.基质和接种污泥的理化特性如表 1所示.
|
|
表 1 基质和接种污泥理化特性 Table 1 Physical and chemical characteristics of the substrate and sludge |
1.3 批试实验
通过添加Na2SO4于基质中, 配制COD/SO42-质量比分别为: 0.25:1、0.5:1、1.5:1、3:1、6:1、12:1的6组TS为10%的基质.批试实验采用容量100 mL血清瓶, 实验有效容积为60 mL.基质与接种污泥按照1:3比例混合, 其中空白实验仅添加接种污泥, 立即充氮气约1 min, 排去瓶中顶空部分氧气后, 迅速塞上橡胶塞, 扣上铝盖, 用封口钳封紧瓶口, 放入35℃恒温水浴振荡槽中振荡, 定期测定瓶中产气量, 以及气体含量.每组实验3个平行, 1个平行用于气体测量, 另外2个平行样用于发酵中间取样, 分别在发酵第2、4、6、8、10、12、14、16、18 d采用注射针取样, 测定发酵液挥发性有机酸(VFAs), 总的、以及溶解态COD、碳水化合物、蛋白质浓度.实验反应完全结束后, 开启瓶盖, 测定发酵液的TS、pH、COD、碳水化合物、蛋白质、总S、S2-、SO42-浓度.
1.4 分析项目与方法pH、TS、VS、悬浮固体(suspended solid, SS)、挥发性悬浮固体(volatile suspended solid, VSS)、COD、S2-采用APHA, 1995标准方法进行; SO42-采用重量法测定(GB 11899-89);总S采用元素分析仪测定(Vario ELⅢ); 有机S=总S-[SO42-]-[S2-]; 碳水化合物采用苯酚-硫酸法; 蛋白质采用Lowy法; 溶解态COD、蛋白质以及碳水化合物的测定方法为:发酵混合液在15 000 r·min-1, 4℃高速冷冻离心机中离心15 min, 取上清液, 过0.45 μm滤膜, 采用上述方法分别测定:溶解态COD、蛋白质以及碳水化合物.颗粒态浓度=总浓度-溶解态浓度.
挥发性有机酸(volatile organic acids, VFAs)采用气相色谱测定(岛津GC-2014, stablilwax-DA色谱柱, 30 m×0.32 mm×0.50 μm, FID检测器); CH4、H2S含量采用气相色谱法测定(岛津GC-2014, P-Q填充柱, 2 m×2 mm, TCD检测器); 产气量利用排气法测量.
1.5 累积曲线动力学参数分析为了进一步探讨COD/SO42-对青霉素菌渣厌氧发酵产气潜能影响的机制, 利用修正的Gompertz公式[式(1)][32], 对不同COD/SO42-青霉素菌渣厌氧发酵累积产气量曲线进行动力学参数分析.
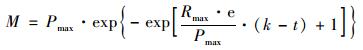
|
(1) |
式中, M为累积甲烷产量(mL·g-1, 以TS计); Pmax为最大产甲烷势(mL·g-1, 以TS计); Rmax为最大产甲烷速率[mL·(g·d)-1, 以TS计]; k为滞后时间(d); t为发酵时间(d).
采用Origin 8.0非线性拟合获得厌氧发酵累积产气量曲线各反应动力学参数值.
1.6 甲烷生成量计算相邻两次测定间隔产生的甲烷体积按式(2)计算:
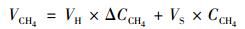
|
(2) |
式中, VCH4为相邻两次测定间隔产生甲烷体积(mL); VH血清瓶顶空体积, 为40 mL; ΔCCH4指相邻两次测定间隔甲烷浓度的变化(%); VS相邻两次测定间隔生成的生物气量(mL); CCH4生物气甲烷含量(%).累积甲烷生成量=各处理累积甲烷产量-接种污泥累积甲烷产量
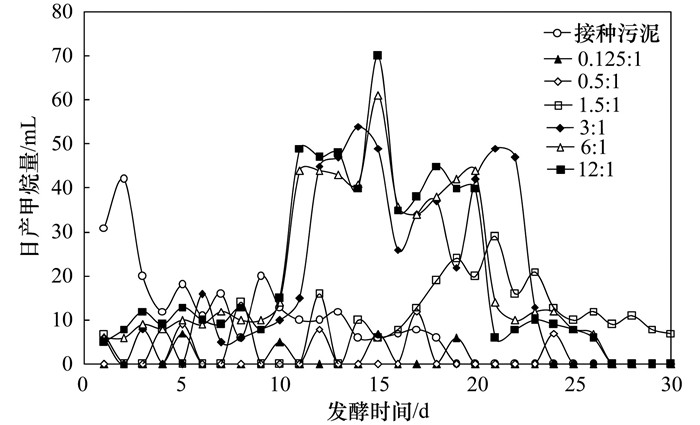
2 结果与讨论 2.1 COD/SO42-对青霉素菌渣厌氧发酵产气潜能的影响 2.1.1 对日产气量的影响图 1为不同COD/SO42-比青霉素菌渣厌氧发酵产甲烷速率变化.从中可知, 在发酵前10 d, 各COD/SO42-比青霉素菌渣厌氧发酵日产甲烷量均比单纯接种污泥发酵低, 各发酵产甲烷速率低的原因可能是由于抗菌素对厌氧污泥的毒性抑制作用所致[33, 34].发酵10 d后, COD/SO42-为3:1、6:1、12:1的发酵日产甲烷速率迅速上升, 表明微生物经过较长时间的适应性驯化, 产气能力逐步恢复[35], 但是COD/SO42-为0.125:1、0.5:1和1.5:1的发酵日产甲烷速率仍旧维持在很低水平, 产生这种现象可能的原因是:在较高的SO42-环境条件下产甲烷菌(MPB)与硫酸盐还原菌(SRB)围绕基质COD的竞争性利用导致SRB菌为优势菌群, MPB菌的活性受到抑制, 产气速率很低. Annachhatre等[36]针对糖蜜废水的研究也发现降低COD:S的比例, 增强了MPB与SRB之间的竞争. COD/SO42-≥3时, 发酵25 d过后最大日产甲烷速率不超过10 mL·d-1. COD/SO42-≤0.5时, 日产甲烷速率在18 d左右就几乎降为0.
|
图 1 不同COD/SO42-比青霉素菌渣发酵日产甲烷量变化 Fig. 1 Effect of the COD/SO42- ratio on daily methane production from the digestion of penicillin bacterial residues |
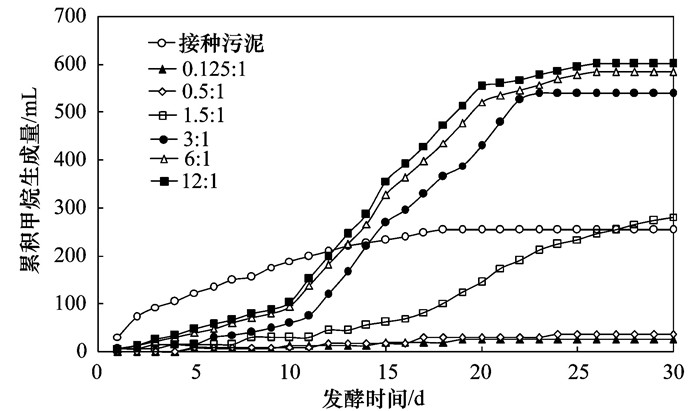
图 2为不同COD/SO42-青霉素菌渣发酵累积产甲烷量变化.从中可知, COD/SO42-≥3时, 累积产甲烷量较高, 扣除污泥背景值后分别为(以TS计): 208、235和246 mL·g-1. COD/SO42-为0.125和0.5时, 累积产甲烷量分别仅为25 mL和35.7 mL(未扣除接种污泥背景值), 远低于接种污泥值232 mL, 说明发酵过程受到完全抑制, 极高SO42-条件下发酵产生的H2S和丙酸对MPB、SRB、也可能两者共同的毒性抑制是其主要原因之一[37].发酵30 d时, COD/SO42-为1.5的处理, 累积产气量为281 mL, 稍高于接种污泥的产甲烷值, 但是远低于COD/SO42-≥3时的累积产甲烷量, 计算表明造成至少49%的产甲烷的抑制. Li等[22]针对磺胺类制药废水的研究也表明COD/SO42-为1.5时会对甲烷菌产生较强的抑制.
|
图 2 不同COD/SO42-比青霉素菌渣发酵累积产甲烷量变化 Fig. 2 Effect of the COD/SO42- ratio on the cumulative methane production from the digestion of penicillin bacterial residues |
Zwietering等[38]报道修正的Gompertz方程是最为适合描述生物产气过程的模型, 对COD/SO42-为1.5、3、6和12的产甲烷累积曲线利用修正的Gompertz方程进行动力学分析, 结果如表 2所示.从中可知, 随着COD比例的增加, 最大产甲烷势和产甲烷速率都增加. COD/SO42-≥3时, 最大产甲烷势分别为(以TS计) 211.52、238.34和243.66 mL·g-1, 模拟结果和实验数据较好吻合.最大反应速率Rmax (以TS计)为0.01 mL·(g·d)-1, Rmax较小的原因是由于抗生素对甲烷发酵的毒性抑制导致的发酵滞后.各COD/SO42-都表现为10 d以上的发酵滞后期k, 发酵微生物发生了适应性驯化, 发酵潜能得以逐步激发, 这较Sari等[35]处理含双氯酚酸废水70 d左右的驯化时间短许多, 可能是由于接种污泥VS浓度较高(表 1), 对抗生素毒性具有较强缓冲所致.在较高SO42-条件下(COD/SO42-为1.5) SRB围绕基质竞争产生的部分抑制导致累积产甲烷势很小[(32.67±1.50)mL·g-1, 以TS计)], 在抗生素毒性抑制耦合基质竞争抑制状态下, 微生物的适应性驯化期也比其他处理更长(15 d).
|
|
表 2 不同COD/SO42-青霉素菌渣厌氧发酵累积产甲烷曲线动力学参数值1) Table 2 Effect of the COD/SO42- ratio on the kinetic parameters for digestion of penicillin bacterial residues obtained from the modified Gompertz model |
2.2 COD/SO42-对青霉素菌渣厌氧发酵物质降解及COD、S平衡的影响 2.2.1 对物质降解效率的影响
在高浓度SO42-条件下, SRB菌相比MPB菌对于乙酸和H2的争夺更具有优势, 导致物质转化效率降低[39].另一方面, 抗生素对厌氧发酵的毒性作用, 也会导致发酵效果的降低.因而对SO42-基质竞争性抑制耦合抗生素毒性抑制风险下的青霉素菌渣厌氧发酵物质转化关系进行探讨. 表 3为不同COD/SO42-青霉素菌渣厌氧发酵物质降解效率.由图 2可知COD/SO42-≤0.5时, 产甲烷受到完全抑制, 结合表 3有机物降解效率低下的结果, 证明此条件下发酵失败. COD/SO42-≥3时, VS、COD、蛋白质、碳水化物的降解效率为60%~90%, 其中COD去除率为75%~84%. SO42-负荷的提高对各类有机物去除效率没有显著影响, 但是SO42-去除率随负荷的提高而减小, 为89.12%~36.03%, 各有机S浓度由1 780 mg·L-1减小为316 mg·L-1左右, 说明水解、酸化过程基质中有机S分解为SO42-的比例较高, 当进一步提高SO42-负荷为COD/SO42-≤1.5时, 由于SRB基质竞争性利用影响, 有机物降解效率迅速降低, 仅为10%~17%, 其中COD去除率为11%~17%. SO42-去除率减小为1.74%~21.85%, 没有发生由于有机S降解而导致的SO42-的积累现象, 发酵液中S2-的浓度由COD/SO42-为12时的1 377 mg·L-1增加为COD/SO42-为0.125时的2 216 mg·L-1, 说明SO42-去除率的低下可能是由于较高的S2-对SRB的抑制作用导致. Barrera等[37]在对废醪液的研究中也证明了高浓度SO42-厌氧消化过程中产生的S2-会对硫酸盐还原过程产生抑制作用.
|
|
表 3 不同COD/SO42-青霉素菌渣厌氧发酵物质降解效率/% Table 3 Effect of the COD/SO42- ratio on the removal rates of organics during the digestion of penicillin bacterial residues/% |
2.2.2 对COD以及S物质平衡的影响
建立以某种物质为基准的化学计量关系可以很方便地表达厌氧发酵过程复杂的物质转化规律.各比例最初的COD投加浓度为102g·L-1, 按照各COD/SO42-比例确定各自的Na2SO42-添加量.依据发酵过程中COD和SO42-的物质转化化学计量关系值(CH4-COD=CH4 mol×64 g; H2S-COD=H2Sgasmol×64 g; S2--COD=Saq2- mol×64 g)[40], 对COD和S的物质平衡进行计算, 结果如图 3所示.当COD/SO42-≥3时, 发酵液中只残留14%~25%的COD未能转化, 75%~86%的COD被厌氧分解, 其中超过71%的COD转化为了甲烷, 由于CO2的COD当量数为0, 所以转化为CO2的COD为0, 未转化为甲烷的COD被硫酸盐还原菌利用来转化SO42-为气体中H2S和溶液中S2-了. COD/SO42-减小到1.5时, COD的甲烷转化率减小为9.1%, COD/SO42-进一步减小到0.5时, 甲烷转化率仅为0.9%, 残留COD相应地增加为83.2%~89.2%, 但是不同比例下用于还原SO42-的COD却没有太大的变化, 基本上在5.0%~9.0%之间[图 3(a)], 说明MPB菌比SRB菌对S2-的毒性更为敏感.
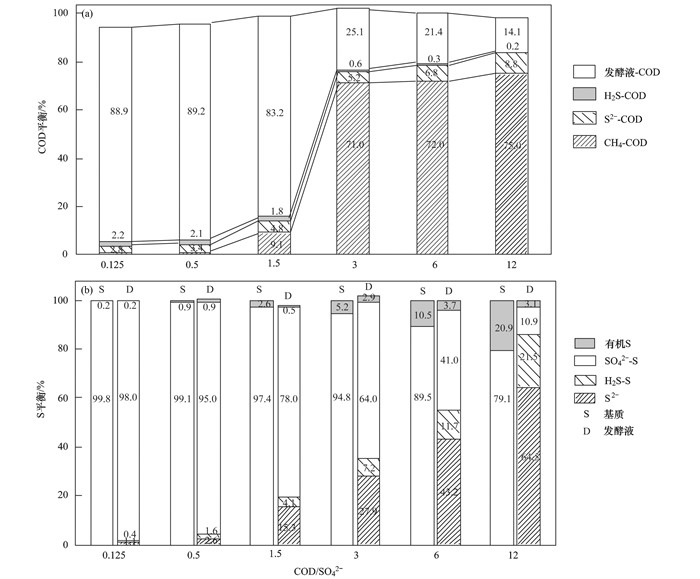
|
图 3 不同COD/SO42-比青霉素菌渣厌氧发酵COD和S的物质平衡 Fig. 3 Effect of the COD/SO42- ratio on the COD and S balances |
图 3(b)为不同COD/SO42-条件S的物质平衡关系.随着SO42-负荷的增加, 残留在发酵液中SO42-比例由10.9%~98%逐步增大, 相应地转化为S2-和H2S的SO42-由86%~1.5%迅速减少.还原的SO42-大部分以S2-的形式存在于发酵液中, 少部分以H2S的形式存在于生物气中, 但其在还原物中的比例随SO42-负荷的增加而增加. COD/SO42-﹤3时, 发酵前后有机S几乎未发生转化, 生成的S2-和H2S全部由SO42-还原转化而来, 当COD/SO42-≥3时, 水解、酸化过程中82%~85%的有机S发生转化, 有机S向SO42-的转化会导致SO42-的积累, 但由表 3可知: SO42-降解率由36.03%提高为89.12%, 说明积累的SO42-在活性逐渐增强的SRB菌的作用下高效去除, 生成的S2-和H2S是SO42-还原和有机S转化共同作用的结果.并且, 当COD/SO42-≥6时, SO42-去除率可达到58.56%以上, 而发酵后残留有机S为总S的3.1%~3.7%, 高于COD/SO42-≤1.5时相应值, 这是由于前者基质中有机S占总S比例是后者10~20倍所致.
2.3 青霉素菌渣厌氧发酵酸生成相物质利用特性 2.3.1 青霉素菌渣厌氧发酵过程VFAs的变化由图 2及表 2知, COD/SO42-≥6时抗生素菌渣厌氧发酵具有较高产气潜能, 所以针对COD/SO42-为6时抗生素菌渣厌氧发酵酸生成相物质利用特性进行探讨.由图 4可知, 发酵2~ 14 d, VFAs浓度由182 mg·L-1逐渐增加为1 634 mg·L-1, 其中乙酸浓度由120mg·L-1逐渐增加为932 mg·L-1, 为主要存在的有机酸形式, 14 d之后, VFAs逐渐转化, 浓度降低, 表明前14 d为有机酸累积过程, 所以, 青霉素菌渣厌氧发酵产酸相为发酵的前14 d.
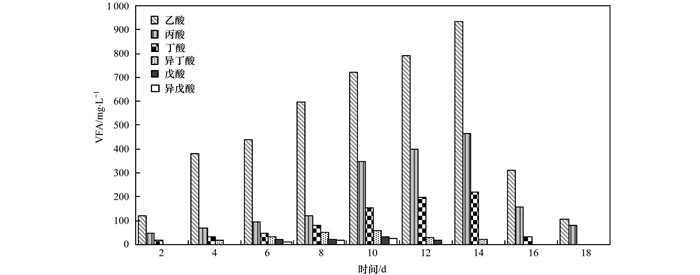
|
图 4 发酵过程VFA的变化 Fig. 4 Variation in VFA content during the digestion of penicillin bacterial residues |
根据图 4可知, 抗生素菌渣厌氧发酵有机酸积累发生在前14 d, 甲烷发酵共分为4个阶段, 即水解、酸化、乙酸化和甲烷化.在产酸相部分细菌通过胞外酶对固形物中碳水化物、蛋白质、脂肪等高分子水解生成结构简单的单糖、氨基酸、丙酮和高级脂肪酸.水解产物可以作为其他细菌的合成物质通过增值反应转变为挥发性脂肪酸(VFAs)[27].一般情况下水解是厌氧发酵限制性步骤, 所以针对这段时期溶解态和颗粒态COD、碳水化物以及蛋白质浓度进行了分析(图 5).
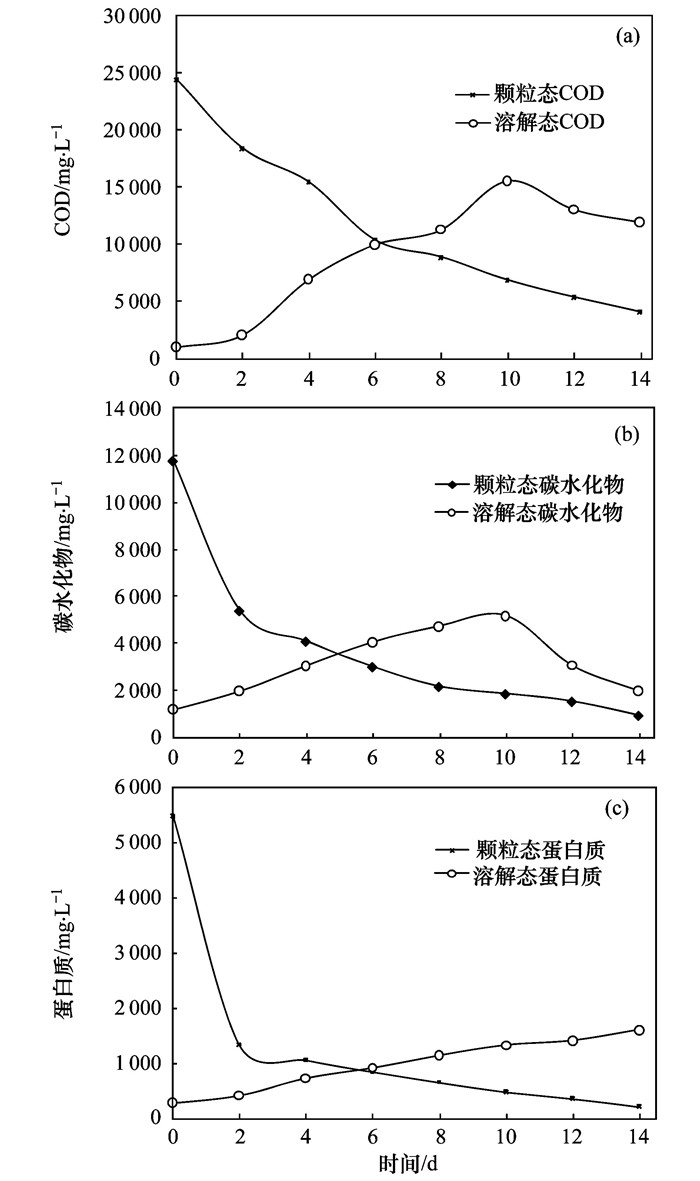
|
图 5 酸生成相颗粒态及溶解态有机物分解特性 Fig. 5 Decomposition characteristics of the particulate and dissolved organics during the acidogenic phase |
在酸生成的前10 d颗粒态COD的浓度很快降低, 同时溶解态的COD快速积累, 总的COD浓度几乎未变, 表明此阶段进行的主要是有机物的水解过程, 很少发生气体的转化, 这也符合产气曲线趋势图 1和图 2. 10 d后颗粒态COD仍快速下降, 但溶解态COD开始降低, 表明溶解性COD开始快速转化为CH4, 且其转化速度大于有机物的水解速度[图 5(a)].通过对颗粒态碳水化物和溶解态碳水化物浓度变化关系的分析[图 5(b)]发现, 碳水化物的变化规律与COD的相同, 并且10 d后溶解态碳水化物的减少量与溶解态COD的减少量大致相等, 表明溶解性COD转换为CH4主要是碳水化合物的转化.由图 5(c)可知, 在整个酸生成相颗粒态蛋白质浓度一直在下降, 溶解态蛋白质浓度一直在上升, 而且两者总量大致不变, 说明在酸生成相溶解性蛋白质没有向甲烷转化.提示青霉素菌渣厌氧发酵过程中首先是碳水化物的甲烷化, 后才开始蛋白质的转化.
3 结论(1) COD/SO42-≥3时, 青霉素菌渣厌氧发酵10 d后产气潜能逐渐恢复, 累积产甲烷量超过(以TS计) 208 mL·g-1; COD去除率为75%~84%, 其中超过71%的COD转化为CH4, 表明发酵微生物完成了适应性驯化; COD/SO42-≤1.5时, 产甲烷分别受到部分或者完全抑制, COD去除率为11%~17%, SO42-的去除率不足5%, 表明较高SO42-负荷条件下MPB与SRB都受到了抑制, 其中转化为CH4的COD不足9.1%, 而还原SO42-的COD保持在5.0%~9.0%, 表明MPB菌比SRB菌对S2-的毒性更为敏感.
(2) 随SO42-负荷的提高还原为S2-和H2S的SO42-由86%减少为1.5%, 还原的SO42-大部分以S2-的形式存在于发酵液中, 少部分以H2S的形式存在于生物气中.
(3) 产酸相累积的溶解性碳水化物10 d后向甲烷转化, 溶解性蛋白质的甲烷化则是在溶解性碳水化物甲烷化之后才开始.
| [1] |
王勇军, 陈平, 韦惠民, 等. 青霉素菌渣厌氧消化技术研究[J]. 中国沼气, 2015, 33(5): 28-31. Wang Y J, Chen P, Wei H M, et al. Anaerobic digestion of penicillin bacterial residue[J]. China Biogas, 2015, 33(5): 28-31. |
| [2] |
洪晨, 杨强, 王志强, 等. 抗生素菌渣与煤混合燃烧特性及其动力学分析[J]. 化工学报, 2017, 68(1): 360-368. Hong C, Yang Q, Wang Z Q, et al. Co-combustion characteristics and kinetic analysis of antibiotic bacterial residue and coal[J]. CIESC Journal, 2017, 68(1): 360-368. |
| [3] | Hu X G, Zhou Q X, Luo Y. Occurrence and source analysis of typical veterinary antibiotics in manure, soli, vegetables and groundwater from organic vegetable bases, northern China[J]. Environmental Pollution, 2010, 158(9): 2992-2998. DOI:10.1016/j.envpol.2010.05.023 |
| [4] | Ramaswamy J, Prasher S O, Patel R M, et al. The effect of composting on the degradation of a veterinary pharmaceutical[J]. Bioresource Technology, 2010, 101(7): 2294-2299. DOI:10.1016/j.biortech.2009.10.089 |
| [5] |
公丕成, 蔡辰, 张博, 等. 我国抗生素菌渣资源化研究新进展[J]. 环境工程, 2017, 35(5): 107-111. Gong P C, Cai C, Zhang B, et al. New progress of research on resource of antibiotic bacterial residue in China[J]. Environmental Engineering, 2017, 35(5): 107-111. |
| [6] |
王慧, 习彦花, 崔冠慧, 等. 头孢菌素菌渣中温厌氧资源化处理[J]. 中国沼气, 2016, 34(1): 19-24. Wang H, Xi Y H, Cui G H, et al. The Resource utilization of cephalosporins bacterial residue by anaerobic digestion under mesophilic temperature[J]. China Biogas, 2016, 34(1): 19-24. |
| [7] |
苏建文, 王俊超, 许尚营, 等. 红霉素菌渣厌氧消化实验研究[J]. 中国沼气, 2013, 31(5): 25-28. Su J W, Wang J C, Xu S Y, et al. Anaerobic digestion of bacterial residues from erythromycin production[J]. China Biogas, 2013, 31(5): 25-28. |
| [8] |
朱培, 张建斌, 陈代杰. 抗生素菌渣处理的研究现状和建议[J]. 中国抗生素杂志, 2013, 38(9): 647-651. Zhu P, Zhang J B, Chen D J. Current research and suggestions for treatment of antibiotic manufacturing biowaste[J]. Chinese Journal of Antibiotics, 2013, 38(9): 647-651. |
| [9] | Zhong W Z, Li Z X, Yang J L, et al. Effect of thermal-alkaline pretreatment on the anaerobic digestion of streptomycin bacterial residues for methane production[J]. Bioresource Technology, 2014, 151: 436-440. DOI:10.1016/j.biortech.2013.10.100 |
| [10] | Zhang G Y, Li C X, Ma D C, et al. Anaerobic digestion of antibiotic residue in combination with hydrothermal pretreatment for biogas[J]. Bioresource Technology, 2015, 192: 257-265. DOI:10.1016/j.biortech.2015.05.014 |
| [11] |
孙效新, 黄栋, 李建民, 等. 抗生素废菌渣液厌氧生物处理试验研究[J]. 中国沼气, 1990, 8(3): 11-14. Sun X X, Huang D, Li J M, et al. Study on the treatment of sewage of antibiotic mycelium by anaerobic digestion[J]. China Biogas, 1990, 8(3): 11-14. |
| [12] |
何品晶, 管冬兴, 吴铎, 等. 氨氮和林可霉素对有机物厌氧消化的抑制效应[J]. 化工学报, 2011, 62(5): 1389-1394. He P J, Guan D X, Wu D, et al. Inhibitory effect of ammonia and lincomycin on anaerobic digestion[J]. CIESC Journal, 2011, 62(5): 1389-1394. |
| [13] | de Oliveira L L, Silveira Duarte I C, Sakamoto I K, et al. Influence of support material on the immobilization of biomass for the degradation of linear alkylbenzene sulfonate in anaerobic reactors[J]. Journal of Environmental Management, 2009, 90(2): 1261-1268. DOI:10.1016/j.jenvman.2008.07.013 |
| [14] |
徐婷, 金艳青, 李勇. 常温下加装脱硫装置的MCAnMBR处理高硫酸盐有机废水试验[J]. 环境科学, 2017, 38(12): 5132-5138. Xu T, Jin Y Q, Li Y. Methane cycle anaerobic membrane bioreactor with desulfurization for treating high sulfate organic wastewater at normal temperature[J]. Environmental Science, 2017, 38(12): 5132-5138. |
| [15] | Maree J P, Strydom W F. Biological sulphate removal in an up-flow packed bed reactor[J]. Water Research, 1985, 19(9): 1101-1106. DOI:10.1016/0043-1354(85)90346-X |
| [16] | Pokorna-Krayzelova L, Mampaet K E, Vannecke T P W, et al. Model-based optimization of microaeration for biogas desulfurization in UASB reactors[J]. Biochemical Engineering Journal, 2017, 125: 171-179. |
| [17] |
姚宏, 何永淼, 吕开雷, 等. 高硫酸盐抗生素废水生物处理[J]. 北京交通大学学报, 2012, 36(3): 97-101. Yao H, He Y M, Lv K L, et al. Biological treatment of high sulfate-containing antibiotics wastewater[J]. Journal of Beijing Jiaotong University, 2012, 36(3): 97-101. |
| [18] | Lopes S I C, Wang X, Capela M I, et al. Effect of COD/SO42- ratio and sulfide on thermophilic (55℃) sulfate reduction during the acidification of sucrose at pH 6[J]. Water Research, 2007, 41(11): 2379-2392. DOI:10.1016/j.watres.2007.02.023 |
| [19] | Lu X Q, Zhen G Y, Ni J L, et al. Effect of influent COD/SO42- ratios on biodegradation behaviors of starch wastewater in an upflow anaerobic sludge blanket (UASB) reactor[J]. Bioresource Technology, 2016, 214: 175-183. DOI:10.1016/j.biortech.2016.04.100 |
| [20] | Shan L L, Zhang Z H, Yu Y L, et al. Performance of CSTR-EGSB-SBR system for treating sulfate-rich cellulosic ethanol wastewater and microbial community analysis[J]. Environmental Science and Pollution Research, 2017, 24(16): 14387-14395. DOI:10.1007/s11356-017-9022-5 |
| [21] | Mazine Kiyuna L S, Fuess L T, Zaiat M. Unraveling the influence of the COD/sulfate ratio on organic matter removal and methane production from the biodigestion of sugarcane vinasse[J]. Bioresource Technology, 2017, 232: 103-112. DOI:10.1016/j.biortech.2017.02.028 |
| [22] | Li W C, Niu Q G, Zhang H, et al. UASB treatment of chemical synthesis-based pharmaceutical wastewater containing rich organic sulfur compounds and sulfate and associated microbial characteristics[J]. Chemical Engineering Journal, 2015, 260: 55-63. DOI:10.1016/j.cej.2014.08.085 |
| [23] | Rinzema A, Lettinga G. The effect of sulphide on the anaerobic degradation of propionate[J]. Environmental Technology Letters, 1988, 9(2): 83-88. DOI:10.1080/09593338809384544 |
| [24] | Ren N Q, Chua H, Chan S Y, et al. Effects of COD/SO42- ratios on an acidogenic sulfate-reducing reactor[J]. Industrial & Engineering Chemistry Research, 2007, 46(6): 1661-1666. |
| [25] | Zhang L H, De Schryver P, De Gusseme B, et al. Chemical and biological technologies for hydrogen sulfide emission control in sewer systems:A review[J]. Water Research, 2008, 42(1-2): 1-12. DOI:10.1016/j.watres.2007.07.013 |
| [26] | Hilton B L, Oleszkiewicz J A. Sulfide-induced inhibition of anaerobic digestion[J]. Journal of Environmental Engineering, 1988, 114(6): 1377-1391. DOI:10.1061/(ASCE)0733-9372(1988)114:6(1377) |
| [27] | Karhadkar P P, Audic J M, Faup G M, et al. Sulfide and sulfate inhibition of methanogenesis[J]. Water Research, 1987, 21(9): 1061-1066. DOI:10.1016/0043-1354(87)90027-3 |
| [28] | Camiloti P R, Mockaitis G, Domingues Rodrigues J A, et al. Innovative anaerobic bioreactor with fixed-structured bed (ABFSB) for simultaneous sulfate reduction and organic matter removal[J]. Journal of Chemical Technology and Biotechnology, 2014, 89(7): 1044-1050. DOI:10.1002/jctb.2014.89.issue-7 |
| [29] | Okabe S, Nielsen P H, Jones W L, et al. Sulfide product inhibition of Desulfovibrio desulfuricans in batch and continuous cultures[J]. Water Reseach, 1995, 29(2): 571-578. DOI:10.1016/0043-1354(94)00177-9 |
| [30] | Li Y F, Jin W, Cheng Y F, et al. Effect of the associated methanogen Methanobrevibacter thaueri on the dynamic profile of end and intermediate metabolites of anaerobic fungus Piromyces sp. F1[J]. Current Microbiology, 2016, 73(3): 434-441. DOI:10.1007/s00284-016-1078-9 |
| [31] | Yang Y, Chen Q, Guo J L, et al. Kinetics and methane gas yields of selected C1 to C5 organic acids in anaerobic digestion[J]. Water Research, 2015, 87: 112-118. DOI:10.1016/j.watres.2015.09.012 |
| [32] | Li Q, Qiao W, Wang X C, et al. Kinetic characterization of thermophilic and mesophilic anaerobic digestion for coffee grounds and waste activated sludge[J]. Waste Management, 2015, 36: 77-85. DOI:10.1016/j.wasman.2014.11.016 |
| [33] |
孙建平, 郑平, 胡宝兰. 多种抗生素对畜禽废水厌氧消化的联合抑制[J]. 环境科学, 2009, 30(9): 2619-2624. Sun J P, Zheng P, Hu B L. Combined effect of antibiotics on anaerobic digestion of piggery wastewater[J]. Environmental Science, 2009, 30(9): 2619-2624. |
| [34] | Mustapha N A, Sakai K, Shirai Y, et al. Impact of different antibiotics on methane production using waste-activated sludge:mechanisms and microbial community dynamics[J]. Applied Microbiology and Biotechnology, 2016, 100(21): 9355-9364. DOI:10.1007/s00253-016-7767-2 |
| [35] | Sari S, Tas D O. Anaerobic degradation of diclofenac under mesophilic conditions[J]. Fresenius Environmental Bulletin, 2014, 23(12): 3048-3053. |
| [36] | Annachhatre A P, Suktrakoolvait S. Biological sulfate reduction using molasses as a carbon source[J]. Water Environment Research, 2001, 73(1): 118-126. DOI:10.2175/106143001X138778 |
| [37] | Barrera E L, Spanjers H, Romero O, et al. Characterization of the sulfate reduction process in the anaerobic digestion of a very high strength and sulfate rich vinasse[J]. Chemical Engineering Journal, 2014, 248: 383-393. DOI:10.1016/j.cej.2014.03.057 |
| [38] | Zwietering M H, Jongenburger I, Rombouts F M, et al. Modeling of the bacterial growth curve[J]. Applied and Environmental Microbiology, 1990, 56(6): 1875-1881. |
| [39] | Percheron G, Bernet N, Moletta R. Start-up of anaerobic digestion of sulfate wastewater[J]. Bioresource Technology, 1997, 61(1): 21-27. DOI:10.1016/S0960-8524(97)00060-6 |
| [40] | Jing Z Q, Hu Y, Niu Q G, et al. UASB performance and electron competition between methane-producing archaea and sulfate-reducing bacteria in treating sulfate-rich wastewater containing ethanol and acetate[J]. Bioresource Technology, 2013, 137: 349-357. DOI:10.1016/j.biortech.2013.03.137 |
 2018, Vol. 39
2018, Vol. 39

