2. 南京工业大学生物与制药工程学院, 南京 211816;
3. 南京工业大学化工学院, 南京 211816;
4. 四川省地质矿产勘查开发局, 成都水文地质工程地质队, 成都 610072
2. College of Biotechnology and Pharmaceutical Engineering, Nanjing Tech University, Nanjing 211816, China;
3. College of Chemical Engineering, Nanjing Tech University, Nanjing 211816, China;
4. Chengdu Hydrogeology and Engineering Geology Team, Sichuan Bureau of Geology & Mineral Resources, Chengdu 610072, China
多环芳烃(PAHs)是由两个或多个苯环以线状、角状或簇状的排列方式构成的有机物, 具有致癌、致畸、致突变性(“三致性”)[1, 2].从航运、石油泄漏、地表径流以及矿物燃料的不完全燃烧等各种活动排放的多环芳烃对环境造成了持续危害[3].这种污染物由于其非常低的水溶性和对海洋沉积物的强吸附力往往在环境中持续存在. PAHs主要是通过挥发、光降解、沉降、化学氧化和微生物降解来去除, 其中微生物降解是最为彻底的多环芳烃去除方法[4], 已成为污染治理的主要研究方向.
在探求多环芳烃降解的过程中, 筛选分离出高效降解菌株是多环芳烃微生物降解技术的关键.相对于低分子量的PAHs, 四环及四环以上的PAHs由于其分子结构复杂、难于被氧化、水溶性差等很难筛选出高效降解的菌株.芘是环境中普遍存在的四环的PAHs, 具有稳定的四苯环对称结构, 是高分子量PAHs的代表化合物.目前发现能够降解芘的典型细菌包括以下几种:不动杆菌属(Acinetobacter sp.)[5]、芽孢杆菌属(Bacillus sp.)[6~9]、克雷伯氏菌属(Klebsiella sp.)[10~12]、解环菌属(Cycloclasticus sp.)[13]、分枝杆菌属(Mycobacterium sp.)[14]、假单胞菌属(Pseudomonas sp.)[11~12, 15]、红球菌属(Rhodococcus sp.)[16]、鞘氨醇单胞菌属(Sphingomonas sp.)[17]、黄杆菌属(Flavobacterium sp.)[18~19]、戈登氏菌属(Gordona sp.)[20~21]、糖丝菌属(Saccharothrix sp.)[22]等. PAHs的微生物降解的难易程度往往与有机物的溶解度、环数、杂环原子性质和取代基的数目、种类、位置有关, 而各种类微生物对不同PAHs降解的机制也各不相同[23].近几年关于萘、菲等简单的PAHs微生物代谢途径的研究较为清晰[24~28], 但对于四环及四环以上的PAHs研究目前还一直是热点, 特别是对四环及四环以上的PAHs的降解途径仍需深入探究[29, 30].
本文从长期被石油污染的石化污泥中分离出1株芘降解菌株, 对其进行了生理生化及分子生物学的鉴定, 考察了菌株的生长及其对芘的降解特性, 并探究了该菌株对芘降解的途径, 以期为多环芳烃污染地区的生物修复和治理提供重要的理论依据.
1 材料与方法 1.1 材料与试剂菌源取自于南京某石化污水厂二沉池活性污泥.
无机盐培养基:NaCl 24 g、KCl 0.7 g、MgSO4·7H20 0.7 g、NH4Cl 1 g、NaNO3 0.5 g、KH2PO4 2 g、Na2HPO4 3 g, 1 000 mL蒸馏水, pH 6~7.芘无机盐培养基:以丙酮配制1 g·L-1的芘丙酮溶液, 取一定量的芘丙酮溶液, 置于已灭菌的无机盐培养基中, 在无菌状态下, 待丙酮挥发完毕.注意芘丙酮溶液加入无机盐培养基时会出现白色浑浊物, 是未挥发的丙酮所致, 待丙酮挥发完全后白色浑浊物消失. LB培养基:NaCl 10 g、蛋白胨10 g、酵母粉5 g. LB培养基中加入琼脂15~20 g制成LB平板培养基.以上培养基均使用高压蒸汽灭菌锅在121℃条件下灭菌20 min.
主要试剂:多环芳烃污染样品芘(Pyene, 质量分数>97%), 购自TCI公司.多环芳烃标准品芘(Pyene, 质量分数99.1%), 购自AccuStandard公司, 有机溶剂纯度均为色谱纯. HTP试剂:按照吡啶:六甲基二硅胺:三甲基氯硅烷比例为7:2:1配置, 混合后-20℃保存备用.
1.2 菌种的筛选与纯化取新鲜的污泥10 g在100 mL 0.85%的生理盐水中振荡20 min, 再取悬液接入以芘(10 mg·L-1)为唯一碳源的无机盐培养基中, 恒温30℃振荡速率为180 r·min-1培养7 d.然后再次转接到20 mg·L-1的芘无机盐培养基中, 继续培养.如此重复, 直到培养基中芘浓度为160mg·L-1.将最后一次的培养液进行10-1~10-9梯度稀释并涂布到LB平板培养基上, 经过培养挑选出生长迅速、丰富的单菌落, 进行平板反复划线分离, 直至得到纯种菌.以1%的接种量转接至三角瓶中, 所有操作在超净工作台中进行.为防止芘被光解, 实验均在暗室培养箱中操作.
1.3 菌种的鉴定通过菌体的形态特征、生理生化特性以及16S rDNA序列结果进行鉴定.
1.3.1 形态特征观察将各纯菌接入LB平板培养基中, 在30℃培养至对数生长期后, 观察细菌的生长状况、菌落形态以及颜色, 同时通过革兰氏染色在显微镜下观察细胞形态.
1.3.2 菌株生理生化特性测定应用美国Biolog公司的Biolog微生物自动分析系统(GEN Ⅲ MicroPlateTM测试面板)对细菌的94种生化表型进行测试, 并鉴定菌种.在94个表型测试中分析了71个碳源利用测定和23个化学敏感性测定. Biolog的微生物鉴定系统软件从GEN Ⅲ MicroPlateTM测试面板的表型模式中鉴定细菌.测试板可以提供微生物的“表型指纹图谱”, 可用于在物种层面识别鉴定[31, 32].所有程序参阅Biolog公司GEN Ⅲ用户指南进行.将获得的纯培养物接种至Biolog所推荐的BUG培养基上, 在IF-A接种液中制备菌悬液, 在Biolog读数仪上读取微孔板数据, 重复3次读数, 最终再与数据库比对鉴定[33, 34].
1.3.3 16S rDNA序列测定鉴定菌株在30℃ LB培养基中生长至对数期, 1.5 mL菌液在转速为12 000 r·min-1下离心收集菌体, 并对该菌株16S rDNA基因序列分析. 16S rDNA扩增的引物为27F AGAGTTTGATCCTGG CTCAG和1492R GGTTACCTTGTTACGACTT[29]. PCR所用酶为ExTaq, PCR反应程序:95℃预变性5 min, 95℃ 30 s, 55℃ 30 s, 72℃ 90 s, 30次循环, 72℃延伸5 min, 4℃保存. DNA纯化和测序工作委托金斯瑞公司完成.测序结果在NCBI上通过Blast程序进行序列同源性比较, 同时下载同源性较高且已知分类菌种的16S rDNA碱基序列, 利用clustalx进行相似性分析, MEGA4软件采用Neighbor-Joining法构建系统发育树图.
1.4 菌株生长曲线动力学研究将活化两次后的种子液以1%接种于LB培养基中, 恒温培养, 每隔1~2 h取样测量其600 nm处的吸光值(D600值).根据菌株在不同时间的生长数据, 选择修正Gompertz模型拟合芘降解菌的生长曲线.通过修正Gompertz模型[35][公式(1)]拟合得到的模型参数, 计算出最大比生长速率U [公式(2)]和微生物生长的延滞期LPD [公式(3)].最终采用R2对建立模型进行评价[36].
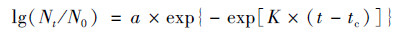
|
(1) |
由此可以得出U和LPD的计算公式如下:
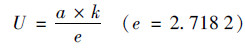
|
(2) |
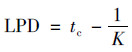
|
(3) |
式中, t为时间; Nt和N0分别表示在时间t时和初始时间微生物数量; a为最大菌数Nmax与初始菌数N0的差值; tc为达到最大生长速率的时间; K为在时间点tc的最大生长速率.
1.5 降解效率的测定将种子液以1%接种量接种于以芘为唯一碳源的无机盐培养基中, 芘浓度为50 mg·L-1, 恒温30℃振荡速率为180 r·min-1下培养, 以含芘而不接菌的无机盐培养基作为空白对照, 记为CK.在培养过程中分别在第0、3、7、11、17、23 d定时取样测定好氧过程中芘的降解率.
取菌液与等体积二氯甲烷混合, 旋转超声, 离心, 重复3次萃取后, 旋转蒸发至萃取液近干.加入无水硫酸钠除去多余水分, 用甲醇定容, 过0.22 μm孔径有机滤膜, 最后进HPLC分析. HPLC测试条件:安捷伦1200系列高效液相色谱, 色谱柱为Water PAHs C-18专用柱, 检测器是紫外检测器, 柱温35℃; 流动相为甲醇, 流速为0.8 mL·min-1, 进样量为10 μL, 检测波长为254 nm.
1.6 中间代谢产物的分析选取LX2降解后的样品进行代谢产物的分析测定.采用等体积的二氯甲烷超声萃取菌液3次, 萃取方法同上.萃取液氮吹至干后, 加入250 μL的HTP试剂进行衍生化反应(120℃下反应30 min), 样品制备好后运用气质联用(GC-MS)检测降解产物. GC-MS条件:进样口温度250℃, 不分流模式, 色谱柱HP-5MS熔融石英柱(30 m×250 μm×0.25 μm).色谱条件:初始柱温70℃, 保留1.5 min, 16℃·min-1升温至250℃, 保留时间10 min.质谱条件为:离子温度200℃, 电子能量70 eV, 扫描范围(m/z)50~400, 载气为氦气, 流速为1 mL·min-1.检索谱库为NIST谱库.空白样为含芘而不接菌的无机盐培养基.
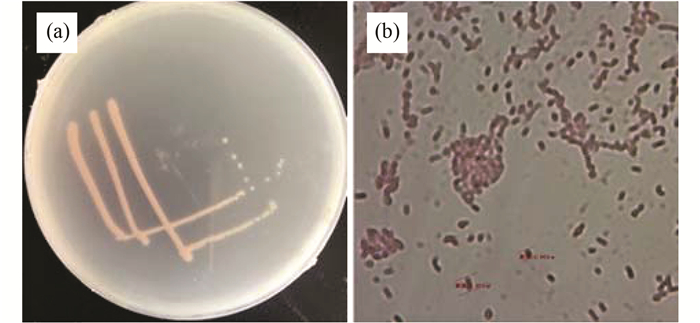
2 结果与讨论 2.1 菌种的鉴定 2.1.1 形态特征观察经过反复筛选、驯化及纯化, 最终筛选出1株好氧降解菌LX2作为研究菌株.菌株LX2在LB培养基中生长良好, 在LB平板培养基6 h后长出黄色菌落, 12 h后菌落变成粉色, 最终变为红色, 为好氧菌.菌株LX2肉眼可看到清晰的单菌落, 菌落较小且扁平, 呈现不规则圆形, 直径0.5~2.0 mm左右, 湿润, 如图 1所示.菌体直或稍弯的杆菌, 菌株大小1.94~2.87 μm左右, 在相差显微镜中观察到菌株活泼的运动, 为革兰氏阴性菌.
|
(a)平板培养,(b)革兰氏染色 图 1 LX2菌株形态 Fig. 1 Morphological observation of the strain LX2 |
由微量分析系统GENIII MicroPlateBiolog确定重要的生物化学特征如表 1所示[33, 37, 38].菌株LX2可有效利用8种碳源, 包括α-D-葡糖、D-果糖-6-磷酸、L-谷氨酸等, 同时化学敏感性测试中, 对14种化学物质敏感, 其中包括夫西地酸、D-丝氨酸、二甲胺四环素、毒素有利福霉素、林肯霉素、万古霉素等.菌株LX2在pH 5.0和6.0都可以很好地生长.经Biolog细菌鉴定系统分析菌株LX2是铜绿假单胞菌属, 可能性为92.0%, 相似性为0.647, 位距为4.180.由此结合菌株LX2的形态特征和Biolog鉴定结果初步确定为铜绿假单胞菌属.
|
|
表 1 生理生化特征测定1) Table 1 Characteristics of physiology and biochemistry |
2.1.3 16S rDNA序列测定
提取菌株的总DNA, 利用16S rDNA通用引物进行PCR扩增得到目的DNA片段, 最终得到1 500 bp的16S rDNA部分序列, 测序结果输入NBCI, 登录号为MF593283.经过BLAST分析比对, 显示菌株LX2与多株铜绿假单胞菌(Pseudomonas aeruginosa)的同源性均为100%.最后将同源性高的菌株的碱基序列依次下载后, 用clustalx进行相似性分析, 并用MEGA 4软件采用Neighbor-Joining法构建系统发育树图(图 2).由图 2可知, 菌株LX2与多株铜绿假单胞菌位于同一分支.综合菌株的形态特征、生理生化特性以及16S rDNA序列结果, 推测LX2属于铜绿假单胞菌, 命名为Pseudomonas sp. LX2.
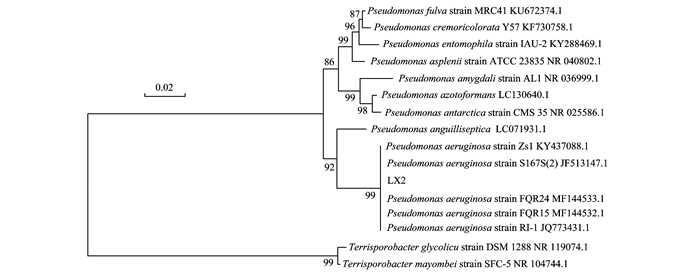
|
图 2 基于16S rDNA基因序列同源性的Pseudomonas sp.LX2系统发育树 Fig. 2 Phylogenetic tree of Pseudomonas sp. LX2based on the 16S rDNA gene homology |
运用Origin软件, 选用SGompertz模型分别拟合菌株LX2的生长曲线(图 3), 得到该模型的拟合参数并计算出各自最大比生长速率U和微生物生长的延滞期LPD(表 2).模型的R2为0.992 32, 说明SGompertz模型可以很好地描述LX2的生长.由表 2可知, 菌株LX2在第6.7 h达到最大生长率速度, LX2的D600值最高达1.794 9±0.019 35, 每小时D600值增长0.16, LX2的最大比增长速率约为0.1, 延迟期大约只有0.6 h, 在2 d后结束对数期.
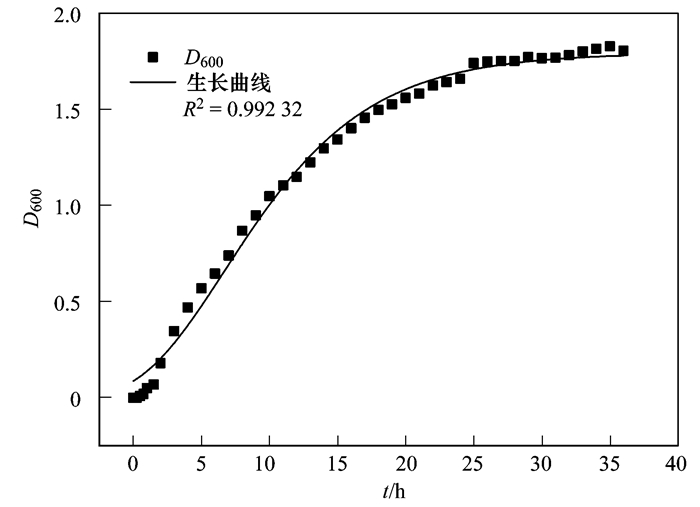
|
图 3 SGompertz模型拟合的Pseudomonas sp. LX2的生长曲线 Fig. 3 Growth curves of Pseudomonas sp. LX2 calculated from the Standard Gompertz model |
|
|
表 2 SGompertz模型得到的Pseudomonas sp. LX2的生长参数1) Table 2 Kinetic growth parameters estimated by the Standard Gompertz model of Pseudomonas sp. LX2 |
2.3 Pseudomonas sp. LX2对芘的降解能力研究
利用HPLC对芘降解效率进行定量分析, 以不接菌的无机盐培养基作为空白对照CK, Pseudomonas sp. LX2在芘初始浓度为50 mg·L-1培养基中对芘的降解效果见图 4.从中可以看出Pseudomonas sp. LX2对芘的降解率随着培养时间的延长而逐渐升高, Pseudomonas sp. LX2在培养第3 d对芘的降解率高达16.6%, 在第17 d时进入衰退期, 降解率在第21 d达到最大为32.1%.在不加菌的空白培养基中, 芘浓度几乎没有变化.由于芘含有4个苯环, 在自然环境中非常难于降解, 且降解菌株在不提供其他碳源的情况下降解通常很低[4].
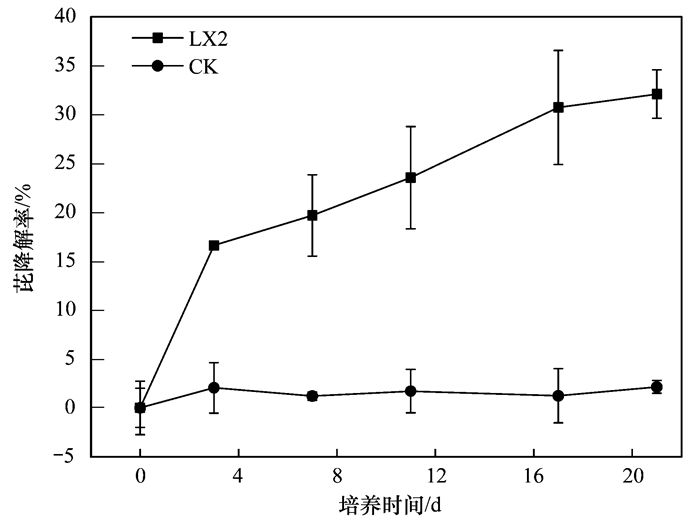
|
图 4 Pseudomonas sp. LX2对芘的降解率 Fig. 4 Rate of pyrene biodegradation by Pseudomonas sp. LX2 |
通过GC-MS进行定性分析, 深入研究Pseudomonas sp. LX2对芘好氧降解过程中的一系列中间代谢产物.提取样品的色谱图表明, Pseudomonas sp. LX2在对芘降解过程中形成了多种代谢产物, 同时以加芘而非加菌的培养基作为空白对照, 结果如图 5所示.从中可知, 与空白对照相比, Pseudomonas sp. LX2的反应体系中在第4 d发现了6种物质. 13.517、13.114和13.336 min的吸收峰经质谱比对分别为芘(C16H10, CAS:129-00-0)、4, 5-二氢芘(C16H12, CAS:6628-98-4)和1, 2, 3, 3a, 4, 5-六氢芘(C16H16, CAS:5385-37-5); 12.697 min和12.947 min的吸收峰经质谱比对分别是氢化肉桂酸(C9H10O2, CAS:501-52-0)和2′-羟基苯丙酮(C9H10O2, CAS:610-99-1)硅烷化后的产物; 氢化肉桂酸和2′-羟基苯丙酮推测为1, 2, 3, 3a, 4, 5-六氢芘经过多次代谢后的产物; 氢化肉桂酸进一步代谢为苯乙酸(C8H8O2, CAS:103-82-2)和苯酚(C6H6O, CAS:108-95-2), 硅烷化后产物被确认停留时间在12.857 min.以上物质分别通过标准品GC/MS分析证实.接种Pseudomonas sp. LX2的反应体系在第7 d的代谢产物中也发现了1, 2, 3, 3a, 4, 5-六氢芘(停留时间为13.343 min), 同时产生了另外两种新的代谢产物, 色谱停留时间分别为10.147 min和10.772 min, 经质谱比对后鉴定为3, 4, 5, 6-四氢苯酐(C8H8O3, CAS:2426-02-0)和原儿茶酸(C7H6O4, CAS:99-50-3), 原儿茶酸为硅烷化后的产物.接种Pseudomonas sp. LX2的反应体系在第11 d的培养物中也发现了原儿茶酸硅烷化后的产物, 另外还发现了邻苯二甲酸二异丙酯(C14H18O4, CAS:605-45-8),色谱停留时间为11.974 min.
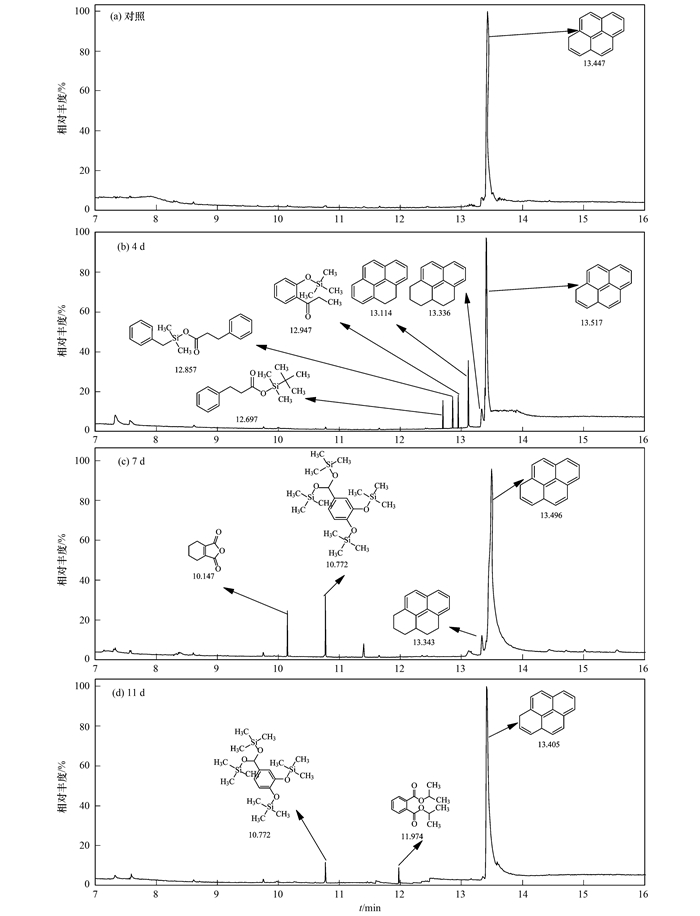
|
图 5 Pseudomonas sp. LX2对芘降解第4、7、11d时中间产物的GC-MS分析 Fig. 5 GC-MS chromatogram of pyrene (4th, 7th, and 11th day) isolates degraded by Pseudomonas sp. LX2 |
根据以上中间产物的分析, 推导出Pseudomonas sp. LX2好氧降解芘的途径如图 6所示.从中可以看出, 氢化还原可能是芘降解的初始活化反应, 芘先通过加入两个氢原子还原成4, 5-二氢芘, 再进一步还原生成1, 2, 3, 3a, 4, 5-六氢芘, 然后饱和碳原子上的碳—碳键切割并氧化最终生成1-羟基-2-萘甲酸.众所周知, 1-羟基-2-萘甲酸被认为是多环芳烃生物降解的关键中间产物[28].氢化还原之后碳—碳键切割是根据假单胞菌降解通过加入氢原子还原苯并芘代谢生成芘进一步生成菲推测而来[39], 随后被氧化最终产生的1-羟基-2-萘甲酸是通过多环芳烃相邻的碳键上发生了羟基化双氧合, 形成氧化产物, 导致苯环裂解.虽然在本研究中没有检测到1-羟基-2-萘甲酸, 但是在许多推定芳烃双加氧酶基因的研究可以支持这一推测[24, 26, 40].已知1-羟基-2-萘甲酸通常可以通过被称为“邻苯二甲酸”或“萘”途径的两种不同途径进行降解[41]. Pseudomonas sp. LX2对芘的降解至少有两个不同的途径.在“邻苯二甲酸”途径中, 1-羟基-2-萘甲酸在脱氢酶、双加氧酶、脱氢异构酶等一系列酶的作用下苯环裂解[42], 生成邻苯二甲酸二异丙酯和2′-羟基苯丙酮, 邻苯二甲酸二异丙酯进一步生成邻苯二甲酸.邻苯二甲酸酐是邻苯二甲酸通过分子内脱水反应的产物, 邻苯二甲酸酐进一步转化生成3, 4, 5, 6-四氢苯酐.通过3, 4, 5, 6-四氢苯酐的存在证明在Pseudomonas sp. LX2对芘的降解过程中产生了邻苯二甲酸[26].邻苯二甲酸和2′-羟基苯丙酮还可以通过“邻苯二甲酸”途径进一步转化为原儿茶酸[27], Qin等[43]在研究苯并[a]芘降解过程中发现, 中间产物菲被氧化成2-甲基-1-萘乙酸, 由此推导出2′-羟基苯丙酮的生成.第二条代谢途径是被称为“萘”的代谢途径, 在培养物中检测到的化合物氢化肉桂酸, 判断其是1-羟基-2-萘甲酸的进一步降解的产物, 从物质的结构来看, 推测氢化肉桂酸是通过1-羟基-2-萘甲酸降解获得的1-萘酚转化而来, 通过1-萘酚代谢1-羟基-2-萘甲酸的替代途径在以前的研究中也有发现[24, 28, 44].氢化肉桂酸依次氧化降解为苯乙酸和苯酚, 苯酚也有可能会被氧化为儿茶酚[45]和原儿茶酸一起经过苯环裂解进入TCA循环, 最终被完全降解[28].本研究中3, 4, 5, 6-四氢苯酐和1-萘酚中间产物的存在表明1-羟基-2-萘甲酸是通过“邻苯二甲酸”和“萘”两条途径进行代谢.邻苯二甲酸可以通过“邻苯二甲酸”途径进一步转化为原儿茶酸[39], 水杨酸可以进一步转化为龙胆酸或儿茶酚[24, 46].虽然在“萘”途径中, 两种产物都可以降解形成TCA循环中间体[28], 但“邻苯二酚”途径比“龙胆酸”途径更常见[47]. Pseudomonas sp. LX2在“邻苯二甲酸”途径中显示出降解为原儿茶酸的能力, 在“萘”途径中推测降解为儿茶酚.
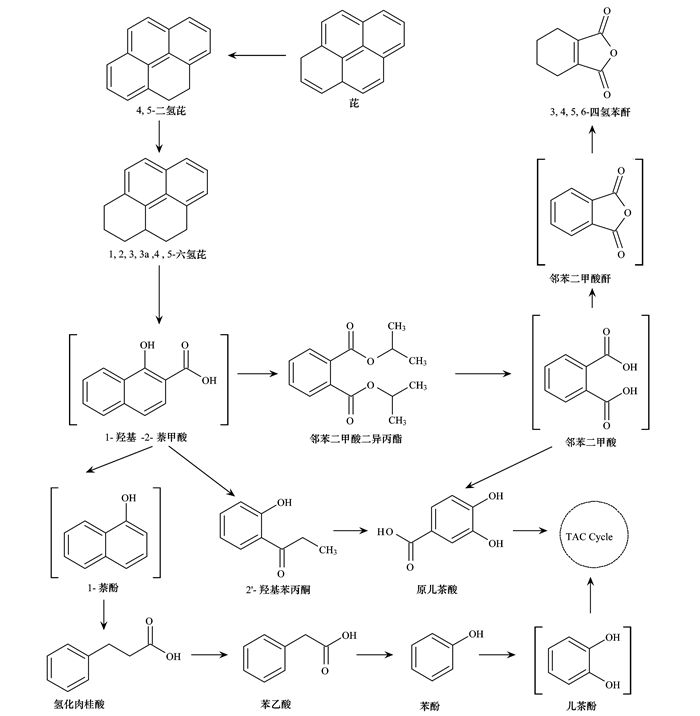
|
括号中的化合物是假设的中间体,但没有检测到 图 6 Pseudomonas sp. LX2对芘好氧降解的途径 Fig. 6 Proposed pathways for aerobic degradation of pyrene by Pseudomonas sp. LX2 |
自南京某石化污水厂二沉池活性污泥中筛选得到1株能以芘为唯一碳源生长的菌株LX2.通过形态鉴定、Biolog及16S rDNA鉴定表明, 菌株属于铜绿假单胞菌(Pseudomonas sp. LX2). Pseudomonas sp. LX2可以有效利用8种碳源, 对14种化学物质敏感, pH值在5.0和6.0时生长良好, 该菌株在LB培养基中生长曲线可以利用SGompertz模型得到很好的描述, 菌株生长迅速, 生长停滞期只有0.6 h, 在7.7 h达到最大生长速率.芘的降解率研究表明, 在以芘为唯一碳源(1%的接菌量, 初始芘浓度为50 mg·L-1)的无机盐培养基中培养21 d后, 芘的降解率高达32.1%.对Pseudomonas sp. LX2在无机盐培养基中降解芘的代谢产物进行GC-MS分析, 检测到一系列的代谢产物, 包括4, 5-二氢芘、1, 2, 3, 3a, 4, 5-六氢芘、氢化肉桂酸、2′-羟基苯丙酮、苯乙酸、苯酚、3, 4, 5, 6-四氢苯酐、原儿茶酚和邻苯二甲酸二异丙酯, 并发现这些物质在降解产物中有一定积累, 从而推测出该菌株可能通过氢化还原开始对芘的初始活化反应, 并通过“邻苯二甲酸”和“萘”两条途径代谢降解芘.
| [1] | Vela N, Martínez-Menchón M, Navarro G, et al. Removal of polycyclic aromatic hydrocarbons (PAHs) from groundwater by heterogeneous photocatalysis under natural sunlight[J]. Journal of Photochemistry and Photobiology A:Chemistry, 2012, 232: 32-40. DOI:10.1016/j.jphotochem.2012.02.003 |
| [2] | Wu B, Zhang Y, Zhang X X, et al. Health risk assessment of polycyclic aromatic hydrocarbons in the source water and drinking water of China:quantitative analysis based on published monitoring data[J]. Science of the Total Environment, 2011, 410-411: 112-118. DOI:10.1016/j.scitotenv.2011.09.046 |
| [3] | 郑博文. 持久性污染物多环芳烃的研究进展[J]. 工业卫生与职业病, 2016, 42(5): 386-390. |
| [4] | Haritash A K, Kaushik C P. Biodegradation aspects of polycyclic aromatic hydrocarbons (PAHs):a review[J]. Journal of Hazardous Materials, 2009, 169(1-3): 1-15. DOI:10.1016/j.jhazmat.2009.03.137 |
| [5] | Zhao Z Y, Wong J W C. Biosurfactants from Acinetobacter calcoaceticus BU03 enhance the solubility and biodegradation of phenanthrene[J]. Environmental Technology, 2009, 30(3): 291-299. DOI:10.1080/09593330802630801 |
| [6] | Li C H, Wong Y S, Wang H Y, et al. Anaerobic biodegradation of PAHs in mangrove sediment with amendment of NaHCO3[J]. Journal of Environmental Sciences, 2015, 30: 148-156. DOI:10.1016/j.jes.2014.09.028 |
| [7] | Yuan S Y, Chang B V. Anaerobic degradation of five polycyclic aromatic hydrocarbons from river sediment in Taiwan[J]. Journal of Environmental Science and Health, Part B, 2007, 42(1): 63-69. DOI:10.1080/03601230601020860 |
| [8] | Chang B V, Chang I T, Yuan S Y. Anaerobic degradation of phenanthrene and pyrene in mangrove sediment[J]. Bulletin of Environmental Contamination and Toxicology, 2008, 80(2): 145-149. DOI:10.1007/s00128-007-9333-1 |
| [9] | Zhang W, Wang H, Zhang R, et al. Bacterial communities in PAH contaminated soils at an electronic-waste processing center in China[J]. Ecotoxicology, 2010, 19(1): 96-104. DOI:10.1007/s10646-009-0393-3 |
| [10] | Xu X Y, Chen X, Su P, et al. Biodegradation potential of polycyclic aromatic hydrocarbons by bacteria strains enriched from Yangtze River sediments[J]. Environmental Technology, 2016, 37(5): 513-520. DOI:10.1080/09593330.2015.1074289 |
| [11] | Li C H, Ye C, Hou X P, et al. Isolation and characterization of polycyclic aromatic hydrocarbon-degrading bacteria with tolerance to hypoxic environments[J]. Journal of Environmental Science and Health, Part A, 2017, 52(7): 581-589. DOI:10.1080/10934529.2017.1293991 |
| [12] | Zafra G, Absalón á E, Cuevas M D C, et al. Isolation and selection of a highly tolerant microbial consortium with potential for PAH biodegradation from heavy crude oil-contaminated soils[J]. Water, Air, & Soil Pollution, 2014, 225(2): 1826. |
| [13] | Wang B J, Lai Q L, Cui Z S, et al. A pyrene-degrading consortium from deep-sea sediment of the west pacific and its key member Cycloclasticus sp. P1[J]. Environmental Microbiology, 2008, 10(8): 1948-1963. DOI:10.1111/emi.2008.10.issue-8 |
| [14] | Cheung P Y, Kinkle B K. Mycobacterium diversity and pyrene mineralization in petroleum-contaminated soils[J]. Applied and Environmental Microbiology, 2001, 67(5): 2222-2229. DOI:10.1128/AEM.67.5.2222-2229.2001 |
| [15] | Ma J, Xu L, Jia L Y. Characterization of pyrene degradation by Pseudomonas sp. strain Jpyr-1 isolated from active sewage sludge[J]. Bioresource Technology, 2013, 140: 15-21. DOI:10.1016/j.biortech.2013.03.184 |
| [16] | Song X H, Xu Y, Li G M, et al. Isolation, characterization of Rhodococcus sp. P14 capable of degrading high-molecular-weight polycyclic aromatic hydrocarbons and aliphatic hydrocarbons[J]. Marine Pollution Bulletin, 2011, 62(10): 2122-2128. DOI:10.1016/j.marpolbul.2011.07.013 |
| [17] | Zhong Y, Luan T G, Lin L, et al. Production of metabolites in the biodegradation of phenanthrene, fluoranthene and pyrene by the mixed culture of Mycobacterium sp. and Sphingomonas sp.[J]. Bioresource Technology, 2011, 102(3): 2965-2972. DOI:10.1016/j.biortech.2010.09.113 |
| [18] |
张宏波, 林爱军, 刘爽, 等. 芘高效降解菌的分离鉴定及其降解特性研究[J]. 环境科学, 2010, 31(1): 243-248. Zhang H B, Lin A J, Liu S, et al. Identification of pyrene degrading strains and the degrading characteristics research[J]. Environment Science, 2010, 31(1): 243-248. |
| [19] |
王蕾, 聂麦茜, 王志盈, 等. 一株芽胞杆菌和一株黄杆菌代谢芘的摄取方式解析[J]. 环境科学学报, 2009, 29(5): 924-929. Wang L, Nie M Q, Wang Z Y, et al. Mode of metabolic pyrene up take by a Bacillus sp. and a Flavobacterium sp.[J]. Acta Science Circumstantiae, 2009, 29(5): 924-929. |
| [20] |
胡凤钗, 李新宇, 苏振成, 等. 三株降解芘的戈登氏菌鉴定及其降解能力[J]. 应用生态学报, 2011, 22(7): 1857-1862. Hu F C, Li X Y, Su Z C, et al. Identification and degradation capability of three pyrene-degrading Gordonia sp. strains[J]. Chinese Journal of Applied Ecology, 2011, 22(7): 1857-1862. |
| [21] |
许丽, 高振, 罗霂, 等. 一株高效降解芘的细菌分离、鉴定及其降解效果[J]. 微生物学报, 2011, 51(3): 313-319. Xu L, Gao Z, Luo M, et al. Isolation and identification of a pyrene-degrading bacterial strain from contaminated soil[J]. Acta Microbiologica Sinica, 2011, 51(3): 313-319. |
| [22] | Hu Y T, Zhou P J, Zhou Y G, et al. Saccharothrix xinjiangensis sp. nov., a pyrene-degrading actinomycete isolated from Tianchi Lake, Xinjiang, China[J]. International Journal of Systematic and Evolutionary Microbiology, 2004, 54(6): 2091-2094. DOI:10.1099/ijs.0.63143-0 |
| [23] |
谭文捷, 李宗良, 丁爱中, 等. 土壤和地下水中多环芳烃生物降解研究进展[J]. 生态环境, 2007, 16(4): 1310-1317. Tan W J, Li Z L, Ding A Z, et al. Biodegradation of polycyclic aromatic hydrocarbons (PAHs) in soil and groundwater:a review[J]. Ecology and Environment, 2007, 16(4): 1310-1317. |
| [24] | Li F, Zhu L Z, Zhang D. Effect of surfactant on phenanthrene metabolic kinetics by Citrobacter sp. SA01[J]. Journal of Environmental Sciences, 2014, 26(11): 2298-2306. DOI:10.1016/j.jes.2014.09.015 |
| [25] | Mnif S, Chebbi A, Mhiri N, et al. Biodegradation of phenanthrene by a bacterial consortium enriched from Sercina oilfield[J]. Process Safety and Environmental Protection, 2017, 107: 44-53. DOI:10.1016/j.psep.2017.01.023 |
| [26] | Huang X, Shi J, Cui C, et al. Biodegradation of phenanthrene by Rhizobium petrolearium SL-1[J]. Journal of Applied Microbiology, 2016, 121(6): 1616-1626. DOI:10.1111/jam.2016.121.issue-6 |
| [27] | Gao S M, Seo J S, Wang J, et al. Multiple degradation pathways of phenanthrene by Stenotrophomonas maltophilia C6[J]. International Biodeterioration & Biodegradation, 2013, 79: 98-104. |
| [28] | Feng T C, Cui C Z, Dong F, et al. Phenanthrene biodegradation by halophilic Martelella sp. AD-3[J]. Journal of Applied Microbiology, 2012, 113(4): 779-789. DOI:10.1111/jam.2012.113.issue-4 |
| [29] | Meena S S, Sharma R S, Gupta P, et al. Isolation and identification of Bacillus megaterium YB3 from an effluent contaminated site efficiently degrades pyrene[J]. Journal of Basic Microbiology, 2016, 56(4): 369-378. DOI:10.1002/jobm.v56.4 |
| [30] | 姚俊, 喻婵, 靳竞男. 一株枯草芽孢杆菌对芘的降解特性及代谢途径的研究[J]. 化学与生物工程, 2014, 31(5): 10-12, 27. |
| [31] |
汪汉成, 李文红, 黄艳飞, 等. Biolog GEN Ⅲ微孔板在烟草青枯病、黑胫病生防细菌鉴定中的应用[J]. 中国烟草学报, 2012, 18(5): 51-55. Wang H C, Li W C, Huang Y F, et al. The application of Biolog GEN Ⅲ Microplate in identifying antagonistic bacteria against tobacco bacterial wilt and tobacco black shank[J]. Acta Tabacaria Sinica, 2012, 18(5): 51-55. |
| [32] |
杨建云, 纪春艳, 凌冰, 等. 黄曲条跳甲幼虫致病菌的鉴定及其对黄曲条跳甲的杀虫活性研究[J]. 中国生物防治学报, 2014, 30(3): 434-440. Yang J Y, Ji C Y, Ling B, et al. Isolation and identification of bacteria from Phyllotreta striolata (fabricius) and determination of its insecticidal bioactivity[J]. Chinese Journal of Biological Control, 2014, 30(3): 434-440. |
| [33] |
徐建红, 李日强, 李红娇, 等. 萘降解菌的分离鉴定以及脱氢酶活性的测定[J]. 山西大学学报(自然科学版), 2013, 36(3): 471-474. Xu J H, Li R Q, Li H J, et al. Isolation and identification of naphthalene-degrading strains and determination of dehydrogenase activity[J]. Journal of Shanxi University (Natural Science Edition), 2013, 36(3): 471-474. |
| [34] | Roach J C M, Levett P N, Lavoie M C. Identification of Streptococcus iniae by commercial bacterial identification systems[J]. Journal of Microbiological Methods, 2006, 67(1): 20-26. DOI:10.1016/j.mimet.2006.02.012 |
| [35] |
宇盛好, 曹慧, 徐斐, 等. 烤鸭中金黄色葡萄球菌生长模型的建立[J]. 食品科学, 2015, 36(13): 154-159. Yu S H, Cao H, Xu F, et al. Predictive models for the growth of staphylococcus aureus in roast duck[J]. Food Science, 2015, 36(13): 154-159. DOI:10.7506/spkx1002-6630-201513029 |
| [36] | Park H S, Sung B Y, Ryu K. Predictive model for growth of Staphylococcus aureus in blanched spinach with seasoning[J]. Journal of the Korean Society for Applied Biological Chemistry, 2012, 55(4): 529-533. DOI:10.1007/s13765-012-2061-1 |
| [37] | Van Assche A, Álvarez-Pérez S, De Breij A, et al. Phylogenetic signal in phenotypic traits related to carbon source assimilation and chemical sensitivity in Acinetobacter species[J]. Applied Microbiology and Biotechnology, 2017, 101(1): 367-379. DOI:10.1007/s00253-016-7866-0 |
| [38] | Safronova V I, Kuznetsova I G, Sazanova A L, et al. Bosea vaviloviae sp. nov., a new species of slow-growing rhizobia isolated from nodules of the relict species Vavilovia formosa (Stev.) Fed[J]. Antonie van Leeuwenhoek, 2015, 107(4): 911-920. DOI:10.1007/s10482-015-0383-9 |
| [39] | Liang L, Song X H, Kong J, et al. Anaerobic biodegradation of high-molecular-weight polycyclic aromatic hydrocarbons by a facultative anaerobe Pseudomonas sp. JP1[J]. Biodegradation, 2014, 25(6): 825-833. DOI:10.1007/s10532-014-9702-5 |
| [40] | Cui C Z, Ma L, Shi J, et al. Metabolic pathway for degradation of anthracene by halophilic Martelella sp. AD-3[J]. International Biodeterioration & Biodegradation, 2014, 89: 67-73. |
| [41] | Deveryshetty J, Phale P S. Biodegradation of phenanthrene by Pseudomonas sp. strain PPD:purification and characterization of 1-hydroxy-2-naphthoic acid dioxygenase[J]. Microbiology, 2009, 155(9): 3083-3091. DOI:10.1099/mic.0.030460-0 |
| [42] |
姜岩, 杨颖, 张贤明. 典型多环芳烃生物降解及转化机制的研究进展[J]. 石油学报(石油加工), 2014, 30(6): 1137-1150. Jiang Y, Yang Y, Zhang X M. Review on the biodegradation and conversion mechanisms of typical polycyclic aromatic hydrocarbons[J]. Acta Petrolei Sinica (Petroleum Processing Section), 2014, 30(6): 1137-1150. |
| [43] | Qin W, Zhu Y, Fan F Q, et al. Biodegradation of benzo(a)pyrene by Microbacterium sp. strain under denitrification:degradation pathway and effects of limiting electron acceptors or carbon source[J]. Biochemical Engineering Journal, 2017, 121: 131-138. DOI:10.1016/j.bej.2017.02.001 |
| [44] | Prabhu Y, Phale P S. Biodegradation of phenanthrene by Pseudomonas sp. strain PP2:novel metabolic pathway, role of biosurfactant and cell surface hydrophobicity in hydrocarbon assimilation[J]. Applied Microbiology and Biotechnology, 2003, 61(4): 342-351. DOI:10.1007/s00253-002-1218-y |
| [45] | Mallick S, Chakraborty J, Dutta T K. Role of oxygenases in guiding diverse metabolic pathways in the bacterial degradation of low-molecular-weight polycyclic aromatic hydrocarbons:a review[J]. Critical Reviews in Microbiology, 2011, 37(1): 64-90. DOI:10.3109/1040841X.2010.512268 |
| [46] | Woo S H, Park J M. Microbial degradation and enhanced bioremediation of polycyclic aromatic hydrocarbons[J]. Journal of Industrial and Engineering Chemistry, 2004, 10(1): 16-23. |
| [47] | Ishiyama D, Vujaklija D, Davies J. Novel pathway of salicylate degradation by Streptomyces sp. strain WA46[J]. Applied and Environmental Microbiology, 2004, 70(3): 1297-1306. DOI:10.1128/AEM.70.3.1297-1306.2004 |
 2018, Vol. 39
2018, Vol. 39


