2. 中国科学院南京土壤所, 南京 210008;
3. 清华大学环境学院, 北京 100084
2. Institute of Soil Science, Chinese Academy of Sciences, Nanjing 210008, China;
3. School of Environment, Tsinghua University, Beijing 100084, China
环丙沙星(ciprofloxacin, CIP)作为第三代氟喹诺酮类抗生素, 因具有广谱抗菌活性而得到广泛应用[1, 2], 这也导致越来越多的CIP进入环境之中.据报道, 近年来CIP在生活污水[3]、制药废水[4]、农业废水[5, 6]和地表水[7~9]中均被频繁检出.该物质化学性质稳定, 很难被微生物降解[10, 11], 因此其抗菌生物活性在环境中残留时间较长.有研究表明[12~14], CIP的大量使用甚至滥用致使致病菌长期处于亚抑菌浓度中, 致病菌很容易突变产生耐药性.因此, 近年来CIP等新型污染物引起的微污染问题已引起了人们的广泛关注[15].
CIP在环境中持续地迁移扩散, 当达到一定浓度后会对某些敏感微生物的生长产生抑制, 甚至直接杀死它们, 从而改变土著微生物区系和数量, 影响微生物群落结构分布[16].同时, 环境中残留的CIP等氟喹诺酮类抗生素能促进抗性基因(antibiotic resistant genes, ARGs)的产生[17].目前, 在河流、湖泊、土壤、底泥等不同介质中就检出了由于抗生素作用而产生的抗性基因和耐药细菌.抗性基因的传播和扩散可能会加快抗药性菌群的大量繁殖[18], 且细菌抗药性一旦生成就很难在短时间内消失[19], 对微生物群落结构形成了潜在的威胁.抗性基因作为一种新型污染物, 其危害比抗生素本身要大[20], 这给人类的身体健康和生态环境安全带来了不可预测的风险[21, 22].近年来, 围绕着CIP对水或土壤中微生物群落的影响开展了较多的研究[23~25], 但大多集中于CIP痕量水平(ng ·L-1或μg ·L-1级别), 关于高浓度CIP(mg ·L-1水平)对活性污泥中微生物群落结构、功能微生物数量及抗性基因丰度的影响还少有报道.
Xie等[26]针对某抗生素生产废水开展研究, 发现废水中CIP浓度很高, 达到5.06 mg ·L-1, 生化处理对CIP去除效果不佳, 处理出水中残留浓度仍高达3.23 mg ·L-1; 且该系统对有机物和氨氮去除效果较差, 推测是废水中的CIP对活性污泥中功能微生物造成了影响.此外, 本实验室前期研究也发现CIP对膜生物反应器(membrane bioreactor, MBR)运行效能会产生负面影响[27].
本研究旨在通过MBR处理CIP模拟废水实验, 考察不同CIP投加浓度下MBR中微生物群落结构和功能微生物丰度的变化; 探讨CIP-ARGs, 包括gyrA、gyrB、parC和parE在CIP选择压力下的变化情况, 以期为CIP生产废水的无害化处理提供数据支撑.
1 材料与方法 1.1 实验用水MBR进水中含有200 mg ·L-1葡萄糖、256.67 mg ·L-1无水乙酸钠、94.17 mg ·L-1硫酸铵和17.50 mg ·L-1磷酸二氢钾(C :N :P=100 :5 :1), 其COD、氨氮和磷浓度分别约为400、20和4 mg ·L-1.整个实验分为4个工况, 共运行132 d, 运行工况1(1~33 d)、2(34~66 d)、3(67~99 d)、4(100~132 d)条件下分别向上述MBR进水中投加CIP浓度为0、5、10和15 mg ·L-1.所用CIP(上海阿拉丁生化科技股份有限公司)、葡萄糖、无水乙酸钠、硫酸铵、磷酸二氢钾等药剂均为分析纯.
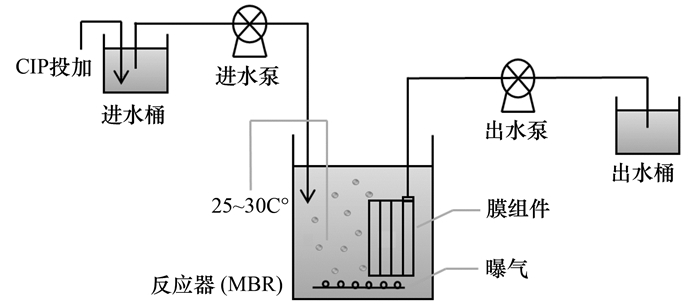
1.2 实验装置与运行条件MBR主要由有机玻璃容器(长方体结构, 有效体积为12 L)、膜组件(日本三菱丽阳株式会社的中空纤维膜, 膜平均孔径为0.1 μm, 膜面积为0.09 m2)和曝气装置组成(图 1).反应器中接种污泥取自某生活污水处理厂好氧膜池, 经稀释水洗和稀释后, 调整反应器中初始MLSS为6.2 g ·L-1.整个MBR装置和进水桶采取遮光处理, 避免CIP发生光解.反应器连续进水, 流速为25 mL ·min-1; 间歇抽吸出水, 每连续出水5 min后停止出水1 min, 以减缓膜污染, 出水流速为30 mL ·min-1.整个实验中, 反应器水力停留时间(HRT)为8 h, 溶解氧为2~4 mg ·L-1, pH为7~8, 水温为25~30℃, COD容积负荷约1.2 kg ·(m3 ·d)-1, 除工况1视情况周期性排泥外其余3个工况几乎没有排泥(检测时所取混合液体积与整个MBR体积相比, 影响甚微, 可忽略此污泥损耗).
|
图 1 实验装置结构示意 Fig. 1 Schematic diagram of MBR |
每个工况下周期性取样分析常规水质指标和CIP的变化情况, 并取污泥样品密封避光保存于-70℃超低温冰箱内, 用于分析微生物群落及其抗性基因的变化.
1.3 分析项目与方法 1.3.1 高通量测序分别于第33 d(运行工况1末)、36 d和66 d(即工况2的第3 d和33 d)、69 d和99 d(即工况3的第3 d和33 d)、102 d和132 d(即工况4的第3 d和33 d)采取污泥样品, 委托生工生物工程(上海)股份有限公司进行高通量测序.
泥水混合物3 000 r ·min-1离心去上清液, 沉淀固体部分使用E.Z.N.ATM Mag-Bind Soil DNA Kit试剂盒(OMEGA)进行DNA提取, 之后用Qubit®2.0荧光计(Invitrogen)检测DNA浓度, 用琼脂糖凝胶(美国UVP)检测DNA完整性, 用Qubit 2.0 DNA检测试剂盒(Life)对基因组DNA精确定量.PCR扩增采用融合了MiSeq测序平台的V3-V4通用引物341F(CCCTACACGACGCTCTTCCGATCTG(barcode) CCTACGGGNGGCWGCAG)和805R(GACTGGAGTTCCTTGGCACCCGAGAATTCCAGACT ACHVGGGTATCTCTA ATCC).PCR扩增、DNA纯化回收和定量混合等步骤参照文献[28].
1.3.2 抗性基因检测分析采用PowerSoil® DNA Isolation Kit试剂盒(MO BIO)并结合液氮冷冻研磨法对样品的基因组DNA进行提取.PCR的扩增引物如表 1所示[29].PCR反应体系:DNA模板1 μL, 2×Taq酶12.5 μL, 上下游引物(10 μmol ·L-1)各0.5 μL, 10.5 μL ddH2O, 反应体系总体积为25 μL.PCR反应程序:94℃预热5 min, 30个循环:94℃变性30 s, 退火温度(gyrA, gyrB:55℃; parC, parE:57℃; 16S rRNA:60℃)30 s, 72℃延伸30 s, 最后72℃停留7 min.再采用Universal DNA Purification Kit(TIANGEN)试剂盒对CIP-ARGs的PCR产物进行纯化回收, 并委托中国科学院南京土壤研究所对纯化的PCR产物进行定量分析.
|
|
表 1 引物信息 Table 1 Primer information |
1.3.3 其它指标检测
CIP浓度采用高效液相色谱仪HPLC(LC-2010A型, 日本岛津)测定:色谱柱为ODS-2, 5 μm 4.6×250 mm(WondaCract, 日本岛津); 检测器为紫外可见吸收检测器(UV-Vis), 波长为277 nm; 流动相为色谱纯乙腈:水(含0.1%甲酸, 色谱纯)=20 :80(体积比); 流速为0.7 mL ·min-1; 温度为35℃; 进样体积为10 μL.COD、氨氮和MLSS分别采用重铬酸钾法、纳氏试剂分光光度法和重量法[30]测定; TOC采用TOC-VCSN总有机碳分析仪(日本岛津)测定.
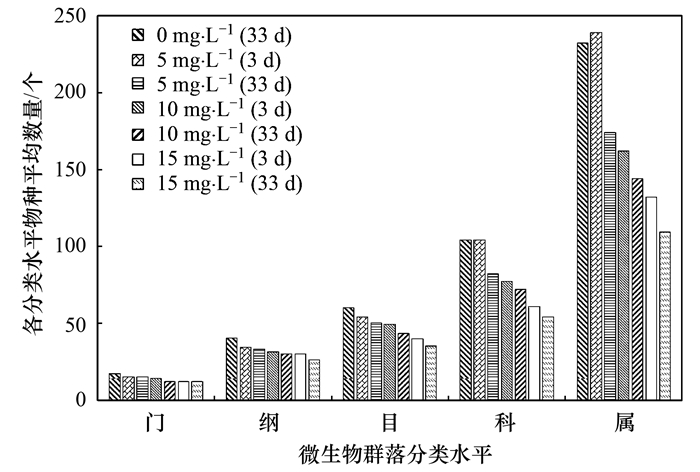
2 结果与讨论 2.1 不同CIP投加浓度下MBR中微生物群落解析 2.1.1 活性污泥的微生物群落结构不同CIP投加浓度下MBR污泥中微生物群落在门、纲、目、科、属等分类水平上的平均种类分布如图 2所示.门、纲、目、科、属等5个分类水平微生物种类数分布大体上都随着CIP投加浓度和运行时间的增加呈现减少的趋势.
|
图 2 各分类水平微生物群落的数量分布 Fig. 2 Distribution of the microbial community at different classification levels |
在工况1下MBR运行稳定时, 微生物群落分析中共检出了17个门、34个纲、60个目、104个科和232个属.
当运行至工况2, MBR进水中CIP浓度为5 mg ·L-1时, 微生物体系突然受到CIP的胁迫, 敏感微生物活性受到抑制而死亡, 使得微生物群落的门、纲和目种类减少; 继续运行至该阶段末, MBR运行逐渐趋于稳定, 此时科和属种类数相较于0 mg ·L-1(33 d)时分别下降了21%和25%.
继续增加MBR进水中CIP浓度至10 mg ·L-1和15 mg ·L-1, 门和纲水平上微生物种类数基本趋于稳定; 但目、科、属水平上微生物种类数却有较大程度减少, 尤其是属水平, 工况4末的属水平种类数相较于工况1末下降了53%.结果显示, CIP的加入对微生物群落的门和纲数量影响不大, 但对目、科、属数量影响较大; 表明了微生物分类单位越小, CIP胁迫所引起的微生物种类变化越明显.
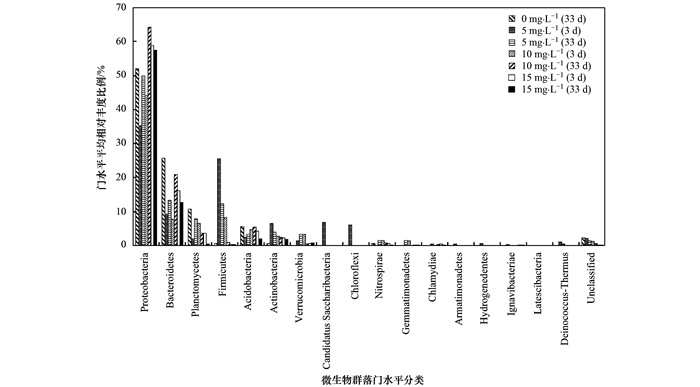
为了深入探究不同CIP浓度对微生物群落结构的影响, 接下来分别从门、科、属水平上进行群落结构分析.门水平上, MBR中微生物群落结构分布如图 3所示.
|
图 3 门水平上微生物群落结构分布 Fig. 3 Distribution of the microbial community at the phylum level |
MBR在工况1中运行稳定时, 微生物群落共检出17个门, 优势菌群为变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、浮霉菌门(Planctomycetes)、酸杆菌门(Acidobacteria)和硝化螺旋菌门(Nitrospirae), 其中变形菌门所占丰度比例最大, 为52%.工况2, MBR进水中开始投加5 mg ·L-1的CIP, 上述优势菌群受到CIP的突然冲击, 其相对丰度比例呈现出不同程度地降低; 厚壁菌门(Firmicutes)、放线菌门(Actinobacteria)、Candidatus Saccharibacteria和绿弯菌门(Chloroflexi)为该工况下新增的优势菌群.
随着运行时间的延长和CIP投加浓度的继续增加, 上述优势菌群的相对丰度比例呈现不同的变化趋势:变形菌门和拟杆菌门的相对丰度比例呈先升高后降低再升高再降低的趋势, 表明这两种菌群对CIP浓度的增加比较敏感; 浮霉菌门、酸杆菌门和硝化螺旋菌门的相对丰度比例均呈先增后减的趋势, 表明这3种菌群能适应一定浓度的CIP环境但在更高浓度的CIP环境中很难生存; 5 mg ·L-1(3 d)时新检出的优势菌群均不能适应CIP的长期胁迫而持续减少, 甚至消失.
在高浓度CIP的长期选择性压力下变形菌门和拟杆菌门成为MBR污泥中的主要菌群, 工况4末二者相对丰度比例分别为57.5%和12.7%, 表明MBR中大部分抗CIP的微生物是属于这两种优势菌群, 这与前人的研究一致[31].
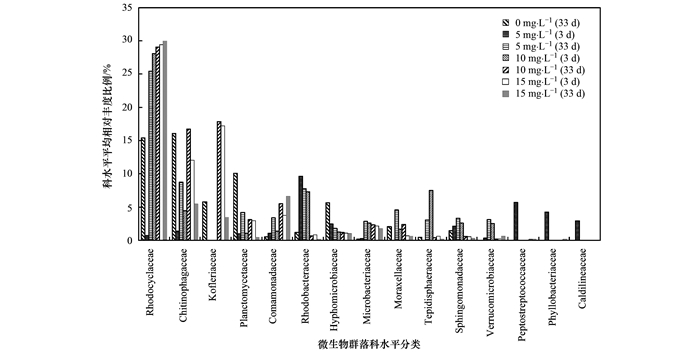
科水平上, MBR中微生物群落结构分布如图 4所示(仅显示整个运行过程中平均相对丰度比例≥2.5%的菌科).
|
图 4 科水平上微生物群落结构分布 Fig. 4 Distribution of the microbial community at the family level |
工况1条件下, MBR中检出的优势菌科有红环菌科(Rhodocyclaceae)、Kofleriaceae、生丝微菌科(Hyphomicrobiaceae)、Chitinophagaceae和浮霉菌科(Planctomycetaceae), 所占比例分别为15.45%、5.80%、5.67%、16.10%和10.13%;前3者均属于变形菌门, 后两者分别隶属于拟杆菌门和浮霉菌门.工况2, 当MBR进水中开始投加CIP时, 这些优势菌科活性和生长受到CIP的抑制, 相对丰度比例均急剧降低; 而红杆菌科(Rhodobacteraceae)、消化链球菌科(Peptostreptococcaceae)和叶杆菌科(Phyllobacteriaceae)比例增大, 成为优势菌科.
当MBR在5~15 mg ·L-1的CIP环境中运行期间, 微生物群落结构产生不同的变化.其中红环菌科逐渐适应CIP环境, 相对丰度比例持续增加; Kofleriaceae、Chitinophagaceae和浮霉菌科的相对丰度比例呈现出先升高后降低再升高再降低的过程, 表现出波动状态, 表明这3种菌科易受到CIP浓度增加的影响, 其中Chitinophagaceae的变化趋势与拟杆菌门完全一致; 生丝微菌科、红杆菌科、消化链球菌科和叶杆菌科相对丰度比例逐渐降低, 表明这4种菌科经不住CIP的长期胁迫.
面对高浓度CIP长时间的选择性压力, 在工况4末期选择出来的优势菌科为红环菌科、Chitinophagaceae和丛毛单胞菌科(Comamonadaceae), 相对丰度比例分别为29.96%、5.44%和6.60%.
属水平上, MBR中微生物群落结构分布如图 5所示(仅显示整个运行过程中平均相对丰度比例≥2.5%的菌属).MBR进水中未投加CIP时, 污泥中的优势菌属为Azospira、Kofleria、Ferruginibacter、动胶菌属(Zoogloea)和生丝微菌属(Hyphomicrobium), 所占比例分别为13.07%、5.80%、10.41%、7.03%和4.48%.工况2, 当MBR进水中开始投加CIP时, 上述优势菌属相对丰度比例均急剧降低, 表明其活性和生长受到了CIP的抑制; 此时优势菌属为动胶菌属(隶属于红环菌科)、Tissierella(隶属于消化链球菌科)、Acetoanaerobium和Roseicitreum.
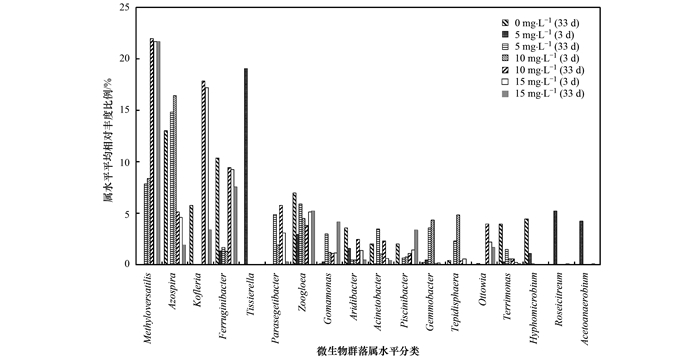
|
图 5 属水平上微生物群落结构分布 Fig. 5 Distribution of the microbial community at the genus level |
由图 5可知, 继续延长CIP作用时间并增加CIP的投加浓度, 微生物群落结构分布产生显著性差异.其中Azospira、Kofleria、Tissierella、生丝微菌属、Acetoanaerobium和Roseicitreum在CIP长期胁迫下很难生存而失去优势; 而Methyloversatilis、Ferruginibacter、动胶菌属和丛毛单胞菌属(Comamonas)在高浓度CIP的选择性压力下成为优势菌属, 工况4末各相对丰度比例分别为21.70%、7.56%、5.24%和4.15%.
门、科、属水平上的微生物群落结构分析表明, 在高浓度CIP的长期胁迫和选择性压力下, 敏感性微生物活性受到抑制而难以生存; 而变形菌门中红环菌科下的Methyloversatilis、Ferruginibacter和动胶菌属及丛毛单胞菌科下的丛毛单胞菌属最终成为了MBR中的优势菌属.
2.1.2 CIP投加浓度对MBR中微生物群落多样性的影响考察CIP投加浓度对MBR中微生物群落丰富度与多样性的影响, 结果如表 2所示.Chao1和ACE指数用来估计微生物群落的丰富度, 而Simpson和Shannon指数代表群落多样性.由表 2可知, 随着MBR进水中CIP投加浓度的增加和反应时间的延长, Chao1指数呈下降趋势, 从0 mg ·L-1(33 d)的1271.7降至15 mg ·L-1(33 d)的469.7, 下降率为63%;ACE指数在整个运行中比较波动, 但整体上呈下降趋势; Simpson指数持续升高, 而Shannon指数持续降低.Chao1、ACE、Shannon指数的降低和Simpson指数的升高表明了MBR污泥中微生物群落受到了CIP的抑制导致部分微生物难以生存而死亡, 使得污泥中微生物群落丰富度和多样性均呈下降趋势, 这与图 2显示的结果一致.
|
|
表 2 CIP投加浓度对微生物群落Chao1、ACE、Simpson和Shannon指数的影响 Table 2 Influence of CIP concentration on Chao1, ACE, Simpson and Shannon indexes |
2.1.3 CIP投加浓度对部分功能微生物群落的影响
考察CIP投加浓度对TOC、氨氮和CIP去除效果的影响(如图 6), 浅析污染物去除率变化与微生物群落结构变化的关系.结合图 5和图 6可知, 随着CIP投加浓度从0 mg ·L-1增加到15 mg ·L-1, TOC平均去除率从97.56%下降至90.84%, 表明CIP对有机物去除有所影响但影响程度不大.众所周知, 菌胶团是活性污泥和生物膜的重要组成部分, 是有机物降解去除的主力军; 动胶菌属作为菌胶团的主要构成体[31], 在CIP长期胁迫下, 其相对丰度比例趋于稳定, 或许TOC去除效果较稳定就与之相关.
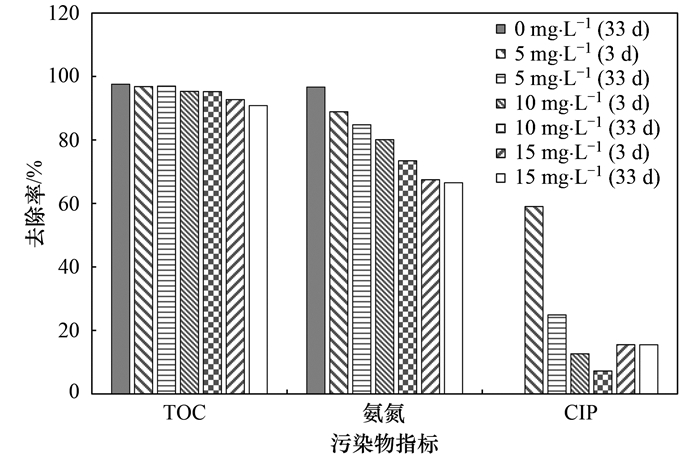
|
图 6 CIP投加浓度对污染物去除率的影响 Fig. 6 Influence of CIP concentration on pollutant removal |
图 5显示, 丛毛单胞菌属也逐渐成为MBR中优势菌属之一, 有研究报道该菌属能利用CIP作为碳源[32]; 但在整个运行期间CIP去除率较低, 除了5 mg ·L-1(3 d)时主要靠污泥吸附去除59.03%的CIP外, 其余时段平均去除率仅为7.24%~24.90%, 表明大部分优势菌种虽可在CIP胁迫下生存和繁殖, 但对CIP利用率不高.
由图 6和图 7可知, 随着CIP投加浓度的增加和运行时间的延长, 亚硝化单胞菌属(Nitrosomonas)和硝化螺旋菌属(Nitrospira)相对丰度呈先降低后升高再降低的趋势; 产碱菌属(Alcaligenes)和硝化杆菌属(Nitrobacter)持续减少, 最后消失.这4种菌属的减少或消失对MBR中氨氮的去除造成了负面影响, 平均去除率由0 mg ·L-1(33 d)的96.67%降低至15 mg ·L-1(33 d)的66.52%.虽然硝化微生物对低浓度CIP(μg ·L-1)可能具有一定的抵抗性和适应性[33], 但在高浓度CIP环境中硝化微生物却很难成为优势菌群, 导致氨氮的去除效率降低.
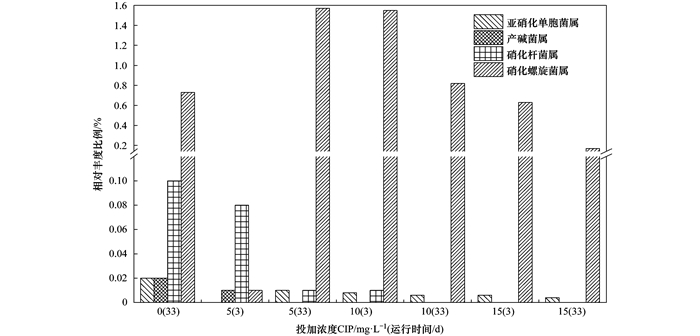
|
图 7 CIP投加浓度对硝化菌属的影响 Fig. 7 Influence of CIP concentration on nitrifying microorganism community |
由图 3~5可知, 在CIP投加浓度为5 mg ·L-1的阶段, MBR中微生物在门、科和属水平上的优势群落结构分布均存在显著性差异.因此选取该阶段为研究对象, 考察CIP-ARGs平均相对丰度随时间的变化情况, 判断ARGs水平是否也存在类似现象, 结果如表 3所示.本研究选取的抗性基因包括DNA促旋酶A亚单位(gyrA)与B亚单位(gyrB)和拓扑异构酶Ⅳ的C亚单位(parC)与E亚单位(parE).为了减少分析检测过程中产生的误差, 采用CIP-ARGs的相对丰度(目标基因拷贝数/ 16S rRNA拷贝数)进行分析[34].表 3显示, CIP的加入对MBR微生物产生了较为显著的影响, 其中内参基因16S rRNA的绝对丰度随CIP暴露时间的增加总体上呈降低趋势, 由3.45×107 copies ·g-1降至33 d的1.68×107 copies ·g-1; 4种CIP-ARGs在运行3 d时, 相对丰度均呈现不同程度的降低, 以gyrA基因降低幅度最大, 原因可能是携带这4种抗性基因的部分菌种对高浓度CIP敏感而被杀死.之后随着运行时间的增加, 这4种CIP-ARGs的相对丰度呈波动状态, 但相较于CIP投加初期均有所增加(parE基因除外), 原因可能在于细菌长期暴露在CIP环境中, 其DNA旋转酶和拓扑异构酶Ⅳ基因的抗性决定区发生基因突变导致细菌对CIP出现抗性作用, 抗药性细菌可能会逐渐被选择成为优势菌群[29], 这与微生物群落结构分布结果相一致.
|
|
表 3 CIP-ARGs在MBR运行中的波动变化(CIP=5 mg ·L-1) Table 3 Fluctuation of CIP-ARGs during the operation of MBR(CIP=5 mg ·L-1) |
3 结论
(1) 通过考察不同CIP投加浓度下MBR污泥中微生物群落结构变化发现, 随着CIP投加浓度和运行时间的增加, 工况4末, 门水平上Proteobacteria和Bacteroidetes仍然为MBR中的优势菌群, 科水平上Rhodocyclaceae、Chitinophagaceae和Comamonadaceae被选择成为优势菌科, 属水平上Methyloversatilis、Ferruginibacter、Zoogloea和Comamonas被选择成为优势菌属.Chao1、ACE、Shannon指数降低和Simpson指数升高表明MBR中微生物丰富度和多样性均降低.高浓度CIP对硝化微生物Nitrosomonas、Nitrospira、Alcaligenes和Nitrobacter的活性具有抑制作用, 导致氨氮去除效果下降.
(2) CIP-ARGs分析表明高浓度CIP的长期胁迫使得MBR中的gyrA、gyrB和parC基因增加.
(3) 本研究仅为CIP对MBR中微生物群落结构特征和抗性基因的影响提供数据支撑, 由于实验规模较小, 未对如何提高耐药菌与抗性基因的削减效果进行探讨, 今后有必要在这一方面, 优化工艺参数与运行条件, 开展放大规模的连续实验, 从技术和经济上进行综合评估.
| [1] |
孙勤寓, 彭逸生, 刘玉, 等. 抗生素环丙沙星(CIP)在两种红树林湿地中的残留及迁移特征[J]. 环境科学学报, 2017, 37(3): 1057-1064. Sun Q Y, Peng Y S, Liu Y, et al. Residues and migration characteristics of antibiotics ciprofloxacin (CIP) in two mangrove wetlands[J]. Acta Scientiae Circumstantiae, 2017, 37(3): 1057-1064. |
| [2] | Zhang Q Q, Ying G G, Pan C G, et al. Comprehensive evaluation of antibiotics emission and fate in the river basins of China:source analysis, multimedia modeling, and linkage to bacterial resistance[J]. Environmental Science & Technology, 2015, 49(11): 6772-6782. |
| [3] | Muñoz I, José G M, Molina-Díaz A, et al. Ranking potential impacts of priority and emerging pollutants in urban wastewater through life cycle impact assessment[J]. Chemosphere, 2008, 74(1): 37-44. DOI:10.1016/j.chemosphere.2008.09.029 |
| [4] | Guney G, Sponza D T. Comparison of biological and advanced treatment processes for ciprofloxacin removal in a raw hospital wastewater[J]. Environmental Technology, 2016, 37(24): 3151-3167. DOI:10.1080/09593330.2016.1179348 |
| [5] |
刘艳萍, 刘鸿雁, 吴龙华, 等. 贵阳市某蔬菜地养殖废水污灌土壤重金属、抗生素复合污染研究[J]. 环境科学学报, 2017, 37(3): 1074-1082. Liu Y P, Liu H Y, Wu L H, et al. Co-contamination of heavy metals and antibiotics in soils under husbandry wastewater irrigation in Guiyang City[J]. Acta Scientiae Circumstantiae, 2017, 37(3): 1074-1082. |
| [6] | 刘畅, 李瑜. 环丙沙星在土壤中的吸附及降解研究进展[J]. 北京农业, 2016(4): 195-199. |
| [7] |
郭睿, 王山军, 昌盛, 等. 嘉兴市饮用水源及城市河网抗生素分布特征[J]. 环境化学, 2016, 35(9): 1842-1852. Guo R, Wang S J, Chang S, et al. The distribution characteristics of antibiotics in Jiaxing drinking water source and urban river[J]. Environmental Chemistry, 2016, 35(9): 1842-1852. DOI:10.7524/j.issn.0254-6108.2016.09.2016020401 |
| [8] | Luo Y, Xu L, Rysz M, et al. Occurrence and transport of tetracycline, sulfonamide, quinolone, and macrolide antibiotics in the Haihe River basin, China[J]. Environmental Science & Technology, 2011, 45(5): 1827-1833. |
| [9] | Zhou L J, Ying G G, Zhao J L, et al. Trends in the occurrence of human and veterinary antibiotics in the sediments of the Yellow River, Hai River and Liao River in northern China[J]. Environmental Pollution, 2011, 159(7): 1877-1885. DOI:10.1016/j.envpol.2011.03.034 |
| [10] | Dorivalgarcía N, Zafra-Gómez A, Navalón A, et al. Removal of quinolone antibiotics from wastewaters by sorption and biological degradation in laboratory-scale membrane bioreactors[J]. Science of the Total Environment, 2013, 442: 317-328. DOI:10.1016/j.scitotenv.2012.10.026 |
| [11] | Liu H, Zhang G P, Liu C Q, et al. The occurrence of chloramphenicol and tetracyclines in municipal sewage and the Nanming River, Guiyang City, China[J]. Journal of Environmental Monitoring, 2009, 11(6): 1199-1205. DOI:10.1039/b820492f |
| [12] | Veldman K, Cavaco L M, Mevius D, et al. International collaborative study on the occurrence of plasmid-mediated quinolone resistance in Salmonella enterica and Escherichia coli isolated from animals, humans, food and the environment in 13 European countries[J]. Journal of Antimicrobial Chemotherapy, 2011, 66(6): 1278-1286. DOI:10.1093/jac/dkr084 |
| [13] |
王小亮, 徐立蒲, 曹欢, 等. 鱼源病原菌对氟喹诺酮类药物的耐药性分析[J]. 中国畜牧兽医, 2013, 40(3): 195-199. Wang X L, Xu L P, Cao H, et al. Analysis of the fluoroquinolone resistance of pathogens isolated from fish[J]. China Animal Husbandry & Veterinary Medicine, 2013, 40(3): 195-199. |
| [14] | Aoike N, Saga T, Sakata R, et al. Molecular characterization of extraintestinal Escherichia coli isolates in Japan:relationship between sequence types and mutation patterns of quinolone resistance-determining regions analyzed by pyrosequencing[J]. Journal of Clinical Microbiology, 2013, 51(6): 1692-1698. DOI:10.1128/JCM.03049-12 |
| [15] |
马驿, 彭金菊, 王芸, 等. 环丙沙星对土壤微生物量碳和土壤微生物群落碳代谢多样性的影响[J]. 生态学报, 2013, 33(5): 1506-1512. Ma Y, Peng J J, Wang Y, et al. Effects of ciprofloxacin on microbial biomass carbon and carbon metabolism diversity of soil microbial communities[J]. Acta Ecologica Sinica, 2013, 33(5): 1506-1512. |
| [16] | Wunder D B, Tan D T, Lapara T M, et al. The effects of antibiotic cocktails at environmentally relevant concentrations on the community composition and acetate biodegradation kinetics of bacterial biofilms[J]. Chemosphere, 2013, 90(8): 2261-2266. DOI:10.1016/j.chemosphere.2012.10.031 |
| [17] | Li J, Wang T, Shao B, et al. Plasmid-mediated quinolone resistance genes and antibiotic residues in wastewater and soil adjacent to swine feedlots:potential transfer to agricultural lands[J]. Environmental Health Perspectives, 2012, 120(8): 1144-1149. DOI:10.1289/ehp.1104776 |
| [18] | Sapkota A R, Curriero F C, Gibson K E, et al. Antibiotic-resistant enterococci and fecal indicators in surface water and groundwater impacted by a concentrated Swine feeding operation[J]. Environmental Health Perspectives, 2007, 115(7): 1040-1045. DOI:10.1289/ehp.9770 |
| [19] | Rodriguez-Mozaz S, Chamorro S, Marti E, et al. Occurrence of antibiotics and antibiotic resistance genes in hospital and urban wastewaters and their impact on the receiving river[J]. Water Research, 2015, 69: 234-242. DOI:10.1016/j.watres.2014.11.021 |
| [20] |
葛伟丽. 长期施用鸡粪农田土壤中典型抗生素耐药细菌及其抗性基因污染分析[D]. 济南: 山东农业大学, 2014. Ge W L. Typical antibiotic resistant bacteria and resistance gene in the farmland soil fertilized with chicken manure chronically[D]. Ji'nan: Shandong Agricultural University, 2014. |
| [21] | Sharma V K, Johnson N, Cizmas L, et al. A review of the influence of treatment strategies on antibiotic resistant bacteria and antibiotic resistance genes[J]. Chemosphere, 2016, 150: 702-714. DOI:10.1016/j.chemosphere.2015.12.084 |
| [22] | Sui Q, Huang J, Deng S B, et al. Occurrence and removal of pharmaceuticals, caffeine and DEET in wastewater treatment plants of Beijing, China[J]. Water Research, 2010, 44(2): 417-426. DOI:10.1016/j.watres.2009.07.010 |
| [23] | Liao X B, Li B X, Zou R S, et al. Biodegradation of antibiotic ciprofloxacin:pathways, influential factors, and bacterial community structure[J]. Environmental Science and Pollution Research, 2016, 23(8): 7911-7918. DOI:10.1007/s11356-016-6054-1 |
| [24] | Gonzalez-Martinez A, Rodriguez-Sanchez A, Martinez-Toledo M V, et al. Effect of ciprofloxacin antibiotic on the partial-nitritation process and bacterial community structure of a submerged biofilter[J]. Science of the Total Environment, 2014, 476-477: 276-287. DOI:10.1016/j.scitotenv.2014.01.012 |
| [25] | Cui H, Wang S P, Fu J, et al. Influence of ciprofloxacin on microbial community structure and function in soils[J]. Biology and Fertility of Soils, 2014, 50(6): 939-947. DOI:10.1007/s00374-014-0914-y |
| [26] | Xie Y W, Chen L J, Liu R. AOX contamination status and genotoxicity of AOX-bearing pharmaceutical wastewater[J]. Journal of Environmental Sciences, 2017, 52: 170-177. DOI:10.1016/j.jes.2016.04.014 |
| [27] |
戴琦, 刘锐, 舒小铭, 等. 环丙沙星对膜生物反应器运行效能的影响及其去除特性[J]. 环境科学, 2018, 39(1): 212-218. Dai Q, Liu R, Shu X M, et al. Removal and influence of ciprofloxacin in a membrane bioreactor[J]. Environmental Science, 2018, 39(1): 212-218. |
| [28] |
魏铭聪. 甲烷渗漏环境中自生矿物形成条件的实验研究[D]. 长春: 吉林大学, 2016. Wei M C. The formation of authigenic minerals during methane seeping in seafloor: insight from laboratory test[D]. Changchun: Jilin University, 2016. |
| [29] | Rubin J, Walker R D, Blickenstaff K, et al. Antimicrobial resistance and genetic characterization of fluoroquinolone resistance of Pseudomonas aeruginosa isolated from canine infections[J]. Veterinary Microbiology, 2008, 131(1-2): 164-172. DOI:10.1016/j.vetmic.2008.02.018 |
| [30] | 国家环境保护总局. 水和废水监测分析方法[M]. 第四版. 北京: 中国环境科学出版社, 2002: 211-213, 277-281. |
| [31] |
何势. 曝气生物滤池中环丙沙星去除行为及其对降解菌群抗药性的诱导作用[D]. 上海: 东华大学, 2016. He S. Removal behavior of ciprofloxacin in the biological aerated filter and its effect on the resistance of bacteria[D]. Shanghai: Donghua University, 2016. |
| [32] | Jung C M, Heinze T M, Strakosha R, et al. Acetylation of fluoroquinolone antimicrobial agents by an Escherichia coli strain isolated from a municipal wastewater treatment plant[J]. Journal of Applied Microbiology, 2009, 106(2): 564-571. DOI:10.1111/jam.2009.106.issue-2 |
| [33] |
何势, 顾超超, 魏欣, 等. 低浓度环丙沙星对曝气生物滤池生物膜硝化过程及硝化微生物的作用影响[J]. 环境科学, 2016, 37(4): 1485-1491. He S, Gu C C, Wei X, et al. Effect of low-concentration ciprofloxacin on the nitrification and nitrifying microorganisms of biofilms in biological aerated filter[J]. Environmental Science, 2016, 37(4): 1485-1491. |
| [34] | Liu L, Liu C X, Zheng J Y, et al. Elimination of veterinary antibiotics and antibiotic resistance genes from swine wastewater in the vertical flow constructed wetlands[J]. Chemosphere, 2013, 91(8): 1088-1093. DOI:10.1016/j.chemosphere.2013.01.007 |
 2018, Vol. 39
2018, Vol. 39


