2. 海洋环境与生态教育部重点实验室, 青岛 266100;
3. 青岛海洋科学与技术国家实验室海洋生态与环境科学功能实验室, 青岛 266071;
4. 中国海洋大学海洋生命学院, 青岛 266003
2. Key Laboratory of Marine Environment and Ecology, Ministry of Education, Qingdao 266100, China;
3. Laboratory for Marine Ecology and Environmental Science, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266071, China;
4. College of Marine Life Science, Ocean University of China, Qingdao 266003, China
海洋沉积物是集化学物质和微生物于一体的特殊生态环境, 广阔的海域面积为微生物提供重要的栖息地, 微生物的分布和代谢活动也影响着沉积物中碳、氮、硫等生源要素的生物地球化学循环.其中硫酸盐还原菌(sulfate-reducing prokaryotes, SRP)和硫氧化菌(sulfur-oxidizing prokaryotes, SOP)是推动海洋硫循环的主要微生物.异化型亚硫酸盐还原酶(dissimilatory sulphite reductase, DSR)在SRP的硫酸盐还原过程中起着关键的催化作用, 其β亚基基因(dsrB)是广泛用于环境样品中SRP分子检测的标记基因[1~4]; soxB基因是硫化物氧化过程中的关键基因, 广泛分布于SOP菌群中[5], 且已被用于多种生境中SOP群落信息的研究[6~9].
20世纪90年代, 相对简便易行的Sanger测序成为第一代测序技术的代表, 以此为基础逐步发展起来的聚丙烯酰胺凝胶(DGGE)技术和克隆文库技术在微生物分子生态学研究中得到了广泛应用.随着测序技术的不断发展, 以Roche 454和Illumina测序平台为主的新一代测序技术(next generation sequencing, NGS), 又称为高通量测序技术, 因其通量高、成本低、准确度高等特点, 已迅速发展成为微生物生态学研究的主要方法, 极大拓宽了环境微生物研究的深度和广度[10~14].
尽管高通量测序技术在测序通量方面具有明显的优势, 但这一技术的出现并不完全排斥第一代测序技术.比较分析不同技术方法的优劣, 是合理进行微生物分子生态学研究的基础.近年来, 已陆续有研究者采用454高通量测序技术、Illumina高通量测序技术和传统分子生物学技术(克隆文库与DGGE技术)对人类粪便[15]、土壤[16]、油田[17]、泥炭藓[18]、生物滤池[19]、烟叶表面[20]、白酒[21, 22]等环境中的微生物群落结构进行了分析, 比较了不同研究方法在微生物群落结构分析中的适用性, 结果表明高通量测序技术能够更加全面和准确地反映环境中微生物群落信息.此外, 贺惠等(未发表数据)通过克隆文库、454和Illumina高通量测序技术研究了海洋沉积物中多样性水平较低的氨氧化细菌群落组成和多样性, 并对3种技术方法所得结果进行比较分析, 结果表明3种测序方法从优到劣依次为Illumina高通量测序技术、454高通量测序技术、克隆文库技术.整体而言, 上述研究均是针对于基因片段较短或多样性水平较低的某种微生物群落(如氨氧化细菌)进行的研究.而不同研究方法在多样性水平较高和基因片段较长的功能菌群群落信息研究中的适用性却未见报道.
在以往的研究中, 根据dsrB基因序列分析, SRP群落可划分成647个OTUs[1], 与其它微生物类群如氨氧化细菌相比, SRP的物种丰富度和多样性水平相对较高[23].而SOP种类繁多, 功能各异, 分布广泛, 且在系统发育和生理代谢方面也具有较高的多样性[24], 与氨氧化细菌相比, SOP同样属于高多样性水平类群. SoxB基因作为多数SOP类群的标记基因, 其PCR扩增片段长度通常为750 bp[5], 与目前常用于高通量测序的基因片段相比, soxB基因片段相对较长.
本研究以dsrB和soxB为目的基因, 分别采用克隆文库技术、454高通量测序技术和Illumina高通量测序技术研究东海表层沉积物中SRP和SOP群落组成、多样性, 比较不同测序技术在分析SRP和SOP菌群特征中的优劣, 以期为环境中高多样性微生物类群的分子生态学研究提供参考.
1 材料与方法 1.1 样品的采集与保存于2011年7月底至8月初乘“润江”号科研考察船对东海海域进行调查, 用箱式采泥器采集沉积物样品, 采样站点为S31(122.55°E, 29.56°N),见图 1.样品采集后, 刮取表层4 cm样品, 分装于无菌密封袋中, 并保存在-80℃用于基因组DNA提取.

|
图 1 东海海域表层沉积物采样站位 Fig. 1 Sampling site of surface sediments from the East China Sea |
将沉积物解冻混匀后, 利用DNA抽提试剂盒PowerSoil® DNA Isolation Kit(MOBIO, 美国)提取总DNA, 并将得到的DNA保存于-20℃冰箱.
1.3 克隆文库构建及生物信息学分析 1.3.1 克隆文库构建将提取的基因组DNA作为PCR扩增的模板, 使用引物DSR-p2060F(5′-CAA CAT CGT YCA YAC CCA GGG)和DSR-4R(5′-GTGTAG CAG TTA CCG CA)[25]扩增dsrB基因, 长度约380 bp. PCR反应条件为:94℃/10 min; 94℃/30 s, 55℃/40 s, 72℃/1 min, 35个循环; 72℃/10 min.反应结束后用1%琼脂糖凝胶电泳检测扩增结果, 并用DNA胶回收试剂盒(宝生物, 大连)纯化目标产物.采用pMD18-T vector Kit(宝生物, 大连)进行连接, 并转化入大肠杆菌(Escherichia coli)Trans 5α感受态细胞, 克隆在Luria-Bertani培养基上培养.
通过蓝白斑方法筛选出阳性克隆后, 用通用引物M13F(-47)和M13R(-48)进行菌落PCR, 1%琼脂糖凝胶电泳检测插入片段大小, 将随机挑选的含有正确插入片段的100个克隆子送去测序(华大基因, 北京).
1.3.2 生物信息学分析将得到的序列首先提交到GenBank数据库, 利用在线检测工具VecScreen检测嵌合体, 用Mothur软件将去除嵌合体后的有效序列按照大于或等于90%的相似性归类到同一OTU, 筛选出每个OTU的代表序列, 并生成稀释曲线, 计算克隆文库的Chao1、Shannon和覆盖度指数.
1.4 454高通量测序及生物信息学分析 1.4.1 454高通量测序分别使用带barcode的特异引物DSR-p2060F/DSR-4R(引物序列同上)和SoxB693F(5′-ATC GGN CAR GCN TTY CCN TA)/SoxB1445B(5′-CAT GTC NCC NCC RTG YTG)[26]扩增dsrB(~380 bp)和soxB(~750 bp)基因. PCR反应条件为:98℃/5 min; 98℃/30 s, 55℃/40 s, 72℃/1 min, 32个循环; 72℃/7 min.使用1.5%浓度的琼脂糖凝胶电泳检测PCR产物, 并使用胶回收试剂盒(Axygen, AP-GX-250)对PCR产物进行纯化, 然后将PCR纯化产物稀释、等量混合.文库制备完成且检测合格后使用Roche 454 GS FLX+测序仪对dsrB和soxB基因进行扩增子高通量测序(派森诺生物科技有限公司, 上海).
1.4.2 数据质控及分析使用QIIME(Version 1.9.0)对原始序列进行质量过滤.将含模糊碱基、引物错配、碱基质量得分小于20及长度较低的序列等予以剔除, 得到高质量序列.使用Uparse在90%序列相似度下对dsrB和soxB基因高质量序列进行OTU聚类, 并选取每个OTU的代表序列.使用QIIME计算每个样品的Chao1、Shannon和覆盖度指数, 并绘制稀释曲线.
1.5 Illumina高通量测序及生物信息学分析 1.5.1 Illumina高通量测序以基因组DNA为模板, 分别使用带barcode特异性引物和高效高保真的酶及酶和缓冲液(Phusion® High-Fidelity PCR Master Mix with GC Buffer, New England Biolabs)对dsrB基因和soxB基因进行扩增(扩增引物同上). PCR反应条件为:98℃/1 min; 98℃/10 s, 50℃/30 s, 72℃/30 s, 30个循环; 72℃/5 min.将PCR产物等浓度混合后使用2%浓度的琼脂糖凝胶进行电泳检测, 并使用胶回收试剂盒(GeneJET, Thermo Scientific)进行纯化.使用建库试剂盒(NEB Next® UltraTM DNA Library Prep Kit)进行文库构建.文库检测合格后, 使用Illumina HiSeq PE250进行上机测序(诺禾致源生物信息科技有限公司, 北京).由于Illumina平台所得序列有效长度通常在400 bp以内, 而本文所使用soxB基因的引物扩增序列长度较长(~750 bp), 因此本研究仅对该基因两端的序列进行测序, 并不对两端序列进行拼接, 并将该测序过程定义为Illumina单端高通量测序.
1.5.2 数据拼接、质控及分析根据dsrB和soxB基因的barcode序列和PCR扩增引物序列拆分下机数据, 去除引物及barcode序列后使用FLASH(V1.2.7)对dsrB基因的双端序列进行拼接.利用Qiime对拼接后的dsrB基因序列和未经拼接的soxB基因的两端序列分别进行质控.将碱基质量得分小于20的序列及片段长度较低的序列等予以剔除, 得到高质量序列.使用Uparse在90%序列相似度下对dsrB和soxB基因高质量序列进行OTU聚类, 并选取每个OTU的代表序列.使用QIIME计算每个样品的Chao1、Shannon和覆盖度指数, 并绘制稀释曲线.
1.6 基因序列登录号将高通量测序所得dsrB和soxB基因序列上传至NCBI Sequnece Read Archive(SRA)数据库, 登录号分别为SRP101534(dsrB)、SRP101417(soxB/HiSeq)和SRP077086(soxB/454).克隆文库测序所得dsrB基因序列提交至GenBank数据库, 登录号为KC567991~KC568065.
2 结果与分析 2.1 硫酸盐还原菌(SRP)群落结构分子多样性研究技术的比较 2.1.1 硫酸盐还原菌(SRP)群落多样性的比较本研究运用3种测序技术对SRP群落多样性及其组成进行研究.由于本研究中Illumina测序平台所获序列条数远高于目前常用测序通量(5万条序列), 因此从Illumina高通量测序所得序列中随机抽取5万条序列作为原始数据, 与上述3种方法所得序列同时进行比较分析.由表 1可知, 高通量测序技术所获序列条数远高于克隆文库技术, 这为充分揭示环境中SRP群落信息奠定了基础.进一步分析发现, 随着测序通量的增加, 检测到的OTU数随之增加, SRP群落丰富度(Chao1)和多样性指数(Shannon)也随之增加.与此类似, 王勋功等(未发表数据)对长江口邻近海域沉积物微生物群落的比较分析及Huse等[27]在对人类粪便样品微生物群落的研究中也发现, 随着序列条数的增加, OTU数随之增加.此外, 对不同研究方法所得覆盖度(表 1)和样品稀释曲线(图 2)进行分析可知, 454和Illumina高通量测序技术所得覆盖度值高达100%且稀释曲线[图 2(b)~2(d)]已趋于饱和, 说明测序数量足够, 能够较全面反映样品中SRP群落的多样性水平, 而克隆文库技术所得覆盖度为79.57%且稀释曲线[图 2(a)]仍处于上升阶段, 说明该测序深度仅涵盖了样品中的部分SRP类群, 不足以全面反映该样品中SRP群落的多样性水平.
|
|
表 1 不同研究方法所得SRP多样性及序列信息 Table 1 Information for the sequences and diversity of SRP obtained from high-throughput sequencing and the traditional clone library |

|
(a)克隆文库技术所得稀释曲线; (b)454高通量测序技术所得稀释曲线; (c)Illumina高通量测序技术测序通量为210 844时所得稀释曲线; (d)Illumina高通量测序技术测序通量为50 000时所得稀释曲线 图 2 不同研究方法所得硫酸盐还原菌菌群稀释曲线 Fig. 2 Rarefaction curves of the SRP community obtained from high-throughput sequencing and the traditional clone library |
将克隆文库技术、454高通量测序技术和Illumina高通量测序技术(210844)所得dsrB基因序列合并, 按照90%相似性进行OTU聚类, 并利用Venn图分析不同研究技术所得OTUs之间的关系[图 3(a)].此外, 本研究从Illumina高通量测序所得dsrB基因序列中随机抽取5万条序列作为原始数据, 与克隆文库技术和454高通量测序技术所得dsrB基因序列合并, 按照上述分析方法同时进行比较分析[图 3(b)].两种研究结果均表明, Illumina高通量测序技术所得OTUs信息均能完整覆盖克隆文库技术所检测到的所有OTUs, 并能覆盖82%以上454高通量测序技术所检测到的OTUs. Illumina测序通量为210844时, Illumina和454高通量测序技术各自独有的OTUs个数分别是333和12个; Illumina测序通量为5万时, Illumina和454高通量测序技术各自独有的OTUs个数分别是163和21个; 进一步分析中发现所有独有OTU的相对丰度均小于1%.由此可见, 两种测序深度下, Illumina高通量测序技术能够检测到更多的SRP类群, 特别是低丰度SRP类群.此外, Illumina测序通量为210844时[图 3(a)], 3种研究方法所检测到的OTUs共有23个, 且其丰度所占比例分别是82.02%(克隆文库)、78.37%(454)、57.35%(Illumina_210844); Illumina测序通量为5万时, 3种研究方法所检测到的OTUs共有20个, 且其丰度所占比例分别是81.40%(克隆文库)、78.15%(454)、57.95%(Illumina_50000).

|
所用研究方法为克隆文库技术、454高通量测序技术和Illumina高通量测序技术, 其中(a)、(b)中IIllumina高通量测序技术的测序通量分别为210844和50000; X:克隆文库技术; Y:454高通量测序技术; Z:Illumina高通量测序技术 图 3 不同研究方法所得OTUs分布Venn图 Fig. 3 Venn graph of OTUs obtained from high-throughput sequencing and the traditional clone library |
分别筛选出3种研究方法所得优势OTUs类群[将高通量测序所得序列丰度低于1%的OTU及克隆文库所得序列数低于2(即相对丰度低于2.25)的OTU归为others], 并将这些优势类群OTU的代表序列与已知SRP进行比对及注释, 进一步分析不同研究方法所得SRP群落组成差异.在科分类水平上(图 4), 3种测序技术的结果均表明该研究海域SRP优势类群为互营杆菌科(Syntrophobacteracea)、互营菌科(Syntrophaceae)、Desulfobulbaceae、脱硫杆菌科(Desulfobacteraceae), 但不同研究方法所得优势类群的相对丰度并不一致.
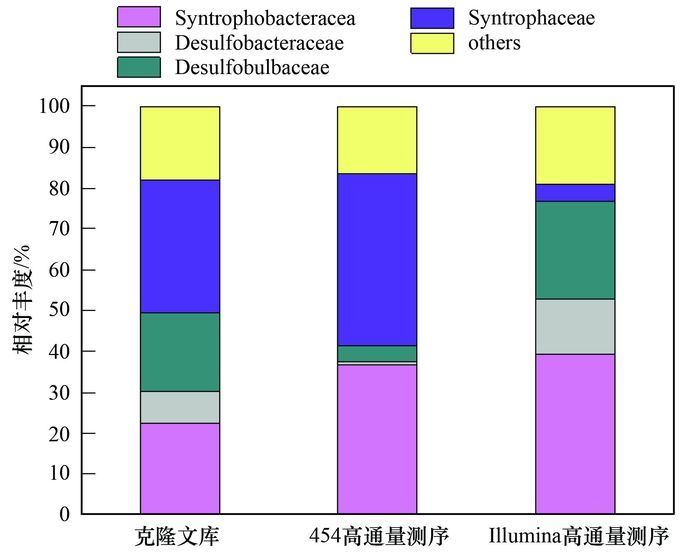
|
others:将高通量测序所得序列相对丰度低于1%的OTU及克隆文库所得序列数低于2(即相对丰度低于2.25)的OTU归为Others 图 4 科分类水平上SRP的相对丰度分析 Fig. 4 Relative abundance of SRP at the family level |
在OTU水平上(表 2), 克隆文库技术检测到的主要优势类群分别是OTU1(21.35%)和OTU90(20.22%), 其中OTU1在454高通量测序技术和Illumina高通量测序技术的结果中也是主要的优势菌群, 且相对丰度分别高达36.57%(454)和35.00%(Illumina).但OTU90在两种高通量测序技术中所检测到的丰度均小于1%, 说明克隆文库技术可能高估了该类群丰度.对于高通量测序技术中所检测到的一些主要类群, 如OTU5、OTU9、OTU16、OTU09、OTU18、OTU19、OTU42、OTU41和OTU45在克隆文库技术中并未检测到, 这可能是由于该技术的测序通量较低, 测序结果存在一定的随机性, 从而不能准确反映SRP群落组成及丰度状况.此外, 454和Illumina高通量测序技术所检测到的SRP菌群也存在一定差异, 如在454高通量测序技术中检测到的部分SRP优势类群, 如OTU10(33.28%)、OTU41(1.90%)、OTU19(1.81%), 在Illumina高通量测序技术中也有发现, 但所检测到的相对丰度均小于1%;在Illumina高通量测序技术中检测到的主要SRP类群(OTU2、OTU4、OTU28和OTU7)则在454高通量测序技术并未检测到.
|
|
表 2 OTU分类水平上SRP群落组成的相对丰度分析1) Table 2 Relative abundance analysis of the SRP community structure at the OTU level |
2.2 硫氧化菌(SOP)群落分子多样性研究技术的比较 2.2.1 硫氧化菌(SOP)群落多样性的比较
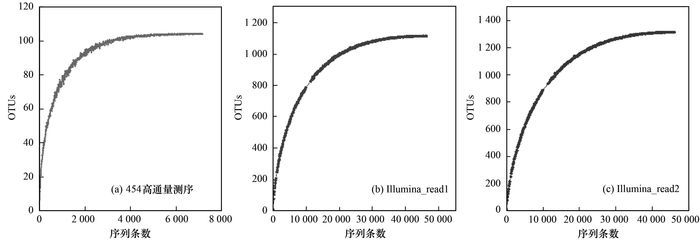
针对SOP的关键功能基因soxB, 通过454和Illumina单端高通量测序技术对SOP群落组成及多样性进行比较分析.不同研究方法所得SOP群落多样性及序列信息如表 3所示. 454高通量测序技术所得序列长度明显高于Illumina单端高通量测序技术, 而Illumina单端高通量测序技术的测序通量远高于454高通量测序技术, 且其所检测到的SOP丰富度及多样性指数也均高于454高通量测序技术.由不同测序平台所得的稀释曲线(图 5)及覆盖度可知, 两种测序技术均能完整有效地反映出样品中SOP的多样性信息.
|
|
表 3 不同研究方法所得硫氧化菌群落多样性及序列信息 Table 3 Information regarding the sequences and diversity of SOP obtained from high-throughput sequencing and the traditional clone library |
|
(a)454高通量测序技术所得稀释曲线; (b)Illumina高通量测序技术所得前端序列(read1)生成的稀释曲线; (c)Illumina高通量测序技术所得后端序列(read2)生成的稀释曲线 图 5 不同研究方法所得硫氧化菌菌群稀释曲线 Fig. 5 Rarefaction curves of the SRP community obtained from high-throughput sequencing and the traditional clone library |
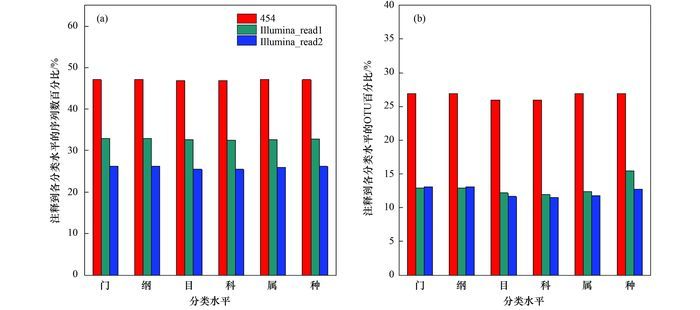
将OTUs代表序列与NCBI(National Center for Biotechnology Information)数据库进行比对注释, 并将两种研究方法检测到的SOP类群进行比较分析(表 4), 结果表明Illumina单端高通量测序方法所检测到的SOP类群更广泛.分析不同研究方法所获序列的物种注释结果发现(图 6), 在454高通量测序结果中, 26.6%的OTUs(丰度为47%)获得了注释信息; 在Illumina单端高通量测结果中, 前端序列(read1)中12.9%的OTUs(丰度为32.7%)获得了注释信息, 末端序列(read2)中12.3%的OTUs(丰度为25.9%)序列获得了注释信息.此外, 两种研究方法所得绝大多数序列(53.0%~74.1%)均未获得注释信息, 并且未得到注释信息的序列既包含丰度较高的优势类群也包含丰度较低的稀有类群.
|
|
表 4 不同研究方法所得硫氧化菌群落信息汇总 Table 4 Taxa information for SOP using different methods |
|
图 6 不同分类水平上获得注释信息序列数和OTU个数百分比 Fig. 6 Percentage of OTUs and abundance at different taxonomic levels |
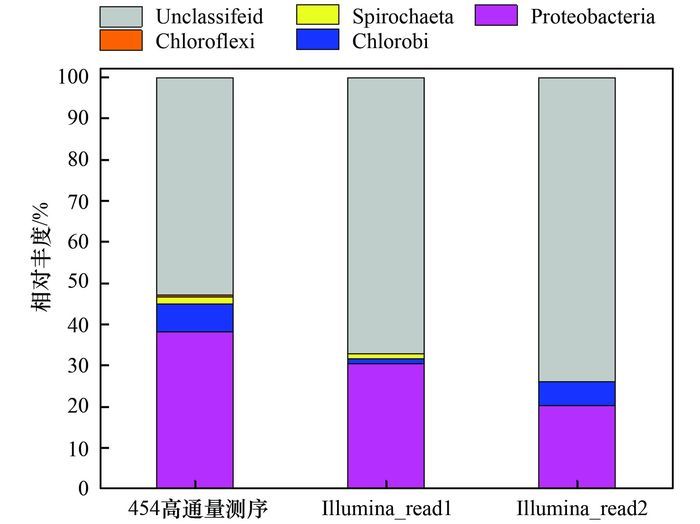
如图 7所示, 在门分类水平上, 两种研究方法均检测到变形菌门(Proteobacteria)、绿菌门(Chlorobi)、螺旋体门(Spirochaeta)和绿弯菌门(Chloroflexi)这4个门类, 其中变形菌门相对丰度最高(20.15%~30.06%), 其它门类相对丰度均较低.变形菌门是由α-Proteobacterium、γ-Proteobacterium和β-Proteobacterium所组成(图 8), 其中α-Proteobacterium在454高通量测序结果中的丰度远高于其在Illumina高通量测序结果中的丰度, 与此相反, γ-Proteobacterium在454高通量测序结果中的丰度远低于其在Illumina高通量测序结果中的丰度.
|
图 7 门分类水平上物种相对丰度 Fig. 7 Relative abundance of species at the phylum level |
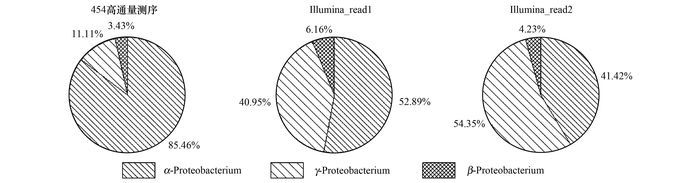
|
图 8 变形菌门纲分类水平上物种相对丰度分布 Fig. 8 Relative abundance of Proteobacteria at the class level |
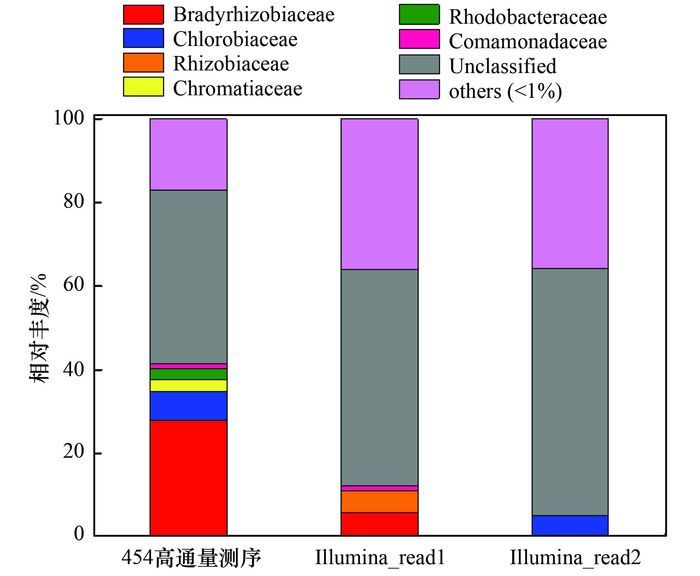
在科分类水平上, 本研究将相对丰度大于1%的SOP类群定义为优势类群, 低于1%的定义为低丰度类群[28].对454和Illumina单端高通量测序所得到的科分类水平上SOP优势类群进行分析(表 5), 结果表明Illumina单端高通量测序结果read1中检测到的优势类群最为丰富(7个), 且454高通量测序技术所检测到的优势类群(5个)在Illumina单端高通量测序结果read1中均有发现, 但Illumina单端高通量序结果read2中所检测到优势类群最少(2个), 且与454高通量测序检测结果以及Illumina高通量测序结果read1差异均较大.
|
|
表 5 不同研究方法所得硫氧化菌优势科信息汇总 Table 5 Information regarding the dominant SOP at the family level obtained by different methods |
对454和Illumina单端高通量测序所得到SOP类群进行OTU水平上的比较分析, 同时将相对丰度大于1%的OTUs定义为优势类群, 低于1%的OTUs统一用others表示, 结果发现454高通量测序共检测到16个优势类群, 是全部OTUs的15.24%, 相对丰度为83.93%; Illumina单端高通量测序所检测到的优势类群分别为12个(read1)和13个(read2), 分别占全部OTUs的1.08%(read1)和0.99%(read2), 相对丰度分别为63.90%(read1)和64.08%(read2).此外, 绝大多数丰度较高的OTUs未获得注释信息, 且两种研究方法所得优势OTUs的分类存在差异(图 9).
|
图 9 硫氧化菌菌群在OTU水平上的相对丰度的分布 Fig. 9 Relative abundance of SOP at the OTU level |
以PCR为基础的DGGE和克隆文库技术是目前常用的微生物分子生态学研究手段, 已经广泛应用于不同生境中SRP和SOP菌群的组成和多样性分析[8, 25, 29~31], 在一定程度上呈现了SRP及SOP菌群的较高多样性及分布的广泛性.但由于克隆文库和DGGE技术所得序列条数通常在几十至一百多之间, 使其对物种丰富度和多样性较高的SRP和SOP菌群的覆盖度较低, 很难全面、真实地反映出环境中SRP和SOP的群落信息.如Jiang等[32]对珠江口沉积物中SRP群落的垂直分布及多样性的研究发现, 利用克隆文库技术所得表层沉积物中dsrAB基因的有效序列仅能覆盖64%的SRP类群; He等[33]和Zhang等[34]对长江口表层沉积物中SRP群落结构的研究发现, 通过克隆文库技术所获得的dsrB基因的有效序列条数分别能覆盖73.49%~87.76%和56%~64%的SRP类群.与此类似, Yousuf等[35]对河口沉积物中SOP群落的研究发现, 通过克隆文库技术所得的aprA基因和soxB基因的序列数分别能覆盖67%~75%和75%~86%的SOP类群. 454和Illumina高通量测序技术所测序列条数通常在几千、甚至几万条以上, 这为全面分析微生物群落信息提供了可能.
在本研究中, 454和Illumina高通量测序技术所获dsrB基因序列条数远远高于克隆文库技术所得的几十条序列, 并且克隆文库技术覆盖度较低(79.57%), 而高通量测序技术覆盖度高达100.00%.此外, 454和Illumina高通量测序技术所检测到的SRP菌群的OTUs数、多样性指数及丰富度指数均高于克隆文库技术.由此可见, 克隆文库技术仅能揭示沉积物中部分SRP菌群的多样性信息, 而高通量测序技术则能更加全面和准确地反映SRP多样性信息.这一结果与以往研究结果相一致[16, 18, 19].值得注意的是, 对于一些多样性水平较低、群落组成相对简单的微生物类群如厌氧氨氧化细菌[36, 37]、氨氧化细菌[38, 39]而言, 克隆文库技术仍是其群落组成和多样性研究的有效方法.
在本研究中, 3种研究方法所检测到的SRP优势类群在门及科分类水平上基本一致, 而在OTU分类水平上差异较大, 如克隆文库技术检测发现OTU90(20.22%)是仅次于OTU1的优势类群, 而在454和Illumina高通量测序技术的检测中的OTU90的相对丰度均小于1%, 说明克隆文库技术可能高估了该类群的丰度.同时, 对454和Illumina高通量测序技术中所检测到的一些主要类群, 在克隆文库技术中并未检测到, 这可能是由于后者的测序通量较低, 且所获序列信息存在一定的随机性, 从而使得部分SRP类群很难被检测到.值得注意的是, 在Illumina高通量测序中检测到的部分主要SRP类群在454高通量测序中并未检测到, 同时在454高通量测序结果中检测到的SRP优势类群OTU10(33.28%)在Illumina高通量测序中其丰度小于1%. Huang等[40]利用16S rRNA基因的454和Illumina高通量测序技术对饮用水中潜在致病菌进行研究发现, 454高通量测序技术并没有检测到低丰度类群, 而测序通量较高的Illumina高通量测序技术可以覆盖低丰度类群.此外, 克隆文库技术、454高通量测序技术和Illumina高通量测序均是基于PCR扩增产物的测序, 而PCR扩增过程也存在一定的偏好性, 这可能也是导致3种研究方法之间差异的原因之一.
整体而言, Illumina高通量测序技术所得OTUs信息能完整覆盖克隆文库技术所检测到的所有OTUs, 并能覆盖82%以上454高通量测序技术所检测到的OTUs. Illumina高通量测序技术所检测到的独有OTUs个数远高于454高通量测序技术, 且所有独有OTU均为低丰度(<1%)类群.由此可见, 对于物种丰富度和多样性水平较高的SRP群落的研究, Illumina高通量测序技术优于454高通量测序技术和克隆文库技术, 特别在低丰度物种的检测方面具有明显优势.
454和Illumina作为目前市场主流的高通量测序平台, 两者各有其优点和不足之处.在测序通量上, Illumina高通量测序技术(通常为5万条序列)优于454高通量测序技术(通常为1万条序列), 而在测序读长上, 454高通量测序技术(通常不高于600 bp)则优于Illumina高通量测序技术(通常不高于400 bp).在本研究中, 454高通量测序技术所得soxB基因平均序列长度(~551 bp)是Illumina单端高通测序所得soxB基因片段长度(~222 bp)的2倍, 而Illumina单端高通测序所得soxB基因序列条数是454高通量测序技术所得soxB基因序列条数的4.5倍.相对较高的测序通量使得Illumina单端高通量测序技术所检测到的SOP的OTUs个数、丰富度及多样性指数也均高于454高通量测序技术, 同时使得Illumina高通量测序方法所检测到的SOP类群更广泛.
本研究中, 在门分类水平上, 454和Illumina单端高通量测序技术所检测到SOP类群相一致, 变形菌门丰度最高(20.15%~30.06%), 绿菌门、绿弯菌门和螺旋体门相对丰度均较低.对科分类水平上SOP优势类群进行分析发现, Illumina单端高通量测序结果read1中检测到的优势类群最为丰富, 且454高通量测序技术所检测到的优势类群在Illumina单端高通量测序结果read1中均有发现, 但Illumina单端高通量测序结果read2与read1及454高通量测序检测结果差异均较大. Claesson等[15]通过454和Illumina高通量测序技术对人体排泄物中细菌群落(16S rRNA基因)组成进行比较分析发现, 在门分类水平上两种研究方法的检测结果基本一致, 而在属分类水平上两种研究方法的检测结果差异较大.此外, Claesson等[15]还发现细菌16S rRNA基因不同扩增区域对高通量测序结果影响要大于不同研究方法对高通量测序结果的影响.与此类似, 在本研究中, soxB基因片段的长度为750 bp, 基于不同研究方法所测基因片段的平均长度可知, 454高通量测序技术所测soxB基因序列是前端551 bp, Illumina单端高通量测序技术所测soxB基因序列分别是前端250 bp(read1)和末端250 bp(read2), 因此, 454高通量测序技术所得序列信息和Illumina单端高通量测序技术的前端序列信息(read1)相似性更高, 因此在群落组成上也更相似.
总体而言, 454高通量测序技术在读长上的优势并未体现出来, 而读长较短, 通量较高的Illumina测序技术在分析SOP群落多样性及物种组成时要优于454高通量测序, 而且Illumina高通量测序技术的前端序列信息(read1)要优于其末端序列信息(read2). Maughan等[41]在利用454和Illumina高通量测序技术对囊胞性纤维症患者的痰样本中细菌群落信息进行比较分析发现, Illumina高通量测序技术在物种注释方面要优于454高通量测序技术; Albertsen等[42]研究也表明Illumina高通量测序平台能够为研究功能基因提供更多的序列信息.此外, 也有报道指出, 尽管454高通量测序平台的测序读长较长, 但其测序错误率是Illumina高通量测序平台的10倍[43, 44]; 且有研究表明, 尽管454高通量测序技术所得基因序列片段较长, 但该长片段包含有高保守性区域, 而这些区域对微生物群落信息的研究并没有很大贡献[41].
4 结论(1) 基于克隆文库技术、454高通量测序技术和Illumina高通量测序技术对高丰富度和高多样性SRP的群落组成及多样性进行比较分析发现, Illumina高通量测序技术优于454高通量测序技术和克隆文库技术, 特别在低丰度物种的检测方面Illumina高通量测序技术具有明显优势.
(2) 以SOP soxB基因作为长片段基因代表, 基于454高通量测序技术和Illumina单端高通量测序技术对SOP群落组成和多样性进行比较分析发现, 454高通量测序技术在读长上的优势并未体现出来, 而Illumina单端高通量测序技术优于454高通量测序技术, 且soxB基因的Illumina高通量测序技术的前端序列信息(read1)要优于末端序列信息(read2).
致谢: “润江”号调查船全体工作人员为调查工作提供了大量帮助和支持, 特此致谢.| [1] | Müller A L, Kjeldsen K U, Rattei T, et al. Phylogenetic and environmental diversity of DsrAB-type dissimilatory (bi) sulfite reductases[J]. The ISME Journal, 2015, 9(5): 1152-1165. DOI:10.1038/ismej.2014.208 |
| [2] | Islamud-Din, El-Latif Hesham A, Ahmad A, et al. PCR-DGGE and real-time PCR dsrB-based study of the impact of heavy metals on the diversity and abundance of sulfate-reducing bacteria[J]. Biotechnology and Bioprocess Engineering, 2014, 19(4): 703-710. DOI:10.1007/s12257-014-0324-x |
| [3] | Pavloudi C, Oulas A, Vasileiadou K, et al. Diversity and abundance of sulfate-reducing microorganisms in a Mediterranean lagoonal complex (Amvrakikos Gulf, Ionian Sea) derived from dsrB gene[J]. Aquatic Microbial Ecology, 2017, 79(3): 209-219. DOI:10.3354/ame01829 |
| [4] | Cui J, Chen X P, Nie M, et al. Effects of spartina alterniflora invasion on the abundance, diversity, and community structure of sulfate reducing bacteria along a successional gradient of coastal salt marshes in China[J]. Wetlands, 2017, 37(2): 221-232. DOI:10.1007/s13157-016-0860-6 |
| [5] | Meyer B, Imhoff J F, Kuever J. Molecular analysis of the distribution and phylogeny of the soxB gene among sulfur-oxidizing bacteria-evolution of the Sox sulfur oxidation enzyme system[J]. Environmental Microbiology, 2007, 9(12): 2957-2977. DOI:10.1111/emi.2007.9.issue-12 |
| [6] | Kojima H, Watanabe T, Iwata T, et al. Identification of major planktonic sulfur oxidizers in stratified freshwater lake[J]. PLoS One, 2014, 9(4): e93877. DOI:10.1371/journal.pone.0093877 |
| [7] | Tourna M, Maclean P, Condron L, et al. Links between sulphur oxidation and sulphur-oxidising bacteria abundance and diversity in soil microcosms based on soxB functional gene analysis[J]. FEMS Microbiology Ecology, 2014, 88(3): 538-549. DOI:10.1111/fem.2014.88.issue-3 |
| [8] | Thomas F, Giblin A E, Cardon Z G, et al. Rhizosphere heterogeneity shapes abundance and activity of sulfur-oxidizing bacteria in vegetated salt marsh sediments[J]. Frontiers in Microbiology, 2014, 5: 309. |
| [9] |
于皓, 陈川, 张莉, 等. 溶解氧对碳氮硫共脱除工艺中微生物群落影响解析[J]. 环境科学, 2013, 34(6): 2368-2374. Yu H, Chen C, Zhang L, et al. Effect of dissolved oxygen on microbial community in simultaneous removal of carbon, nitrogen and sulfur process[J]. Environmental Science, 2013, 34(6): 2368-2374. |
| [10] | Zhu D C, Tanabe S H, Yang C, et al. Bacterial community composition of South China Sea sediments through pyrosequencing-based analysis of 16S rRNA genes[J]. PLoS One, 2013, 8(10): e78501. DOI:10.1371/journal.pone.0078501 |
| [11] | Jiang X T, Peng X, Deng G H, et al. Illumina sequencing of 16S rRNA tag revealed spatial variations of bacterial communities in a mangrove wetland[J]. Microbial Ecology, 2013, 66(1): 96-104. DOI:10.1007/s00248-013-0238-8 |
| [12] |
杨浩, 张国珍, 杨晓妮, 等. 16S rRNA高通量测序研究集雨窖水中微生物群落结构及多样性[J]. 环境科学, 2017, 38(4): 1704-1716. Yang H, Zhang G Z, Yang X N, et al. Microbial community structure and diversity in cellar water by 16S rRNA high-throughput sequencing[J]. Environmental Science, 2017, 38(4): 1704-1716. |
| [13] |
乔沙沙, 周永娜, 柴宝峰, 等. 关帝山森林土壤真菌群落结构与遗传多样性特征[J]. 环境科学, 2017, 38(6): 2502-2512. Qiao S S, Zhou Y N, Chai B F, et al. Characteristics of fungi community structure and genetic diversity of forests in Guandi Mountains[J]. Environmental Science, 2017, 38(6): 2502-2512. |
| [14] |
邸琰茗, 王广煊, 黄兴如, 等. 再生水补水对河道底泥细菌群落组成与功能的影响[J]. 环境科学, 2017, 38(2): 743-751. Di Y M, Wang G X, Huang X R, et al. Effect of reclaimed water on bacterial community composition and function in urban river sediment[J]. Environmental Science, 2017, 38(2): 743-751. |
| [15] | Claesson M J, Wang Q, O'Sullivan O, et al. Comparison of two next-generation sequencing technologies for resolving highly complex microbiota composition using tandem variable 16S rRNA gene regions[J]. Nucleic Acids Research, 2010, 38(22): e200. DOI:10.1093/nar/gkq873 |
| [16] |
夏围围, 贾仲君. 高通量测序和DGGE分析土壤微生物群落的技术评价[J]. 微生物学报, 2014, 54(12): 1489-1499. Xia W W, Jia Z J. Comparative analysis of soil microbial communities by pyrosequencing and DGGE[J]. Acta Microbiologica Sinica, 2014, 54(12): 1489-1499. |
| [17] |
伊丽娜. 新疆油田典型区块内源微生物菌群分析及功能菌群研究[D]. 廊坊: 中国科学院研究生院(渗流流体力学研究所), 2014. Yi L N. Analysis on indigenous microbial community and functional bacteria of typical block in Xinjiang Oilfield[D]. Langfang:University of Chinese Academy of Sciences (Institute of Porous Flow and Fluid Mechanics), 2014. http://cdmd.cnki.com.cn/Article/CDMD-80027-1015646147.htm |
| [18] |
向兴. 神农架大九湖泥炭湿地细菌和古菌多样性研究[D]. 武汉: 中国地质大学, 2015. Xiang X. Biodiversity of bacteria and archaea in Dajiuhu peatland, Shennongjia[D]. Wuhan:China University of Geosciences, 2015. http://cdmd.cnki.com.cn/Article/CDMD-10491-1016061146.htm |
| [19] |
蔡言安, 李冬, 毕学军, 等. 基于不同测序技术的生物群落结构及功能菌分析[J]. 中国环境科学, 2016, 36(6): 1830-1834. Cai Y A, Li D, Bi X J, et al. Analysis of microbial community structure and functional bacteria in a biofilter with different sequencing technologies[J]. China Environmental Science, 2016, 36(6): 1830-1834. |
| [20] |
龚俊, 刘玉配, 李媛媛. 烟叶表面微生物类群两种检测方法的比较研究[J]. 华东师范大学学报(自然科学版), 2016(3): 92-101. Gong J, Liu Y P, Li Y Y. Comparative analysis of microbial communities on tobacco leaves between clone library and high-throughput sequencing[J]. Journal of East China Normal University (Natural Science), 2016(3): 92-101. |
| [21] |
陈玲, 袁玉菊, 曾丽云, 等. 16S rDNA克隆文库法与高通量测序法在浓香型大曲微生物群落结构分析中的对比研究[J]. 酿酒科技, 2015(12): 33-36, 40. Chen L, Yuan Y J, Zeng L Y, et al. 16S rDNA clone library vs. high-throughput sequencing method in the analysis of the microbial communities in Nongxiang Daqu[J]. Liquor-Making Science & Technology, 2015(12): 33-36, 40. |
| [22] |
黄莹娜, 熊小毛, 胡远亮, 等. 基于PCR-DGGE和高通量测序分析白云边酒窖泥细菌群落结构与多样性[J]. 微生物学通报, 2017, 44(2): 375-383. Huang Y N, Xiong X M, Hu Y L, et al. Bacterial community and diversity in pit mud of Baiyunbian liquor analyzed by PCR-DGGE and high-throughput sequencing[J]. Microbiology China, 2017, 44(2): 375-383. |
| [23] | 贺惠. 乳山湾邻近海域沉积物中好氧氨氧化微生物分子生态学研究[D]. 青岛: 中国海洋大学, 2016. |
| [24] | Ghosh W, Dam B. Biochemistry and molecular biology of lithotrophic sulfur oxidation by taxonomically and ecologically diverse bacteria and archaea[J]. FEMS Microbiology Reviews, 2009, 33(6): 999-1043. DOI:10.1111/j.1574-6976.2009.00187.x |
| [25] | Geets J, Borremans B, Diels L, et al. DsrB gene-based DGGE for community and diversity surveys of sulfate-reducing bacteria[J]. Journal of Microbiological Methods, 2006, 66(2): 194-205. DOI:10.1016/j.mimet.2005.11.002 |
| [26] | Petri R, Podgorsek L, Imhoff J F. Phylogeny and distribution of the soxB gene among thiosulfate-oxidizing bacteria[J]. FEMS Microbiology Letters, 2001, 197(2): 171-178. DOI:10.1111/fml.2001.197.issue-2 |
| [27] | Huse H M, Welch D B M, Sogin M L. The Science and Applications of Microbial Genomics[M]. Washington, DC: National Academies Press, 2012: 188-207. |
| [28] | Galand P E, Casamayor E O, Kirchman D L, et al. Ecology of the rare microbial biosphere of the Arctic Ocean[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(52): 22427-22432. DOI:10.1073/pnas.0908284106 |
| [29] | Luo J F, Lin W T, Guo Y. Functional genes based analysis of sulfur-oxidizing bacteria community in sulfide removing bioreactor[J]. Applied Microbiology and Biotechnology, 2011, 90(2): 769-778. DOI:10.1007/s00253-010-3061-x |
| [30] | Guan J, Zhang B L, Mbadinga S M, et al. Functional genes (dsr) approach reveals similar sulphidogenic prokaryotes diversity but different structure in saline waters from corroding high temperature petroleum reservoirs[J]. Applied Microbiology and Biotechnology, 2014, 98(4): 1871-1882. DOI:10.1007/s00253-013-5152-y |
| [31] |
罗丽, 刘永军, 王晓昌. 石油集输系统中硫酸盐还原菌的分布和多样性[J]. 环境科学, 2010, 31(9): 2160-2165. Luo L, Liu Y J, Wang X C. Distribution and diversity of sulfate-reducing bacteria in a crude oil gathering and transferring system[J]. Environmental Science, 2010, 31(9): 2160-2165. |
| [32] | Jiang L J, Zheng Y P, Peng X T, et al. Vertical distribution and diversity of sulfate-reducing prokaryotes in the Pearl River estuarine sediments, southern China[J]. FEMS Microbiology Ecology, 2009, 70(2): 249-262. DOI:10.1111/fem.2009.70.issue-2 |
| [33] | He H, Zhen Y, Mi T Z, et al. Community composition and distribution of sulfate-and sulfite-reducing prokaryotes in sediments from the Changjiang estuary and adjacent East China Sea[J]. Estuarine, Coastal and Shelf Science, 2015, 165: 75-85. DOI:10.1016/j.ecss.2015.09.005 |
| [34] | Zhang Y, Zhen Y, Mi T Z, et al. Molecular characterization of sulfate-reducing bacteria community in surface sediments from the adjacent area of Changjiang Estuary[J]. Journal of Ocean University of China, 2016, 15(1): 107-116. DOI:10.1007/s11802-016-2781-7 |
| [35] | Yousuf B, Kumar R, Mishra A, et al. Unravelling the carbon and Sulphur metabolism in coastal soil ecosystems using comparative cultivation-independent genome-level characterisation of microbial communities[J]. PLoS One, 2014, 9(9): e107025. DOI:10.1371/journal.pone.0107025 |
| [36] | Fu B B, Liu J W, Yang H M, et al. Shift of anammox bacterial community structure along the Pearl Estuary and the impact of environmental factors[J]. Journal of Geophysical Research:Oceans, 2015, 120(4): 2869-2883. DOI:10.1002/2014JC010554 |
| [37] | Hou L J, Zheng Y L, Liu M, et al. Anaerobic ammonium oxidation (anammox) bacterial diversity, abundance, and activity in marsh sediments of the Yangtze estuary[J]. Journal of Geophysical Research:Biogeosciences, 2013, 118(3): 1237-1246. DOI:10.1002/jgrg.v118.3 |
| [38] | Chen Y Y, Zhen Y, He H, et al. Diversity, abundance, and spatial distribution of ammonia-oxidizing β-Proteobacteria in sediments from Changjiang estuary and its adjacent area in East China Sea[J]. Microbial Ecology, 2014, 67(4): 788-803. DOI:10.1007/s00248-013-0341-x |
| [39] | He H, Zhen Y, Mi T Z, et al. Community composition and abundance of ammonia-oxidizing archaea in sediments from the Changjiang estuary and its adjacent area in the East China Sea[J]. Geomicrobiology Journal, 2016, 33(5): 416-425. DOI:10.1080/01490451.2014.986695 |
| [40] | Huang K L, Zhang X X, Shi P, et al. A comprehensive insight into bacterial virulence in drinking water using 454 pyrosequencing and Illumina high-throughput sequencing[J]. Ecotoxicology and Environmental Safety, 2014, 109: 15-21. DOI:10.1016/j.ecoenv.2014.07.029 |
| [41] | Maughan H, Wang P W, Caballero J D, et al. Analysis of the cystic fibrosis lung microbiota via serial Illumina sequencing of bacterial 16S rRNA hypervariable regions[J]. PLoS One, 2012, 7(10): e45791. DOI:10.1371/journal.pone.0045791 |
| [42] | Albertsen M, Hansen L B S, Saunders A M, et al. A metagenome of a full-scale microbial community carrying out enhanced biological phosphorus removal[J]. The ISME Journal, 2012, 6(6): 1094-1106. DOI:10.1038/ismej.2011.176 |
| [43] | Glenn T C. Field guide to next-generation DNA sequencers[J]. Molecular Ecology Resources, 2011, 11(5): 759-769. DOI:10.1111/j.1755-0998.2011.03024.x |
| [44] | Luo C W, Tsementzi D, Kyrpides N, et al. Direct comparisons of Illumina vs. Roche 454 sequencing technologies on the same microbial community DNA sample[J]. PLoS One, 2012, 7(2): e30087. DOI:10.1371/journal.pone.0030087 |
 2018, Vol. 39
2018, Vol. 39


