环烷酸类物质(naphthenic acids, NAs)是石油的天然成分之一, 由分子结构中含有一个或多个饱和环的环烷基直链羧酸组成, 可用通用分子式CnH2n+zO2表示[1].随着世界能源需求的增长, 大量环烷酸类物质随着石油开采、运输及加工等过程进入水体[2]、沉积物[3]和土壤[4]等环境中.其所具有的高稳定、难挥发、半衰期长以及类表面活性剂物质等特点, 使其极易进入并积累于陆生、水生生物体内, 从而进入食物链, 对生态系统和人类健康形成具大的潜在威胁[1, 5]. Scarlett等[6]发现NAs对斑马鱼幼体暴露96 h的半致死浓度为13.1 mg·L-1, 对两栖类豹蛙[7]暴露72 h的NAs半致死浓度仅为4.10 mg·L-1.越来越多的研究证明, 在众多石油污染物中, NAs是除多环芳烃外最具生态毒性的污染物[4, 8].
从20世纪70年代开始, 国内外学者们就NAs的物理化学性质[1~3]、毒性[6, 7]、分析测量手段[4, 5]、降解技术[8, 9]及影响因素[10, 11]等开展了一系列研究, 为了解NAs的生态环境效应, 提供了基础.降解及去除NAs毒性的研究, 主要集中在筛选和利用单一或混合微生物对水环境中NAs的降解[9~14].如Clemente等[9]利用油砂尾矿池微生物对好氧状态下商用NAs的降解潜能进行了探究, 发现油砂尾矿池微生物对NAs具有良好的降解能力, 10 d内降解率达到90%以上, 且培养上清液毒性显著降低. Yue等[13]从油砂尾矿废水中分离出以NAs为唯一碳源的苍白杆菌(Ochrobactrum)、短波单胞菌(Brevundimonas)和芽孢杆菌(Bacillus), 分别用于商用NAs的降解, 发现3种菌均有较强的NAs降解能力.此外, 部分学者还通过结合生物反应器[15]和模拟湿地[16]等工程技术, 进一步研究优化微生物的NAs降解能力.然而, 这些研究主要集中于对水环境中NAs污染物的去除, 较少关注土壤中NAs的降解.实际上, 除海洋石油泄漏外, 大部分的石油污染物都是先进入陆地生态系统, 然后通过地表径流、污水污泥排放等进入水体.因此, 了解外源NAs在土壤中的降解过程, 对于防止NAs在生态系统中的迁移, 控制和管理NAs污染范围, 具有重要的意义.
本文拟通过研究外源NAs在土壤中的降解动力学过程, 探索NAs自然降解潜能及其降解的微生物调控机制, 以期为石油污染土壤的生态修复提供科学支撑支持.
1 材料与方法 1.1 土壤样品本实验所用自然洁净土壤采自北京大学未名湖西岸(N 39°59′, E 116°18′), 土壤的基本理化性质见表 1.其有机质和营养元素含量能保证微生物的正常生长需求.
|
|
表 1 土壤的基本理化性质 Table 1 Essential physiochemical properties of soil |
1.2 化学试剂
本实验所用药剂:商用NAs, 美国Acros Organics公司; MAX小柱(200 mg, 6 mL), 美国Waters公司; 色谱级甲醇, 二氯甲烷, 乙酸乙酯, 均购自美国Fisher公司; 甲酸(色谱纯), 氨水(分析纯), 吡啶, 国药集团化学试剂北京有限公司; 1-EDC-HCl[1-ethyl-3-(3-dimethylaminopropyl) carbodiimide hydrochloride, 1-EDC-HCl], 美国Sigma公司; 2-NPH(2-nitrophenylhydrazine, 2-NPH), ICN生物医学有限公司.
1.3 土壤制备和实验设计土壤在室温条件下自然风干后研磨, 过1 mm尼龙筛备用.为了消除环境改变造成的影响, 预处理后土壤于避光恒温条件下静置两周, 保证微生物群落结构稳定.实验共设置灭菌对照组A和自然降解组B, 每组设置3个重复.预处理土壤作为自然降解组B土壤.灭菌对照组A采用高压灭菌锅进行土壤灭菌.将上述风干研磨过筛后的土壤, 置于高压锅中(121℃)灭菌30 min, 取出置于25℃培养箱避光培养6 h(使土壤中芽孢萌发), 之后再于上述条件下灭菌, 重复3次, 获得灭菌对照组A土壤备用.
根据前期研究[4], 我国主要产油区石油污染土壤中NAs最高污染浓度约180 mg·kg-1左右, 因而本研究设置土壤的NAs污染浓度为180 mg·kg-1.将商用NAs溶解于二氯甲烷中制备成环烷酸-二氯甲烷储备液, 分别均匀加入上述预处理过的土壤中, 使土壤中NAs含量达到180 mg·kg-1, 并于通风橱中搅拌以挥发二氯甲烷.待二氯甲烷完全挥发后, 分别取100 g土壤于50 cm×50 cm培养皿中, 用无菌透气封口膜封口, 置于30℃培养箱中避光培养30 d, 实验期间土壤含水率控制在10%左右并定期搅拌, 每隔5 d搅拌均匀后取样, 样品于-20℃冰箱中保存.
1.4 土壤NAs提取土壤中NAs的提取方法参考文献[4]修改而成:取10 g样品溶于100 mL 0.1 mol·L-1NaOH溶液中, 于70℃水浴锅中静置10 min, 取出于涡旋振荡器振荡10 min, 10 000 r·min-1离心20 min后, 将上清液转移至干净的大试管中.对固体残留物重复以上操作, 合并萃取液.利用固相萃取柱(solid-phase extraction, SPE)对NAs进行萃取. SPE柱经1倍柱体积的甲醇和水活化后, 将萃取液转移至SPE柱内, 控制流速在1 mL·min-1.待萃取液全部通过SPE柱后, 加入6 mL 5%的氨水溶液和6 mL甲醇, 抽真空干燥. SPE柱经纸边固定, 7 000 r·min-1离心20 min后, 分两次向SPE柱内加入5%的甲酸-乙酸乙酯洗脱液, 收集洗脱液, 氮吹干燥后待用.分别制备0、20、40、100和180 mg·kg-1的NAs污染土壤, 按上述方法提取后, 得到了NAs的回收率为(104.38%±9.54%), 满足本研究的分析要求.
1.5 NAs提取物衍生化衍生化方法参考Yen等[17]的研究结果:样品过0.22 μm滤膜后用3 mol·L-1 HCl调节pH到8~10, 取200 μL样品到1.5 mL液相色谱小瓶中, 加入80 μL 2-NPH, 80 μL 1-EDC-HCL, 60℃水浴20 min后加入40 μL KOH, 再次水浴15 min后于-20℃保存以待测定.衍生化试剂的配置参考文献[17]完成.
1.6 NAs衍生物仪器测定及定量分析使用岛津液相色谱仪SPD-10A对NAs衍生物进行分析, 所用色谱柱为迪马C18反向色谱柱(250 mm×4.6 mm×5 μm), 测试及积分条件参考文献[17]修改而成.测试时, 色谱柱保持在30℃恒温条件下, 流速为1.5 mL·min-1, 进样体积为20 μL.
定量分析方法参考文献[17]修改完成.以Acros Organics公司购买的商用NAs为标准物质, 分别配置0、20、40、80、160 mg·L-1的NaOH-NAs溶液进行测定, 以峰面积与浓度建立标准曲线, 结果为:y=650.33x+1694.3(R2=0.996).为确定此方法对土壤中NAs的检测下限, 取不同含量的NAs加入到10 g洁净土壤中, 经提取、萃取、仪器分析后, 确定了此方法的检测下限为12 mg·kg-1.
1.7 土壤DNA提取、扩增及高通量测序分析对实验第0、5和30 d时自然降解组的土样使用Power Soil DNA Isolation Kit(MO BIO Laboratories, Carlsbad, CA)对土壤样品进行总DNA提取, 提取结果经1%琼脂糖凝胶电泳检测合格后, 以微生物总DNA为模板, 采用16S rDNA通用引物338F/806R扩增细菌的V3-V4区. PCR反应条件如下: 95℃ 5 min, 30×(95℃ 40 s, 50℃ 50 s, 72℃ 45 s), 72℃ 10 min. PCR反应采用50 μL体系:10×Pyrobest Buffer 5 μL; 2.5 μmol·L-1 dNTPs 4 μL; 10 μmol·L-1 P1 (338F) 2 μL; 10 μmol·L-1 P2 (806R) 2 μL; 2.5 U·μL-1 Pyrobest DNA Polymerase 0.3 μL; DNA模板3 μL; ddH2O 36.7 μL.
所得PCR产物用AxyPrepDNA凝胶回收试剂盒(AXYGEN公司)纯化, 经1%琼脂糖凝胶电泳检测, 参照电泳初步定量结果, 将PCR产物用QuantiFluorTM-ST蓝色荧光定量系统(Promega公司)进行检测定量, 之后按照每个样本的测序量要求, 进行相应比例的混合.构建MiSeq文库, 通过Illumina MiSeq PE300平台进行测序(委托北京奥维森基因科技有限公司完成).对得到的原始数据进行质量控制, 获得最终用于分析的序列.应用Qiime(quantitative insights into microbial ecology)根据序列相似度获得操作分类单元OTU(operational taxonomic unit), 并采用RDP classifier贝叶斯算法对97%相似水平的OTU代表序列进行分类学分析, 在各个分类水平上统计样品的群落组成.
2 结果与讨论 2.1 外源NAs在土壤中的降解过程测定灭菌对照组和自然降解组中NAs在土壤中的剩余量, 绘制的降解动态见图 1.
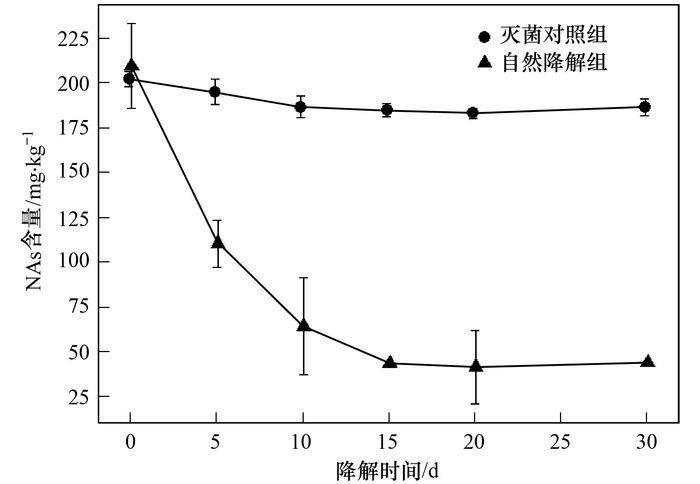
|
图 1 外源NAs在土壤中的降解 Fig. 1 Degradation of exogenous NAs in soil |
虽然灭菌对照组排除了生物降解, 实验期间土壤NAs含量却出现了10%左右的降低.有研究表明, NAs对光照敏感, 且波长为254 nm的紫外线照射能够显著改变NAs的结构组成和毒性[18].考虑到本研究在实验期间采取了恒温避光培养, 其10%的非生物降解可能为其它因素, 如老化作用[19]、有机物螯合等.
自然降解组中NAs的降解活动主要集中在实验前15 d.实验第5 d, 有50%的NAs已经被降解, 到第15 d时, 被降解的NAs达80%, 之后降解活动基本停止, 实验结束, 约有20%的NAs残留在土壤中.污染物在土壤中的自然降解受土壤理化性质、微生物群落组成和污染物化学结构等诸多因素的影响[20].以往的研究显示, 微生物优先降解低环数、低分子量、结构相对较为简单的NAs, 而高环数、高分子量、高度分枝化的NAs则较难被生物利用而残留下来[1, 9, 12, 21].如C原子数低于17的NAs组分相较于高分子量NAs更易被生物降解, 本实验所选用的商用NAs, C原子数在10~25之间.因此, 这部分残留可能为其中较难被生物利用的组分.然而, 现有研究结果主要来源于对水体中NAs的降解, 考虑到土壤颗粒对污染物的吸附[22]及有机污染物在土壤中的老化[19], 本研究中的NAs残留, 也有可能通过氢键结合、静电吸附以及范德华力等作用吸附于土壤颗粒表面或分配进入土壤有机质中[1, 23], 不易解析, 较难被微生物利用, 成为新的持久性难降解有机物.
2.2 NAs降解过程中细菌群落动态变化 2.2.1 NAs污染土壤中细菌群落结构分别提取实验第0、5、30 d时自然降解组土样(标记为day 0、day 5和day 30) 的DNA, 采用Illumina进行高通量测序分析.序列按97%相似性进行聚类, 共得到826个OTU, 分属于25个门, 47个纲, 161个属.在门的分类水平上, 细菌分布在24个已知细菌门.其中, 放线菌门(Actinobacteria)、变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、绿弯菌门(Chloroflexi)、酸杆菌门(Acidobacteria)、疣微菌门(Verrucomicrobia)、浮霉菌门(Planctomycetes)、TM7和厚壁菌门(Firmicutes)9种为优势门类, 其相对丰度之和在3个土样中均占到土壤细菌总量的98%以上.纲水平上, 放线菌门中的放线菌纲(Actinobacteria)、变形菌门中的α变形菌纲(α-Proteobacteria)以及拟杆菌门中的鞘脂杆菌纲(Sphingobacteriia)为3种优势菌纲.而属水平上, 相对丰度最高的3种菌属则分别为未鉴定属、变形菌门中的Skermanella属以及放线菌门中的Blastococcus属.
在这些优势菌群中, 酸杆菌门、疣微菌门、浮霉菌门和厚壁菌门是全球碳循环过程的主要参者[24~26].这些研究显示, 作为石油组成成分之一的NAs, 在自然土壤中具有大量可利用该物质的细菌.
2.2.2 土壤细菌群落的OTU分布统计多个样本中所共有和独有的OTU数目, 可以较为直观地表现出样本OTU数目组成的相似性及重叠情况.为此, 对3个土样在97%的相似水平上进行统计(图 2):共得到826个OTU, 其中共有OTU数目为653, 约为79%, 在OTU总数中占据了绝对优势比例; 不同土样特有OTU数目呈现出较大差异, 降解开始(day 0), 土样中特有OTU数目仅为4种, 降解第5 d(day 5), 特有OTU数目从4种迅速增加到了37种, 占总OTU数量的4.5%, 降解结束(day 30), 土样中特有OTU数目又减少为8种.对土样中的特有OTU进一步分析发现:day 0土样中, 除1种特有OTU属于拟杆菌门外, 其余3种均为变形菌门; 而day 5土样中, 特有OTU分布在未鉴定门、拟杆菌门、变形菌门、TM7、厚壁菌门、疣微菌门、酸杆菌门、迷踪菌门(Elusimicrobia)、蓝藻门(Cyanobacteria)、芽单胞菌门(Gemmatimonadetes)、OD1以及BD1-5共12种门类中, 以未鉴定门(8种OTU)、拟杆菌门(6种OTU)和变形菌门(6种OTU)所包含的特有OTU数目最多; day 30土样中, 特有OTU主要分布在未鉴定门, 且在属水平上均为未鉴定属.
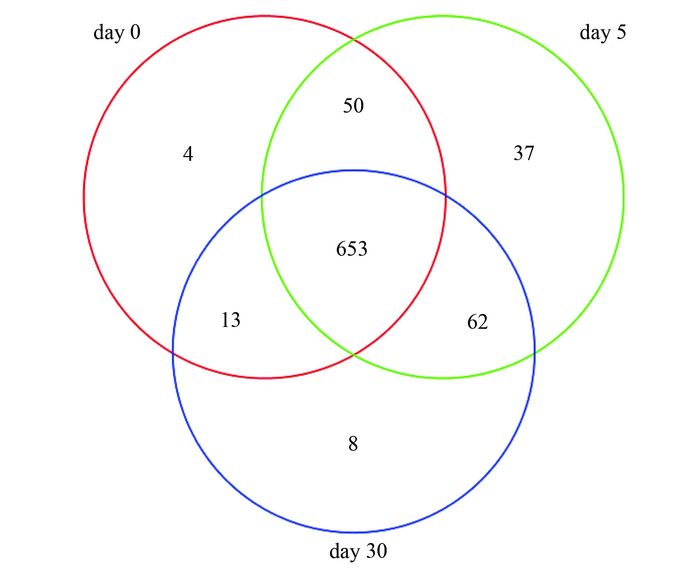
|
图 2 OTU分布Venn图 Fig. 2 Venn diagram showing the distribution of OTU |
实验开始到第5 d, 土壤中的NAs含量减至103 mg·kg-1, 降解量接近50%, 在此期间, 土样中未鉴定门、变形菌门、拟杆菌门等多门类特有OTU数量则迅速增长; 到实验第30 d, NAs含量降至42 mg·kg-1时, 这部分特有OTU则消失, 降解曲线趋于平缓, 说明这些门类中的细菌很可能以NAs为唯一碳源, 是潜在的NAs降解菌.
2.2.3 优势菌群分布及变化具体到微生物类型, 从门水平上看到(图 3), 降解开始和结束时, 第一优势门类均为放线菌门, 而降解第5 d, 第一优势门类则变成变形菌门.与降解开始土样相比, 降解第5 d, 放线菌门相对丰度明显降低, 降幅高达24.8%, 而变形菌门则基本不变.降解结束, 放线菌门和变形菌门相对丰度均升高.
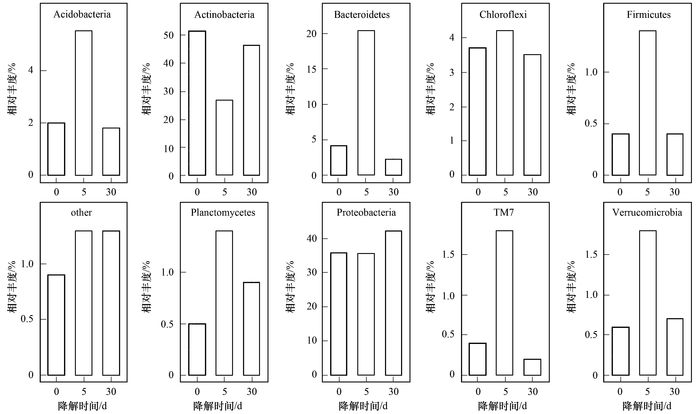
|
图 3 门水平细菌群落结构及分布 Fig. 3 Bacteria community structure and distribution at phylum level |
对放线菌门和变形菌门在纲水平上相对丰度大于1%的菌群进一步分析(图 4), 降解第5 d, 放线菌门在所有纲水平上, 相对丰度均降低, 其中放线菌纲相对丰度的大幅下降是造成放线菌门含量降低的主要原因; 降解结束, 放线菌纲相对丰度又升高为降解初始水平, 其他纲类含量也有不同程度的增长.而变形菌门相对丰度在降解第5 d基本保持不变, 但优势地位由第二优势门类变为第一优势门类, 在纲水平上, 除α变形菌纲相对丰度降低外, 其余变形菌纲相对丰度均增加.降解结束, α变形菌纲和γ变形菌纲(γ-Proteobacteria)相对丰度升高, 而β变形菌纲(β-Proteobacteria)和δ变形菌纲(δ-Proteobacteria)含量则降低, 变形菌门相对丰度整体升高了6.8%.
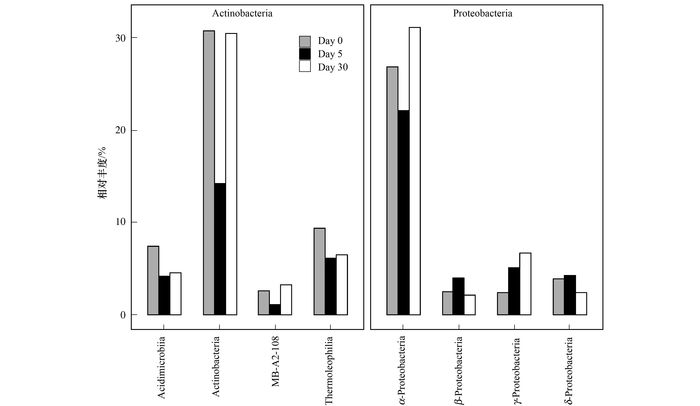
|
图 4 放线菌门和变形菌门在纲水平上的细菌组成 Fig. 4 Bacteria composition of Actinobacteria and Proteobacteria at the class level |
实验开始到第5 d, 土样中的放线菌门在高含量NAs(大于103 mg·kg-1)污染胁迫下, 相对丰度明显降低, 且由第一优势菌门变为第二优势菌门, 而到降解第30 d, NAs污染胁迫减弱时(42 mg·kg-1), 其相对丰度又升高并重新成为第一优势菌门, 表明放线菌门对高含量NAs污染耐受性较差, 而在低含量NAs污染时则可以正常生长繁殖.尽管降解第5 d, 变形菌门在门水平上相对丰度基本不变, 但在纲水平上, β变形菌纲和γ变形菌纲含量则升高.其中, β变形菌纲在高NAs含量(大于103 mg·kg-1)下丰度升高, 低含量(42 mg·kg-1)下则降低, 表明β变形菌纲在高含量NAs污染胁迫下更具竞争优势.而在NAs污染胁迫下, γ变形菌纲则呈现出相对丰度不断增长的趋势.分析NAs降解过程土样中的特有OTU也发现, 降解第5 d土样中出现了属于γ变形菌纲的特有OTU.研究发现, γ变形菌纲细菌具有NAs降解能力[14, 27], 本研究结果进一步说明了, γ变形菌纲对高含量NAs污染具有较强耐受性和潜在的降解能力.
与放线菌门和变形菌门的变化趋势不同, 酸杆菌门、拟杆菌门、绿弯菌门、厚壁菌门、浮霉菌门、TM7、疣微菌门以及其他菌门在降解第5 d, NAs含量高达103 mg·kg-1时, 其相对丰度均增加(图 3).其中酸杆菌门和拟杆菌门增幅最为明显, 分别升高了3.5%和16.1%.而当NAs含量降至42 mg·kg-1时, 各门类相对丰度又降低为降解初始土样水平.
统计酸杆菌门和拟杆菌门在纲和属水平上相对丰度大于1%的菌属, 如表 2所示.酸杆菌门在纲和属水平上, 相对丰度大于1%的菌属中, 分别包含了1种酸杆菌纲(Acidobacteria)和1种Blastocatella属.其中, 酸杆菌纲在降解第5 d, 相对丰度升高了3.4%;而属于酸杆菌纲的Blastocatella属, 其相对丰度在降解第5 d仅增加了1.4%, 说明土样中还存在着大量属于酸杆菌纲的未鉴定属.拟杆菌门在纲水平上包含了鞘脂杆菌纲和噬纤维菌纲(Cytophagia)2种纲, 两者在降解第5 d相对丰度分别升高了10.6%和4.8%;属水平上, 4种菌属相对丰度在降解第5 d均增加, 共升高了6.8%, 远远低于拟杆菌门在门水平上相对丰度的增长.
|
|
表 2 酸杆菌门和拟杆菌门在纲和属水平上的细菌组成 Table 2 Bacteria composition of Acidobacteria and Bacteroidetes at the class and genus level |
有研究发现, 酸杆菌门是优秀的石油降解菌[24], 拟杆菌门则可以降解复杂的复合物[26].该实验中, 土壤添加高含量NAs后, 拟杆菌门和酸杆菌门物种丰度在短时间内出现大幅度升高, 说明这些门中的细菌能够被NAs诱导, 以NAs作为碳源进行细胞增殖.尤其在土壤中NAs含量高达103 mg·kg-1时, 依然表现出活跃的生长, 显示了较高的NAs耐受性, 具有潜在的利用价值.降解第5 d土样中, 酸杆菌门和拟杆菌门中特有OTU的出现, 也证明了这一猜测.
3 结论本文通过向自然洁净土壤添加180 mg·kg-1高含量NAs污染, 探究了外源NAs在土壤中的降解过程及微生物群落动态变化.研究发现:自然洁净土壤对高含量NAs具有较强的降解能力, 15 d后降解量稳定在80%左右, 但NAs含量低于42 mg·kg-1后难以被生物利用; 高含量NAs污染明显改变了土壤细菌群落结构, 土样中特有OTU数目增加, 拟杆菌门、酸杆菌门以及变形菌门中的γ变形菌纲相对丰度短期内明显升高, 是潜在的NAs降解菌.
| [1] | Clemente J S, Fedorak P M. A review of the occurrence, analyses, toxicity, and biodegradation of naphthenic acids[J]. Chemosphere, 2005, 60(5): 585-600. DOI:10.1016/j.chemosphere.2005.02.065 |
| [2] | Holowenko F M, Mackinnon M D, Fedorak P M. Characterization of naphthenic acids in oil sands wastewaters by gas chromatography-mass spectrometry[J]. Water Research, 2002, 36(11): 2843-2855. DOI:10.1016/S0043-1354(01)00492-4 |
| [3] | Wan Y, Wang B L, Khim J S, et al. Naphthenic acids in coastal sediments after the Hebei Spirit oil spill:a potential indicator for oil contamination[J]. Environmental Science & Technology, 2014, 48(7): 4153-4162. |
| [4] | Jie W, Cao X F, Chai L W, et al. Quantification and characterization of naphthenic acids in soils from oil exploration areas in China by GC/MS[J]. Analytical Methods, 2015, 7(5): 2149-2154. DOI:10.1039/C4AY02658F |
| [5] | Demeter M A, Lemire J, George I, et al. Harnessing oil sands microbial communities for use in ex situ naphthenic acid bioremediation[J]. Chemosphere, 2014, 97: 78-85. DOI:10.1016/j.chemosphere.2013.11.016 |
| [6] | Scarlett A G, Reinardy H C, Henry T B, et al. Acute toxicity of aromatic and non-aromatic fractions of naphthenic acids extracted from oil sands process-affected water to larval zebrafish[J]. Chemosphere, 2013, 93(2): 415-420. DOI:10.1016/j.chemosphere.2013.05.020 |
| [7] | Melvin S D, Trudeau V L. Growth, development and incidence of deformities in amphibian larvae exposed as embryos to naphthenic acid concentrations detected in the Canadian oil sands region[J]. Environmental Pollution, 2012, 167: 178-183. DOI:10.1016/j.envpol.2012.04.002 |
| [8] | Giesy J P, Anderson J C, Wiseman S B. Alberta oil sands development[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(3): 951-952. DOI:10.1073/pnas.0912880107 |
| [9] | Clemente J S, MacKinnon M D, Fedorak P M. Aerobic biodegradation of two commercial naphthenic acids preparations[J]. Environmental Science & Technology, 2004, 38(4): 1009-1016. |
| [10] | Yue S Q, Ramsay B A, Wang J X, et al. Biodegradation and detoxification of naphthenic acids in oil sands process affected waters[J]. Science of the Total Environment, 2016, 572: 273-279. DOI:10.1016/j.scitotenv.2016.07.163 |
| [11] | Gunawan Y, Nemati M, Dalai A. Biodegradation of a surrogate naphthenic acid under denitrifying conditions[J]. Water Research, 2014, 51: 11-24. DOI:10.1016/j.watres.2013.12.016 |
| [12] | Folwell B D, McGenity T J, Price A, et al. Exploring the capacity for anaerobic biodegradation of polycyclic aromatic hydrocarbons and naphthenic acids by microbes from oil-sands-process-affected waters[J]. International Biodeterioration & Biodegradation, 2016, 108: 214-221. |
| [13] | Yue S Q, Ramsay B A, Ramsay J A. Biodegradation of naphthenic acid surrogates by axenic cultures[J]. Biodegradation, 2015, 26(4): 313-325. DOI:10.1007/s10532-015-9736-3 |
| [14] | Demeter M A, Lemire J A, Yue G, et al. Culturing oil sands microbes as mixed species communities enhances ex situ model naphthenic acid degradation[J]. Frontiers in Microbiology, 2015, 6: 936. |
| [15] | Islam M S, Zhang Y Y, McPhedran K N, et al. Granular activated carbon for simultaneous adsorption and biodegradation of toxic oil sands process-affected water organic compounds[J]. Journal of Environmental Management, 2015, 152: 49-57. |
| [16] | Toor N S, Franz E D, Fedorak P M, et al. Degradation and aquatic toxicity of naphthenic acids in oil sands process-affected waters using simulated wetlands[J]. Chemosphere, 2013, 90(2): 449-458. DOI:10.1016/j.chemosphere.2012.07.059 |
| [17] | Yen T W, Marsh W P, Mackinnon M D, et al. Measuring naphthenic acids concentrations in aqueous environmental samples by liquid chromatography[J]. Journal of Chromatography A, 2004, 1033(1): 83-90. DOI:10.1016/j.chroma.2004.01.030 |
| [18] | McMartin D W, Headley J V, Friesen D A, et al. Photolysis of naphthenic acids in natural surface water[J]. Journal of Environmental Science and Health, Part A, 2004, 39(6): 1361-1383. DOI:10.1081/ESE-120037839 |
| [19] | An X J, Xiao B H, Di X Y, et al. Research progress on aging of organic pollutants in geosorbents:a review[J]. Acta Geochimica, 2017, 36(1): 27-43. DOI:10.1007/s11631-016-0129-z |
| [20] |
宋宁慧, 单正军, 石利利, 等. 多重环境因子对氟胺磺隆在土壤中降解的影响[J]. 环境科学, 2012, 33(12): 4400-4405. Song N H, Shan Z J, Shi L L, et al. Effects of multiple environmental factors on triflulsulfuron-methyl degradation in soils[J]. Environmental Science, 2012, 33(12): 4400-4405. |
| [21] | Han X M, Scott A C, Fedorak P M, et al. Influence of molecular structure on the biodegradability of naphthenic acids[J]. Environmental Science & Technology, 2008, 42(4): 1290-1295. |
| [22] | Misiti T M. Fate and effect of naphthenic acids in biological systems[D]. Georgia:Georgia Institute of Technology, 2012. https://www.ncbi.nlm.nih.gov/pubmed/23863388 |
| [23] | Janfada A, Headley J V, Peru K M, et al. A laboratory evaluation of the sorption of oil sands naphthenic acids on organic rich soils[J]. Journal of Environmental Science and Health, Part A, 2006, 41(6): 985-997. DOI:10.1080/10934520600620105 |
| [24] | Yu S L, Li S G, Tang Y Q, et al. Succession of bacterial community along with the removal of heavy crude oil pollutants by multiple biostimulation treatments in the Yellow River Delta, China[J]. Journal of Environmental Sciences, 2011, 23(9): 1533-1543. DOI:10.1016/S1001-0742(10)60585-2 |
| [25] | Janssen P H. Identifying the dominant soil bacterial taxa in libraries of 16S rRNA and 16S rRNA genes[J]. Applied and Environmental Microbiology, 2006, 72(3): 1719-1728. DOI:10.1128/AEM.72.3.1719-1728.2006 |
| [26] | Wagner M, Horn M. The Planctomycetes, Verrucomicrobia, Chlamydiae and sister phyla comprise a superphylum with biotechnological and medical relevance[J]. Current Opinion in Biotechnology, 2006, 17(3): 241-249. DOI:10.1016/j.copbio.2006.05.005 |
| [27] | Johnson R J, Smith B E, Rowland S J, et al. Biodegradation of alkyl branched aromatic alkanoic naphthenic acids by Pseudomonas putida KT2440[J]. International Biodeterioration & Biodegradation, 2013, 81: 3-8. |
 2017, Vol. 38
2017, Vol. 38


