2. 清华大学环境学院, 北京 100084;
3. 浙江省水质科学与技术重点实验室, 浙江清华长三角研究院生态环境研究所, 嘉兴 314050;
4. 北京大学环境科学与工程学院, 北京 100871
2. School of Environment, Tsinghua University, Beijing 100084, China;
3. Zhejiang Provincial Key Laboratory of Water Science and Technology, Department of Environmental Technology and Ecology, Yangtze Delta Region Institute of Tsinghua University, Zhejiang, Jiaxing 314050, China;
4. College of Environmental Sciences and Engineering, Peking University, Beijing 100871, China
抗生素抗性能够使抗生素对细菌失效, 一旦致病菌获得抗生素抗性, 可能使目前一些易治愈的常规传染病变得极为致命[1, 2].目前, 大量致病菌抗生素抗性的获得与传播已经对人类健康造成了巨大威胁[3].因此, 抗生素抗性的产生与传播必须关注并加以控制.
生物体使用抗生素后, 首先对肠道微生物造成选择压力[4], 产生的抗生素抗性菌(antibiotic resistance bacteria, ARBs)及未被吸收利用的抗生素随着排泄物进入自然环境或污水处理系统, 而污水处理系统大多将净化后的废水再排入自然环境.如此, 不仅ARBs被输移扩散, 其所携带或释放出的抗生素抗性基因(antibiotic resistance genes, ARGs)还将转移传播.为控制ARGs经废水排放而扩散传播, 对污水处理系统中ARGs赋存特征开展研究十分必要.
目前对污水处理系统中ARGs赋存情况已有不少报道[5~10], 但研究基本上采用0.22 μm滤膜过滤的预处理方法收集DNA, 即关注了细胞态ARGs, 而游离DNA携带ARGs的特征还鲜见报道. ARGs在环境中可通过接合、转化、转导这3种水平基因转移(horizontal gene transfer, HGT)的方式在不同细菌间传播扩散[3]; 其中, 转化正是细菌从环境中直接吸收游离DNA而获得新基因的方式.忽略了对游离ARGs的评估, 污水处理系统ARGs的风险水平可能会被低估.本文选取两座污水处理系统为研究对象, 采用荧光定量PCR技术, 对污水处理系统中的细胞态及游离态ARGs的丰度特征开展分析, 以期为揭示游离态ARGs在污水处理系统中的动态变化, 并评估游离态ARGs对排水受纳环境ARGs传播扩散的潜在风险提供研究积累.
1 材料与方法 1.1 污水处理系统及样品采集选取一座处理规模1 000 m3·d-1、缺氧-好氧膜生物反应器工艺的生活污水处理系统(编号M)和一座处理规模4 800 m3·d-1、缺氧-好氧-好氧-混凝-砂滤组合工艺的焦化废水处理系统(编号C)为研究对象, 工艺流程及采样点位置如图 1所示, 其中MBR工艺中的膜为0.02 μm孔径的中空纤维超滤膜.
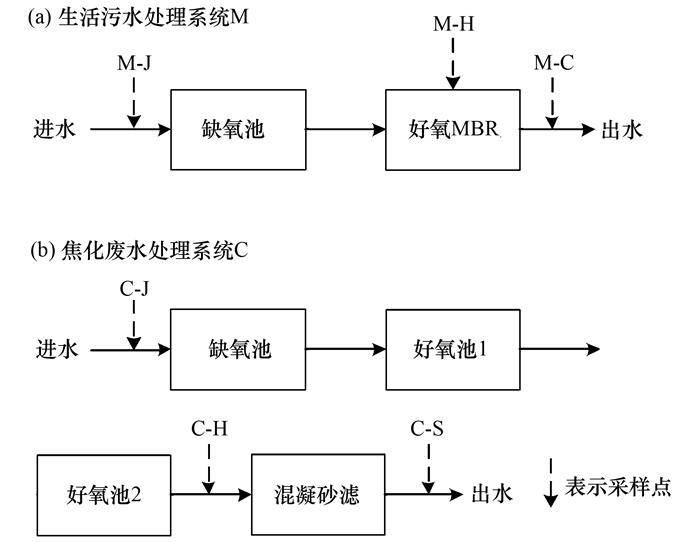
|
图 1 污水处理系统工艺流程及采样点示意 Fig. 1 Treatment process of the wastewater treatment systems and the sampling sites |
在系统M中, 采集了进水(M-J)、好氧池混合液(M-H)、出水(M-C)这3个样品; 在系统C中, 采集了进水(C-J)、好氧池2出水(C-H)、混凝-砂滤出水(C-S)这3个样品.样品采集及保存使用5L聚乙烯桶, 所有器皿在使用前经过10%漂白剂浸泡以去除DNA残留[11], 随后依次使用自来水、去离子水和Millipore超纯水冲洗干净.样品采集后于3 h内运送回实验室, 4℃冰箱保存, 并立即进行水质分析和DNA提取.此外, 将两座污水处理系统的最终出水M-C和C-S于25℃培养箱内避光静置, 对放置5 d和10 d后的系统M出水(M-C-d5和M-C-d10) 及放置5 d后的系统C出水(C-S-d5) 也进行水质分析、DNA提取和ARGs分析.样品水质情况如表 1所示.
|
|
表 1 样品水质情况 Table 1 Water quality of the samples |
1.2 DNA提取 1.2.1 细胞态DNA提取
细胞态DNA的收集采用0.22 μm滤膜截留的方法.根据样品中悬浮物的浓度, 15 mL M-J、0.5 mL M-H、21 mL C-J、15 mL C-H、350 mL C-S和350 mL C-S-d5分别以0.22 μm滤膜过滤, 截留在膜上的悬浮物使用PowerSoil DNA Isolation试剂盒(MoBio, 美国)完成DNA提取.
1.2.2 游离态DNA提取取400 mL样品(M-J、M-H、C-J、C-H、C-S、C-S-d5) 进行30 min的静置, 使悬浮物基本沉于水体底部.移取上清液并由0.22 μm滤膜过滤, 以去除水体中残留的悬浮物和细胞.去除细胞的滤出液采用酒精沉淀法[12, 13]进行游离态DNA的收集:将726 mL无水乙醇和33mL的3M乙酸钠加入到装有330 mL滤出液的蓝盖瓶中, 密封混匀; 将混合溶液置于-20℃冷藏过夜, 再将混合液于10 000 r·min-1离心10 min, 弃除上清液; 收集到的沉淀使用PowerSoil DNA Isolation试剂盒(MoBio, 美国)完成DNA提取.
1.2.3 总DNA提取对于MBR出水样品(M-C、M-C-d5、M-C-d10), 由于只有极少量的悬浮物残留在水体中(浊度 < 0.2 NTU), 认为所含的微生物细胞量很低, 故细胞态DNA没有单独收集提取, 而是与游离态DNA一起提取, 从而获取总DNA.取330 mL未经0.22 μm滤膜过滤的M-C、M-C-d5、M-C-d10, 直接与726 mL无水乙醇和33 mL的3M乙酸钠混合, -20℃冷藏过夜; 离心收集沉淀后, 使用PowerSoil DNA Isolation试剂盒(MoBio, 美国)提取样品总DNA.
所有样品的DNA提取均使用两个生物样品重复, 所获DNA样品由NanoDrop 2000(Thermo Fisher, 美国)测定浓度.
1.3 ARGs的定量测定选取sul Ⅱ(磺胺类)、tetC(四环素类)、blaPSE-1(β-内酰胺类)和ermB(大环内酯类)这4种针对不同抗生素的抗性基因, 对污水处理系统中ARGs的丰度特征进行研究.常规PCR对样品中该4种ARGs定性检测的结果显示:系统M中存在该4种ARGs, 系统C中仅存在sul Ⅱ1种ARG, 因此对系统M样品进行了4种ARGs的荧光定量PCR分析, 对系统C样品进行sul Ⅱ基因的定量分析.
对ARGs的荧光定量PCR测定采用SYBR Green Ⅰ方法, 在CFX-96定量PCR仪(Bio-Rad, 美国)上完成.所使用的引物和升温程序如表 2所示.定量PCR的反应体系为:10 μL SYBRs Premix Ex TaqTM (Takara, 日本), 2.0 μL DNA模板, 各0.4 μL前、后引物, 7.2 μL ddH2O.标准曲线采用10倍梯度稀释的已知浓度标准质粒为模板扩增获得, 标准质粒采用对目标基因进行克隆的方法构建, 具体过程参考Bai等的研究[14].所有标准曲线线性拟合相关系数均>0.98, 扩增效率在93.6%~106.4%之间.定量PCR检测均采样3个测试重复.
|
|
表 2 定量PCR引物及升温程序 Table 2 Primers and thermal programs for real-time PCR |
1.4 统计分析
使用SPSS 17.0对两座污水处理系统不同处理阶段样品中检测到的总ARGs绝对丰度进行ANOVA分析, 采用方法为Student-Newman-Keuls, 显著性水平α=0.05.
2 结果与讨论 2.1 DNA含量共提取了6个细胞态DNA、6个游离态DNA和3个总DNA样品, 提取的DNA质量和计算的样品DNA含量如表 3所示.可知, 提取的DNA在(315.0±84.9)~(13245±480.8) ng之间, 满足后续定量PCR分析的需求.
|
|
表 3 提取的DNA量和样品中DNA的含量 Table 3 Extracted DNA amount and DNA concentration in the samples |
从进水DNA组成看, 细胞态DNA在M-J和C-J中的含量分别为(148.2±33.7) ng·mL-1和(17.4±3.7) ng·mL-1, 而游离态DNA在两个进水中的含量分别为(4.0±0.6) ng·mL-1和(1.0±0.3) ng·mL-1, 可见在废水中存在着一定的游离DNA, 但细胞态DNA是主要的DNA形态.在系统M的混合液样品M-H中, 细胞态DNA含量[(18 290.0±297.0) ng·mL-1)]远远高于其它样品, 这是混合液中高浓度的MLSS所造成的.而M-H及系统C生物处理出水样品C-H中游离态DNA含量均较进水中的游离态DNA含量有大幅上升, 可见生物处理可使废水中游离态DNA的含量有所上升.系统M的膜出水M-C中, 总DNA含量为(1.6±0.7) ng·mL-1, 不仅明显低于膜池混合液M-H中细胞态DNA的含量, 而且大幅低于M-H中游离态DNA的含量[(19.2±1.8) ng·mL-1], 表明MBR的膜过滤作用不仅可以截留池内的活性污泥, 更可有效削减水体中的游离DNA.比较系统C混凝-砂滤前后水体C-H和C-S中的DNA含量发现, C-S中细胞态DNA含量[(10.8±1.8) ng·mL-1]和游离态DNA含量[(3.4±0.2) ng·mL-1]均较C-H中细胞态DNA含量[(89.3±6.6) ng·mL-1]和游离态DNA含量[(40.1±1.5) ng·mL-1]有大幅降低, 说明混凝-砂滤在去除水体中的悬浮颗粒的同时, 也能够有效削减游离态DNA.但混凝-砂滤出水C-S中残留的总DNA量比膜出水M-C中残留的总DNA量高出近8倍, 也说明混凝-砂滤对两种类型DNA的去除能力明显弱于膜过滤.
将两座污水处理系统出水置于培养箱恒温静置避光保存, 检测保存后水体中的DNA含量及ARGs丰度的变化, 为分析污水处理厂出水保存及排放过程中细胞态和游离态ARGs的可能变化提供参考.系统M出水静置后, 水中总DNA含量随着静置时间延长而上升.由于游离DNA无法自我扩增, 这说明水中残留的微生物在不断增殖.系统C出水静置5d后, C-S-d5中细胞态DNA[(7.0±1.0) ng·mL-1]较原出水C-S中细胞态DNA[(10.8±1.8) ng·mL-1]有所下降; 而游离态DNA[(7.6±1.3) ng·mL-1]则比原出水C-S中游离态DNA[(3.4±0.2) ng·mL-1]上升2倍多, 然而两种类型DNA总含量保持一致.说明系统C出水在静置条件下, 微生物不但没有增殖, 反而发生了消亡, 从而导致了细胞裂解和游离态DNA含量的增加, 这可能与系统C出水的水质情况有关.焦化废水含有大量有毒有害和难降解化学物质, 经过生物处理后, 残留于水中的物质可能是一些难生物利用的物质, 并可能存在一定的毒性, 不利于细胞的增殖; 再经过混凝-砂滤处理后, 出水C-S的COD有93.5 mg·L-1, 且残留的活性细胞比例可能也不高.
2.2 ARGs丰度本研究以相对丰度和绝对丰度来考察ARGs丰度.以16S rRNA基因数量作为参照是大量研究中较常用的一种相对丰度评估方式, 但本研究中涉及游离态DNA, 考虑到游离态DNA存在降解的情况, ARGs与16S rRNA基因的降解率未必一致, 而部分降解的基因无法通过定量PCR测得, 但仍能反映在DNA量上, 因此认为以DNA量作为参照能更好地评估基因的相对丰度.所以, 本研究中相对丰度以单位质量DNA所含的ARGs拷贝数(copies·ng-1, 以DNA计, 下同)进行评估, 文中细胞态ARGs的相对丰度为单位细胞态DNA所含的ARGs数量, 游离态ARGs的相对丰度为单位游离态DNA所含的ARGs数量, 总ARGs的相对丰度为单位总DNA所含的ARGs数量; 而绝对丰度为单位体积样品中所含有的ARGs拷贝数(copies·L-1).两座污水处理系统中ARGs的丰度情况如图 2所示.
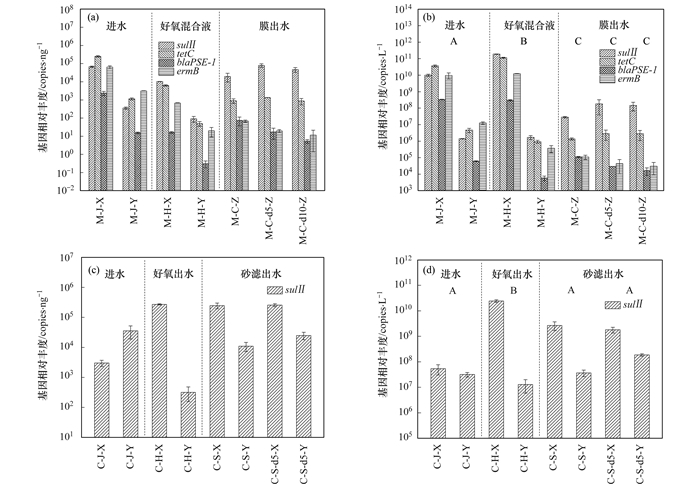
|
(a)系统M中ARGs的相对丰度; (b)系统M中ARGs的绝对丰度; (c)系统C中ARGs的相对丰度; (d)系统C中ARGs的绝对丰度; 横坐标的样品编号中, 末字母X代表细胞态、Y代表游离态、Z代表总(即细胞态+游离态); 图(b)和图(d)中不同的字母代表样品间总ARGs绝对丰度的显著性差异, α=0.05 图 2 两座污水处理系统中ARGs的丰度 Fig. 2 ARGs abundances in two wastewater treatment systems |
从图 2(a)可知, 进水M-J中4种ARGs细胞态的相对丰度为2.3×103~2.5×105 copies·ng-1, 高低顺序为tetC>sul Ⅱ>ermB>blaPSE-1; 在好氧混合液M-H中, 4种ARGs细胞态的相对丰度为1.6×102~1.0×104 copies·ng-1, 较进水中的相对丰度降低1个数量级, 说明在该生活污水处理过程中, ARBs在微生物间竞争中不占优势, 其在微生物群落中的比例有所下降.此外, M-H中4种ARGs细胞态的相对丰度sul Ⅱ>tetC>ermB>blaPSE-1, 说明携带sul Ⅱ基因的ARBs在系统M中比携带tetC基因的ARBs更具竞争优势.
从游离态ARGs看, M-J和M-H中的游离态ARGs相对丰度高低顺序与对应的细胞态ARGs相对丰度高低顺序基本一致, 但均较对应的细胞态ARGs相对丰度低1~2个数量级.推测这主要是由于细胞裂解产生游离态DNA后, 游离态DNA受到吸附和降解作用[19], 使得游离DNA中的基因组成和细胞态DNA基因组成有所差异; 游离态DNA中较低的ARGs相对丰度可能是由于游离的DNA受到了部分降解, 部分基因片段化, 无法通过PCR检测到, 但仍可以通过紫外分光光度计检测到该部分DNA的质量.
膜出水M-C中, 总ARGs的相对丰度范围为6.8×102~1.9×104 copies·ng-1, 与好氧混合液M-H中细胞态ARGs的相对丰度相当, 高于M-H中游离态ARGs的相对丰度, 说明M-C总DNA中仍然含有一定量的细胞态DNA, 使得其总ARGs的相对丰度较高.此外, 表 3数据可以看出, M-C总DNA含量为(1.6±0.7) ng·mL-1, 大幅低于M-H中游离态DNA的含量[(19.2±1.8) ng·mL-1], 显示膜过滤对游离态DNA有较好的截留作用, 那么能透过膜孔而残留在水中的DNA应为较小的片段, 如碎片化的核区/染色体DNA、质粒、噬菌体等.研究已证实质粒可能比核区/染色体DNA编码更多的ARGs[20], 而噬菌体也被认为是ARGs的一个基因库[21], 因此膜的选择透过性可能造成膜出水中游离态ARGs相对丰度的升高.经静置之后, sul Ⅱ和tetC基因的相对丰度较M-C中有小幅上升, 而blaPSE-1和ermB基因的相对丰度则有小幅下降, 这可能与不同ARBs的特性有关.
图 2(b)显示了系统M中ARGs绝对丰度的变化.进水M-J中4种ARGs细胞态的绝对丰度为3.4×108~3.7×1010 copies·L-1, 绝对丰度的高低顺序与相对丰度一致.在好氧混合液M-H中, 虽然细胞态ARGs较进水中的相对丰度低, 但由于混合液MLSS很高, 细胞态ARGs的绝对丰度仍高于进水.出水M-C中总ARGs得到有效的去除, 其绝对丰度与M-H中游离态ARGs接近.系统M对sul Ⅱ、tetC、blaPSE-1和ermB这4种ARGs总的去除率分别为2.54、4.42、3.48和4.95 logs.已报道的生活污水处理系统也表现出较好的ARGs去除能力, 如我国北方两座A2/O污水处理厂对ARGs(细胞态)的去除率为89.9%~99.8%[22], 另一座A2/O及一座三沟式氧化沟污水处理厂对ARGs(细胞态)的去除率分别为1.16~2.44 logs和1.10~2.25 logs[23].去除细胞态ARGs的关键是出水中悬浮污泥的控制, 系统M比上述污水处理系统具有更高的ARGs去除效果, 主要是由于膜对悬浮污泥的截留作用.
2.2.2 系统C中的ARGs丰度图 2(c)显示了系统C中sul Ⅱ基因的相对丰度变化.进水C-J中细胞态sul Ⅱ的相对丰度为3.0×103 copies·ng-1, 显著低于系统M进水中细胞态sul Ⅱ的相对丰度(6.7×104 copies·ng-1), 可见焦化废水与生活污水相比, 不仅检测到的ARGs种类较少, 而且ARGs相对丰度也较低; 然而, 经过生物处理的好氧出水C-H中, 细胞态sul Ⅱ的相对丰度升高至2.7×105 copies·ng-1, 意味着系统C的生物处理工艺对废水中携带sul Ⅱ的ARBs产生富集效应; 混凝-砂滤出水C-S中, 细胞态sul Ⅱ的相对丰度与C-H中基本持平.进水C-J中游离态sul Ⅱ的相对丰度为3.5×104 copies·ng-1, 超过了细胞态sul Ⅱ的相对丰度; 好氧出水C-H中游离态sul Ⅱ的相对丰度为3.2×102 copies·ng-1, 较C-J中下降了两个数量级; 但是, 游离态sul Ⅱ相对丰度在混凝-砂滤出水C-S中有所回升(1.1×104 copies·ng-1), 并且随着出水的静置继续上升(2.5×104 copies·ng-1).
从图 2(d)的绝对丰度看, 进水C-J中细胞态sul Ⅱ为5.4×107 copies·L-1, 略高于游离态sul Ⅱ(3.2×107 copies·L-1).好氧出水C-H中, 细胞态sul Ⅱ绝对丰度上升到2.4×1010 copies·L-1, 游离态sul Ⅱ绝对丰度仍保持在107 copies·L-1数量级; 而混凝-砂滤出水C-S中, 细胞态sul Ⅱ绝对丰度下降了1个数量级, 而游离态sul Ⅱ绝对丰度则小幅上升; 静置5 d后, 水体中细胞态sul Ⅱ绝对丰度继续下降, 而游离态sul Ⅱ则继续上升.与相对丰度及提取的DNA量对照分析, 混凝-砂滤出水中游离态sul Ⅱ上升的原因可能是:游离DNA的降解受到水质、温度等多种环境因素的影响[11], 混凝-砂滤过程中细胞死亡释放的游离DNA降解速度可能低于好氧曝气过程, 使得混凝-砂滤出水中游离sul Ⅱ的相对丰度大幅升高, 进而使水体中游离sul Ⅱ的绝对量有所上升.
由于系统C进水中细胞态ARGs的量较少, 经A/O/O组合生物处理系统处理后, 出水中含有一定量的悬浮污泥, 并具有较高的ARGs相对丰度, 使得A/O/O对废水中ARGs的去除率为负(-2.66 logs), 但是对游离态ARGs有少量去除(0.39 logs).经过混凝-砂滤工艺对生物处理出水中悬浮物的进一步去除, 系统C对细胞态ARGs的去除率仍然为负(-1.70 logs), 同时游离态ARGs的去除率也为负(-0.06 logs).系统C对ARGs去除能力的不足, 一方面是由于焦化废水进水中细胞态ARGs较低, 而出水含有一定量的悬浮污泥, 另一方面可能是焦化废水处理系统存在某些选择性因子, 或促进了ARGs的HGT, 或造成了某些ARBs的竞争优势, 使污泥中ARBs得到了富集, 这显示了生活污水处理和工业废水处理对ARGs传播的不同作用.
为了控制环境中ARGs的增殖与传播, 一方面需要有效减少可能在环境中造成选择压力的抗生素输入, 另一方面需要加强控制ARGs在不同场所与环境中的输移, 从而降低通过HGT传播扩散ARGs的可能性.污水处理系统将最终出水排入到自然环境, 其出水中的ARGs可能直接提高受纳环境中ARGs的水平, 并增加经HGT而传播ARGs的可能性, 因此有效控制污水处理系统出水中的ARGs显得尤为重要.通过沉淀、过滤等工艺, 可有效降低出水中的细胞态ARGs水平, 也使游离态ARGs在总ARGs中的比例得到提高.如本研究系统C中, 游离态sul Ⅱ在总sul Ⅱ中的比重从好氧出水C-H的0.05%, 提高到混凝-砂滤出水C-S中的1.33%.此外, 消毒工艺可对出水中残留的ARBs产生较好的灭活作用[24, 25], 然而随着死亡ARBs的细胞发生裂解, 胞内ARGs将被释放到水体中, 形成游离态ARGs.如本研究中C-S静置5 d后, 游离态sul Ⅱ在总sul Ⅱ中的比例上升至9.31%.这就意味着水中细胞态ARGs被去除后, 水中残留的游离态ARGs以及ARBs灭活后释放出的游离态ARGs仍然将进入受纳环境中.对于游离态DNA经HGT途径传播ARGs的风险, 应开展更深入地研究与评估, 以指导污水处理系统出水中游离态ARGs的控制对策.
3 结论(1) 游离态ARGs与细胞态ARGs共同赋存于污水处理系统的各个阶段废水中.
(2) 生活污水处理系统M, 进水中细胞态sul Ⅱ、tetC、blaPSE-1和ermB这4种ARGs的相对丰度和绝对丰度均远高于游离态; 生物处理系统未对ARBs产生富集效应, 并使游离态ARGs丰度有所下降; MBR膜过滤不仅有效截留悬浮污泥, 也有效削减水中的游离DNA, A/O-MBR工艺对总ARGs的去除率达到2.54~4.95 logs.
(3) 焦化废水处理系统C, 进水中细胞态sul Ⅱ基因的相对丰度低于游离态, 但绝对丰度略高于游离态sul Ⅱ; A/O/O工艺对sul Ⅱ携带的ARBs产生了富集效应; 混凝-砂滤工艺通过去除生物处理出水中的悬浮污泥, 使水中细胞态sul Ⅱ的量下降了1个数量级, 但游离态sul Ⅱ量有所上升.混凝-砂滤对生物处理出水中悬浮污泥的去除、出水中细胞的消亡裂解, 使得游离态sul Ⅱ在总sul Ⅱ中的比例持续上升.在ARBs被深度去除或灭活后, 污水处理系统出水中游离态ARGs介导受纳环境ARGs传播扩散的风险有待后续研究进行评估.
| [1] | De Leener E, Martel A, Decostere A, et al. Distribution of the erm (B) gene, tetracycline resistance genes, and Tn1545-like transposons in macrolide-and lincosamide-resistant enterococci from pigs and humans[J]. Microbial Drug Resistance, 2004, 10(4): 341-345. DOI:10.1089/mdr.2004.10.341 |
| [2] | Kim S R, Nonaka L, Suzuki S. Occurrence of tetracycline resistance genes tet(M) and tet(S) in bacteria from marine aquaculture sites[J]. FEMS Microbiology Letters, 2004, 237(1): 147-156. DOI:10.1111/fml.2004.237.issue-1 |
| [3] | Andersson D I, Hughes D. Antibiotic resistance and its cost:is it possible to reverse resistance?[J]. Nature Reviews Microbiology, 2010, 8(4): 260-271. |
| [4] | Caucci S, Karkman A, Cacace D, et al. Seasonality of antibiotic prescriptions for outpatients and resistance genes in sewers and wastewater treatment plant outflow[J]. FEMS Microbiology Ecology, 2016, 92(5): fiw060. DOI:10.1093/femsec/fiw060 |
| [5] | Tao C W, Hsu B M, Ji W T, et al. Evaluation of five antibiotic resistance genes in wastewater treatment systems of swine farms by real-time PCR[J]. Science of the Total Environment, 2014, 496: 116-121. DOI:10.1016/j.scitotenv.2014.07.024 |
| [6] | Yang Y, Li B, Zou S C, et al. Fate of antibiotic resistance genes in sewage treatment plant revealed by metagenomic approach[J]. Water Research, 2014, 62: 97-106. DOI:10.1016/j.watres.2014.05.019 |
| [7] | Tao W D, Zhang X X, Zhao F Z, et al. High Levels of Antibiotic resistance genes and their correlations with bacterial community and mobile genetic elements in pharmaceutical wastewater treatment bioreactors[J]. PLoS One, 2016, 11(6): e0156854. DOI:10.1371/journal.pone.0156854 |
| [8] | Rodriguez-Mozaz S, Chamorro S, Marti E, et al. Occurrence of antibiotics and antibiotic resistance genes in hospital and urban wastewaters and their impact on the receiving river[J]. Water Research, 2015, 69: 234-242. DOI:10.1016/j.watres.2014.11.021 |
| [9] |
覃彩霞, 佟娟, 申佩弘, 等. 螺旋霉素制药废水处理过程中耐药菌和抗性基因的转归特征[J]. 环境科学, 2015, 36(9): 3311-3318. Qin C X, Tong J, Shen P H, et al. Fate of ARB and ARGs during wastewater treatment process of spiramycin production[J]. Environmental Science, 2015, 36(9): 3311-3318. |
| [10] |
黄福义, 李虎, 安新丽, 等. 城市生活污水和生活垃圾渗滤液抗生素抗性基因污染的比较研究[J]. 环境科学, 2016, 37(10): 3949-3954. Huang F Y, Li H, An X L, et al. Comparative investigation of antibotic resistance genes between wastewater and landfill leachate[J]. Environmental Science, 2016, 37(10): 3949-3954. |
| [11] | Eichmiller J J, Best S E, Sorensen P W. Effects of temperature and trophic state on degradation of environmental DNA in lake water[J]. Environmental Science & Technology, 2016, 50(4): 1859-1867. |
| [12] | Ficetola G F, Miaud C, Pompanon F, et al. Species detection using environmental DNA from water samples[J]. Biology Letters, 2008, 4(4): 423-425. DOI:10.1098/rsbl.2008.0118 |
| [13] | Eichmiller J J, Miller L M, Sorensen P W. Optimizing techniques to capture and extract environmental DNA for detection and quantification of fish[J]. Molecular Ecology Resources, 2016, 16(1): 56-68. DOI:10.1111/1755-0998.12421 |
| [14] | Bai Y H, Sun Q H, Wen D H, et al. Abundance of ammonia-oxidizing bacteria and archaea in industrial and domestic wastewater treatment systems[J]. FEMS Microbiology Ecology, 2012, 80(2): 323-330. DOI:10.1111/fem.2012.80.issue-2 |
| [15] | Jiang L, Hu X L, Xu T, et al. Prevalence of antibiotic resistance genes and their relationship with antibiotics in the Huangpu River and the drinking water sources, Shanghai, China[J]. Science of the Total Environment, 2013, 458-460: 267-272. DOI:10.1016/j.scitotenv.2013.04.038 |
| [16] | Chen B W, Liang X M, Huang X P, et al. Differentiating anthropogenic impacts on ARGs in the Pearl River Estuary by using suitable gene indicators[J]. Water Research, 2013, 47(8): 2811-2820. DOI:10.1016/j.watres.2013.02.042 |
| [17] | Stoll C, Sidhu J P S, Tiehm A, et al. Prevalence of clinically relevant antibiotic resistance genes in surface water samples collected from Germany and Australia[J]. Environmental Science & Technology, 2012, 46(17): 9716-9726. |
| [18] | Huerta B, Marti E, Gros M, et al. Exploring the links between antibiotic occurrence, antibiotic resistance, and bacterial communities in water supply reservoirs[J]. Science of the Total Environment, 2013, 456-457: 161-170. DOI:10.1016/j.scitotenv.2013.03.071 |
| [19] | Romanowski G, Lorenz M G, Wackernagel W. Adsorption of plasmid DNA to mineral surfaces and protection against DNase I[J]. Applied and Environmental Microbiology, 1991, 57(4): 1057-1061. |
| [20] | Li A D, Li L G, Zhang T. Exploring antibiotic resistance genes and metal resistance genes in plasmid metagenomes from wastewater treatment plants[J]. Frontiers in Microbiology, 2015, 6: 1025. |
| [21] | Subirats J, Sànchez-Melsió A, Borrego C M, et al. Metagenomic analysis reveals that bacteriophages are reservoirs of antibiotic resistance genes[J]. International Journal of Antimicrobial Agents, 2016, 48(2): 163-167. DOI:10.1016/j.ijantimicag.2016.04.028 |
| [22] | Mao D Q, Yu S, Rysz M, et al. Prevalence and proliferation of antibiotic resistance genes in two municipal wastewater treatment plants[J]. Water Research, 2015, 85: 458-466. DOI:10.1016/j.watres.2015.09.010 |
| [23] | Li J N, Cheng W X, Xu L K, et al. Occurrence and removal of antibiotics and the corresponding resistance genes in wastewater treatment plants:effluents' influence to downstream water environment[J]. Environmental Science and Pollution Research, 2016, 23(7): 6826-6835. DOI:10.1007/s11356-015-5916-2 |
| [24] | Zhang Y Y, Zhuang Y, Geng J J, et al. Inactivation of antibiotic resistance genes in municipal wastewater effluent by chlorination and sequential UV/chlorination disinfection[J]. Science of the Total Environment, 2015, 512-513: 125-132. DOI:10.1016/j.scitotenv.2015.01.028 |
| [25] | Ferro G, Guarino F, Castiglione S, et al. Antibiotic resistance spread potential in urban wastewater effluents disinfected by UV/H2O2 process[J]. Science of the Total Environment, 2016, 560-561: 29-35. DOI:10.1016/j.scitotenv.2016.04.047 |
 2017, Vol. 38
2017, Vol. 38


