在水处理过程中,消毒过程能灭活水体中的致病菌和微生物,降低水传播疾病的风险.水处理过程常采用紫外线消毒和次氯酸钠消毒,然而其消毒机制存在明显的差异,紫外线消毒作为典型的物理消毒剂,主要通过破坏微生物的遗传系统 (DNA、RNA) 而达到消毒的目的[1, 2],然而次氯酸钠作为一种化学消毒剂,更多的损害其他的物质 (包括细胞结构、蛋白质、脂质和内部的酶系统)[3, 4].
病原菌和病毒是水传播疾病的主要病原微生物,它们的形态和核酸结构,以及对不利环境的抵抗能力有明显差异[5],因此消毒方法对它们的作用效果目前尚存争议.要阐明消毒效果就必须选用典型的微生物进行对比研究.大肠杆菌是原核生物的模式生物,常作为水质卫生学的指示菌[6],而脊髓灰质炎病毒则是小RNA病毒科肠道病毒属的典型代表[7, 8].它们在污水中都广泛存在,可以分别作为水中DNA病原菌的代表性细菌[9]和RNA病毒的代表性病毒.
目前,病原微生物常用的检测方法主要包括传统的培养法和定量PCR方法,常用于对消毒效果的评价.基于培养的检测方法主要用于微生物活性的检测,其缺点不能用于检测处于活的但不可培养 (viable but non-culturable, VBNC) 状态的微生物[10],而定量PCR方法具有较高的灵敏度和专一性,是从分子水平 (DNA、RNA) 对微生物消毒效果进行分析,解决了上述缺点,使快速定量检测无法培养的微生物成为可能[11].迄今为止,微生物消毒过程中培养方法和分子生物学检测方法之间的关系是不确定的.一些研究表明两种方法结果是一致的[12, 13],例如Jin等[14]采用全基因组分段PCR方法检测肠道病毒消毒过程核酸的损伤,得到5′NCR非编码区核酸的损伤与病毒感染性消失是一致的.但也有一些研究者持否定态度.Li等[15]研究了氯消毒对轮状病毒的灭活, 认为PCR方法低估了微生物灭活.因此确定两种检测方法之间的关系,选择合适的评价方法是至关重要的.
本研究选用大肠杆菌和脊髓灰质炎病毒作为代表性微生物,利用培养法和定量PCR检测,对比研究紫外线消毒和次氯酸钠消毒对细菌和病毒的作用特点.
1 材料与方法 1.1 典型微生物从美国菌种保藏中心购买的大肠杆菌 (ATCC25922),接种于液体LB培养基中,于37℃摇床中200 r·min-1培养12~16 h,8 000 r·min-1离心10 min,得到细胞悬浊液,用磷酸缓冲液 (PBS,pH=7.2) 洗脱两次,进行10倍稀释得到不同浓度的大肠杆菌菌液,作为后续指示性细菌.
脊髓灰质炎病毒提取于脊髓灰质炎疫苗糖丸 (人二倍体细胞).提取过程:将人喉癌上皮细胞 (Hep-2) 接种于5 mL的DMEM低糖培养液 (Hyclone,北美),加入10% FBS胎牛血清 (GIBCO,美国)、3.75 g·L-1的碳酸氢钠、100 μg·mL-1链霉素 (生工生物公司,上海)、100 U·mL-1青霉素 (生工生物公司,上海),放入5%的CO2培养箱中37℃培养至细胞长到80%待用.将糖丸经DMEM 10倍稀释,接种于长至80%的Hep-2细胞,出现细胞病变后反复冻融3次,800 r·min-1离心10 min,上清液即为脊髓灰质炎病毒.
1.2 消毒实验在40 mL的PBS缓冲液中分别接种大肠杆菌和脊髓灰质炎病毒.经次氯酸钠消毒和紫外线消毒后,采用培养法对微生物活性进行分析,采用定量PCR方法对核酸损伤进行分析,每个实验重复3次.
1.2.1 紫外线消毒本实验所用紫外灯的功率为15 W (波长253.7 nm),实验中紫外线强度为0.10 mW·cm-2.向直径为90 mm的培养皿中加入接种微生物的40 mL PBS水样,把培养皿放到准平行光紫外线照射器的紫外灯下,培养皿下置磁力搅拌器,轻轻搅拌水样.紫外线剂量 (mJ·cm-2)=紫外线强度 (mW·cm-2)×辐射时间 (s), 通过调节紫外线照射时间达到不同的紫外线剂量,辐射一定时间后将水样取出,4℃保存,用于后续分析.
1.2.2 次氯酸钠消毒本实验中向接种微生物的40 mL PBS水样中分别投加0.5、1、1.5、2、2.5、5、10、15、20、25 mg·L-1次氯酸钠 (碘量法测定浓度),接触反应30 min后,用0.1%(质量浓度) 硫代硫酸钠终止反应,保存于4℃用于后续分析.
1.3 培养法检测大肠杆菌和脊髓灰质炎病毒采用膜过滤平板计数方法检测大肠杆菌的活性,10倍稀释水样经直径50 mm、孔径0.22 μm的混合纤维膜过滤后,置于MFC培养基上,于37℃培养24 h后进行菌落计数分析.
采用半数细胞感染剂量法 (TCID50法) 确定脊髓灰质炎病毒的感染性,将病毒液或水样采用DMEM进行连续10倍稀释 (10-1~10-10),接种于长至100% Hep-2细胞的96孔板中,每个稀释度接种一纵排共8个平行样,每孔接种病毒100 μL.设正常细胞对照组,正常对照组位于最后一排 (100 μL DMEM).接种1.5 h后 (每隔30 min摇晃充分混匀),弃掉病毒液,加入2% FBS的DMEM细胞维持液,逐日观察细胞病变并记录,采用Reed-Muench法计算病毒浓度 (TCID50·mL-1)[16],其中1 TCID50·mL-1=0.693 PFU·mL-1(病毒的空斑形成单位,plaque forming units).
1.4 定量PCR方法检测大肠杆菌和脊髓灰质炎病毒 1.4.1 DNA和cDNA提取大肠杆菌的水样经14 000 r·min-1 4℃离心20 min,弃上清液,收集菌体,参照细菌基因组DNA提取试剂盒 (北京百泰克生物技术有限公司,DP2001) 说明提取总DNA作为模板.对脊髓灰质炎病毒水样采用RNA柱式提取试剂盒 (QlAamp Viral RNA Mini Kit,Qiagen,CA52906) 提取总RNA,并参照PrimeScript® RT reagent Kit with gDNA Eraser (Takara,DRR047A) 进行RT反应,得到cDNA作为后续反应模板.
1.4.2 定量PCR检测方法为了确定消毒过程中核酸的损伤程度,采用SYBR® Primix Dimer EraserTM perfect real-time kit (Takara,DRR091A) 试剂对大肠杆菌的uidA核酸 (表 1) 进行定量分析.定量PCR反应体系为:12.5 μL的SYBR Premix DimerEraserTM (2×),0.5 μL的ROX Reference Dye Ⅱ,400nmol·L-1的上游引物URL-301和下游引物URR-432,2 μL的DNA,用超纯水补至25 μL.定量PCR的反应程序为:95℃预变性30 s;95℃ 15 s,60℃ 30 s,72℃ 60 s,80℃ 10 s收集荧光信号,扩增40个循环.
|
|
表 1 大肠杆菌和脊髓灰质炎病毒的扩增引物 Table 1 Primers used in this study to amplify Escherichia coli and poliovirus |
采用Primix the Ex TaqTM kit (Takara, DRR039) 试剂对脊髓灰质炎病毒的5′NCR核酸 (表 1) 进行扩增.定量PCR反应体系为:12.5 μL Primix Ex TaqTM,0.5 μL的ROX Reference Dye Ⅱ,400nmol·L-1的上游引物EV1和下游引物EV2,400nmol·L-1的TaqMan探针EVP,2 μL的cDNA,用超纯水补至25 μL.反应程序:预变性95℃ 30 s;95℃ 10 s,60℃ 34 s,扩增40个循环.
通过质粒标准曲线推测核酸的含量,定量PCR标线的建立:PCR产物经纯化后连接到pMD-19T载体 (TaKaRa) 上,再将连接产物转化到DH5α感受态细胞中.经PCR鉴定筛选的阳性菌落于AMP的LB培养基中37℃培养16 h,采用质粒提取试剂盒提取重组质粒 (生工生物公司,上海),并通过Nanodrop 2000(Thermo) 测定质粒浓度,将含有目的片段的重组质粒进行10倍梯度稀释建立标准曲线.
1.5 数据分析采用线性拟合对微生物的去除进行分析,并推断去除率达到90% (1-log) 所需的消毒剂量 (紫外线剂量或次氯酸钠浓度):
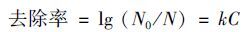
|
式中,k为反应常数;N0为消毒前微生物的浓度 (培养法:CFU·mL-1或TCID50·mL-1,定量PCR:copies·mL-1);N为消毒后微生物的浓度 (培养法:CFU·mL-1或TCID50·mL-1,定量PCR:copies·mL-1);C为消毒剂剂量 (次氯酸钠消毒:mg·L-1,紫外线消毒:mJ·cm-2).
采用SPSS Ver.20软件进行单因素方差分析,比较不同消毒剂量下微生物浓度是否存在显著性影响 (P<0.05).
2 结果与分析 2.1 定量PCR检测大肠杆菌和脊髓灰质炎病毒的敏感性和可靠性经计算大肠杆菌和脊髓灰质炎病毒重组质粒标准品的浓度分别为1.1×1010 copies·μL-1和2.93×1010 copies·μL-1,将标准品质粒经10倍稀释,进行定量PCR反应建立标准曲线如图 1所示.重组质粒浓度的对数与对应的CT值 (定量PCR扩增反应中荧光信号达到设定阈值时所经历的循环数) 存在良好的线性关系.由图 1可知,大肠杆菌质粒相关系数R2为0.998,PCR扩增效率98.5%(E%=10-1/S-1,S为斜率),脊髓灰质炎病毒质粒相关系数为R2=0.994,PCR扩增效率E%=96.9%,得到基因标记物的检测灵敏度分别为11 copies·μL-1和29.3 copies·μL-1.
|
图 1 质粒浓度和循环数CT相关关系 Fig. 1 Correlation between CT and plasmid concentration |
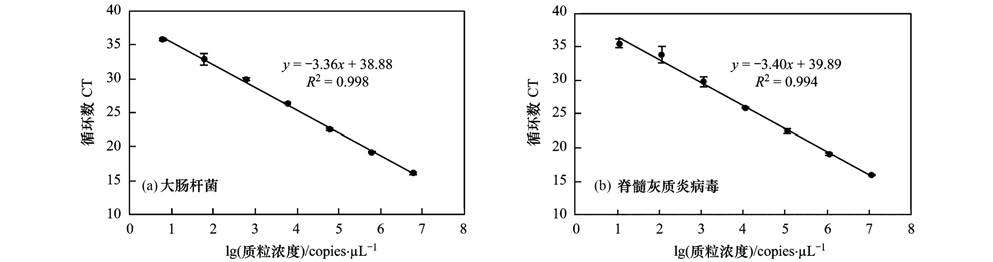
紫外线和次氯酸钠对大肠杆菌的消毒作用呈明显的剂量反应关系,如图 2所示.培养法结果显示:当紫外线剂量为2 mJ·cm-2时,大肠杆菌的浓度为 (8.1±0.71)×104 CFU·mL-1明显低于起始浓度 (3.52±2.08)×105 CFU·mL-1(P<0.05),然而更高的紫外线剂量 (>5 mJ·cm-2),检测大肠杆菌的浓度与2 mJ·cm-2时浓度无显著性差异 (P>0.05) 如图 2(a)所示,即紫外线灭活曲线由两阶段构成:灭活初期低紫外线剂量灭活速率较快 (反应常数k=0.55, R2=0.94),随反应进行紫外线剂量呈明显的拖尾状态 (k=0.11, R2=0.99),拖尾可能是由于消毒过程微生物表面性质的变化和胞外聚合物的产生导致的微生物聚集引起的[19].当紫外线剂量达到城镇污水处理一级A标准的20 mJ·cm-2,大肠杆菌完全被灭活.如图 2(c)所示次氯酸钠能迅速灭活大肠杆菌 (0.025<P<0.041),且当次氯酸钠剂量达到2 mg·L-1时,接触反应30 min,大肠杆菌完全灭活.次氯酸钠消毒过程呈一阶剂量反应关系 (k=2.96, R2=0.95).
|
图 2 大肠杆菌的紫外线和次氯酸钠剂量反应曲线 Fig. 2 UV and sodium hypochlorite dose-response curves for Escherichia coli |
定量PCR结果显示:当紫外线剂量照射达到50 mJ·cm-2时,大肠杆菌的uidA基因浓度为 (1.69±0.64)×105 copies·mL-1低于起始浓度 (3.35±1.17)×105 copies·mL-1(P=0.006),然而,当紫外线剂量达到400 mJ·cm-2(远远高于城镇污水处理的一级A标准的20 mJ·cm-2和中水回用的紫外线剂量标准80 mJ·cm-2紫外线剂量),仍能检测 (1.46±0.42)×104 copies·mL-1的基因存在[图 2(b)].当0.5、1.0、1.5、2.0、5.0 mg·L-1次氯酸钠接触反应30 min后,核酸浓度分别为 (5.11±1.3)×105、(5.12±2.03)×105、(2.22±1.72)×105、(1.6±0.49)×105以及 (0.67±0.11)×105 copies·mL-1,较起始的 (5.14±2.0)×105 copies·mL-1低剂量次氯酸钠对大肠杆菌uidA核酸的损伤很少 (P为1.0、1.0、0.283、0.109、0.426),高浓度次氯酸钠 (8 mg·L-1) 对大肠杆菌起始拷贝数有明显的减少,为 (4.59±1.8)×103 copies·mL-1(P=0.006).
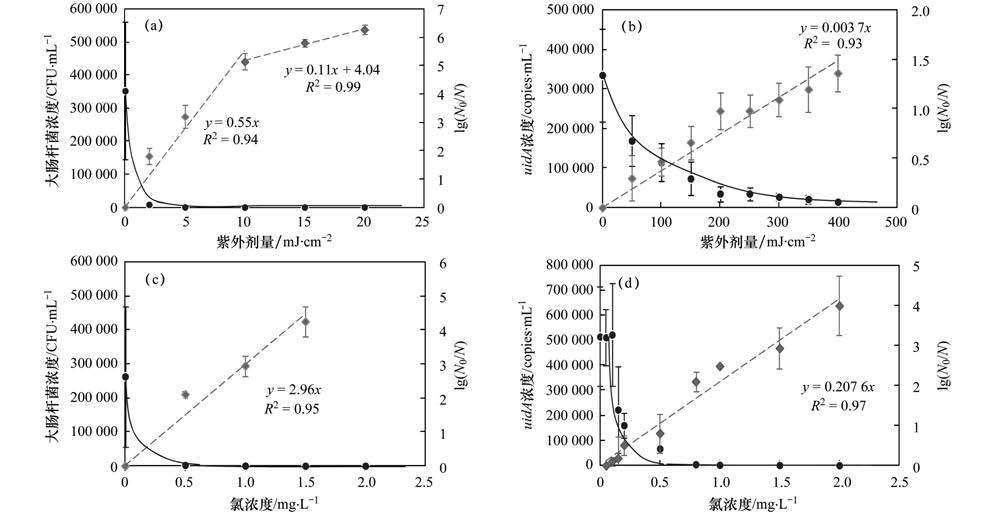
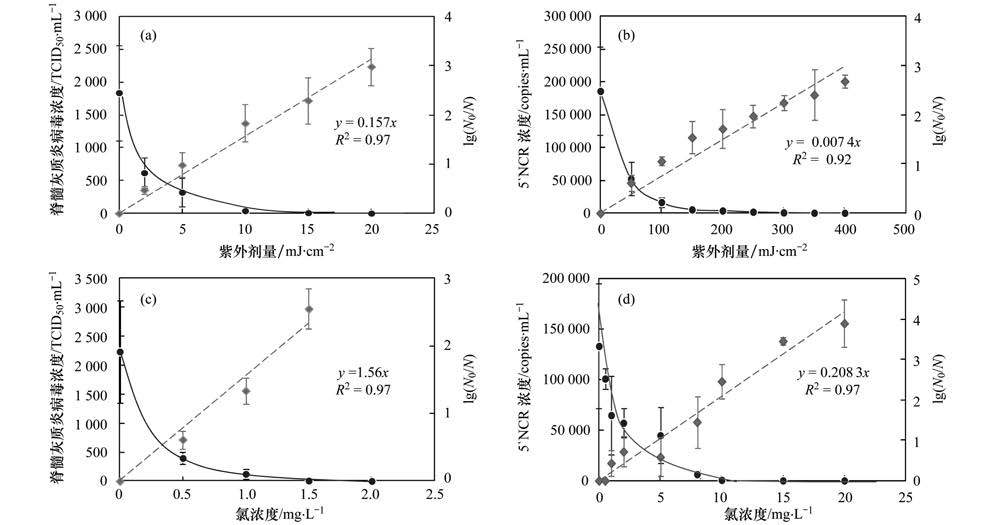
2.3 紫外线消毒和次氯酸钠消毒对脊髓灰质炎病毒的影响紫外线 (TCID50方法: k=0.157, R2=0.97;定量PCR: k=0.007 4, R2=0.92) 和次氯酸钠 (TCID50方法: k=1.56, R2=0.97;定量PCR: k=0.208 3, R2=0.97) 对脊髓灰质炎病毒呈一阶剂量反应关系 (图 3).随消毒剂量的增加脊髓灰质炎病毒迅速被灭活,当紫外线剂量超过20 mJ·cm-2,起始次氯酸钠量为2 mg·L-1时接触反应30 min,脊髓灰质炎病毒被完全灭活.定量PCR结果显示,随紫外线剂量增加50 mJ·cm-2,脊髓灰质炎病毒的5′NCR基因拷贝数从起始的 (1.86±0.67)×105 copies·mL-1减少到 (5.24±2.6)×104 copies·mL-1,发生明显的核酸损伤 (P<0.05)[图 3(b)],然而低剂量次氯酸钠含量0.5、1.0、2.0 mg·L-1对5′NCR区产生轻微的影响 (P=0.963、0.139、0.061)[图 3(d)],当次氯酸钠浓度达到5 mg·L-1以上时,脊髓灰质炎病毒的5′NCR拷贝数较起始时有明显降低 (P=0.001~0.025),当紫外线剂量达到400 mJ·cm-2、次氯酸钠量达到20 mg·L-1时,脊髓灰质炎病毒的基因拷贝数低于检出限水平.
|
(a) 和 (c) TCID50法检测脊髓灰质炎病毒;(b) 和 (d) 采用定量PCR法检测脊髓灰质炎病毒 图 3 脊髓灰质炎病毒的紫外线消毒和次氯酸钠消毒剂量反应曲线 Fig. 3 UV and sodium hypochlorite dose-response curves for Poliovirus |
通过线性拟合剂量反应关系,推算微生物去除率达到1-log所需的紫外线剂量和接触30 min时的氯消毒ct值 (氯浓度×接触时间,mg·L-1·min),培养法检测结果显示:紫外线消毒过程中,在较低的紫外线辐射剂量下 (1.81 mJ·cm-2) 大肠杆菌活性即可达到1-log去除率,而脊髓灰质炎病毒丢失感染性所需的剂量为6.37 mJ·cm-2,为前者的3.5倍,说明病毒的灭活比细菌的灭活需要更多紫外线剂量 (图 4),氯消毒过程,培养法检测达到1-log减少时,脊髓灰质炎病毒和大肠杆菌所需氯剂量分别为4.82 mg·L-1和0.338 mg·L-1,ct值分别为19.2 mg·L-1·min和10.14 mg·L-1·min,与紫外线消毒结果一致,即病毒比细菌更难被灭活.因此采用大肠杆菌作为水质卫生学指标存在一定的健康风险.而定量PCR检测结果显示:紫外线消毒过程,基因拷贝数减少1-log,脊髓灰质炎病毒和大肠杆菌所需的紫外线辐射剂量分别为135 mJ·cm-2和270.3 mJ·cm-2,氯消毒所需的次氯酸钠浓度也大幅度提高分别为4.8 mg·L-1和4.82 mg·L-1,ct值为144 mg·L-1·min和144.6 mg·L-1·min.定量PCR方法检测大肠杆菌和脊髓灰质炎病毒达到1-log的去除率所需的紫外线剂量和ct值要比培养法高出1~2数量级,特别是对于大肠杆菌,紫外线剂量高达270.3 mJ·cm-2时基因的损伤才达到1-log,是培养法1.81 mJ·cm-2的149倍.因此常规的消毒技术无法有效地去除病原微生物的核酸,而剩余的核酸进入下游水域,微生物间能通过水平基因转移的方式获得相应的基因[20],可能导致致病基因的传播和扩散,引发严重的健康风险.
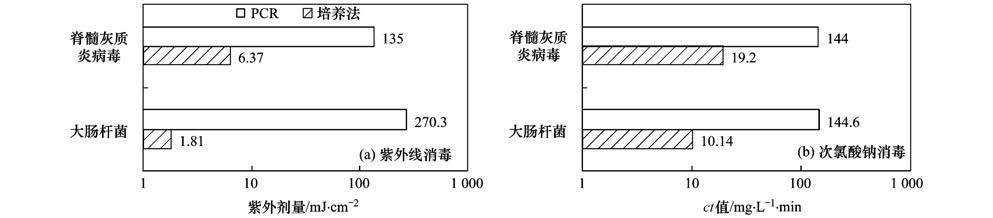
|
图 4 达到1-log大肠杆菌和脊髓灰质炎病毒去除所需的紫外线剂量和ct值 Fig. 4 Comparison of UV dose andct values of chlorination for 1-log removal of Escherichia coliand Poliovirus reduction |
迄今为止所做的微生物灭活研究,但大多数集中在微生物的灭活动力学研究方面,而有关消毒过程中核酸的损伤则鲜见报道.定量PCR方法能用于检测核酸的损伤包括核酸链的破坏、碱基的氧化以及形成嘧啶二聚体[21, 22].本研究采用定量PCR检测消毒过程中核酸损伤,结果发现:核酸损伤达到1-log所需的消毒剂量比用培养法检测的1-log微生物数量减少所需的剂量要高出1~2数量级 (图 4).造成这种显著差异的原因可能包括以下两个方面:首先是培养法检测的局限性.在消毒过程中,微生物常处于有活性但不可培养 (VBNC) 状态[10],其仍保持新陈代谢能力,在适当的环境条件下还能恢复感染性和致病性[23, 24],而这些微生物是无法通过培养法检测到的.PCR则是针对核酸片段的检测方法,完全脱离了培养条件的束缚,是从遗传信息破坏的角度来评价微生物的灭活.显然,能够被PCR方法检测到的微生物数量高于培养法的.其次,PCR结果对微生物灭活的低估可能与检测片段的长度和位置有关[7].待测核酸片段越长,越易检测到消毒的影响.本研究中,大肠杆菌待测核酸片段长度为153 bp,达到1-log核酸损伤所需的紫外线剂量为270.3 mJ·cm-2,而McKinney等[25]针对1 kb片段进行研究,仅需50~100 mJ·cm-2就造成了1-log核酸损伤.
如图 5所示,细菌长度为1 μm明显大于病毒的直径20~30 nm,其与消毒剂接触反应的几率更大,且细菌的结构较复杂包括胶囊、细胞壁、细胞膜、细胞质、酶系统、DNA等复杂结构对消毒剂更敏感,因此病毒对消毒剂的耐性强于细菌.紫外线消毒主要与微生物的核酸发生光化学反应,引起磷酸二酯键和氢键的断裂、形成嘧啶二聚体 (例如:DNA损伤T-T CPDs和6-4PPS;RNA损伤形成尿嘧啶环丁烷二聚体、尿嘧啶水合物、胞嘧啶水合物) 导致病毒的复制能力的消失[1, 26],并引起少量细菌的细胞膜蛋白以及内部酶系统或病毒衣壳的损伤 (图 5),其共同作用导致微生物的灭活,因此与活性相比核酸损伤需要更多的紫外线剂量.值得注意的是,紫外线消毒过程中脊髓灰质炎病毒的RNA损伤比大肠杆菌的DNA损伤更严重 (图 4),该结果与培养法病毒对紫外线的耐性更强的结果正好相反.这主要与两种微生物的结构有关,大肠杆菌作为代表性细菌,是原核生物存在明显的细胞结构,而脊髓灰质炎病毒由核酸和衣壳蛋白组成,无明显的细胞结构 (图 5),因此紫外线进入细菌内比进入病毒更难.其次,细菌的DNA核酸不容易被损伤,主要由于其二维结构[27],PCR检测方法的原理是利用单链的碱基配原则进行体外复制的过程,即使DNA二维结构氢键断裂损伤发生解链过程,然而采用定量PCR方法无法检测,另外研究表明RNA的磷酸二酯键更容易受到损伤[28],因此单链线性RNA对紫外线更敏感.
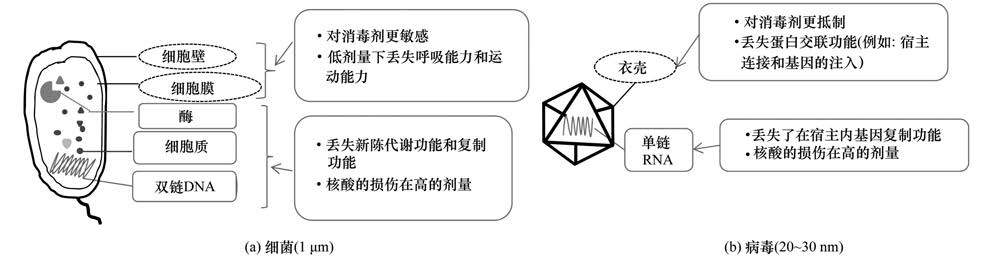
|
图 5 细菌和病毒的损伤机制示意 Fig. 5 Scheme for the killing and destruction of the bacteria and virus |
次氯酸钠消毒过程形成HOCl作为一种强氧化剂,具有较强的细胞渗透力,首先损伤细胞膜使其通透性增强,从而使其呼吸作用和运动功能受阻失去可培养能力,然后进入内部干扰酶系统,导致细胞内容物泄漏,最后导致蛋白物质和核酸的损伤,使细菌的新陈代谢和复制功能丧失使细菌完全死亡[29].次氯酸钠消毒对病毒的灭活过程,首先损害病毒的衣壳蛋白,使其蛋白交联作用丢失 (病毒丢失了宿主连接以及基因的注入的能力) 感染性消失,病毒的完整性被破坏丢失保护核酸的能力,然后进一步氧化核酸形成5-氯胞啶 (5-chlorocytidine) 及较少的8-氯鸟嘌呤 (8-chloroguanosine) 和8-氯腺嘌呤 (8-chloroadenosine)[30],使病毒丢失复制功能.因此,次氯酸钠消毒过程中,低浓度的氯在影响核酸之前使微生物失去了活性,微生物完全灭活时核酸仍保持一定完整性,核酸损伤需要更多的次氯酸钠剂量.
4 结论(1) 紫外线消毒和次氯酸钠对大肠杆菌和脊髓灰质炎病毒呈明显的剂量反应关系,当紫外线剂量达到20 mJ·cm-2时, 20 mg·L-1次氯酸钠接触反应30 min,大肠杆菌和脊髓灰质炎病毒丢失可培养性,然而脊髓灰质炎病毒对消毒剂的耐性强于大肠杆菌.
(2) 紫外线消毒对RNA的损伤明显高于DNA,由于RNA的磷酸二酯键更容易受到损伤.常规的消毒技术无法有效地去除微生物的核酸.
(3) 定量结果显示核酸损伤需要更多的消毒剂量,达到1-log的核酸损伤所需的消毒剂量高于培养法的1~2个数量级,这可能由于紫外线和次氯酸钠能使微生物处于有活性但不可培养 (VBNC) 状态,另一方面消毒过程存在除核酸损伤外的其他的机制,紫外线消毒通过破坏微生物的核酸 (DNA/RNA) 达到灭活的目的,然而仍能引起少量蛋白、膜的损伤.而次氯酸钠消毒更多的损害细菌的细胞膜和病毒的衣壳蛋白,使其失去活性.
| [1] | Rastogi R P, Richa, Kumar A, et al. Molecular mechanisms of ultraviolet radiation-induced DNA damage and repair[J]. Journal of Nucleic Acids, 2010, 2010: 592980. |
| [2] | 李静, 王大宁, 曲红梅, 等. 紫外线对大肠杆菌的损伤机制研究[J]. 军事医学, 2016, 40(9): 725–728. Li J, Wang D N, Qu H M, et al. Mechanism of Escherichia coli injury under UV disinfection[J]. Military Medical Sciences, 2016, 40(9): 725–728. |
| [3] | Virto R, Mañas P, Álvarez I, et al. Membrane damage and microbial inactivation by chlorine in the absence and presence of a chlorine-demanding substrate[J]. Applied and Environmental Microbiology, 2005, 71(9): 5022–5028. DOI: 10.1128/AEM.71.9.5022-5028.2005 |
| [4] | Cho M, Kim J, Kim J Y, et al. Mechanisms of Escherichia coli inactivation by several disinfectants[J]. Water Research, 2010, 44(11): 3410–3418. DOI: 10.1016/j.watres.2010.03.017 |
| [5] | Sinton L W, Hall C H, Lynch P A, et al. Sunlight inactivation of fecal indicator bacteria and bacteriophages from waste stabilization pond effluent in fresh and saline waters[J]. Applied and Environmental Microbiology, 2002, 68(3): 1122–1131. DOI: 10.1128/AEM.68.3.1122-1131.2002 |
| [6] | Edberg S C, Rice E W, Karlin R J, et al. Escherichia coli:the best biological drinking water indicator for public health protection[J]. Journal of Applied Microbiology, 2000, 88(S1): 106S–116S. DOI: 10.1111/jam.2000.88.issue-S1 |
| [7] | Simonet J, Gantzer C. Degradation of the Poliovirus 1 genome by chlorine dioxide[J]. Journal of Applied Microbiology, 2006, 100(4): 862–870. DOI: 10.1111/jam.2006.100.issue-4 |
| [8] | Simonet J, Gantzer C. Inactivation of Poliovirus 1 and F-specific RNA phages and degradation of their genomes by UV irradiation at 254 nanometers[J]. Applied and Environmental Microbiology, 2006, 72(12): 7671–7677. DOI: 10.1128/AEM.01106-06 |
| [9] | Cho M, Chung H, Choi W, et al. Different inactivation behaviors of MS-2 phage and Escherichia coli in TiO2 photocatalytic disinfection[J]. Applied and Environmental Microbiology, 2005, 71(1): 270–275. DOI: 10.1128/AEM.71.1.270-275.2005 |
| [10] | Oliver J D. Recent findings on the viable but nonculturable state in pathogenic bacteria[J]. FEMS Microbiology Reviews, 2010, 34(4): 415–425. DOI: 10.1111/j.1574-6976.2009.00200.x |
| [11] | Wong K, Fong T T, Bibby K, et al. Application of enteric viruses for fecal pollution source tracking in environmental waters[J]. Environment International, 2012, 45: 151–164. DOI: 10.1016/j.envint.2012.02.009 |
| [12] | 刘艳芝, 薛斌, 陈照立, 等. 二氧化氯灭活水中肠道腺病毒及其分子机制研究[J]. 环境与健康杂志, 2013, 30(2): 95–98. Liu Y Z, Xue B, Chen Z L, et al. Mechanism and regularity of inactivation of adenovirus by chlorine dioxide in water[J]. Journal of Environment and Health, 2013, 30(2): 95–98. |
| [13] | Trombert A, Irazoqui H, Martín C, et al. Evaluation of UV-C induced changes in Escherichia coli DNA using repetitive extragenic palindromic-polymerase chain reaction (REP-PCR)[J]. Journal of Photochemistry and Photobiology B:Biology, 2007, 89(1): 44–49. DOI: 10.1016/j.jphotobiol.2007.08.003 |
| [14] | Jin M, Shan J Y, Chen Z L, et al. Chlorine dioxide inactivation of enterovirus 71 in water and its impact on genomic targets[J]. Environmental Science & Technology, 2013, 47(9): 4590–4597. |
| [15] | Li D, Gu A Z, Zeng S, et al. Evaluation of the infectivity, gene and antigenicity persistence of rotaviruses by free chlorine disinfection[J]. Journal of Environmental Sciences, 2011, 23(10): 1691–1698. DOI: 10.1016/S1001-0742(10)60623-7 |
| [16] | Reed L J, Muench H. A simple method of estimating fifty per cent endpoints[J]. American Journal of Epidemiology, 1938, 27(3): 493–497. DOI: 10.1093/oxfordjournals.aje.a118408 |
| [17] | Maheux A F, Picard F J, Boissinot M, et al. Analytical comparison of nine PCR primer sets designed to detect the presence of Escherichia coli/Shigella in water samples[J]. Water Research, 2009, 43(12): 3019–3028. DOI: 10.1016/j.watres.2009.04.017 |
| [18] | Monpoeho S, Dehée A, Mignotte B, et al. Quantification of enterovirus RNA in sludge samples using single tube real-time RT-PCR[J]. Biotechniques, 2000, 29(1): 88–93. |
| [19] | Kollu K, Örmeci B. UV-induced self-aggregation of E. coli after low and medium pressure ultraviolet irradiation[J]. Journal of Photochemistry and Photobiology B:Biology, 2015, 148: 310–321. DOI: 10.1016/j.jphotobiol.2015.04.013 |
| [20] | Rodriguez-Mozaz S, Chamorro S, Marti E, et al. Occurrence of antibiotics and antibiotic resistance genes in hospital and urban wastewaters and their impact on the receiving river[J]. Water Research, 2015, 69: 234–242. DOI: 10.1016/j.watres.2014.11.021 |
| [21] | Sikorsky J A, Primerano D A, Fenger T W, et al. DNA damage reduces Taq DNA polymerase fidelity and PCR amplification efficiency[J]. Biochemical and Biophysical Research Communications, 2007, 355(2): 431–437. DOI: 10.1016/j.bbrc.2007.01.169 |
| [22] | Chatzisymeon E, Droumpali A, Mantzavinos D, et al. Disinfection of water and wastewater by UV-A and UV-C irradiation:application of real-time PCR method[J]. Photochemical & Photobiological Sciences, 2011, 10(3): 389–395. |
| [23] | 景明, 王磊. 污水回用中主要病原菌解析及其紫外消毒效应[J]. 环境科学, 2016, 37(2): 622–629. Jing M, Wang L. Analysis of pathogenic bacteria in reclaimed water and impact of UV disinfection on the removal of pathogenic bacteria[J]. Environmental Science, 2016, 37(2): 622–629. |
| [24] | Sinha R P, Häder D P. UV-induced DNA damage and repair:a review[J]. Photochemical & Photobiological Sciences, 2002, 1(4): 225–236. |
| [25] | McKinney C W, Pruden A. Ultraviolet disinfection of antibiotic resistant bacteria and their antibiotic resistance genes in water and wastewater[J]. Environmental Science & Technology, 2012, 46(24): 13393–13400. |
| [26] | Sikorsky J A, Primerano D A, Fenger T W, et al. Effect of DNA damage on PCR amplification efficiency with the relative threshold cycle method[J]. Biochemical and Biophysical Research Communications, 2004, 323(3): 823–830. DOI: 10.1016/j.bbrc.2004.08.168 |
| [27] | McKillip J, Jaykus L A, Drake M. rRNA Stability in Heat-Killed and UV-Irradiated Enterotoxigenic Staphylococcus aureus and Escherichia coli O157:H7[J]. Applied and Environmental Microbiology, 1998, 64(11): 4264–4268. |
| [28] | Ceuppens S, Li D, Uyttendaele M, et al. Molecular methods in food safety microbiology:interpretation and implications of nucleic acid detection[J]. Comprehensive Reviews in Food Science and Food Safety, 2014, 13(4): 551–577. DOI: 10.1111/1541-4337.12072 |
| [29] | Suquet C, Warren J J, Seth N, et al. Comparative study of HOCl-inflicted damage to bacterial DNA ex vivo and within cells[J]. Archives of Biochemistry and Biophysics, 2010, 493(2): 135–142. DOI: 10.1016/j.abb.2009.10.006 |
| [30] | Sigstam T, Gannon G, Cascella M, et al. Subtle Differences in virus composition affect disinfection kinetics and mechanisms[J]. Applied and Environmental Microbiology, 2013, 79(11): 3455–3467. DOI: 10.1128/AEM.00663-13 |
 2017, Vol. 38
2017, Vol. 38


