多环芳烃(polycyclic aromatic hydrocarbons, PAHs)是一类广泛分布于环境中的由两个或两个以上苯环以线状、角状或簇状排列的稠环化合物,因其具有强烈的致突变、致癌及致畸作用,美国环境保护署和欧盟均已经将PAHs列入典型污染物之列[1, 2].由于其毒性、生物蓄积性及半挥发性并能在环境中持续存在,也被列入典型持久性有机污染物(persistent organic pollutants, POPs).化石燃料的燃烧是PAHs的主要来源,由于人类对化石产品的不断开发利用,PAHs持续向环境中排放,高温过程形成的PAHs大多排放到大气中,随着大气环流、大洋环流等循环而不断扩散从而导致空气、土壤及水体甚至南极、高山冰川等都不同程度受到PAHs的污染[3~5].自然条件下的多环芳烃很难降解,且其抗降解能力随着分子中苯环数和分子量的增加而增强. PAHs在环境中的降解或消除途径主要包括挥发、光氧化、生物富集、化学氧化、土壤吸附及微生物降解等[2].其中微生物的降解在土壤、水体及沉积物等环境中的PAHs去除中起到了主导作用,是环境中PAHs衰减的主要途径之一[6]. PAHs的微生物降解可以在有氧、微氧及厌氧的条件下进行,其中有氧条件下微生物的降解已经得到大量研究,其降解效率高,降解机制也相对明确.然而,在土壤、水体、沉积物等环境中,多数PAHs可能处于微氧或厌氧状态,因此,PAHs的微氧及厌氧代谢在上述环境中极有可能起到了不可忽视的作用[6].
在厌氧条件下,NO3-、Fe3+、CO2及SO42-等均可作为与PAHs降解过程相偶联的电子受体,通过氧化还原反应将PAHs逐级降解[7].由于硝酸盐通常在土壤、水体等环境中大量存在,因此PAHs的厌氧代谢过程与以其为电子受体的反硝化过程的偶联可能在缺氧或厌氧环境中PAHs的降解中起到重要的作用.反硝化过程是反硝化微生物在一定条件下,将硝酸盐、亚硝酸盐逐级还原,最终将氮以一氧化氮(NO)、氧化亚氮(N2O)或分子态氮(N2)的形式释放出来的过程.这一系列酶促反应分别由硝酸盐还原酶(nitrate reductase, Nar)、亚硝酸盐还原酶(nitrite reductase, Nir)、氧化氮还原酶(nitric oxidereductase, Nor)和氧化亚氮还原酶(nitrous oxide reductase, Nos)所催化[8].编码这些酶的相关功能基因也被广泛用作研究环境中反硝化微生物群落组成的分子标识物[9, 10]. Mihelcic等[11]在1988年首次报道了典型PAHs (萘)可在反硝化条件下降解,此后,反硝化条件下PAHs的降解逐渐被人们所关注,一些研究也相继报道了多种大分子的复杂PAHs在反硝化条件下降解[12~14].目前的研究表明,反硝化条件下PAHs的降解与NO3-的含量存在明显的相关性[15~17].一些以反硝化过程中的功能基因作为分子标识的分子生态学研究进一步表明,PAHs的降解与反硝化微生物的丰度和群落结构密切相关[9, 18].由此可见,电子受体(NO3-)的浓度及反硝化相关的微生物群落结构均是反硝化条件下PAHs厌氧代谢的关键影响因子.
土壤是PAHs重要的汇,源于大气、水体及人为的原油泄漏均会造成土壤PAHs污染,同时土壤的复杂性也决定了厌氧的PAHs降解在PAHs的衰减中可能起到了不可忽视的作用.当前,有不少关于土壤反硝化富集培养物厌氧降解PAHs的报道,针对田间自然土壤或沉积物中PAHs含量及相关反硝化功能微生物关系也逐渐为人们所关注.如吴艳阳等[18]以narG、nirS和nosZ基因为分子标识,探究了沉积物中PAHs对反硝化功能基因垂直分布的影响.而Guo[9]等以nirK、nirS和nosZ为分子标识物,研究了芘对农业土壤中反硝化微生物群落的影响,并发现土壤中芘的降解率与反硝化速率及反硝化功能基因的丰度之间具有密切的关联.因此,本文选择具有50多年历史的江汉油田区域为研究对象,以反硝化的功能基因nirK及nirS为分子标识,利用定量PCR及克隆文库结合限制性末端多态性(terminal restriction fragment length polymorphism, T-RFLP)的方法,来探讨该油田区域土壤PAHs含量等环境因子与反硝化微生物群落结构之间的关系,以期为进一步深入研究土壤PAHs反硝化降解的微生物机制提供一定的理论依据.
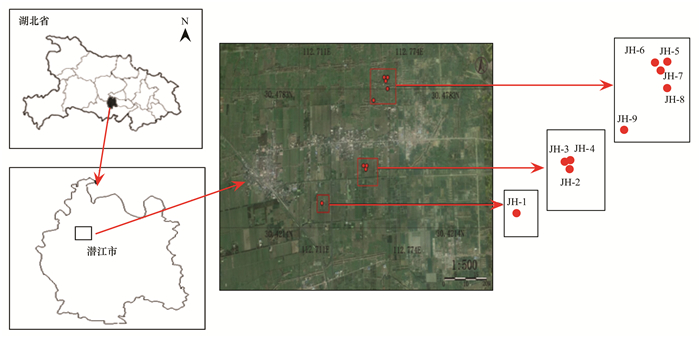
1 材料与方法 1.1 土壤样品的采集江汉油田位于湖北省的潜江市境内,已有50多年的油气开发历史,是新中国最早开发的油气田之一.本研究的土壤样品在2014年7月采集于江汉油田区域不同出油井附近的农田(水稻-油菜轮作)表层(0~20 cm).共选择了9个采样点(112°29′E~113°01E,30°04N~30°49′N),编号为JH-1~JH-9,在每个采样点中心周围30 m2的范围内,随机选择9个点,进行多点混合并初步剔除石块和杂物,以完成单个土样的采集,采样点的地理信息如图 1所示.采集好的样品装于自封袋后,置于放有冰袋的箱子中运回实验室.随后,过2 mm筛后分装,部分保存于4℃用于基本性质测定,部分置于-20℃冰箱用于DNA的提取,剩余部分风干后用于有机质等的测定.
|
图 1 土壤样品的地理信息 Fig. 1 Geographic information of soil samples |
土壤的pH值采用2.5 :1的水土比,用“PHS-3C型”pH仪测定.总有机质、有效磷及含水量的测定参照文献[19]进行,分别采用重铬酸钾氧化还原滴定法、钼锑抗比色法及比重法进行.氨氮和硝氮分别采用靛酚蓝分光光度法和萘乙二胺分光光度法测定[19].土壤PAHs的测定参照曹启民等[20]的方法进行,主要步骤如下:称取10 g预先于-80℃冷冻干燥的过100目筛的土壤样品于烧杯中,随后加入10 g无水硫酸钠和2 g铜粉至烧杯中混合均匀;无损移入滤纸桶,加入70 mL丙酮-正己烷(1 :1)混合液浸泡土壤样品12 h;将滤纸桶置于索氏提取器中75℃提取6 h,再将提取液于~46 kPa,45℃下旋转蒸发浓缩至1~2 mL,随后过装有1 cm石英砂、12 cm活化硅胶、6 cm中性氧化铝及1 cm无水硫酸钠的层析柱净化;以15 mL色谱级正己烷预洗后,用70 mL二氯甲烷和正己烷的混合液(3 :7)洗脱;最后,收集洗脱液旋转蒸发浓缩并用甲醇定容至1 mL,用高压液相色谱HITACHI L-7100(Hitachi High-Technologies Corporation, Japan)测定各样品PAHs含量.
1.3 土壤DNA的提取及nirK、nirS基因的定量PCR本实验中土壤样品DNA的提取,参照试剂盒说明书,使用Fast DNA® SPIN Kit for Soil (MP Biomedicals, USA)试剂盒进行.提取到的样品总DNA,用1%的琼脂糖凝胶电泳进行检测,并用DU 800 spectrophotometer (Beckman Coulter, USA)测定其浓度,最后保存于-20℃冰箱备用.
本研究中,nirK及nirS基因丰度的检测用SYBR GREEN法,在荧光定量PCR仪(ABI 7500, Applied Biosystems, USA)上进行.各基因扩增所用的引物序列及具体的扩增程序见表 1.定量PCR按照Premix Ex TaqTM(TaKaRa Biotechnology, Japan)的说明书进行,反应采用25 μL体系,其中含上下游引物各1 μL,DNA模板1 μL.实验中的标准曲线参照文献[21]的方法进行,在各反应完成后,设置了溶解曲线程序,用来检测real-time PCR产物的特异性,最后通过ABI 7500 Software (version 2.0.6)来分析所得的数据.
|
|
表 1 扩增基因用引物及PCR扩增程序 Table 1 Primer sets and PCR procedures |
1.4 克隆文库的构建及T-RFLP分析
为了全面解析所采土样中nirK及nirS基因的类群,本研究中分别构建了nirK及nirS基因的混合克隆文库,通过对混合克隆文库的分析,可为后续T-RFLP分析中限制性内切酶的选择提供依据,同时也有助于确定T-RFLP分析中所获取的各末端片段所对应的类群.现以nirK基因混合克隆文库的构建流程为例,简述如下:用不带FAM荧光标记的引物(见表 1)分别PCR扩增各样品的nirK基因,扩增采用25 μL体系,利用EX-Taq (Takara, Japan),按说明书进行;分别取各样品的nirK基因产物5 μL,混合均匀,从而得到该基因的混合PCR产物;再以混合的PCR产物为对象,纯化后连接至pGEM-T Easy载体(Promega, USA),转化大肠杆菌感受态细胞JM109(Takara, Japan),通过蓝白斑筛选,随机挑取300个左右的阳性克隆子,送上海生物工程有限公司进行测序,从而完成nirK基因混合克隆文库的构建.获取的序列用DNAMAN (Version 6.0.3.48)软件进行同源性分析,将同源性高于95%的序列归入同一操作分类单元(operational taxonomic unit, OTU),选取各OTU的代表性序列,采用邻位相邻法(Neighbor-Joining),用MEGA (Version 6.0)软件构建系统发育树[22].之后,用DNAMAN (Version 6.0.3.48)对各代表性OTU的序列进行限制性酶切位点分析,结果表明选择性内切酶HaeⅢ(Takara, Japan)能够较好地对nirK及nirS基因克隆文库中各代表性OTU进行分型,可作为后续T-RFLP分析中的选择性内切酶.将所获得的序列提交GenBank并获得相应的序列号,nirK: KX149075-KX149093,nirS: KX149049-KX149074.
在混合克隆文库构建完成的基础上,利用5′端带有FAM荧光标记的引物(表 1),分别对各样品的nirK及nirS基因进行PCR扩增,获取的目标产物经纯化后,在37℃用HaeⅢ酶切4 h,使用核酸测序仪ABI PRISM3700(Applied Biosystems, USA)进行基因扫描,用GeneMapper (Applied Biosystems, USA)软件对T-RFLP图谱进行分析.
1.5 数据处理实验数据的统计分析在SPSS 17.0软件中的单因素方差分析的S-N-K (Student-Newman-Keuls test)检验计算不同样品之间的差异显著性(P < 0.05).相关性用直线相关分析(Bivariate过程),采用Spearman相关系数计算,双尾显著性检验.文中nirK及nirS基因群落结构与环境因子关系的分析用CANOCO 4.5软件中的冗余分析(Redundancy analysis, RDA)来完成.
2 结果与讨论 2.1 土壤样品的基本性质土壤样品的基本性质如表 2所示.有机质含量在不同样品间存在显著差异,其中JH-5有机质含量最高(101.56 g ·kg-1),其次为JH-1、JH-3、JH-4和JH-8,它们的含量在38~41 g ·kg-1之间,其它各样品有机质含量均低于30 g ·kg-1.样品pH值的测定结果表明,各样品均为碱性土壤.有效磷含量较高的样品为JH-4(28.07 mg ·kg-1)和JH-8(25.38 mg ·kg-1),其次为JH-6(15.94 mg ·kg-1)和JH-7(11.39 mg ·kg-1),其余各样品的有效磷含量均低于10 mg ·kg-1.铵态氮含量在各样点土壤样品间的变化不大,其含量范围为4.03~4.60 mg ·kg-1.硝态氮含量较高的为JH-6(21.84 mg ·kg-1)和JH-8(29.91 mg ·kg-1),其次为JH-7(10.81 mg ·kg-1),其余各样品含量均低于10.00 mg ·kg-1. PAHs含量在各样点间有明显的不同,其中JH-4中的含量(8.10 mg ·kg-1)最高,其次为JH-8(2.01 mg ·kg-1)、JH-5(1.65 mg ·kg-1)和JH-2(1.23 mg ·kg-1),JH-6、JH-7和JH-9分别为0.80、0.63和0.61 mg ·kg-1,含量最少的为JH-1(0.22 mg ·kg-1)和JH-3(0.21 mg ·kg-1).据报道,长江上游重庆金佛山土壤16种优先控制PAHs的含量范围为0.24~2.12 mg ·kg-1[25].而对下游的长江三角洲区域土壤PAHs含量的调查表明,该区域土壤总PAHs含量范围为0.021~3.579 mg ·kg-1[26].由此可见,除样品JH-4外,本研究多数样点土壤中PAHs含量均在上述调查结果的范围之内.
|
|
表 2 土壤样品的基本性质1) Table 2 Soil basic properties |
2.2 土壤样品nirK及nirS基因的丰度
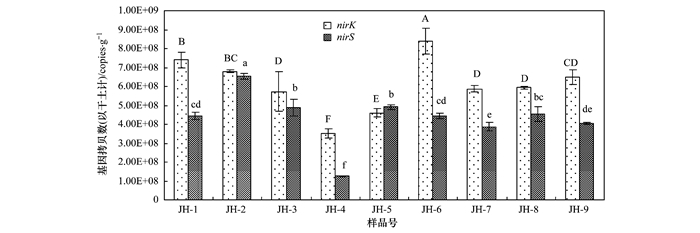
定量PCR的结果见图 2.从中可知,各土壤样品中nirK基因的丰度(以干土计,下同)在3.51×108~8.38×108 copies ·g-1之间,其中JH-6丰度最高(8.38×108 copies ·g-1),其次为JH-1(7.39×108 copies ·g-1)和JH-2(6.38×108 copies ·g-1),而样品JH-3、JH-7、JH-8、JH-9中该基因的丰度差异不显著,含量在5.7×108~6.9×108 copies ·g-1之间,样品JH-4中nirK基因丰度显著低于其它各样品.由图 2还可看出,油田土壤样品中nirS基因拷贝数在1.25×108~6.54×108 copies ·g-1之间,且各样点土壤样品中nirS基因的丰度均低于nirK基因.在样品JH-2中最高(6.54×108 copies ·g-1),与nirK基因相同,样品JH-4中nirS基因的丰度也最低(1.25×108 copies ·g-1),而其它各样点土壤样品中该基因的丰度均在3.86×108~4.9×108 copies ·g-1之间.
|
误差线为标准偏差(n=3),大写和小写字母分别指示nirK及nirS基因在各土壤样品间的差异显著(P < 0.05) 图 2 土壤样品中nirK及nirS基因拷贝数 Fig. 2 Copy numbers of nirK and nirS in soil samples |
从上述结果可以看出,本研究中各样点土壤中nirK基因的拷贝数均高于nirS基因,表明在江汉油田区域土壤中前者可能在亚硝酸盐还原酶基因中占有主导地位. Yoshida等[27]在研究稻田土壤中反硝化微生物丰度时,也发现其中nirK基因丰度显著高于nirS基因.一般认为土壤的基本性质,如有机质和硝酸根含量等[28],或水文等诸多因素均能影响到nirK及nirS基因的相对丰度[29].此外,在PAHs含量最高的样品JH-4中,nirK及nirS基因的丰度均显著低于其它各样品,这表明过高的PAHs含量可能会抑制土壤反硝化微生物的生长.而土壤反硝化功能基因拷贝数与PAHs含量的相关性分析也表明它们之间存在明显的负相关(nirK: R2=0.54, P < 0.05;nirS: R2=0.58, P < 0.05). Guo等[9]在研究芘对农业土壤中反硝化微生物群落组成时也发现,随着芘浓度的升高,土壤中nirK及nirS基因拷贝数均随之减少,这与本实验结果一致.由此看来,土壤反硝化相关nirK及nirS型反硝化微生物可能均对PAHs污染较为敏感,能够被PAHs所抑制.
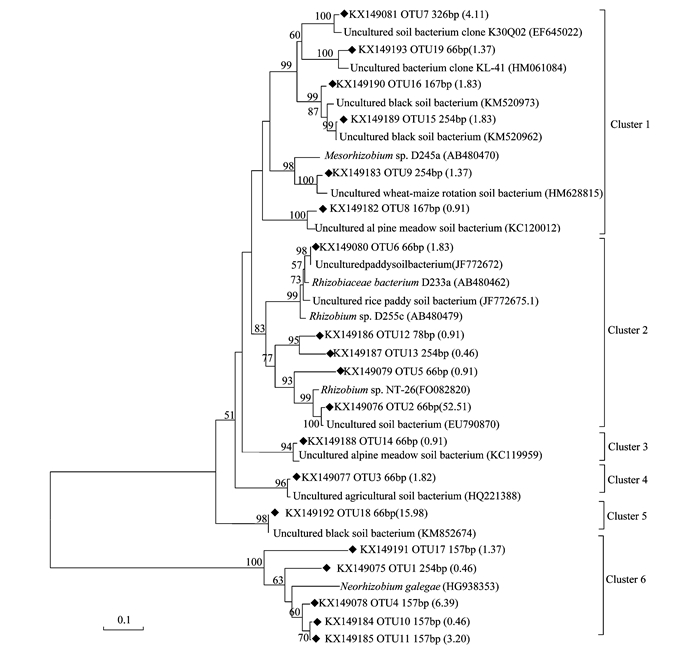
2.3 混合的nirK及nirS克隆文库分析分别从nirK及nirS的混合克隆文库中随机挑选出300及264个阳性克隆进行测序,最终分别获得219个nirK和177个nirS有效序列.以95%的序列相似性划分操作分类单元(operational taxonomic units, OTUs),获取的nirK序列可分为19个OTUs,nirS则可分为26个OTUs.最后,从每个OTU中挑选1个代表性序列来构建nirK(图 3)及nirS(图 4)基因的系统发育树.如图 3所示,油田区域土壤中的nirK基因可分为6个Clusters,占克隆文库中序列数比例较高的有Cluster 1(254bp、167bp,11.42%),Cluster 2(66bp,56.62%)和Cluster 6(157bp,11.88%),它们所含的序列共占克隆文库总序列数的82%,且分别与中生根瘤菌属、根瘤菌属、及新根瘤菌属根瘤菌有较高的同源性.而Clusters 3~5共占克隆序列总数的18.71%,分别与高山草甸、农田和黑土土壤中的未培养nirK序列具有较高的同源性.可见,该油田区域土壤中的优势的nirK型反硝化微生物为根瘤菌科细菌.
|
实验获得的序列信息(序列号、酶切片段大小)用◆标记,括号中的数字为该序列占总序列的百分比(%) 图 3 基于nirK基因克隆文库构建的系统发育树 Fig. 3 Neighbour-joining phylogenetic tree of nirK based on the clone library |
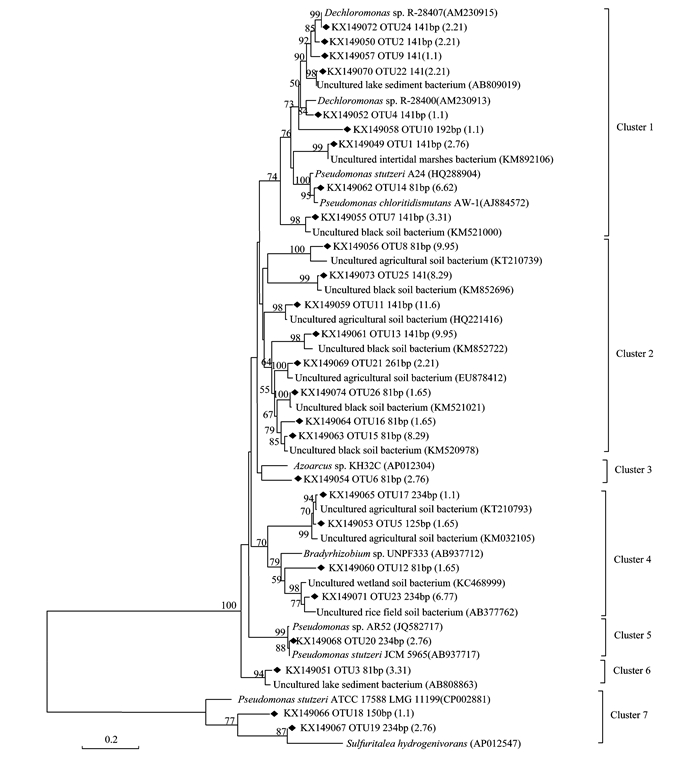
由nirS基因系统发育树(图 4)可知,该油田区域土壤中nirS型微生物可分为7个Clusters.其中Cluster 2(81 bp、141 bp)所含序列占克隆文库总序列数的53.59%,且为未培养的土壤nirS型微生物.其它含量较高的依次为Cluster 1(141 bp、192 bp),Cluster 5(234 bp)和Cluster 4(81、125、234 bp),所含序列分别占克隆文库序列总数的22.26%、15.98%和11.17%,且分别与红环菌科的脱氯单胞菌属、假单胞菌属及慢生根瘤菌属有较高的同源性.其中,红环菌科的nirS型微生物被报道为沼泽和水稻土中的优势nirS型微生物类群[30, 31].所含序列比例较少的为Cluster 3(81 bp)、Cluster 6(81 bp)和Cluster 7(150 bp、234 bp),共占序列总数的9.93%,它们分别与固氮弓菌属、未培养的nirS微生物及硫酸盐还原菌具有较高的同源性.通过上述对nirK、nirS混合克隆文库及构建的系统发育树的分析,可为后续T-RFLP中确认和分析各样点土壤样品中不同长度的限制性末端片段所代表的nirK及nirS类群提供依据.
|
实验获得的序列信息(序列号、酶切片段大小)用◆标记,括号中的数字为该序列占总序列的百分比(%) 图 4 基于nirS基因克隆文库构建的系统发育树 Fig. 4 Neighbour-joining phylogenetic tree of nirS based on the clone library |
各样点土壤样品中nirK基因末端限制性片段图谱的组成(图 5)分析表明,样品JH-1、JH-2、JH-3、JH-5、JH-7中nirK基因的群落组成较为相似,157 bp的限制性末端片段所代表的类群为它们的最优势nirK类群,在上述样品中的比例分别为76%、70%、70%、100%和75%.参照nirK基因系统发育树(图 3)可知,157 bp所代表的根瘤菌科的新根瘤菌属为上述样点土壤样品中的优势nirK类群.而样点JH-6、JH-8和JH-9中157 bp的末端片段所占比例大幅下降,且出现了特异性的78 bp (未培养的nirK类群)和167 bp (中生根瘤菌属)的片段.此外,PAHs含量最高的JH-4具有与其它各样点不同的nirK基因末端片段组成,134 bp的片段所代表的类群(未培养的nirK类群)为其优势nirK种类,占其nirK基因T-RFLP末端片段总量的45%.
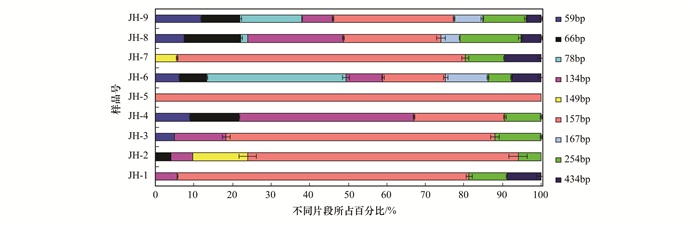
|
图 5 土壤样品中nirK基因群落组成 Fig. 5 Community composition of nirK in soil samples |
将土壤的理化性质及T-RFLP中的各限制性末端片段分别作为环境和物种变量,利用CANOCO 4.5软件对土壤环境因子与nirK型反硝化微生物群落结构进行冗余分析(图 6).结果表明,样品JH-1、JH-2、JH-3、JH-5和JH-7之间的距离较近,JH-6和JH-9间的距离较近,而PAHs含量较高的样品JH-4和JH-8在一个象限,这与上述T-RFLP图谱组成分析的结果基本一致.由图 6还可看出,除能够作为微生物氮源及磷源的铵态氮和有效磷外,土壤PAHs含量同样也是影响该区域土壤nirK基因群落组成最重要的环境因子.此外,RDA分析还可看出,134 bp (不确定类群)和66 bp (根瘤菌属)所代表的nirK类群与土壤PAHs含量呈正相关,表明它们可能在PAHs反硝化代谢中起到作用,其中根瘤菌属已经被证实能够在芘污染土壤中得到富集[22].如Keum等[32]从PAHs污染土壤中分离出根瘤菌,并发现其能够以菲为单一碳源生长,且能够有效降解萘和菲.
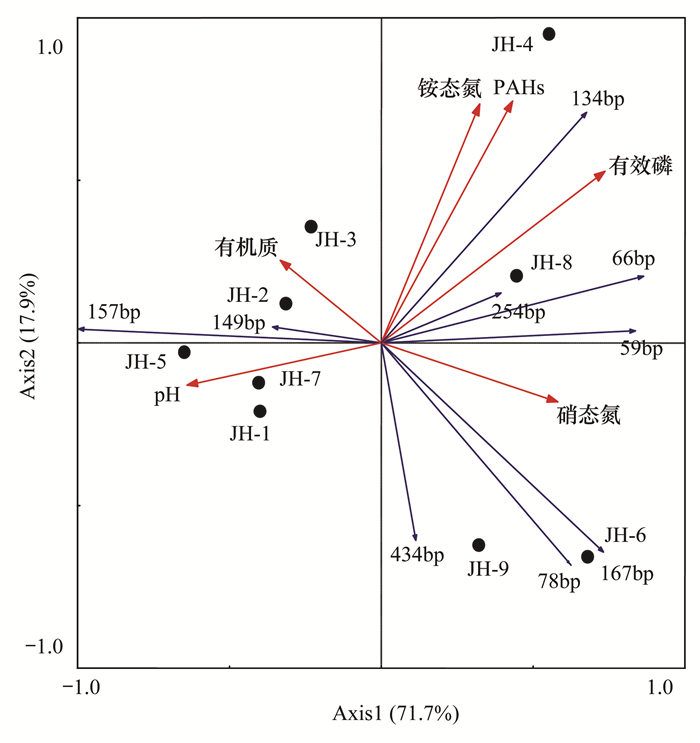
|
图 6 土壤样品中nirK基因群落与土壤基本性质的RDA分析 Fig. 6 RDA analysis based on the T-RFLP profiles and soil properties of nirK |
由基于nirS基因T-RFLP剖面的末端片段组成分析(图 7)可知,相较于nirK,nirS基因群落组成在不同样点土壤样品中的差异并不明显,除JH-8全部为141 bp所代表的类群(脱氯单胞菌属)组成外,其余样品间均具有3种以上共有的末端片段类群.此外,样品JH-1和JH-7中检测到了特异性的192 bp (假单胞菌属),样品JH-2、JH-3和JH-4检测到特异性的161 bp (不确定类群).而在PAHs含量较高的样品JH-2和JH-4中检测到了特异性的234 bp类群(假单胞菌属),且在后者中比例最高.基于nirS基因群落组成及土壤基本性质的RDA分析(图 8)表明,样品JH-1、JH-3、JH-5和JH-9在一个象限,JH-2、JH-4及JH-7在一个象限,JH-6和JH-8位于一个象限.由此看来,PAHs含量较高的JH-4和JH-8中nirS基因群落组成与其它样点的差异并不明显.影响各样点土壤样品中nirS基因群落组成的主要环境因子为土壤硝酸盐和有效磷含量,硝酸盐是反硝化微生物电子传递链的末端电子受体,而有效磷是微生物生长不可或缺的大量元素之一,因此它们均有可能是限制土壤nirS型微生物群落结构的关键因子.此外,由图 8还可看出,141、161和234 bp所代表的nirS类群与土壤PAHs含量呈正相关,除161 bp为不确定类群外,141 bp为脱氯单胞菌属,234 bp为假单胞菌属,这些属的微生物已经被证实能够在有氧及厌氧条件下对PAHs进行降解.当前的研究表明,假单胞菌属在有机物污染环境中普遍存在,它可以以这些有机污染物作为碳源和能源,在有氧及厌氧条件下对这些有机污染物进行降解[33],且假单胞菌属对典型PAHs (萘、蒽)的降解也已经为人们所证实[34~36].脱氯单胞菌属经常从被污染物污染的地方分离得到,能够在厌氧及微氧条件下分解苯和甲苯[37].由此看来,尽管nirS基因的群落结构对PAHs的响应并不积极,但nirS型的假单胞菌有可能在油田区土壤PAHs的反硝化代谢中起到不可忽视的作用.
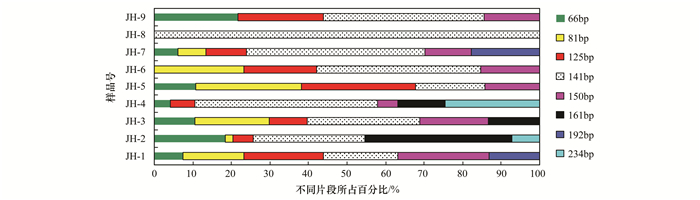
|
图 7 土壤样品中nirS基因群落组成 Fig. 7 Community composition of nirS in soil samples |
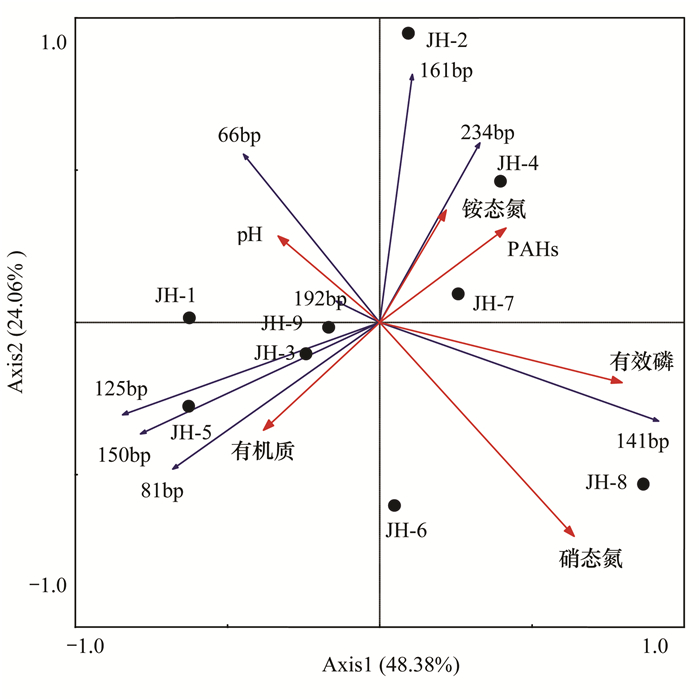
|
图 8 土壤样品中nirS基因群落与土壤基本性质的RDA分析 Fig. 8 RDA analysis based on the T-RFLP profiles and soil properties of nirS |
(1)油田区土壤中nirK基因丰度均高于nirS,且它们均与土壤的PAHs含量呈显著负相关.
(2)油田区土壤中nirK型反硝化微生物的主要类群为根瘤菌科,且土壤PAHs含量是影响nirK型反硝化微生物群落组成的关键因子.
(3)与nirK型反硝化微生物相比较,土壤PAHs含量并不是影响油田区nirS基因群落组成的关键因子,硝酸盐及有效磷含量对其有显著影响,但重要的有机污染物降解菌-nirS型假单胞菌与土壤PAHs含量呈正相关,表明nirS型假单胞菌在潜在PAHs污染的油田区土壤中PAHs的反硝化代谢中可能起到重要作用.
| [1] | Eggen T, Majcherczyk A. Removal of polycyclic aromatic hydrocarbons (PAH) in contaminated soil by white rot fungus Pleurotus ostreatus[J]. International Biodeterioration & Biodegradation, 1998, 41(2) : 111–117. |
| [2] | Wild S R, Jones K C. Polynuclear aromatic hydrocarbons in the United Kingdom environment: a preliminary source inventory and budget[J]. Environmental Pollution, 1995, 88(1) : 91–108. DOI: 10.1016/0269-7491(95)91052-M |
| [3] | Zhou Q X, Hua T. Bioremediation: A review of applications and problems to be resolved[J]. Progress in Natural Science, 2004, 14(11) : 937–944. DOI: 10.1080/10020070412331344601 |
| [4] | Bamforth S M, Singleton I. Bioremediation of polycyclic aromatic hydrocarbons: current knowledge and future directions[J]. Journal of Chemical Technology and Biotechnology, 2005, 80(7) : 723–736. DOI: 10.1002/(ISSN)1097-4660 |
| [5] | Mulligan C N, Yong R N, Gibbs B F. Surfactant-enhanced remediation of contaminated soil: a review[J]. Engineering Geology, 2001, 60(1-4) : 371–380. DOI: 10.1016/S0013-7952(00)00117-4 |
| [6] | Li C H, Zhou H W, Wong Y S, et al. Vertical distribution and anaerobic biodegradation of polycyclic aromatic hydrocarbons in mangrove sediments in Hong Kong, South China[J]. Science of the Total Environment, 2009, 407(21) : 5772–5779. DOI: 10.1016/j.scitotenv.2009.07.034 |
| [7] | Lu X Y, Zhang T, Fang H H P. Bacteria-mediated PAH degradation in soil and sediment[J]. Applied Microbiology and Biotechnology, 2011, 89(5) : 1357–1371. DOI: 10.1007/s00253-010-3072-7 |
| [8] | Philippot L. Denitrifying genes in bacterial and Archaeal genomes[J]. Biochimica et Biophysica Acta (BBA)-Gene Structure and Expression, 2002, 1577(3) : 355–376. DOI: 10.1016/S0167-4781(02)00420-7 |
| [9] | Guo G X, Deng H, Qiao M, et al. Effect of pyrene on denitrification activity and abundance and composition of denitrifying community in an agricultural soil[J]. Environmental Pollution, 2011, 159(7) : 1886–1895. DOI: 10.1016/j.envpol.2011.03.035 |
| [10] | 梁丽华, 左剑恶. 反硝化功能基因--检测反硝化菌种群结构的分子标记[J]. 微生物学通报, 2009, 36(4) : 627–633. Liang L H, Zuo J E. Denitrifying functional genes-the molecular marker for detection of denitrifying community structure[J]. Microbiology China, 2009, 36(4) : 627–633. |
| [11] | Mihelcic J R, Luthy R G. Microbial degradation of acenaphthene and naphthalene under denitrification conditions in soil-water systems[J]. Applied and Environmental Microbiology, 1988, 54(5) : 1188–1198. |
| [12] | Mcnally D L, Mihelcic J R, Lueking D R. Biodegradation of three-and four-ring polycyclic aromatic hydrocarbons under aerobic and denitrifying conditions[J]. Environment Science & Technology, 1998, 32(17) : 2633–2639. |
| [13] | Eriksson M, Sodersten E, Yu Z T, et al. Degradation of polycyclic aromatic hydrocarbons at low temperature under aerobic and nitrate-reducing conditions in enrichment cultures from northern soils[J]. Applied and Environmental Microbiology, 2003, 69(1) : 275–284. DOI: 10.1128/AEM.69.1.275-284.2003 |
| [14] | Chang B V, Chang S W, Yuan S Y. Anaerobic degradation of polycyclic aromatic hydrocarbons in sludge[J]. Advances in Environmental Research, 2003, 7(3) : 623–628. DOI: 10.1016/S1093-0191(02)00047-3 |
| [15] | Dou J F, Liu X, Ding A Z. Anaerobic degradation of naphthalene by the mixed bacteria under nitrate reducing conditions[J]. Journal of Hazardous Materials, 2009, 165(1-3) : 325–331. DOI: 10.1016/j.jhazmat.2008.10.002 |
| [16] | Lu X Y, Zhang T, Fang H H P, et al. Biodegradation of naphthalene by enriched marine denitrifying bacteria[J]. International Biodeterioration & Biodegradation, 2011, 65(1) : 204–211. |
| [17] | Yang X N, Ye J X, Lyu L M, et al. Anaerobic biodegradation of pyrene by Paracoccus denitrificans under various nitrate/nitrite-reducing conditions[J]. Water, Air, & Soil Pollution, 2013, 224 : 1578–1578. |
| [18] | 吴艳阳, 吴群河, 黄珊, 等. 沉积物中多环芳烃对反硝化功能基因垂直分布的影响[J]. 环境科学, 2012, 33(10) : 3592–3597. Wu Y Y, Wu Q H, Huang S, et al. Effect of Polycyclic Aromatic Hydrocarbons on the Vertical Distribution of Denitrifying Genes in River Sediments[J]. Environmental Science, 2012, 33(10) : 3592–3597. |
| [19] | 杨剑虹, 王成林, 代亨林. 土壤农化分析与环境监测[M]. 北京: 中国大地出版社, 2008. |
| [20] | 曹启民, 陈桂珠, 缪绅裕. 多环芳烃的分布特征及其与有机碳和黑碳的相关性研究--以汕头国际湿地示范区三种红树林湿地表层沉积物为例[J]. 环境科学学报, 2009, 29(4) : 861–868. Cao Q M, Chen G Z, Liao S Y. Distribution correlations of polycyclic aromatic hydrocarbOns with organic carbon and black carbon in surface sediments of three mangrove wetlands in the Shantou Wetland Demonstration Site, China[J]. Acta Scientiae Circumstantiae, 2009, 29(4) : 861–868. |
| [21] | He J Z, Shen J P, Zhang L M, et al. Quantitative analyses of the abundance and composition of ammonia-oxidizing bacteria and ammonia-oxidizing archaea of a Chinese upland red soil under long-term fertilization practices[J]. Environmental Microbiology, 2007, 9(9) : 2364–2374. DOI: 10.1111/emi.2007.9.issue-9 |
| [22] | Tamura K, Dudley J, Nei M, et al. MEGA4: Molecular evolutionary genetics analysis (MEGA) software version 4.0[J]. Molecular Biology and Evolution, 2007, 24(8) : 1596–1599. DOI: 10.1093/molbev/msm092 |
| [23] | Braker G, Fesefeldt A, Witzel K P. Development of PCR primer systems for amplification of nitrite reductase genes (nirK and nirS) to detect denitrifying bacteria in environmental samples[J]. Applied and Environmental Microbiology, 1998, 64(10) : 3769–3775. |
| [24] | Michotey V, Mejean V, Bonin P. Comparison of methods for quantification of cytochrome cd (1)-denitrifying bacteria in environmental marine samples[J]. Applied and Environmental Microbiology, 2000, 66(4) : 1564–1571. DOI: 10.1128/AEM.66.4.1564-1571.2000 |
| [25] | 师阳, 孙玉川, 梁作兵, 等. 重庆金佛山土壤中PAHs含量的海拔梯度分布及来源解析[J]. 环境科学, 2015, 36(4) : 1417–1424. Shi Y, Sun Y C, Liang Z B, et al. Altitudinal gradient distribution and source analysis of PAHs content of topsoil in Jinfo Mountian, Chongqing[J]. Environmental Science, 2015, 36(4) : 1417–1424. |
| [26] | 李静雅, 吴迪, 许芸松, 等. 长江三角洲区域表土中多环芳烃的近期分布与来源[J]. 环境科学, 2016, 37(1) : 253–261. Li J Y, Wu D, Xu Y S, et al. Recent distribution and sources of polycyclic aromatic hydrocarbons in surface soils from Yangtze River Delta[J]. Environmental Science, 2016, 37(1) : 253–261. |
| [27] | Yoshida M, Ishii S, Otsuka S, et al. Nirk-harboring denitrifiers are more responsive to denitrification-inducing conditions in rice paddy soil than nirS-harboring bacteria[J]. Microbes and Environments, 2010, 25(1) : 45–48. DOI: 10.1264/jsme2.ME09160 |
| [28] | Bárta J, Melichová T, Vaněk D, et al. Effect of pH and dissolved organic matter on the abundance of nirK and nirS denitrifiers in spruce forest soil[J]. Biogeochemistry, 2010, 101(1-3) : 123–132. DOI: 10.1007/s10533-010-9430-9 |
| [29] | Levy-Booth D J, Winder R S. Quantification of nitrogen reductase and nitrite reductase genes in soil of thinned and clear-cut Douglas-Fir stands by using Real-time PCR[J]. Applied and Environmental Microbiology, 2010, 76(21) : 7116–7125. DOI: 10.1128/AEM.02188-09 |
| [30] | Saito T, Ishii S, Otsuka S, et al. Identification of novel Betaproteobacteria in a succinate-assimilating population in denitrifying rice paddy soil by using stable isotope probing[J]. Microbes and Environments, 2008, 23(3) : 192–200. DOI: 10.1264/jsme2.23.192 |
| [31] | Priemé A, Braker G, Tiedje J M. Diversity of nitrite reductase (nirK and nirS) gene fragments in forested upland and wetland soils[J]. Applied and Environmental Microbiology, 2002, 68(4) : 1893–1900. DOI: 10.1128/AEM.68.4.1893-1900.2002 |
| [32] | Keum Y S, Seo J S, Hu Y T, et al. Degradation pathways of phenanthrene by Sinorhizobium sp. C4[J]. Applied Microbiology and Biotechnology, 2006, 71(6) : 935–941. DOI: 10.1007/s00253-005-0219-z |
| [33] | Ridgway H F, Safarik J, Phipps D, et al. Identification and catabolic activity of well-derived gasoline-degrading bacteria from a contaminated aquifer[J]. Applied and Environmental Microbiology, 1990, 56(11) : 3565–3575. |
| [34] | Rockne K J, Chee-Sanford J C, Sanford R A, et al. Anaerobic naphthalene degradation by microbial pure cultures under nitrate-reducing conditions[J]. Applied and Environmental Microbiology, 2000, 66(4) : 1595–1601. DOI: 10.1128/AEM.66.4.1595-1601.2000 |
| [35] | Lee K, Park J W, Ahn I S. Effect of additional carbon source on naphthalene biodegradation by Pseudomonas putida G7[J]. Journal of Hazardous Materials, 2003, 105(1-3) : 157–167. DOI: 10.1016/j.jhazmat.2003.08.005 |
| [36] | Jacques R J S, Santos E C, Bento F M, et al. Anthracene biodegradation by Pseudomonas sp. isolated from a petrochemical sludge landfarming site[J]. International Biodeterioration & Biodegradation, 2005, 56(3) : 143–150. |
| [37] | Wolterink A, Kim S, Muusse M, et al. Dechloromonas hortensis sp. nov. and strain ASK-1, two novel (per) chlorate-reducing bacteria, and taxonomic description of strain GR-1[J]. International Journal of Systematic and Evolutionary Microbiology, 2005, 55(5) : 2063–2068. DOI: 10.1099/ijs.0.63404-0 |
 2016, Vol. 37
2016, Vol. 37


