2. 同济大学环境科学与工程学院, 上海 200092
2. College of Environmental Science and Engineering, Tongji University, Shanghai 200092, China
污水回用是解决我国水资源短缺的主要途径,消毒是保证污水回用卫生学安全的主要工艺,紫外消毒具有广谱的杀菌作用,且较少形成消毒副产物[1, 2],生态风险小,已经成为污水回用的常用消毒方法. 紫外消毒的作用机理是在波长为200-300 nm的紫外线照射下,微生物DNA分子上的胸腺嘧啶形成嘧啶二聚体,抑制了基因的转录和翻译过程[3],从而杀死细胞. 目前,污水回用紫外消毒的研究主要集中在水质对消毒效果的影响[4],而紫外消毒对微生物的作用主要集中在饮用水处理方面[5, 6]. 污水中微生物浓度相对较高,含有多种病原菌包括沙门氏菌、 分枝杆菌、 军团菌等[7],此外二级出水中化学污染物的浓度相对较高,成分相对复杂[4],因此紫外消毒对水中微生物的影响与饮用水不同. 目前,水环境细菌的检测仍以培养法为主. 无法较好地指示活性菌的存在,qPCR技术能够较好地指示细菌的存在,但无法区分活性菌与非活性菌,高估了样品中活性病原菌的浓度进而无法很好地评估污水回用的风险[8]. RNA半衰期较短,在非活性菌内会快速降解而在活性体内稳定存在,可以很好地指示活性菌的存在. 有学者建立的基于RNA的Q-RT-PCR技术能快速、 灵敏地检测活性菌[9]. 已有研究中,通过揭示二级出水中微生物(特别是病原菌)群落结构,进而考察紫外消毒对污水回用中主要活性病原菌存在情况的研究较少.
鉴于此,本研究首先采用454焦磷酸测序技术,分析二级出水中微生物(特别是病原菌)群落结构特性,揭示二级出水中常见的几种病原菌; 然后采用培养法、 qPCR及Q-RT-PCR方法考察了紫外消毒对该污水厂二级除水中指示菌(大肠杆菌)和病原菌(沙门氏菌、 分枝杆菌)的去除特性,以期为污水回用中病原菌的去除工艺提供一些参考,保障污水回用的卫生学安全.
1 材料与方法 1.1 样品采集及实验设置本研究样品采自南方某生活污水处理厂二级出水,该厂采用传统活性污泥法,二级处理采用空气曝气活性污泥法,该厂进水与出水的水质参数情况如表 1所示. 二级出水的取样体积为5 L. 样品取好后放置于采样箱(内置冰块)中,于2 h内运回实验室,1 L水样用于群落结构分析. 共采样5次.
|
|
表 1 本实验用再生水的水质参数 Table 1 Water quality parameters of reclaimed waters used in this study |
紫外消毒对病原菌的影响在实验室进行. 紫外辐射采用超净台中的紫外灯管(苏信净化设备生产厂,产品型号:YJ 1320)进行. 经紫外辐射计测定,本研究采用的紫外灯管对实验中水样放置区域的紫外照射强度均匀. 实验中采用校准的紫外辐射计对玻璃杯处的紫外强度为 0.1mW ·cm-2. 将1.5 L水样倒入2L烧杯中,放入磁性转子,将烧杯放在磁力搅拌器上,打开超净台的紫外灯,使得水样接受紫外照射. 实验所需的紫外线剂量(mJ ·cm-2)=紫外线强度(mW ·cm-2)×照射时间(s).
《城镇给排水紫外线消毒设备》标准(GB/T 19857-2005)规定:城镇生活饮用水的技术标准为40 mJ ·cm-2,中水回用的技术标准为80 mJ ·cm-2,城镇污水的技术标准为15 mJ ·cm-2(一级B标准),城镇污水的技术标准为20 mJ ·cm-2(一级A标准). 紫外线消毒设备应用于城市杂用水、 景观环境用水时,应分别达到GB-T 18920和GB-T 50335中的卫生学指标要求. 再生水作为景观环境用水时粪大肠菌群要求小于200 CFU ·L-1. 本研究中,设定紫外线剂量20、 40、 60、 80 mJ ·cm-2时,分别用培养法检测粪大肠菌群的浓度水平,考察不同紫外剂量对本样品中微生物的去除水平,以选择合适的紫外剂量.
经选择,考虑经济成本,最终设定紫外线剂量为60mJ ·cm-2,即照射时间为10 min. 照射后在黑暗条件下放置12 h. 本实验在室温环境下进行,反应终止后采用培养法和qPCR及Q-RT-PCR检测消毒前后大肠杆菌、 沙门氏菌及分枝杆菌的浓度.
1.2 二级出水中微生物多样性分析 1.2.1 DNA的提取将1L水样经醋酸纤维素滤膜过滤,将过滤下的物质于滤膜一同转入15 mL无菌离心管中,加入10 mL灭菌后的PBS在漩涡振荡器上洗脱过滤截留的颗粒物. 将滤膜取出,离心管置于4℃离心10 min,转速设为10 000 r ·min-1(日立,型号:CF16RN). 弃去上清液,将底部物质转移到2 mL离心管中,于4℃离心5 min,转速为12 000 r ·min-1. 弃去上清液,管内留500 μL液体进行DNA的提取. DNA提取采用FastDNA@ Spin Kit for Soil(MP Biomedicals,产品编号:6560200)试剂盒,具体操作过程按说明书进行. 最后得到DNA体积为50 μL.对得到的DNA进行纯化,采用Universal DNA 纯化回收试剂盒(天根,产品编号:DP214). 提取后的DNA用于454焦磷酸测序.
1.2.2 PCR扩增PCR扩增区域为细菌16S rRNA的V1-V3区,扩增长度为500 bp. 上游引物为:27F:5′- 融合 A-标签-CA接头-AGAGTTTGATCMTGGCTCAG-3′,下游引物为:533R:5′- 融合 B-TC 接头-TTACCGCGG CTGCTGGCAC-3′. 为了区分同一体系中的不同样品,在引物前端加入一个10 bp的标签. PCR反应体系为50 μL:5×FastPfu Buffer 10 μL; 2.5 μmol ·L-1 dNTPs 5 μL; 5 μmol ·L-1上下游引物2 μL; FastPfu Polymerase 1 μL; DNA模板100-200 ng; 其余用双蒸水补足50 μL. PCR扩增反应程序为:先95℃ 2 min; 然后进行30个循环(95℃ 30 s,55℃ 30 s,72℃ 30 s),最后72℃ 10 min. 将PCR产物进行2%琼脂糖凝胶电泳在500 bp位置处检测到条带,将PCR产物用TaKaRaMiniBEST Agarose Gel DNA Extraction凝胶回收试剂盒(TaKaRa,产品号:9762)回收PCR产物. 采用分光光度计测定DNA的浓度及质量(Nanodrop-2000c).
1.2.3 454文库构建和测序应用高通量测序平台GS 454 FLX Titanium(美国Roche公司)对标记的PCR产物进行测序. 测序完成后对所得序列进行处理,处理原则如下:①检测标签的完整性,弃去标签不完整的序列(即使只有一个碱基对发生错误); ②弃去片段长度低于200 bp的序列; ③去除尾部质量数低于20的末端序列; ④保证每个样品系列中80%以上碱基的序列质量数大于20[10].
1.2.4 测序结果分析及分类学分析对1.2.3节得到的有效序列比对至SILVA数据库(http://www.mothur.org/w/images/9/98/Silva.bacteria.zip)的16S rRNA序列上,去除目标区域以外的序列. 利用mothur软件(http://www.mothur.org),根据每组中的重复序列,去除嵌合体(chimera)序列,通过对RDP数据库中(http://www.mothur.org/w/images/5/59/Trainset9_032012.pds.zip)的PDS序列进行比对,排除叶绿体、 线粒体、 古细菌、 真核序列的干扰. 根据序列的相似性,将之归为多个OUT. 选择相似水平为0.03的进行后续分析,构建稀疏曲线. 利用mothur软件计算样本的Chao丰度指数(http://www.mothur.org/wiki/Chao),Shannon多样性指数(http://www.mothur.org/wiki/Shannon),Simpson多样性指数(http://www.mothur.org/wiki/Simpson),覆盖度指数(http://www.mothur.org/wiki/Coverage)及Pielou均匀度指数(http://en.wikipedia.org/wiki/Species_evenness).
1.3 细菌的检测 本研究中选择常用的指示菌大肠杆菌作为目标指示菌. 对测序结果进行分析,最终选择沙门氏菌和分枝杆菌作为待检测的病原菌. 分别采用培养法、 qPCR及Q-RT-PCR方法检测水样中细菌的浓度水平. 1.3.1 培养法大肠杆菌、 沙门氏菌及分枝杆菌的培养法检测参考文献[11]进行.
1.3.2 DNA提取消毒前病原菌的分析采用1.2.1节提取得到的DNA. 消毒后浓缩1L待测样品,提取过程按照1.2.1节进行.
1.3.3 qPCR(1)qPCR引物及探针的选择
本研究中大肠杆菌和分枝杆菌的qPCR检测采用SYBR® Green qPCR. 采用Taqman® qPCR定量检测沙门氏菌. 所采用的目的基因和相应引物序列见表 2.
|
|
表 2 定量PCR采用的目的基因和相应引物序列 Table 2 Gene targets and primers used for qPCR detection |
(2)qPCR标准品的制备
标准品采用质粒标准品,制作方法参见文献[15],制备含有uidA、 invA和hsp65基因的质粒DNA.
大肠杆菌标准品构建过程中采用大肠杆菌和与uidA基因相应的引物,沙门氏菌和与invA基因相应的引物,分枝杆菌标准品的构建采用分枝杆菌和hsp65基因相应的引物,目标基因经PCR扩增之后将特异性产物连接入pCR 2.1-TOPO载体(Invitrogen) 中,经过酶切鉴定测序分析和质粒提取等步骤获得大肠杆菌DNA标准品. 采用核酸蛋白仪测量DNA的浓度. 将得到的质粒DNA按10倍梯度稀释,制得qPCR标准品.
(3)qPCR测定
大肠杆菌及分枝杆菌qPCR(TaKaRa,Code:DRR041A)反应体系为(20 μL):SYBR® Premix Ex TaqTM 10 μL,上下游引物各0.5 μL(终浓度0.25 μmol ·L-1),DNA(标准品或样品)5 μL,双蒸水4 μL. 沙门细菌qPCR(TaKaRa,Code:DRR039A)反应体系为(25 μL):Premix Ex TaqTM 12.5 μL,上下游引物各2.5 μL(终浓度1.0 μmol ·L-1),Taqman® 荧光探针1 μL(终浓度0.25 μmol ·L-1),DNA(标准品或样品)5 μL,双蒸水1.5 μL补足体积至25 μL.
大肠杆菌反应程序如下:1个循环:95℃,1 min; 40个循环:95℃,20 s; 60℃,20 s; 72℃,15 s(收集荧光); 熔解曲线的过程为:95℃,1 min,从60℃开始每30 s温度升高0.5℃,总共进行71个循环,结束温度为95℃,反应结束之后4℃保存.
沙门氏菌反应程序如下:1个循环:95℃,10 min; 40个循环:95℃,20 s; 60℃,30 s; 72℃,25 s(收集荧光); 不设置熔解曲线,反应结束之后4℃保存.
分枝杆菌反应程序如下:1个循环:94℃,1 min; 40个循环:94℃,60 s; 60℃,60 s; 72℃ 60 s(收集荧光); 熔解曲线过程为同大肠杆菌.
每次定量PCR反应都设空白对照(以相同体积的双蒸水取代DNA),每次测定设2个平行样.
1.3.4 Q-RT-PCRQ-RT-PCR标准品的制备参考文献[7].
样品的浓缩参照1.2.1. RNA的提取采用Fast RNA® Pro Soil-Direct Kit(MP Biomedical,产品编 号:116070050).
2 结果与讨论 2.1 二级出水中微生物群落结构组成 2.1.1 微生物多样性分析通过对5个批次样品16S rRNA基因文库进行焦磷酸测序,经修剪去杂后,共获得1 128、 1 299、 1 292、 1 019和1 462条优化序列,序列平均长度为545 bp. 将优化序列截齐后与SILVA比对后进行聚类,在97%相似性下分别获得252、 264、 375、 315和554 OTUs,稀疏曲线如图 1所示. 从中可知,当测序数量超过1 000时,仍有新的OUT可被测出. 稀疏曲线随测序序列增加趋向平坦,表明本研究样本取样量合理. 表 3列出了α多样性分析各样品的多样性指数.
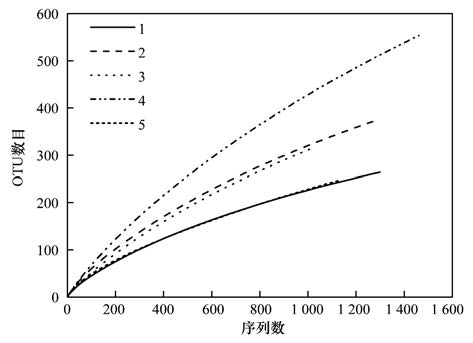
|
图 1 二级出水样品的稀疏曲线 Fig. 1 Rarefaction curves of secondary effluent samples |
|
|
表 3 二级出水中微生物物种丰富度和多样性评价 Table 3 Richness and diversity estimators of bacteria in secondary effluent samples |
对测序结果进行门的组成进行分析(对5个样品的测得结果进行累计,当细菌门比例小于1%时,划入其他类别),分析结果如图 2所示. 相似性为97%时,样品中共检测到21个门,包括酸杆菌门(Acidobacteria)、 放线菌门(Actinobacteria)、 (Armatimonadetes)、 拟杆菌门(Bacteroidetes)、 绿弯菌门(Chloroflexi)、 蓝细菌门(Cyanobacteria)、 Deinococcus-Thermus、 厚壁菌门(Firmicutes)、 梭杆菌门(Fusobacteria)、 Gemmatimonadete、 硝化螺旋菌门(Nitrospira)、 浮霉菌门(Planctomycetes)、 变形菌门(Proteobacteria)、 螺旋菌门(Spirochaetes)、 互养菌门(Synergistetes)和疣微菌门(Verrucomicrobia). 样品中有794条序列不能被分入已知菌门,这些细菌可能是未知菌种.
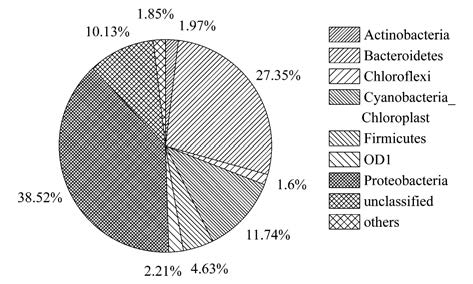
|
图 2 样品中细菌群落结构门比例组成 Fig. 2 Proportion of bacterial community structure in the sample |
由图 2可知,样品中包括蓝细菌门(Cyanobacteria)、 拟杆菌门(Bacteroidetes)、 厚壁菌门(Firmicutes)、 变形菌门(Proteobacteria)这4个主要门及未定菌(OD1)和其它未分类菌. Ye等[16]在研究香港一座采用活性污泥法的污水厂微生物群落结构中发现,污水厂二级出水中变形菌门(Proteobacteria)是最主要的细菌群. Lee等[17]在研究首尔不同污水厂进水及活性污泥的微生物群落结构中同样也发现,变形菌门(Proteobacteria)、 拟杆菌门(Bacteroidetes)及厚壁菌门(Firmicutes)是依次最主要的3个细菌群. Shu等[18]在研究陕西6座污水处理厂(包括工业污水处理厂及市政污水处 理厂)厌氧消化污泥的微生物群落结构中,也得到了同样的结论,变形菌门(Proteobacteria)、 拟杆菌门(Bacteroidetes)、 绿弯菌门(Chloroflexi)及厚壁菌门(Firmicutes)是依次主要的4个细菌群. 本研究得到的结论与这些研究结果相一致.
对两个主要门变形菌门(Proteobacteria)和拟杆菌门(Bacteroidetes)进行进一步分析,结果如图 3所示.
由图 3(a)可知,变形菌门(Proteobacteria)主要由α-、 β-和γ-Proteobacteria这3个主要亚纲组成,分别为23.9%、 43.5%和12.9%. 值得注意的是β-Proteobacteria亚纲包含许多种类的致病菌,在本研究中也检测到. 由图 3(b)可知,拟杆菌门(Bacteroidetes)主要由Flavobacteria、 Sphingobacteria和Bacteroidia这3个主要纲组成,分别为79.5%、 11.6%和2.5%. 上述研究结果与Hu等[10]的研究结果相一致.

|
图 3 样品中细菌群落结构纲水平比例组成 Fig. 3 Proportion of bacterial community structure at class level in the sample |
病原菌的去除是保障污水回用卫生学安全的重要组成. 综合国内外研究病原菌分类[19, 20],本研究共发现11种病原菌(表 4),其中梭菌属(Clostridium)、 弓形杆菌属(Arcobacter)和分支杆菌(Mycobacterium)的含量较高,此外常见的病原菌埃希氏菌和志贺氏菌(Escherichia和Shigella)和军团菌(Legionella)也均检测到. 弓形杆菌属(Arcobacter)是革兰氏阴性螺旋形微生物,属于弯曲杆菌科[21],会引起腹泻、 腹痛等症状. 对一些消毒剂(如紫外消毒)的耐受性较强,在深度处理过程中较难去除[22]. 分枝杆菌属(Mycobacterium)大部分种类是机会致病菌,可能会引起系列人类感染疾病(如支原体肺炎)[23]. 分枝杆菌是革兰氏阳性菌,其特殊的生理性能(包括生长缓慢、 细胞膜较厚、 疏水性的细胞膜)使得其对各种消毒剂具有较好的抗性[24]. 此外,该细菌可以寄生于阿米巴虫身上,进一步增强了他们的耐消毒性[25]. 在污水回用中需采用别的处理方法(如膜处理等)进行去除. 梭菌属(Clostridium)是革兰氏阳性菌,在污水处理厂中普遍存在[19],会引起破伤风等各类疾病. 梭菌属为专性厌氧微生物,能形成孢子,使其对氧化性消毒剂具有极强的耐受性[26]. 但是,梭菌属对紫外消毒则较为敏感[27, 28],这主要由于紫外消毒并不是通过氧化作用破外细胞结构,而是直接导致核酸突变,阻碍其复制和转录,封锁及阻碍蛋白质的合成同时产生的自由基可引起光电离,最终导致微生物死亡[29, 30]. 不同病原菌的生理特性不同,在深度处理过程中应结合不同病原菌的生理特性设置处理来去除污水中的病原菌.
|
|
表 4 样品中各病原菌比例 Table 4 Proportion of pathogenic bacteria in the sample |
2.2 紫外消毒对指示菌与病源菌的杀灭特性细菌的影响
不同紫外剂量下,大肠杆菌的去除效果如图 4所示. 从中可知,当紫外剂量为60 mJ ·cm-2时,对大肠杆菌的去除率达到3.2个数量级,出水浓度为113 CFU ·L-1能够满足再生水用作景观用水时的卫生学要求. 因此,本研究考察紫外剂量为60 mJ ·cm-2对不同病原菌的去除效果.
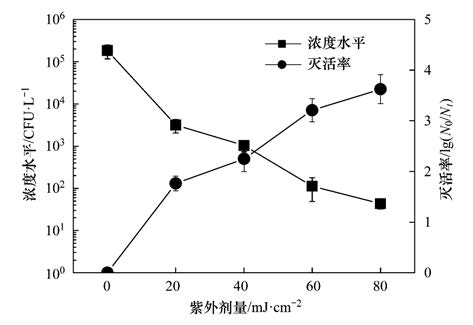
|
图 4 大肠杆菌的紫外消毒曲线 Fig. 4 UV inactivation curves of E.coli |
本研究中qPCR的线性检测区间如下:大肠杆菌为3×102~3×108 copies ·reaction-1; 沙门氏菌为6×102~6×108 copiese ·reaction-1; 分枝杆菌为8×101~8×108 copies ·reaction-1. 大肠杆菌、 沙门氏菌与分枝杆菌的线性相关系数均>0.99,PCR扩增效率在95%-110%之间. 大肠杆菌及分枝杆菌的熔解曲线呈现单一的熔点峰,峰值为87℃±0.5℃. 表明本研究所采用的qPCR标准曲线线性相关性较好,扩增效率较高,对细菌的特异性较强.
本研究中Q-RT-PCR的线性监测区间如下:大肠杆菌、 沙门氏菌和分枝杆菌均为1×102-1×109copies ·reaction-1. 大肠杆菌与分枝杆菌的线性相关系数均>0.99,PCR扩增效率在95%-115%之间.
采用培养法、 qPCR及Q-RT-PCR方法检测水源地水样消毒前后大肠杆菌、 沙门氏菌与分枝杆菌的浓度水平,检测结果如图 5所示. 从中可知,60 mJ ·cm-2剂量紫外消毒对可培养大肠杆菌及沙门氏菌的去除率约为99.9%. 靳慧霞等人在研究紫外消毒对大肠杆菌灭活效率的研究中发现,当消毒剂量为60 mJ ·cm-2时,大肠杆菌的去除率为99.99%[31]. 本研究相同剂量下,可培养大肠杆菌的去除率较低,这可能是由于紫外消毒会受到水体浊度、 有机物等的影响[32]. 但是,相同剂量的紫外消毒对可培养分枝杆菌的去除率不足90%,这与分枝杆菌特殊的细胞结构有关. 分枝杆菌的细胞膜富含脂类,细胞膜厚,具有疏水性,使得分枝杆菌对外界环境的抵抗性较强[33, 34].
qPCR检测消毒前后大肠杆菌、 沙门氏菌及分枝杆菌的浓度水平的变化并不显著. 这表明紫外消毒对细菌基因片段的破坏速率远小于其对细菌可培养性的破坏速率. qPCR检测的是目标菌的目的基因片段而不是整个基因组,由于检测的目的基因的片段较短,无法很好地指示整个基因组的受损情况. Q-RT-PCR检测结果显示,经60 mJ ·cm-2剂量紫外消毒后,活性大肠杆菌、 沙门氏菌及分枝杆菌的浓度水平下降不显著. 结合培养法的结果,细菌虽然无法在培养基上形成菌落,但是仍具有逆转录活性,这表明细菌仍具有一定活性. 这可能是由于经过紫外照射后细菌可能进入VBNC(viable but nonculturable)状态,无法在培养基上生长,但仍具有细胞活性,可在一定条件下恢复其可培养性[35, 34]. 值得注意的是,分枝杆菌qPCR及Q-RT-PCR的检测结果较培养法的检测结果高2个数量级. 有学者指出,由于分枝杆菌种类较多,生长条件不同,一种培养基可能无法检测到所有的分枝杆菌,采用qPCR的检测结果会比培养法高1个多数量级[36]. 此外,Q-RT-PCR的检测较qPCR略低,这是由于qPCR检测到的是所有具有目的基因的DNA片段(包括非活性及活性菌),而Q-RT-PCR检测到的仅是具有逆转录活性的RNA片段,因此Q-RT-PCR比qPCR较好地指示污水中的活性病原菌的浓度水平.
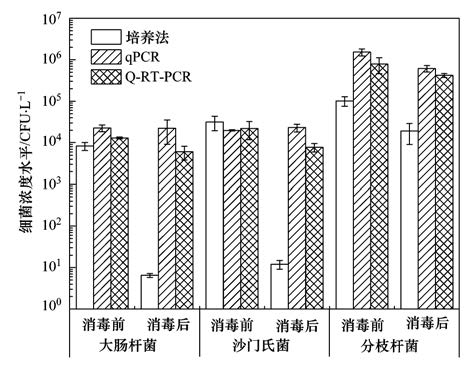
|
图 5 培养法、 qPCR及Q-RT-PCR对不同指示菌的检测结果 Fig. 5 Results of culture-based method,qPCR and Q-RT-PCR detection for different indicator bacterial strains |
上述研究结果表明,紫外消毒能够抑制大肠杆菌及沙门氏菌的可培养性,但这些细菌尚能保持逆转录活性. 与投加化学消毒剂的消毒工艺相比,紫外消毒工艺没有持久的消毒能力,一些仍具有活性的微生物在经受紫外照射灭活后能够再次复活,恢复其可培养性. 已被证实能够产生复活现象的微生物有:大肠埃希氏菌、 洋葱假单胞杆菌、 粪链球菌、 蓝藻等[37]. 此外,紫外消毒对分枝杆菌的影响较小,降低了紫外消毒的效果,给污水回用带来了安全隐患,需结合其他工艺(如投加余氯等)来保障污水回用的卫生学安全.
3 结论(1)454焦磷酸测序研究污水厂二级出水中微生物群落结构,共发现12个门的细菌,其中拟杆菌门(Bacteroidetes)和变形菌门(Proteobacteria)是主要的门类,且拟杆菌门(Bacteroidetes)的主要组成部位为Flavobacteria,变形菌门(Proteobacteria)的主要组成部位为α-、 β-和γ-Proteobacteria. 本研究共发现11种病原菌,其中梭菌属(Clostridium)和分支杆菌(Mycobacterium)是主要的属类,此外还有军团菌(Legionella)等常见的致病性较高的病原菌.
(2)研究比较60mJ ·cm-2剂量紫外消毒对大肠杆菌、 沙门氏菌及分枝杆菌的去除效果. 结果表明,该计量的紫外消毒能去除3个数量级可培养的大肠杆菌及沙门氏菌,对分枝杆菌的去除效果不显著. Q-RT-PCR结果表明,虽然经过紫外消毒后,大部分大肠杆菌及沙门氏菌失去了可培养性,但仍具有逆转录活性,需结合其他深度处理工艺进一步去除活性病原菌以保障污水回用的卫生学安全.
| [1] | Liu W, Andrews S A, Bolton J R, et al. Comparison of disinfection byproduct (DBP) formation from different UV technologies at bench scale[J]. Water Supply, 2002, 2 (5-6): 515-521. |
| [2] | Craik S A, Weldon D, Finch G R, et al. Inactivation of cryptosporidium parvum oocysts using medium-and low-pressure ultraviolet radiation[J]. Water Research, 2001, 35 (6): 1387-1398. |
| [3] | Meulemans C C E. The basic principles of UV-disinfection of water[J]. 1987, 9 (4): 299-313. |
| [4] | 王秀娟, 胡学香, 胡春. 水质对紫外消毒在两种典型再生水中应用的影响[J]. 环境工程学报, 2012, 6 (12): 4289-4293. |
| [5] | 张永吉, 刘文君. 紫外线对自来水中微生物的灭活作用[J]. 中国给水排水, 2005, 21 (9): 1-4. |
| [6] | 楼朝刚, 夏东升, 赵帆, 等. 微波无极紫外光对自来水中微生物的灭活作用[J]. 环境工程, 2008, 26 (3): 10-12. |
| [7] | 林怡雯. 再生水系统中VBNC病原菌复活特性与风险的研究[D]. 北京: 清华大学, 2013. |
| [8] | Nocker A, Camper A K. Novel approaches toward preferential detection of viable cells using nucleic acid amplification techniques[J]. FEMS Microbiology Letters, 2009, 291 (2): 137-142. |
| [9] | Fey A, Eichler S, Flavier S, et al. Establishment of a real-time PCR-based approach for accurate quantification of bacterial RNA targets in water, using Salmonella as a model organism[J]. Applied and Environmental Microbiology, 2004, 70 (6): 3618-3623. |
| [10] | Hu M, Wang X H, Wen X H, et al. Microbial community structures in different wastewater treatment plants as revealed by 454-pyrosequencing analysis[J]. Bioresource Technology, 2012, 117 : 72-79. |
| [11] | 仝铁铮. PMA-qPCR选择性检测水中活性菌方法的研究与应用[D]. 北京: 清华大学, 2010. |
| [12] | Heijnen L, Medema G. Quantitative detection of E. coli, E. coli O157 and other shiga toxin producing E. coli in water samples using a culture method combined with real-time PCR[J]. Journal of Water and Health, 2006, 4 (4) : 487-498. |
| [13] | Jjemba P K, Weinrich L A, Cheng W, et al. Regrowth of potential opportunistic pathogens and algae in reclaimed-water distribution systems[J]. Applied and Environmental Microbiology, 2010, 76 (13): 4169-4178. |
| [14] | Pusterla N, Byrne B A, Hodzic E, et al. Use of quantitative real-time PCR for the detection of Salmonella spp. in fecal samples from horses at a veterinary teaching hospital[J]. The Veterinary Journal, 2010, 186 (2): 252-255. |
| [15] | 胡秀华, 何苗, 刘丽, 等. 水中轮状病毒实时定量 PCR 外标准品的构建[J]. 环境科学, 2008, 29 (2): 380-385. |
| [16] | Ye L, Zhang T. Bacterial communities in different sections of a municipal wastewater treatment plant revealed by 16S rDNA 454 pyrosequencing[J]. Applied Microbiology and Biotechnology, 2013, 97 (6): 2681-2690. |
| [17] | Lee S H, Kang H J. Park H D. Influence of influent wastewater communities on temporal variation of activated sludge communities[J]. Water Research, 2015, 73 : 132-144. |
| [18] | Shu D T, He Y L, Yue H, et al. Microbial structures and community functions of anaerobic sludge in six full-scale wastewater treatment plants as revealed by 454 high-throughput pyrosequencing[J]. Bioresource Technology, 2015, 186 : 163-172. |
| [19] | Ye L, Zhang T. Pathogenic bacteria in sewage treatment plants as revealed by 454 pyrosequencing[J]. Environmental Science & Technology, 2011, 45 (17): 7173-7179. |
| [20] | Bibby K, Viau E, Peccia J. Pyrosequencing of the 16S rRNA gene to reveal bacterial pathogen diversity in biosolids[J]. Water Research, 2010, 44 (14): 4252-4260. |
| [21] | Lehner A, Tasara T, Stephan R. Relevant aspects of Arcobacter spp. as potential foodborne pathogen[J]. International Journal of Food Microbiology, 2005, 102 (2): 127-135. |
| [22] | 许小红. 城市水体中新型病原细菌生态学特征及其溯源研究[D]. 镇江: 江苏大学, 2008. |
| [23] | Tortoli E. Impact of genotypic studies on mycobacterial taxonomy: the new mycobacteria of the 1990s[J]. Clinical Microbiology Reviews, 2003, 16 (2): 319-354. |
| [24] | Kim C J, Kim N H, Song K H, et al. Differentiating rapid- and slow-growing mycobacteria by difference in time to growth detection in liquid media[J]. Diagnostic Microbiology and Infectious Disease, 2013, 75 (1): 73-76. |
| [25] | Thomas V, Loret J F, Jousset M, et al. Biodiversity of amoebae and amoebae-resisting bacteria in a drinking water treatment plant[J]. Environmental Microbiology, 2008, 10 (10): 2728-2745. |
| [26] | Tyrrell S A, Rippey S R, Watkins W D. Inactivation of bacterial and viral indicators in secondary sewage effluents, using chlorine and ozone[J]. Water Research, 1995, 29 (11): 2483-2490. |
| [27] | Gilboa Y, Friedler E. UV disinfection of RBC-treated light greywater effluent: Kinetics, survival and regrowth of selected microorganisms[J]. Water Research, 2008, 42 (4-5): 1043-1050. |
| [28] | Doan L, Forrest H, Fakis A, et al. Clinical and cost effectiveness of eight disinfection methods for terminal disinfection of hospital isolation rooms contaminated with Clostridium difficile 027[J]. Journal of Hospital Infection, 2012, 82 (2): 114-121. |
| [29] | Rajala-Mustonen R L, Toivola P S, Heinonen-Tanski H. Effects of peracetic acid and UV irradiation on the inactivation of coliphages in wastewater[J]. Water Science and Technology, 1997, 35 (11-12): 237-241. |
| [30] | Sanz E N, Dávila I S, Balao J A A, et al. Modelling of reactivation after UV disinfection: Effect of UV-C dose on subsequent photoreactivation and dark repair[J]. Water Research, 2007, 41 (14): 3141-3151. |
| [31] | 靳慧霞, 董滨, 孙颖, 等. 超声协同紫外灭活大肠杆菌实验研究[J]. 环境科学与技术, 2010, 33 (11): 22-27, 42. |
| [32] | 赵琳, 王晓昌. 紫外与次氯酸钠消毒效果及影响因素研究[J]. 环境污染与防治, 2014, 36 (7): 41-45. |
| [33] | Brennan P J, Nikaido H. The envelope of mycobacteria[J]. Annual Review of Biochemistry, 1995, 64 (1): 29-63. |
| [34] | Daffé M, Draper P. The envelope layers of mycobacteria with reference to their pathogenicity[J]. Advances in Microbial Physiology, 1997, 39 : 131-203. |
| [35] | Gin K Y H, Goh S G. Modeling the effect of light and salinity on viable but non-culturable (VBNC) Enterococcus[J]. Water Research, 2013, 47 (10): 3315-3328. |
| [36] | Zuccheri G, Asproulis N. Detection of pathogens in water using micro and nano-technology[M]. London: IWA publishing, 2012. |
| [37] | Liltved H, Landfald B. Influence of liquid holding recovery and photoreactivation on survival of ultraviolet-irradiated fish pathogenic bacteria[J]. Water Research, 1996, 30 (5): 1109-1114. |
 2016, Vol. 37
2016, Vol. 37


