三氯乙烯(trichloroethylene,TCE)作为一种典型的有机氯代溶剂,已被广泛应用于五金、 电子、 化工、 制革、 干洗、 医药等多个行业[1]. 然而,随着三氯乙烯制品的广泛使用、 相关废弃物的随意处置及工厂三氯乙烯泄漏事故的发生,氯乙烯污染地下水的事件时有出现,TCE目前已成为我国和很多其它国家(如美国、 加拿大、 德国、 日本和泰国等)土壤和地下水中最为常见的污染物之一[2, 3, 4, 5, 6]. 由于TCE具有较高的生物毒性和较强的“致癌、 致畸、 致突变”效应,容易通过食物链在生物体内富集,对人体和动物的肝、 肾、 心血管和胃肠等器官都有极强的毒害作用[7, 8, 9],如果TCE污染的土壤和地下水没有得到及时的治理,势必会严重威胁到人类及其它生物的生命安全.
近年来,由于越来越多的有机和无机污染物进入水体以及这些污染物在水环境中的长期积累和暴露,水体污染的复合性特征也表现得越来越突出[10]. 高存荣等对全国31个省的69个城市地下水有机污染检测结果表明,除了TCE等卤代烃有较高的检出率外,还同时检测到大量的氯代苯类、 单环芳烃及多环芳烃等有毒污染物[3]. 在地下水中,TCE很少以单独污染的形式出现,它经常与其它卤代烃及芳香族化合物在一起形成一个复合的污染羽,这在一定程度上加大了TCE污染的治理难度. 目前,纳米铁还原[11, 12]及氧化剂氧化TCE[13, 14]是研究和应用较多的原位修复技术,然而这些技术都或多或少地存在作用半径小、 会带来二次污染等问题,再加上地下水和土壤多为密闭的厌氧环境,这些因素使得越来越多的科学家们将注意力转向了更加经济、 环保和有效的厌氧生物修复技术.
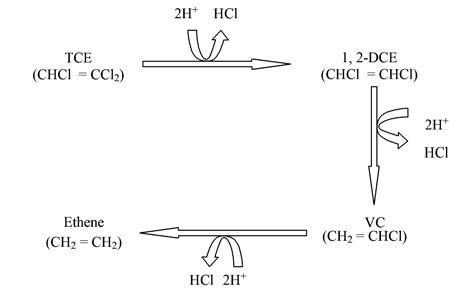
在厌氧条件下(如图 1),TCE作为电子受体,其氯原子逐步被氢原子(电子供体)取代生成1,2-二氯乙烯(1,2-DCE)、 VC和乙烯(ETH). 目前已报道的具有这种完全脱氯能力的菌株只有DHC[15],而DHC只能在特定的碳源和电子供体的情况下才能正常的生长并发挥脱氯功能[16, 17, 18]. 为了能更好地提高TCE污染地下水的自我修复能力,人们已经尝试选择一些释氢基质(如甲醇、 乙醇、 乙酸、 丙酸、 乳酸、 丁酸及苯甲酸等)来代替H2作为DHC的电子供体[18, 19]. 而在这些释氢基质中,苯甲酸又恰好是苯、 扁桃酸、 甲苯等多种芳香族化合物厌氧降解过程的中间体[20, 21],如果能驯化出一批以苯甲酸为碳源的厌氧还原脱氯体系,则很可能在一定程度上可以解决TCE等卤代烃与苯系化合物的复合污染问题. 围绕这个目的,本文结合气相色谱与高通量测序及实时荧光定量PCR技术对苯甲酸盐驯化的三氯乙烯降解体系展开研究,希望从生态学的角度探讨厌氧还原脱氯过程中的协同修复特性,以期为有机复合污染环境的生物修复提供理论依据.
|
图 1 TCE的厌氧还原脱氯原理[20] Fig. 1 Reductive dechlorination principle of TCE in the anaerobic condition |
从厦门一污水处理厂现场用一次性无菌注射器抽取活性污泥并转移至预先灭过菌的血清瓶中,用冰盒保存并带回实验室.
1.2 实验方法 1.2.1 TCE降解菌的富集培养BAV1培养基(培养基配方参照文献[22]的方法)如下.
①矿物盐: (g ·L-1)50×储液(1 L培养基中加入20 mL); ②微量元素溶液(g ·L-1)1 000×储液 (1 L培养基中加入1 mL); ③Se/Wo solution (g ·L-1) 1 000×储液 (1 L培养基中加入1 mL); ④维生素混合溶液(g ·L-1) 1 000×储液 (1 L培养基中加入1 mL); ⑤还原缓冲液(g ·L-1)(1 L培养基中加入8 mL).
将上述①、 ②、 ③、 ⑤ 这4种溶液按所表示比例加入1L培养液中,培养液中事先加入苯甲酸钠并加热至煮沸约15 min后,通入99.99%氮气进行冷却,调节pH值至7.2-7.3,将配制好的完全培养基分装至血清瓶中,盖好铝盖,经高压灭菌后,向瓶中加入④溶液及10 μmol TCE(分析纯)作为电子受体,摇匀,按10%的比例接种活性污泥,于恒温培养箱中倒置培养,定期用气相色谱仪(Agilent 7890)的FID检测器检测培养过程中TCE浓度的变化,选择降解效果最好的驯化体系进行传代培养,直至获得具有稳定降解效果的培养体系.
1.2.2 TCE降解过程的测定(1)气相色谱仪器配置
仪器: 气相色谱仪; 进样方式: 顶空手动进样; 色谱柱: DB-624 (60 m×0.32 mm×1.8 μm); 检测器: FID检测器; 载气: 高纯氮(99.999%).
(2)仪器分析条件
柱温: 50℃,进样口温度200℃; 检测器(FID)温度: 220℃; 载气为高纯氮(99.999%); 柱流速: 6 mL ·min-1; 分流比: 2:1; 尾气吹扫流量: 50 mL ·min-1; 空气流量: 500 mL ·min-1; 氢气流量: 50 mL ·min-1; 压力: 145.13 kPa; 进样体积: 100 μL.
(3)驯化体系对TCE降解特性的测定
取上述已经驯化好的具有稳定降解效果的培养体系,按10%的比例平行转接至新鲜培养基中,根据先前驯化体系的降解特性定期检测体系中氯乙烯类物质的浓度,并同时采集该时刻的培养液,冻存于-20℃冰箱中以备下一步实验使用(所有培养体系均设置重复和对照).
1.2.3 实时荧光定量PCR(1)引物和探针的设计
引物和探针的设计参照文献[23]. 本实验中主要考察了3种与TCE降解相关的还原脱氯酶编码基因tceA、 vcrA及bvcA的数量变化情况. 其中,tceA基因所编码的还原脱氯酶主要催化TCE到DCE和VC的转化; vcrA基因所编码的还原脱氯酶主要催化DCE到VC的转化; 而bvcA基因所编码的还原脱氯酶主要催化VC到ETH的转化[23].
(2)DNA的提取
由于每个驯化体系的体积(约40 mL)比较小,且TCE的主要降解菌Dehalococcoides spp. 生长非常缓慢,如果取的菌液过多会影响体系的稳定性及降解效率,加之Dehalococcoides spp. 对O2非常敏感,如果取样过于频繁,很容易给体系造成不可逆的冲击,所以在做荧光定量的时候,只取初始与终了两个时间点的样品来分析. 取样时,用5 mL的一次性针管从充分混匀的驯化污泥体系中抽取4 mL菌液,用0.22 μm的混合纤维素酯微孔滤膜过滤,将膜剪碎后存于1.5 mL的无菌EP管中,DNA提取步骤按照EZNA® Soil DNA Kit(Omega Bio-Tek,USA)提示的方法进行操作.
(3)标准质粒的提取
选用带功能基因的环境样品,通过PCR获取目的基因片段,再连接到已知载体质粒PMD18-T(Takara公司)上并转化到感受态细胞DH5α(Takara公司)中,将携带有功能基因片段的克隆子进行富集培养,按照EZNA® Plasmid Mini Kit Ⅱ(Omega Bio-Tek,USA)的操作方法提取质粒,并用Nanondrop分光光度计(Thermo Fisher公司)对质粒进行定量并计算其拷贝数,具体步骤参照文献[23].
(4)PCR条件及程序
通过预实验对实时荧光定量PCR反应体系和扩增条件进行优化,确定最佳体系及条件.
总反应体系为20 μL: 正、 反向引物(10 μmol ·L-1)0.4 μL和探针0.8 μL、 TaqMan Mix 10 μL、 模板DNA 2 μL、 ROX Reference Dye 0.4 μL、 Nuclease-Free Water 6 μL.
荧光定量PCR扩增条件(采用两步法):
预变性95℃ 30 s,1个循环; 95℃ 5 s,60℃ 34 s,40个循环.
同时以Nuclease-Free Water为模板作为阴性对照.
在荧光定量PCR(ABI 7500,美国)运行过程中,借助ABI公司的SDS软件实时观察PCR荧光扩增曲线、 采集分析数据.
1.2.4 基于16S rRNA基因的高通量测序(1)测序样品的预处理
提取TCE完全降解时培养菌液的DNA样品,用细菌16S rRNA基因V1-V3高可变区27F/534R[24]引物对采集样品的DNA进行扩增,F引物5′端带有10 bp序列标签用于样品区分. PCR反应程序如下: 94℃预变性5 min; 94℃变性30 s,55℃退火30 s,72℃延伸90 s,共25个循环; 最终72℃延伸7 min. PCR产物用胶纯化试剂盒(Qiagen公司)纯化,回收产物浓度用Qubit dsDNA BR Assay Kit(Life Technologies公司)进行精确定量. 回收产物等量混合后送至深圳华大基因科技公司完成454(GS-FLX Titanium System)焦磷酸测序,序列数据递交至NCBI SRA数据库(序列号: SRX193676).
(2)生物信息学分析
454测序数据用QIIME软件进行分析. 为保证生物信息学分析质量,本研究对原始数据进行多步质控,包括: ①去除序列长度小于150 bp的序列; ②去除含有模糊碱基N的序列; ③去除平均测序精确度低于99.7%的序列; ④去除PCR引物和识别标签中含有模糊碱基的序列; ⑤去除含有超过6个连续同聚体(相同碱基)的序列. 以97%序列相似度划分操作分类单元(operational taxonomic unit,OTU). 序列用RDP Classifier完成种属分类,并应用CREST软件完成细菌类群的分类.
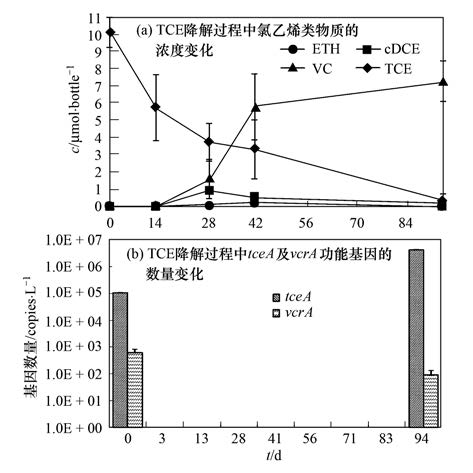
2 结果与讨论 2.1 驯化体系对TCE的降解在厌氧条件下,三氯乙烯作为电子受体,其氯原子被氢原子(电子供体)取代,并逐步脱氯生成1,2-DCE、 VC和ETH. 如图 2所示,实验组体系中的TCE(初始量为10 μmol)在94 d内被完全去除; 随着TCE的降解,VC逐渐累积,在TCE完全降解时达到峰值; 1,2-DCE有两种同分异构体: 顺式1,2-DCE(cis-DCE)和反式1,2-DCE(trans-DCE),检测结果显示,在体系培养两周后出现cis-DCE,在第四周时达到峰值(≤1 μmol),随着反应的进行cis-DCE逐渐消失,反应过程中未发现trans-DCE,这个现象与Futagami等[25]的研究结果较一致; 在TCE降解过程中有非常少量的ETH产生,但在反应终体系中却未检测到ETH,取而代之的是大量的甲烷(图中未给出). 这个现象说明体系中至少存在两类菌,一类属于DHC,这类菌在还原脱氯反应中发挥了主要作用; 另一类属于产甲烷菌,它们在体系中会与DHC争夺电子供体[19],从而抑制DHC的生长并导致TCE脱氯不完全. 但也有研究报道,在厌氧环境中,产甲烷菌和其它微生物常携带有大量的还原性过渡金属辅助因子(reduced transition-metal cofactors),能通过共代谢作用将TCE还原脱氯,但这种共代谢作用会随着逐步脱氯而逐级递减,导致最终的产物停留在cis-DCE和VC[26]. 体系中的还原脱氯过程究竟是在哪一类菌的作用下完成的呢?可能还需要通过进一步的实验来证实.
|
图 2 体系中氯乙烯类物质浓度与功能基因(tceA及vcrA)数量的关系 Fig. 2 Relation between the concentration of VOCs and copies of functional gene (tceA and vcrA) in enrichment cultures |
DHC对TCE的还原脱氯过程是在位于该类菌细胞膜上的还原脱氯酶的催化作用下完成的[16],不同种类的还原脱氯酶所执行的脱氯功能也是不同的,如tceA基因所编码的还原脱氯酶(存在于Dehalococcoides 195和FL2菌株中)主要催化TCE转化为cis-DCE和VC; vcrA基因所编码的还原脱氯酶(存在于Dehalococcoides VS和GT菌株中)主要催化cis-DCE到VC的转化; 而bvcA基因所编码的还原脱氯酶(存在于Dehalococcoides VS和GT菌株中)主要催化VC到ETH的转化[27]. 前期的预实验发现体系中只检测到tceA和vcrA两种功能基因,没有检测到bvcA基因,所以后期的实验没有对bvcA基因进行定量. 从图 2(b)中可以看出,随着TCE的完全降解,携带有还原脱氯基因tceA的功能菌的数目显著增加(从1×105 copies ·L-1上升到4×106 copies ·L-1),而携带有还原脱氯基因vcrA的功能菌的数目不但没有增加,反而减少(从6×102 copies ·L-1下降到9×101 copies ·L-1),从而可以推测出该反应体系中发挥还原脱氯作用的主要功能菌为携带tecA功能基因的DHC.
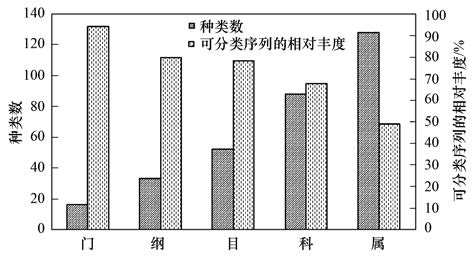
2.2 驯化体系的微生物群落结构分析454焦磷酸测序共获得4 195条原始序列,经多步质控后获得2 133条高质量序列信息,经RDP分类得到594个OTU. 从图 3的RDP分析结果来看,这些序列可归入16个门,33个纲,52个目,88个科和129个属,随着分类等级的降低,可归类到确切的纲、 目、 科、 属的序列数比例也在减少,分别为94.1%、 79.8%、 78.3%、 67.6%和48.8%,这说明该体系中还有很大一部分微生物的分类地位还不明确,很可能存在一些未知功能菌.
|
图 3 驯化体系中微生物群落的RDP分类结果 Fig. 3 RDP classification results of microbial community |
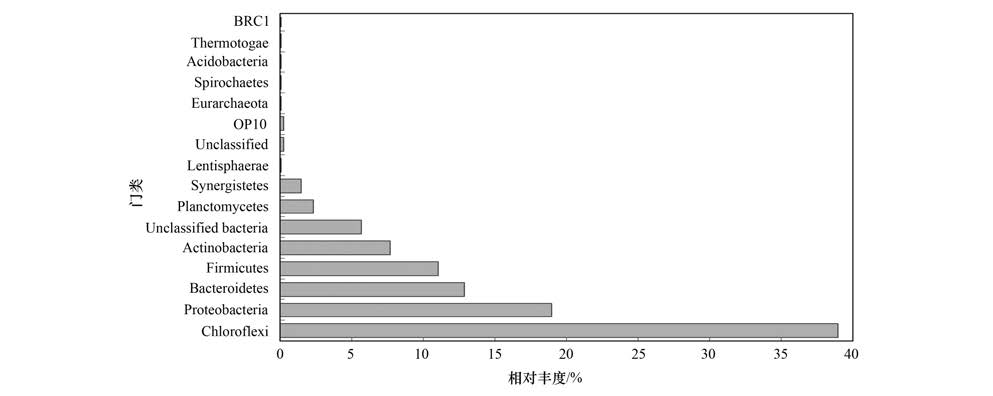
在门类划分水平上(图 4),体系中占主导地位的门类为Chloroflexi、 Proteobacteria、 Bacteroidetes、 Firmicutes、 Actinobacteria和细菌域内的未知门,所占比例分别为39.0%、 19.0%、 12.9%、 11.0%、 7.7%和5.7%; 在纲划分水平上,体系中占主导地位的纲类为Anaerolineae、 Bacteroidetes门类的未知纲、 Clostridia、 Actinobacteria、 Deltaproteobacteria、 Gammaproteobacteria、 细菌域内的未知纲、 Dehalococcoidetes、 Caldilineae及Betaproteobacteria,所占比例分别为29.1%、 11.1%、 10.2%、 7.7%、 7.6%、 6.2%、 5.7%、 3.9%、 3.5%和3.1%; 在目划分水平上,体系中占主导地位的目类为Anaerolineales、 Bacteroidetes门类的未知目及Clostridiales,所占比例分别为29.1%、 11.1%和9.5%; 在科划分水平上,体系中占主导地位的科类为Anaerolineaceae、 Bacteroidetes门类的未知科、 细菌域内的未知科、 Desulfobulbaceae、 Dehalococcoides目类的未知科、 Caldilineaceae及Moraxellaceae,所占比例分别为29.1%、 11.1%、 5.7%、 4.2%、 3.9%、 3.5%和3.5%; 而在属划分水平上,体系中优势属为Anaerolineaceae科内的未知属、 Bacteroidetes门内的未知属、 Leptolinea、细菌域内的未知属、 Desulfobulbus、 Dehalococcoides目内的未知属、 Longilinea、 Caldilinea、 Acinetobacter及Bellilinea,它们在体系中所占的比例分别为13.9%、 11.1%、 8.0%、 5.7%、 4.2%、 3.9%、 3.6%、 3.5%、 3.5%和3.0%.
|
图 4 驯化体系中微生物群落的门类结构 Fig. 4 Phylum distribution of microbial community |
为了更好地理解优势菌的系统进化地位,将这些优势菌(百分比≥1%)的代表OTU与已报道可培养菌的最相似序列(摘自http://www. ezbiocloud.net/eztaxon)进行比对,并构建系统发育树. 从亲缘关系上看(图 5),除了部分OTU (unclassified group 1、 group 2和group 3)不能比对到相应的种属外,其余大部分OTU均能比对到相近的种属; 部分OTU (6145、 12124、 959、 12244和1997)与已报道过的还原脱氯功能菌DHC的进化地位十分接近,荧光定量PCR的结果[图 2(b)]也证实了这一类菌的存在,可以判定DHC类群菌在体系的还原脱氯作用中发挥了主要的作用; 体系中除了DHC外,还有一些OTU(6150、 8477和2892)与Geobacter 的亲缘关系很近,而这类菌也具有一定的还原脱氯功能[17, 28],但这类菌是否参与了TCE的还原脱氯过程,还有待进一步验证; 在厌氧降解体系中,属于δ-Proteobacteria纲的Syntrophus和Desulfovibrio及Clostridia纲的Syntrophomonas和 Desulfotomaculum经常作为初级代谢者为体系中的降解菌提供有效的碳源及电子供体[29, 30],体系中系统进化关系与Desulfovibrio相近的OTU(4032、 11550及8480)以及与Syntrophomonas相近的OTU(13630、 5371及11484)很可能作为DHC的共生菌辅助DHC完成还原脱氯作用; 已有研究表明,维生素B12是还原脱氯酶(RDases)的必需辅助因子,然而,由于DHC本身缺乏咕啉环(合成维生素B12的重要前体物)合成途径,只能通过它的共生菌(如Pelosinus、 Dendrosporobacter、 Sporotalea和Clostridium)来提供[31, 32],体系中与Clostridium相近的OTU(13800、 450、 10836、 9319及11798)很可能在辅助DHC进行还原脱氯作用方面起到了一定的作用; 另外,体系中还有一部分OTU(约占2.3%)的亲缘关系与Thauera相接近,据报道,此类菌多参与苯环类化合物的降解过程[29, 30],很可能这类菌与体系中的苯甲酸代谢相关,通过代谢苯甲酸间接为DHC提供碳源或电子供体; 在比对结果中还发现,有少部分OTU(约占1%)与产甲烷古菌Methanosaeta和Methanobacterium相似度很高,虽然含量不高,但很可能这类菌在后期的碳源及电子供体竞争中取得优势,从而抑制了DHC的完全脱氯作用,导致体系中VC和甲烷的大量累积.
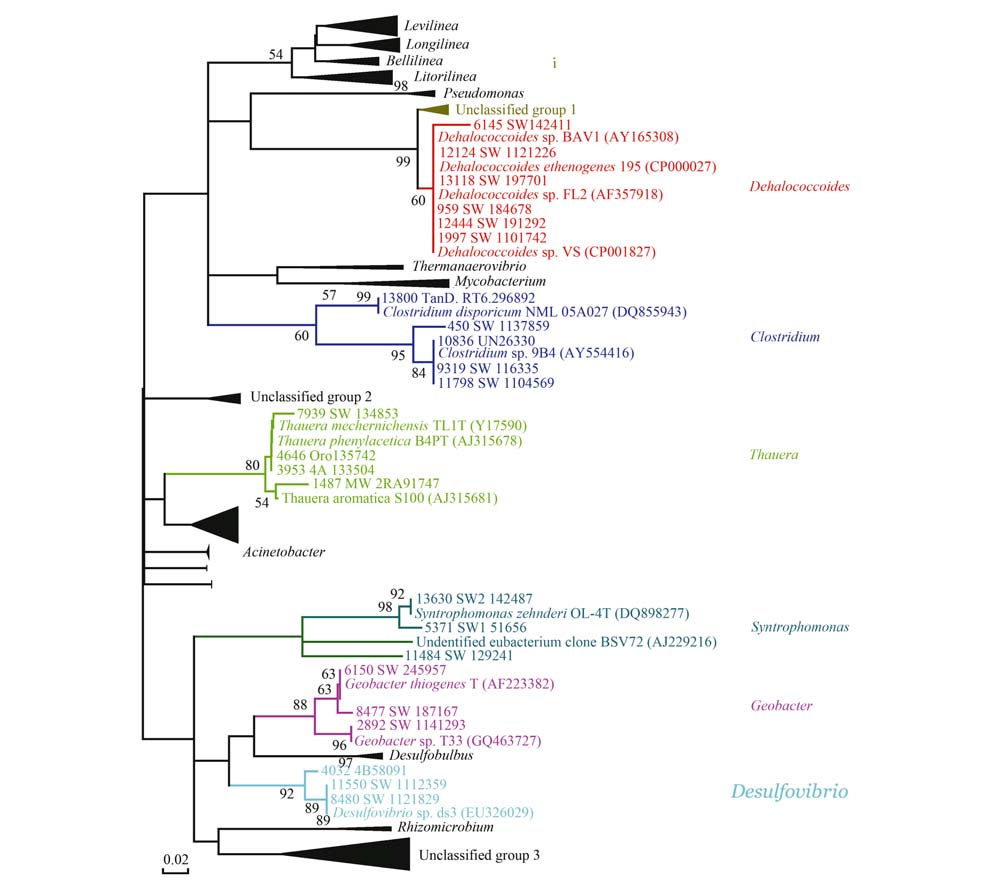
|
图 5 驯化体系中优势菌属的系统发育树 Fig. 5 Phylogenetic tree of dominant genera in the enrichment cultures |
TCE厌氧降解体系中的微生物群落结构是十分复杂的,从体系的代谢及微生物群落结构特征,可以推断出体系中各类菌群之间的大致代谢关系(图 6),体系中一些发酵型细菌(如Thauera)能直接利用苯甲酸生成次级代谢产物(如乙酸和H2),而这些产物为DHC提供了直接碳源或电子供体,辅助它们进行生长代谢; 另一类菌(如Desulfovibrio、 Syntrophomonas及 Desulfotomaculum等)与DHC构成共生关系,它们在进行生长代谢时,能合成还原脱氯作用所需的V12或电子供体,辅助DHC进行脱氯; 还有一类菌(如产甲烷菌)与DHC构成竞争关系,它们会与DHC竞争碳源和电子供体,从而抑制DHC的脱氯功能. TCE的还原脱氯过程其实是还原脱氯菌与其它功能菌共同作用的结果,至于它们之间的共生或竞争机制如何演绎,还需要通过进一步的实验来佐证.
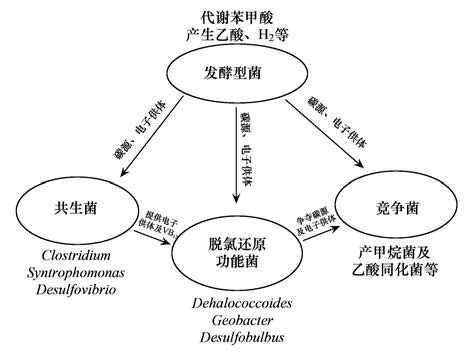
|
图 6 TCE降解过程中各菌群的代谢关系 Fig. 6 Metabolic interactions in the process of TCE-degradation |
(1)以苯甲酸盐为唯一碳源和电子供体的活性污泥驯化体系能够将TCE逐步脱氯还原至VC,同时伴有大量的甲烷产生.
(2)体系中起主要还原脱氯作用的是携带tceA功能基因的DHC,其数量随着TCE的降解而不断增加.
(3)体系中的微生物多样性很高,涵盖了16个门、 33个纲、 52个目、 88个科和129个属,而且随着分类等级的降低,可归类到确切的纲、 目、 科、 属的序列数比例也在减少,约有51.2%的序列的分类地位尚未明确,说明体系中存在着很多未知的功能菌.
(4)体系对TCE的降解过程是以DHC为主导的,在Desulfovibrio、 Syntrophomonas及 Desulfotomaculum等共生菌的辅助下完成的还原脱氯过程.
| [1] | 钱翌, 岳飞飞, 褚衍洋.三氯乙烯环境污染修复技术研究进展[J].环境化学, 2012, 31 (9): 1335-1343. |
| [2] | Suttinun O, Luepromchai E, Müller R.Cometabolism of trichloroethylene: concepts, limitations and available strategies for sustained biodegradation[J].Reviews in Environmental Science and Bio/Technology, 2013, 12 (1): 99-114. |
| [3] | 高存荣, 王俊桃.我国69个城市地下水有机污染特征研究 [J].地球学报, 2011, 32 (5): 581-591. |
| [4] | 孟凡生, 王业耀.三氯乙烯污染地下水和土壤的修复[J].中国给水排水, 2005, 21 (4): 34-36. |
| [5] | 俞光明, 刘红樱, 张泰丽, 等.杭州市浅层地下水有机污染及其风险初步评价[J].资源调查与环境, 2007, 28 (3): 198-204. |
| [6] | 张凤君, 王斯佳, 马慧, 等.三氯乙烯和四氯乙烯在土壤和地下水中的污染及修复技术[J].科技导报, 2012, 30 (18): 65-72. |
| [7] | Bácsi I, Török T, B-Béres V, et al.Laboratory and microcosm experiments testing the toxicity of chlorinated hydrocarbons on a cyanobacterium strain (Synechococcus PCC 6301) and on natural phytoplankton assemblages[J].Hydrobiologia, 2013, 7 10 (1): 189-203. |
| [8] | Fang Z Z, Krausz K W, Tanaka N, et al.Metabolomics reveals trichloroacetate as a major contributor to trichloroethylene-induced metabolic alterations in mouse urine and serum[J].Archives of Toxicology, 2013, 87 (11): 1975-1987. |
| [9] | Rusyn I, Chiu W A, Lash L H, et al.Trichloroethylene: Mechanistic, epidemiologic and other supporting evidence of carcinogenic hazard[J].Pharmacology & Therapeutics, 2014, 141 (1): 55-68. |
| [10] | 曲久辉.我国水体复合污染与控制[J].科学对社会的影响, 2000, (1): 35-39. |
| [11] | Elliott D W, Zhang W X.Field assessment of nanoscale bimetallic particles for groundwater treatment[J].Environmental Science and Technology, 2001, 35 (24): 4922-4926. |
| [12] | 王薇, 金朝晖, 李铁龙.包覆型纳米铁的制备及其去除三氯乙烯的研究[J].中国环境科学, 2009, 29 (8): 811-815. |
| [13] | 刘晓文, 李荣飞, 李霞, 等.过硫酸钠原位修复三氯乙烯污染土壤的模拟研究[J].环境科学学报, 2013, 33 (11): 2935-2940. |
| [14] | 吴嘉怡, 蔡信德, 靖元孝, 等.高锰酸钾氧化去除砂壤土中三氯乙烯的试验研究[J].中国环境科学, 2011, 31 (5): 810-814. |
| [15] | Matturro B, Rossetti S.GeneCARD-FISH: detection of tceA andvcrA reductive dehalogenase genes in Dehalococcoides mccartyi by fluorescence in situ hybridization[J].Journal of Microbiological Methods, 2015, 110: 27-32. |
| [16] | Brisson V L, West K A, Lee P K, et al.Metagenomic analysis of a stable trichloroethene-degrading microbial community[J].The ISME Journal, 2012, 6 (9): 1702-1714. |
| [17] | Pant P, Pant S.A review: Advances in microbial remediation of trichloroethylene (TCE)[J].Journal of Environmental Sciences, 2010, 22 (1): 116-126. |
| [18] | Schneidewind U, Haest P J, Atashgahi S, et al.Kinetics of dechlorination by Dehalococcoides mccartyi using different carbon sources[J].Journal of Contaminant Hydrology, 2014, 157: 25-36. |
| [19] | Löffler F E, Ritalahti K M, Zinder S H.Dehalococcoides and reductive dechlorination of chlorinated solvents[A].In: Stroo H F, Leeson A, Ward C H, (eds.).Bioaugmentation for Groundwater Remediation[M].New York: Springer, 2013.39-88. |
| [20] | Bunge M, Kleikemper J, Miniaci C, et al.Benzoate-driven dehalogenation of chlorinated ethenes in microbial cultures from a contaminated aquifer[J].Applied Microbiology and Biotechnology, 2007, 76 (6): 1447-1456. |
| [21] | 张晓云, 盖忠辉, 台萃, 等.微生物降解苯甲酸的研究进展[J].微生物学通报, 2012, 39 (12): 1808-1816. |
| [22] | He J Z, Ritalahti K M, Yang K L, et al.Detoxification of vinyl chloride to ethene coupled to growth of an anaerobic bacterium[J].Nature, 2003, 424 (6944): 62-65. |
| [23] | Holmes V F, He J Z, Lee P K H, et al.Discrimination of multiple Dehalococcoides strains in a trichloroethene enrichment by quantification of their reductive dehalogenase genes[J].Applied and Environmental Microbiology, 2006, 72 (9): 5877-5883. |
| [24] | Hu A Y, Yang X Y, Chen N W, et al.Response of bacterial communities to environmental changes in a mesoscale subtropical watershed, Southeast China[J].Science of the Total Environment, 2014, 472: 746-756. |
| [25] | Futagami T, Morono Y, Terada T, et al.Dehalogenation activities and distribution of reductive dehalogenase homologous genes in marine subsurface sediments[J].Applied and Environmental Microbiology, 2009, 75 (21): 6905-6909. |
| [26] | Gantzer C J, Wackett L P.Reductive dechlorination catalyzed by bacterial transition-metal coenzymes[J].Environmental Science & Technology, 1991, 25 (4): 715-722. |
| [27] | Do Dgˇ an-Subal E, Bastiaens L, Leys N, et al.Quantitative and functional dynamics of Dehalococcoides spp.and its tceA and vcrA genes under TCE exposure[J].Biodegradation, 2014, 25 (4): 493-504. |
| [28] | Maillard J, Charnay M P, Regeard C, et al.Reductive dechlorination of tetrachloroethene by a stepwise catalysis of different organohalide respiring bacteria and reductive dehalogenases[J].Biodegradation, 2011, 22 (5): 949-960. |
| [29] | Leuthner B, Leutwein C, Schulz H, et al.Biochemical and genetic characterization of benzylsuccinate synthase from Thauera aromatica: a new glycyl radical enzyme catalysing the first step in anaerobic toluene metabolism[J].Molecular Microbiology, 1998, 28 (3): 615-628. |
| [30] | Mechichi T, Stackebrandt E, Gad'on N, et al.Phylogenetic and metabolic diversity of bacteria degrading aromatic compounds under denitrifying conditions, and description of Thauera phenylacetica sp.nov., Thauera aminoaromatica sp.nov., and Azoarcus buckelii sp.nov[J].Archives of Microbiology, 2002, 178 (1): 26-35. |
| [31] | He J Z, Holmes V F, Lee P K H, et al.Influence of vitamin B12 and cocultures on the growth of Dehalococcoides isolates in defined medium[J].Applied and Environmental Microbiology, 2007, 73 (9): 2847-2853. |
| [32] | Men Y J, Lee P K H, Harding K C, et al.Characterization of four TCE-dechlorinating microbial enrichments grown with different cobalamin stress and methanogenic conditions[J].Applied Microbiology and Biotechnology, 2013, 97 (14): 6439-6450. |
| [33] | Morris B E L, Henneberger R, Huber H, et al.Microbial syntrophy: interaction for the common good[J].FEMS Microbiology Reviews, 2013, 37 (3): 384-406. |
| [34] | Shukla A K, Upadhyay S N, Dubey S K.Current trends in trichloroethylene biodegradation: a review[J].Critical Reviews in Biotechnology, 2014, 34 (2): 101-114. |
| [35] | Watanabe K, Kouzuma A.Syntrophic interactions in biodegradative consortia[A].In: Nojiri H M, Tsuda M, Fukuda M, et al (Eds.).Biodegradative Bacteria[M].Tokyo: Springer Verlag, 2014.267-277. |
 2015, Vol. 36
2015, Vol. 36


