2. 同济大学污染控制与资源化研究国家重点实验室, 上海 200092;
3. 中国科学院重庆绿色智能技术研究院, 水库水环境重点实验室, 重庆 400714
2. State Key Laboratory of Pollution Control and Resource Reuse, Tongji University, Shanghai 200092, China;
3. Key Laboratory of Reservoir Aquatic Environment, Chongqing Institute of Green and Intelligent Technology, Chinese Academy of Sciences, Chongqing 400714, China
抗生素大量使用进入环境介质中诱导抗性基因产生,导致大量耐药细菌出现,威胁公众安全. 2006年,Pruden等[1]提出将抗生素抗性基因作为一种新型污染物. 抗性基因已经在不同的环境介质中(河流[2]、 土壤[3]、 污水处理厂[4]及污泥[5]等)得到广泛检测,但是在垃圾填埋场中还没有相关的报道. 过期或者不用的抗生素不可避免地进入环境,因此需要相关的法律法规来减少其对环境的污染. 美国很多州规定过期或者不用的抗生素必须丢弃到下水道[6],但是这种方式会导致污水处理厂抗性基因升高. 在我国,过期或者不用的抗生素的去向没有明确的路线,但是很多过期或者不用的抗生素随着生活垃圾进入到垃圾填埋场中(尚无具体数据),使垃圾填埋场成为潜在的抗生素抗性基因储存库. 同时,填埋场中抗生素抗性基因的存在,会通过基因水平转移[7]、 雨水冲刷、 垃圾渗滤液、 地表径流等方式污染周边的土壤、 地下水和地表水.
抗生素抗性基因与抗生素水平[8]显著相关,同时还与环境介质本身的理化性质(温度、 光照、 溶解氧等)以及污染物等(如有机物、 重金属[9]等)密切相关. Engemann等[10]研究发现高温可使抗生素降解速率加快,诱导产生的抗性也随之减少. Peak等[11]研究了养牛场蓄粪池中6种四环素抗性基因在夏秋两季的含量水平,结果显示,抗性基因的浓度在秋季明显高于夏季. Pei等[12]检测了经过不同温度生物处理后的牛奶厂废水中4种抗生素抗性基因(tetW、 tetO、 sul Ⅰ 、 sul Ⅱ )的含量,结果发现20℃处理后的抗性基因含量显著低于4℃处理后的含量. Rysz等[13]评估了氧气可利用率对四环素抗性基因增值速率的影响,研究发现厌氧条件可以消减抗性基因. Graham等[14]对哈瓦那西部Almendares河流底泥中抗性基因与环境因子关联进行了调查,发现底泥中抗性基因的丰度与抗生素、 重金属(特别是Cu)的含量呈显著正相关性. 了解环境因子与抗性基因的关系,有助于有效防控环境介质中抗性基因.
本研究以西安江村沟垃圾填埋场为对象,遵循国标采取3个地点3个深度的垃圾样品,分析垃圾的理化指标,用荧光实时定量PCR技术调查了5种抗生素抗性基因的存在和污染水平,同时以相关性分析探讨了抗性基因与垃圾理化性质的相关关系,以期为我国垃圾填埋场中抗生素抗性基因的污染状况提供信息. 1 材料与方法 1.1 样品采集与处理
垃圾采集于西安江村沟垃圾填埋场,如图 1所示取3点分别为S1、 S2、 S3,然后每一点分3层取样,P1、 P2、 P3分别代表垃圾填埋场表层、 中层和深层的土样. P1采集自地表下0~5 m,P2采集自地表下10~15 m,P3采集自地表下20~35 m. S1区是2期扩建区,填埋龄为1~3 a. S2和S3区域为10~15 a填埋区,表层5米区域为填埋场2期扩建新填埋的垃圾,相应的填埋龄S2表层为0.3 a,而S3表层为1 a. 3个取样点遵循填埋龄随着深度增加而增加的规律. 样品采集遵照国标生活垃圾填埋场岩土工程技术规范(CJJ 176-2012),用XY-100型液压型钻机进行钻孔,相应的套筒收集相应深度部位的垃圾.
 | 图 1 西安江村沟垃圾填埋场采样点
Fig. 1 Sampling locations at Jiangcungou landfill,Xian China
|
所采集的样品带回实验室后混匀后一部分用于测定pH和含水率,一部分超低温(-80℃)保存,以备样品DNA提取,一部分低温(-4℃)保存,用于FDA水解酶活性的测定,剩余的垃圾固体放在实验室通风处进行风干后过140目筛,用于其他理化性质的测定. 1.2 垃圾固体理化性质的测定
垃圾固体理化性质的检测按照文献[15]的方法进行检测. 60℃烘箱内烘干24 h后恒重测定含水率[16]; pH值按1 ∶5加水过滤后用pH计测定; 有机质采用重铬酸钾外加热法; 总氮采用半微量凯氏定氮; 总磷采用氢氧化钠熔融法; 铵态氮采用靛酚比色法; 硝态氮采用酚二磺酸比色法; FDA水解酶活性以荧光素二乙酸酯为底物培养垃圾微生物,用分光光度法检测荧光素浓度变化[17]. 1.3 垃圾固体DNA提取
称取5 g垃圾于25 mL离心管中,加入20 mL PBS缓冲液涡旋振荡10 min,以12000 r ·min-1转速离心3 min,倒掉上清液,重复3次后,采用试剂盒PowerSoil DNA Isolation Kit(MO-BIO,USA),按照生产厂商说明的方法提取0.25 g垃圾固体中所含微生物DNA. 提取的DNA用超微量紫外分光光度计NanoVue(GE,USA)检测浓度. 1.4 PCR 实验选择了磺胺类抗生素抗性基因(sul Ⅰ 和sul Ⅱ )、 抗氯霉素类抗生素抗性基因(cat)、 β-内酰胺类抗生素抗性基因(bla-SHV)、 以及四环素类抗生素抗性基因(tetW)等5种抗生素抗性基因,这5种抗生素抗性基因的引物、 退火温度以及片段大小见表 1.
| 表 1 抗生素抗性基因引物 Table 1 Antibiotic resistance genes used in this research |
PCR反应体系为: 10×Taq DNA聚合酶buffer(Mg2+),5 μL; MgSO4(25mmol ·L-1),3 μL; dNTPs,每种脱氧核苷酸2.5mmol ·L-1; rTaq DNA酶,每个反应2.5 Unit; DNA模板,2 μL; ddH2O,34.95 μL; 总体积为50 μL.
PCR反应程序为: 95℃预变性10 min; 然后95℃15 s,退火30 s,72℃ 30 s,35个循环; 接着72℃ 7 min; 4℃保存. 1.5 目的基因片段测序及序列比对
将PCR扩增出来的目的基因,采用琼脂糖凝胶回收试剂盒(Tiangen,Beijing)进行切胶回收后,连接到pMD19 T-vector(TaKaRa,Japan)载体上,然后转化感受态大肠杆菌细胞(DH5a,Biomed). 将转化的感受态细胞涂平板后于37℃培养16 h左右,放入冰箱4℃ 2 h后,挑选出白斑,用含有Ampicillin的 LB(Ampicillin终浓度50 μg ·mL-1)培养液(3~5 mL)扩大培养,取1 mL 菌液送样至北京六合华大基因科技股份有限公司进行测序,其余菌液用来提取质粒,稀释标线备用. 测序结果用DNAStar软件对序列进行拼接、 编辑后,在NCBI网站(http://www.ncbi.nlm.nih.gov/blast/)上进行BLASTn同源性检索比对. 1.6 荧光定量PCR
使用Realplex2定量PCR仪(Eppendorf,Germany)对目标抗性基因进行SYBR Green荧光定量. 定量PCR反应程序: 95℃,15 min; [95℃ 15s(变性),Tm 30 s(引物退火),72℃ 30 s (引物延伸)]×40 次; 95℃ 15 s,60℃ 15 s,升温20 min,95℃ 15 s(溶解曲线添加). 反应体系为20 μL,包含Go Taq Q-PCR Master Mix 10 μL,引物(10 μmol ·L-1)的各0.4 μL,DNA模板1 μL,Nuclease-Free Water 8.2 μL.
标准曲线的制备: 将制备的菌液用质粒提取试剂盒MiniBEST Plasmid Purification Kit Ver 2.0(TaKaRa,Japan)按照生产商说明的方法进行提取,然后将提取的质粒用超微量紫外分光光度计(Nanodrop,UK)检测其浓度. 然后以此浓度的质粒DNA10倍梯度稀释制定标准曲线. 5种标准曲线的系数R2 在0.993~0.999 之间; 扩增效率E在75.8%~112.7%之间; 斜率在-3.05~4.08 之间,表明线性相关良好,可用于计算各基因的拷贝数.
拷贝数计算公式:
gene copy DNA=(c×6.022×1023)/(660×N)
式中,c代表DNA浓度(ng ·μL-1),N代表DNA片段的长度. 1.7 统计分析所得的结果用Spss 19.0进行分析.
2 结果与讨论 2.1 垃圾理化性质
实验测得垃圾固体的理化性质由表 2所示数据可以看出: 随着填埋深度的增加,含水率、 pH、 TN、 TP、 有机质、 硝态氮、 铵态氮含量以及FDA水解酶活性都呈现出无规律变化. 为了进一步清晰地描述垃圾填埋场不同深度垃圾理化性质的综合差异,应用主成分分析(principal components analysis,PCA)对各样点的8个理化指标进行分析,提取出特征值贡献最大的两个主成分(PC1和PC2,贡献率分别为47.64%和29.52%)来作为样点垃圾固体理化特性的变量,不同样点第1和第2主成分得分的散布图如图 2所示. 同时计算理化因子的载荷,如表 3所示(因子载荷是理化因子与相应主成分之间的相关系数,其绝对值就越大,对主成分的得分影响越大): PC1代表含水率,pH值,TN,有机质,硝态氮,铵态氮以及FDA水解酶活性的综合指标,PC2主要代表TP的综合指标. 图 2同样显示了垃圾填埋场不同深度垃圾的理化因子没有明显的规律. 垃圾理化性质的无规律变化是由于垃圾成分复杂不均一的原因导致的,Barlaz等[20]总结美国垃圾的特点也得出相同的结果.
| 表 2 垃圾固体的理化性质结果与5种抗生素抗性基因拷贝数对数值 Table 2 Physicochemical properties and copies of five antibiotic resistance genes of refuse |
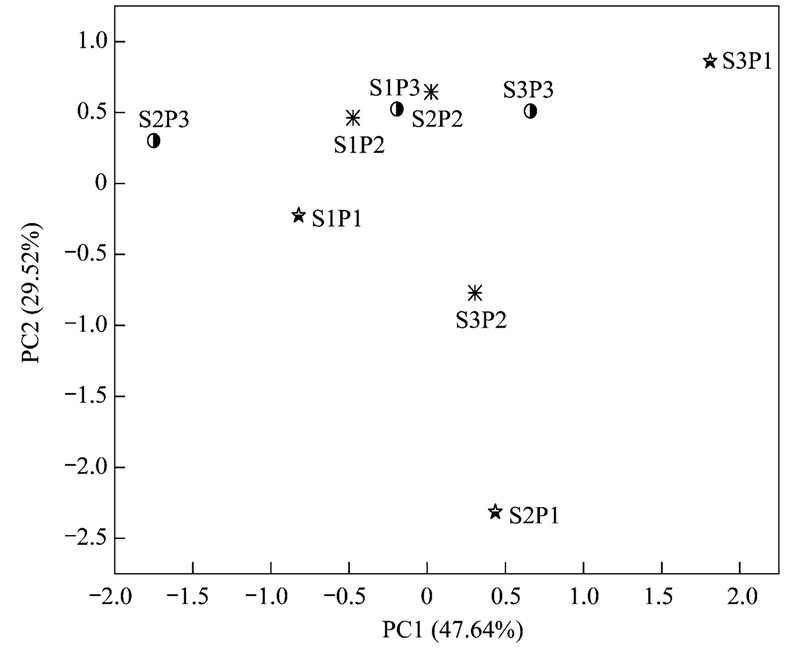 | 图 2 垃圾填埋场不同深度理化性质主成分分析的散布
Fig. 2 PCA analysis of different depths landfill based on physicochemical properties
|
| 表 3 各个理化因子的载荷矩阵 Table 3 Physicochemical factors of loading |
2.2 抗生素抗性基因分析
为了证明荧光定量PCR检测结果的可靠性,将所制的目标抗性基因质粒标准品测序结果与NCBI数据库进行对比,如表 4所示,实验所选引物具有极好的专一性,所得到的基因序列与已有报道大小一致,相似度达99%以上.
| 表 4 5种抗生素抗性基因的BLASTn结果 Table 4 BLASTn results of five antibiotic resistance genes |
以证明的抗性基因引物对垃圾样品中抗性基因进行荧光定量扩增,实验结果如图 3所示. 可以看出,所采的垃圾中都检测到了这5种抗生素抗性基因,说明这5种抗生素抗性基因广泛存在于西安江村沟垃圾填埋场中. 在这5种抗生素抗性基因中,磺胺类抗性基因(sul Ⅰ 和sul Ⅱ )含量最高,四环素类抗性基因次之,而抗氯霉素类抗性基因和β-内酰胺类抗性基因含量较低. 其中sul Ⅱ 基因拷贝数(以干土计,下同)最高可达(3.70±0.06)×108 copies ·g-1,sul Ⅰ 基因拷贝数最高亦可达(9.33±0.06)×106 copies ·g-1,tetW最高基因拷贝数为(2.27±0.08)×105 copies ·g-1,bla-SHV基因拷贝数最高为(3.68±0.09)×104 copies ·g-1,而cat基因拷贝数最高为(1.39±0.10)×104 copies ·g-1. 同时,这5种基因的基因拷贝数最高值都出现在表层垃圾中,可能原因是表层垃圾尚处于稳定化早期,仅有小量抗生素被降解或者迁移转化而保持较大的选择压力,有利于ARGs的增长传播.
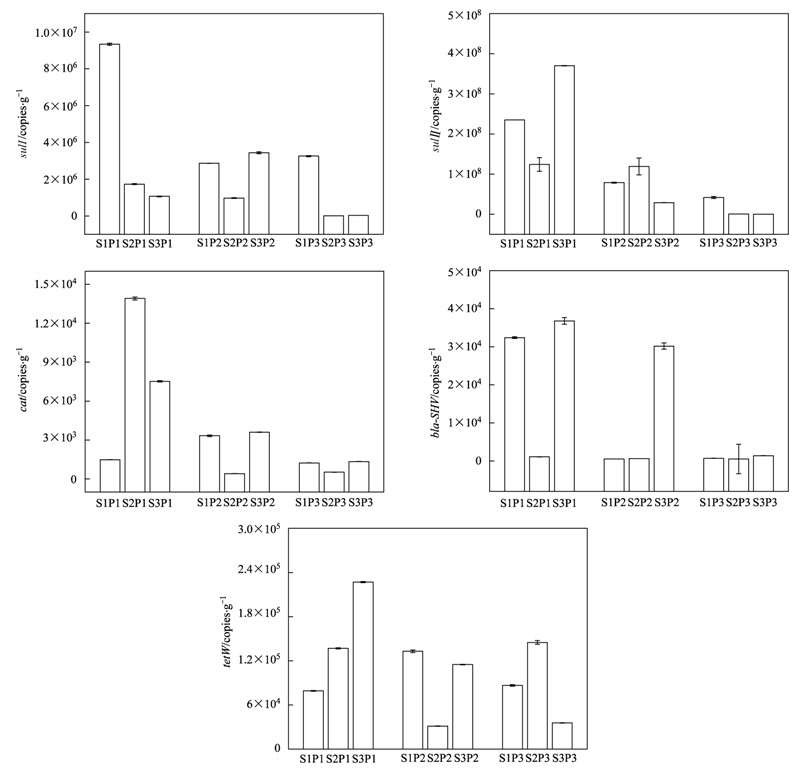 | 图 3 垃圾固体中5种抗生素抗性基因拷贝数
Fig. 3 Copies of five antibiotic resistance genes in landfill
|
抗生素抗性基因普遍存在于中国的环境介质中,尤其是磺胺类抗生素抗性基因与四环素类抗生素抗性基因. Gao等[21]对海河水体和沉积物进行了抗性基因的检测,发现sul Ⅰ 和sul Ⅱ 在沉积物中拷贝数为109~1012 copies ·g-1,是水环境中的120~2000倍. Wu等[22]对畜禽养殖场周边土壤进行了抗性基因的检测,检测出15种四环素类抗性基因,建立了四环素类抗性基因从养殖场到周边土壤迁移的途径. 黄智婷[23]调查了上海两个垃圾中转站渗滤液中抗生素抗性基因的分布,结果表明磺胺类抗生素抗性基因与四环素类抗生素抗性基因均存在于渗滤液中,其中sul Ⅱ 含量最高可达到1011 copies ·mL-1.
中国是世界最大的抗生素生产和消费国,2007年抗生素产量21万t,其中46.1% 用于畜禽养殖[24]. 过期或者不用的抗生素以及不被动物吸收的抗生素进入环境介质,在环境中迁移转化. 当抗生素进入垃圾填埋场后,导致抗性基因在填埋场的大量出现. 同时一些非法进入垃圾填埋场的医疗垃圾或者污水厂污泥由于本身携带大量的抗生素与抗生素抗性基因,也会导致抗性基因在垃圾填埋场的传播. 例如垃圾中转站渗滤液中检测到抗生素抗性基因是其进入垃圾填埋场的直接证据[23]. 2.3 抗生素抗性基因丰度与理化性质相关关系分析
实验测得垃圾固体的理化性质的均值以及5种抗生素抗性基因copies均数的对数值如表 2所示,记垃圾固体理化性质的数据位x1~x8,5种抗生素抗性基因的数据位y1~y5. 采用统计分析软件Spass 19.0对表 2中的数据进行Pearson相关性分析,结果如表 5所示,可以发现抗性基因sul Ⅰ 、 sul Ⅱ 和cat与含水率呈明显的正相关; 同时sul Ⅰ 和cat基因含量与pH值呈负相关. 目前关于环境因子与抗性基因关联的研究尚处于起步阶段,一些研究证明抗性基因与抗性素的传播具有一致性和相似性[8],但研究也表明环境因子会影响抗性基因的传播,例如环境介质中的重金属等污染物对抗性基因的水平转移有一定促进作用[9]. 而在本实验中含水率与pH影响抗性基因的丰度,但是具体机制尚不清楚.
| 表 5 垃圾固体5种抗生素抗性基因与理化性质相关性分析结果 1) Table 5 Correlation analysis between physicochemical properties and copies of five antibiotic resistance genes of refuse |
(1)5种抗生素抗性基因(sul Ⅱ 、 sul Ⅰ 、 tetW、 bla-SHV和 cat) 均存在于垃圾填埋场垃圾中,显示垃圾填埋场是抗生素抗性基因的一个重要储存库.
(2)磺胺类抗性基因含量最高,四环素类抗性基因次之.
(3)抗性基因sul Ⅰ 、 sul Ⅱ 和cat与含水率呈明显的正相关,同时sul Ⅰ 和cat基因含量与pH值呈负相关.
| [1] | Pruden A, Pei R T, Storteboom H, et al. Antibiotic resistance genes as emerging contaminants: studies in northern Colorado[J]. Environmental Science & Technology, 2006, 40 (23): 7445-7450. |
| [2] | 杨颖. 北江水环境中抗生素抗性基因污染分析[D]. 广州: 中山大学, 2010. |
| [3] | Knapp C W, Dolfing J, Ehlert P A I, et al. Evidence of increasing antibiotic resistance gene abundances in archived soils since 1940[J]. Environmental Science & Technology, 2010, 44 (2): 580-587. |
| [4] | 李佳楠. 城市污水系统中典型抗生素、抗性基因和抗性细菌的丰度与关联性研究[D]. 杭州: 浙江大学, 2014. |
| [5] | Zhang T, Zhang M, Zhang X X, et al. Tetracycline resistance genes and tetracycline resistant lactose-fermenting Enterobacteriaceae in activated sludge of sewage treatment plants[J]. Environmental Science & Technology, 2009, 43 (10): 3455-3460. |
| [6] | Kotchen M, Matthew J, Kallaos K, et al. Pharmaceuticals in wastewater: Behavior, preferences, and willingness to pay for a disposal program[J]. Journal of Environmental Management, 2009, 90 (3): 1476-1482. |
| [7] | Hill K E, Top E M. Gene transfer in soil systems using microcosms[J]. FEMS Microbiology Ecology, 1998, 25 (4): 319-329. |
| [8] | 周启星, 罗义, 王美娥, 等. 抗生素的环境残留、生态毒性及抗性基因污染[J]. 生态毒理学报, 2007, 2 (3): 243-251. |
| [9] | Knapp C W, McCluskey S W, Singh B K, et al. Antibiotic resistance gene abundances correlate with metal and geochemical conditions in archived Scottish soils[J]. PLoS One, 2011, 6 (11): e27300. |
| [10] | Engemann C A, Keen P L, Knapp C W, et al. Fate of tetracycline resistance genes in aquatic systems: Migration from the water column to peripheral biofilms[J]. Environmental Science & Technology, 2008, 42 (14): 5131-5136. |
| [11] | Peak N, Knapp C W, Yang R K, et al. Abundance of six tetracycline resistance genes in wastewater lagoons at cattle feedlots with different antibiotic use strategies[J]. Environmental Microbiology, 2007, 9 (1): 143-151. |
| [12] | Pei R T, Kim S C, Carlson K H, et al. Effect of river landscape on the sediment concentrations of antibiotics and corresponding antibiotic resistance genes (ARG)[J]. Water Research, 2006, 40 (12): 2427-2435. |
| [13] | Rysz M, Alvarez P J J. Amplification and attenuation of tetracycline resistance in soil bacteria: aquifer column experiments[J]. Water Research, 2004, 38 (17): 3705-3712. |
| [14] | Graham D W, Olivares-Rieumont S, Knapp C W, et al. Antibiotic resistance gene abundances associated with waste discharges to the Almendares River near Havana, Cuba[J]. Environmental Science & Technology, 2011, 45 (2): 418-24. |
| [15] | 中国科学院南京土壤研究所. 土壤理化分析[M]. 上海: 上海科学技术出版社, 1978. |
| [16] | 朱勇. 基于异化铁还原作用的准好氧填埋场CH4控制技术研究[D]. 北京: 清华大学, 2011. |
| [17] | 马星竹. 长期施肥土壤的FDA水解酶活性[J]. 浙江大学学报(农业与生命科学版), 2010, 36 (4): 451-455. |
| [18] | Xi C W, Zhang Y L, Marrs C Y, et al. Prevalence of antibiotic resistance in drinking water treatment and distribution systems [J]. Applied and Environmental Microbiology, 2009, 75 (17): 5714-5718. |
| [19] | Chee-Sanford J C, Aminov R I, Krapac I J, et al. Occurrence and diversity of tetracycline resistance genes in lagoons and groundwater underlying two swine production facilities[J]. Applied and Environmental Microbiology, 2001, 67 (4): 1494-1502. |
| [20] | Barlaz M A. Microbial studies of landfills and anaerobic refuse decomposition[J]. Manual of Environmental Microbiology, 1997. 541-557. |
| [21] | Gao P P, Mao D Q, Luo Y, et al. Occurrence of sulfonamide and tetracycline-resistant bacteria and resistance genes in aquaculture environment[J]. Water Research, 46 (7): 2355-2364. |
| [22] | Wu N, Qiao M, Zhang B, et al. Abundance and diversity of tetracycline resistance genes in soils adjacent to representative swine feedlots in China[J]. Environmental Science & Technology, 2010, 44 (18): 6933-6939. |
| [23] | 黄智婷. 抗生素残留及抗性基因在生活垃圾转运填埋过程中的迁移转化和去除研究[D]. 上海: 华东师范大学, 2014. |
| [24] | Hvistendahl M. China takes aim at rampant antibiotic resistance[J]. Science, 2012, 336 (6083): 795-795. |
 2015, Vol. 36
2015, Vol. 36


