2. 环境保护部南京环境科学研究所, 南京 210042;
3. 国家环境保护农药环境评价与污染控制重点实验室, 南京 210042
2. Nanjing Institute of Environmental Sciences, MEP, Nanjing 210042, China;
3. State Environmental Protection Pesticide Environmental Assessment and Pollution Control Key Laboratory, Nanjing 210042, China
随着畜牧养殖业的集约化发展,抗生素已广泛用于养殖业达50 a之久[1]. 其中四环素由于具有较强的抗炎、 免疫作用且价格低廉[2],常被作为饲料添加剂及防病药物在畜牧业尤其养猪行业中广泛使用[3]. 畜禽养殖中大量使用的四环素多会以原药的形式随粪便及尿液排出动物体外[4]. 由于畜禽粪便中残留的四环素类药物在非特定条件下降解较为缓慢[5],当被持续施加到农田后就直接导致了土壤污染. 近年来,国外有报道指出环境中抗生素的残留可诱导环境中抗性基因产生,并可维持其含量水平[6]. Pruden等[7]指出,抗生素污染所诱导的抗性基因与抗生素一样,也可能成为一种新型环境污染物,而且其风险要比抗生素药物本身的污染风险更高. 此外,土壤抗生素污染还可通过地表径流和淋滤作用进一步造成水体的抗生素污染[8],并引发细菌耐药性,威胁动物与人类健康. 鉴于四环素残留可能造成的环境和健康危害,很多国家已经在养殖业中制定了四环素使用规范并强制进行禽畜粪便的无害化处置[9]. 然而我国在此方面的研究才刚刚开始,所暴露出来的某些环境问题却日趋严重[10],因此,了解四环素残留对耐药菌及抗性基因形成的影响,对于降低四环素耐药菌及抗性基因的环境污染,制定养殖行业四环素的使用规范,控制我国有机肥料使用中的环境风险,保护人类的身体健康显得尤为重要.
1 材料与方法 1.1 土壤的采集
实验所用土壤采集于江苏省沭阳市某年出栏量为3 000头左右的养猪场周边种植耕地,已持续施用该养猪场的未经任何处理的猪粪8 a,每年施加猪粪肥2~3次. 土壤性质为黄棕壤. 本研究中采样分为两次,2011年11月(秋季)不同点采集土壤样品7个,分别标记为A1~A7,2012年7月(夏季)采集4个土壤样品,分别标记为S1~S4. 其中S1、 S2、 S3为A1、 A2、 A3的同点重复采样. 同时于2011年11月采集当地远离养猪场,从未施加过猪粪的耕地土壤作为空白对照,标记为CK. 土壤采集于地表以下10~15 cm处,采集后于4℃冰箱中冷藏保存[11].
1.2 猪粪的采集
采集该养猪场内新鲜猪粪若干,置于干净的塑料袋中,于4℃冰箱中保存.
1.3 土壤和猪粪中四环素残留量的分析
采用LC-MS/MS法[12]测定土壤和猪粪中四环素残留量,HPLC测定条件:色谱柱:ACQUITY UPLC BEH C18 柱(1.7 μm,2.1 mm×100 mm; Waters); 柱温:35℃; 流动相:乙腈(A)和0.1%甲酸/水(体积比)(B),流速0.2mL ·min-1. MS检测条件:电喷雾离子源(ESI),离子源温度为120℃,脱溶剂温度为350℃,脱溶剂气和锥孔气为氮气,脱溶剂气流速为600 L ·h-1,锥孔气流速为50 L ·h-1,碰撞气为高纯氩气,采用多反应监测模式(MRM)检测. 测定使用ESI正离子模式,进样5 μL. 土壤中四环素的检出率为100%,回收率为70.4%±5.7%,检测限为0.2 ng ·g-1. 粪便中四环素的检出率为100%,回收率为68.3%±6.6%,检测限为0.3 ng ·g-1.
1.4 土壤的处理 1.4.1 土壤总DNA的提取
分别取各采样点土壤0.25 g,按照PowerSoil-DNA Isolation Kit(MoBio,美国)试剂盒说明书提取土壤总DNA.
1.4.2 土壤中可培养细菌和耐药菌丰度分析
每个土壤样品称取5 g,分别加入到装有45 mL无菌生理盐水和若干小玻璃珠的三角瓶中,37℃下200 r ·min-1振荡30 min制成土壤悬液,记为稀释度1,然后按照10倍倍比稀释的方法将样品梯度稀释至10-2、 10-3、 10-4、 10-5. 分别取上述稀释梯度的土壤悬液200 μL涂布于牛肉膏蛋白胨平板培养基和含16 μg ·mL-1四环素的牛肉膏蛋白胨平板培养基上,各稀释度制备3个平行板. 平板倒置于生化培养箱中,37℃恒温培养2~5 d,根据菌落生长情况确定培养时间(H). 计数每个平板上的菌落数,从而计算每克土壤样品中的菌落形成单位(CFU). 计算公式为:菌落数/g土=(9×平均菌落数×稀释倍数)/0.2.
1.4.3 耐药菌的分离
利用平板划线法纯化抗性平板上长出的耐药菌. 挑取纯化后的单菌落接种至肉汤培养基(含四环素16 μg ·mL-1)中培养,培养好的耐药菌置于甘油管中,于-70℃冰箱中保存,以作后续研究. 从秋季土壤样品中分离出59株耐药菌,A1~A7分别为14、 19、 8、 4、 5、 4、 5株,夏季样品中分离出35株耐药菌,其中S1 8株,S2 14株,S3 6株,S4 7株,对照组CK中分离出3株耐药菌.
1.4.4 耐药菌基因组DNA和质粒DNA的提取
将纯化好的耐药菌接种于肉汤培养基(含四环素16μg ·mL-1)中,在37℃,200 r ·min-1摇床中培养过夜,分别利用TIANamp Bacteria DNA Kit(Tiangen,中国)和AxyPrep Plasmid Miniprep Kit(Axygen,美国)提取耐药菌基因组DNA和质粒DNA.
1.5 耐药菌的鉴定
以提取的耐药菌基因组DNA作为模板,选取16S rDNA通用引物27F(5′-AGAGTTTGATCAT GGCTCAG-3′)和1492R(5′-TACGGYTACCTTG TTACGACTT-3′)进行PCR扩增. 扩增体系:100~200 ng 基因组DNA,10×Taq plus buffer(1.5 mmol MgCl2),2.5 mmol dNTP,1.2 U Taq plus DNA聚合酶,10 μmol 上/下游引物,补足ddH2O至25 μL. 扩增条件:94℃预变性5 min,94℃变性1 min,58℃退火30 s,72℃延伸90 s,30个循环,最后72℃延伸10 min完成扩增. 设置双蒸水为模版的阴性对照. 将扩增好的PCR产物送往南京金斯瑞生物科技有限公司测序. 从Internet 进入NCBI站点后,用“Nucleotide Blast”程序将测序序列与GenBank中已知的各种微生物16S rDNA进行相似性搜索,寻找同源序列,确定其所在菌属.
1.6 tetA、 tetC、 tetE抗性基因的检测 1.6.1 基因组DNA和质粒DNA为模版的PCR扩增
抗性基因tetA、 tetC和tetE 的引物设计来源于文献[13, 14],引物序列如表 1. 建立25 μL扩增体系,其中含100~200 ng 基因组DNA或质粒DNA、 10×Taq plus buffer(1.5 mmol MgCl2)、 1.5 U Taq plus DNA聚合酶、 2.5 mmol dNTP、 10 μmol 上/下游引物、 ddH2O补充体积至25 μL. 混匀上述反应液后,94℃预变性5 min,然后按94℃变性1 min,60℃、 59℃、 56℃(tetA、 tetC、 tetE)退火30 s,72℃延伸45 s,最后于72℃延伸7 min结束反应. 选取基因组DNA A202和质粒DNA A206的扩增产物送往南京金斯瑞生物科技有限公司测序,经比对,确定基因组DNA A202上含有tetA和tetC基因,质粒DNA A206上含有tetE基因,故将其作为检测的阳性对照; E.coli DH5α不含tet基因,被当作阴性对照. PCR扩增产物经1.5%琼脂糖凝胶电泳检测.
1.6.2 土壤总DNA上tetA和tetC基因的RT-PCR定量分析
从GenBank数据库中获取tetA和tetC基因的cDNA序列,通过NCBI的Primer-Blast(http://www.ncbi.nlm.nih.gov/tools/primer-blast)软件设计RT-PCR引物,引物序列见表 1. 参照Ultra SYBR mixture(CWbio,中国)试剂盒说明书在CFX96 TouchTM Real-Time PCR Detection System (Bio-Rad,美国)仪器上进行RT-PCR扩增,扩增条件为95℃热变性10 min,后进行40个循环,每个循环包括95℃变性15 s,60℃反应1 min,最后60℃升到95℃,其间每隔0.5℃采集一次荧光以生成溶解曲线,根据溶解曲线的变化检测扩增结果的特异性.
| 表 1 引物设计Table 1 Primer for PCR |
选取基因组DNA A202(特异性扩增产物送往南京金斯瑞生物科技有限公司测序,经比对,确定基因组DNA A202上含有tetA和tetC基因)作为阳性模版,提取出的基因组DNA用琼脂糖凝胶电泳及紫外分光光度计(UV-8000PC)检测含量以及纯度,A260/A280值在1.8~2.0之间,表明提取的基因组DNA纯度较高. 基因拷贝数按以下公式计算:拷贝数=(质量/相对分子质量)×6.02×1023. 提取的基因组DNA进行10倍浓度梯度稀释作为反应模版,以拷贝数的log值为横坐标,Ct值为纵坐标绘制标准曲线. 建立的标准曲线如图 1、 图 2.
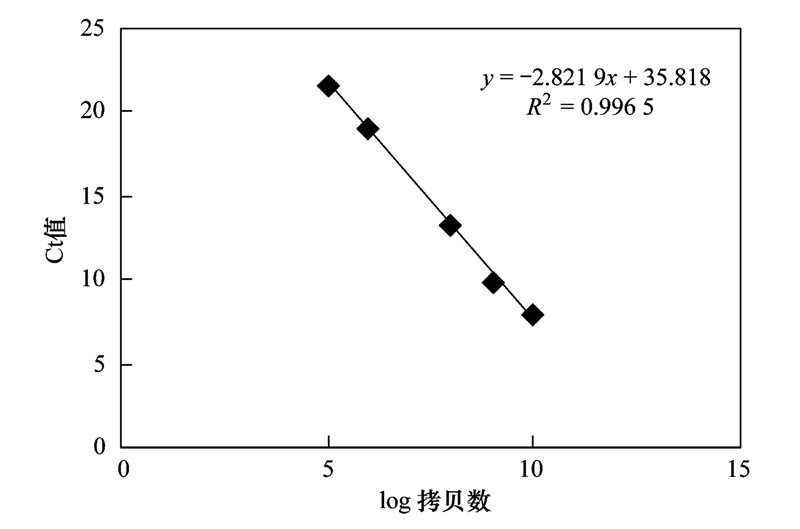 | 图 1 tetA基因标准曲线 Fig. 1 Standard curve of tetA gene |
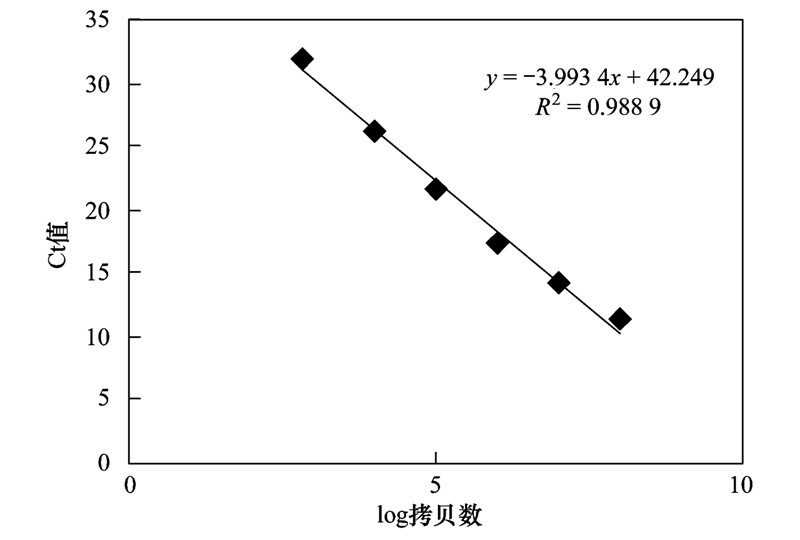 | 图 2 tetC基因标准曲线 Fig. 2 Standard curve of tetC gen |
2 结果与讨论 2.1 长期施用四环素残留猪粪后土壤中四环素的残留情况
从表 2中可以看出,采集的新鲜猪粪中四环素的残留量(干重)为908.2μg ·kg-1. 秋季土壤和夏季土壤中四环素的残留含量为41.1~61.9 μg ·kg-1,这与Zhu等[15]检测的北京等地农场附近使用禽畜粪便后土壤中的四环素残留量较为接近,在同一个数量级别. 在对照组CK中未检出四环素残留.
| 表 2 四环素残留量与土壤中耐药菌、 可培养细菌的丰度Table 2 Tetracycline residues and the amount of drugresistant bacteria and cultivable bacteria in soil |
2.2 长期施用四环素残留猪粪对土壤中耐药菌种类及丰度的影响 2.2.1 长期施用四环素残留猪粪后土壤微生物的四环素抗性水平
利用涂布平板法分别统计了各土壤样品中四环素耐药菌菌落数和可培养细菌菌落数,结果显示施用过猪粪肥和未施用过猪粪肥土壤中可培养细菌菌落数(以CFU表示)介于2.83×107~10.24×107之间,耐药菌菌落数介于0.0032×105~8.56×105(表 2),这表明采集的所有土壤中都有着丰富的细菌及耐药菌. 秋季和夏季各土壤样品中四环素耐药菌菌落数都达到了105 CFU ·g-1,远高于对照组中四环素耐药菌菌落数(0.003 2×105 CFU ·g-1). 同时秋季和夏季土壤中四环素耐药菌菌落数占可培养细菌菌落数的比例分别为0.14%~1.08%、 0.58%~0.71%,都明显高于对照组中四环素耐药菌所占的比例(0.001%). 这说明长期施用含四环素残留的猪粪能够提高土壤中微生物的四环素抗性水平. 另外土壤中可培养细菌及四环素耐药菌的数量巨大,可能会为耐药菌和抗性基因的传播与扩散提供良好的来源与介质[16],具有较高的环境风险性.
2.2.2 长期施用四环素残留猪粪对耐药菌形成的影响
利用涂布平板法从采集的12个土壤样品中共分离到97株耐药菌,其中94株来自于11个施用过猪粪肥的土壤样品,3株来自于未施用过猪粪肥的土壤样品. 根据菌落主要形态特征和16S rDNA序列分析结果,对纯化后的这些耐药菌菌株进行了鉴定,发现它们分别属于16个属. 各样品中耐药菌的组成如图 3所示,在所分析的12个土壤样品中,A2样品中的耐药菌多样性最为明显,该土壤中共分离鉴定到7个菌属的耐药菌; 其次为A5和 S1,都具有5个属的耐药菌(图 3). 从秋季土壤样品中分离出的59株耐药菌属于13种菌属,夏季分离出的35株耐药菌属于10种菌属,而对照组中的耐药菌只属于一种菌属(Streptomyces),明显低于前两者,这些结果都表明长期施用四环素残留猪粪有助于增加土壤中耐药菌的多样性. 耐药菌中出现较多的为Bacillus属(27株)和Streptomyces 属(17株),这两个菌属都是土壤中的土著微生物,这与Jensen 等[17]的研究结果较为一致,其发现原本土壤中就已经大量存在的细菌产生了耐药性,且占据了很大比例,这也更好地说明了土壤中大量的四环素耐药菌由猪粪直接带入或者其他途径进入土壤的几率较小,而主要是通过四环素诱导作用生成的. 以上结果都表明长期施加含有四环素残留的猪粪能够明显诱导生成大量且种类复杂的四环素耐药菌.
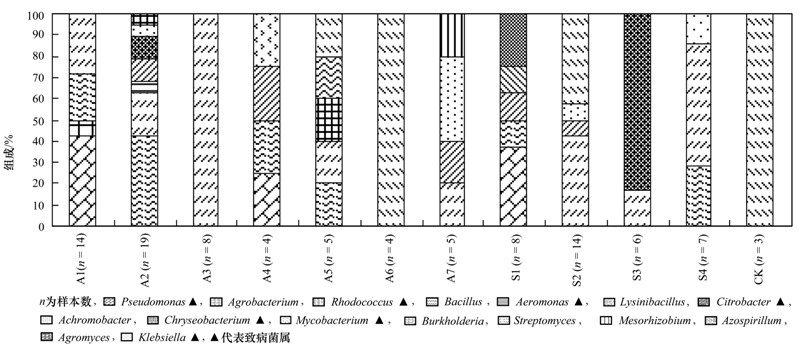 | 图 3 土壤中耐药菌的组成 Fig. 3 Composition of tetracycline-resistant bacteria in the soil microcosms |
2.2.3 长期施用四环素残留猪粪对致病菌形成的影响
在97株耐药菌中可以检测到37株致病菌,占总耐药菌数的38.14%. 致病菌分别属于7种菌属,其中含量较多的为耐药型Rhodococcus(15株)、 Pseudomonas(10株)等. 致病菌对人类健康具有一定的危害性,如Rhodococcus近年来作为人体病菌越来越引起人们的关注[18]; Pseudomonas更是一种抗药性极强的细菌,能引起化脓性病变[19]. 数量如此多、 种类如此繁杂的耐药性致病菌一方面可能来源于动物体内,由粪便带入[20],或者由于蚊蝇滋生,从外界带入病原菌[21],另一面更可能是来源于土壤中四环素的直接诱导作用. 这些具有抗药性的致病菌一旦进入环境后,将对有接触的居民的健康产生明显的不利影响,容易造成疾病的传播. 结果表明长期施加的含有四环素残留的猪粪是土壤中抗药性致病菌的重要来源和促进其生成的主要推力,如果粪便资源化再利用时,兽药抗生素的残留问题得不到重视的话,将会严重危害人畜健康.
2.3 长期施用四环素残留猪粪对耐药菌中tetA、 tetC、 tetE抗性基因分布的影响
利用tetA、 tetC和tetE特异性引物对分离到的97株(其中94株分离自施用过猪粪的土壤,3株分离自未施用过猪粪的土壤)耐药菌基因组DNA和质粒DNA上的四环素抗性基因进行了分析. 统计结果显示,前94株四环素耐药菌的基因组DNA上均检测到四环素抗性基因,质粒DNA上抗性基因出现的几率为95.7%,而对照组的3株耐药菌的基因组DNA和质粒DNA上均未检测到抗性基因. 进一步分析可知,有92株耐药菌的基因组DNA被检测出含有tetC基因,其次为tetA,45株,没能在基因组DNA上检测到tetE基因,这与文献中报道的tetE往往存在于大片段的质粒DNA上相符合[22]. 在质粒DNA上,3种抗性基因都能被检出,tetC(91株)和tetA(41株)基因保持着较高的检出率,检出率比较为tetC>tetA>tetE,3种抗性基因的检出率与Zhang 等[23]报道的检出率相当. 有研究表明,耐药菌可以通过质粒交换等水平基因转移的方式将抗性基因传播给环境微生物[24],造成抗性基因的扩散,而在本研究中所检测的四环素抗性基因tetA、 tetC和tetE均为用于编码外排泵蛋白的基因,这些外排泵基因又常与携带不同抗性基因甚至不同来源的质粒相连,这无疑加剧了抗性基因在细菌群体中的转移并使细菌易于产生多重耐药性,值得注意的是被鉴定出的37株致病菌的质粒DNA上都能检测出抗性基因,这些抗性基因如果通过质粒交换等方式在环境中进行传播,将可提高环境对四环素类抗生素的抗性水平. 研究结果说明,长期施用四环素残留猪粪能够诱导耐药菌的基因组DNA和质粒DNA上产生大量的抗性基因,同时存在于质粒DNA上的抗性基因又可能通过基因水平转移的方式扩散,将引发巨大的潜在危害,并威胁到环境及人类健康.
2.4 长期施用四环素残留猪粪对土壤中tetA、 tetC抗性基因量的影响
利用荧光定量法测定了施用过猪粪肥与未施用过猪粪肥的土壤总DNA上tetA和tetC基因的含量,结果发现施用过猪粪肥的土壤总DNA上tetA的含量范围为2.81×105~5.25×105 copies ·g-1,明显低于tetC的含量范围1.82×105~32.17×105 copies ·g-1,这与邹世春等[25]检测的养殖场土壤中tetA和tetC基因的含量及规律较为一致,而在未施用过猪粪肥的土壤总DNA上则未能检测出抗性基因的残留量. 这些结果表明养猪场周边的土壤由于受到四环素的持续性输入,导致了tetA和tetC基因在土壤中被诱导生成,从而造成抗性基因对土壤的污染. 将各样品中的四环素残留量与相对应的四环素抗性基因含量进行比对与拟合,发现各样品中四环素抗性基因(tetA和tetC)总含量和与四环素的残留量呈现良好的正相关线性关系(R2=0.981 7),拟合的线性方程如图 4. 这也表明在一定的四环素残留量范围内,可以通过四环素的残留量来粗略计算抗性基因的含量,从而判断土壤中抗性基因的污染水平,为抗性基因污染的鉴定提供了一条便捷途径.
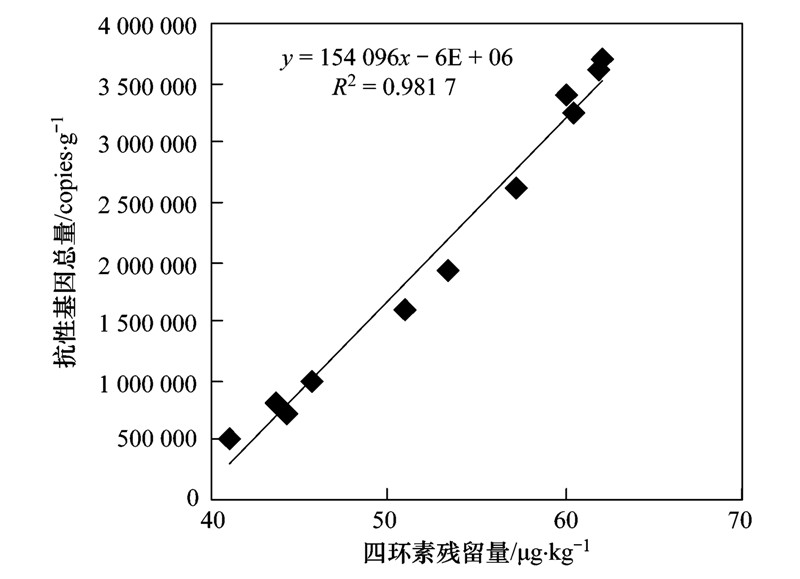 | 图 4 四环素残留量与抗性基因总量的线性方程Fig. 4 Liner equation of tetracycline residues and the total amount of resistance genes |
2.5 气候变化对耐药菌及抗性基因形成的影响
在本研究中S1、 S2、 S3为A1、 A2、 A3的同点重复采样,从A1、 A2和A3中分离出41株耐药菌,分别属于10个菌属,各土壤样品中耐药菌菌落数占可培养细菌菌落数的比例为0.84%~1.08%; 从S1、 S2和S3中分离出28株耐药菌,分别属于9种菌属,土壤中耐药菌菌落数占可培养细菌菌落数比例为0.35%~0.71%,比较发现秋季耐药菌的种类和数量都明显高于夏季. A1、 A2、 A3土壤中抗性基因(tetA和tetC)的总含量范围为1.93×106~3.59×106 copies ·g-1,高于S1、 S2、 S3抗性基因的含量范围5.13×105~9.94×105 copies ·g-1,这些结果都表明秋季较夏季更适合耐药菌和抗性基因的生成,究其原因可能是由于秋季凉爽的气候较夏季的闷热高温更适合耐药菌和抗性基因的生成[26]; 另一方面可能由于夏季降雨量增加,加快了土壤中四环素的流失,在本研究中得到了验证,A1、 A2、 A3的四环素残留量平均值为57.5μg ·kg-1,显著高于S1、 S2、 S3的平均值43.7μg ·kg-1. 当夏季土壤中四环素浓度降低,诱导产生的耐药菌和抗性基因的量也相应下降. 这表明气候条件的变化对四环素耐药菌及抗性基因的形成具有一定的影响,可以通过改变一定的气候条件来有效抑制四环素耐药菌及抗性基因的形成.
3 结论
(1)长期施加四环素残留猪粪能促进包括致病菌在内的耐药菌及抗性基因的形成,对人体健康将会产生明显的不利影响,同时畜禽养殖场附近的土壤等环境也成为了抗性基因的一个重要储存库. 因此兽禽粪便利用前的抗生素风险评估与无害化处置,对于控制环境中抗生素耐药菌和抗性基因的形成显得尤为重要.
(2)在目前四环素残留量的水平下,土壤中四环素残留量与其诱导的抗性基因的含量存在着显著的相关关系,为快速判断抗性基因的污染水平提供了便捷途径.
(3)适宜的气候条件更适合耐药菌和抗性基因的生成,通过人为改变一定的气候条件可以有效抑制土壤中耐药菌及抗性基因的生成.
| [1] | Looft T, Johonson T A, Allen H K, et al. In-feed antibiotic effects on the swine intestinal microbiome[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109 (5): 1691-1696. |
| [2] | 王秋霞, 赵刚, 吴洪涛, 等. 四环素的研究进展[J]. 现代畜牧兽医, 2012, (10): 67-68. |
| [3] | 闾幸,余卫娟,兰亚琼,等.嘉兴市地表水中兽用抗生素的污染现状调查[J].环境科学,2013, 34 (9):3368-3373. |
| [4] | Chee-Sanford J C, Mackie R I, Koie S, et al. Fate and transport of antibiotic residues and antibiotic resistance genes following land application of manure waste[J]. Journal of Environmental Quality, 2009, 38 (3): 1086-1108. |
| [5] | 童子林, 刘元璐, 胡真虎, 等. 四环素类抗生素污染畜禽粪便的厌氧消化特征[J]. 环境科学, 2012, 33 (3): 1028-1032. |
| [6] | McGowan E. Comment on "antibiotic resistance genes as emerging contaminants studies in Northern Colorado"[J]. Environmental Science & Technology, 2007, 41 (7): 2651-2652. |
| [7] | Pruden A, Pei R T, Storteboom H, et al. Antibiotic resistance genes as emerging contaminants, studies in Northern Colorado[J]. Environmental Science & Technology, 2006, 40 (23): 7445-7450. |
| [8] | Weiss K, Schüssler W, Porzelt M. Sulfamethazine and flubendazole in seepage water after the sprinkling of manured areas[J]. Chemosphere, 2008, 72 (9): 1292-1297. |
| [9] | Kim K R, Owens G, Kown S I, et al. Occurrence and environmental fate of veterinary antibiotics in the terrestrial environemnt[J]. Water, Air, & Soil Pollution, 2011, 214 (1-4): 163-174. |
| [10] | 尹春艳, 骆永明, 滕应, 等. 典型设施菜地土壤抗生素污染特征与积累规律研究[J]. 环境科学, 2012, 33 (8): 2810-2816. |
| [11] | Wu N, Qiao M, Zhang B, et al. Abundance and diversity of tetracycline resistance genes in soils adjacent to representative swine feedlots in China[J]. Environmental Science & Technology, 2010, 44 (18): 6933-6939. |
| [12] | 邰义萍, 莫测辉, 李彦文, 等. 长期施用粪肥菜地土壤中四环素类抗生素的含量与分布特征[J]. 环境科学, 2011, 32 (4): 1182-1187. |
| [13] | Ng L K, Martin I, Alfa M, et al. Multiplex PCR for the detection of tetracycline resistant genes[J]. Molecular and Cellular Probes, 2001, 15 (4): 209-215. |
| [14] | Glenn L S M, Lindsey R L, Folster J P, et al. Antimicrobial resistance gens in multidrug-resistant salmonella enterica isolated from animals, retail meats, and humans in the United States and Canada[J]. Microbial Drug Resistance, 2013, 19 (3): 1175-184. |
| [15] | Zhu Y G, Johnson T A, Su J Q, et al. Diverse and abundant antibiotic resistance genes in Chinese swine farms[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110 (9): 3435-3440. |
| [16] | Luo Y, Xu L, Rysz M, et al. Occurrence and transport of tetracycline, sulfonamide, quinolone, and macrolide antibiotics in the Haihe River Basin, China[J]. Environmental Science & Technology, 2011, 45 (5): 1827-1833. |
| [17] | Jensen L B, Baloda S, Boye M, et al. Antimicrobial resistance among Pseudomonas spp. and the Bacillus cereus group isolated from Danish agricultural soil[J]. Environment International, 2001, 26 (7-8): 581-587. |
| [18] | 华苟根, 郭坚华. 红球菌属的分类及应用研究进展[J]. 微生物学通报, 2003, 30 (4): 107-108. |
| [19] | Tuon F F, Gortz L W, Rocha J L. Risk factors for pan-resistant Pseudomonas aeruginosa bacteremia and the adequacy of antibiotic therapy[J]. The Brazilian Journal of Infectious Diseases, 2012, 16 (4): 351-356. |
| [20] | Jiang H X, Tang D, Liu Y H, et al. Prevalence and characteristics of β-lactanmase and plasmid-mediated quinolone resistance genes in Escherichia coil isolated from farmed fish in China[J]. Journal of Antimicrobial Chemotherapy, 2012, 67 (10): 2350-2353. |
| [21] | 叶小梅, 常志州, 陈欣, 等. 畜禽养殖场排放物病原微生物危险性调查[J]. 生态与农村环境学报, 2007, 23 (2): 66-70. |
| [22] | Call D R, Bakko M K, Krug M J, et al. Identifying antimicrobial resistance genes with DNA microarrays[J]. Antimicrobial Agents and Chemotherapy, 2003, 47 (10): 3290-3295. |
| [23] | Zhang T, Zhang M, Zhang X X, et al. Tetracycline resistance genes and tetracycline resistant Lactose-Fermenting Enterobacteriaceae in activated sludge of sewage treatment plants[J]. Environmental Science & Technology, 2009, 43 (10): 3455-3460. |
| [24] | Ansari M I, Grohmann E, Malik A. Conjugative plasmids in multi-resistant bacterial isolates from Indian soil[J]. Journal of Applied Microbiology, 2008, 104 (6): 1774-1781. |
| [25] | 邹世春, 李青, 贺竹梅. 禽畜养殖场土壤抗生素抗性基因污染的初步研究[J]. 中山大学学报, 2012, 51 (6): 87-91. |
| [26] | 徐冰洁, 罗义, 周启星, 等. 抗生素抗性基因在环境中的来源、传播扩散及生态风险[J]. 环境化学, 2010, 29 (2): 169-178. |
 2014, Vol. 35
2014, Vol. 35


