2. 中国环境科学研究院西南分院, 重庆 401147;
3. 成都大学建筑与土木工程学院, 成都 610106;
4. 百色学院农业与食品工程学院, 百色 533000
2. Southwest Branch of Chinese Research Academy of Environmental Sciences, Chongqing 401147, China;
3. School of Architecture and Civil Engineering, Chengdu University, Chengdu 610106, China;
4. Faculty of Agriculture and Food Engineering, Baise University, Baise 533000, China
邻苯二甲酸酯(phthalic acid esters, PAEs)生产成本低, 缺乏廉价的替代品, 长期作为增塑剂等被添加到塑料农膜、食品包装、装修材料、化妆品、玩具、医用口罩和手套等产品中[1~3].在以上产品生产、使用和废物处置过程中, PAEs与聚合物高分子之间的氢键易断裂, 从而被释放到环境中并持久存在[1].人类通过日常消费塑料产品直接接触PAEs[2, 3], 也可通过饮食途径摄入土壤中的PAEs[4], 长期暴露会导致内分泌、呼吸和生殖系统疾病, 甚至引发癌症[4].PAEs作为高关注的新污染物, 其使用受到限制, 但塑料农膜的不合理回收和再生利用等仍造成我国农田土壤和作物中PAEs含量有增无减[4, 5], 其中邻苯二甲酸正丁酯(DBP)和邻苯二甲酸二(2-乙基己基)酯(DEHP)检出率和含量均最高, 是主要的PAEs污染物[6~9].因此, 面对我国新污染物治理和污染土壤精准修复的迫切需求, 强化农田土壤中PAEs的风险管控和去除具有重要基础意义和支撑作用.
土壤微生物是土壤生态系统物质循环和能量流动的驱动者[10].当土壤暴露在有机污染中, 土壤微生物群落迅速对环境扰动进行调节和适应, 并降解污染物, 从而维持生态系统的稳定[11, 12].有研究表明, PAEs污染会改变土壤微生物群落结构和功能, 并扰乱土壤生态系统:DBP会降低土壤细菌群落多样性、碳代谢和氮代谢等功能活性, 导致土著微生物群落朝着不可逆的方向演替[13~15];DEHP同样会降低细菌群落多样性, 但能显著提升降解菌丰度以及编码酯酶和羧酸酯酶等降解酶的基因丰度[16, 17].然而, 先前研究过多关注土壤微生物群落结构的改变, 未深入探究群落中关键种的核心作用及其潜在的代谢功能.有研究表明识别关键物种并重建其代谢潜力, 可以揭示生态系统的物质循环过程[18];也可以利用关键种丰度的变化指示污染程度[19].关于PAEs修复机制的研究大多只关注某种污染物, 且剂量较高.因此, 厘清土壤中微生物群落对PAEs污染的去除机制及关键物种的作用, 对农田合理利用和保护十分重要, 也是拓展农业微生物资源的重要方法, 探寻不同类型PAEs在低剂量下修复机制的共性对于PAEs治理修复技术的推广也十分迫切.
紫色土富含矿物质, 是我国川渝丘陵地区重要的农业土壤资源[20].本文以紫色土为环境介质, 选取DEHP和DBP为研究对象, 利用宏基因组学技术揭示土壤土著微生物群落结构与功能对低剂量下不同程度PAEs污染的响应机制, 并识别PAEs降解过程的关键细菌及其代谢潜力, 通过揭示紫色土中不同类型PAEs去除中的共性机制, 以期为保护紫色土和相关环境基准的制定提供基础数据支撑.
1 材料与方法 1.1 样品采集供试土壤采自重庆市某农田(105°49′ E, 28°28′ N)表层土壤(0~20 cm), 去除石砾和植物残根等杂物, 研磨过2 mm筛备用.土壤类型为紫色湿润雏形土, 土壤质地(美国制)为粉砂壤土, 土壤pH为6.89, 含水量为15.73%, ω(总有机碳)为8.32 g·kg-1, ω(溶解性有机碳)为14.37 mg·kg-1, ω(全氮)为0.81g·kg-1, ω(全磷)为0.85 g·kg-1, ω(∑PAEs)为0.14 mg·kg-1(只有DEHP检出).
1.2 实验设计DBP、DEHP、同位素内标DBP-D4和色谱级丙酮等有机试剂购自德国Merck公司.本实验开始前, 将DBP和DEHP分别溶于丙酮中制备成250、500和1 000 mg·L-1的工作液, 于4℃冰箱内避光保存备用.向每个干净的玻璃烧杯中装入称好的1 kg土壤, 加入适当去离子水, 调整土壤湿度到田间最大持水量的60%, 在恒温恒湿培养箱(温度25℃, 相对湿度70%)避光培养7 d, 以恢复并稳定土壤微生物活性.分别将0.02 L的3种浓度的DBP工作液添加到土壤中, 混合均匀, 制备梯度为5、10和20 mg·kg-1的DBP污染土壤, 并用同样的方法制备DEHP污染土壤.另将0.02 L丙酮加入土壤, 制备未污染土壤, 作为空白对照.每个处理设置3个平行.将每个样品于空气中放置3 h, 使丙酮挥发.用保鲜膜封口以减少水分散失, 并用针刺约20个小孔, 保持通气, 置于恒温恒湿培养箱避光培养, 于第7、15、30、60和90 d取样测定PAEs.参考先前文献中两种物质的半衰期[13~17, 21], 同时考虑测序成本, 选取7 d和15 d的样品分别混合均匀后进行宏基因组测序, 样品名称见表 1.
|
|
表 1 实验设计 Table 1 Experiment design |
1.3 土壤PAEs测定
使用美国Thermo fisher公司的Trace 1300/TSQ 9000气相色谱-三重四极杆质谱联用仪测定PAEs, 样品前处理方法参考GB/T 39234-2020[22].毛细管色谱柱类型为TG-17(30 m×0.25 mm×0.25 μm), 载气为高纯氦气, 流速为1.2 mL·min-1, 采用选择性离子检测器模式, 无脉冲不分流进样, 进样体积为1 μL, 程序升温设置如下:50℃保持1 min, 以15℃·min-1升温至200℃保持1 min, 再以8℃·min-1升至280 ℃保持3 min.
测定前, 所有玻璃器皿浸在重铬酸钾洗液中过夜, 用自来水和去离子水重复洗3次, 烘干, 再用丙酮冲洗, 测定时没有使用塑料物品.实验前检查仪器空白和试剂空白, 每10个样品添加1个空白样品, 并测定样品平行样, 最终得到的每个样品含量, 取平行样的均值减去空白值, 处理组样品再减去空白对照样品的含量.实验前进行空白基质加标, 方法回收率为82.6%~112.3%, 内标回收率为86.5%~107.2%, 实验过程中样品加标回收率为78.3%~109.1%, 仪器检出限(3倍信噪比计)为0.13~0.51 μg·L-1, 方法检出限定义为平行空白样品中DBP和DEHP的均值加上3倍标准偏差, 分别为0.21 μg·kg-1和0.39 μg·kg-1.
1.4 土壤DNA提取和序列拼装土壤宏基因组DNA的提取和测序委托上海美吉生物医药科技有限公司, 采用QIAamp DNA试剂盒(Qiagen, 德国)提取DNA, 随后利用TBS-380微型荧光计(Turner biosystems, 德国)、NanoDrop 2000分光光度计(Thermo fisher, 美国)和1%的琼脂糖凝胶电泳检测DNA浓度和质量.检测合格的DNA样品使用NovaSeq平台(Illumina, 美国)进行PE150双端测序, 每个样品获得不少于12 GB的数据量.
使用FASTP[23](https://github.com/OpenGene/fastp)对reads的3'端和5'端进行质量剪切, 去除剪切后长度 < 50 bp、平均碱基质量值< 20以及含N碱基的reads, 获得Clean reads.使用MEGAHIT V1.1.2[24](https://github.com/voutcn/megahit)对Clean reads进行拼接和组装, 然后根据长度为k的核苷酸序列(kmer值设定为47~97)之间的重叠关系, 构建德布鲁因图, 获取重叠群(contigs), 把reads映射到contigs, 根据reads之间的双末端关系把contigs拼接成scaffolds.将scaffolds打断成新的contigs, 优选长度≥300 bp的contigs作为最终的组装结果.使用MetaGene[25](http://metagene.cb.k.u-tokyo.ac.jp/)对contigs进行开放阅读框预测, 将长度≥100bp的基因序列翻译为氨基酸序列.采用CD-HIT V4.6.1[26](http://www.bioinformatics.org/cd-hit/)进行基因聚类, 每类取最长的基因作为代表序列, 构建非冗余基因集.使用SOAPaligner V2.21[27](http://soap.genomics.org.cn/), 将每个样品的Clean reads与非冗余基因集进行比对(相似度95%以上), 统计基因在对应样品中的丰度信息.使用BLASTP(http://blast.ncbi.nlm.nih.gov/Blast.cgi, evalue≤1×10-5)将非冗余基因集序列分别与NCBI-NR和KEGG数据库进行比对, 分别获得物种和功能注释.测序数据已提交至国家微生物科技数据中心.
1.5 数据分析利用Origin 2018软件对PAEs测定数据进行一级动力学拟合, 方程如下:
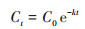
|
(1) |
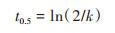
|
(2) |
式中, Ct为时间t时土壤中PAEs残留量(mg·kg-1), C0为PAEs初始含量(mg·kg-1), k为降解速率常数, t为降解时间(d), t0.5为降解半衰期(d).
使用R V4.1.2软件的“vegan”包计算两两样本间Bray-Curtis距离, 进行UPGMA(unweighted pair group method with arithmetic mean)聚类分析和非度量多维尺度(nonmetric multidimensional scaling, NMDS)分析, 并利用ANOSIM相似性分析(analysis of similarities)评估样本间微生物群落组成的差异.Origin软件用于单因素方差分析、普通最小二乘回归(ordinary least squares, OLS)、Mann-Whitney U和Kruskal-Wallis非参数检验等, 显著性水平P < 0.05和P < 0.01作为显著性和极显著性判断标准.群落中关键物种/功能的筛选基于Python软件的Networkx工具包, 为了避免虚假相关性, 选择属水平上相对丰度前50的物种/功能进行网络分析, 使用Spearman相关性来评估物种/功能之间的关联(|R|≥0.5, P < 0.05), 并使用错误发现率对P值多重检验校正, 以网络节点的度中心性(degree centrality)、紧密中心性(closeness centrality)和介数中心性(betweenness centrality)这3个指标的总和作为定义关键物种/功能的阈值[28].
2 结果与讨论 2.1 PAEs在土壤中的降解特征PAEs在土壤中的降解特征如表 2所示, 紫色土中DEHP和DBP降解动态均符合一级动力学降解模型, 半衰期介于17.0~38.2 d之间, 60 d基本达到平台期, 90 d时仍未完全降解.5 mg·kg-1 和20 mg·kg-1处理组中, DBP半衰期均快于DEHP, 60 d后DEHP残留量显著高于DBP.10 mg·kg-1处理组中, 二者残留量没有显著差异, DBP半衰期慢于DEHP.另一方面, DBP的降解速率会随初始含量的增加而降低.而DEHP处理后, 初始含量为10 mg·kg-1自然降解速率最快, 20 mg·kg-1自然降解速率最慢.
|
|
表 2 土壤中DBP和DEHP含量和降解参数1) Table 2 Content and degradation parameters of DBP和DEHP in soils after different treatments |
有研究表明, 20 mg·kg-1 DBP在潮土和红壤中自然降解迅速, 其半衰期分别为0.3 d和1.2 d[13], 远远快于本研究.40 mg·kg-1的DBP的半衰期也小于5 d[19].此外, 红壤中DBP降解速率会随初始含量的增加而降低, 这与本研究的结果一致, 在潮土中相反[13].由于DEHP的高疏水性, 其自然降解速率通常较慢, 5~40 mg·kg-1的DEHP在培养35 d后降解量均不到一半[29], 200 mg·kg-1的DEHP半衰期也超过35 d[30], 本研究中DEHP降解速率同样较慢.由此可推断出, PAEs降解速率的差异可能是由于土壤性质、PAEs类型和剂量的不同, 导致土壤微生物群落发生不同的响应变化.此外, 本研究紫色土中腐殖酸含量高, 会导致PAEs的生物有效性降低[21], 并且土壤本身PAEs残留水平低, 土壤微生物群落未经驯化, 因此生物降解能力相比其它研究较低.
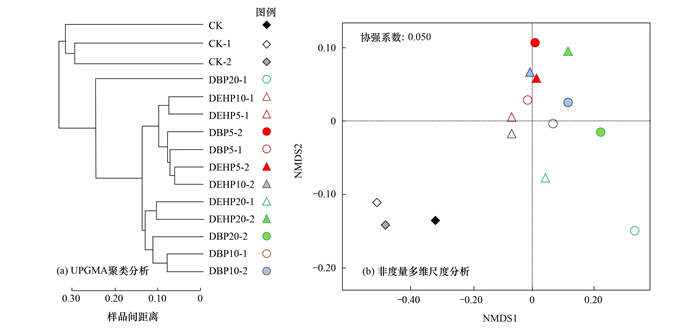
2.2 土壤细菌群落结构对PAEs污染的响应各个样品质控后获得了5.2×107 ~ 6.8×107条clean reads, 拼装获得3.4×105 ~ 5.8×105条contigs, 总长度为2.3×108 ~ 3.8×108 bp, 对contigs进行评估, N50值介于526~862 bp, 表明contigs组装结果较好.根据种水平物种丰度进行UPGMA聚类分析和NMDS分析(图 1).UPGMA聚类分析发现, PAEs残留量越相近的样品中, 群落结构相似度越高, 表明PAEs对土壤细菌群落的影响与其残留量有关.NMDS排序的胁强系数为0.05, 排序结果较好.未污染空白样本位于图左侧, PAEs污染处理样本多位于图右侧, 两种PAEs处理的样本间距离较近, 群落相似度较高, 但第7 d和15 d样本的分布相对分离(ANOSIM检验:R = 0.64, P < 0.001), 表明PAEs残留量对土壤细菌群落结构差异的贡献大于其类型.
|
图 1 基于种分类水平的UPGMA聚类分析和NMDS分析 Fig. 1 UPGMA clustering analysis and NMDS analysis of bacterial community structure at species levels |
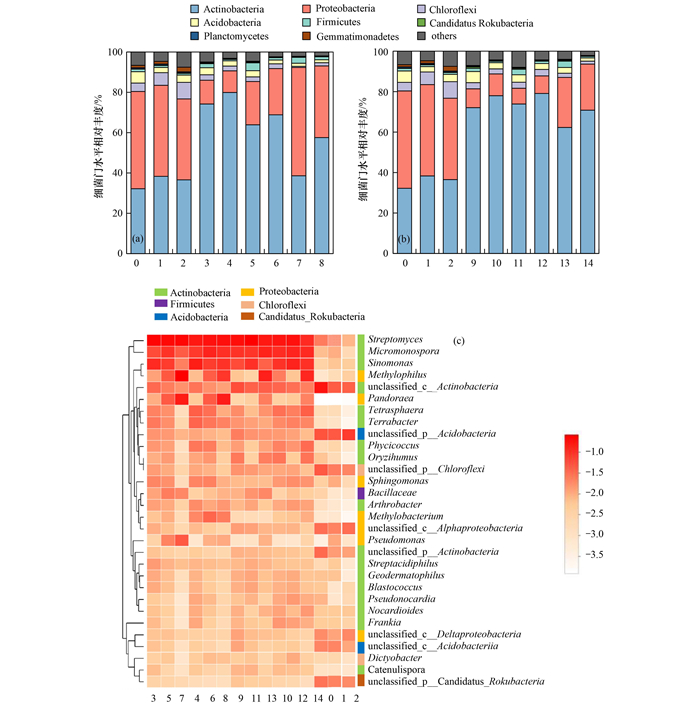
物种注释后发现, 门水平上的细菌主要包括放线菌门(Actinobacteria)、变形菌门(Proteobacteria)、绿弯菌门(Chloroflexi)、酸杆菌门(Acidobacteria)和厚壁菌门(Firmicutes)等.初始土壤(CK)中, 变形菌门(48.2%)为优势门, 其次为放线菌门(32.2%).DBP染毒第7 d时[图 2(a)], 5、10和20 mg·kg-1处理后放线菌门相对丰度分别提升到74.2%、63.9%和39.0%, 第15 d时继续增加到79.9%、68.8%和57.6%, 成为优势门, 且相对丰度随DBP残留量的减少而增加.同样, 5、10和20 mg·kg-1的DEHP染毒7 d后[图 2(b)], 放线菌门相对丰度依次提高到72.1%、73.9%和62.3%, 第15 d增加到78.0%、79.1%和70.9%, 且相对丰度大小与其降解速率的高低顺序一致.因此, 放线菌门可能在PAEs自然降解中扮演重要角色.相对丰度前30的细菌属中[图 2(c)], 污染样品中放线菌门的Streptomyces、Micromonospora和Sinomonas以及变形菌门的Methylophilus和Pandoraea等细菌相对丰度均高于未污染样品.由此可见, DBP和DEHP污染改变了紫色土土著细菌群落结构, Streptomyces和Pandoraea等细菌随PAEs残留量的减少在群落中的优势地位不断提高, 具有指示PAEs污染程度的潜力.Streptomyces等放线菌被报道在DBP[31]和DEHP[17]污染环境中具有显著生存优势, 这类细菌能降解纤维素和几丁质等高分子聚合物, 为土壤提供养分, 因此在极端环境下保持较高的活性和DNA修复能力.变形菌门是细菌域数量和种类最大的一个门, 在不同环境中均能占据生态位[32].Methylophilus可以利用含碳有机物作为碳源生长繁殖, 通过去甲基化减少PAEs侧链长度[33].Pandoraea属是一种革兰氏阴性菌, 可以降解农药和苯系物等有机物[34~37].因此, 这些物种在土壤细菌群落去除PAEs过程中扮演重要角色, 可能直接参与PAEs降解或者调节物种互作网络从而影响群落构建, 进而导致PAEs降解效率的差异.
|
(a)DBP污染后, (b)DEHP污染后, (c)属水平细菌群落结果;0.CK, 1.CK-1, 2.CK-2, 3.DBP5-1, 4.DBP5-2, 5.DBP10-1, 6.DBP10-2, 7.DBP20-1, 8.DBP20-2, 9.DEHP5-1, 10.DEHP5-2, 11.DEHP10-1, 12.DEHP10-2, 13.DEHP20-1, 14.DEHP20-2;热图按相对丰度对数值显示颜色 图 2 土壤细菌群落结构柱状图和热图 Fig. 2 Histogram and heatmap of soil bacterial community structure |
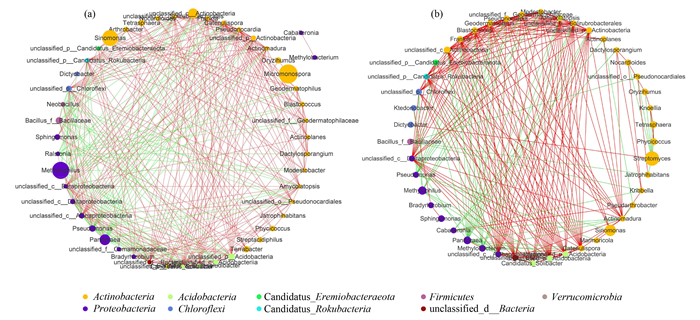
基于样本间群落结构相似度, 分为第7 d和15 d两组进行网络分析, 以确定PAEs不同降解阶段的关键物种.如图 3和表 3所示, 第7 d时, Pandoraea、Pseudomonas、unclassified_c__Alphaproteobacteria、unclassified_d__Bacteria、unclassified_p__Acidobacteria和unclassified_p__Chloroflexi这6个属位于共现网络的中心位置, 其综合得分最高, 同时拥有高度中心性(0.48)、高紧密中心性(0.53)和低介数中心性(0.02), 表明这些物种通过与其它物种之间紧密的合作关系或激烈的竞争关系, 形成复杂的互作网络, 这些物种的缺失可能会造成整个群落互作网络稳定性的下降.同样地, Pandoraea、Amycolatopsis、Catenulispora、Frankia、unclassified_c__Acidobacteriia、unclassified_c__Actinobacteria和unclassified_o__Solirubrobacterales这7个属被确定为第15 d时细菌群落的关键物种.
|
(a)第7 d样品优势细菌互作网络, (b)第15 d样品优势细菌互作网络;节点颜色反映门分类水平, 节点大小反映物种丰度, 红色连线表示节点之间正相关, 绿色连线表示负相关, 线越粗, 表示相关性越高 图 3 属水平优势物种共现网络 Fig. 3 Co-occurrence network at the genus level |
|
|
表 3 物种共现网络特征参数1) Table 3 Characteristic parameters of co-occurrence network |
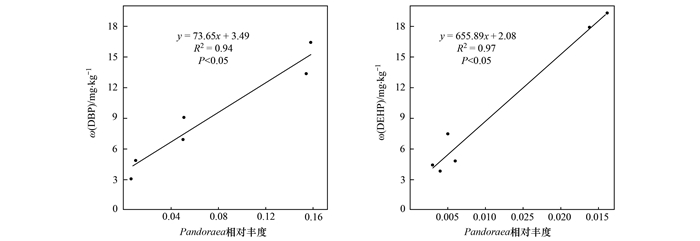
Pandoraea是两组样本中共有的关键物种.如图 3所示, 7 d时, Pandoraea与Methylophilus和Pseudomonas等4个物种有显著的正相关关系, 与其余19个节点显著负相关, 在15 d时, 仅与Methylobacterium显著正相关, 与其它19个属显著负相关, 表明Pandoraea与其他物种主要存在竞争关系, 从而维持互作网络的稳定[38].OLS回归模型进一步表明(图 4), Pandoraea的相对丰度与DBP和DEHP均呈现显著的线性正相关关系(R2 > 0.80, P < 0.05), 暗示Pandoraea可用于预测紫色土中DBP或DEHP污染趋势.关键类群作为污染指示剂正在引起国内外学者的关注[39, 40], 但少有研究报道PAEs污染土壤核心菌及其生态作用.Kong等[19]认为放线菌门的Arthrobacter和Sphingomonas等是40 mg·kg-1 DBP污染农田中细菌群落的关键菌, 可以用于预测细菌群落对DBP污染的响应.不同的是, 本研究中Pandoraea来自变形菌门, 并且可以指示两种污染物.尽管Pandoraea不是丰度最高的类群, 但占据网络枢纽位置, 其存在或灭绝均会影响群落功能[41~43].有研究表明, 污染胁迫下, 核心菌会分泌抗生素、毒素或代谢物等与中间类群或有效类群强烈相互作用, 调节土著细菌群落结构使其适应环境胁迫[41].DBP或DEHP的添加或降解, 使得Pandoraea的丰度相应地增加或减少, 导致网络中与之协作的物种发生类似的变化, 与之互斥的物种得以灭亡或繁殖, 从而维持网络的稳定和PAEs降解等生态系统功能.
|
图 4 普通最小二乘回归 Fig. 4 Ordinary least squares regression |
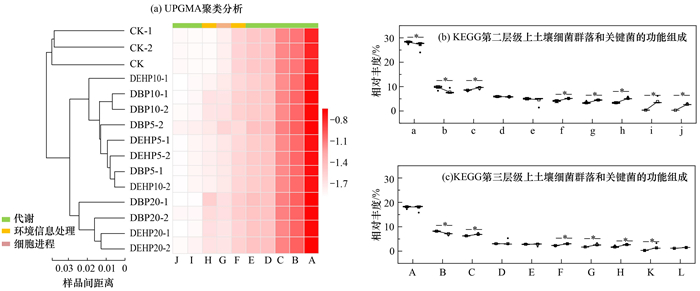
将基因序列与KEGG数据库进行比对注释, 并分类为不同层级的代谢通路.对第三层级上代谢通路的基因丰度进行UPGMA聚类分析[图 5(a)], 结果显示, 污染样品间排序距离短, 表明不同程度PAEs污染后的土壤细菌群落功能较为相似, 群落中大部分基因被注释到代谢途径(16.1%~18.6%)、次级代谢物的生物合成(7.3%~8.3%)和不同环境的微生物代谢(5.6%~6.4%)等通路上.
|
(a)热图按相对丰度的对数值显示颜色;(b)和(c)同一代谢途径对应的两个箱体表示两种分类水平, 左箱体表示所有样本细菌群落水平上某一代谢途径相对丰度, 右箱体表示所有样本Pandoraea属细菌某一代谢途径相对丰度;*表示在P < 0.05水平上显著;A.代谢途径, B.次级代谢物的生物合成, C.不同环境的微生物代谢, D.碳代谢, E.氨基酸的生物合成, F.ABC转运, G.群体感应, H.双组分系统, I.丙酮酸代谢, J.嘌呤代谢, K.苯甲酸代谢, L.乙醛酸及二羧酸代谢;a.全局与总览图, b.碳水化合物代谢, c.氨基酸代谢, d.能量代谢, e.辅助因子和维他命代谢, f.膜运输, g.信号传导, h.原核生物细胞群落, i.异源物质生物降解和代谢, j.其它氨基酸的代谢;所展示功能的相对丰度超过1% 图 5 土壤细菌群落功能组成的UPGMA聚类分析和箱线图 Fig. 5 UPGMA clustering analysis and boxplot of bacterial community function |
此外, 在第二层级上[图 5(b)], Pandoraea属所包含的膜运输、信号传导、异源物质生物降解和代谢以及其它氨基酸代谢等代谢通路上的相对丰度显著高于土壤细菌群落整体水平, 第三层级上[图 5(c)], ABC转运、群体感应、双组分系统和苯甲酸代谢等代谢通路相对丰度也显著高于群落整体水平.ABC转运可以将糖、氨基酸和离子等通过细胞膜转运至活细胞, 促进细胞对营养的吸收;双组分系统是细菌信号传导系统;群体感应是原核生物细胞间交流的重要机制;异源物质生物降解和代谢途径中, 包含众多催化含苯环有机物降解途径的酶编码基因, 如多环芳烃降解和苯甲酸降解等, 均是PAEs生物降解的重要途径[17].
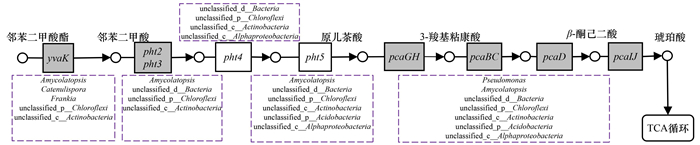
通过构建包含yvaK、pht和pca家族的基因集发现, Pandoraea含有PAEs降解的多个酶基因(图 6), 例如编码羧酸酯酶的yvaK基因, 编码4, 5-邻苯二甲酸双加氧酶的pht2和pht3基因, 以及催化原儿茶酸开环的pca家族基因簇等, 但不含pht4和pht5基因, 表明Pandoraea不能独自将PAEs彻底矿化, 与之协同合作的Pseudomonas等细菌只能催化原儿茶酸开环.然而, 与Pandoraea存在显著竞争关系的unclassified_p__Chloroflexi和unclassified_c__Actinobacteria等其它关键微生物, 拥有催化PAEs完全矿化的代谢途径.
|
圆表示PAEs及其中间产物, 箭头表示降解方向, 灰色和白色填充方框分别表示Pandoraea中含有的和不含的催化PAEs降解的相关酶基因, 虚线方框表示含有对应酶基因的其它关键微生物;改自KEGG数据库 图 6 PAEs降解途径关键节点中涉及的酶编码基因及其来源 Fig. 6 Enzyme coding genes involved in PAEs degradation pathways and their sources |
先前研究只简单关注PAEs污染下群落水平上碳氮代谢、糖代谢和水解酶等功能基因的响应[15, 44, 45], 未深究其微生物来源及其生态作用, 本研究利用宏基因组揭示了Pandoraea作为细菌群落核心物种的作用.在PAEs降解前期, Pandoraea大量繁殖将其转化为中间代谢产物邻苯二甲酸后, 其丰度随PAEs的降解减少, 但仍可以通过分泌、扩散和交换信号分子来刺激群落中部分基因表达、物质运输和细胞通讯来调节群落密度, 并开始促进其它物种利用邻苯二甲酸来生长繁殖;当邻苯二甲酸降解为原儿茶酸后, 也可以反馈供Pandoraea再次利用, 从而将PAEs彻底矿化.由此可见, Pandoraea可能主导着紫色土PAEs上下游的生物降解, 并且通过细胞通讯来提高菌群的完全降解能力, 以此抵抗不利环境并维持群落结构的稳定, 保护土壤健康.由此可见, 在今后PAEs治理时, 可以在人工构建PAEs高效降解菌群时考虑对Pandoraea的定向分离培养, 从而提高降解菌群的效率, 为新污染物的治理修复提供技术支撑.
3 结论(1)紫色土中DBP和DEHP降解动态均符合一级动力学降解模型, 不同单体物质结构及其初始含量的差异均影响其生物降解效率.
(2)宏基因组测序分析发现, 第7 d和15 d土壤样品间细菌群落结构存在显著差异, PAEs对土壤细菌群落的影响主要与其残留量有关.放线菌门在污染土壤中为优势门, Streptomyces等细菌为优势属.放线菌门相对丰度随时间不断提升, 不同污染条件下DBP或DEHP半衰期越小, 放线菌门相对丰度越高, 因此, 放线菌门在PAEs自然降解中扮演重要角色.
(3)紫色土中不同PAEs污染条件下共有的关键菌是Pandoraea属, 与大多优势物种存在竞争关系, 具有指示DBP和DEHP污染程度的潜力, 在群落中负责苯甲酸降解、群体感应、ABC转运和双组分系统等功能, 可以促进细菌间信息交流和其它PAEs降解菌的繁殖, 从而将PAEs彻底矿化并维持群落互作网络的稳定.
| [1] | Das M T, Kumar S S, Ghosh P, et al. Remediation strategies for mitigation of phthalate pollution: Challenges and future perspectives[J]. Journal of Hazardous Materials, 2021, 409. DOI:10.1016/j.jhazmat.2020.124496 |
| [2] | De-la-Torre G E, Dioses-Salinas D C, Dobaradaran S, et al. Release of phthalate esters (PAEs) and microplastics (MPs) from face masks and gloves during the COVID-19 pandemic[J]. Environmental Research, 2022, 215. DOI:10.1016/j.envres.2022.114337 |
| [3] | Li Y Y, Zheng N, Li Y, et al. Exposure of childbearing-aged female to phthalates through the use of personal care products in China: An assessment of absorption via dermal and its risk characterization[J]. Science of the Total Environment, 2022, 807. DOI:10.1016/j.scitotenv.2021.150980 |
| [4] | Lü H X, Mo C H, Zhao H M, et al. Soil contamination and sources of phthalates and its health risk in China: a review[J]. Environmental Research, 2018, 164: 417-429. DOI:10.1016/j.envres.2018.03.013 |
| [5] | Wang Y, Wang F, Xiang L L, et al. Risk assessment of agricultural plastic films based on release kinetics of phthalate acid esters[J]. Environmental Science & Technology, 2021, 55(6): 3676-3685. |
| [6] | Wang J, Chen G C, Christie P, et al. Occurrence and risk assessment of phthalate esters (PAEs) in vegetables and soils of suburban plastic film greenhouses[J]. Science of the Total Environment, 2015, 523: 129-137. DOI:10.1016/j.scitotenv.2015.02.101 |
| [7] | Zeng L J, Huang Y H, Chen X T, et al. Prevalent phthalates in air-soil-vegetable systems of plastic greenhouses in a subtropical city and health risk assessments[J]. Science of the Total Environment, 2020, 743. DOI:10.1016/j.scitotenv.2020.140755 |
| [8] | Ma J, Lu Y G, Teng Y, et al. Occurrence and health risk assessment of phthalate esters in tobacco and soils in tobacco-producing areas of Guizhou province, southwest China[J]. Chemosphere, 2022, 303. DOI:10.1016/j.chemosphere.2022.135193 |
| [9] | Li Y T, Wang J, Yang S, et al. Occurrence, health risks and soil-air exchange of phthalate acid esters: A case study in plastic film greenhouses of Chongqing, China[J]. Chemosphere, 2021, 268. DOI:10.1016/j.chemosphere.2020.128821 |
| [10] | Delgado-Baquerizo M, Maestre F T, Reich P B, et al. Microbial diversity drives multifunctionality in terrestrial ecosystems[J]. Nature Communications, 2016, 7(1). DOI:10.1038/ncomms10541 |
| [11] | Fierer N. Embracing the unknown: Disentangling the complexities of the soil microbiome[J]. Nature Reviews Microbiology, 2017, 15(10): 579-590. DOI:10.1038/nrmicro.2017.87 |
| [12] | Tezel U, Pavlostathis S G. Quaternary ammonium disinfectants: microbial adaptation, degradation and ecology[J]. Current Opinion in Biotechnology, 2015, 33: 296-304. DOI:10.1016/j.copbio.2015.03.018 |
| [13] | Cheng J J, Liu Y N, Wan Q, et al. Degradation of dibutyl phthalate in two contrasting agricultural soils and its long-term effects on soil microbial community[J]. Science of the Total Environment, 2018, 640-641: 821-829. |
| [14] | Wang Z G, Liu S, Xu W H, et al. The microbiome and functions of black soils are altered by dibutyl phthalate contamination[J]. Applied Soil Ecology, 2016, 99: 51-61. DOI:10.1016/j.apsoil.2015.11.024 |
| [15] | Zhu F X, Yan Y Y, Doyle E, et al. Microplastics altered soil microbiome and nitrogen cycling: The role of phthalate plasticizer[J]. Journal of Hazardous Materials, 2022, 427. DOI:10.1016/j.jhazmat.2021.127944 |
| [16] | Wang L, Wang L H, Chang Q, et al. Effects of di-(2-ethylhexyl) phthalate on microbial biomass carbon and microbial community structural diversity in a Mollisol[J]. European Journal of Soil Science, 2017, 68(6): 897-908. DOI:10.1111/ejss.12471 |
| [17] | Zhu F X, Doyle E, Zhu C Y, et al. Metagenomic analysis exploring microbial assemblages and functional genes potentially involved in di (2-ethylhexyl) phthalate degradation in soil[J]. Science of the Total Environment, 2020, 715. DOI:10.1016/j.scitotenv.2020.137037 |
| [18] | Chen Z W, Zhong X, Zheng M Y, et al. Indicator species drive the key ecological functions of microbiota in a river impacted by acid mine drainage generated by rare earth elements mining in South China[J]. Environmental Microbiology, 2022, 24(2): 919-937. DOI:10.1111/1462-2920.15501 |
| [19] | Kong X, Bai Z B, Jin T, et al. Arthrobacter is a universal responder to di-n-butyl phthalate (DBP) contamination in soils from various geographical locations[J]. Journal of Hazardous Materials, 2022, 422. DOI:10.1016/j.jhazmat.2021.126914 |
| [20] | 王智慧. 不同pH紫色土中硝化作用及硝化微生物宏基因组研究[D]. 重庆: 西南大学, 2017. |
| [21] | Bai H C, Lu P L, Zhang L L, et al. New insight into the contribution of humic substance to degradation of phthalate acid esters in soil: Link batch experiment to model study[J]. Journal of Environmental Chemical Engineering, 2022, 10(6). DOI:10.1016/j.jece.2022.108731 |
| [22] | GB/T 39234-2020, 土壤中邻苯二甲酸酯测定气相色谱-质谱法[S]. |
| [23] | Chen S F, Zhou Y Q, Chen Y R, et al. fastp: an ultra-fast all-in-one FASTQ preprocessor[J]. Bioinformatics, 2018, 34(17): i884-i890. DOI:10.1093/bioinformatics/bty560 |
| [24] | Li D H, Liu C M, Luo R B, et al. MEGAHIT: an ultra-fast single-node solution for large and complex metagenomics assembly via succinct de Bruijn graph[J]. Bioinformatics, 2015, 31(10): 1674-1676. DOI:10.1093/bioinformatics/btv033 |
| [25] | Noguchi H, Park J, Takagi T. MetaGene: prokaryotic gene finding from environmental genome shotgun sequences[J]. Nucleic Acids Research, 2006, 34(19): 5623-5630. DOI:10.1093/nar/gkl723 |
| [26] | Fu L M, Niu B F, Zhu Z W, et al. CD-HIT: accelerated for clustering the next-generation sequencing data[J]. Bioinformatics, 2012, 28(23): 3150-352. DOI:10.1093/bioinformatics/bts565 |
| [27] | Li R Q, Li Y R, Kristiansen K, et al. SOAP: short oligonucleotide alignment program[J]. Bioinformatics, 2008, 24(5): 713-714. DOI:10.1093/bioinformatics/btn025 |
| [28] | Banerjee S, Schlaeppi K, Van Der Heijden M G A. Keystone taxa as drivers of microbiome structure and functioning[J]. Nature Reviews Microbiology, 2018, 16(9): 567-576. DOI:10.1038/s41579-018-0024-1 |
| [29] |
王蕾, 枉靖宜, 张颖, 等. 不同添加浓度DEHP在黑土中的降解特点及对酶活性的影响[J]. 土壤通报, 2022, 53(1): 144-151. Wang L, Wang J Y, Zhang Y, et al. Degradation characteristics of DEHP added into mollisols at different concentrations and its effect on enzyme activities[J]. Chinese Journal of Soil Science, 2022, 53(1): 144-151. |
| [30] | Zhu F X, Zhu C Y, Doyle E, et al. Fate of di (2-ethylhexyl) phthalate in different soils and associated bacterial community changes[J]. Science of the Total Environment, 2018, 637-638: 460-469. DOI:10.1016/j.scitotenv.2018.05.055 |
| [31] | Xu W H, You Y M, Wang Z G, et al. Dibutyl phthalate alters the metabolic pathways of microbes in black soils[J]. Scientific Reports, 2018, 8(1). DOI:10.1038/s41598-018-21030-8 |
| [32] | Spain A M, Krumholz L R, Elshahed M S. Abundance, composition, diversity and novelty of soil Proteobacteria[J]. The ISME Journal, 2009, 3(8): 992-1000. DOI:10.1038/ismej.2009.43 |
| [33] | Kumar V, Maitra S S. Biodegradation of endocrine disruptor dibutyl phthalate (DBP) by a newly isolated Methylobacillus sp. V29b and the DBP degradation pathway[J]. 3 Biotech, 2016, 6(2). DOI:10.1007/s13205-016-0524-5 |
| [34] | Peeters C, De Canck E, Cnockaert M, et al. Comparative genomics of Pandoraea, a genus enriched in xenobiotic biodegradation and metabolism[J]. Frontiers in Microbiology, 2019, 10. DOI:10.3389/fmicb.2019.02556 |
| [35] | Okeke B C, Siddique T, Arbestain M C, et al. Biodegradation of γ-hexachlorocyclohexane (Lindane) and α-hexachlorocyclohexane in water and a soil slurry by a Pandoraea species[J]. Journal of Agricultural and Food Chemistry, 2002, 50(9): 2548-2555. DOI:10.1021/jf011422a |
| [36] | Wang X Q, Wang Q L, Li S J, et al. Degradation pathway and kinetic analysis for p-xylene removal by a novel Pandoraea sp. strain WL1 and its application in a biotrickling filter[J]. Journal of Hazardous Materials, 2015, 288: 17-24. DOI:10.1016/j.jhazmat.2015.02.019 |
| [37] | Yang J, Guo C L, Liu S S, et al. Characterization of a di-n-butyl phthalate-degrading bacterial consortium and its application in contaminated soil[J]. Environmental Science and Pollution Research, 2018, 25(18): 17645-17653. DOI:10.1007/s11356-018-1862-0 |
| [38] | Palmer J D, Foster K R. Bacterial species rarely work together[J]. Science, 2022, 376(6593): 581-582. DOI:10.1126/science.abn5093 |
| [39] | Zhang H J, Yang L, Li Y, et al. Pollution gradients shape the co-occurrence networks and interactions of sedimentary bacterial communities in Taihu Lake, a shallow eutrophic lake[J]. Journal of Environmental Management, 2022, 305. DOI:10.1016/j.jenvman.2021.114380 |
| [40] | Veloso S, Amouroux D, Lanceleur L, et al. Keystone microbial taxa organize micropollutant-related modules shaping the microbial community structure in estuarine sediments[J]. Journal of Hazardous Materials, 2023, 448. DOI:10.1016/j.jhazmat.2023.130858 |
| [41] | Zafra G, Taylor T D, Absalón A E, et al. Comparative metagenomic analysis of PAH degradation in soil by a mixed microbial consortium[J]. Journal of Hazardous Materials, 2016, 318: 702-710. DOI:10.1016/j.jhazmat.2016.07.060 |
| [42] | Jiao S, Lu Y H, Wei G H. Soil multitrophic network complexity enhances the link between biodiversity and multifunctionality in agricultural systems[J]. Global Change Biology, 2022, 28(1): 140-153. DOI:10.1111/gcb.15917 |
| [43] | Hu W G, Ran J Z, Dong L W, et al. Aridity-driven shift in biodiversity-soil multifunctionality relationships[J]. Nature Communications, 2021, 12(1). DOI:10.1038/s41467-021-25641-0 |
| [44] | Chen W J, Wang Z G, Xu W H, et al. Dibutyl phthalate contamination accelerates the uptake and metabolism of sugars by microbes in black soil[J]. Environmental Pollution, 2020, 262. DOI:10.1016/j.envpol.2020.114332 |
| [45] | You Y M, Wang Z G, Xu W H, et al. Phthalic acid esters disturbed the genetic information processing and improved the carbon metabolism in black soils[J]. Science of the Total Environment, 2019, 653: 212-222. DOI:10.1016/j.scitotenv.2018.10.355 |
 2024, Vol. 45
2024, Vol. 45


