2. 佐治亚州南方大学生物系, 佐治亚州斯泰茨伯勒, 美国
2. Department of Biology, Georgia Southern University, Statesboro, Georgia, USA
土壤作为森林生态系统生命活动的主要场所, 除了为地上植被的生长提供养分外, 也孕育着数量庞大, 种类多样的微生物.土壤微生物在调节生物地球化学循环和维持生态系统功能等方面发挥重要作用, 是森林生态系统稳定性与可持续性的驱动者[1].大多数关于微生物群落的研究多集中在30 cm以上的表层土壤中[2].据报道, 土壤中30%~35%的微生物分布在25 cm以下的土壤层中[3], 对土壤养分和有机碳循环发挥重要作用[4, 5], 驱动土壤碳库的结构及碳源汇过程[6].因此, 了解土壤剖面不同深度下微生物群落的组成和特征, 有助于全面理解土壤地下生态系统过程.
微生物对土壤环境变化敏感[7], 例如pH、含水量[8]和土壤养分含量的改变等均会影响微生物群落的结构和多样性[9].由于森林类型的多样性、结构的复杂性以及森林对干扰和变化环境响应的不确定性, 对森林土壤生态系统中微生物群落维持机制的认识还十分有限.土壤剖面深度被视作土壤性质的生态过滤器[10], 沿剖面形成了异质性的梯度环境.土壤有机碳在不同土层的分布受淋溶作用和植物根系的共同影响, 深层土壤的活性碳含量显著低于表层土壤[11], 而深层土壤储存了大量有机碳[12, 13], 比表层土壤碳周转时间更长[14], 因此深层土壤丰富的碳含量对微生物群落构建的作用不容忽视[15].深层土壤中的微生物被认为在土壤发育和生物地球化学循环中起着重要的作用, 因为它们接近母质, 对土壤形成的影响更强[16].有机碳库和碳储存量随土层深度发生变化[17], 几乎所有的土壤因素通常会随着土壤深度的变化而变化[18], 但目前对存在于较深土壤处的微生物群落的特征尚未得到很好的描述, 而这些群落所表现出的空间特征仍然知之甚少[19].
华北落叶松(Larix principis-rupprechtii)是我国北方温带地区亚高山森林类型的重要树种之一, 其天然林在华北地区广泛分布.芦芽山天然次生华北落叶松林是华北地区保存较好的森林生态系统.本研究采集华北落叶松林土壤剖面不同深度的土壤样品, 应用Illumina MiSeq测序技术分析细菌群落分布特征, 通过了解土壤剖面细菌群落丰度、多样性和组成的变化规律及其驱动因子, 以期阐明森林深层土壤微生物群落的构建机制.
1 材料与方法 1.1 研究区概况与样品采集研究地位于山西省忻州市宁武县境内芦芽山国家级自然保护区, 样地设置于“山西亚高山草地生态系统教育部野外观测研究站”(111°54′39″E, 38°44′11″N)内的华北落叶松林地, 平均海拔2 294 m, 属暖温带半湿润山区气候.于2021年8月, 在1 hm2标准样地(100 m×100 m)中选取4个采样点挖掘土壤剖面, 按腐殖质(P0)、0~10(P1)、10~20(P2)、20~40(P3)和40~80 cm(P4)分层采集土壤样品.剔除可见的石头和植物残体等杂物后, 将样品分为2份装于无菌自封袋后带回实验室.一份土壤保存于4℃, 用于土壤理化指标的测定, 另一份放置于-20℃冰箱中, 用于微生物高通量测序.
1.2 土壤理化性质测定样品的全碳(TC)、全氮(TN)和全硫(TS)通过元素分析仪(Elementar Vario MACRP, 德国)测定; 土壤105℃烘干, 枯落物、腐殖质经65℃烘干至恒重, 测定其含水率; pH用酸度计(HANNA, 意大利)测定(土壤与水的比例1∶2.5, 枯落物与水的比例为1∶20, 质量浓度).采用间断元素分析仪(CleverChem 380, 德国) 测定土壤铵态氮(NH4+-N)和硝态氮(NO3--N).全磷(TP)含量用全自动间断化学分析仪(CleverChem Anna, DeChem-Tech, 德国)测定.
1.3 土壤微生物DNA提取、PCR扩增和测序称取0.25 g土壤样品, 使用E.Z.N.A.® soil DNA kit (Omega Bio-tek, Norcross, GA, U.S.)试剂盒, 按说明书进行微生物群落总DNA抽提.PCR引物选择带Barcode的特异引物515F (5′-GTGCCAG CMGCCGCGGTAA-3′)和806R(5′-GGACTACHVG GGTWTCTAAT-3′), 对土壤细菌16S rRNA V3-V4区对应的序列进行PCR扩增.将同一样本的PCR产物混合后, 使用2%琼脂糖凝胶回收PCR产物, 利用AxyPrep DNA Gel Extraction Kit (Axygen Biosciences, Union City, CA, USA) 进行回收产物纯化.利用Illumina公司的MiSeq PE 300平台对扩增产物进行测序(上海美吉生物医药科技有限公司).
1.4 数据分析使用UPARSE软件, 根据97%相似度对序列进行OTU聚类并剔除嵌合体.利用RDP classifier对每条序列进行物种分类注释, 比对Silva 16S rRNA数据库(version 138), 设置比对阈值为70%.利用Microsoft Excel对数据进行整理, 利用SPSS 19.0对腐殖质层、不同深度土壤的理化性质及细菌多样性进行单因素方差分析(One-way ANOVA); 采用非度量多维尺度分析(nonmetric multidimensional scaling analysis, NMDS)研究样本间群落组成的相似性或差异性.通过冗余分析(RDA) 分析不同环境因子对群落结构的影响程度; 利用Spearman相关系数衡量土壤性质与群落α多样性以及主要类群(门或纲水平)的相关关系.利用共现性网络分析不同深度土壤细菌群落物种的共存关系, 指标包括节点(node)、边(edge)、平均度(average degree)、模块化(modularity)和平均聚类系数(average clustering coefficient)等, 利用R4.0软件(psych、reshape2包)计算相关数据并导入Gephi 0.9.3实现分子生态网络可视化.最后, 通过Tax4Fun进行细菌群落功能预测.
2 结果与分析 2.1 土壤剖面理化性质华北落叶松林不同深度的土壤理化参数如表 1所示.腐殖质层(P0)的TN、TC和NH4+显著高于其它不同深度土壤层, 且0~80 cm的4个不同深度土层之间无显著差异.土壤TP含量沿垂直深度呈先减后增的趋势, 其中在40~80 cm土层(P4)中含量最多, 达到1.02 g·kg-1, 而在10~20 cm土层(P2)最低, 为0.62 g·kg-1.土壤整体偏酸性, pH沿土层深度呈增加趋势.
|
|
表 1 沿土壤剖面的土壤化学性质比较1) Table 1 Comparison of soil chemical properties along soil profiles |
2.2 土壤剖面细菌群落组成
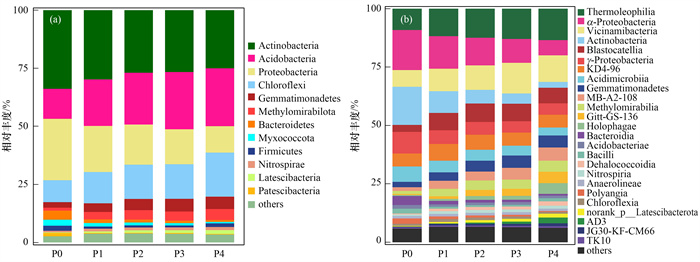
通过优化过滤去除低质量序列后, 共得到土壤细菌群落的有效序列总数为1 891 719, 序列平均长度417 bp.共包括37门, 125纲, 296目, 447科, 771属, 5 718个OTU.将相对丰度大于1%的分类群定义为优势类群, 其余合并为其他(others).土壤剖面细菌群落优势门有12个, 分别为放线菌门(Actinobacteria)、酸杆菌门(Acidobacteria)、变形菌门(Proteobacteria)、绿弯菌门(Chloroflexi)、芽单胞菌门(Gemmatimonadetes)、甲基肌酐菌门(Methylomirabilota)、拟杆菌门(Bacteroidetes)、粘菌门(Myxococcota)、厚壁菌门(Firmicutes)、硝化螺旋菌门(Nitrospirae)、Latescibacteria和髌骨菌门(Patescibacteria), 其中放线菌门(25%~34%)、变形菌门(11%~26%)、酸杆菌门(13%~25%)和绿弯菌门(10%~19%)的丰度占整个土壤剖面细菌群落80%以上[图 1(a)].放线菌门、变形菌门和拟杆菌门随着土壤深度增加相对丰度显著降低, 酸杆菌门和绿弯菌门则呈现相反的变化趋势.土壤剖面细菌群落的优势纲有25个[图 1(b)], 相对丰度较高的前11个分别是属于酸杆菌门的Vicinamibacteria和芽球菌纲(Blastocatellia); 放线菌门的嗜热油菌纲(Thermoleophilia)、放线菌纲(Actinobacteria)、酸微菌纲(Acidimicrobiia)和MB-A2-108; 绿弯菌门的KD4-96和Gitt-GS-136; 变形菌门的γ-变形菌纲(γ-proteobacteria)和α-变形菌纲(α- proteobacteria); 芽单胞菌门的芽单胞菌纲(Gemmatimonadetes).
|
(a)门水平, (b)纲水平 图 1 土壤剖面细菌群落物种组成及相对丰度 Fig. 1 Species composition and relative abundance of bacterial community in soil profile |
对土壤剖面细菌群落中优势类群在门和纲水平上的相对丰度进行Wilcoxon秩和检验.12个优势细菌门中有8个优势门的相对丰度在土壤剖面存在显著差异[图 2(a)].其中变形菌门、绿弯菌门、拟杆菌门、Latescibacteria和粘菌门在不同土层间具有极显著差异(P < 0.01), 放线菌门、酸杆菌门、芽单胞菌门和硝化菌门具有显著差异(P < 0.05).在纲水平, 25个优势纲中有11个优势纲的相对丰度在土壤剖面细菌群落中存在显著差异[图 2(b)].其中α-变形菌纲、放线菌纲、酸微菌纲和拟杆菌纲随土壤深度增加相对丰度逐渐减少, 而MB-A2-108和Gitt-GS-136随深度增加而增加.

|
(a)门水平, (b)纲水平; *为P < 0.05, 显著差异, **为P < 0.01, 极显著差异 图 2 沿土壤剖面细菌群落在门和纲水平的组间差异分析 Fig. 2 Differences analysis of relative abundance of bacteria at phylum and class level along soil profile |
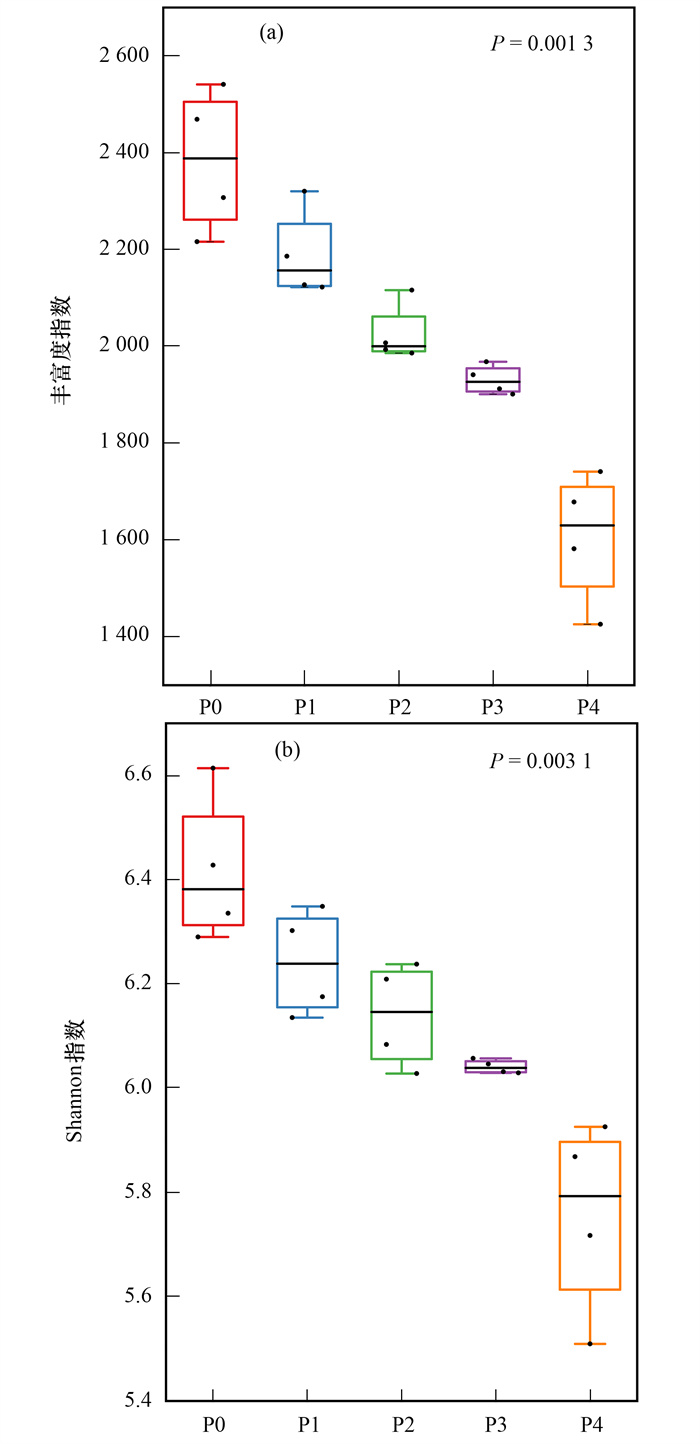
土壤剖面细菌群落的α多样性分析结果显示, 土壤剖面腐殖质层(P0)细菌群落Shannon指数最高, 为6.42; 40~80 cm(P4)层最低, 为5.76.丰富度指数与Shannon指数的变化趋势一致, 各层之间具有显著差异(图 3).随着土层深度的变化, 细菌群落α多样性呈显著单调降低趋势.
|
图 3 土壤剖面细菌群落α多样性分析 Fig. 3 Analysis of α diversity of bacterial community along soil profile |
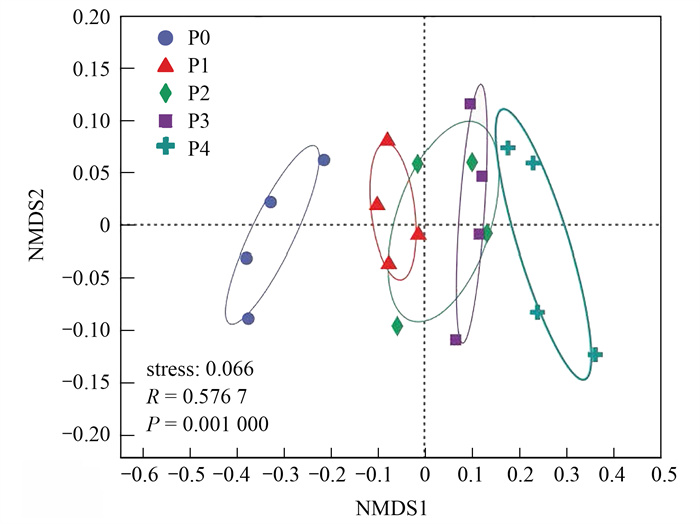
基于Bray-Curtis距离的非度量多维尺度排序(NMDS)分析土壤剖面细菌群落β多样性变化.分析结果的应力函数值(stress)为0.033, 小于0.05, 具有很好的代表性(图 4), 显示土壤深度显著影响了细菌群落结构.Adonis分析(组间差异显著R=0.444 8, P < 0.001)显示, P0和P4层与其他3层之间存在明显差异(图 4).
|
图 4 非度量多维尺度分析土壤细菌群落结构随土层深度的变化 Fig. 4 NMDS analysis of variation in soil bacterial community structure along with depth |
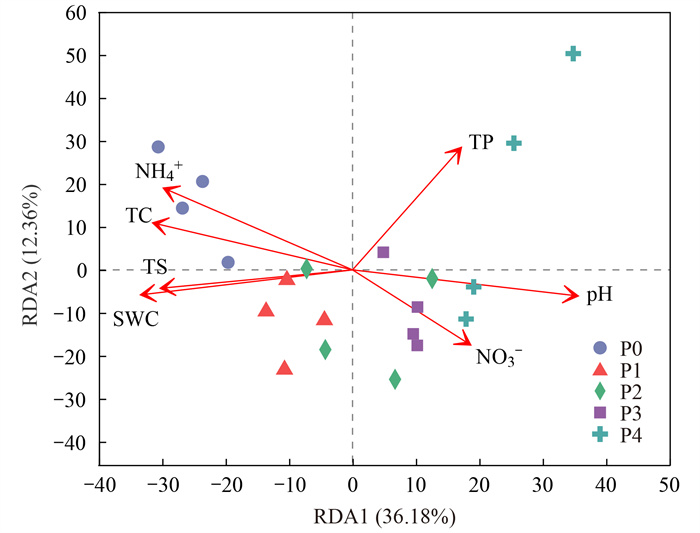
根据方差膨胀因子筛选出VIF小于10的环境因子, 然后利用RDA约束排序分析确定土壤剖面细菌群落空间结构变化的驱动因子.结果显示, 第一排序轴的解释率为36.18%, 第二排序轴的解释率为12.36%, 前两个轴的总解释率达48.54%.土壤NH4+、TC、TS、WCS、pH、NO3-和TP是导致细菌群落结构发生变化的重要因子(图 5), 其中pH对土壤细菌群落具有显著影响.变形菌门、放线菌门、拟杆菌门和粘菌门与pH呈显著负相关, 酸杆菌门、绿弯菌门、芽单胞菌门、甲基肌酐菌门和硝化菌门与pH呈显著正相关; α-变形菌纲、放线菌纲、酸微菌纲、γ-变形菌纲与pH呈显著负相关, 嗜热油菌纲、Vicinamibacteria、芽单胞菌纲和MB-A2-108与pH呈显著正相关(图 6).
|
图 5 土壤剖面细菌群落与生态因子关系的冗余分析 Fig. 5 Redundancy analysis of dominant phyla and ecological factors of bacterial community in soil profile |

|
(a)门水平, Act: Actinobacteria, Aci: Acidobacteria, Pro: Proteobacteria, Chl: Chloroflexi, Gem: Gemmatimonadetes, Met: Methylomirabilota, Bac: Bacteroidetes, Myx:Myxococcota, Fir: Firmicutes, Nit: Nitrospirota; (b)纲水平, The: Thermoleophilia, Alp: α-proteobacteria, Vic: Vicinamibacteria, Act: Actinobacteria, Bla: Blastocatellia, Gam: γ-proteobacteria, KD4: KD4-96, Aci: Acidimicrobiia, Gem: Gemmatimonadetes, MB: MB-A2-108 图 6 土壤理化性质与前10个细菌门或纲的相对丰度之间的关系 Fig. 6 Correlation between soil physicochemical factors and relative abundance of top ten phyla or class of bacteria |
分子生态网络分析结果显示, 不同土壤剖面细菌群落的复杂性存在一定差异(图 7).如表 2所示, P0的节点数(222)和边数(1 886)最多, P2层群落的节点数较低, 但边数较高(1 831), 其平均度达到11.302, 网络密度达到0.07, 为最大值, 模块化程度最低为0.368.而深层土(P4)细菌群落的节点数和边数都最低, 平均度也最低(5.129), 群落的复杂性相对较低[图 7(e)].变形菌门、酸杆菌门、绿弯菌门和放线菌门包含了网络中80%以上的节点, 这与门水平细菌群落组成中相对丰度一致.
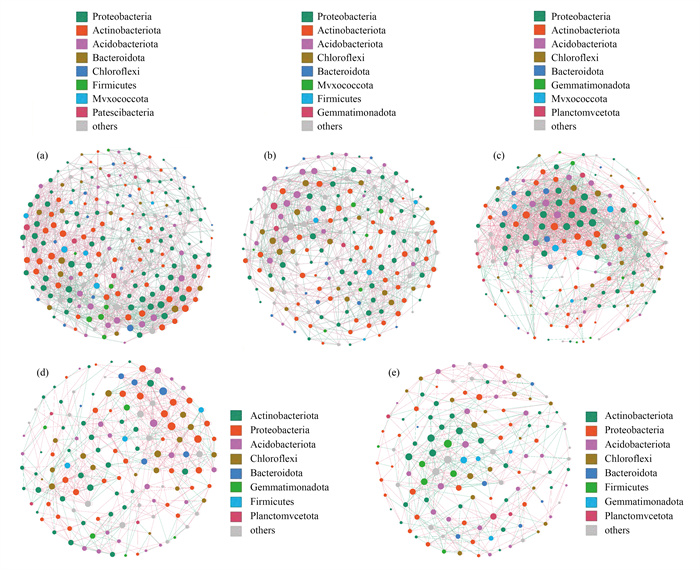
|
(a)腐殖质层(P0), (b)0~10 cm层(P1), (c) 10~20 cm层(P2), (d) 20~40 cm层(P3), (e) 40~80 cm层(P4) 图 7 土壤剖面细菌群落的共现网络 Fig. 7 Co-occurrence networks of bacterial communities along soil profiles |
|
|
表 2 细菌共现性网络拓扑学性质 Table 2 Topology properties of bacterial co-occurrence network |
2.6 沿土壤剖面细菌群落的潜在功能
根据细菌群落16S序列的分类结果, 基于Tax4Fun进行细菌群落功能预测, 并通过与KEGG数据库对比, 共获得6类KEGG一级代谢通路功能基因以及一级代谢通路下的19个二级代谢通路功能基因(图 8).一级代谢功能主要有细胞过程(cellular processes)、环境信息处理(environmental information processing)、遗传信息处理(genetic information processing)、人类疾病(human diseases)、代谢(metabolism)和有机系统(organismal systems)这6大类[图 8(a)].3个主要通路为代谢、遗传信息处理和环境信息处理.基于KEGG数据对土壤剖面细菌群落二级功能层预测结果主要为跨膜运输(membrane transport)、碳水化合物代谢(carbohydrate metabolism)、氨基酸代谢(amino acid metabolism)、信号转导(signal transduction)、能量代谢(energy metabolism)、辅助因子和维生素代谢(metabolism of cofactors and vitamins)、核苷酸代谢(nucleotide metabolism)、异生物素降解和代谢(xenobiotics biodegradation and metabolism)、转化(translation)、复制与修复(replication and repair)、脂类代谢(lipid metabolism)、萜类化合物和聚酮类药物的代谢(metabolism of terpenoids and polyketides)、其他氨基酸代谢(metabolism of other amino acids)、细胞活力(cell motility)、聚糖生物合成与代谢(glycan biosynthesis and metabolism)、细胞生长和死亡(cell growth and death)、蛋白质的分子折叠(folding)、细菌性传染病(infection disease of bacteria)、其他次级代谢物的生物合成(biosynthesis of other secondary metabolites)[图 8(b)].含量较高的功能基因分别是碳水化合物代谢、跨膜运输、氨基酸代谢、信号转导、能量代谢和维生素的代谢等, 对功能基因进行热图分析显示, 膜运输和碳水化合物代谢活性相关基因丰度随着土壤深度增加而降低; 而细胞运动和信号转导相关基因在深层土壤具有较高的丰度(图 9).
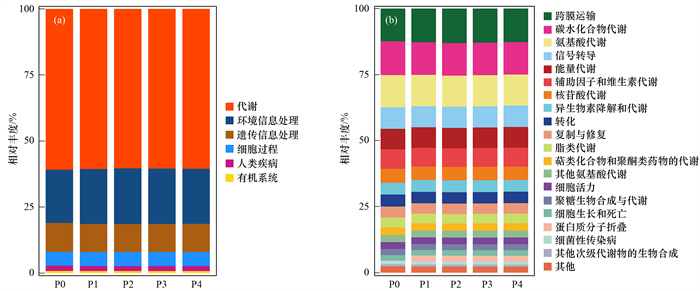
|
(a)一级通路分析, (b)二级通路分析 图 8 土壤剖面细菌一级和二级通路分析 Fig. 8 Analysis of bacterial primary and secondary pathways in soil profile |
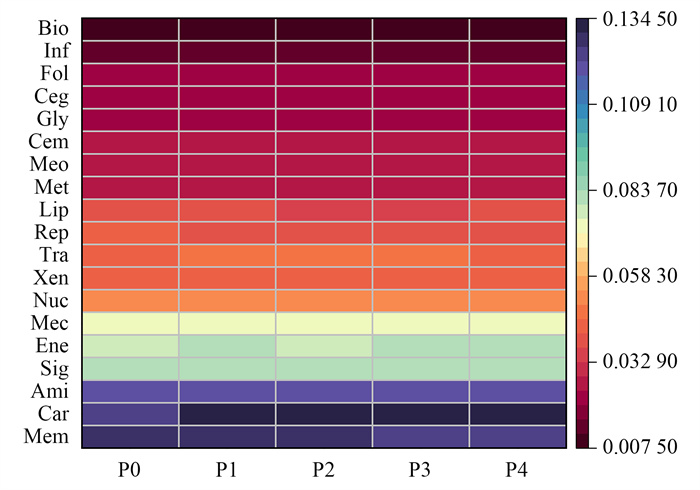
|
Mem: 跨膜运输, Car: 碳水化合物代谢, Ami: 氨基酸代谢, Sig: 信号转导, Ene: 能量代谢, Mec: 辅助因子和维生素的代谢, Nuc: 核苷酸代谢, Xen: 异生物素降解和代谢, Tra: 转化, Rep: 复制与修复, Lip: 脂类代谢, Met: 萜类化合物和聚酮类药物的代谢, Meo: 其他氨基酸的代谢, Cem: 细胞活力, Gly: 聚糖生物合成与代谢, Ceg: 细胞生长和死亡, Fol: 蛋白质的分子折叠, Inf: 细菌性传染病, Bio: 其他次级代谢物的生物合成 图 9 土壤剖面不同深度土层细菌群落功能的KEGG二级代谢通路基因热图分析 Fig. 9 Heat map analysis of KEGG secondary metabolic pathway genes of soil bacterial communities at different depths of soil profile |
本研究结果表明, 华北落叶松林土壤剖面细菌群落丰富度和多样性随深度增加而显著降低, 这与Eilers等[15]在美国科罗拉多州山地的Pinus ponderosa和P. contorta森林土壤细菌和古菌群落的研究结果一致, 这种变化趋势还体现在农田生态系统[12].与深层土壤相比, 表层土壤的气候条件和有机质资源的多样性变化较大, 为微生物创造了多样化栖息地[20].本研究中, 腐殖质层和40~80 cm深度土壤细菌群落结构与其他土壤层的差异显著, 而0~10、10~20和20~40 cm土壤层的细菌群落结构则相似, 其原因可能是由于这3个土壤层养分的主要来源都是植物根系[21, 22], 养分含量差异不显著, 形成了相对均匀的微环境.腐殖质层由于凋落物的输入以及受到外界环境因素影响[23], 而40~80 cm处土壤已经表现出土质矿化, 空气稀缺, 部分细菌无法适应该生境, 导致细菌群落结构在这两个生境发生更多的分化[24].本研究中pH是华北落叶松林土壤剖面细菌群落组成和主要物种相对丰度的主控因素, 这与诸多的研究结果一致, pH值对微生物群落结构和多样性具有重要的影响[25, 26, 27], 其可作为不同深度土壤细菌群落结构的最佳预测因子[28].
在物种组成上, 不同细菌类群的丰度往往随着土壤深度表现出不同的变化趋势[29], 如本研究中的主要优势菌群变形菌门、拟杆菌门、放线菌门、绿弯菌门和酸杆菌门等的变化.拟杆菌和变形菌通常生活在营养丰富的环境中[30].变形菌门的相对丰度随土壤深度的增加呈减少的趋势[26].α-变形菌大多都是革兰氏阴性菌, 其群落生存是“r-策略”, 碳源底物利用效率低, 偏好碳源丰富的环境[31].随着深度增加, 营养物质减少, 变形菌门的含量逐渐降低.大多数放线菌喜欢富氧环境, 因而其相对丰度随土壤深度增加而降低.另一方面, MB-A2-108能够适应低营养含量的土壤, 具有极高的耐受不良环境的能力, 其相对丰度随土层深度增加而增加.绿弯菌门在有机质相对较低的环境中占优势[32], 因此深层土壤为其提供了适宜的环境, 其相对丰度较高[26].酸杆菌门与土壤pH值的变化有关, 在酸性土壤中其丰度通常增加[26, 33].以往研究中, 其相对丰度随着土层深度增加而减少[34].但在本研究中, 酸杆菌随土层深度呈增加趋势, 可能的原因是酸杆菌门为寡营养型细菌, 营养物质对酸杆菌的丰度具有更重要的影响.总之, 生活方式和土壤特征等因素可能决定细菌对不同土壤深度的环境适应性[6, 35].
采用分子生态网络可以分析群落中各物种之间的相互作用关系[36]. 网络中的正相关关系可能反映物种之间存在协同合作或者生态位重叠, 负相关关系可能意味着竞争作用或者生态位分化[37, 38].有研究表明, 在更深的土壤中微生物的相互作用强度增加[39].本研究中, 40~80 cm深的土层中网络负边数比例较高, 说明该层细菌群落物种间存在更大的竞争, 这可能与营养物质的缺乏有关.整个土壤剖面细菌群落物种间的正连接数远高于负连接数, 这与Zhou等[40]对细菌群落共现网络的研究结果一致, 说明细菌群落间的协同作用在华北落叶松林土壤生态系统中发挥主导作用.10~80 cm平均聚类系数较高, 而平均路径距离较短, 说明该网络中物种之间相互作用更紧密, 对干扰的响应更加敏感[41, 42].腐殖质层与0~10 cm处土壤细菌群落的拓扑特征则呈相反趋势, 群落的稳定性对外界环境的变化更为耐受.在腐殖质层、10~20 cm处细菌网络的平均度较高, 可能是这两个土壤层的营养物质丰富, 细菌群落拥有更加高效的养分利用效率[43], 且物种间具有紧密的依存关系.
表层土壤微生物的大部分功能基因相对丰度高于深层土壤[44], 例如膜转运、糖代谢、碳水化合物代谢相关的基因等, 这主要是由于表层土壤营养物质较充足, 细菌群落的物种多样性较高, 有更高的功能多样性[45].细胞运动和信号转导等基因在深层土壤具有较高的丰度, 这可能是细菌在面对环境变化时, 通过改变细胞间的信息交流和物种间的相互作用来应对土壤深度的变化引起的营养物质含量的变化[17].
4 结论(1) 芦芽山华北落叶松林土壤细菌群落的组成和多样性沿土壤剖面存在显著差异.多样性随土层深度呈显著降低趋势, pH是导致细菌群落结构发生变化的最主要的环境因子.
(2) 整体上, 华北落叶松林土壤细菌群落的种间呈现正相关的协同关系, 随着土壤深度的增加细菌群落的种间关系更为紧密, 深层土壤中负相关的竞争关系更为明显.腐殖质层和0~10 cm土壤细菌群落相较于深层土壤群落结构更为稳定.
(3) 受到营养物质含量的影响, 沿着土壤剖面随深度的增加, 微生物代谢活性呈下降趋势.
| [1] | Serna-Chavez H M, Fierer N, Van Bodegom P M. Global drivers and patterns of microbial abundance in soil[J]. Global Ecology and Biogeography, 2013, 22(10): 1162-1172. DOI:10.1111/geb.12070 |
| [2] | Ko D, Yoo G, Yun S T, et al. Bacterial and fungal community composition across the soil depth profiles in a fallow field[J]. Journal of Ecology and Environment, 2017, 41(1). DOI:10.1186/s41610-017-0053-0 |
| [3] | Schnecker J, Wild B, Takriti M, et al. Microbial community composition shapes enzyme patterns in topsoil and subsoil horizons along a latitudinal transect in western Siberia[J]. Soil Biology and Biochemistry, 2015, 83: 106-115. DOI:10.1016/j.soilbio.2015.01.016 |
| [4] | Shigyo N, Umeki K, Hirao T. Seasonal dynamics of soil fungal and bacterial communities in cool-temperate montane forests[J]. Frontiers in Microbiology, 2019, 10. DOI:10.3389/fmicb.2019.01944 |
| [5] | Buckeridge K M, Mason K E, McNamara N P, et al. Environmental and microbial controls on microbial necromass recycling, an important precursor for soil carbon stabilization[J]. Communications Earth & Environment, 2020, 1(1). DOI:10.1038/s43247-020-00031-4 |
| [6] | Hao J J, Chai Y N, Lopes L D, et al. The effects of soil depth on the structure of microbial communities in agricultural soils in Iowa (United States)[J]. Applied and Environmental Microbiology, 2021, 87(4). DOI:10.1128/AEM.02673-20 |
| [7] | Liu M H, Sui X, Hu Y B, et al. Microbial community structure and the relationship with soil carbon and nitrogen in an original Korean pine forest of Changbai Mountain, China[J]. BMC Microbiology, 2019, 19(1). DOI:10.1186/s12866-019-1584-6 |
| [8] | Liu Z F, Fu B J, Zheng X X, et al. Plant biomass, soil water content and soil N: P ratio regulating soil microbial functional diversity in a temperate steppe: a regional scale study[J]. Soil Biology and Biochemistry, 2010, 42(3): 445-450. DOI:10.1016/j.soilbio.2009.11.027 |
| [9] |
刘晓华, 魏天兴. 高通量测序分析黄土高原退耕还林区土壤细菌群落特征[J]. 环境科学, 2021, 42(9): 4489-4499. Liu X H, Wei T X. High-throughput sequencing analysis of soil bacterial community in the grain for green project areas of the Loess Plateau[J]. Environmental Science, 2021, 42(9): 4489-4499. |
| [10] | Tripathi B M, Kim M, Kim Y, et al. Variations in bacterial and archaeal communities along depth profiles of Alaskan soil cores[J]. Scientific Reports, 2018, 8(1). DOI:10.1038/s41598-017-18777-x |
| [11] |
蔡文良, 谢艳云, 唐雯. 海南尖峰岭热带山地雨林土壤有机碳储量和垂直分布特征[J]. 生态环境学报, 2019, 28(8): 1514-1521. Cai W L, Xie Y Y, Tang W. Vertical distribution of soil organic carbon storage of tropical mountain rainforest in Jianfengling, Hainan[J]. Ecology and Environmental Sciences, 2019, 28(8): 1514-1521. |
| [12] | Upton R N, Sielaff A C, Hofmockel K S, et al. Soil depth and grassland origin cooperatively shape microbial community co-occurrence and function[J]. Ecosphere, 2020, 11(1). DOI:10.1002/ecs2.2973 |
| [13] | Gross C D, Harrison R B. The case for digging deeper: soil organic carbon storage, dynamics, and controls in our changing world[J]. Soil Systems, 2019, 3(2). DOI:10.3390/soilsystems3020028 |
| [14] | Rumpel C, Kögel-Knabner I. Deep soil organic matter—a key but poorly understood component of terrestrial C cycle[J]. Plant and Soil, 2011, 338(1-2): 143-158. DOI:10.1007/s11104-010-0391-5 |
| [15] | Eilers K G, Debenport S, Anderson S, et al. Digging deeper to find unique microbial communities: the strong effect of depth on the structure of bacterial and archaeal communities in soil[J]. Soil Biology and Biochemistry, 2012, 50: 58-65. DOI:10.1016/j.soilbio.2012.03.011 |
| [16] | Wu W X, Lu H P, Sastri A, et al. Contrasting the relative importance of species sorting and dispersal limitation in shaping marine bacterial versus protist communities[J]. The ISME Journal, 2018, 12(2): 485-494. DOI:10.1038/ismej.2017.183 |
| [17] | Hobley E, Wilson B, Wilkie A, et al. Drivers of soil organic carbon storage and vertical distribution in eastern Australia[J]. Plant and Soil, 2015, 390(1-2): 111-127. DOI:10.1007/s11104-015-2380-1 |
| [18] |
张子怡, 李卓然, 阮爱东, 等. 新疆荒漠地带裸土剖面微生物群落结构和功能分析[J]. 生物资源, 2022, 44(3): 275-284. Zhang Z Y, Li Z R, Ruan A D, et al. Analysis of microbial community structure and function of bare soil profile in desert area of Xinjiang[J]. Biotic Resources, 2022, 44(3): 275-284. |
| [19] | Xu J S, Gao W, Zhao B Z, et al. Bacterial community composition and assembly along a natural sodicity/salinity gradient in surface and subsurface soils[J]. Applied Soil Ecology, 2021, 157. DOI:10.1016/j.apsoil.2020.103731 |
| [20] |
王雪, 刘晋仙, 柴宝峰, 等. 宁武亚高山湖泊细菌群落的时空格局及驱动机制[J]. 环境科学, 2019, 40(7): 3285-3294. Wang X, Liu J X, Chai B F, et al. Spatio-temporal patterns of microbial communities and their driving mechanisms in subalpine lakes, Ningwu, Shanxi[J]. Environmental Science, 2019, 40(7): 3285-3294. |
| [21] | Peixoto L, Elsgaard L, Rasmussen J, et al. Decreased rhizodeposition, but increased microbial carbon stabilization with soil depth down to 3.6 m[J]. Soil Biology and Biochemistry, 2020, 150. DOI:10.1016/j.soilbio.2020.108008 |
| [22] | Thorup-Kristensen K, Halberg N, Nicolaisen M, et al. Digging deeper for agricultural resources, the value of deep rooting[J]. Trends in Plant Science, 2020, 25(4): 406-417. DOI:10.1016/j.tplants.2019.12.007 |
| [23] |
刘坤和, 薛玉琴, 竹兰萍, 等. 嘉陵江滨岸带不同土地利用类型对土壤细菌群落多样性的影响[J]. 环境科学, 2022, 43(3): 1620-1629. Liu K H, Xue Y Q, Zhu L P, et al. Effect of different land use types on the diversity of soil bacterial community in the coastal zone of Jialing River[J]. Environmental Science, 2022, 43(3): 1620-1629. |
| [24] | Stone M M, Deforest J L, Plante A F. Changes in extracellular enzyme activity and microbial community structure with soil depth at the Luquillo critical zone observatory[J]. Soil Biology and Biochemistry, 2014, 75: 237-247. |
| [25] | Tereshchenko L G, Sur S, Meoni L, et al. Increased Qt variability index is associated with mortality in incident hemodialysis patients[J]. Journal of the American College of Cardiology, 2015, 65(10). DOI:10.1016/S0735-1097(15)60338-2 |
| [26] | Mundra S, Kjønaas O J, Morgado L N, et al. Soil depth matters: shift in composition and inter-kingdom co-occurrence patterns of microorganisms in forest soils[J]. FEMS Microbiology Ecology, 2021, 97(3). DOI:10.1093/femsec/fiab022 |
| [27] | Naylor D, McClure R, Jansson J. Trends in microbial community composition and function by soil depth[J]. Microorganisms, 2022, 10(3). DOI:10.3390/microorganisms10030540 |
| [28] |
贺纪正, 葛源. 土壤微生物生物地理学研究进展[J]. 生态学报, 2008, 28(11): 5571-5582. He J Z, Ge Y. Recent advances in soil microbial biogeography[J]. Acta Ecologica Sinica, 2008, 28(11): 5571-5582. |
| [29] | Baldrian P, Kola Dřík M, Štursová M, et al. Active and total microbial communities in forest soil are largely different and highly stratified during decomposition[J]. The ISME Journal, 2012, 6(2): 248-258. |
| [30] | Goldfarb K C, Karaoz U, Hanson C A, et al. Differential growth responses of soil bacterial taxa to carbon substrates of varying chemical recalcitrance[J]. Frontiers in Microbiology, 2011, 2. DOI:10.3389/fmicb.2011.00094 |
| [31] | Blagodatskaya E V, Blagodatsky S A, Anderson T H, et al. Priming effects in chernozem induced by glucose and N in relation to microbial growth strategies[J]. Applied Soil Ecology, 2007, 37(1-2): 95-105. |
| [32] |
彭雯, 谭玲, 明安刚, 等. 南亚热带典型人工纯林土壤剖面细菌群落组成差异分析[J]. 土壤通报, 2018, 49(6): 1361-1369. Peng W, Tan L, Ming A G, et al. Bacterial community composition in soil profile of typical monoculture plantations in south subtropical China[J]. Chinese Journal of Soil Science, 2018, 49(6): 1361-1369. |
| [33] | Jones R T, Robeson M S, Lauber C L, et al. A comprehensive survey of soil acidobacterial diversity using pyrosequencing and clone library analyses[J]. The ISME Journal, 2009, 3(4): 442-453. |
| [34] | Tveit A, Schwacke R, Svenning M M, et al. Organic carbon transformations in high-Arctic peat soils: key functions and microorganisms[J]. The ISME Journal, 2013, 7(2): 299-311. |
| [35] |
刘晋仙, 李毳, 景炬辉, 等. 中条山十八河铜尾矿库微生物群落组成与环境适应性[J]. 环境科学, 2017, 38(1): 318-326. Liu J X, Li C, Jing J H, et al. Composition and environmental adaptation of microbial community in Shibahe copper tailing in Zhongtiao Mountain in Shanxi[J]. Environmental Science, 2017, 38(1): 318-326. |
| [36] | Faust K, Raes J. Microbial interactions: from networks to models[J]. Nature Reviews Microbiology, 2012, 10(8): 538-550. |
| [37] | Deng Y, Zhang P, Qin Y J, et al. Network succession reveals the importance of competition in response to emulsified vegetable oil amendment for uranium bioremediation[J]. Environmental Microbiology, 2016, 18(1): 205-218. |
| [38] |
张梅婷, 刘晋仙, 苏嘉贺, 等. 苦草叶表附生和水体浮游细菌群落多样性格局及其影响因素[J]. 环境科学, 2023, 44(1): 252-261. Zhang M T, Liu J X, Su J H, et al. Diversity patterns and influencing factors of epibiotic in Vallisneria natans and planktonic bacteria communities[J]. Environmental Science, 2023, 44(1): 252-261. |
| [39] |
黄兰婷, 倪浩为, 李新宇, 等. 典型红壤水稻土剖面细菌和真菌分子生态网络特征研究[J]. 土壤学报, 2021, 58(4): 1018-1027. Huang L T, Ni H W, Li X Y, et al. Molecular ecological network of bacteria and fungi in paddy soil profile of typical red soil[J]. Acta Pedologica Sinica, 2021, 58(4): 1018-1027. |
| [40] | Zhou H, Gao Y, Jia X H, et al. Network analysis reveals the strengthening of microbial interaction in biological soil crust development in the Mu Us Sandy Land, Northwestern China[J]. Soil Biology and Biochemistry, 2020, 144. DOI:10.1016/j.soilbio.2020.107782 |
| [41] | Nguyen B A T, Chen Q L, He J Z, et al. Livestock manure spiked with the antibiotic Tylosin significantly altered soil protist functional groups[J]. Journal of Hazardous Materials, 2022, 427. DOI:10.1016/j.jhazmat.2021.127867 |
| [42] |
李冰, 李玉双, 魏建兵, 等. 不同土地利用方式对土壤细菌分子生态网络的影响[J]. 环境科学, 2020, 41(3): 1456-1465. Li B, Li Y S, Wei J B, et al. Effects of different land use typess on the molecular ecological network of soil bacteria[J]. Environmental Science, 2020, 41(3): 1456-1465. |
| [43] | Wagg C, Schlaeppi K, Banerjee S, et al. Fungal-bacterial diversity and microbiome complexity predict ecosystem functioning[J]. Nature Communications, 2019, 10(1). DOI:10.1038/s41467-019-12798-y |
| [44] | Young J M, Skvortsov T, Kelleher B P, et al. Effect of soil horizon stratigraphy on the microbial ecology of alpine paleosols[J]. Science of the Total Environment, 2019, 657: 1183-1193. |
| [45] |
吴玲玲, 冀瑞卿, 徐洋, 等. 蒙古栎纯林内根系及栖息地土壤细菌群落结构及功能预测[J]. 吉林农业大学学报, 2022. Wu L L, Ji R Q, Xu Y, et al. Community structure and functional prediction of bacteria of the root and the habitat soil in the Quercus mongolica pure forest[J]. Journal of Jilin Agricultural University, 2022. DOI:10.13327/j.jjlau.2022.1785 |
 2023, Vol. 44
2023, Vol. 44


