2. 岳阳市农业科学研究院, 农业农村部岳阳农业环境科学观测实验站, 岳阳 414000
2. Yueyang Agricultural Environment Scientific Experiment Station, Ministry of Agriculture and Rural Affairs, Yueyang Academy of Agricultural Sciences, Yueyang 414000, China
由于含砷矿产的开采、含砷农药滥用和砷超标地下水灌溉等造成国内外稻田砷污染现象突出[1].水稻籽粒相对于其他作物可食部位更易于积累砷, 可通过食物链方式严重威胁人体健康[2].近年来研究发现, 水稻颖壳累积过量的二甲基砷(DMAs)容易引起水稻直穗病[3], 后者是一种生理性病害, 水稻抽穗后不能灌浆结实, 颖壳畸形, 稻穗直立, 严重者不能抽穗, 导致水稻减产, 甚至绝产[4, 5].然而水稻植株本身不能将砷甲基化, 水稻体内DMAs主要来源于对土壤DMAs的吸收作用[6].因此关注稻田土壤中砷的甲基转化过程, 明确环境因子影响砷甲基化效率变化, 对于今后水稻直穗病防控具有重要科学意义.
稻田土壤中砷存在形态主要包括无机砷(iAs)和甲基态砷, 其中无机砷包括还原态砷[As(Ⅲ)]和氧化态砷[As(Ⅴ)], 甲基砷主要包括单甲基砷酸盐(MMAs)、二甲基砷酸盐(DMAs)和三甲基砷(TMAsO)[7].微生物参与土壤中砷的氧化、还原、甲基化和去甲基化过程[8].微生物对砷的甲基化被认为是一个解毒过程, 无机砷通过连续的还原和甲基化过程形成具有挥发性的还原态甲基砷, 进而释放到微生物胞外[9].微生物对砷的甲基化主要由功能基因arsM编码生成的S-腺苷甲硫氨酸(SAM)甲基转移酶调控[10].有研究表明, 稻田土壤中的砷甲基化主要由硫酸盐还原菌(dsrA)驱动, 而产生的DMAs可以被一些营养型产甲烷菌(mcrA)通过代谢甲基基团为CO2的方式发生去甲基反应[11].因此, 稻田土壤中砷的甲基化和去甲基化动态平衡控制着土壤中DMAs的浓度水平, 进而在很大程度上影响着水稻直穗病的发生[11].外源添加有机物料和含硫物质、环境温度等均会显著影响土壤Eh、pH状况和可溶性总有机碳(TOC)浓度, 改变砷甲基化相关微生物区系, 从而影响砷甲基化效率[12~14].外源添加有机物质有助于提高稻田土壤微生物砷甲基化效率, 且不同来源的有机物质提升砷甲基化效率的能力亦不同[12, 15].持续性稻田淹水促进稻田土壤中微生物砷甲基化基因arsM拷贝数和籽粒甲基砷浓度明显增加[16].外源添加硫酸盐和提高温度有利于提高硫酸盐还原菌丰度和活性, 进而促进砷甲基转化[17].稻田落干是水稻生产中常见且必要的农艺措施, 水稻分蘖晚期通过排水落干控制水稻有效分蘖数量, 促进产量提升; 而灌浆晚期通过稻田落干可为后期水稻收获提供便利条件.稻田落干过程伴随着孔隙水TOC、无机砷和砷甲基化相关微生物丰度与多样性等的同步变化, 进而造成稻田土壤砷甲基化效率改变.以往研究更加关注单一环境因子对砷甲基化效率的影响, 而稻田落干过程中砷甲基化效率变化和关键因素影响还鲜见报道.
本研究通过室内模拟稻田落干过程, 分析落干过程中孔隙水砷形态、TOC含量、土壤中砷甲基化微生物(arsM)、硫酸盐还原菌(dsrA)、产甲烷菌(mcrA)丰度和arsM微生物群落变化, 揭示稻田落干过程砷甲基化效率变化规律, 探究环境因子(如Eh、pH、TOC)和砷甲基化相关微生物丰度与群落变化影响砷甲基化效率的可能过程, 相关结果有助于丰富稻田砷甲基化转化的理论认知, 以期为今后水稻直穗病科学防控提供指导.
1 材料与方法 1.1 供试土壤和水稻秸秆本实验土壤取自贵州兴仁县(XR)砷污染稻田土壤(25°24′N, 105°15′ E)和广西南丹县(ND)砷污染稻田土壤(25°08′N, 107°30′ E).供试土壤类型分别为发育自第四纪红黏土的黄壤性水稻土和发育自砂页岩的红壤性水稻土, 均取自0~20 cm耕层.实验前土壤先去除石砾和植物残根, 风干磨碎, 过200目筛后备用.土壤理化性质见表 1, 各指标测定方法见文献[18].水稻秸秆由湖南省岳阳市农业科学研究院提供, 实验前水稻秸秆切碎并研磨成粉末, 并利用γ射线物理灭菌, 避免因秸秆添加带入的可能生物污染.
|
|
表 1 供试土壤基本理化性质 Table 1 Geochemical property of experimental soils |
1.2 土壤培养实验
将供试土壤称重90 g放入50 mL离心管中.每种供试土壤设置对照(CK)和外源添加秸秆处理(RS).对照为无秸秆添加处理.外源添加秸秆处理为每90 g土壤添加2.25 g秸秆.秸秆添加量以碳输入量为土体重量的1%计算获得.秸秆和土壤混合均匀装进50 mL离心管中.各处理设置3个重复.孔隙水采样器(19.21.22F, MacroRhizon, 荷兰)事先垂直埋放于供试土壤中, 以减少后期采样对土壤的干扰.离心管外壁锡纸覆盖, 以保持高于土表 2 cm静水层模拟稻田淹水状况开展淹水培养.培养全过程温度控制在25℃.前期实验表明, 淹水培养21 d后孔隙水砷形态、Eh和pH等关键指标变化不显著, 供试土壤进入相对稳定的淹水状态.本实验中淹水培养21 d后停止水分补充, 以自然落干开始模拟稻田水分落干过程.实验在落干培养的第0、24、36、48和60 h, 利用Pt/Ag-AgCl电极和pH计(Thermo Orion 4 Star, Beverly, 美国)分别测定土表以下3 cm处Eh电位和pH值; 利用孔隙水采样器采集土壤孔隙水样, 于-20℃冰箱保存备用; 采集第0、36和60 h新鲜土壤于-80℃冰箱保存, 用于分子生物学指标分析.落干时间为60 h时, 离心管内表层土壤没有明显干裂, 且下层孔隙水仍可通过采样器收集, 对实验结果影响有限.
|
|
表 2 供试土壤、有无秸秆、时间和其交互作用对于各指标的多因素方差分析 Table 2 MANOVA of soil, rice straw, drying time, and their interactions influencing each response factor |
1.3 化学指标分析
土壤孔隙水样品过0.22 μm滤膜后, 取1 mL待测液添加200 μL H2O2(30%)氧化样品15 min, 使用高效液相电感耦合等离子体质谱仪(HPLC-ICP-MS, Agilent ICP-MS 7900, 美国)测定各砷形态浓度.为提高砷形态分析质量, HPLC-ICP-MS上机测样时, 每间隔10个样品检测一次As(Ⅲ)单标溶液(GBW08666, 中国计量科学研究院), 各批次单标溶液回收率达97%.为了更直观地表示砷甲基化效率, 基于各砷形态结果计算砷甲基化效率, 公式如下:

|
式中, c为该形态砷的浓度; TMAsO、DMAs和MMAs分别结合有3、2和1个甲基基团.此外, 第0、36和60 h的孔隙水样利用TOC仪(ELEMENTAL, 德国)测定TOC浓度.
1.4 DNA提取、qPCR和Illumina MiSeq高通量测序土壤DNA提取采用DNA提取试剂盒[天根生化科技(北京)有限公司]进行.分析细菌16S rRNA[19]、古菌16S rRNA[20]、arsM[21]、dsrA[22]和mcrA[17]绝对定量拷贝数, 目的基因引物和反应条件见相关研究.质粒克隆后, qPCR反应体系为2×Master Mix 10 μL, 10 μmol·L-1的PCR特异引物F和R, 加水至总体积分别为18 μL和2 μL DNA.将质粒标准品按10倍梯度稀释, 每个梯度取2 μL做模板建立标准曲线.将混合好的PCR体系置于Realtime PCR仪上进行PCR反应.扩增反应结束后建立PCR产物熔融曲线, 确定反应特异性.
选择落干过程中砷甲基化效率变化较为显著的供试土壤为对象, 以其功能基因arsM PCR扩增产物引物构建MiSeq文库, 于Illumina MiSeq_PE300平台进行Paired-end测序.对测得的Fastq数据进行质控处理, 最终得到优质的Fasta数据.下机数据(Raw PE)经去除拼接, 进一步去除嵌合体和短序列后得到优质clean_tags, 采用聚类uparse方法, 以97%相似水平对所有序列进行OTU(Operational Taxonomic Units)划分.通过与GenBank non-redundant nucleotide database (nt)数据库使用Blast算法进行比对, 设置1e-5的阈值, 得到每个OTU对应的物种分类信息.
1.5 数据分析采用Excel 2016对所有数据进行基本统计分析, 采用Origin 2019进行数据作图, 处理间方差分析采用LSD-t检验在0.05水平下进行.所有数据均表示为平均值±标准误(SE).采用多因素方差分析的方法, 于SPSS 21软件上分析不同处理和交互作用下各指标变化显著性.采用Canoco 5.0中的冗余分析(RDA)来确定环境因子、功能基因丰度与砷化学形态之间关联.对于高通量测序数据, 使用QIIME(v1.8.0)软件进行α多样指数分析(包括Shannon和Chao1等指数).基于物种注释及相对丰度结果, 使用R(v3.6.0)软件进行物种组成柱状图分析.使用QIIME1(v1.8.0)计算β多样性距离矩阵, 使用R(v3.6.0)软件进行最小二乘法判别分析(PLS-DA分析).使用Phython(v2.7)软件开展比较LDA效应大小(LEfSe)分析, 以区分不同处理分组微生物丰度有显著差异的物种.
2 结果与分析 2.1 稻田土壤落干过程Eh、pH和孔隙水TOC浓度变化伴随落干过程, 土壤Eh、pH(图 1)和孔隙水TOC浓度(图 2)变化显著.随落干时间延长, XR和ND土壤Eh值显著升高, 总体来看土壤Eh状态由完全淹水状态下的-300~-200 mV向落干后的-150~-50 mV变迁[图 1(a)].相同土壤不同处理间存在一定差异, 即落干后期RS处理Eh值高于CK处理.特别是XR土壤, CK处理在落干的第0和24 h时Eh值高于RS处理, 而在第36、48和60 h时则RS处理Eh值明显高于CK; 供试土壤落干过程pH值随着时间变化规律不明显, pH值变化在6.5~7.7范围内[图 1(b)].总体来看, CK处理土壤pH值整体呈现上升趋势, 且变幅较大, 而RS处理特别是ND土壤pH值呈下降趋势, 变幅较小; 供试土壤RS处理TOC浓度均高于CK处理(图 2), 其中XR土壤RS处理在各采样时间下均显著高于CK(P<0.05), 而ND土壤仅在培养时间为60 h时显著高于CK(P<0.05).除ND-CK处理外, 各处理土壤TOC浓度均随落干时间延长呈现不同程度增加, 但仅在ND土壤RS处理中存在显著差异.
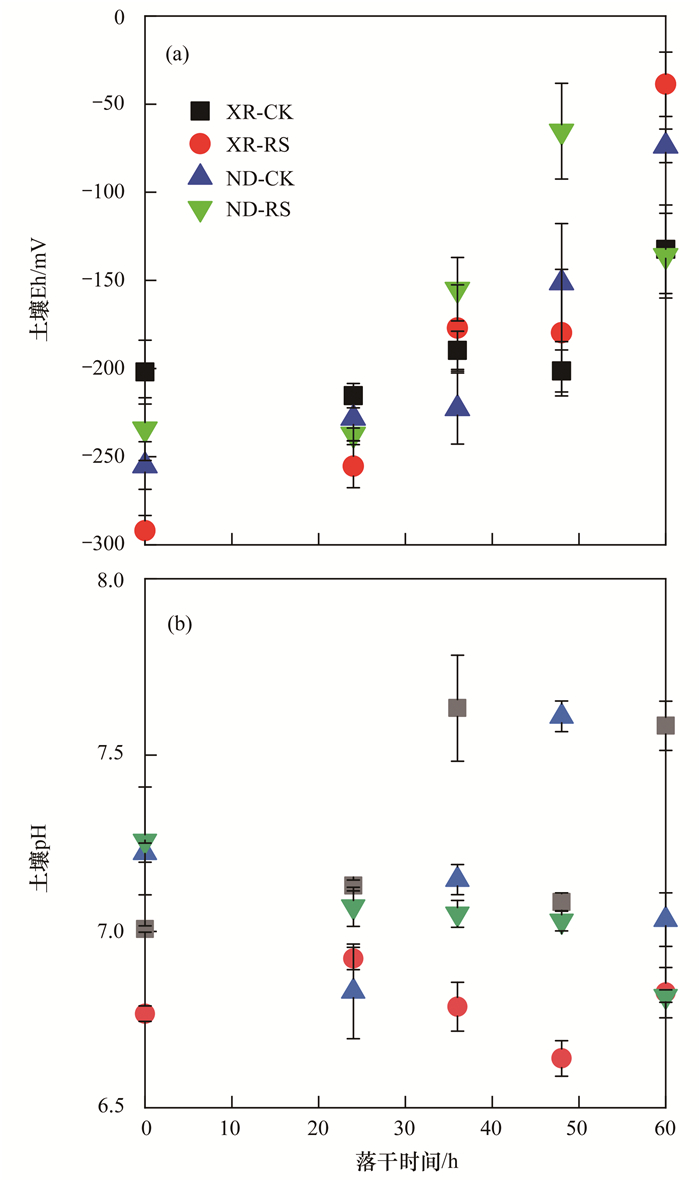
|
图 1 供试土壤落干过程中Eh和pH变化 Fig. 1 Changes in soil Eh and pH with paddy soil drying |

|
图 2 兴仁土壤和南丹土壤落干过程孔隙水TOC含量变化 Fig. 2 Changes in TOC content in porewater during drying of Xingren soil and Nandan soil |
供试土壤各处理的孔隙水iAs和DMAs浓度随落干过程变化更为显著(图 3).随落干时间延长, XR-CK处理iAs浓度整体呈现增加趋势, 而其它形态砷变化不显著; ND-CK、XR-RS和ND-RS处理iAs浓度先增加后降低, 而DMAs浓度随培养时间延长而明显增加.供试土壤CK处理iAs浓度均显著高于其它有机砷[图 3(a)和3(b)], 而RS处理DMAs浓度均显著高于iAs、MMAs和TMAsO[图 3(c)和3(d)].两种供试土壤比较来看, ND土壤无论是否添加秸秆处理均具有更高的DMAs浓度.

|
图 3 随落干时间延长稻田土壤孔隙水各砷形态变化 Fig. 3 Changes in As species in porewater with paddy soil drying |
供试稻田落干过程中砷甲基化效率变化存在差异(图 4).随落干时间延长, XR-CK和XR-RS处理土壤砷甲基化效率没有显著变化; 而ND-CK和ND-RS处理土壤砷甲基化效率则显著增加.当培养时间为60 h时, XR-CK和XR-RS处理砷甲基化效率分别提高9%和0.6%, 而ND-CK处理土壤砷甲基化效率显著高于培养初期(0 h, P<0.05), 提高约61.8%; 当培养时间大于36 h时, ND-RS处理土壤砷甲基化效率显著高于培养初期(0 h, P<0.05), 培养时间为60 h时, 提高约23.2%.处理间比较来看, RS处理的砷甲基化效率均显著高于CK处理(P<0.05).两种供试土壤比较来看, ND-CK处理的砷甲基化效率显著高于XR-CK, ND土壤相对具有更高的砷甲基化效率.

|
不同小写字母表示同一处理不同时间点之间差异显著, 不同大写字母表示同一时间点不同处理之间差异显著, P<0.05(LSD-t test) 图 4 随落干时间延长稻田土壤孔隙水砷甲基化效率变化 Fig. 4 Changes in As methylation efficiency in porewater with paddy soil drying |
各砷甲基化相关功能基因拷贝数随落干过程发生显著变化(图 5).随落干时间延长, 除XR-CK处理外, 细菌16S rRNA和产甲烷菌mcrA拷贝数均表现出不同程度下降.特别是XR-RS和ND-RS处理mcrA拷贝数, 当落干时间为60 h时显著低于培养初期(0 h, P<0.05).砷甲基化基因arsM和硫酸盐还原菌dsrA拷贝数则随落干时间延长表现出一定的增加趋势, 而古细菌拷贝数保持相对稳定.供试土壤的CK和RS处理比较来看, 添加秸秆处理后各基因拷贝数均明显增加, 特别是ND土壤的细菌16S rRNA、古细菌和dsrA拷贝数增加显著(P<0.05).两种供试土壤比较来看, ND土壤具有更高的细菌16S rRNA、古细菌、arsM和mcrA拷贝数, 而XR土壤含有更高的dsrA拷贝数.

|
不同小写字母表示相同处理不同落干时间下差异显著, 不同大写字母表示相同时间不同处理间差异显著 图 5 土壤全细菌、古菌和砷转化相关功能基因拷贝数随落干时间变化 Fig. 5 Changes in copy numbers of bacteria, archaea, and other genes associated with As methylation with paddy soil drying |
不同土壤、有无秸秆、落干时间和交互作用显著影响各因变量变化(表 2).不同土壤处理对于孔隙水TOC浓度、各砷形态浓度、砷甲基化效率和细菌、古菌、功能基因(arsM、mcrA、dsrA)拷贝数均具有显著影响(P<0.05).有无秸秆添加对土壤pH、TOC浓度、细菌、古菌、功能基因(arsM、mcrA、dsrA)、各砷形态浓度和砷甲基化效率有显著影响(P<0.05).时间处理对于环境因子(Eh)、功能基因(mcrA)、砷形态(DMAs)和砷甲基化效率有显著影响(P<0.05).土壤和有无秸秆交互作用对于环境因子(pH)、细菌、古菌、功能基因(arsM、mcrA、dsrA)、砷化学形态(iAs、MMAs、DMAs、TMAsO)和砷甲基化效率存在显著影响(P<0.05).土壤和时间交互作用对于环境因子(pH)、功能基因(mcrA)、砷化学形态(MMAs、DMAs)和砷甲基化效率有显著影响(P<0.05).有无秸秆与落干时间交互作用对于环境因子(pH)、功能基因(mcrA)和砷化学形态(MMAs、DMAs)有显著影响(P<0.05).土壤、有无秸秆和落干时间三者交互作用对于环境因子(Eh)、功能基因(mcrA)和砷化学形态(MMAs、DMAs)有显著影响(P<0.05).
利用RDA分析探讨环境因子(TOC、Eh、pH)、功能基因丰度和砷化学形态之间的关系(图 6).RDA 1解释度为65.64%, RDA 2解释度为16.51%, 总解释度为82.15%, 由此可见, RDA轴Ⅰ和轴Ⅱ能够较好地反映环境因子、功能基因丰度与As化学形态之间关联.功能基因dsrA(F=55.8, P=0.002)、arsM(F=42.4, P=0.002)、古菌(F=26.6, P=0.002)、细菌(F=13.2, P=0.002)、mcrA(F=7.9, P=0.004)、环境因子TOC(F=10.9, P=0.002)和pH(F=6.8, P=0.004)对砷化学形态影响显著(P<0.05).功能基因dsrA、arsM、mcrA和古菌、细菌分别解释不同化学形态As的62.1%、55.5%、18.9%和43.9%, 28.0%; 环境因子TOC和pH分别解释不同化学形态As的24.2%和16.7%.总体来看, 环境TOC与Eh变化和砷转化相关功能基因与甲基态砷呈现正相关联, 与无机砷iAs呈负相关联.

|
红色箭头表示环境因子和功能基因丰度, 蓝色箭头表示砷化学形态 图 6 利用RDA分析环境因子、功能基因丰度和砷化学形态之间关联 Fig. 6 RDA analysis of the relationship among environmental factors, functional genes, and As species |
由于ND土壤孔隙水砷甲基化效率随落干时间延长而显著增加, XR土壤砷甲基化效率变化不显著, 故进一步以ND土壤落干初期(0 h)和落干60 h样本为研究对象, 基于功能基因测序分析arsM功能微生物群落变化.相同处理不同落干时间比较, ND土壤arsM功能微生物α多样性指标如Chao1、Observed_species、PD_whole_tree、Shannon、clean_reads和OTUs无显著差异(P>0.05); 相同落干时间不同处理比较来看, 上述指标整体上亦未发现显著差异(P>0.05)(数据未列).利用基于OTU的偏最小二乘法判别分析(PLS-DA)ND土壤arsM功能微生物β多样性差异, 结果表明ND土壤添加秸秆处理与对照存在显著差异, 且对照处理arsM功能微生物多样性随时间变化存在显著差异, 而RS处理则变化较小[图 7(a)].进一步物种注释分析发现, ND土壤处理间arsM功能微生物门水平组成均主要以undentified、Actinobacteria和Proteobacteria为主, 且ND-CK处理相比ND-RS含有相对更高丰度的Gemmatimonadetes[图 7(b)].利用线性判别分析比较各处理间和落干时间下arsM功能微生物差异物种影响力大小, 发现添加秸秆和落干均能显著改变砷甲基化相关微生物群落[图 7(c)].以纲水平arsM功能微生物差异物种变化为例, ND-CK土壤落干初期以α-Proteobacteria为主要差异物种, 而落干60 h后主要差异物种为Rubrobacteria和Fimbriimonadia; 添加秸秆后ND-RS土壤落干初期以Actinobacteria为主要差异物种, 而落干60 h后主要差异物种为Clostridia和Acidobacterii.

|
(a) 不同处理和落干时间下arsM微生物群落变化主成分分析, (b)不同处理和落干时间下arsM微生物门水平群落组成, (c)不同处理和落干时间下显著差异arsM微生物LEfSe分析; (a)和(b)中图例分别表示南丹土壤CK处理落干的第0、36和60 h的样本, RS秸秆处理落干的第0、36和60 h样本; (c)中图例表示南丹土壤CK处理和RS秸秆处理第0 h和60 h样本; ND表示采集自广西南丹的砷污染水稻土, CK表示未添加秸秆的对照, RS表示添加秸秆处理 图 7 基于OTU的ND土壤不同处理arsM功能微生物β多样性和LEfSe分析 Fig. 7 The β diversity of arsM-carrying bacteria of the paddy soil under different treatments and the comparative LEfSe analysis |
水稻颖壳累积过量的DMAs是水稻直穗病发生的直接因素[3], 稻田土壤中砷甲基转化是诱导水稻直穗病发生的关键.水稻生长的分蘖晚期和灌浆晚期, 为了减少无效分蘖, 使籽粒灌浆更充分, 稻田排水晒田是常见的农艺手段[23].本研究采用的两种供试土壤田间淹水原位分析(表土下15 cm处)其水稻根际Eh为-250 mV, 经排水落干至表土出现裂痕时其Eh为-40 mV.本研究通过室内模拟稻田土壤落干获得的Eh变幅为-300~-50 mV, 表明该模拟条件能相对较好地反映稻田真实条件下根际的Eh变化.以往研究认为, 淹水环境更利于稻田砷甲基化转化[24], 而本研究中随落干过程延长, 供试土壤Eh变化并不会降低孔隙水中砷的甲基化效率, 对于ND土壤甚至存在显著提高, ND-CK和ND-RS处理分别提高61.8%和23.2%.该结果表明, 尽管稻田落干造成土壤Eh状况显著提升, 但其根际Eh可能仍处于有利于砷甲基化反应的还原性环境.研究还发现两种供试土壤间砷甲基化效率存在差异, 秸秆添加显著提高土壤砷甲基化效率, 而落干过程砷甲基化功能微生物arsM和硫酸盐还原菌dsrA丰度增加, 产甲烷菌mcrA丰度下降, 且砷甲基化功能微生物群落更替可能是造成砷甲基化效率变化的重要生物学原因.
供试土壤无论有无秸秆添加其落干过程中均未发现孔隙水砷甲基化效率下降, XR土壤落干过程中孔隙水砷甲基化效率有一定提升, 但差异不显著, 而ND土壤则显著提高(P<0.05).稻田土壤落干过程水分逐渐减少, Eh值逐渐升高, 由-300~-200 mV范围向-150~-50 mV范围变化.硫酸盐还原菌生长的最适Eh值为-100 mV[25], 产甲烷菌最适Eh值为-330 mV[26].因此, 本研究模拟的落干过程可能存在由产甲烷菌的最适生境向硫酸盐还原菌最适生境的转变, 进而影响砷甲基化相关微生物群落.本研究中砷甲基化功能微生物arsM和硫酸盐还原菌dsrA丰度伴随落干过程呈现不同程度提高, 而产甲烷菌基因mcrA丰度显著下降.硫酸盐还原菌和产甲烷菌被认为分别是参与砷甲基化和去甲基化的关键微生物[11].落干过程中砷甲基化相关微生物丰度增加, 而去甲基化相关微生物丰度减少, 可能是砷甲基化效率提高的重要生物学原因.随落干时间延长ND-RS处理孔隙水TOC含量显著增加, 可能为土壤中砷甲基化微生物生长提供了丰富碳源, 进而促进该处理孔隙水砷甲基化效率提升.此外, 无机砷浓度变化亦可能影响落干过程砷甲基化效率变化.本研究中, XR-RS、ND-CK和ND-RS处理孔隙水iAs浓度随土壤落干呈现先增加后下降的变化趋势.iAs作为砷甲基化的底物形态其浓度增加有助于促进砷甲基化进程[27].另外, 本研究中落干过程可能造成的浓缩效应对孔隙水DMAs浓度提高的贡献基本可以忽略, 因为XR-CK、ND-CK和ND-RS处理孔隙水TMAsO浓度并未发生明显变化.
不论ND土壤还是XR土壤, 添加秸秆后孔隙水砷甲基化效率明显高于CK处理.秸秆添加显著促进孔隙水中TOC浓度提升, 进而激发了土壤中砷甲基化微生物的数量和活性[28].秸秆添加后砷甲基化基因arsM和硫酸盐还原菌dsrA基因拷贝数均高于CK处理, 很好地解释了秸秆添加提高砷甲基化效率背后的生物学原因.秸秆还田使得土壤孔隙水中TOC浓度提升, 可以直接或间接地促进iAs从土壤胶体表面释放, 特别是在较高的TOC浓度下[15, 29].DOM浓度的提升还会使土壤Eh氧化还原电位下降, 由于H+消耗, pH值上升, 促进土壤iAs向溶液释放[30], 进而增加砷甲基化效率.然而本研究中, 添加秸秆处理孔隙水iAs含量相比对照则显著下降, 这可能是因为添加秸秆处理中形成了DOM-Fe-As的三元络合物[31]以及iAs向甲基态砷特别是DMAs转化导致.此外, 硫素添加可以通过促进硫酸盐还原菌丰度增加的方式促进砷甲基转化[17].秸秆硫素含量相对较低, 其腐解后产生的DOM中硫含量约6.1 mg·L-1[15].因此本研究因秸秆添加带入硫素对砷甲基化效率的影响较为有限.有研究发现, DOC丰度和其碳分子特征可能差异性影响稻田土壤砷甲基化进程, 活性碳官能团丰度相对更高的DOC有利于激发砷甲基化功能微生物活性, 进而提高土壤砷甲基化转化[15].本研究中, 不同供试土壤秸秆腐解造成的DOC碳分子差异是否与稻田土壤砷甲基化进程相关联还需进一步研究.此外, 两种供试土壤比较来看, 其甲基化效率亦存在明显差异.落干过程ND-CK处理砷甲基化效率均高于XR-CK处理, 可能原因包括:①相比XR土壤, ND土壤中可能有相对更高的砷甲基化功能微生物丰度.本研究中ND土壤arsM基因拷贝数显著高于XR土壤; ②ND土壤本底iAs浓度高于XR土壤, 从而为砷甲基化提供更多底物.本研究中, ND土壤不论有无秸秆添加其iAs浓度均高于XR土壤; ③ND土壤孔隙水含有更高的TOC浓度.ND土壤孔隙水TOC浓度约为XR土壤的2~4倍, 高浓度TOC有助于激发砷甲基化功能微生物活性, 进而提高砷甲基化效率[28].两种土壤CK处理中孔隙水总砷(各形态加和法)含量比较来看, XR土壤孔隙水总砷(27.3~45.7 μg·L-1)明显低于ND土壤(57.7~82.8 μg·L-1), 这可能与ND(0.54 mg·kg-1)相比XR(0.38 mg·kg-1)土壤较高的有效态砷含量有关.此外, ND土壤相对更高的土壤pH及有机质含量, 亦有利于增加土壤中砷的活性.
本研究模拟的稻田落干过程可以显著改变砷甲基化功能微生物多样性, 进而影响砷甲基化效率.以ND土壤为研究对象, 发现随落干过程延长, arsM功能微生物α多样性指标并无显著差异, 而基于OTU的β多样性和线性判别分析(LDA)存在显著差异.该结果表明, 伴随稻田落干过程arsM功能微生物群落结构改变显著影响砷甲基化效率.基于LEfSe分析结果发现, ND-CK土壤落干初期以α-Proteobacteria为主要差异的arsM基因携带物种, 而落干60 h后主要差异的arsM基因携带物种为Rubrobacteria和Fimbriimonadia.携带arsM基因的α-Proteobacteria物种被报道广泛发现于中国东部砷累积严重的沉积物和含砷废水中, 并参与环境中砷的甲基化过程[32, 33].添加秸秆后ND-RS土壤落干初期以Actinobacteria为主要差异arsM基因携带物种, 而落干60 h后主要差异物种为Clostridia和Acidobacteriia.具有抗砷能力的Actinobacteria被发现广泛存在于砷污染土壤, 同时参与土壤中砷的甲基转化[34]; 分离自砷污染稻田的隶属Clostridia的多株厌氧硫酸盐还原菌被发现具备将土壤中As(Ⅲ)甲基化为MMAs(Ⅴ)和DMAs(Ⅴ)的能力[35].稻田落干过程中伴随的土壤Eh、TOC等环境因子变化可能影响部分关键arsM功能微生物群落的更替, 该过程中arsM功能微生物对环境Eh-pH变化和TOC丰度与分子特征的响应机制仍需进一步关注.
4 结论本研究通过模拟稻田落干过程发现, 当土壤Eh由-300~-200 mV范围向-150~-50 mV范围变化时, 并未降低稻田土壤砷甲基化效率, 相反ND土壤砷甲基化效率显著提高, 而XR土壤孔隙水砷甲基化效率变化不显著.伴随落干稻田Eh增加, 砷甲基化微生物和硫酸还原菌拷贝数增加, 而砷去甲基化相关微生物产甲烷菌拷贝数显著下降, 砷甲基化功能微生物群落结构更替, 可能是ND土壤孔隙水砷甲基化效率提高的重要生物学原因.此外, 添加秸秆显著增加孔隙水TOC含量, 提高arsM携带微生物和硫酸盐还原菌dsrA基因拷贝数, 进而提高孔隙水甲基化砷特别是DMAs含量和砷甲基化效率.研究结果有助于提升稻田落干过程中砷甲基化变化的认知, 为今后水稻直穗病防控提供指导.
| [1] | Yang Y P, Zhang H M, Yuan H Y, et al. Microbe mediated arsenic release from iron minerals and arsenic methylation in rhizosphere controls arsenic fate in soil-rice system after straw incorporation[J]. Environmental Pollution, 2018, 236: 598-608. DOI:10.1016/j.envpol.2018.01.099 |
| [2] | Zeng X B, Bai L Y, Gao X, et al. Agricultural planning by selecting food crops with low arsenic accumulation to efficiently reduce arsenic exposure to human health in an arsenic-polluted mining region[J]. Journal of Cleaner Production, 2021, 308. DOI:10.1016/j.jclepro.2021.127403 |
| [3] | Tang Z, Wang Y J, Gao A X, et al. Dimethylarsinic acid is the causal agent inducing rice straighthead disease[J]. Journal of Experimental Botany, 2020, 71(18): 5631-5644. DOI:10.1093/jxb/eraa253 |
| [4] | Yan W G, Dilday R H, Tai T H, et al. Differential response of rice germplasm to straighthead induced by arsenic[J]. Crop Science, 2005, 45(4): 1223-1228. DOI:10.2135/cropsci2004.0348 |
| [5] | Rahman M A, Hasegawa H, Rahman M M, et al. Straighthead disease of rice (Oryza sativa L.) induced by arsenic toxicity[J]. Environmental and Experimental Botany, 2008, 62(1): 54-59. DOI:10.1016/j.envexpbot.2007.07.016 |
| [6] | Huang J H, Hu K N, Decker B. Organic arsenic in the soil environment: speciation, occurrence, transformation, and adsorption behavior[J]. Water, Air, & Soil Pollution, 2011, 219(1-4): 401-415. |
| [7] | Akter K F, Owens G, Davey D E, et al. Arsenic speciation and toxicity in biological systems[J]. Reviews of Environmental Contamination and Toxicology, 2005, 184: 97-149. |
| [8] | Bhattacharya P, Welch A H, Stollenwerk K G, et al. Arsenic in the environment: biology and chemistry[J]. Science of the Total Environment, 2007, 379(2-3): 109-120. DOI:10.1016/j.scitotenv.2007.02.037 |
| [9] | Zhu Y G, Xue X M, Kappler A, et al. Linking genes to microbial biogeochemical cycling: lessons from arsenic[J]. Environmental Science & Technology, 2017, 51(13): 7326-7339. |
| [10] | Zhao F J, Harris E, Yan J, et al. Arsenic methylation in soils and its relationship with microbial arsM abundance and diversity, and As speciation in rice[J]. Environmental Science & Technology, 2013, 47(13): 7147-7154. |
| [11] | Chen C, Li L Y, Huang K, et al. Sulfate-reducing bacteria and methanogens are involved in arsenic methylation and demethylation in paddy soils[J]. The ISME Journal, 2019, 13(10): 2523-2535. DOI:10.1038/s41396-019-0451-7 |
| [12] | Huang H, Jia Y, Sun G X, et al. Arsenic speciation and volatilization from flooded paddy soils amended with different organic matters[J]. Environmental Science & Technology, 2012, 46(4): 2163-2168. |
| [13] | Jia Y, Sun G X, Huang H, et al. Biogas slurry application elevated arsenic accumulation in rice plant through increased arsenic release and methylation in paddy soil[J]. Plant and Soil, 2013, 365(1): 387-396. |
| [14] | Mohapatra D, Mishra D, Chaudhury G R, et al. Removal of arsenic from arsenic rich sludge by volatilization using anaerobic microorganisms treated with cow dung[J]. Soil and Sediment Contamination: An International Journal, 2008, 17(3): 301-311. DOI:10.1080/15320380802007069 |
| [15] | Yan M M, Zeng X B, Wang J, et al. Dissolved organic matter differentially influences arsenic methylation and volatilization in paddy soils[J]. Journal of Hazardous Materials, 2020, 388. DOI:10.1016/j.jhazmat.2019.121795 |
| [16] | Wang M, Tang Z, Chen X P, et al. Water management impacts the soil microbial communities and total arsenic and methylated arsenicals in rice grains[J]. Environmental Pollution, 2019, 247: 736-744. DOI:10.1016/j.envpol.2019.01.043 |
| [17] | Chen C, Yang B Y, Shen Y, et al. Sulfate addition and rising temperature promote arsenic methylation and the formation of methylated thioarsenates in paddy soils[J]. Soil Biology and Biochemistry, 2021, 154. DOI:10.1016/j.soilbio.2021.108129 |
| [18] | 鲍士旦. 土壤农化分析[M]. (第三版). 北京: 中国农业出版社, 2000. |
| [19] | Reid M C, Maillard J, Bagnoud A, et al. Arsenic methylation dynamics in a rice paddy soil anaerobic enrichment culture[J]. Environmental Science & Technology, 2017, 51(18): 10546-10554. |
| [20] | Yin F B, Dong H M, Zhang W Q, et al. Antibiotic degradation and microbial community structures during acidification and methanogenesis of swine manure containing chlortetracycline or oxytetracycline[J]. Bioresource Technology, 2018, 250: 247-255. DOI:10.1016/j.biortech.2017.11.015 |
| [21] | Jia Y, Huang H, Zhong M, et al. Microbial arsenic methylation in soil and rice rhizosphere[J]. Environmental Science & Technology, 2013, 47(7): 3141-3148. |
| [22] | Schippers A, Neretin L N. Quantification of microbial communities in near-surface and deeply buried marine sediments on the Peru continental margin using real-time PCR[J]. Environmental Microbiology, 2006, 8(7): 1251-1260. DOI:10.1111/j.1462-2920.2006.01019.x |
| [23] |
杨军, 黄淑娥, 张金恩, 等. 晒田土壤含水量对水稻有效分蘖的影响[J]. 中国农学通报, 2016, 32(24): 36-42. Yang J, Huang S E, Zhang J E, et al. Effects of soil water content after field drying on rice effective tillers[J]. Chinese Agricultural Science Bulletin, 2016, 32(24): 36-42. DOI:10.11924/j.issn.1000-6850.casb16050037 |
| [24] | Zhao F J, Zhu Y G, Meharg A A. Methylated arsenic species in rice: geographical variation, origin, and uptake mechanisms[J]. Environmental Science & Technology, 2013, 47(9): 3957-3966. |
| [25] | Kushkevych I. Isolation and purification of sulfate-reducing bacteria[EB/OL]. https://cdn.intechopen.com/pdfs/69731.pdf, 2019-10-24. |
| [26] |
蔡祖聪, 沈光裕, 颜晓元, 等. 土壤质地、温度和Eh对稻田甲烷排放的影响[J]. 土壤学报, 1998, 35(2): 145-154. Cai Z C, Shen G Y, Yan X Y, et al. Effects of soil texture, soil temperature and Eh on methane emissions from rice paddy fields[J]. Acta Pedologica Sinica, 1998, 35(2): 145-154. |
| [27] | Ascar L, Ahumada I, Richter P. Influence of redox potential (Eh) on the availability of arsenic species in soils and soils amended with biosolid[J]. Chemosphere, 2008, 72(10): 1548-1552. DOI:10.1016/j.chemosphere.2008.04.056 |
| [28] | Wang Y H, Li P, Jiang Z, et al. Diversity and abundance of arsenic methylating microorganisms in high arsenic groundwater from Hetao plain of inner Mongolia, China[J]. Ecotoxicology, 2018, 27(8): 1047-1057. DOI:10.1007/s10646-018-1958-9 |
| [29] | Avneri-Katz S, Young R B, McKenna A M, et al. Adsorptive fractionation of dissolved organic matter (DOM) by mineral soil: Macroscale approach and molecular insight[J]. Organic Geochemistry, 2017, 103: 113-124. DOI:10.1016/j.orggeochem.2016.11.004 |
| [30] | Bennett W W, Teasdale P R, Panther J G, et al. Investigating arsenic speciation and mobilization in sediments with DGT and DET: a mesocosm evaluation of oxic-anoxic transitions[J]. Environmental Science & Technology, 2012, 46(7): 3981-3989. |
| [31] | Borggaard O K, Holm P E, Strobel B W. Potential of dissolved organic matter (DOM) to extract As, Cd, Co, Cr, Cu, Ni, Pb and Zn from polluted soils: a review[J]. Geoderma, 2019, 343: 235-246. |
| [32] | Guo T, Li L G, Zhai W W, et al. Distribution of arsenic and its biotransformation genes in sediments from the East China Sea[J]. Environmental Pollution, 2019, 253: 949-958. DOI:10.1016/j.envpol.2019.07.091 |
| [33] | Zhai W W, Qin T Y, Li L G, et al. Abundance and diversity of microbial arsenic biotransformation genes in the sludge of full-scale anaerobic digesters from a municipal wastewater treatment plant[J]. Environment International, 2020, 138. DOI:10.1016/j.envint.2020.105535 |
| [34] | Retamal-Morales G, Mehnert M, Schwabe R, et al. Detection of arsenic-binding siderophores in arsenic-tolerating Actinobacteria by a modified CAS assay[J]. Ecotoxicology and Environmental Safety, 2018, 157: 176-181. DOI:10.1016/j.ecoenv.2018.03.087 |
| [35] | Wang P P, Bao P, Sun G X. Identification and catalytic residues of the arsenite methyltransferase from a sulfate-reducing bacterium, Clostridium sp. BXM[J]. FEMS Microbiology Letters, 2015, 362(1): 1-8. |
 2022, Vol. 43
2022, Vol. 43


